Method Article
Ökaryotik mRNA-Protein kompleksleri kurtarmak için bir Tandem oligonükleotid tabanlı RNA izolasyon yordamı
Bu Makalede
Özet
Endogenously kurulan mRNA-protein kompleksleri kurtarılması için bir tandem RNA izolasyon yordam (seyahat) açıklanmıştır. Özellikle, çapraz vivo içindeRNA-protein kompleksleri vardır, poliadenile RNA'ların özleri ile oligo(dT) boncuk etkilenmezsiniz ve belirli mRNA'ların değiştirilmiş RNA antianlamlı oligonucleotides ile yakalanır. MRNA'ların için bağlı proteinler immunoblot analizi ile tespit edilir.
Özet
RNA bağlanıcı proteinler (RBPs) çoğu Gen ifadesinin kontrolünde önemli rol oynarlar. Bu nedenle, mRNA-protein kompleksleri biyokimyasal karakterizasyonu etkileşen proteinler veya kodlamayan RNA'ların değişkenden mRNA yönetmelik anlamak için gerekli. Burada, biz endogenously kurulan mRNA-protein kompleksleri hücresel özleri gelen arıtma sağlayan bir tandem RNA izolasyon (seyahat) açıklayınız. İki adım protokolü bir mRNA sonra ile izole ilgi ile 3'-biotinylated 2'-O-metillenmiş antianlamlı RNA oligonucleotides, mRNA'ların antianlamlı oligo(dT) boncuk ve sonraki yakalama poliadenile yalıtım içerir streptavidin boncuk. GEZİ vivo içinde çapraz mRNA-Ribonükleoprotein (mRNP) kompleksleri Maya, nematodlar ve daha fazla RNA ve protein analiz insan hücreleri kurtarmak için kullanıldı. Böylece, gezi adapte edilebilir çok yönlü bir yaklaşım poliadenile RNA'ların hücre içi ya da çevresel cues empoze mRNPs dinamik yeniden düzenleme çalışmaya organizmalar arasında her türlü olacaktır.
Giriş
Çoğu gen düzenlemesi tahrik RNA bağlanıcı proteinler (RBPs), kodlamayan RNA'ların (ncRNA'lar) ve mRNA'ların arasındaki etkileşimleri, hangi doğrudan işleme, yerelleştirme, çeviri ve her transkript hücreleri1 içinde çürüme , 2. RBPs ve ncRNA belirli mRNA'ların ne Ribonükleoprotein kompleksleri/tanecik olarak (RNPs), bilinen kurulması ile etkileşim tanımlaması bu nedenle mRNA'ların ve gen ifadesi denetim kaderi anlama anahtarıdır. Tamamlayıcı yaklaşımlar RNP kompleksleri3,4biyokimyasal karakterizasyonu için üstlenilen: "protein-merkezli" yaklaşım belirli RBPs arınma dayalı iken, "RNA-merkezli" yaklaşım içerir yalıtım altgrupları veya bireysel RNA'ların ve etkileşen protein veya RNA'ların daha sonraki analiz. Son zamanlarda, RNA-merkezli bir yaklaşım poliadenile ile etkileşim RBP repertuar tanımlanması için (poly(A)) RNA'ların5 giderek popüler olmuştur protein-RNA dengelemek için bir ultraviyole (UV) ışık crosslinking adım dahil ederek etkileşimleri önceki RBPs Katalog insan hücreleri6,7,8,9,10,11' deki önemli ölçüde genişletilmiş mRNA izolasyonu, Caenorhabditis elegans 12, saccharomyces cerevisiae12,13,14ve diğer organizmalar15,16,17,18, 19,20. Ancak, eşleme proteinler ve/veya belirli transkript monte ncRNA'lar hala büyük bir mücadeledir. Bu amaçla, şu anda iki ana yaklaşım kullanılır: bir yandan, RNA aptamer Etiketler benzeşme arıtma etkinleştirmek için faiz RNA erimiş. Böylece,21,22,23,24Tobramisin ve streptomisin dahil olmak üzere aminoglikosite antibiyotik veya kat protein gibi proteinler yüksek benzeşimli RNA aptamers bağlama R17/MS2 bakteriyofaj veya bakteriyel streptavidin S125,26,27,28,29. Her ne kadar bu yaklaşım nispeten sağlam ve çok yönlü olduğu gösterilmiştir, tasarım RNA soruşturma altında klonlama gerektirir ve böylece doğal mRNA'ların yakalamak için kullanılamaz. Öte yandan, antianlamlı oligonucleotides (ASOs) erken kurtarma için kullanıldı ve karakterizasyonu yüksek oranda yerli RNPs30,31 ve viral RNA'ların32dile getirdi. Daha yakın zamanlarda, ASOs uzun olmayan kodlama RNA'ların33,34,35 yakalamak için uygulanan ve vitro mRNPs36kurdu. Tüm RNA merkezli yaklaşımları ile büyük bir sınırlayıcı faktör düşük ifade mRNA'ların kurtarma daha sorunlu hale RNA ilgi, kopya sayısıdır. Bu sınırlama tepki geliștirme tarafından üstesinden gelebilir; Ancak, bu potansiyel olarak arka plan artmasına neden olabilir.
Burada, bizim son zamanlarda geliştirilen ASO tabanlı tandem RNA izolasyon yordamı (seyahat) yüksek seçicilik37 (Şekil 1). ile yerli mRNA-protein kompleksleri yalıtmak için tarif Protokol iki sıralı ASO tabanlı arıtma adımdan oluşur, yani özel olarak tasarlanmış kısa biotinylated 2'-metoksi kullanarak belirli mRNA yakalayarak takip boncuk poli(a) mRNA'ların piyasada bulunan oligo(dT) ile yalıtım birleşince RNA ASOs. Bu iki adım yordam bulaşıcı proteinlerin ortadan kaldırmak yardımcı olur ve ayarlama ve en iyi duruma getirme için fırsatlar ekler. Bu örnekte, gezi üzerinde seçili mRNA'ların Maya, nematod, uygulandı ve in vivo onaylamak için insan hücreleri mRNA-protein kompleksleri kurdu.
Protokol
1. tasarım antianlamlı Oligonucleotides (ASOs)
- MRNA veya kullanılabilir çevrimiçi araçlar38kullanarak bir parça ikincil yapısını analiz. Bu nedenle, nükleotid (nt) dizisi boş kutuya girin. Temel seçenekler kutusunda en az serbest enerji (MFE) ve bölümleme işlevi ve izole baz çifti önlemekseçin. Çıkış seçenekleri kutusunda etkileşimli RNA ikincil yapı Arsa seçin ve nihayet tıkırtı devam etmek düğme. Yeni bir pencere olacaktır faiz mRNA ikincil yapısını görüntüler pop-up.
- En az üç farklı ilgi tercihen bölgeleri tespit ikincil yapılar eksik mRNA içinde 21-24 nts uzun serileri seçin (Örn., yapılandırılmamış döngülerinde) ve 3' çevrilmemiş bölgeleri (UTR).
Not: ASOs sıralarını 3' çevrilmemiş bölgelerde (UTR) tavlama en iyi performansı gösterdiği gözlendi. Bir ihtimal kodlama dizisi (CD) bağlı ribozomlarda bazen ASOs tavlama engel. 5'-UTRs tavlama ASOs test değil.- Guanidin/sitozin oranı % 50'e yakın bölgeleri seçin ve nükleotid tandem eksik saç tokaları veya kendini tavlama potansiyel oluşumunu önlemek için yinelenir.
- El ile değişiklik tasarım 2'-metoksi adlı 3', tam olarak istenen içinde seçili bölgeler (adım 1.2) için tamamlayıcı bir biotin yan taşıyan RNA oligonucleotides mRNA hedef.
Not: 2'-metoksi değişiklikler RNA dayanıklı hücresel RNases için render ve erime sıcaklığı, sıkı yıkamak için izin veren çift yönlü artırır. Biotin yan ASOs streptavidin ile yakalamak için gereklidir.- ~ 60-65 için RNA melez erime sıcaklığı ayarlamak ° C ve yüksek dil sıra karmaşıklığı olduğundan emin olun (> % 60) uygun online araçlar39ile belirlendiği gibi.
- Temel yerel hizalama arama aracı40 potansiyel ASO çapraz-hibridizasyon ile diğer mRNA'ların transcriptome içinde aramak için kullanın. Nükleotit patlama, dizileri boş kutuya eklemek ve faiz organizmanın seçin. Kalan parametreleri varsayılan tutmak ve patlama' ı tıklatın.
Not: 8-10 nts bile kısmi sürekli hizalamasını çapraz-hibridizasyon ve kurtarma bu mRNA yol açabilir.
2. hücre kültürü, UV radyasyon vermeliyiz ve hücre lizis
Not: S. cerevisiaemayası, nematodlar C. elegansve insan embriyonik böbrek hücreleri (HEK293) için yordamı aşağıda, tasvir edilmiştir. Yine de, her ne kadar açıkça henüz test değil de, diğer organizmalar için adapte edilebilir.
- S. cerevisiae
- Maya hücreleri büyümek (Örn., zorlanma BY4743 modeli; MATa/α his3Δ1/his3Δ1 leu2Δ0/leu2Δ0 LYS2/lys2Δ0 met15Δ0/MET15 ura3Δ0/ura3Δ0 PFK2:TAP / PFK2) YPD medya 500 ml (%1 maya ekstresi, % 2 pepton, % 2 D-glikoz) 30 ° C'de sabit 220 devir / dakikada sallayarak ile.
- Orta günlük aşamada hücreleri toplamak (600 nm (OD)600 optik yoğunluk ~ 0,6) 0,45 µm naylon filtreleri kullanarak filtreleme veya Santrifüjü 3000 × g (RT) Oda sıcaklığında 2 min için de. Akış yoluyla veya süpernatant, sırasıyla atmak.
- 0,45 µm naylon filtre kullanarak hücreleri üç kez fosfat-arabelleğe alınmış-tuz (PBS) 25 mL ile yıkayın veya Santrifüjü RT, 2 dk 3000 × g de tarafından toplamak ve süpernatant atın. Mümkün olduğunca çabuk empoze stres RNA-protein etkileşimleri üzerinde bir etkiye sahip olarak hücreleri toplamak.
- PBS 25 mL hücrelerde resuspend ve süspansiyon bir 15 cm Petri kabına dökün. Çanak buza koyun, kapağını kaldırmak ve üç kez ile 254 nm UV ışık UV crosslinker nazik karıştırma ile her pozlama döngüsü arasında buzda 2-min sonlu 400 mJ cm-2 hücreleri göstermek.
- Hücreleri bir 50 mL tüp aktarmak ve 3000 × g 4 ° C'de 3 dk de Santrifüjü tarafından hücreleri toplamak Süpernatant atın ve Pelet koruyun.
Not: Hücreler sıvı azot içinde donmuş ve-80 ° C'de depolanan ek olabilir - Soğutulmuş lizis arabelleği A 4 mL hücrelerde resuspend (LB-A; 100 mM Tris-HCl, pH 7.5, 500 mM LiCl, 10 mM EDTA, % 1 Triton X-100, 5 mM DTT, 20 U ml-1 ben 100 U ml-1 RNasin, proteaz inhibitörü kokteyl EDTA ücretsiz tam DNaz).
- Hücreleri iki 2 mL tüpler içine aktarın. Max ekleyin. ⅔ hacimleri soğutulmuş cam boncuk ve 4 ° C'de 10 dakika 30 Hz değerinde bir doku lyser hücrelerde bozabilir
- Sıcak bir iğne ile tüpün dibinde delik ve lysate bir taze 1,5 mL tüp tarafından Santrifüjü 600 × g 30 için de transfer s.
- Lysate 3 dk, 3000 × g de 4 ° C'de üç sıralı centrifugations tarafından o zaman 5000 × g ve 10.000 × g 5 min için temizleyin.
Not: Özler olabilir ek donmuş sıvı azot ve-80 ° C'de depolanan
- C. elegans
- 20 ° C'de kültür Bristol N2 solucanlar tabaklarda OP50 Escherichia coli gerilme ile seribaşı Yuvarlak solucanlar büyüme orta (NGM) (%0.3 NaCl, %1,7 agar, % 0.25 pepton, 1 mM CaCl2, 5 µg/mL kolesterol, 1 mM MgSO4, 25 mM KPO4 arabellek, pH 6.0) 41 .
- M9 arabellek41 (% 0,3 KH2PO4, %0.6 Na2HPO4, %0,5 NaCl, 1 mM MgSO4) 5 mL her plakasına eklemek, solucanlar resuspend ve 15 mL tüp solucanları aktarmak için plaka salla. Bir slayt içinde süspansiyon 2 µL yerleştirin ve süspansiyon de solucan mikroskopla saymak. Sonra kurt başı plaka sayısını hesaplamak için faktör geçerlidir.
- Toplamak ~ 120.000 solucanlar Santrifüjü oda sıcaklığında (400 × g, 2 dk, RT) tarafından ve süpernatant atın.
- Solucanlar üç kez yıkayın. Bu nedenle, M9 arabellek 10 mL ekleyin, INVERSION tarafından mix ve solucanlar tarafından Santrifüjü 400 × g RT. 2 min için de toplamak
- Solucanlar için M9 arabellek 15 mL ekleyin ve 15 dakika RT., çevirme bir tekerlek üzerine yerleştirin
- Solucanlar NGM tabak (~ 4.000 kurt başı plaka) aktarmak ve UV-ışığa maruz (254 nm), UV crosslinker 300 mJ cm-2 .
- M9 arabellek 5 mL doğrudan plaka ekleyin ve solucanlar arabellekte resuspend için plaka sallamak. Bir pipet ile süspansiyon 15 mL tüp aktarmak ve solucanlar tarafından Santrifüjü 400 × g RT. 2 min için de toplamak
- Solucanlar 2 mL lizis arabellek B resuspend (LB-B; 100 mM Tris-HCl, pH 8.0, 150 mM NaCl, 1 mM EDTA, % 0.75 IGEPAL, 1 mM DTT, 20 U mL-1 ben 100 U mL-1 RNasin, proteaz inhibitörü kokteyl EDTA ücretsiz tam DNaz).
- Sıvı azot ile dolu bir harç de solucan eziyet. 50 mL tüp ve tezcan, RT. toz toplamak
Not: Güvenlik prosedürleri (Örneğin, duman hood ve emanet eldiven) göre sıvı azot ele alınması gerekir - Lysate yağ tabakası tarafından Santrifüjü 14.000 × g 10 min için de dahil olmak üzere temizleyin ve daha sonra açıklanan geçmek bir şırınga ile 0,45 mikron filtre aracılığıyla lysate.
- İnsan kültürlü hücreleri
Not: Aşağıdaki açıklamalar HEK293 hücre pGL3-CDKN1B-3 geçici transfection dayanır ' UTR muhabir plazmid CDKN1B/27 aşağı ateş böceği luciferase gen423′UTR ifade eder.- Kültür HEK293 hücreleri içinde Dulbecco'nın modifiye kartal Orta (25 mM glikoz ve 1 mM sodyum pyruvate, içeren DMEM) 100 U mL-1 penisilin, 100 µg mL-1 streptomisin ve % 10 fetal Sığır serum (FBS) ile desteklenmiş ve 37 ° C'de, kuluçkaya bir oksijen odası (kuluçka) içeren % 5 CO2.
- Tohum ~ 3 × 106 HEK293 hücrelerde bir standart 10 cm doku kültürü transfection önceki gün çanağı. Bir hemasitometre içeren hücreleri saymak.
- Mix 2 µg muhabir gen (Örn., pGL3-p27-3'UTR) transfection reaktif 20 µL ile ve % 70 confluency (~ 7 × 106 hücreleri), hücre transfect.
- Hücreleri daha fazla 48 saat için bir kuluçka-37 ° C'de hasat önce yerleştirin.
- Serolojik pipet ile orta kaldırmak ve hızlı bir şekilde iki kez önceden 37 ° C'de ısındı PBS 10 mL içeren hücreleri yıkayın PBS serolojik pipet ile kaldırın.
- Yemek için PBS 6 mL ekleyin, buza koyun ve UV ışığı hücrelere sunarsınız (254 nm) adlı bir UV crosslinker 100 mJ cm-2 .
Not: Plaka UV ışık pozlama sırasında buz üstünde tutulmalıdır. - PBS ve 15 mL tüp transferi (hücrelerdeki toplam ~ 107 ) hücrelerde kapalı kazımak.
- Spin aşağı 250 × g 4 ° C'de 10 dakika için hücreleri ve süpernatant bir pipet ile kaldır.
Not: Hücre Pelet ek donmuş sıvı azot ve-80 ° C'de kadar kullanmak depolanır. - Önceden soğutulmuş lizis arabelleğinin 2 mL hücrelerde resuspend (LB-A) tarafından yukarı ve aşağı için pipetting 5 - 6 kez, süre tüp buz üzerinde tutmak.
- Lysate bir pipet ile buz üzerinde yerleştirilen bir 5 mL tüp aktarın.
- Lysate üç tur 30 ile 10 mikron genlik, 20 s patlamaları oluşan sonication için konu dönemlere ilişkin buz soğutma s.
Not: parçaları DNA ve hücre lizis tamamlar gibi Sonication önerilir. - Lysate 2 mL tüpler ve 4 ° C'de 10 dak için 15.000 × g, santrifüj transfer (= Özü) süpernatant toplamak ve yeni bir tüp aktarmak. Daha fazla protein ve RNA analizi için analitik bir örnek (5-%10) kaldırın.
Not: Bu adım kalan herhangi bir hücre artıkları kaldırmak için çok önemlidir ve tekrar edilebilir.
3. ilk adım: Poli(a) RNA izolasyon
- 1 mg oligo(dT)25equilibrate-500 µL ilgili lizis arabelleği manyetik boncuklar birleştiğinde.
- 5 mg, 10 mg veya 4 mg (protein) S. cerevisiae, C. elegans veya HEK293 özler, sırasıyla, oligo(dT)25 1 mg ile birleştirmek-manyetik boncuklar birleştiğinde. Örnekleri şiddetle için bir karıştırıcı 25 ° C'de 10 dakika karıştırın.
Not: Protein konsantrasyonları özler Sığır serum albumin (BSA) bir referans standart olarak kullanarak Bradford tahlil ile belirlenebilir. Dinç örnekleri sallayarak sedimantasyon boncuk engeller. MRNA yalıtım özgüllük değerlendirmek için bir denetim deney paralel olarak aşırı (20 µg) polyadenylic asit (pA) ekleyerek yapılabilir. - 10 s ve çıkarmak için manyetik bir stand tüpler yer süpernatant. Süpernatant kurtarma (adım 3.7) sonraki tur için buz üzerinde tutun.
- Yıkama 500 µL tampon A Ekle (10 mM Tris-HCl, pH 7.5, 600 mM LiCl, 1 mM EDTA, % 0,1 Triton X-100) boncuk ve 5 için girdap s.
Not: LiCl konsantrasyon yıkama arabelleklerindeki değişebilir. 600 mM LiCl tavsiye edilir ama düşük konsantrasyonlarda da (500 mM HEK293 için 300 mM C. elegansiçin) test edildi. - Toplamak bir mıknatıs ile boncuk ve boncuk 500 µL yıkama arabelleği B (WB; 10 mM Tris-HCl, pH 7.5, 600 mM LiCl, 1 mM EDTA) ile iki kez yıkayın.
- 10 mm Tris-HCl, pH 7.5 için sürekli (1000 devir/dakika) sallayarak ile 2 dk 80 ° C'de 30 µL RNA bir karıştırıcı elute. Hemen tüp manyetik tribünde yer ve eluate 10'dan sonra toplamak s. kaydetme arınma ek vizit boncuk.
Not: eluate mRNA'ların daha düşük sıcaklıklarda boncuk için potansiyel yeniden birleştirme önlemek için mümkün olduğunca hızlı bir şekilde toplamak. - Boncuk önceki adım adım 3.3 toplanan süpernatant ekleyin ve yakalama, çamaşır ve mRNA'ların, elüsyon iki kez (adım 3.3-3.6) tekrarlayın. Eluates tekrarlanan tur üzerinden sonra birleştirilir ve-80 ° C'de depolanan
4. ikinci adım: 3'-Biotinylated 2'-metoksi modifiye antianlamlı RNA Oligonucleotides ile belirli bir yakalama mRNA'ların
Not: ASOs uygunluğu test etmek için aşağıdaki yordamı ile hücreleri izole toplam RNA'ların yapılabilir.
- Streptavidin birleştiğinde manyetik boncuklar bağlama 1 ml 30 µL equilibrate ve yıkama arabellek (B & W arabellek; 10 mM Tris-HCl, pH 7.5, 150 mM NaCl, 0,5 mM EDTA, pH 8.0) bir rotator için 1 h üzerinde 0.1 mg mL-1Escherichia coli transfer RNA (tRNA) içeren RT.
- Boncuk üç kez B & W arabelleği 750 µL ile yıkayın.
- Boncuk B & W arabellek 30 µL içinde resuspend ve buz üzerinde kullanımda kadar devam.
- ~ 35 µg önceki poli(a) yalıtım üzerinden toplam protein seyreltik (3 bakınız) B & W arabelleği bir 1,5 mL tüp 100 µL içinde.
Not: 600 ASOs toplam RNA ile özgüllük test etmek için ekleme B & W arabelleği 100 µL için saflaştırılmış toplam RNA'ın ng. - İlgili ASO 200 pmol ekleyin ve 5 min için 70 ° C'de kuluçkaya.
Not: Yüksek sıcaklık ASO tavlama kolaylaştırmak RNA ikincil yapılarda giderir. - Tüm ısı-blok aygıttan kaldırmak ve RT yavaş yavaş soğuması için 10 dakika için yerleştirin.
- Streptavidin birleştiğinde dengelenmiş manyetik boncuklar 30 µL adımından 4.1 için örnek ekleyin.
- Karışımı bir karıştırıcı 950 rpm sabit sallayarak ile 30 dk 25 ° c için kuluçkaya.
Not: Her 10 min tüpler sedimantasyon boncuk önlemek için fiske önerilir. - Tüpler manyetik kürsüye getirin, süpernatant kaldırmak ve boncuk üç kez ile önceden ısıtılmış B & W arabelleği 750 µL 55 ° C'de yıkama
Not: En uygun yıkama sıcaklığı biraz farklı/farklı ASOs arasında değişebilir. Bu çapraz olmayan toplam RNA önceki hücre özleri ile deneme gerçekleştirme ile bu koşul ayarlamak için tavsiye edilir. - 10 mm Tris-HCl, pH 7.5 ile bir mikser. 950 devirde sabit sallayarak 10 dk 90 ° C'de 20 µL RNA elute Tüpler manyetik tribünde yer ve hemen eluate toplamak.
Not: Protein analizi için 20 µL 1 × Laemmli arabelleği için boncuk ekleyin ve 5 dk. proteinler Standart laboratuvar protokolleri takip immunoblot analiz tarafından izlenir için 95 ° C'de kuluçkaya.
Sonuçlar
Biz üç farklı organizmalar37--dan onların ilişkili proteinler ile belirli mRNA'ların yakalamak için bir yolculuk, olarak adlandırılan bir RNA ASO tabanlı yalıtım stratejisi geliştirdi. Esasen, RNA-protein kompleksleri çapraz vivo içinde olduğunu tarafından UV-ışınlama 254 nm ve poli(a) hücrelerin RNA'ların piyasada bulunan oligo (dT) ile birleştiğinde-manyetik boncuklar ele geçirildi, sonra mRNA ilgi ile izole edildi 3'-biotinylated 2-0'-metoksi antianlamlı RNA oligonucleotides (Şekil 1) değiştiren. Bu nedenle Maya, C. elegans ve insan bölgelere seçili mRNA birkaç 21-24 değiştiren nts ASOs tam tamamlayıcılık ile tasarlanmış ve mRNA (astar ve ASOs listesini tablo verilir ilgi kurtarmak için uygunluk test 1). verimliliği ve bireysel ASOs özgüllüğü ilk ilgili organizma izole etmek toplam RNA ile değerlendirildi. Bu deneylerde, RNA ASOs streptavidin Birleşik paramagnetic boncuk birleştiğinde ve çapraz toplam RNA ilgili organizma hazırlanan ile inkübe edildi. Boncuk üzerinden yakalanan mRNA'ların yayımlandıktan sonra mRNA varlığı de ilişkisiz denetim mRNA'ların ters Transkripsiyon (RT) tarafından izlenen hedefler-polimeraz zincir reaksiyonu (PCR)37. Biz bu iki değişken, tuz konsantrasyonu ve sıcaklık yıkama arabellekleri, üç farklı ASOs (Şekil 2A) ile test edildi Maya dan PFK2 mRNA yakalama verimliliği önemli roller oynadı fark ettim. 25 mM için tuz (NaCl) konsantrasyon düşürücü azaltılmış kurtarma olumsuz ve tüm ASOs ile non-detectable düzeylere mRNA (PFK1) kontrol, ama aynı zamanda istenen kurtarma azaltılmış mRNA hedef PFK2 (giriş 10-%15). Bunun tersi olarak, fizyolojik düzeyleri (150 mM NaCl) tuz konsantrasyonları artış % 75'e kadar ile ASO PFK2-bu PFK1 denetiminin aşan 2, PFK2 mRNA'ların kurtarılması arttı mRNA tarafından en az 5-fold (Şekil 2A). Daha fazla dikkat, farklı ASOs büyük değişim mRNA hedef yakalama efficacies fizyolojik tuz konsantrasyonları ASOs. ampirik doğrulama ihtiyacını vurgulayan gösterdi MRNA kurtarma bağımlılığını yıkama arabellek sıcaklığı C. elegans cep-1 ve Maya ilgili ASOs (Tablo 1) kullanarak RPS20 mRNA'ların örneklenir. Biz en uygun yıkama sıcaklığı 50 ° C ve ilgisiz mRNA'ların ve mRNA'ların hedef (2B rakam) verimli kurtarma düşük arka plan belirgin olarak 55 ° C arasında gözlendi. Bu noktada, çapraz-hibridizasyon ASOs ile diğer mRNA'ların olasılığı vurgulamak istiyoruz. Örneğin, DNM1 ASO tam olarak tamamlayıcı bir sıra sıra kodlama DNM1 içinde ama aynı zamanda kısmen ACT1 mRNA ile anneals. DNM1 ASOs yıkama sıcaklığı ne olursa olsun her iki mRNA'ların çapraz-hibridizasyon (Şekil 2C) için güçlü eğilimi gösteren kurtarıldı. Son olarak, biz-si olmak kullanılmış yukarıda açıklanan testleri ve S. cerevisiae, C. elegans ve insan hücreleri (Şekil 2B izole RNA ilgili hedef mRNA'ların kurtarılması için uygun üç ASOs seçmek için en iyi duruma getirmeleri toplam ).
Uygun ASOs in vitro sonra ilk seçim UV-çapraz organizmalar/hücrelerden (Şekil 1) elde edilen hücre özleri ile yolculuk yapılır. Özellikle, üç farklı mRNA'ların üç farklı organizmalar gelen kurtarma test: PFK2 mRNA S. cerevisiae, cep-1 nematodunun C. elegans, ve bir muhabir Maya dan mRNA (pGL3-CDKN1B-3'UTR) 3' UTR taşıyan insan CDKN1B/p27 mRNA dizisi için geçici ifade insan HEK293 hücrelerinde luciferase (luc) muhabir için erimiş. RNA izolasyon özgüllük denetlemek için biz birkaç alakasız mRNA'ların kurtarma izlenen ve oligo(dT)25 için hücresel mRNA'ların bağlayıcı ile rekabet hücre özleri, pA aşırı ilavesi rekabet deneyler gerçekleştirdiği boncuk ilk adım arıtma sırasında. Olarak daha önce görüldü çapraz toplam RNA örnekleri (resim 2), RT-PCR doğruladı ile ilgili mRNA'ların zenginleştirme hedef mRNA yolculuk sırasında birkaç denetim ilgili olmayan mRNA'lar değil zenginleştirilmiş Oysa (Şekil 3A). Ayrıca, ben de mRNA'ların boncuk ASOs, belirsiz bağlama önlemek uygun engelleme işlemleri gösteren olmadan üzerinde algılandı. Çapraz-hibridizasyon belirli mRNA'ların ve ilişkili proteinler inferred yakalanması için potansiyel sonra dikkate alınacak olsa da bu satırda ilgisiz/şifreli ASOs da denetimi olarak kullanılabilir. Son eluate proteinlerin neredeyse gümüş lekeli polyacrylamide jeller üzerinde (veri gösterilmez) görüntülenmeyecektir beri daha önce bilinen mRNA etkileşen proteinlerin varlığı daha fazla immunoblot analiz tarafından değerlendirildi. Bu Pfk2p seçerek PFK2 mRNA bir ribozom bağımsız şekilde12bağlar S. cerevisiae' dan C. elegans GLD-1, 3' UTR dizileri cep - 1 bağlar bir kanonik RBP mRNA için translasyonel yönetmelik43; ve HuR, mRNA kararlılığı ve çeviri p27/CDKN1B mRNA44düzenleyen bir RBP. Beklendiği gibi tüm bu proteinler Western Blot Analizi (Şekil 3B) ile ilgili mRNA'ların gezi eluates tespit edilmiştir.
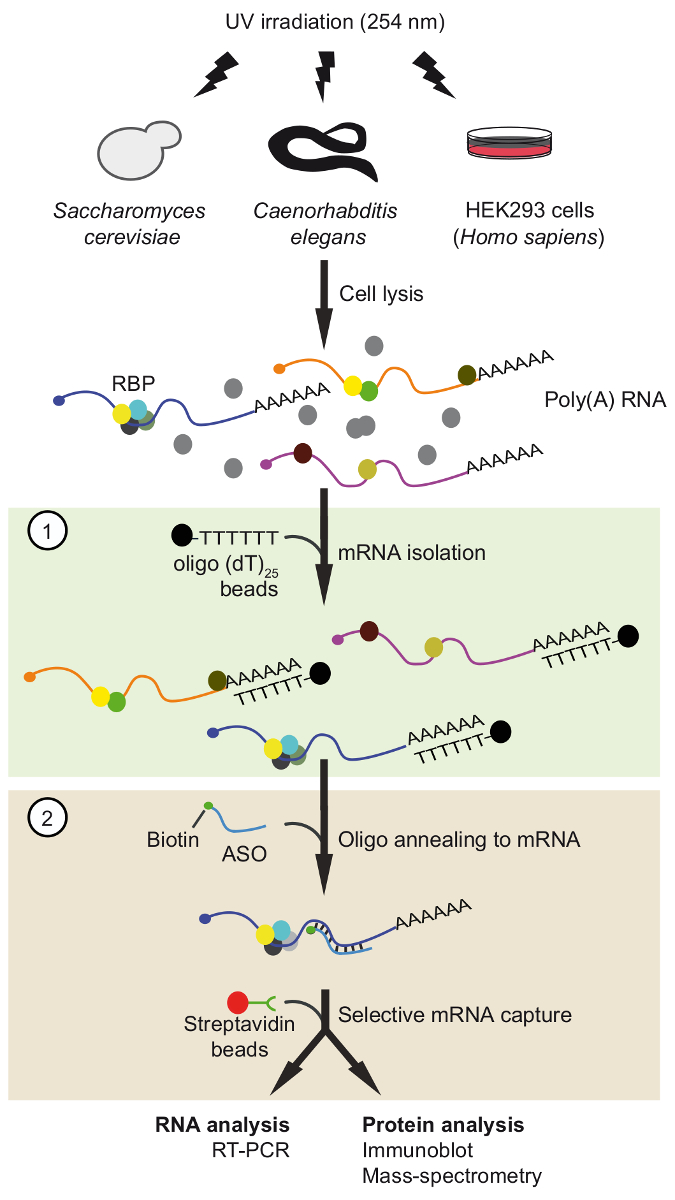
Şekil 1. Şematik Gösterim gezisi. Etmek için RNA vivo içinde UV-ışınlama tarafından proteinlerdir. İlk adımda (yeşil ışıklı), poli(a) RNA-protein kompleksleri ilişkisiz proteinler kaldırmak için sıkı çamaşır koşullar uygulayarak oligo(dT)25 boncuk ile kazanılmaktadır. İkinci adımda (pembe kutu), hedef mRNP çekti-biotinylated antianlamlı RNA oligonucleotides ve streptavidin boncuklarla out. Saf mRNPs sonra RT-PCR ve immunoblot/kütle-spektrometresi (RNA ve proteinler sırasıyla faiz, mRNA ile etkileşim tanımlamak için MS) tarafından incelenir. Rakam üzerinden önceki yayın37 izni ile güncellenmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
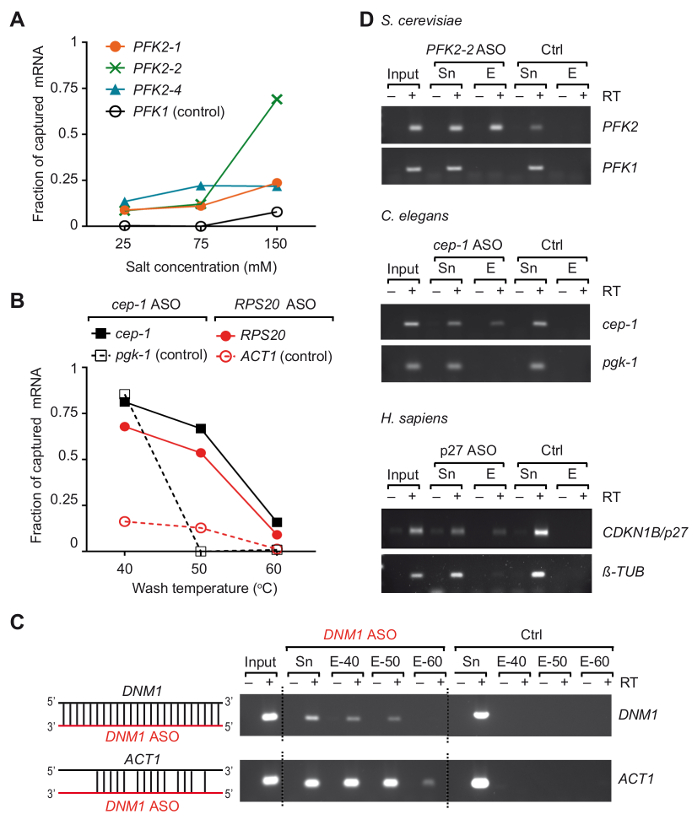
Şekil 2. Çapraz olmayan hücreler/organizmaların kullanarak toplam RNA üzerinden seçili mRNA izolasyonu değiştiren antisens RNA yakalama probları. (A)tuz konsantrasyonları Maya PFK2 mRNA ASOs ile yakalama verimliliği üzerine etkisi. Toplam RNA Maya hücreleri ile belirtilen ASOs kombine ve 55 ° C'de belirtilen tuz (NaCl) konsantrasyon içeren bir arabellek ile yıkanmış Yeşil, mavi ve turuncu satırları temsil eden PFK2 mRNA kurtarma RT-PCR37tarafından belirtildiği gibi farklı ASOs ile: PFK2-1 ve PFK2 -2 tavlama içinde 3' UTR, PFK2-4 belgili tanımlık CDS '. PFK1 bu olumsuz bir mRNA kontrol. PCR ile 32 amplifikasyon devir yapılan ve yukarıda açıklanan37sayılabilir. C. eleganscep-1 ve Maya RPS20 ASO (B) kısmı siyah ve kırmızı çizgilerin içinde sırasıyla temsil farklı yıkama sıcaklıklarda mRNA'ların bağlı. C. eleganspgk-1 ve Maya ACT1 sigara-hedef (kontrol) mRNA'ların olur. 30 ve 32 PCR döngüleri Maya ve C. elegans mRNA tespiti için sırasıyla uygulandı. (C) DNM1 ile ACT1 mRNA DNM1 mRNA hem de potansiyel çapraz-hibridizasyon serileri ile (kırmızı) ASO hibridizasyon şematik gösterimi sola gösterilir. RT-PCR reaksiyonları (30 döngüleri) boncuk eluted Maya DNM1 ve ACT1 mRNA tespiti için urun gösterilen bir özel jel sağda gösterilir. Giriş, toplam RNA; Sn, ASOs ile kuluçka sonra süpernatant; E, eluates, yıkanmış boncuk üzerinden sıcaklıklar öncesinde elüsyon (40 ° C, 50 ° C ve 60 ° C) gösterilir. Bir denetim deney (Ctrl) ASO eklemeden paralel olarak gerçekleştirildi. (D) özel jel RT-PCR ürünleri (sağda) S. cerevisiaeMaya, C. elegansve insan HEK293 hücreleri (H. sapiens) Toplam RNA yakalanan mRNA tespiti için gösterilen. Giriş, toplam RNA; Sn, süpernatant; E, boncuk üzerinden eluates. PCR Maya mRNA'ların 30 amplifikasyon devredir, 32 C. elegans mRNA'ların devredir tarafından gerçekleştirilmiş ve 28 ve 30 insan tübülin ve p27 mRNA'ların, sırasıyla döngüleri. Rakam üzerinden önceki yayın37 izni ile güncellenmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
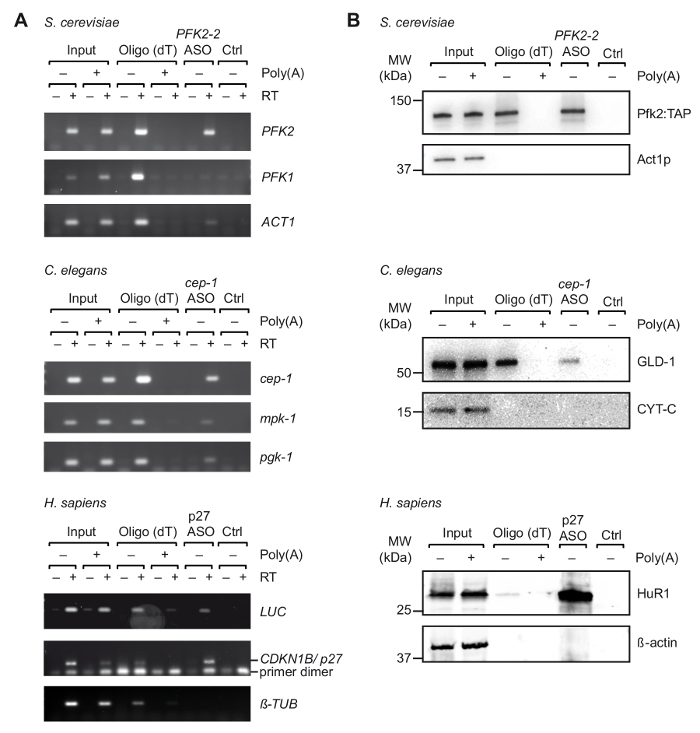
Şekil 3. Belirli mRNA-protein kompleksleri özleri gelen yakalanması elde edilen gezi ile UV çapraz hücrelerden. (A)özel jelleri mRNA'ların (sağda) RT-PCR ile oligo(dT) ve belirtilen ASOs S. cerevisiae, C. elegans ve insan (H. sapiens) hücre özleri ile yakalama üzerine tespiti için. Giriş, çapraz hücre/organizmalar üzerinden toplam RNA; CTRL, denetim ASO olmadan. Poli(a), poli(a) ile rekabet. RT-PCR olarak açıklanan37 35 amplifikasyon çevrimleri ile LUCiçin p27, 32 devredir gerçekleştirildi ve 29 tübüliniçin döngüleri. (B) mRNA bağlı proteinler belirtilen antikorlar (sağda) ile Immunoblot analizi. Yüklenen kesirler şunlardır: %0,1, % 2.5 ve %1 Maya için yuvarlak ve insan girişleri; % 10, % 10 ve % 5'i Maya, yuvarlak ve insan oligo(dT) şerit; ve tüm ASOs yolları için % 66'ya. Moleküler Ağırlık (MW) kilodaltons (kDa) gösterilir. Şekil izni37ile yayınlanacaktır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
| Astar adı | Sıra | Hedef | Boyutu | |
| Pfk2_Fwd | GTGTTAAGGGTTCACATGTCG | PFK2 S. cerevisiae | 133 bp | |
| Pfk2_Rev | CTTCCAACCAAATGGTCAGC | PFK2 S. cerevisiae | 133 bp | |
| Pfk1_Fwd | GGTGATTCTCCAGGTATGAATG | PFK1 S. cerevisiae | 97 bp | |
| Pfk1_Rev | CTTCGTAACCTTCGTAAACAGC | PFK1 S. cerevisiae | 97 bp | |
| Act1_Fwd | GTCTGGATTGGTGGTTCTATC | ACT1 S. cerevisiae | 85 bp | |
| Act1_Rev | GGACCACTTTCGTCGTATTC | ACT1 S. cerevisiae | 85 bp | |
| Dnm1_Fwd | CTGTGTTCGATGCATCAGAC | DNM1 S. cerevisiae | 156 bp | |
| Dnm1_Rev | CGCACTCCAATTCTTCTCTC | DNM1 S. cerevisiae | 156 bp | |
| Rps20_Fwd | CGCTGAACAACACAACTTGG | RPS20 S. cerevisiae | 228 bp | |
| Rps20_Rev | GGAAGCAACAACAACTTCGAC | RPS20 S. cerevisiae | 228 bp | |
| Cep1_Fwd | CGATGAAGAGAAGTCGCTGT | Cep-1 C. elegans | 110 bp | |
| Cep1_Rev | ATCTGGGAACTTTTGCTTCG | Cep-1 C. elegans | 110 bp | |
| Pgk1_Fwd | GCGATATTTATGTCAATGATGCTTTC | PGK-1 C. elegans | 74 bp | |
| Pgk1_Rev | TGAGTGCTCGACTCCAACCA | PGK-1 C. elegans | 74 bp | |
| Mpk1_Fwd | TGCTCAGTAATCGGCCATTG | MPK-1 C. elegans | 74 bp | |
| Mpk1_Rev | TCCAACAACTGCCAAAATCAAA | MPK-1 C. elegans | 74 bp | |
| p27_Fwd | TTTAAAAATACATATCGCTGACTTCATGG | p27 H. sapiens | 212 bp | |
| p27_Rev | CAAAGTTTATGTGCTACATAAAAGGTAAAAA | p27 H. sapiens | 212 bp | |
| Luc_Fwd | AATGGCTCATATCGCTCCTGGAT | Luciferase P. pγralis | 117 bp | |
| Luc_Rev | TGGACGATGGCCTTGATCTTGTCT | Luciferase P. pγralis | 117 bp | |
| Β-TUBULIN_Fwd | CTGAACCACCTTGTCTCAGC | Β-TÜBÜLİN H. sapiens | 136 bp | |
| Β-TUBULIN_Rev | AGCCAGGCATAAAGAAATGG | Β-TÜBÜLİN H. sapiens | 136 bp | |
| PFK2-1 ASO | AAUAGAAAGUGUAAUAAAAGGUCAU | 3' UTR PFK2 S. cerevisiae | - | |
| PFK2-2 ASO | GUUUCAUGGGGUAGUACUUGU | 3' UTR PFK2 S. cerevisiae | - | |
| PFK2-4 ASO | CUUGAAGAGGAGCGUUCAUA | ORF PFK2 S. cerevisiae | - | |
| DNM1 ASO | UCGGUCAGUGGAGGUUCAGCGUUU | ORF DNM1 S. cerevisiae | - | |
| RPS20 ASO | GUCGGUAAUAGCCUUCUCAUUCUUG | ORF RPS20 S. cerevisiae | - | |
| cep-1 ASO | GUGAGAAAUGCGGUGCUUUGAAA | 3' UTR cep-1 C. elegans | - | |
| p27 ASO | UCAUACCCCGCUCCACGUCAGUU | 3' UTR p27 H. sapiens | - | |
Tablo 1. Oligonükleotid dizileri. PCR astar ve bu iş, astar sıralama, gen hedef ve beklenen parça boyutu amplifikasyon sonra kullanılan ASOs listesi.
Tartışmalar
GEZİ belirli mRNA'ların vivo içinde biyokimyasal anlamına gelir ile bağlı proteinler analizine izin verir. Belirli RBPs etkileşim mRNA hedefleri farklı organizmalar/hücrelerdeki doğrulamak için yöntemi kullandık, yolculuk da poli(a) RNA'lar, sitoplazmik uzun ncRNA'lar gibi diğer türleri çalışmak için uygun olabilir. Ayrıca, MS veya RNA sıralama ile ilişkili proteinler ve/veya RNA'ların sistematik Analizi bir yukarı ölçekleme tarafından yordam elde edilebilir. Bu bağlamda, ön veriler 1 litre maya kültürleri ve en az 100 milyon insan HEK293 hücrelerinin yeterli Başlangıç materyali iyi ifade mRNA'ların için güvenilir MS veri elde etmek için (yayınlanmamış) sonuçlar sağlayabilir gösteriyor.
Açıklanan gezi Protokolü 254, UV ışığıyla hücreleri ışınlama içerir nm crosslink RNA-protein etkileşimleri. Bu sıkı yıkama koşulları uygulama arıtma sırasında sağlar. Yine de, açıkça test rağmen crosslinking isteğe bağlıdır ve etkin RNPs aynı zamanda fizyolojik arabellekleri ile kurtarılabilir Not istiyoruz. Ancak, bu durumda, protein ve RNA'ların veya potansiyel yeniden düzenleme hedef RNA lysates dikkate alınması. UV ya da diğer crosslinking yordamlar Uygulamalı (Örneğin, formaldehit) ise, RNA yıkımı azalmış mRNPs kurtarılması için neden olabilir gibi tersine, RNA'ın bütünlük değerlendirilmelidir. Ayrıca, UV-crosslinking oldukça verimsiz (~ %5) olduğunu ve daha az çok çift iplikçikli RNA ile etkileşim proteinler için hangi bazı sınıflar RBPs12tespiti için önyargı tanıştırabilirim. Son olarak, UV ışınlama için veri yorumu45,46düşünülmesi gereken protein-protein ve protein-DNA Glossar tetikleyebilir. Bu nedenle, alternatif crosslinking yordamlarla, örneğin formaldehit, aynı zamanda daha büyük protein derlemeler mRNA'ların üzerinde yakalamak için özel ilgi olabilir.
Bir ana gezi ASO tabanlı arıtma yordamı belirli RNA'ların, böylece artan seçicilik ve Co kirleticiler arındırıcı olasılık azalan kurtarmak için iki adım özelliğidir. Örneğin, doğrudan biotin bağlanıcı proteinler ortak arıtma için yol açabilecek hücre özleri biotinylated ASOs ekleme için bir endişe ile ilgilidir. Bu RNA-protein interactome yakalama için bitmiş gibi sıkı arıtma koşulları sağlayan arıtma kovalent eşleşmiş oligo-(dT)25 manyetik boncuklar, kullanarak poli(a) RNA'ların ilk turunda uygulayarak ve hile. Ayrıca, poli(a) RNA seçime ribozomal RNA'ların ve tRNA, gibi son derece bol ncRNA'lar kaldırır ve bu nedenle örnek ve çapraz-hibridizasyon ve kirlenme için potansiyel kaynakları karmaşıklığı azaltır. İkinci adımda, belirli mRNA'ların biotinylated ile yeniden elde etmek ve mRNA hedeflerde bölgeleriyle tavlama ASOs değiştirilebilir. Alternatif olarak, değiştirilmiş ASOs da doğrudan kovalent bir amin bağlayıcı üzerinden manyetik boncuklar için biotin proteinler yakalama eğilimi azaltarak bağlanması. Ancak, deneyim, streptavidin boncuk eklenmesi önce poli(a) RNA kesir ASOs ile kuluçka mRNPs ASO birleştiğinde boncuk doğrudan ilavesi daha verimli bir şekilde kurtarıldı. Belki de, ücretsiz oligos kinetically ile karşılaştırıldığında immobilize oligos yapılandırılmış RNA dizilerinde daha iyi erişim ile tercih. Bu nedenle, ASOs kovalent kaplin boncuk örnekle kuluçka önce için RNA hedef kurtarma için zararlı olabilir ve ampirik olarak test edilmesi gerekiyor.
ASO dayalı yöntemleri ile bir dezavantajı bazı hedef mRNA'ların verimsiz değildir. Bu nedenle transkript farklı bölgelerinde tavlama birkaç ASOs değerlendirilmesi önerilir. Özellikle, biz 3'-UTR'nin serilerinde ile TAV 2-3 ASOs veya CD mRNA ilgi tasarımını öneririz. Her ASO ilgili mRNA hedef kurtarma için farklı bir verimlilik sergileyebilirler ve ampirik olarak olmalıdır (Şekil 2A, B, verileri gösterilmez) test. Sonunda, 3' UTRs tavlama ASOs daha iyi performans için CD tavlama olanlar ile karşılaştırıldığında bulduk. Bu ribozomlara CD içinde verimli hibridizasyon engel, ancak Binding UTRs sitelerde ribozomlar, çeviri yokluğu nedeniyle artan erişilebilirlik nedeniyle olabilir. Biz aynı zamanda deneyimli birkaç ASOs birleşimi için artan kirlilik bol sigara hedef mRNA'ların neden olabilir ve aşağı açılır verimliliği azalır. Her durumda, seçilen oligos selectivity rekabet deneyler tarafından kontrol edilebilir (Örn., rakip oligos ilavesi).
Biz diğer mRNA'ların ile o bile kısmi hibridizasyon gözlemlediği gibi daha endişe potansiyel çapraz-hibridizasyon ilgisiz RNA'lar ile ilgilidir bu mRNA (Şekil 2C) kurtarma için yol açabilir. Tasarlanmış ASOs uygunluğu değerlendirmek için bu nedenle ilk vitro test en iyi tuz konsantrasyonları ve yıkama sıcaklık arabellekleri için toplam RNA'ları ile gerçekleştirme öneririz. Bizim ellerde streptavidin birleştiğinde manyetik boncuklar 150 mM içeren arabellekleri yıkama NaCl 55 ° C, en iyi performansı gösterdiği, ancak her tasarlanmış ASO için koşullardan bağımsız testler öneririz. Önemli, biz tüm ASOs içinde vitro deneyler (2D rakam) seçili daha fazla içinde geçerliliğini kanıtlayıcı çapraz hücre özleri (Şekil 3), anılan sıraya göre mRNA hedeften yakalamak için uygun bulundu in vitro test. MRNA yakalama için uygun ASOs tasarımı yanı sıra, ek etkenler seçicilik üzerinde etkisi olabilir. Bu nedenle, BSA ve/veya tRNA ile kapsamlı engelleme yordamlar boncuk türü belirtimlerine göre boncuk belirsiz bağlama engelleyebilirsiniz. Belirsiz proteinlerin emilimini daha da azaltmak için Ayrıca Lo-bind tüpler, kullanımı özellikle düşük miktarda örnekleri ile çalışırken öneririz.
Genel olarak, gezi RNA'ların ASOs ile yakalamak için daha önce kurulan yaklaşımlar tamamlayabilir. Örneğin, kodlamayan RNA Xist nükleer özler tüm transkript kaplı uzun (90-mer) biotinylated DNA oligonucleotides bir dizi kullanarak 200-800 milyon UV çapraz hücrelerden türetilmiş gelen ilişkili proteinler ile izole edildi 34,35. Ancak, seçtiğiniz döşeme yaklaşım ışığında çapraz-hibridizasyon ilgisiz RNA'lar ile için büyük bir potansiyel sorunlu haline gelebilir ve belirli transkript, e.gseçmek için kullanılamaz., alternatif olarak formlar spliced. Son zamanlarda, özel olarak tasarlanmış kilitli nükleik asit (LNA) veya DNA ASO mixmers kovalent vitro transkript veya son derece ifade ribozomal RNA'ların boş hücre özleri gelen kurtarmak için manyetik bir reçine bağlı; Ancak, vivo içinde oluşan mRNA-protein kompleksleri36kurtarma için test. Çoğu gen kontrol RBPs ve ncRNA tarafından artan tanıma ışığında, biz gezi kompozisyon ve RNP kompleksleri hücrelerde hücre içi ve çevre ipuçları ve etkileri üzerine dinamikleri araştırmak için yararlı bir yöntem olduğuna inanıyorum Sağlık ve hastalık.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Dr. Jonathan Hall ve Mauro Zimmermann (ETH Zürih) PFK2 ve cep-1 ASOs, Dr Maikel Wouters p27/CDKN1B ASOs ve Dr Rafal Ciosk (Friedrich seiliti Biyomedikal Araştırma Enstitüsü, tasarımı için sentezi için sana şükrediyoruz Basel) anti-GLD-1 karşı antikorlar. Bu eser Biyoteknoloji ve Biyolojik Bilimler Araştırma Konseyi (1/K009303/BB) ve bir Royal Society Wolfson araştırma Merit Ödülü (WM170036) A.P.G. tarafından desteklenmiştir
Malzemeler
| Name | Company | Catalog Number | Comments |
| MaxQ 5000 Large Incubated and Refrigerated Orbital Shaker | Thermo Fisher Scientific | SHKE5000-8CE | Floor shaker to grow yeast cells |
| BBD 6220 | Thermo Fisher Scientific | CO2 incubator to grow human cells | |
| Sonicator Soniprep150 | MSE | MSS150.CX4.5 | Sonicator/cell disruptor. Used to shear DNA in human cell lysates |
| Stratalinker 1800 | Stratagene | Stratalinker to expose cells to UV light at 254 nm | |
| Tissue Lyser | Qiagen | RETSCH MM200 | Device to mechanically disrupt yeast cells |
| Refrigerated Centrifuge | Eppendorf | 5810R | Centrifuge to spin down cells, lysates |
| Shaking incubator, Thriller | Peqlab | Thermoshaker for 1.5 mL tubes | |
| Glass beads 0.5mm | Stratech Scientific Limited | 11079105 | Lysis of yeast cells |
| Nylon Filter, 0.41 μm | Millipore | NY4104700 | Collection of yeast cells |
| Millex-HA Filter, 0.45 µm | EMD Millipore | SLHA02510 | Filter to clear nematode lysate |
| Eppendorf LoBind microcentrifuge tubes Protein 1.5 mL | Sigma-Aldrich | 22431081 | Minimise protein loss |
| 2 mL microfuge tube | Ambion | AM12425 | Yeast extract preparation and other applications |
| 5 mL tube | Thermo Fisher Scientific | 129A | Collection of HEK293 cell lysate |
| 15 mL tube | Sarstedt | 62.547.254 | Collection C. elegans |
| 50 mL plastic tube | Sarstedt | 62.554.502 | Collection yeast cells |
| DMEM, high glucose, pyruvate | Thermo Fisher Scientific | 41966 | Media for culturing HEK293 cells |
| Penicillin-Streptomycin | Sigma-Aldrich | P4333 | Used to supplement cell culture media to control bacterial contamination |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F7524 | Used to supplement cell culture media |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| RQ1 RNase-Free DNase | Promega | M6101 | DNAse I to degrade DNA from cell lysate |
| RNasin Plus RNase Inhibitor | Promega | N2611 | RNase inhibitor to avoid RNA degradation |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitor cocktail to avoid protein degradation |
| Dynabeads Oligo (dT)25 | Thermo Fisher Scientific | 61011 | Magnetic beads required for the first step of mRNA-protein complex purification |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | Magnetic beads required for the second step of mRNA-protein complex purification |
| E. coli tRNA | Sigma-Aldrich | 10109550001 | transfer RNA from E. coli used for blocking streptavidin beads and avoid unsepecific interactions |
| Quick Start Bradford 1x Dye Reagent | BioRad | 5000205 | To determine protein concentration within lysates, using BSA as reference |
| Bovine Serum Albumin | Sigma | A7906-100G | Used to perform the standard curve that will be used as reference for protein concentration determination |
| mouse anti-Act1 | MP Biomedicals | 869100 | Antibody for detection of yeast actin in Western blot (1:2,500) |
| mouse anti-Act1 | Sigma | A1978 | Antibody for detection of human actin in Western blot (1:2,000) |
| mouse anti-HuR | Santa Cruz | sc-5261 | Antibody for detection of HuR in Western blot (1:500) |
| mouse anti–CYC-1 | Invitrogen | 456100 | Antibody for detection of Cyc-1 in Western blot (1:1,000) |
| peroxidase anti-peroxidase soluble complex | Sigma | P1291 | Detection of Pfk2:TAP in Western blot (1:5,000) |
| HRP-conjugated sheep anti-mouse IgG | Amersham | NXA931 | HRP-coupled secondary antibody |
Referanslar
- Mitchell, S. F., Parker, R. Principles and properties of eukaryotic mRNPs. Molecular Cell. 54 (4), 547-558 (2014).
- Iadevaia, V., Gerber, A. P. Combinatorial Control of mRNA Fates by RNA-Binding Proteins and Non-Coding RNAs. Biomolecules. 5 (4), 2207-2222 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Matia-González, A. M., Gerber, A. P., Sesma, A., von der Haar, T. Ch. 14. Fungal RNA Biology. , 347-370 (2014).
- Tsvetanova, N. G., Klass, D. M., Salzman, J., Brown, P. O. Proteome-wide search reveals unexpected RNA-binding proteins in Saccharomyces cerevisiae. PLoS One. 5 (9), (2010).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Castello, A., et al. System-wide identification of RNA-binding proteins by interactome capture. Nature Protocols. 8 (3), 491-500 (2013).
- Liao, Y., et al. The Cardiomyocyte RNA-Binding Proteome: Links to Intermediary Metabolism and Heart Disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Liepelt, A., et al. Identification of RNA-binding Proteins in Macrophages by Interactome Capture. Molecular Cell Proteomics. 15 (8), 2699-2714 (2016).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212 (2016).
- Matia-Gonzalez, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural and Molecular Biology. 22 (12), 1027-1033 (2015).
- Mitchell, S. F., Jain, S., She, M., Parker, R. Global analysis of yeast mRNPs. Nature Structural and Molecular Biology. 20 (1), 127-133 (2013).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127 (2015).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128 (2016).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42 (2016).
- Reichel, M., et al. In Planta Determination of the mRNA-Binding Proteome of Arabidopsis Etiolated Seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Koster, T., Marondedze, C., Meyer, K., Staiger, D. RNA-Binding Proteins Revisited - The Emerging Arabidopsis mRNA Interactome. Trends Plant Sciences. 22 (6), 512-526 (2017).
- Hamasaki, K., Killian, J., Cho, J., Rando, R. R. Minimal RNA constructs that specifically bind aminoglycoside antibiotics with high affinities. Biochemistry. 37 (2), 656-663 (1998).
- Bachler, M., Schroeder, R., von Ahsen, U. StreptoTag: a novel method for the isolation of RNA-binding proteins. RNA. 5 (11), 1509-1516 (1999).
- Vazquez-Pianzola, P., Urlaub, H., Rivera-Pomar, R. Proteomic analysis of reaper 5' untranslated region-interacting factors isolated by tobramycin affinity-selection reveals a role for La antigen in reaper mRNA translation. Proteomics. 5 (6), 1645-1655 (2005).
- Hartmuth, K., Vornlocher, H. P., Luhrmann, R. Tobramycin affinity tag purification of spliceosomes. Methods Molecular Biology. 257, 47-64 (2004).
- Beach, D. L., Keene, J. D. Ribotrap : targeted purification of RNA-specific RNPs from cell lysates through immunoaffinity precipitation to identify regulatory proteins and RNAs. Methods Molecular Biology. 419, 69-91 (2008).
- Slobodin, B., Gerst, J. E. A novel mRNA affinity purification technique for the identification of interacting proteins and transcripts in ribonucleoprotein complexes. RNA. 16 (11), 2277-2290 (2010).
- Slobodin, B., Gerst, J. E. RaPID: an aptamer-based mRNA affinity purification technique for the identification of RNA and protein factors present in ribonucleoprotein complexes. Methods Molecular Biology. 714, 387-406 (2011).
- Yoon, J. H., Gorospe, M. Identification of mRNA-Interacting Factors by MS2-TRAP (MS2-Tagged RNA Affinity Purification). Methods Molecular Biology. 1421, 15-22 (2016).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Blencowe, B. J., Sproat, B. S., Ryder, U., Barabino, S., Lamond, A. I. Antisense probing of the human U4/U6 snRNP with biotinylated 2'-OMe RNA oligonucleotides. Cell. 59 (3), 531-539 (1989).
- Lingner, J., Cech, T. R. Purification of telomerase from Euplotes aediculatus: requirement of a primer 3' overhang. Proceedings of the National Academy of Sciences U S A. 93 (20), 10712-10717 (1996).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Molecular Cell Proteomics. 12 (6), 1539-1552 (2013).
- Imig, J., et al. miR-CLIP capture of a miRNA targetome uncovers a lincRNA H19-miR-106a interaction. Nature Chemical Biology. 11 (2), 107-114 (2015).
- Chu, C., et al. Systematic discovery of Xist RNA binding proteins. Cell. 161 (2), 404-416 (2015).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Rogell, B., et al. Specific RNP capture with antisense LNA/DNA mixmers. RNA. 23 (8), 1290-1302 (2017).
- Matia-Gonzalez, A. M., Iadevaia, V., Gerber, A. P. A versatile tandem RNA isolation procedure to capture in vivo formed mRNA-protein complexes. Methods. , 93-100 (2017).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic Acids Research. 36 (Web Server issue), W70-W74 (2008).
- Kalendar, R., Khassenov, B., Ramankulov, Y., Samuilova, O., Ivanov, K. I. FastPCR: An in silico tool for fast primer and probe design and advanced sequence analysis. Genomics. 109 (3-4), 312-319 (2017).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Kedde, M., et al. A Pumilio-induced RNA structure switch in p27-3' UTR controls miR-221 and miR-222 accessibility. Nature Cell Biology. 12 (10), 1014-1020 (2010).
- Schumacher, B., et al. Translational repression of C. elegans p53 by GLD-1 regulates DNA damage-induced apoptosis. Cell. 120 (3), 357-368 (2005).
- Ziegeler, G., et al. Embryonic lethal abnormal vision-like HuR-dependent mRNA stability regulates post-transcriptional expression of cyclin-dependent kinase inhibitor p27Kip1. Journal of Biological Chemistry. 285 (20), 15408-15419 (2010).
- Itri, F., et al. Femtosecond UV-laser pulses to unveil protein-protein interactions in living cells. Cellular and Molecular Life Sciences. 73 (3), 637-648 (2016).
- Zhang, L., Zhang, K., Prandl, R., Schoffl, F. Detecting DNA-binding of proteins in vivo by UV-crosslinking and immunoprecipitation. Biochemical and Biophysical Research Communications. 322 (3), 705-711 (2004).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır