Method Article
Um procedimento de isolamento de RNA em Tandem do Oligonucleotide-baseado para recuperar complexos mRNA-proteínas eucariotas
Neste Artigo
Resumo
Um procedimento de isolamento de RNA em tandem (viagem) para recuperação de complexos mRNA-proteínas endogenamente formado é descrito. Especificamente, complexos de RNA-proteína são quitosana na vivo, polyadenylated RNAs são isolados de extratos com grânulos de oligo (descolamento) e particulares mRNAs são capturados com modificados oligonucleotides antisentidos do RNA. Proteínas vinculadas a mRNAs são detectadas pela análise de immunoblot.
Resumo
Proteínas de ligação de RNA (RBPs) desempenham papéis-chave no controle pós-transcricional da expressão gênica. Portanto, a caracterização bioquímica de complexos mRNA-proteínas é essencial para a compreensão de regulamento de mRNA inferido pela interação de proteínas ou RNA não-codificante. Aqui, descrevemos um procedimento de isolamento de RNA do tandem (viagem) que permite a purificação dos complexos mRNA-proteínas endogenamente formado de extratos celulares. O protocolo em duas etapas envolve o isolamento do polyadenylated mRNAs com grânulos antisentido oligo (descolamento) e subsequente captura de um mRNA de interesse com biotinilado 3'-2'-O-misturado do RNA oligonucleotides antisentido, que então pode ser isolada com grânulos de estreptavidina. VIAGEM foi usada para recuperar em vivo complexos mRNA-ribonucleoprotein (mRNP) de quitosana de levedura, nematoides e células humanas para posterior análise do RNA e proteína. Assim, a viagem é uma abordagem versátil que pode ser adaptada para todos os tipos de polyadenylated RNAs em organismos para estudar o dinâmico re-arranjo de mRNPs imposta por sinais intracelulares ou ambientais.
Introdução
Regulação gênica pós-transcricional é conduzida através de interações entre proteínas RNA-obrigatórias (RBPs), não-codificantes RNAs (ncRNAs) e mRNAs, que direcionar o processamento, localização, tradução e decaimento de cada transcrição dentro pilhas1 , 2. a identificação do RBPs e ncRNA interagindo com particulares mRNAs, que estabelece o que é conhecido como ribonucleoprotein complexos/partículas (RNPs), é, portanto, a chave para compreender o destino do controle de expressão de mRNAs e gene. Abordagens complementares são realizadas para a caracterização bioquímica da RNP complexos3,4: enquanto a abordagem "centrada em proteína" baseia-se na purificação do RBPs específicos, a abordagem "Centrada no RNA" envolve o isolamento de subpopulações ou RNAs individuais e posterior análise de interação de proteínas ou RNAs. Recentemente, uma abordagem centrada no RNA para a identificação do repertório RBP interagindo com polyadenylated (poly(A)) RNAs5 tornou-se cada vez mais popular, incorporando um passo de reticulação de luz ultravioleta (UV) para estabilizar a proteína-RNA prévia de interações do isolamento de mRNAs, que dramaticamente prorrogado o catálogo da RBPs em células humanas6,7,8,9,10,11, Caenorhabditis elegans 12, saccharomyces cerevisiae12,13,14e outros organismos15,16,17,18, 19,20. No entanto, o mapeamento de proteínas e/ou montado em particulares transcrições de ncRNAs ainda é um grande desafio. Para o efeito, duas principais abordagens são usadas atualmente: por um lado, o RNA aptamer etiquetas são fundidas para o RNA de interesse para permitir a purificação da afinidade. Desse modo, os RNA aptamers ligam com alta afinidade para os antibióticos aminoglicosidos incluindo tobramicina e estreptomicina21,22,23,24, ou proteínas, tais como a proteína do revestimento partir do R17/MS2 bacteriófago ou bacteriana streptavidin S125,26,,27,28,29. Embora essa abordagem foi mostrada para ser relativamente robusto e versátil, ele requer a clonagem para o RNA de design sob investigação e, portanto, não pode ser usado para capturar os mRNAs naturais. Por outro lado, oligonucleotides antisentido (ASOs) foram usados desde o início para a recuperação e caracterização de altamente expresso nativo RNPs30,31 e de RNAs virais32. Mais recentemente, ASOs foram aplicados para capturar o tempo não codificação RNAs33,34,35 e em vitro formou mRNPs36. Um factor limitante principal com todas as estratégias centralizadas de RNA é o número de cópia do RNA de interesse, fazendo com que a recuperação de mRNAs expressos por baixo mais problemático. Esta limitação pode ser superada pelo "upscaling" a reação; no entanto, isto pode potencialmente levar a aumento do plano de fundo.
Aqui, descrevemos nosso tandem recentemente desenvolvido baseados em ASO procedimento de isolamento de RNA (viagem) para isolar complexos mRNA-proteínas nativas com alta seletividade37 (Figura 1). O protocolo envolve duas etapas de purificação baseada em ASO sequencial, ou seja o isolamento dos mRNAs poli com oligo (descolamento) comercialmente disponível juntamente grânulos seguidos pela captura de mRNAs específicos usando especificamente projetado biotinilado curto 2'-metoxi ASOs de RNA. Este procedimento de duas etapas ajuda a eliminar as proteínas contaminantes e adiciona oportunidades para ajuste e otimização. Nesta instância, viagem foi aplicada na mRNAs selecionados do fermento, nemátodos, e formaram de células humanas para confirmar na vivo complexos mRNA-proteínas.
Protocolo
1. projeto Oligonucleotides antisentido (ASOs)
- Analise a estrutura secundária do mRNA ou um fragmento usando suas ferramentas disponíveis on-line38. Portanto, digite a sequência de nucleotídeos (nt) na caixa vazia. Na caixa de opções básicas, seleccione mínimo a energia livre (MFE) e função de partição e evitar pares de bases isoladas. Na caixa de opções de saída, selecione a trama de estrutura secundária do RNA interativo e finalmente clique no botão seguir . Uma nova janela aparecerá que exibe a estrutura secundária do mRNA do interesse.
- Selecione pelo menos três diferentes 21-24 nts longas sequências dentro do mRNA do interesse, preferencialmente nas regiões falta detectáveis estruturas secundárias (ex., em loops não estruturados) e em 3' não traduzidas regiões (UTR).
Nota: Tem sido observado que a ASOs recozimento para sequências nas regiões 3' não (UTR) melhor realizada. Uma possibilidade é que ribossomos ligados à sequência de codificação (CDS) ocasionalmente podem obstruir o recozimento da ASOs. ASOs recozimento para 5' UTRs não foram testados.- Selecione as regiões com um rácio de guanidina/citosina cerca de 50% e falta em tandem do nucleotide repete para evitar a potencial formação de grampos ou self recozimento.
- Manualmente o projeto 2'-metoxi modificado oligonucleotides RNA tendo um moiety biotina na 3', totalmente complementares para as regiões selecionadas (etapa 1.2) dentro do desejado alvo do mRNA.
Nota: 2'-metoxi modificações processar o RNA resistente para celular RNases e aumenta o duplex temperatura, que permite a lavagem rigorosa de fusão. A fracção de biotina é necessária para capturar ASOs com estreptavidina.- Ajustar a temperatura de fusão dos híbridos RNA ~ 60-65 ° C e certifique-se de que tem uma complexidade alta sequência linguística (> 60%) conforme determinado com ferramentas on-line apropriado39.
- Use a ferramenta de busca Local básica de alinhamento40 para procurar potenciais ASO Cruz-hibridação com outros mRNAs no transcriptoma. Selecione o Nucleotide BLAST, insira as sequências na caixa vazia e o organismo de interesse. Manter os parâmetros restantes como padrão e clique em explosão.
Nota: O alinhamento contínuo mesmo parcial de 8-10 nts pode levar a Cruz-hibridação e recuperação deste mRNA.
2. cultura, irradiação UV e célula de pilha Lysis
Nota: A seguir, o procedimento é descrito por brotamento levedura S. cerevisiae, nemátodo c. eleganse células de rim humano embrionário (HEK293). No entanto, pode ser adaptado para outros organismos, bem como, apesar de que não foi explicitamente testado ainda.
- S. cerevisiae
- Desenvolvem-se células de levedura (EG., estirpe BY4743 derivado; MATa/α his3Δ1/his3Δ1 leu2Δ0/leu2Δ0 LYS2/lys2Δ0 met15Δ0/MET15 ura3Δ0/ura3Δ0 PFK2:TAP / PFK2) em 500 mL de mídia YPD (extrato de levedura 1%, 2% de peptona, 2% D-glicose) a 30 ° C, com agitação constante a 220 rpm.
- Coletar as células na fase log de mid (densidade óptica em 600 nm (OD)600 ~ 0.6) por filtração utilizando filtros de nylon 0,45 µm ou por centrifugação a 3.000 x g por 2 min à temperatura ambiente (RT). Descarte o escoamento ou o sobrenadante, respectivamente.
- Lavar as células três vezes com 25 mL de fosfato--salino (PBS) usando o filtro de nylon 0,45 µm ou recolher por centrifugação a 3.000 x g por 2 min em RT e descartar o sobrenadante. Colete as células tão rapidamente quanto possível, o estresse imposto poderia ter um impacto sobre interações de RNA-proteína.
- Ressuspender as células em 25 mL de PBS e em seguida, despeje a suspensão em uma placa de Petri de 15 cm. Coloque o prato no gelo, retire a tampa e expor as células três vezes com 400 mJ cm-2 de luz de UV 254 nm em um UV-crosslinker com intervalos de 2 min no gelo entre cada ciclo de exposição com uma mistura suave.
- Transfira as células para um tubo de 50 mL e coletar as células por centrifugação a 3.000 x g por 3 min a 4 ° C. Desprezar o sobrenadante e manter a pelota.
Nota: As células podem ser snap congelado em nitrogênio líquido e armazenado a-80 ° C. - Ressuspender as células em 4 mL de tampão de Lise refrigerados A (LB-A; 100 mM Tris-HCl, pH 7,5, 500mm LiCl, 10 mM de EDTA, 1% Triton X-100, 5 mM DTT, U 20 ml-1 , U 100 ml-1 RNasin, completar coquetel de inibidor de protease EDTA livre de DNase).
- Transferi as células para dois tubos de 2 mL. Adicione o max. Volumes ⅔ de refrigerados grânulos de vidro e perturbam as células em um lyser tecido a 30 Hz durante 10 minutos a 4 ° C.
- Um buraco no fundo do tubo com uma agulha quente e transfira o lisado para um tubo 1,5 mL por centrifugação a 600 × g por 30 s.
- Limpe o lisado por três sequenciais centrifugações a 4 ° C, a 3.000 × g por 3 min, em seguida, 5.000 × g e 10.000 × g por 5 min cada.
Nota: Extractos podem ser snap-congelado em nitrogênio líquido e armazenados a-80 ° C.
- C. elegans
- Vermes de Bristol N2 cultura a 20 ° C em placas de nematódeo crescimento médio (NGM) (0,3% de NaCl, ágar de 1,7%, 0,25% de peptona, 1 mM CaCl2, colesterol 5 µ g/mL, 1 mM de MgSO4, 25mm KPO4 tampão, pH 6.0) inoculadas com a estirpe OP50 Escherichia coli 41 .
- Adicionar 5 mL de M9 reserva41 (0,3% KH2PO4, 0,6% Na2HPO4, 0,5% de NaCl, 1 mM de MgSO4) para cada placa, agitar a placa para Ressuspender as minhocas e transferir os vermes para um tubo de 15 mL. Coloque 2 µ l da suspensão em um slide e contar os vermes na suspensão com um microscópio. Em seguida, aplica o fator para calcular o número de vermes por placa.
- Coletar ~ 120.000 vermes por centrifugação à temperatura ambiente (400 × g, 2 min, RT) e descartar o sobrenadante.
- Lave os vermes três vezes. Portanto, adicionar 10 mL de tampão de M9, misturar por inversão e coletar os vermes por centrifugação a 400 × g por 2 min em RT
- Adicionar 15 mL de tampão de M9 para as minhocas e coloque sobre uma roda rotatórios durante 15 min à RT
- Transferir os vermes para placas NGM (~ 4.000 vermes por placa) e expor à luz UV (254 nm) em 300 mJ cm-2 em um UV-crosslinker.
- Adicionar 5 mL de tampão de M9 diretamente à placa e agitar a placa para Ressuspender os vermes no buffer. A suspensão com uma pipeta de transferência para um tubo de 15 mL e coletar os vermes por centrifugação a 400 × g por 2 min em RT
- Resuspenda as minhocas em 2 mL de tampão de Lise B (LB-B; 100 mM Tris-HCl, pH 8.0, 150 mM de NaCl, 1 mM EDTA, 0,75% IGEPAL, 1 milímetro DTT, U 20 mL-1 , U 100 mL-1 RNasin, completar coquetel de inibidor de protease EDTA livre de DNase).
- Moa as minhocas num almofariz preenchidos com nitrogênio líquido. Recolher o pó em um tubo de 50 mL e degelo no RT
Nota: O nitrogênio líquido deve ser manuseado de acordo com os procedimentos de segurança (por exemplo, das emanações hood e segurança luvas) - Limpar o lisado, incluindo a camada de gordura por centrifugação a 14.000 × g por 10 min e posteriormente passar a esclareceu lisado através de um filtro de 0,45 μm com uma seringa.
- Culturas de células humanas
Nota: A seguinte descrição baseia-se na transitória transfecção de células HEK293 com pGL3-CDKN1B-3'UTR repórter do que expressa a 3′UTR de CDKN1B/27 a jusante a luciferase de vaga-lume gene42.- As células HEK293 de cultura em modificado águia de Dulbecco médio (DMEM) contendo 25 mM glicose e piruvato de sódio de 1 mM, suplementado com 100 U o penicilina de mL-1 , o 100 µ g mL-1 estreptomicina e a 10% de soro fetal bovino (FBS) e incubar a 37 ° C, em um umidificado câmara (incubadora) contendo 5% de CO2.
- Semente ~ 3 × 106 HEK293 células em cultura de tecido padrão de 10 cm do prato no dia anterior o transfeccao. Conte as células com um hemocytometer.
- Misture 2 µ g do gene repórter (EG., pGL3-p27-3'UTR) com 20 µ l de reagente de transfeccao e transfect as células na confluência de 70% (~ 7 × 106 células).
- Coloca as células em uma incubadora a 37 ° C por mais de 48 h antes da colheita.
- Remover o meio de uma pipeta sorológica e lavar rapidamente as células duas vezes com 10 mL de PBS pré aquecido a 37 ° C. Remova o PBS com uma pipeta sorológica.
- Adicionar 6 mL de PBS para o prato, coloque no gelo e em seguida, expor as células à luz UV (254 nm) em 100 mJ cm-2 em um UV-crosslinker.
Nota: A placa deve ser mantida no gelo durante a exposição de luz UV. - Raspar as células (células de7 ~ 10 total) em PBS e transferir para um tubo de 15 mL.
- Gire para baixo as células a 250 × g por 10 min a 4 ° C e em seguida, remover o sobrenadante com uma pipeta.
Nota: O centrifugado pode ser snap-congelado em nitrogênio líquido e armazenado a-80 ° C até o uso. - Ressuspender as células em 2 mL de tampão de Lise pre-refrigerados (LB-A) pipetando para cima e para baixo para 5 - 6 vezes, mantendo o tubo no gelo.
- Transfira o lisado com uma pipeta para um tubo de 5 mL colocado no gelo.
- Sujeitar o lisado de três rodadas de sonication consistindo de 20 s rajadas em 10 μm amplitude com 30 s de resfriamento períodos no gelo.
Nota: Sonication recomenda-se que ele conclui a Lise das células e fragmentos de DNA. - Transfira o lisado para tubos de 2 mL e centrifugar 15.000 × g por 10 min a 4 ° C. Recolher o sobrenadante (= extrair) e transferir para um tubo novo. Remova uma amostra para análise (5-10%) para análise do RNA e mais proteína.
Nota: Este passo é fundamental para remover os detritos de célula restante e pode ser repetido.
3. primeiro passo: Isolamento de RNA poli (a)
- Equilibrar a 1 mg de oligo (descolamento)25-acoplado grânulos magnéticos em 500 µ l do respectivo Lise.
- Combine 5 mg, 10 mg ou 4 mg (proteína) de S. cerevisiae, c. elegans ou extractos HEK293, respectivamente, com 1 mg de oligo (descolamento)25 -acoplado grânulos magnéticos. Misture as amostras vigorosamente por 10 min a 25 ° C, em um misturador.
Nota: As concentrações de proteína dos extratos podem ser determinadas com ensaio de Bradford usando albumina de soro bovino (BSA) como um padrão de referência. Agitação vigorosa de amostras previne sedimentação dos grânulos. Para avaliar a especificidade do isolamento do mRNA, um experimento de controle podem ser executado em paralelo com a adição de um excesso (20 µ g) de ácidos polyadenylic (pA). - Coloque os tubos num suporte magnético para 10 s e remover o sobrenadante. Manter o sobrenadante no gelo para rondas subsequentes de recuperação (etapa 3.7).
- Adicionar 500 µ l de lavagem tampão A (10 mM Tris-HCl, pH 7,5, 600mm LiCl, 1 mM EDTA, 0,1% Triton X-100) para as contas e vórtice para 5 s.
Nota: A concentração de LiCl nos buffers de lavagem pode variar. 600 mM LiCl é recomendado, mas concentrações inferiores também foram testadas (500 mM para c. elegans, 300 mM para HEK293). - Recolher os grânulos com um imã e em seguida lave os grânulos duas vezes com 500 µ l de tampão de lavagem B (WB; 10 mM Tris-HCl, pH 7,5, 600mm LiCl, 1 mM EDTA).
- Eluir o RNA em 30 µ l de 10 mM Tris-HCl, pH 7,5 a 80 ° C por 2 min com agitação contínua (1.000 rpm) em um misturador. Imediatamente, coloque o tubo no suporte magnético e recolher o eluato após 10 s. Salve os grânulos para rodadas adicionais de purificação.
Nota: Recolha o eluato mais rapidamente possível para evitar potenciais de religação dos mRNAs para os grânulos em temperaturas mais baixas. - Adicionar os grânulos da etapa anterior para o sobrenadante coletado na etapa 3.3 e repetir a captura, a lavagem e eluição dos mRNAs duas vezes (passos 3.3-3.6). Os eluídos de repetidas rodadas são então combinados e podem ser armazenados a-80 ° C.
4. segunda etapa: captura de específico mRNAs com biotinilado 3'-2'-metoxi RNA antisenso modificada Oligonucleotides
Nota: Para testar a adequação da ASOs, o procedimento a seguir pode ser executado com totais RNAs isolados de células.
- Equilibrar a 30 µ l de grânulos magnéticos streptavidin-acoplado em 1 mL de vinculação e lavar buffer (buffer de B & W; 10 mM Tris-HCl, pH 7,5, 150 mM NaCl, 0.5 mM EDTA, pH 8.0) contendo 0,1 mg mL-1Escherichia coli RNA de transferência (tRNA) em rotacao por 1h no RT.
- Lave os grânulos três vezes com 750 µ l de tampão B & W.
- Resuspenda as contas em 30 µ l de tampão B & W e manter no gelo até o uso.
- Diluir ~ 35 µ g de proteína total do isolamento poli anterior (ver 3) em 100 µ l de tampão B & W em um tubo de 1,5 mL.
Nota: Para testar a especificidade da ASOs com RNA total, adicionar 600 ng do RNA total purificado a 100 µ l de tampão B & W. - Adicionar 200 pmol do respectivo ASO e incubar a 70 ° C por 5 min.
Nota: A temperatura elevada resolve estruturas secundárias no ARN facilitando o recozimento de ASO. - Retire todo o calor-bloco do dispositivo e colocá-lo na RT para 10 min refrigerar para baixo lentamente.
- Adicione 30 µ l de equilibrada grânulos magnéticos streptavidin-acoplado da etapa 4.1 para a amostra.
- Incube a mistura por 30 min a 25 ° C, com agitação constante a 950 rpm em um misturador.
Nota: Recomenda-se agite os tubos a cada 10 min para evitar a sedimentação dos grânulos. - Coloque os tubos no stand magnético, remover o sobrenadante e lave os grânulos três vezes com 750 µ l de tampão B & W pré-aquecido a 55 ° C.
Nota: A temperatura de lavagem ideal ligeiramente diferentes/variam entre diferentes ASOs. Recomenda-se ajustar a esta condição com prévio de RNA total não-quitosana realizando o experimento com extratos de células. - Eluir o RNA em 20 µ l de 10 mM Tris-HCl, pH 7,5 a 90 ° C por 10 min com agitação constante a 950 rpm em um misturador. Coloque os tubos no suporte magnético e imediatamente recolher o eluato.
Nota: Para análise de proteínas, adicionar 20 µ l de tampão de Laemmli × 1 para os grânulos e incubar a 95 ° C por 5 min. proteínas são monitoradas pela análise de immunoblot seguindo protocolos padrão de laboratório.
Resultados
Desenvolvemos uma estratégia de isolamento de RNA baseado no ASO, denominada viagem, para capturar os mRNAs particulares com suas proteínas limite de três organismos diferentes37. Essencialmente, complexos de RNA-proteína foram quitosana na vivo por UV-irradiação de células em 254 nm e poli RNAs foram recuperados com grânulos magnéticos acoplados comercialmente disponível oligo (dT) e, em seguida, o mRNA do interesse foi isolado com 3'-biotinilado 2-0'-metoxi modificado oligonucleotides antisentido de RNA (Figura 1). Nós, portanto, desenvolvido vários 21-24 nts modificado ASOs com cheio-complementaridade para regiões nos mRNAs selecionados de levedura, c. elegans e humanos e testado sua adequação para recuperar o mRNA do interesse (uma lista de primers e ASOs é dado na tabela de 1). a eficiência e a especificidade da ASOs individuais foram avaliados primeiro com ligação cruzada não RNA total isolado do respectivo organismo. Nesses experimentos, RNA ASOs foram acopladas a estreptavidina conjugada paramagnéticos grânulos e incubadas com o RNA total não-quitosana, preparado a partir do organismo correspondente. Após a liberação de mRNAs capturados de grânulos, a presença de mRNA alvo, bem como independente controle mRNAs foi monitorado por transcrição reversa (RT)-polimerase reação em cadeia (PCR)37. Notamos que duas variáveis, a concentração de sal e a temperatura de lavagem buffers, desempenhado papéis importantes na eficiência da captura de mRNA PFK2 do fermento que foi testado com três diferentes ASOs (Figura 2A). Diminuindo a concentração de sal (NaCl) a 25 mM reduzido a recuperação do negativo controlar mRNA (PFK1) para níveis não-detectáveis com todas ASOs, mas também reduziu a recuperação do desejado alvo mRNA PFK2 (10-15% da entrada). Por outro lado, um aumento das concentrações sal para níveis fisiológicos (150 mM NaCl) aumentou a recuperação de mRNAs PFK2 até 75% com o ASO PFK2-2, superior do controle PFK1 mRNA pelo menos 5-fold (Figura 2A). Ainda mais notável, as ASOs diferentes mostraram grande variação de mRNA eficácias de captura do alvo em sal-concentrações fisiológicas, enfatizando a necessidade de validação empírica da ASOs. A dependência da recuperação do mRNA na temperatura do tampão de lavagem é exemplificada por cep-1 do c. elegans e levedura RPS20 mRNAs, utilizando os respectivos ASOs (tabela 1). Observamos que a temperatura de lavagem ideal foi entre 50 ° C e 55 ° C, como é evidente do fundo baixo com mRNAs independentes e a recuperação eficiente de mRNAs alvo (Figura 2B). Neste momento, gostaríamos de enfatizar a possibilidade da Cruz-hibridação da ASOs com outros mRNAs. Por exemplo, a ASO DNM1 é totalmente complementar uma sequência dentro o DNM1 sequência de código, mas é também parcialmente anneals com ACT1 mRNA. ASOs DNM1 recuperado ambos os mRNAs independentemente da temperatura de lavagem, mostrando forte propensão para a Cruz-hibridação (Figura 2). Finalmente, nós usamos os testes descritos acima e otimizações para selecionar três ASOs que foram adequados para a recuperação do alvo respectivos mRNAs de total RNA isolado de S. cerevisiae, c. elegans e células de origem humanas (Figura 2D ).
Após a seleção inicial de adequado ASOs in-vitro, realizamos a viagem com célula de extratos obtidos de UV-quitosana organismos/células (Figura 1). Especificamente, nós testamos a recuperação de três diferentes mRNAs de três organismos diferentes: PFK2 mRNA de levedura S. cerevisiae, cep-1 desde o nemátodo c. elegans e um repórter mRNA (pGL3-CDKN1B-3'UTR) tendo o 3' UTR sequência de mRNA de CDKN1B/p27 humano fundido a um repórter do luciferase (luc) para expressão transiente em células HEK293 humanas. Para controlar a especificidade do isolamento de RNA, temos monitorado a recuperação de vários mRNAs independentes, e realizamos experimentos de competição pela adição de um excesso de pA para extratos de células, que compete com a ligação do celulares mRNAs de oligo (descolamento)25 grânulos durante o primeiro passo a passo de purificação. Como visto anteriormente com ligação cruzada não amostras do RNA totais (Figura 2), RT-PCR confirmou o enriquecimento dos respectivos mRNAs direcionar mRNA durante a viagem, Considerando que vários mRNAs não relacionados ao controle não foram enriquecidos (Figura 3A). Além disso, nenhum dos mRNAs foram detectados em grânulos sem ASOs, indicando procedimentos adequados de bloqueio que evitar a vinculação inespecíficas. Nesta linha, ASOs independentes/mexidos podem também ser usados como controle, embora o potencial de cruz-hibridação com certos mRNAs e captura inferida de proteínas acopladas tem então de ser tidos em conta. Desde que proteínas no eluato final poderiam ser dificilmente visualizadas em gel de poliacrilamida prata manchada (dados não mostrados), a presença de proteínas de interação de mRNA anteriormente conhecidas mais foi avaliada por análise de immunoblot. Isso inclui Pfk2p de S. cerevisiae, que se liga seletivamente aos PFK2 mRNA em uma forma independente de Ribossoma12; GLD-1 c. elegans , um RBP canônico que vincula a 3' UTR as sequências de cep-1 mRNA para regulamento translacional43; e HuR, um RBP que regula a estabilidade do mRNA e tradução do RNAm de p27/CDKN1B 44. Como esperado, todas estas proteínas foram identificadas nos eluídos viagem dos respectivos mRNAs por análise de Western Blot (Figura 3B).
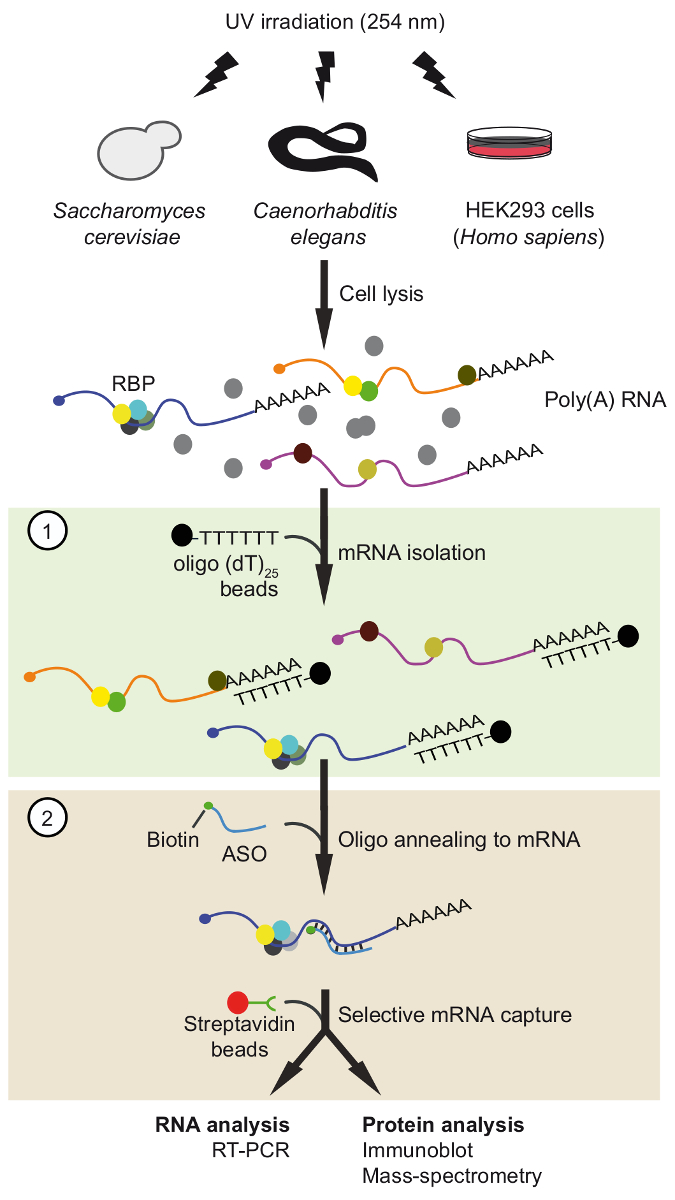
Figura 1. Representação esquemática da viagem. As proteínas são quitosana para RNA na vivo por irradiação UV. Na primeira etapa (caixa de luz verde), complexos poli RNA-proteína são recuperados com grânulos de25 oligo (descolamento) aplicando condições rigorosas de lavagem para remover as proteínas unbound. Na segunda etapa (caixa-de-rosa), mRNP o alvo é puxado-para fora com oligonucleotídeos de RNA antisenso biotinilado e grânulos de estreptavidina. Os mRNPs purificadas são então analisados por RT-PCR e immunoblot /-espectrometria de massa (MS) para identificar RNAs e proteínas que interagem com o mRNA do interesse, respectivamente. A figura foi modificada da anterior publicação37 com permissão. Clique aqui para ver uma versão maior desta figura.
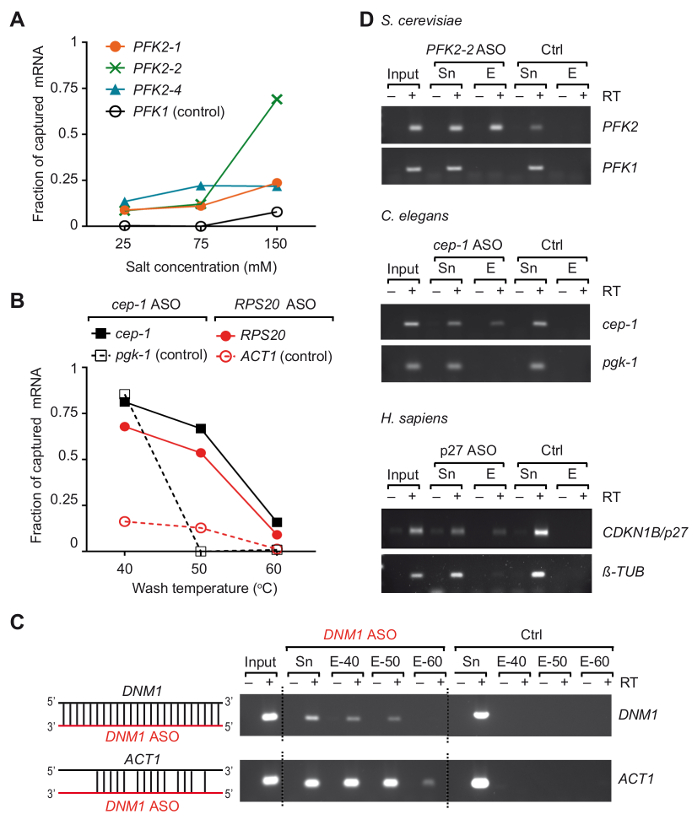
Figura 2. Isolamento de mRNAs selecionados de RNA total de não-quitosana células/organismos usando modificado antisentido RNA captura sondas. (A) impacto de concentrações de sal sobre a eficiência de captura da levedura PFK2 mRNA com ASOs. O RNA total de células de levedura foi combinado com as ASOs indicados e lavado com tampão contendo a especificado concentração de sal (NaCl) a 55 ° C. Verde, azuis e laranja linhas representam PFK2 recuperação de mRNA com diferentes ASOs conforme determinado por RT-PCR37: PFK2-1 e PFK2 -2 anneal na 3' UTR, PFK2-4 em CDS. PFK1 é um negativo controlar mRNA. PCR foi executado com 32 ciclos de amplificação e quantificado como descrito anteriormente,37. (B) fração de c. eleganscep-1 e leveduras RPS20 ASO vinculado mRNAs em temperaturas diferentes de lavagem, representadas nas linhas de pretas e vermelhas, respectivamente. C. eleganspgk-1 e levedura ACT1 são mRNAs não-alvo (controle). 30 e 32 ciclos PCR foram aplicados para detecção de levedura e mRNAs de c. elegans , respectivamente. (C) representação esquemática da hibridação de DNM1 ASO (vermelho) com sequências no DNM1 mRNA, bem como o potencial da Cruz-hibridação com ACT1 mRNA é mostrada à esquerda. Um gel de agarose mostrando produtos de reações de RT-PCR (30 ciclos) para a detecção de leveduras DNM1 e ACT1 mRNAs eluídos de missanga é mostrado à direita. Entrada, o RNA total; Sn, sobrenadante após incubação com ASOs; E, eluídos de missanga lavadas indicaram eluição prévia de temperaturas (40 ° C, 50 ° C e 60 ° C). Um experimento de controle (Ctrl) foi realizado em paralelo sem adição de ASO. (D) gel do Agarose mostrando produtos de RT-PCR para a detecção de mRNAs (à direita) capturados do RNA total de levedura S. cerevisiae, c. eleganse células HEK293 humanas (H. sapiens). Entrada, o RNA total; Sn, sobrenadante; E, eluídos de grânulos. PCR foi realizado por 30 ciclos de amplificação para levedura mRNAs, 32 ciclos de mRNAs de c. elegans , e 28 e 30 ciclos para humano tubulina e p27 mRNAs, respectivamente. A figura foi modificada da anterior publicação37 com permissão. Clique aqui para ver uma versão maior desta figura.
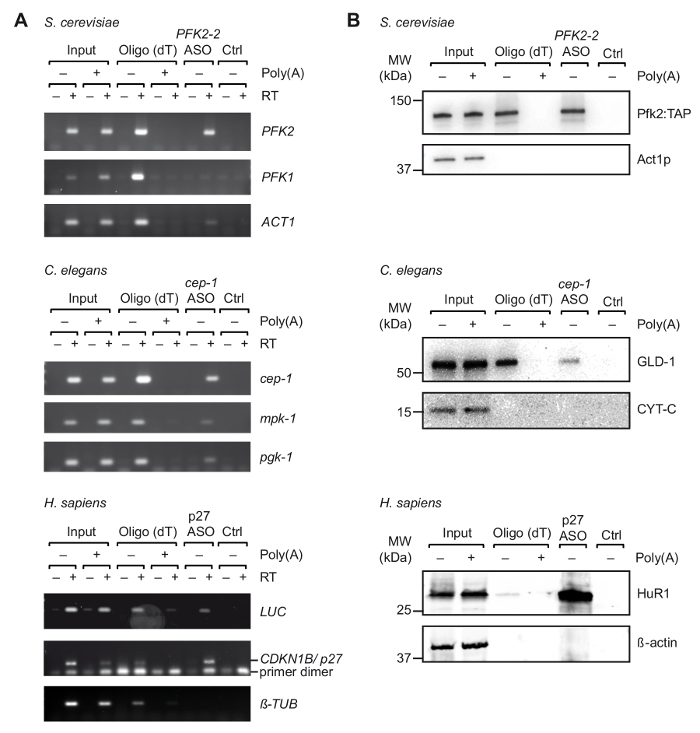
Figura 3. Captura de complexos mRNA-proteínas específicas dos extratos de derivadas de células de quitosana UV com viagem. Gel de Agarose (A) para a detecção de mRNAs (à direita) com RT-PCR após captura com oligo (descolamento) e ASOs indicados de S. cerevisiae, c. elegans e extractos de célula humanos (H. sapiens). Entrada, o RNA total de células/organismos de ligação cruzada; CTRL, controle sem ASO. Poli, competição com poli (a). RT-PCR foi realizada como descrito37 com 35 ciclos de amplificação para LUC, 32 ciclos para p27, e os ciclos de 29 para tubulina. (B) análise de Immunoblot de mRNA-limite de proteínas com anticorpos indicados (à direita). Carregado de frações são as seguintes: 0,1%, 2,5% e 1% de fermento, entradas de nematoides e humanas; 10%, 10% e 5% para o fermento, pistas de oligo (descolamento) nematoides e humana; e 66% para todas as faixas da ASOs. Pesos moleculares (MW) são indicados em kilodaltons (kDa). Figura republicada com permissão37. Clique aqui para ver uma versão maior desta figura.
| Nome da primeira demão | Sequência de | Alvo | Tamanho | |
| Pfk2_Fwd | GTGTTAAGGGTTCACATGTCG | PFK2 S. cerevisiae | 133 bp | |
| Pfk2_Rev | CTTCCAACCAAATGGTCAGC | PFK2 S. cerevisiae | 133 bp | |
| Pfk1_Fwd | GGTGATTCTCCAGGTATGAATG | PFK1 S. cerevisiae | 97 bp | |
| Pfk1_Rev | CTTCGTAACCTTCGTAAACAGC | PFK1 S. cerevisiae | 97 bp | |
| Act1_Fwd | GTCTGGATTGGTGGTTCTATC | Act1 S. cerevisiae | 85 bp | |
| Act1_Rev | GGACCACTTTCGTCGTATTC | Act1 S. cerevisiae | 85 bp | |
| Dnm1_Fwd | CTGTGTTCGATGCATCAGAC | DNM1 S. cerevisiae | 156 bp | |
| Dnm1_Rev | CGCACTCCAATTCTTCTCTC | DNM1 S. cerevisiae | 156 bp | |
| Rps20_Fwd | CGCTGAACAACACAACTTGG | RPS20 S. cerevisiae | 228 bp | |
| Rps20_Rev | GGAAGCAACAACAACTTCGAC | RPS20 S. cerevisiae | 228 bp | |
| Cep1_Fwd | CGATGAAGAGAAGTCGCTGT | CEP-1 c. elegans | 110 bp | |
| Cep1_Rev | ATCTGGGAACTTTTGCTTCG | CEP-1 c. elegans | 110 bp | |
| Pgk1_Fwd | GCGATATTTATGTCAATGATGCTTTC | PGK-1 c. elegans | 74 bp | |
| Pgk1_Rev | TGAGTGCTCGACTCCAACCA | PGK-1 c. elegans | 74 bp | |
| Mpk1_Fwd | TGCTCAGTAATCGGCCATTG | MPK-1 c. elegans | 74 bp | |
| Mpk1_Rev | TCCAACAACTGCCAAAATCAAA | MPK-1 c. elegans | 74 bp | |
| p27_Fwd | TTTAAAAATACATATCGCTGACTTCATGG | P27 H. sapiens | 212 bp | |
| p27_Rev | CAAAGTTTATGTGCTACATAAAAGGTAAAAA | P27 H. sapiens | 212 bp | |
| Luc_Fwd | AATGGCTCATATCGCTCCTGGAT | Luciferase pγralis p. | 117 bp | |
| Luc_Rev | TGGACGATGGCCTTGATCTTGTCT | Luciferase pγralis p. | 117 bp | |
| Β-TUBULIN_Fwd | CTGAACCACCTTGTCTCAGC | Β-TUBULINA H. sapiens | 136 bp | |
| Β-TUBULIN_Rev | AGCCAGGCATAAAGAAATGG | Β-TUBULINA H. sapiens | 136 bp | |
| ASO PFK2-1 | AAUAGAAAGUGUAAUAAAAGGUCAU | 3' UTR PFK2 S. cerevisiae | - | |
| ASO PFK2-2 | GUUUCAUGGGGUAGUACUUGU | 3' UTR PFK2 S. cerevisiae | - | |
| ASO PFK2-4 | CUUGAAGAGGAGCGUUCAUA | ORF PFK2 S. cerevisiae | - | |
| DNM1 ASO | UCGGUCAGUGGAGGUUCAGCGUUU | ORF DNM1 S. cerevisiae | - | |
| RPS20 ASO | GUCGGUAAUAGCCUUCUCAUUCUUG | ORF RPS20 S. cerevisiae | - | |
| CEP-1 ASO | GUGAGAAAUGCGGUGCUUUGAAA | 3' UTR cep-1 c. elegans | - | |
| P27 ASO | UCAUACCCCGCUCCACGUCAGUU | 3' UTR p27 H. sapiens | - | |
Tabela 1. Sequências do oligonucleotide. Lista de iniciadores de PCR e ASOs usadas neste trabalho, sequência da primeira demão, alvo de gene e tamanho do fragmento esperado após amplificação.
Discussão
VIAGEM permite a análise de proteínas vinculada de mRNAs específicos na vivo com meios bioquímicos. Enquanto usamos o método para validar a interação do RBPs particulares com alvos de mRNA de diferentes organismos/células, viagem também pode ser aplicável para estudar outros tipos de poli RNAs, tais como citoplasmática ncRNAs longo. Além disso, a análise sistemática de proteínas acopladas e/ou RNAs com sequenciamento MS ou RNA poderia ser alcançado por uma extrapolação do procedimento. A este respeito, nossos dados preliminares indicam que 1 L de culturas de leveduras e pelo menos 100 milhões de células HEK293 humanas poderiam fornecer matérias-primas suficientes obter dados fiáveis de MS para mRNAs bem expressos (resultados inéditos).
O protocolo descrito de viagem inclui a irradiação de células com luz UV de 254 nm para interações crosslink RNA-proteína. Isto permite a implementação de condições rigorosas de lavagem durante a purificação. No entanto, embora não explicitamente testado, desejamos notar que reticulação é opcional e RNPs ativos também podem ser recuperados com buffers fisiológicas. Todavia, neste caso, o potencial re-arranjo das proteínas e/ou RNAs no alvo do RNA em lysates deve ser tida em conta. Por outro lado, se UV ou outros procedimentos de reticulação são aplicada (por exemplo, formaldeído), a integridade do RNA deve ser avaliada como degradação de RNA poderia levar a recuperação diminuiu de mRNPs. Além disso, UV-reticulação é bastante ineficiente (~ 5%) e ainda menos então para proteínas interagindo com double-stranded do RNA, que poderia apresentar viés para a detecção de determinadas classes de RBPs12. Finalmente, a irradiação UV também pode induzir as referências cruzadas da proteína-proteína e proteína-ADN que devem ser consideradas para dados interpretação45,46. Portanto, procedimentos alternativos de reticulação, por exemplo com formaldeído, também podem ser de particular interesse para capturar o maiores conjuntos de proteína na mRNAs.
Uma característica chave da viagem é o procedimento de purificação baseada em ASO para recuperar RNAs particulares, aumentando assim a seletividade e diminuindo a probabilidade de purificação co contaminantes Two-Step. Por exemplo, uma preocupação refere-se à adição de ASOs biotinilado diretamente a extratos de células, que poderiam levar a co purificação de proteínas de ligação de biotina. Isso é contornado em viagem através da implementação de uma primeira rodada de purificação de RNAs poli usando covalentemente acoplado oligo-(dT)25 grânulos magnético, que permite a condições rigorosas de purificação como feito para captura de interactome RNA-proteína. Além disso, a seleção de RNA poli (a) remove ncRNAs altamente abundantes, tais como o RNA ribossomal e tRNAs e, portanto, reduz a complexidade da amostra e fontes potenciais para Cruz-hibridação e contaminação. Na segunda etapa, particulares mRNAs são recuperados com biotinilado e modificado ASOs que recozem com regiões sobre os alvos do mRNA. Alternativamente, ASOs modificados poderiam também ser diretamente ligado covalentemente ligados à grânulos magnéticos através de uma amina-linker, reduzindo a propensão para capturar proteínas biotina. No entanto, em nossa experiência, a incubação da fração de RNA poli (a) com ASOs antes da adição de grânulos streptavidin recuperado mRNPs mais eficientemente do que a adição direta de grânulos ASO-acoplado. Possivelmente, oligos livre cineticamente são favorecidos com melhor acesso para sequências no ARN estruturado em comparação com os oligos imobilizados. Daí, o acoplamento covalente da ASOs talões antes da incubação com a amostra poderia ser prejudicial para a recuperação do alvo do RNA e tem que ser testado empiricamente.
Um inconveniente com métodos ASO baseado é ineficiente recuperação de alguns mRNAs de alvo. Recomendamos, portanto, a avaliação de vários ASOs que recozem para diferentes regiões da transcrição. Especificamente, nós sugerimos que o projeto de 2-3 ASOs que recozem com sequências no 3'-UTR ou os CDS do mRNA do interesse. Cada ASO podem apresentar uma eficiência diferente para a recuperação do respectivo destino mRNA e deve ser empiricamente testadas (Figura 2A, B, dados não mostrados). No final, nós encontramos que ASOs recozimento para os 3' UTRs tiveram melhor desempenho em comparação com os de recozimento para CDS. Isto pode ser devido à maior acessibilidade de ligação locais em UTRs devido à ausência de traduzir ribossomas, Considerando que os ribossomos podem obstruir hibridização eficiente dentro de CDS. Também tivemos que combinação de vários ASOs poderia levar à contaminação de aumentada de mRNAs abundantes do não-alvo e redução na eficiência de suspenso. Em qualquer caso, a seletividade de oligos o escolhido pode ser controlada pelos experimentos de competição (EG., adição de oligos concorrentes).
Mais preocupação diz respeito ao potencial Cruz-hibridação com RNAs não relacionados, como observamos que mesmo parcial da hibridação com outros mRNAs pode levar à recuperação do que mRNA (Figura 2). Para avaliar a adequação da ASOs projetado, portanto, recomendamos executar inicial em vitro testes com totais RNAs para otimizar as concentrações de sal e temperatura de lavagem de buffers. Em nossas mãos, lavagem de grânulos magnéticos streptavidin-acoplado em buffers contendo 150 mM NaCl a 55 ° C executada melhor, mas recomendamos testes independentes das condições de cada ASO projetado. Notável, achamos que todas ASOs, selecionados a partir de experimentos in vitro (Figura 2D) foram apropriadas para capturar o respectivo alvo de mRNA de extratos de células de ligação cruzada (Figura 3), justificando ainda mais a validade da em Vitro testes. Além de projetar ASOs adequados para captura de mRNA, fatores adicionais poderiam impactar na seletividade. Portanto, procedimentos de bloqueio abrangentes com BSA e/ou tRNA de acordo com as especificações de tipo de grânulo podem impedir a ligação inespecíficas de grânulos. Para reduzir ainda mais inespecíficas absorção de proteínas, também recomendamos o uso de tubos de ligação-Lo, especialmente quando se trabalha com pequenas quantidades de amostras.
Geralmente, a viagem pode complementar abordagens previamente estabelecidas para capturar RNAs com ASOs. Por exemplo, o RNA não-codificante Xist foi isolado com limite proteínas de extratos nucleares derivados de células de quitosana UV 200 milhões por meio de uma matriz de tempo (90-mer) biotinilado DNA os oligonucleotides que cobriu a transcrição inteira 34,35. No entanto, a abordagem escolhida a telha pode se tornar problemática, tendo em conta o grande potencial para a Cruz-hibridação com RNAs independentes, e não pode ser usado para selecionar a transcrição específica, por exemplo., alternativamente emendados formulários. Recentemente, projetado especificamente bloqueados de ácidos nucleicos (LNA) ou DNA ASO mixmers foram covalentemente ligados a uma resina magnética para recuperar em vitro transcrição ou altamente expressas RNAs ribossomal de extratos sem célula; no entanto, não foi testado para a recuperação de na vivo formado complexos mRNA-proteínas36. Tendo em conta o maior reconhecimento do controle pós-transcricional gene por RBPs e ncRNA, acreditamos que a viagem é um método útil para investigar a composição e a dinâmica dos complexos de RNP dentro de células após sinais intracelulares e ambientais e seu impacto na saúde e na doença.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Nós estamos gratos ao Dr. Jonathan Hall e Mauro Zimmermann (ETH Zurich) para a síntese de PFK2 e cep-1 ASOs, Dr. Maikel Wouters para o projeto da ASOs p27/CDKN1B e Dr. Rafal Ciosk (Instituto de pesquisas biomédicas, Friedrich Miescher Basileia) para detecção de anticorpos anti-GLD-1. Este trabalho foi apoiado pela biotecnologia e Conselho de pesquisa de ciências biológicas (BB/K009303/1) e um prêmio Royal Society Wolfson Research mérito (WM170036) para A.P.G.
Materiais
| Name | Company | Catalog Number | Comments |
| MaxQ 5000 Large Incubated and Refrigerated Orbital Shaker | Thermo Fisher Scientific | SHKE5000-8CE | Floor shaker to grow yeast cells |
| BBD 6220 | Thermo Fisher Scientific | CO2 incubator to grow human cells | |
| Sonicator Soniprep150 | MSE | MSS150.CX4.5 | Sonicator/cell disruptor. Used to shear DNA in human cell lysates |
| Stratalinker 1800 | Stratagene | Stratalinker to expose cells to UV light at 254 nm | |
| Tissue Lyser | Qiagen | RETSCH MM200 | Device to mechanically disrupt yeast cells |
| Refrigerated Centrifuge | Eppendorf | 5810R | Centrifuge to spin down cells, lysates |
| Shaking incubator, Thriller | Peqlab | Thermoshaker for 1.5 mL tubes | |
| Glass beads 0.5mm | Stratech Scientific Limited | 11079105 | Lysis of yeast cells |
| Nylon Filter, 0.41 μm | Millipore | NY4104700 | Collection of yeast cells |
| Millex-HA Filter, 0.45 µm | EMD Millipore | SLHA02510 | Filter to clear nematode lysate |
| Eppendorf LoBind microcentrifuge tubes Protein 1.5 mL | Sigma-Aldrich | 22431081 | Minimise protein loss |
| 2 mL microfuge tube | Ambion | AM12425 | Yeast extract preparation and other applications |
| 5 mL tube | Thermo Fisher Scientific | 129A | Collection of HEK293 cell lysate |
| 15 mL tube | Sarstedt | 62.547.254 | Collection C. elegans |
| 50 mL plastic tube | Sarstedt | 62.554.502 | Collection yeast cells |
| DMEM, high glucose, pyruvate | Thermo Fisher Scientific | 41966 | Media for culturing HEK293 cells |
| Penicillin-Streptomycin | Sigma-Aldrich | P4333 | Used to supplement cell culture media to control bacterial contamination |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F7524 | Used to supplement cell culture media |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| RQ1 RNase-Free DNase | Promega | M6101 | DNAse I to degrade DNA from cell lysate |
| RNasin Plus RNase Inhibitor | Promega | N2611 | RNase inhibitor to avoid RNA degradation |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitor cocktail to avoid protein degradation |
| Dynabeads Oligo (dT)25 | Thermo Fisher Scientific | 61011 | Magnetic beads required for the first step of mRNA-protein complex purification |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | Magnetic beads required for the second step of mRNA-protein complex purification |
| E. coli tRNA | Sigma-Aldrich | 10109550001 | transfer RNA from E. coli used for blocking streptavidin beads and avoid unsepecific interactions |
| Quick Start Bradford 1x Dye Reagent | BioRad | 5000205 | To determine protein concentration within lysates, using BSA as reference |
| Bovine Serum Albumin | Sigma | A7906-100G | Used to perform the standard curve that will be used as reference for protein concentration determination |
| mouse anti-Act1 | MP Biomedicals | 869100 | Antibody for detection of yeast actin in Western blot (1:2,500) |
| mouse anti-Act1 | Sigma | A1978 | Antibody for detection of human actin in Western blot (1:2,000) |
| mouse anti-HuR | Santa Cruz | sc-5261 | Antibody for detection of HuR in Western blot (1:500) |
| mouse anti–CYC-1 | Invitrogen | 456100 | Antibody for detection of Cyc-1 in Western blot (1:1,000) |
| peroxidase anti-peroxidase soluble complex | Sigma | P1291 | Detection of Pfk2:TAP in Western blot (1:5,000) |
| HRP-conjugated sheep anti-mouse IgG | Amersham | NXA931 | HRP-coupled secondary antibody |
Referências
- Mitchell, S. F., Parker, R. Principles and properties of eukaryotic mRNPs. Molecular Cell. 54 (4), 547-558 (2014).
- Iadevaia, V., Gerber, A. P. Combinatorial Control of mRNA Fates by RNA-Binding Proteins and Non-Coding RNAs. Biomolecules. 5 (4), 2207-2222 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Matia-González, A. M., Gerber, A. P., Sesma, A., von der Haar, T. Ch. 14. Fungal RNA Biology. , 347-370 (2014).
- Tsvetanova, N. G., Klass, D. M., Salzman, J., Brown, P. O. Proteome-wide search reveals unexpected RNA-binding proteins in Saccharomyces cerevisiae. PLoS One. 5 (9), (2010).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Castello, A., et al. System-wide identification of RNA-binding proteins by interactome capture. Nature Protocols. 8 (3), 491-500 (2013).
- Liao, Y., et al. The Cardiomyocyte RNA-Binding Proteome: Links to Intermediary Metabolism and Heart Disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Liepelt, A., et al. Identification of RNA-binding Proteins in Macrophages by Interactome Capture. Molecular Cell Proteomics. 15 (8), 2699-2714 (2016).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212 (2016).
- Matia-Gonzalez, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural and Molecular Biology. 22 (12), 1027-1033 (2015).
- Mitchell, S. F., Jain, S., She, M., Parker, R. Global analysis of yeast mRNPs. Nature Structural and Molecular Biology. 20 (1), 127-133 (2013).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127 (2015).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128 (2016).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42 (2016).
- Reichel, M., et al. In Planta Determination of the mRNA-Binding Proteome of Arabidopsis Etiolated Seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Koster, T., Marondedze, C., Meyer, K., Staiger, D. RNA-Binding Proteins Revisited - The Emerging Arabidopsis mRNA Interactome. Trends Plant Sciences. 22 (6), 512-526 (2017).
- Hamasaki, K., Killian, J., Cho, J., Rando, R. R. Minimal RNA constructs that specifically bind aminoglycoside antibiotics with high affinities. Biochemistry. 37 (2), 656-663 (1998).
- Bachler, M., Schroeder, R., von Ahsen, U. StreptoTag: a novel method for the isolation of RNA-binding proteins. RNA. 5 (11), 1509-1516 (1999).
- Vazquez-Pianzola, P., Urlaub, H., Rivera-Pomar, R. Proteomic analysis of reaper 5' untranslated region-interacting factors isolated by tobramycin affinity-selection reveals a role for La antigen in reaper mRNA translation. Proteomics. 5 (6), 1645-1655 (2005).
- Hartmuth, K., Vornlocher, H. P., Luhrmann, R. Tobramycin affinity tag purification of spliceosomes. Methods Molecular Biology. 257, 47-64 (2004).
- Beach, D. L., Keene, J. D. Ribotrap : targeted purification of RNA-specific RNPs from cell lysates through immunoaffinity precipitation to identify regulatory proteins and RNAs. Methods Molecular Biology. 419, 69-91 (2008).
- Slobodin, B., Gerst, J. E. A novel mRNA affinity purification technique for the identification of interacting proteins and transcripts in ribonucleoprotein complexes. RNA. 16 (11), 2277-2290 (2010).
- Slobodin, B., Gerst, J. E. RaPID: an aptamer-based mRNA affinity purification technique for the identification of RNA and protein factors present in ribonucleoprotein complexes. Methods Molecular Biology. 714, 387-406 (2011).
- Yoon, J. H., Gorospe, M. Identification of mRNA-Interacting Factors by MS2-TRAP (MS2-Tagged RNA Affinity Purification). Methods Molecular Biology. 1421, 15-22 (2016).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Blencowe, B. J., Sproat, B. S., Ryder, U., Barabino, S., Lamond, A. I. Antisense probing of the human U4/U6 snRNP with biotinylated 2'-OMe RNA oligonucleotides. Cell. 59 (3), 531-539 (1989).
- Lingner, J., Cech, T. R. Purification of telomerase from Euplotes aediculatus: requirement of a primer 3' overhang. Proceedings of the National Academy of Sciences U S A. 93 (20), 10712-10717 (1996).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Molecular Cell Proteomics. 12 (6), 1539-1552 (2013).
- Imig, J., et al. miR-CLIP capture of a miRNA targetome uncovers a lincRNA H19-miR-106a interaction. Nature Chemical Biology. 11 (2), 107-114 (2015).
- Chu, C., et al. Systematic discovery of Xist RNA binding proteins. Cell. 161 (2), 404-416 (2015).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Rogell, B., et al. Specific RNP capture with antisense LNA/DNA mixmers. RNA. 23 (8), 1290-1302 (2017).
- Matia-Gonzalez, A. M., Iadevaia, V., Gerber, A. P. A versatile tandem RNA isolation procedure to capture in vivo formed mRNA-protein complexes. Methods. , 93-100 (2017).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic Acids Research. 36 (Web Server issue), W70-W74 (2008).
- Kalendar, R., Khassenov, B., Ramankulov, Y., Samuilova, O., Ivanov, K. I. FastPCR: An in silico tool for fast primer and probe design and advanced sequence analysis. Genomics. 109 (3-4), 312-319 (2017).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Kedde, M., et al. A Pumilio-induced RNA structure switch in p27-3' UTR controls miR-221 and miR-222 accessibility. Nature Cell Biology. 12 (10), 1014-1020 (2010).
- Schumacher, B., et al. Translational repression of C. elegans p53 by GLD-1 regulates DNA damage-induced apoptosis. Cell. 120 (3), 357-368 (2005).
- Ziegeler, G., et al. Embryonic lethal abnormal vision-like HuR-dependent mRNA stability regulates post-transcriptional expression of cyclin-dependent kinase inhibitor p27Kip1. Journal of Biological Chemistry. 285 (20), 15408-15419 (2010).
- Itri, F., et al. Femtosecond UV-laser pulses to unveil protein-protein interactions in living cells. Cellular and Molecular Life Sciences. 73 (3), 637-648 (2016).
- Zhang, L., Zhang, K., Prandl, R., Schoffl, F. Detecting DNA-binding of proteins in vivo by UV-crosslinking and immunoprecipitation. Biochemical and Biophysical Research Communications. 322 (3), 705-711 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados