Method Article
הליך בידוד RNA טנדם מבוססי Oligonucleotide לשחזר את מתחמי ה-mRNA האיקריוטים-חלבון
In This Article
Summary
טנדם RNA בידוד שגרה (טיול) עבור שחזור של mRNA בנוי endogenously-חלבון מתחמי מתואר. באופן ספציפי, מתחמי RNA-חלבון תפור ויוו, polyadenylated RNAs מבודדים של תמציות עם חרוזים oligo(dT), mRNAs מסוים נלכדים עם ששונה oligonucleotides antisense RNA. חלבונים מאוגדים mRNAs מזוהים על-ידי ניתוח immunoblot.
Abstract
RNA-מחייב חלבונים (RBPs) לשחק בתפקידי מפתח שליטה post-transcriptional של ביטוי גנים. לכן, איפיון ביוכימי של מתחמי mRNA-חלבון חיוני להבנת תקנה mRNA להסיק על ידי חלבונים אינטראקציה או אי קידוד RNAs. במסמך זה, אנו מתארים טנדם RNA בידוד הליך (טיול) המאפשרת הטיהור של מתחמי mRNA-חלבון endogenously בנוי מתמציות הסלולר. פרוטוקול שני שלבים כרוך בידודו של polyadenylated mRNAs עם antisense oligo(dT) חרוזים וזו שלאחריה ללכוד של mRNA עניין עם 3'-biotinylated 2'-O-מפוגל antisense RNA oligonucleotides, אשר אז יכול להיות מבודדת עם streptavidin חרוזים. הטיול נעשה שימוש להתאושש ויוו תפור mRNA-ribonucleoprotein (mRNP) מתחמי פטריות, נמטודות, תאים אנושיים לצורך ניתוח RNA וחלבון נוסף. לפיכך, הטיול הוא בגישה רב-תכליתי הניתן לכל סוגי polyadenylated RNAs מעבר אורגניזמים ללמוד הסידור מחדש דינאמית של mRNPs שהוטלו על ידי רמזים תאיים או סביבתית.
Introduction
הכונה post-transcriptional מונעת דרך האינטראקציות בין ה-RNA מחייב חלבונים (RBPs) ללא קידוד RNAs (ncRNAs), mRNAs, אשר ישיר את העיבוד, לוקליזציה, תרגום, דעיכה של כל תעתיק בתוך תאים1 , 2. הזיהוי של RBPs ושל ncRNA אינטראקציה עם mRNAs מסוים, הקמת מה שמכונה ribonucleoprotein מתחמי/חלקיקים (RNPs), ולכן הוא מפתח להבנת גורל mRNAs ובקרת ביטוי גנים. גישות משלימות מבוצעת על איפיון ביוכימי RNP מתחמי3,4: בעוד הגישה "חלבון ממוקדת" מבוססת על הטיהור של RBPs ספציפיים, הגישה "RNA ממוקדת" כוללת בידוד subpopulations, לאחר מכן ניתוח של חלבונים אינטראקציה או RNAs או RNAs בודדים. לאחרונה, גישה ממוקדת RNA לצורך זיהוי הרפרטואר RBP אינטראקציה עם polyadenylated (poly(A)) RNAs5 הפך יותר ויותר פופולרי על ידי שילוב צעד crosslinking אור אולטרה סגול (UV) כדי לייצב חלבון-RNA אינטראקציות מראש ניתוקה של mRNAs, אשר באופן דרמטי להרחיב את הקטלוג של RBPs תאים אנושיים6,7,8,9,10,11, Caenorhabditis elegans 12,13,12, האפייה14, ו אחרים אורגניזמים15,16,17,18, 19,20. עם זאת, המיפוי של חלבונים ו/או ncRNAs התאספו על תעתיקים מסוים הוא עדיין אתגר גדול. לשם כך, שתי גישות עיקריות משמשים כיום: מצד אחד, תגים aptamer RNA הם התמזגו הרנ א עניין כדי לאפשר זיקה טיהור. ובכך, לאגד aptamers RNA עם זיקה גבוהה לאנטיביוטיקה aminoglycoside כולל tobramycin, סטרפטומיצין21,22,23,24, או חלבונים, כגון החלבון המעיל מ R17/MS2 bacteriophage או בקטריאלי streptavidin S125,26,27,28,29. אף-על-פי גישה זו הוצגה להיות תכליתי ועמיד יחסית, זה מצריך שיבוט לעצב הרנ א תחת חקירה, ולכן לא ניתן להשתמש כדי ללכוד את mRNAs טבעי. מצד שני, antisense oligonucleotides (אסוס) שימשו בשלב מוקדם ההתאוששות והביע אפיון מאוד מקורית RNPs30,31 ו RNAs ויראלי32. לאחרונה, אסוס הוחלו ללכוד זמן ללא קידוד RNAs33,34,35 ויצרו במבחנה mRNPs36. אחד הגורם המגביל העיקרי עם כל הגישות ממוקדת RNA הוא המספר עותק של RNA של עניין, שהופך את ההתאוששות של mRNAs לידי ביטוי נמוך יותר בעייתי. מגבלה זו ניתן להתגבר על ידי upscaling התגובה; עם זאת, זה יכול העלול להוביל להגדיל את הרקע.
במסמך זה, אנו מתארים שלנו פיתחה לאחרונה מבוססי אסו טנדם הליך בידוד RNA (טיול) כדי לבודד את מתחמי ה-mRNA מקורי-חלבון עם סלקטיביות גבוהה37 (איור 1). הפרוטוקול כרוכה בשני שלבים רציפים טיהור מבוססי אסו, כלומר ניתוקה של poly(A) mRNAs עם oligo(dT) זמינים מסחרית בשילוב חרוזים ואחריו לכידה של mRNAs ספציפי באמצעות במיוחד מעוצב biotinylated קצר 2'-מתוקסי אסוס RNA. הליך זה בשני שלבים מסייעת סילוק מזהמים חלבונים ומוסיף הזדמנויות עבור התאמת ואופטימיזציה. במקרה זה, הטיול היה מוחל על mRNAs הנבחר של שמרים, נמטודות, והקים תאים אנושיים כדי לאשר ויוו מתחמי mRNA-חלבון.
Protocol
1. עיצוב Antisense Oligonucleotides (אסוס)
- לנתח את מבנה שניוני של mRNA או קטע ממנו באמצעות כלים מקוונים הזמינים38. לכן, להזין את רצף נוקלאוטיד (nt) התיבה הריקה. בתיבה אפשרויות בסיסיות, בחר מינימום אנרגיה חופשית (MFE), פונקציית החלוקה , להימנע בסיסים מבודדים. בתיבה אפשרויות פלט, בחר RNA אינטראקטיבי מבנה שניוני מגרש , ולבסוף ללחוץ על הכפתור להמשיך . חלון חדש יהיה חלון המציג שמבנה mRNA עניין משני.
- בחר לפחות שלושה שונים 21-24 nts רצפים ארוכים בתוך ה-mRNA של עניין, מעדיפים באזורים חסר לזיהוי מבנים משניים (למשל., בלולאות לא מובנה) וב 3' ללא תרגום אזורים (UTR).
הערה: זה נצפתה אסוס ועוברים תהליך ריפוי כדי רצפים באזורים 3' ללא תרגום (UTR) המבוצעת בצורה הטובה ביותר. אפשרות אחת היא כי הריבוזומים מאוגד רצף קידוד (תקליטורים) עשוי לעתים להכשיל את חישול של אסוס. אסוס ועוברים תהליך ריפוי כדי 5' UTRs לא נבדקו.- בחר את האזורים עם יחס guanidine/ציטוזין קרוב 50% וחוזר חסר נוקלאוטיד טנדם למנוע יצירת פוטנציאל סיכות או ריפוי עצמי.
- באופן ידני עיצוב 2'-מתוקסי שונה oligonucleotides RNA הנושאת moiety של ביוטין-3', משלים לחלוטין אזורים שנבחרו (שלב 1.2) בתוך הרצוי יעד ה-mRNA.
הערה: 2'-מתוקסי שינויים רינדור עמידים RNases הסלולר של RNA ומגביר את דופלקס נמס בטמפרטורה, מה שמאפשר עבור כביסה מחמירים. Moiety ביוטין נדרש עבור לכידת אסוס עם streptavidin.- להתאים את טמפרטורת ההיתוך של כלאיים RNA ל ~ 60-65 ° C ולהבטיח שיש המורכבות רצף לשוני גבוה (> 60%) כפי שנקבע עם כלים מקוונים מתאימים39.
- השתמש את בסיסי בכלי החיפוש המקומי של יישור40 לחיפוש פוטנציאליים אסו קרוס-הכלאה עם אחרים mRNAs, transcriptome. בחר נוקלאוטיד הפיצוץ, להוסיף את רצפי בתיבת ריק ובחר האורגניזם עניין. תמשיך הנותרים הפרמטרים כברירת מחדל ולחץ על הפיצוץ.
הערה: אפילו חלקית רציפה היישור של 8-10 nts יכול להוביל קרוס-הכלאה והשחזור של mRNA הזה.
2. התא תרבות, הקרנת UV ותא פירוק
הערה: בחלק זה, התהליך מתואר ניצני שמרים cerevisiae ס, נמטודות C. elegansשל תאי הכליה עובריים אנושיים (HEK293). בכל זאת, זה יכול להיות מותאם אורגניזמים אחרים, אם כי לא במפורש נוסה עדיין.
- Cerevisiae ס
- לגדל תאי שמרים (למשל., זן BY4743 derivate; MATa/α his3Δ1/his3Δ1 leu2Δ0/leu2Δ0 LYS2/lys2Δ0 met15Δ0/MET15 ura3Δ0/ura3Δ0 PFK2:TAP / PFK2) ב- 500 מ"ל של YPD מדיה (תמצית שמרים 1%, peptone 2%, 2% D-גלוקוז) ב 30 מעלות צלזיוס ברעידות קבוע ב 220 סל"ד.
- לאסוף את התאים בשלב אמצע יומן (צפיפות אופטית על 600 nm (OD)600 ~ 0.6) על-ידי סינון באמצעות מסננים ניילון מיקרומטר 0.45 או צנטריפוגה ב g × 3,000 למשך 2 דקות בטמפרטורת החדר (RT). למחוק את הזרימה דרך או את תגובת שיקוע, בהתאמה.
- לשטוף את התאים שלוש פעמים עם 25 מ ל תמיסת פוספט-באגירה-(PBS) באמצעות מסנן ניילון מיקרומטר 0.45 או לאסוף על ידי צנטריפוגה ב g × 3,000 למשך 2 דקות ב RT וזורקים את תגובת שיקוע. לאסוף את התאים מהר ככל האפשר כפי הלחץ שנכפה עליו יכול להשפיע על אינטראקציות חלבון ה-RNA.
- Resuspend התאים 25 מ של PBS, ואז לשפוך את המתלים ב 15 ס מ פטרי. מניחים את המנה על קרח, להסיר את המכסה, לחשוף את התאים שלוש פעמים עם 400 ס"מ mJ-2 של 254 ננומטר UV באור UV-crosslinker עם הפסקות 2-מין על הקרח בין כל מחזור חשיפה עם ערבוב עדין.
- להעביר את התאים שפופרת 50 מ ל ולאסוף את התאים על ידי צנטריפוגה ב g × 3,000 דקות 3-4 מעלות צלזיוס. למחוק את תגובת שיקוע ולשמור בגדר.
הערה: תאים ניתן snap קפוא חנקן נוזלי ומאוחסנים ב-80 מעלות צלזיוס. - Resuspend תאי 4 מ של פירוק צוננת מאגר A (LB-A; 100 מ מ טריס-HCl, pH 7.5, 500 מ מ LiCl, 10 מ מ EDTA, 1% טריטון X-100, 5 מ מ DTT, 20 U מ ל-1 DNase אני, 100 U מ ל-1 RNasin, להשלים את קוקטייל מעכב פרוטאז נטולת EDTA).
- העבר את התאים לתוך שני צינורות 2 מ"ל. הוסף מקס. ⅔ נפחי מקורר חרוזי זכוכית ולשבש את התאים lyser רקמות ב 30 הרץ למשך 10 דקות ב 4 º C.
- לחורר בתחתית הצינורית במחט חמה ולהעביר את lysate צינור mL 1.5 טריים על ידי צנטריפוגה ב g × 600 ל 30 s.
- נקה lysate על ידי שלוש centrifugations רציפים ב 4 ° C ב g × 3,000 למשך 3 דקות, ואז 5,000 × g ו g × 10,000 למשך 5 דקות.
הערה: תמציות ניתן snap-קפוא חנקן נוזלי, המאוחסנים ב- 80 ° c
- C. elegans
- תולעים בריסטול N2 תרבות ב 20 מעלות צלזיוס על צלחות תולעים נימיות צמיחה בינוני (NGM) (0.3% NaCl אגר 1.7%, peptone 0.25%, 1 מ"מ CaCl2, כולסטרול µg 5/mL, 1 מ MgSO4, 25 מ מ KPO4 מאגר, pH 6.0) עם המתח OP50 Escherichia coli 41 .
- הוסף 5 מ של M9 מאגר41 (0.3% ח'2PO4, 0.6% נה2HPO4, 0.5% NaCl, MgSO 1 מ4) כל צלחת, ללחוץ את הצלחת resuspend התולעים ולהעביר את התולעים ל צינור 15 מ"ל. מקום 2 µL של ההשעיה שקופית ולספור את התולעים ההשעיה עם מיקרוסקופ. לאחר מכן להחיל הגורם כדי לחשב את המספר של תולעים לכל צלחת.
- לאסוף ~ תולעים 120,000 על ידי צנטריפוגה בטמפרטורת החדר (400 × g, 2 דקות, RT) ולמחוק את תגובת שיקוע.
- לשטוף את התולעים שלוש פעמים. לכן, הוספת 10 מ"ל M9 המאגר, לערבב על-ידי היפוך, לאסוף את התולעים על ידי צנטריפוגה ב g × 400 למשך 2 דקות ב- RT.
- להוסיף 15 מ"ל M9 המאגר את התולעים ומניחים על גלגל על כל ההזמנות למשך 15 דקות ב- RT.
- להעביר את התולעים NGM צלחות (~ 4,000 תולעים לכל צלחת) ולחשוף לאור-UV (254 ננומטר)-300 ס מ mJ-2 ב UV-crosslinker.
- הוסף 5 מ של מאגר M9 ישירות לצלחת ומנערים את הצלחת כדי resuspend את התולעים במאגר. להעביר את הבולם עם פיפטה צינור 15 מ"ל ולאסוף את התולעים על ידי צנטריפוגה ב g × 400 למשך 2 דקות ב- RT.
- Resuspend התולעים ב 2 מ של פירוק מאגר B (LB-B; 100 מ מ טריס-HCl, pH 8.0, 150 מ מ NaCl, 1 מ"מ EDTA, 0.75% IGEPAL, 1 מ"מ DTT, 20 U מ ל-1 DNase אני, 100 U מ ל-1 RNasin, להשלים את קוקטייל מעכב פרוטאז נטולת EDTA).
- לטחון תולעים מרגמה מלא עם חנקן נוזלי. לאסוף את האבקה שפופרת 50 מ ל, הפשרה-RT.
הערה: חנקן נוזלי להיות מטופלת על-פי נהלי בטיחות (למשל, fume הוד ובטיחות כפפות) - נקה את lysate כולל שכבת השומן על ידי צנטריפוגה-14,000 g × 10 דקות, לאחר מכן לעבור את שקופה lysate דרך מסנן 0.45 μm עם מזרק.
- בתרבית תאים אנושיים
הערה: התיאור הבא מבוסס על תרביות תאים ארעית של תאים HEK293 עם pGL3-CDKN1B-3'UTR כתב פלסמיד המבטאת את 3′UTR של CDKN1B/27 במורד הזרם של גחלילית לוציפראז ג'ין42.- תרבות HEK293 תאים בינוני (DMEM ששינה הנשר של Dulbecco) המכילה גלוקוז 25 מ מ ו פירובט נתרן 1 מ מ, בתוספת 100 U מ ל-1 פניצילין, 100 µg מ ל-1 סטרפטומיצין, 10% סרום שור עוברית (FBS), דגירה-37 מעלות צלזיוס humidified תא (חממה) המכילה 5% CO2.
- זרע ~ 3 × 10 תאים6 HEK293 בתרבות רקמה רגיל 10 ס מ לחלק את יום לפני תרביות תאים. לספור את התאים עם hemocytometer.
- לערבב 2 µg של הגן כתב (למשל., pGL3-p27-3'UTR) עם µL 20 של ריאגנט תרביות תאים, transfect את התאים ב- 70% confluency (~ 7 × 106 תאים).
- מקם את התאים בתוך אינקובטור ב 37 מעלות צלזיוס במשך 48 שעות נוספות לפני הקטיף.
- להסיר את המדיום עם פיפטה סרולוגית ושוטפים במהירות את התאים פעמיים עם 10 מ"ל של PBS להתחמם מראש ב- 37 מעלות צלזיוס. הסר PBS עם פיפטה סרולוגית.
- להוסיף 6 מ של PBS המנה, מניחים על קרח, ואז לחשוף את התאים לאור אולטרא סגול (254 ננומטר)-100 ס מ mJ-2 ב UV-crosslinker.
הערה: הצלחת יש לשמור על הקרח במהלך החשיפה UV-אור. - לגרד את התאים (בתאים7 ~ 10 סה כ) PBS ואת העברת צינור 15 מ"ל.
- ספין למטה התאים ב 250 g × 10 דקות ב 4 ° C ולאחר מכן להסיר את תגובת שיקוע עם פיפטה.
הערה: בגדר התא יכול להיות snap-קפוא חנקן נוזלי ומאוחסנים ב-80 מעלות צלזיוס עד השימוש. - Resuspend התאים 2 מ של מאגר פירוק טרום מקורר (LB-A) על ידי pipetting למעלה ולמטה במשך 5 - 6 פעמים, תוך שמירה על הצינור על קרח.
- להעביר את lysate עם פיפטה שפופרת 5 מ ל הניח על קרח.
- נושא את lysate כדי שלושה סבבי sonication בהיקף של 20 s לפרצי ב 10 משרעת μm עם 30 s קירור תקופות על קרח.
הערה: Sonication מומלץ בזמן שהוא משלים את פירוק של תאים ושל מקטעי ה-DNA. - להעביר את lysate 2 mL צינורות, צנטריפוגה ב 15,000 × g 10 דקות ב 4 º C. לאסוף את תגובת שיקוע (= תמצית), העברת צינור חדש. הסר דוגמה אנליטי (5-10%) עוד יותר חלבון וניתוח RNA.
הערה: שלב זה הוא קריטי כדי להסיר את כל שאריות תאים הנותרים, ניתן לחזור.
3. שלב הראשון: בידוד Poly(A) RNA
- Equilibrate 1 מ ג של oligo(dT)25-מצמידים beads מגנטי ב 500 µL של המאגר פירוק בהתאמה.
- לשלב 5 מ"ג, 10 מ ג או 4 מ"ג (חלבון) של cerevisiae ס, C. elegans או תמציות HEK293, בהתאמה, עם 1 מ ג של oligo(dT)25 -מצמידים beads מגנטי. מערבבים את הדגימות נמרצות במשך 10 דקות 25 ° c במיקסר.
הערה: ניתן לקבוע ריכוז חלבון של תמציות עם ברדפורד assay באמצעות אלבומין שור (BSA) כסטנדרט הפניה. רעד נמרצת של דגימות מונע שקיעת של חרוזים. כדי להעריך את ייחודה של ה-mRNA בידוד, ניתן לבצע ניסוי שליטה במקביל על-ידי הוספת עודף (20 µg) של חומצות polyadenylic (פי אי). - במקום הצינורות על דוכן מגנטי עבור 10 s והסר תגובת שיקוע. שמור את תגובת שיקוע על הקרח לביקור עוקבות של התאוששות (שלב 3.7).
- להוסיף 500 µL של שטיפת מאגר A (10 מ מ טריס-HCl, pH 7.5, 600 מ מ. LiCl, 1 מ"מ EDTA, 0.1% טריטון X-100) חרוזים, מערבולת עבור 5 s.
הערה: ריכוז LiCl במאגרי שטיפת יכול להשתנות. 600 מ מ LiCl זה מומלץ אך ריכוז נמוך יותר נבדקו גם (500 מ מ של C. elegans, 300 מ"מ עבור HEK293). - לאסוף את החרוזים עם מגנט ולאחר מכן לשטוף את החרוזים פעמיים עם 500 µL שטיפת מאגר B (WB; 10 מ מ טריס-HCl, pH 7.5, 600 מ מ. LiCl, 1 מ מ EDTA).
- Elute את הרנ א ב 30 µL 10 מ מ טריס-HCl, pH 7.5 ב 80 מעלות צלזיוס למשך 2 דקות עם רציף רועד (1,000 סל ד) במיקסר. מיד למקם את הצינור ביציע מגנטי ולאסוף את eluate אחרי 10 ס להציל את החרוזים לביקור נוסף של טיהור.
הערה: לאסוף את eluate מהר ככל האפשר כדי למנוע איגוד מחדש פוטנציאלית של mRNAs החרוזים בטמפרטורות נמוכות. - להוסיף את החרוזים מהשלב הקודם תגובת שיקוע שנאספו בשלב 3.3, וחזור על לכידת, כביסה, • תנאי של mRNAs פעמיים (שלבים 3.3-3.6). Eluates של סיבובים חוזרים ונשנים משולבים מכן ויש שניתן לאחסן ב- 80 ° c
4. שנית שלב: mRNAs לכידה של ספציפי עם 3'-Biotinylated 2'-מתוקסי השתנה Antisense RNA Oligonucleotides
הערה: כדי לבדוק את ההתאמה של אסוס, ההליך הבא יכול להתבצע עם הכולל RNAs מבודד מן התאים.
- Equilibrate 30 µL של streptavidin מצמידים beads מגנטי ב 1 מ"ל של איגוד ולשטוף מאגר (מאגר B & W; 10 מ מ טריס-HCl, pH 7.5, 150 מ מ NaCl, 0.5 מ מ EDTA, pH 8.0) המכיל 0.1 מ ג מ ל-1Escherichia coli העברה RNA (tRNA) ב מסובב על 1 h RT.
- לשטוף את החרוזים שלוש פעמים עם 750 µL מאגר B & W.
- Resuspend את החרוזים ב 30 µL של B & W מאגר ולשמור בקירור עד השימוש.
- לדלל µg ~ 35 סך החלבון מן הבידוד poly(A) הקודם (ראו 3) ב- µL 100 של מאגר B & W בצינור 1.5 מ ל.
הערה: כדי לבדוק יחודיות של אסוס עם RNA הכולל, להוסיף 600 ng של RNA הכולל מטוהרים כדי µL 100 מאגר B & W. - להוסיף 200 pmol של אסו בהתאמה, דגירה-70 מעלות צלזיוס למשך 5 דקות.
הערה: הטמפרטורה מוגברות פותר משני מבנים הרנ א הקלת חישול של אסו. - החום-בניין שלם להסיר ההתקן ומקם אותו ב RT 10 דקות להתקרר לאט.
- להוסיף 30 µL של beads מגנטי מצמידים streptavidin equilibrated מ שלב 4.1 המדגם.
- דגירה את התערובת למשך 30 דקות ב- 25 ° C עם טלטול מתמיד ב- 950 סל ד במיקסר.
הערה: מומלץ כדי לסתור את הצינורות בכל 10 דקות למניעת שיקוע של חרוזים. - למקם את הצינורות על הדוכן מגנטי, להסיר את תגובת שיקוע ולשטוף את החרוזים שלוש פעמים עם 750 µL מראש ומחוממת מאגר B & W ב- 55 מעלות צלזיוס.
הערה: הטמפרטורה שטיפת אופטימלית עשוי מעט שונים/להשתנות בין אסוס שונים. מומלץ להתאים את המצב הזה עם הלא-תפור הכולל RNA לפני ביצוע הניסוי עם תמציות תא. - Elute את הרנ א ב µL 20 10 מ מ טריס-HCl, pH 7.5 ב 90 מעלות צלזיוס למשך 10 דקות עם טלטול מתמיד ב- 950 סל ד במיקסר. למקם את הצינורות הדוכן מגנטי ולאסוף מיד את eluate.
הערה: לצורך ניתוח חלבון, להוסיף µL 20 1 × Laemmli מאגר החרוזים, דגירה ב 95 מעלות צלזיוס במשך 5 דק חלבונים המפוקחים על-ידי ניתוח immunoblot בעקבות פרוטוקולי מעבדה סטנדרטיים.
תוצאות
פיתחנו אסטרטגיית בידוד מבוסס אסו RNA, כינה את הטיול, כדי ללכוד mRNAs מסוים עם שלהם חלבונים מאוגד של אורגניזמים שונים 337. בעיקרו של דבר, ה-RNA-חלבון מתחמי היו תפור ויוו על ידי הקרנת UV של תאים 254 ננומטר, ואת poly(A) RNAs ששוחזרו עם חרוזים בשילוב-מגנטית oligo זמינים מסחרית (dT), ואז ה-mRNA של עניין היה מבודד עם 3'-biotinylated 2-0'-מתוקסי ששינה antisense RNA oligonucleotides (איור 1). אנחנו ולכן שנועדה מספר 21-24 nts ששינה אסוס עם מלא-משלימים את החסר אזורים ב- mRNAs הנבחר שמרים, C. elegans , האדם, ונבדק להתאמתם כדי לשחזר את ה-mRNA של עניין (רשימה של תחל אסוס הוא נתון טבלה 1). יעילות וספציפיות של אסוס בודדים הוערכו קודם עם הלא-תפור RNA הכולל מבודד מן האורגניזם בהתאמה. בניסויים אלה, אסוס RNA היו מצמידים streptavidin מצומדת חרוזים פאראמגנטיים, מודגרות עם הלא-תפור RNA הכולל מקריסטלים של האורגניזם המתאים. לאחר שחרורו של mRNAs בשבי של החרוזים, הנוכחות של mRNA מטרות הבקרה גם הכי לא קשורים mRNAs נוטרה על ידי שעתוק במהופך (RT)-פולימראז תגובת שרשרת (PCR)37. שמנו לב כי שני משתנים, ריכוז המלחים טמפרטורה של מאגרי לשטוף, שגילמה תפקידים חיוניים היעילות של לכידתו של mRNA PFK2 של שמרים שבו נבדקה עם שלושה שונים אסוס (איור 2 א). הורדת הריכוז (NaCl) מלח כדי 25mm מופחתת ההתאוששות של התשליל שליטה mRNA (PFK1) לרמות נסתרים עם כל אסוס, אך זה גם צמצמה את ההתאוששות של הרצוי היעד mRNA PFK2 (10-15% הקלט). לעומת זאת, גידול של ריכוז מלח פיזיולוגית רמות (150 מ מ NaCl) גדל ההתאוששות של PFK2 mRNAs עד 75% עם אסו PFK2-2, העולה על זה של הפקד PFK1 mRNA מאת 5-fold לפחות (איור 2 א). ראוי עוד לציין, אסוס שונים הראו וקצרה mRNA efficacies ללכידת המטרה-פיזיולוגיים ריכוזי מלח, מדגישים את הצורך באימות אמפירי של אסוס. התלות של mRNA שחזור הטמפרטורה של המאגר שטיפת הוא דוגמה עבור C. elegans cep-1 ושמרים mRNAs RPS20 , שימוש בהתאמה אסוס (טבלה 1). הבחנו כי הטמפרטורה שטיפת אופטימלית היה בין 50 ° C ל- 55 ° C, כפי שיתבקש מהרקע נמוך עם mRNAs שאינם קשורים ושחזור יעיל mRNAs היעד (איור 2B). בשלב זה, אנו רוצים להדגיש את האפשרות קרוס-הכלאה של אסוס עם mRNAs אחרים. לדוגמה, אסו DNM1 מלא משלימים רצף בתוך DNM1 קידוד רצף, אבל זה גם חלקית anneals עם ACT1 mRNA. אסוס DNM1 התאוששה בשני mRNAs ללא קשר הטמפרטורה כביסה, מראה נטייה חזקה קרוס-הכלאה (איור 2C). לבסוף, השתמשנו המבחנים שתואר לעיל, אופטימיזציות לבחירת אסוס שלושה שהיו מתאים ההתאוששות של יעד בהתאמה mRNAs מ סך RNA מבודד cerevisiae ס, C. elegans , תאים אנושיים (איור דו-ממדי ).
לאחר הבחירה הראשונית של מתאימים אסוס במבחנה, ביצענו טיול עם תמציות התא המתקבל UV-תפור אורגניזמים/תאים (איור 1). באופן ספציפי, בדקנו את ההתאוששות של שלוש mRNAs שונות של אורגניזמים שונים שלוש: PFK2 mRNA של השמרים cerevisiae ס, cep 1 של נמטודות C. elegans, ואף כתב mRNA (pGL3-CDKN1B-3'UTR) הנושאת את 3' UTR רצף של האדם CDKN1B/p27 mRNA התמזגו עם כתב לוציפראז (לוק) להבעה ארעי התאים HEK293 של האדם. כדי לשלוט יחודיות של הבידוד RNA, אנחנו במעקב ההתאוששות של מספר mRNAs לא קשורה, אנחנו ביצע ניסויים תחרות על ידי התוספת של עודף של הרשות הפלסטינית על תמציות התא, אשר מתחרה עם הכריכה של mRNAs הסלולר כדי oligo(dT)25 חרוזים במהלך השלב הראשון שלב טיהור. כפי שראינו בעבר עם הלא-תפור הכולל RNA דגימות (איור 2), RT-PCR אישר העשרת של mRNAs בהתאמה היעד mRNA במהלך הטיול, ואילו מספר שליטה שאינם קשורים mRNAs היו לא מועשר (איור 3 א). יתר על כן, גם אני לא mRNAs אותרו על חרוזים בלי אסוס, המציינת את נהלי חסימה המתאים להימנע איגוד לא ספציפי. בקו זה, אסוס לא קשור/מקושקשות עשוי לשמש גם שליטה, למרות הפוטנציאל של צלב-הכלאה עם mRNAs מסוימים, לכידת הנגזרת של חלבונים מאוגד יש אז יש לקחת בחשבון. מאז חלבונים ב- eluate הסופי יכול להיות דמיינו בקושי על ג'לים לזיהוי שהוכתמו כסף (נתונים לא מוצג), נוכחותם של חלבונים שמעצבת ידועים של mRNA הוערך עוד יותר על ידי ניתוח immunoblot. זה כולל את Pfk2p של cerevisiae ס, אשר נקשר באופן סלקטיבי PFK2 mRNA באופן עצמאי ריבוזום12; C. elegans GLD-1, RBP הקנוני המאגד 3' UTR רצפים של cep-1 זה mRNA עבור translational תקנה43; חור, RBP המסדירה את יציבות ה-mRNA ותרגום של mRNA p27/CDKN1B 44. כצפוי, כל החלבונים האלה אותרו ב- הטיול eluates של mRNAs בהתאמה על ידי ניתוח המערבי כתם (איור 3B).
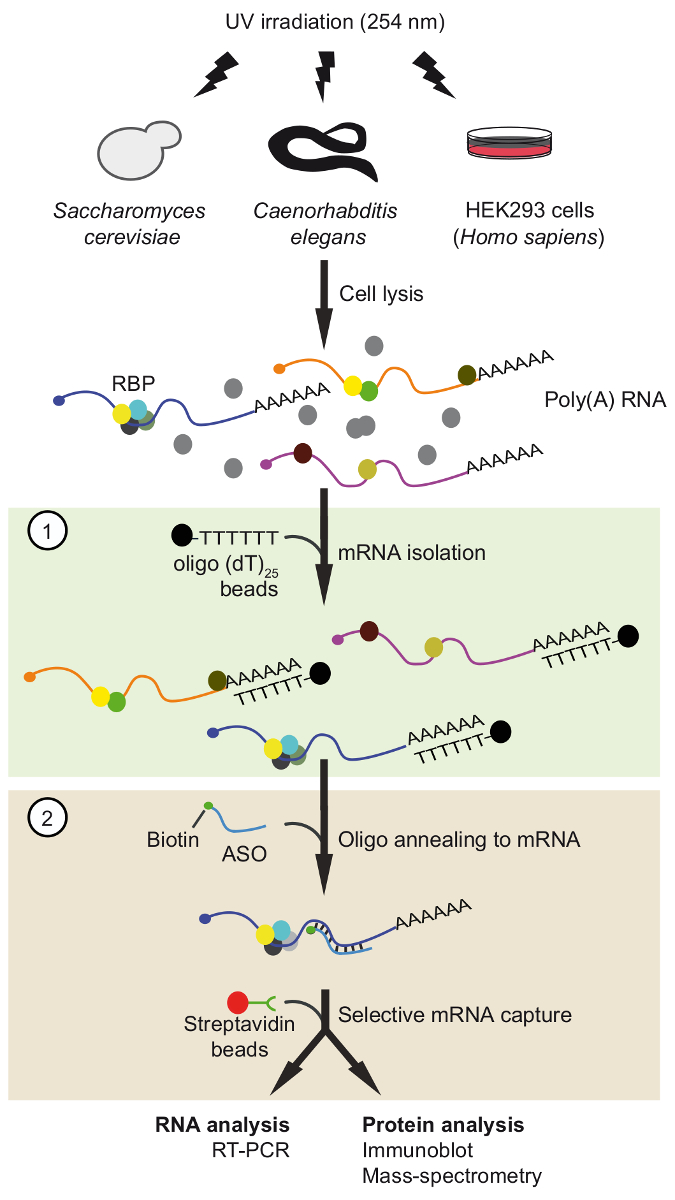
איור 1. ייצוג סכמטי של הטיול- חלבונים הם תפור RNA ויוו על ידי הקרנת UV. בשלב הראשון (תיבת ירוק בהיר), poly(A) RNA-חלבון מתחמי התאוששו עם חרוזים25 oligo(dT) בהחלת תנאים מחמירים כביסה כדי להסיר חלבונים לא מאוגד. בשלב השני (תיבת ורוד), mRNP היעד הוא הוציא החוצה עם biotinylated antisense RNA oligonucleotides וחרוזים streptavidin. MRNPs מטוהרת הם מכן נותחו על ידי RT-PCR ו- immunoblot/פלאנארית (MS) כדי לזהות RNAs וחלבונים אינטראקציה עם ה-mRNA של עניין, בהתאמה. הדמות שונה מן הפרסום הקודם37 עם הרשאה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
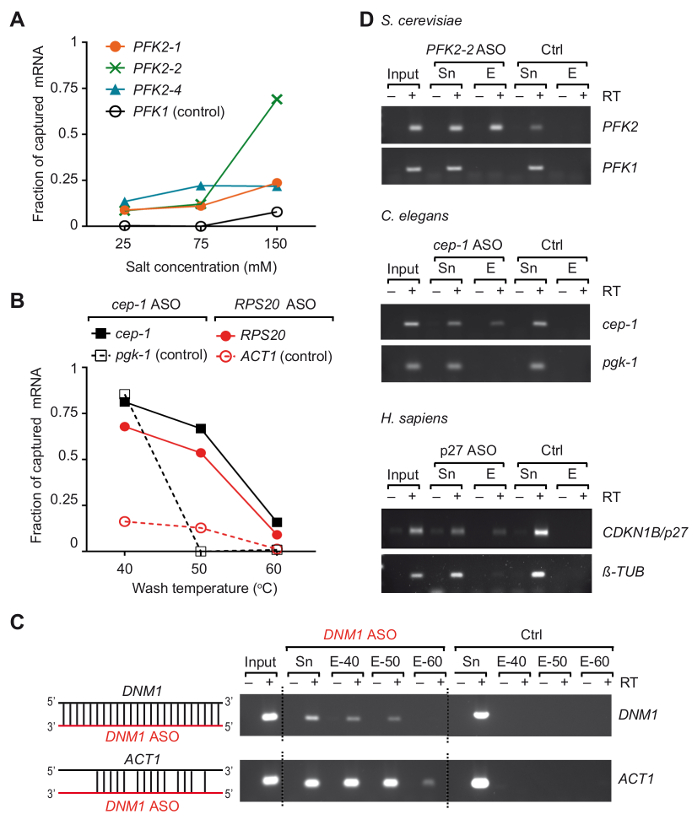
באיור 2. בידוד של mRNAs שנבחרה מ- RNA הכולל של התאים הלא-תפור/באמצעות אורגניזמים ששינה antisense RNA לכידת הגששים. (א) ההשפעה של ריכוזי מלח על יעילות לכידת של השמרים PFK2 mRNA עם אסוס. סה כ RNA מתאי שמרים בשילוב של אסוס המצוין ושטף עם מאגר המכיל את הריכוז (NaCl) מלח שצוין ב- 55 מעלות צלזיוס. ירוק, כחול וכתום קווים מייצגים PFK2 שחזור mRNA עם אסוס שונים כפי שנקבע על-RT-PCR37: PFK2-1 ו- PFK2 -2 anneal ב 3' UTR, PFK2-4 את התקליטורים. PFK1 הוא נגטיבי לשלוט mRNA. PCR הופיעה עם מחזורים הגברה 32, לכמת כמו שתואר לעיל37. (B) חלק ג eleganscep-1 ושמרים RPS20 אסו מאוגדים mRNAs בטמפרטורות שונות לשטוף, מיוצגים בקווים אדומים ושחורים, בהתאמה. Eleganspgk ג - 1 שמרים ACT1 mRNAs הלא-יעד (בקרה) בקרב אנשי עסקים ותיירים כאחד. מחזורים PCR 30 ו- 32 הוחלו לגילוי של שמרים, mRNAs C. elegans , בהתאמה. (ג) ייצוג סכמטי של הכלאה DNM1 אסו (אדום) עם רצפי DNM1 mRNA, כמו גם פוטנציאל קרוס-הכלאה עם ACT1 mRNA מוצגת משמאל. ג'ל agarose מציג מוצרים מ- RT-PCR תגובות (30 מחזורים) עבור זיהוי שמרים DNM1 ו ACT1 mRNAs eluted של חרוזים מוצג מימין. קלט, RNA הכולל; Sn, תגובת שיקוע לאחר דגירה עם אסוס; E, eluates של חרוזים שטפו אותם במקום המצוין • מוקדמת תנאי הטמפרטורות ( 40 ° צלזיוס, 50 ° C ו- 60 ° C). ניסוי שליטה (Ctrl) בוצעה במקביל מבלי להוסיף אסו. (ד) Agarose ג'ל מציג מוצרים RT-PCR איתור mRNAs (מימין) שנלקחו. RNA הכולל של שמרים cerevisiae ס, C. elegans, תאים HEK293 אנושיים (ח' סאפיינס). קלט, RNA הכולל; Sn, supernatant; E, eluates של החרוזים. PCR בוצע על ידי 30 מחזורי הגברה עבור שמרים mRNAs, מחזורי 32 עבור mRNAs C. elegans , 28 ו- 30 מחזורים האנושי טובולין , p27 mRNAs, בהתאמה. הדמות שונה מן הפרסום הקודם37 עם הרשאה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
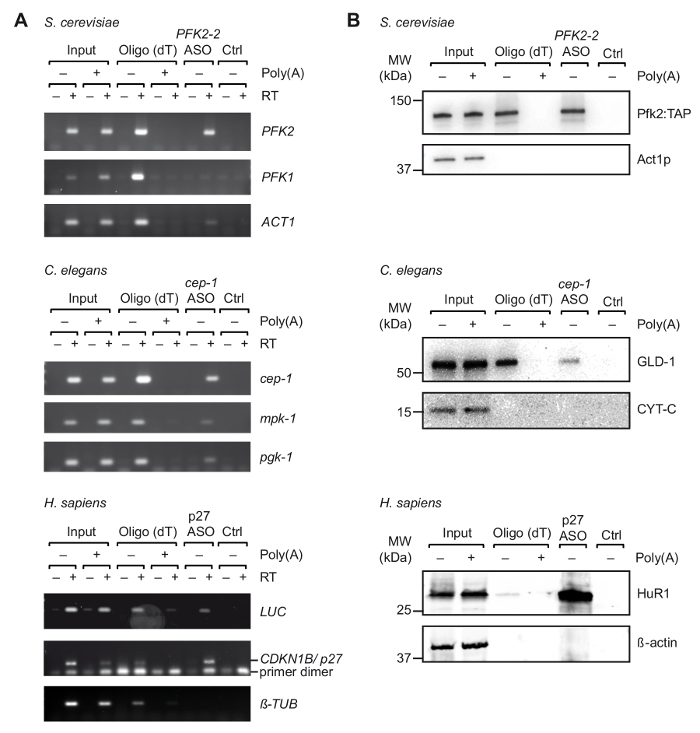
איור 3. לכידתו של mRNA ספציפי-חלבון קומפלקסים של תמציות נגזר מתאי תפור UV עם טריפ. Agarose (א) ג ' לים, הגילוי של mRNAs (מימין) עם RT-PCR על לכידת עם oligo(dT) של אסוס המצוין cerevisiae ס, C. elegans , תמציות האנושי של תא (ח' סאפיינס). קלט, RNA הכולל של תפור תאים/אורגניזמים; Ctrl, שליטה ללא אסו. Poly(A), תחרות עם poly(A). RT-PCR בוצע כפי שמתואר37 עם מחזורים הגברה 35 עבור לוק, מחזורי 32 עבור p27, ו 29 מחזורים עבור טובולין. (B) ניתוח Immunoblot של חלבונים mRNA מכורך עם נוגדנים המצוין (מימין). שברים טעון הם כדלקמן: 0.1%, 2.5%, 1% עבור שמרים, תשומות נמטודות ואנושי; 10%, 10% ו-5% עבור שמרים, מסלולים oligo(dT) נמטודות ואנושי; 66% עבור כל הנתיבים אסוס. מולקולרית משקל (MW) מסומנים ב- kilodaltons (kDa). דמות מחדש עם הרשאה37. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| פריימר שם | רצף | היעד | גודל | |
| Pfk2_Fwd | GTGTTAAGGGTTCACATGTCG | PFK2 cerevisiae ס | 133. לחץ דם | |
| Pfk2_Rev | CTTCCAACCAAATGGTCAGC | PFK2 cerevisiae ס | 133. לחץ דם | |
| Pfk1_Fwd | GGTGATTCTCCAGGTATGAATG | PFK1 cerevisiae ס | 97. לחץ דם | |
| Pfk1_Rev | CTTCGTAACCTTCGTAAACAGC | PFK1 cerevisiae ס | 97. לחץ דם | |
| Act1_Fwd | GTCTGGATTGGTGGTTCTATC | ACT1 cerevisiae ס | 85. לחץ דם | |
| Act1_Rev | GGACCACTTTCGTCGTATTC | ACT1 cerevisiae ס | 85. לחץ דם | |
| Dnm1_Fwd | CTGTGTTCGATGCATCAGAC | DNM1 cerevisiae ס | 156. לחץ דם | |
| Dnm1_Rev | CGCACTCCAATTCTTCTCTC | DNM1 cerevisiae ס | 156. לחץ דם | |
| Rps20_Fwd | CGCTGAACAACACAACTTGG | RPS20 cerevisiae ס | 228. לחץ דם | |
| Rps20_Rev | GGAAGCAACAACAACTTCGAC | RPS20 cerevisiae ס | 228. לחץ דם | |
| Cep1_Fwd | CGATGAAGAGAAGTCGCTGT | cep-1 C. elegans | 110. לחץ דם | |
| Cep1_Rev | ATCTGGGAACTTTTGCTTCG | cep-1 C. elegans | 110. לחץ דם | |
| Pgk1_Fwd | GCGATATTTATGTCAATGATGCTTTC | pgk-1 C. elegans | 74. לחץ דם | |
| Pgk1_Rev | TGAGTGCTCGACTCCAACCA | pgk-1 C. elegans | 74. לחץ דם | |
| Mpk1_Fwd | TGCTCAGTAATCGGCCATTG | mpk-1 C. elegans | 74. לחץ דם | |
| Mpk1_Rev | TCCAACAACTGCCAAAATCAAA | mpk-1 C. elegans | 74. לחץ דם | |
| p27_Fwd | TTTAAAAATACATATCGCTGACTTCATGG | p27 ה ספיינס | 212. לחץ דם | |
| p27_Rev | CAAAGTTTATGTGCTACATAAAAGGTAAAAA | p27 ה ספיינס | 212. לחץ דם | |
| Luc_Fwd | AATGGCTCATATCGCTCCTGGAT | לוציפראז pγralis פ | 117. לחץ דם | |
| Luc_Rev | TGGACGATGGCCTTGATCTTGTCT | לוציפראז pγralis פ | 117. לחץ דם | |
| Β-TUBULIN_Fwd | CTGAACCACCTTGTCTCAGC | Β-טובולין ה ספיינס | 136. לחץ דם | |
| Β-TUBULIN_Rev | AGCCAGGCATAAAGAAATGG | Β-טובולין ה ספיינס | 136. לחץ דם | |
| אסו PFK2-1 | AAUAGAAAGUGUAAUAAAAGGUCAU | 3' UTR cerevisiae ס PFK2 | - | |
| אסו PFK2-2 | GUUUCAUGGGGUAGUACUUGU | 3' UTR cerevisiae ס PFK2 | - | |
| אסו PFK2-4 | CUUGAAGAGGAGCGUUCAUA | ORF cerevisiae ס PFK2 | - | |
| DNM1 אסו | UCGGUCAGUGGAGGUUCAGCGUUU | ORF cerevisiae ס DNM1 | - | |
| RPS20 אסו | GUCGGUAAUAGCCUUCUCAUUCUUG | ORF cerevisiae ס RPS20 | - | |
| cep-1 אסו | GUGAGAAAUGCGGUGCUUUGAAA | 3' UTR cep-1 C. elegans | - | |
| p27 אסו | UCAUACCCCGCUCCACGUCAGUU | 3' UTR p27 ה ספיינס | - | |
טבלה 1. רצפים oligonucleotide. רשימת תחל PCR ואסוס בשימוש זה בעבודה, פריימר רצף, ג'ין היעד של גודל המקטע הצפוי לאחר הגברה.
Discussion
הטיול מאפשר הניתוח של חלבונים קשורה ספציפית mRNAs ויוו באמצעים ביוכימיים. כאשר השתמשנו בשיטת כדי לאמת את האינטראקציה של RBPs מסוים עם מטרות mRNA אורגניזמים/תאים שונים, הטיול גם ייתכן החל ללמוד סוגים אחרים של poly(A) RNAs, כגון cytoplasmic ncRNAs ארוך. יתר על כן, ניתוח שיטתי של חלבונים המאוגד ו/או RNAs עם רצף MS או רנ א יכולה להיות מושגת על ידי למעלה-קנה מידה של ההליך. בהקשר זה, הראשוניים שלנו מציין כי 1 ליטר של תרביות שמרים ולפחות 100 מיליון תאים אנושיים HEK293 יכולה לספק מספיק חומר המוצא כדי לקבל נתונים MS אמין עבור ביטוי היטב mRNAs (פורסם תוצאות).
הפרוטוקול המתואר הטיול כולל את ההקרנות של תאים עם אור UV-254 ננומטר על אינטראקציות crosslink RNA-חלבון. פעולה זו מאפשרת ליישום תנאים מחמירים שטיפת במהלך טיהור. ובכל זאת, אם כי לא במפורש נבדק, אנו מאחלים לשים לב כי crosslinking הוא אופציונלי, גם עשוי להיות התאושש RNPs פעיל עם מאגרי פיזיולוגיים. עם זאת, במקרה זה, הסידור מחדש פוטנציאלית של חלבונים ו/או RNAs על המטרה RNA ב lysates צריכים להילקח בחשבון. לעומת זאת, אם UV או הליכים אחרים crosslinking יישומית (למשל, פורמלדהיד), בשלמות הרנ א צריך להעריך משום השפלה RNA עלול לגרום לשחזור מופחתת של mRNPs. בנוסף, UV-crosslinking נמצא יעיל למדי (~ 5%), פחות חלבונים אינטראקציה עם כפול גדילי RNA, אשר יכול להטיה איתור לשיעורים מסוימים של RBPs12. לבסוף, הקרנת UV יכול גם לגרום crosslinks חלבון, חלבון-DNA הנחשבות עבור נתונים לפרשנות45,46. לכן, נהלים חלופיים crosslinking, לדוגמה עם פורמלין, יכול להיות גם עניין מיוחד כדי ללכוד ההרכבות חלבון גדול יותר mRNAs.
אחת התכונות המרכזיות של הטיול הוא שני צעדים טיהור מבוססי אסו הליך לשחזר RNAs מסוים, ובכך מגדיל סלקטיביות, להקטין את ההסתברות של שיתוף טיהור מזהמים. למשל, הדאגה העיקרית מתייחסת הוספת biotinylated אסוס ישירות אל התא תמציות, אשר יכול להוביל טיהור משותף של ביוטין מחייב חלבונים. זה הוא מצויין בטיול על-ידי יישום בסיבוב הראשון של טיהור של RNAs poly(A) באמצעות oligo-(dT) covalently בשילוב25 beads מגנטי, מה שמאפשר עבור תנאים מחמירים לטיהור כפי שנעשה עבור חלבון ה-RNA interactome לכידת. יתר על כן, הבחירה RNA poly(A) מסירה ncRNAs שופע במיוחד, כגון ribosomal RNAs ו- tRNAs, והיא ולפיכך מפחית את המורכבות של המדגם, מקורות פוטנציאליים על הצלב-הכלאה וזיהום. בשלב השני, mRNAs מסוים התאוששו עם biotinylated ולשנות אסוס זה anneal עם אזורים על המטרות mRNA. לחלופין, אסוס ששונה יכול להיות ישירות covalently מצורף גם beads מגנטי דרך-אמין-לינקר, צמצום של הנטייה ללכוד ביוטין מחייב חלבונים. עם זאת, מניסיוננו, הדגירה של השבר RNA poly(A) עם אסוס לפני התוספת של חרוזים streptavidin התאוששה mRNPs ביעילות רבה יותר מאשר תוספת ישירה של חרוזים מצמידים אסו. ייתכן, oligos חינם העדיף kinetically עם גישה טובה יותר רצפי RNA מובנית לעומת oligos קיבוע. לפיכך, קשרי ערכיות של אסוס כדי להשתלב חרוזים לפני הדגירה עם הדגימה יכולה להיות מזיקה להתאוששות של המטרה RNA ויש להיבדק מדעית.
חסרון אחד עם אסו המבוסס על שיטות הוא שחזור לא יעיל של כמה mRNAs היעד. לכן, אנו ממליצים הערכת מספר אסוס זה anneal לאזורים שונים של התעתיק. באופן ספציפי, אנו מציעים את העיצוב של אסוס 2-3 זה anneal עם רצפי 3'-UTR או את הדיסקים של ה-mRNA עניין. כל אסו עשוי להפגין על יעילות שונות עבור ההתאוששות של המטרה mRNA בהתאמה, צריך להיות מדעית נבדק (איור 2 א, ב', נתונים לא מוצג). לבסוף, מצאנו אסוס ועוברים תהליך ריפוי UTRs 3' ביצעו טוב יותר בהשוואה לאלה חישול לתקליטורים. זה יכול להיות בגלל הנגישות מוגברת של איגוד אתרים UTRs בהעדר לתרגם את הריבוזומים, ואילו הריבוזומים עלול להכשיל הכלאה יעילה בתוך תקליטורים. גם שחווינו זה שילוב של אסוס מספר יכול להוביל לזיהום מוגבר של mRNAs-יעד שופע, מופחתת יעילות נפתחים. בכל מקרה, מידת הבררנות של oligos שבחרת. יכול להיות נשלט על ידי תחרות ניסויים (למשל., תוספת של oligos מתחרים).
נוספים הדאגה מתייחס פוטנציאליים קרוס-הכלאה עם RNAs לא קשורים, כפי הבחנו כי הכלאה אפילו חלקית עם אחרים mRNAs יכול להוביל ההתאוששות של mRNA הזה (איור 2C). להערכת ההתאמה של אסוס תוכנן, לכן מומלץ ביצוע הראשונית במבחנה בדיקות עם RNAs הכולל כדי למטב את ריכוזי מלח וטמפרטורה כביסה של מאגרים. בידיים שלנו, שטיפה של מצמידים streptavidin beads מגנטי במאגרי המכיל 150 מ מ NaCl ב 55 ° C שבוצעה בצורה הטובה ביותר, אך אנו ממליצים על בדיקות עצמאיות של תנאי כל אסו מעוצב. ראוי לציין, מצאנו כי כל אסוס שנבחר מתוך ניסויים בתוך חוץ גופית (איור דו-ממדי) היו המתאים ללכידת המטרה mRNA בהתאמה של תמציות תא תפור (איור 3), substantiating עוד יותר את תוקפו של ב חוץ גופית בדיקות. מלבד עיצוב אסוס מתאימים עבור לכידת mRNA, גורמים נוספים יכול להשפיע על סלקטיביות. לכן, נהלי חסימה מקיף עם BSA ו/או tRNA בהתאם למפרטים סוג חרוז יכול למנוע קשירה לא ספציפי חרוזים. כדי לצמצם עוד יותר את ספיגת חלבונים לא ספציפי, אנחנו גם ממליצים על השימוש Lo-bind צינורות, במיוחד כאשר עובדים עם כמויות נמוכות של דגימות.
באופן כללי, הטיול יכול להשלים הגישות שנוצרו קודם לכן כדי ללכוד RNAs עם אסוס. למשל, ה-RNA ללא קידוד Xist היה מבודד עם חלבונים מאוגד מן הגרעין תמציות נגזר 200-800 מיליון UV תפור תאים באמצעות מערך של זמן (90-mer) biotinylated DNA oligonucleotides שכיסה את התעתיק כולו 34,35. עם זאת, הגישה ריצוף שבחרת שיכול להיות בעייתי לאור הפוטנציאל הגדול של צלב-הכלאה עם RNAs לא קשורים, זה לא יכול לשמש כדי לבחור פרוטוקול ספציפי, למשל., לחלופין משולבים טפסים. לאחרונה, שתוכננה במיוחד חומצת גרעין נעול (מגבר קדם) או DNA אסו mixmers חוברו covalently שרף מגנטי להתאושש במבחנה תעתיק או ביטוי מאוד ribosomal RNAs. התא ללא תמציות; עם זאת, לא נוסה על ההתאוששות של ויוו נוצר חלבון ה-mRNA מתחמי36. לאור ההכרה מוגברת של ג'ין post-transcriptional שליטה על-ידי RBPs ncRNA, אנו מאמינים כי הטיול היא שיטה שימושית כדי לחקור את ההרכב ואת הדינמיקה של מתחמי RNP בתוך התאים על רמזים תאיים וסביבתיים והשפעתו ב. בחולי ובבריאות
Disclosures
המחברים אין לחשוף.
Acknowledgements
אנחנו אסירי תודה ד ר ג'ונתן הול, מאורו צימרמן (ETH ציריך) לסינתזה של PFK2 ו- cep-1 אסוס, ד ר מייקל ואוטרס על עיצוב האתר של אסוס p27/CDKN1B, ואת ד ר Rafal Ciosk (פרידריך מישר מכון למחקר ביורפואי, באזל) עבור נוגדנים anti-GLD-1. עבודה זו נתמכה על ידי הביוטכנולוגיה, מחקר מדעי הביולוגיה המועצה (BB/K009303/1), המלכותי החברה וולפסון למחקר פרס הצטיינות (WM170036) כדי A.P.G.
Materials
| Name | Company | Catalog Number | Comments |
| MaxQ 5000 Large Incubated and Refrigerated Orbital Shaker | Thermo Fisher Scientific | SHKE5000-8CE | Floor shaker to grow yeast cells |
| BBD 6220 | Thermo Fisher Scientific | CO2 incubator to grow human cells | |
| Sonicator Soniprep150 | MSE | MSS150.CX4.5 | Sonicator/cell disruptor. Used to shear DNA in human cell lysates |
| Stratalinker 1800 | Stratagene | Stratalinker to expose cells to UV light at 254 nm | |
| Tissue Lyser | Qiagen | RETSCH MM200 | Device to mechanically disrupt yeast cells |
| Refrigerated Centrifuge | Eppendorf | 5810R | Centrifuge to spin down cells, lysates |
| Shaking incubator, Thriller | Peqlab | Thermoshaker for 1.5 mL tubes | |
| Glass beads 0.5mm | Stratech Scientific Limited | 11079105 | Lysis of yeast cells |
| Nylon Filter, 0.41 μm | Millipore | NY4104700 | Collection of yeast cells |
| Millex-HA Filter, 0.45 µm | EMD Millipore | SLHA02510 | Filter to clear nematode lysate |
| Eppendorf LoBind microcentrifuge tubes Protein 1.5 mL | Sigma-Aldrich | 22431081 | Minimise protein loss |
| 2 mL microfuge tube | Ambion | AM12425 | Yeast extract preparation and other applications |
| 5 mL tube | Thermo Fisher Scientific | 129A | Collection of HEK293 cell lysate |
| 15 mL tube | Sarstedt | 62.547.254 | Collection C. elegans |
| 50 mL plastic tube | Sarstedt | 62.554.502 | Collection yeast cells |
| DMEM, high glucose, pyruvate | Thermo Fisher Scientific | 41966 | Media for culturing HEK293 cells |
| Penicillin-Streptomycin | Sigma-Aldrich | P4333 | Used to supplement cell culture media to control bacterial contamination |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F7524 | Used to supplement cell culture media |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| RQ1 RNase-Free DNase | Promega | M6101 | DNAse I to degrade DNA from cell lysate |
| RNasin Plus RNase Inhibitor | Promega | N2611 | RNase inhibitor to avoid RNA degradation |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitor cocktail to avoid protein degradation |
| Dynabeads Oligo (dT)25 | Thermo Fisher Scientific | 61011 | Magnetic beads required for the first step of mRNA-protein complex purification |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | Magnetic beads required for the second step of mRNA-protein complex purification |
| E. coli tRNA | Sigma-Aldrich | 10109550001 | transfer RNA from E. coli used for blocking streptavidin beads and avoid unsepecific interactions |
| Quick Start Bradford 1x Dye Reagent | BioRad | 5000205 | To determine protein concentration within lysates, using BSA as reference |
| Bovine Serum Albumin | Sigma | A7906-100G | Used to perform the standard curve that will be used as reference for protein concentration determination |
| mouse anti-Act1 | MP Biomedicals | 869100 | Antibody for detection of yeast actin in Western blot (1:2,500) |
| mouse anti-Act1 | Sigma | A1978 | Antibody for detection of human actin in Western blot (1:2,000) |
| mouse anti-HuR | Santa Cruz | sc-5261 | Antibody for detection of HuR in Western blot (1:500) |
| mouse anti–CYC-1 | Invitrogen | 456100 | Antibody for detection of Cyc-1 in Western blot (1:1,000) |
| peroxidase anti-peroxidase soluble complex | Sigma | P1291 | Detection of Pfk2:TAP in Western blot (1:5,000) |
| HRP-conjugated sheep anti-mouse IgG | Amersham | NXA931 | HRP-coupled secondary antibody |
References
- Mitchell, S. F., Parker, R. Principles and properties of eukaryotic mRNPs. Molecular Cell. 54 (4), 547-558 (2014).
- Iadevaia, V., Gerber, A. P. Combinatorial Control of mRNA Fates by RNA-Binding Proteins and Non-Coding RNAs. Biomolecules. 5 (4), 2207-2222 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Matia-González, A. M., Gerber, A. P., Sesma, A., von der Haar, T. Ch. 14. Fungal RNA Biology. , 347-370 (2014).
- Tsvetanova, N. G., Klass, D. M., Salzman, J., Brown, P. O. Proteome-wide search reveals unexpected RNA-binding proteins in Saccharomyces cerevisiae. PLoS One. 5 (9), (2010).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Castello, A., et al. System-wide identification of RNA-binding proteins by interactome capture. Nature Protocols. 8 (3), 491-500 (2013).
- Liao, Y., et al. The Cardiomyocyte RNA-Binding Proteome: Links to Intermediary Metabolism and Heart Disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Liepelt, A., et al. Identification of RNA-binding Proteins in Macrophages by Interactome Capture. Molecular Cell Proteomics. 15 (8), 2699-2714 (2016).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212 (2016).
- Matia-Gonzalez, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural and Molecular Biology. 22 (12), 1027-1033 (2015).
- Mitchell, S. F., Jain, S., She, M., Parker, R. Global analysis of yeast mRNPs. Nature Structural and Molecular Biology. 20 (1), 127-133 (2013).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127 (2015).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128 (2016).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42 (2016).
- Reichel, M., et al. In Planta Determination of the mRNA-Binding Proteome of Arabidopsis Etiolated Seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Koster, T., Marondedze, C., Meyer, K., Staiger, D. RNA-Binding Proteins Revisited - The Emerging Arabidopsis mRNA Interactome. Trends Plant Sciences. 22 (6), 512-526 (2017).
- Hamasaki, K., Killian, J., Cho, J., Rando, R. R. Minimal RNA constructs that specifically bind aminoglycoside antibiotics with high affinities. Biochemistry. 37 (2), 656-663 (1998).
- Bachler, M., Schroeder, R., von Ahsen, U. StreptoTag: a novel method for the isolation of RNA-binding proteins. RNA. 5 (11), 1509-1516 (1999).
- Vazquez-Pianzola, P., Urlaub, H., Rivera-Pomar, R. Proteomic analysis of reaper 5' untranslated region-interacting factors isolated by tobramycin affinity-selection reveals a role for La antigen in reaper mRNA translation. Proteomics. 5 (6), 1645-1655 (2005).
- Hartmuth, K., Vornlocher, H. P., Luhrmann, R. Tobramycin affinity tag purification of spliceosomes. Methods Molecular Biology. 257, 47-64 (2004).
- Beach, D. L., Keene, J. D. Ribotrap : targeted purification of RNA-specific RNPs from cell lysates through immunoaffinity precipitation to identify regulatory proteins and RNAs. Methods Molecular Biology. 419, 69-91 (2008).
- Slobodin, B., Gerst, J. E. A novel mRNA affinity purification technique for the identification of interacting proteins and transcripts in ribonucleoprotein complexes. RNA. 16 (11), 2277-2290 (2010).
- Slobodin, B., Gerst, J. E. RaPID: an aptamer-based mRNA affinity purification technique for the identification of RNA and protein factors present in ribonucleoprotein complexes. Methods Molecular Biology. 714, 387-406 (2011).
- Yoon, J. H., Gorospe, M. Identification of mRNA-Interacting Factors by MS2-TRAP (MS2-Tagged RNA Affinity Purification). Methods Molecular Biology. 1421, 15-22 (2016).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Blencowe, B. J., Sproat, B. S., Ryder, U., Barabino, S., Lamond, A. I. Antisense probing of the human U4/U6 snRNP with biotinylated 2'-OMe RNA oligonucleotides. Cell. 59 (3), 531-539 (1989).
- Lingner, J., Cech, T. R. Purification of telomerase from Euplotes aediculatus: requirement of a primer 3' overhang. Proceedings of the National Academy of Sciences U S A. 93 (20), 10712-10717 (1996).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Molecular Cell Proteomics. 12 (6), 1539-1552 (2013).
- Imig, J., et al. miR-CLIP capture of a miRNA targetome uncovers a lincRNA H19-miR-106a interaction. Nature Chemical Biology. 11 (2), 107-114 (2015).
- Chu, C., et al. Systematic discovery of Xist RNA binding proteins. Cell. 161 (2), 404-416 (2015).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Rogell, B., et al. Specific RNP capture with antisense LNA/DNA mixmers. RNA. 23 (8), 1290-1302 (2017).
- Matia-Gonzalez, A. M., Iadevaia, V., Gerber, A. P. A versatile tandem RNA isolation procedure to capture in vivo formed mRNA-protein complexes. Methods. , 93-100 (2017).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic Acids Research. 36 (Web Server issue), W70-W74 (2008).
- Kalendar, R., Khassenov, B., Ramankulov, Y., Samuilova, O., Ivanov, K. I. FastPCR: An in silico tool for fast primer and probe design and advanced sequence analysis. Genomics. 109 (3-4), 312-319 (2017).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Kedde, M., et al. A Pumilio-induced RNA structure switch in p27-3' UTR controls miR-221 and miR-222 accessibility. Nature Cell Biology. 12 (10), 1014-1020 (2010).
- Schumacher, B., et al. Translational repression of C. elegans p53 by GLD-1 regulates DNA damage-induced apoptosis. Cell. 120 (3), 357-368 (2005).
- Ziegeler, G., et al. Embryonic lethal abnormal vision-like HuR-dependent mRNA stability regulates post-transcriptional expression of cyclin-dependent kinase inhibitor p27Kip1. Journal of Biological Chemistry. 285 (20), 15408-15419 (2010).
- Itri, F., et al. Femtosecond UV-laser pulses to unveil protein-protein interactions in living cells. Cellular and Molecular Life Sciences. 73 (3), 637-648 (2016).
- Zhang, L., Zhang, K., Prandl, R., Schoffl, F. Detecting DNA-binding of proteins in vivo by UV-crosslinking and immunoprecipitation. Biochemical and Biophysical Research Communications. 322 (3), 705-711 (2004).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved