Method Article
Una procedura di isolamento del RNA basati su Oligonucleotide Tandem recuperare mRNA-proteina eucariotica complessi
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
È descritta una procedura di isolamento di RNA (viaggio) tandem per il recupero dei complessi in modo endogeno formato mRNA-proteina. In particolare, complessi RNA-proteina sono reticolate in vivo, poliadenilazione RNAs sono isolati dagli estratti con i branelli di oligo e particolare mRNA vengono catturati con oligonucleotidi antisenso RNA modificati. Proteine legate a mRNA vengono rilevati dall'analisi del immunoblot.
Abstract
Le proteine RNA-leganti (RBPs) giocano un ruolo chiave nel controllo post-trascrizionale dell'espressione genica. Pertanto, caratterizzazione biochimica dei complessi di mRNA-proteina è essenziale per comprendere il regolamento mRNA derivato da proteine interagenti o RNA non codificanti. Qui, descriviamo una procedura di isolamento di RNA di tandem (viaggio) che permette la purificazione di complessi proteina mRNA in modo endogeno formato da estratti cellulari. Il protocollo in due fasi comporta l'isolamento di poliadenilazione mRNA con oligo antisenso perline e successiva acquisizione di un mRNA di interesse con 3'-biotinylated 2'-O-metilato RNA antisenso, che può quindi essere isolato con Perline di streptavidina. VIAGGIO è stato utilizzato per recuperare in vivo complessi di mRNA-ribonucleoproteina (mRNP) di reticolato da lievito, nematodi e cellule umane per ulteriori analisi di RNA e proteine. Così, il viaggio è un approccio versatile che può essere adattato a tutti i tipi di poliadenilazione RNAs attraverso organismi per studiare la dinamica riassetto della mRNPs imposto da segnali intracellulari o ambientale.
Introduzione
Regolazione genica post-trascrizionale è guidato attraverso le interazioni tra le proteine RNA-leganti (RBPs), non-codificazione RNAs (ncRNAs) e mRNA, che dirigere l'elaborazione, localizzazione, traduzione e degradazione di ogni trascrizione all'interno di cellule1 , 2. l'identificazione del RBPs e ncRNA interagendo con particolare mRNA, stabilire ciò che è noto come ribonucleoproteina complessi/particelle (RNP), è quindi la chiave per comprendere il destino del controllo di espressione di mRNA e gene. Due approcci complementari sono intrapresi per caratterizzazione biochimica di RNP complessi3,4: mentre l'approccio "proteina-centrica" si basa sulla purificazione del RBPs specifico, l'approccio "RNA-centrica" implica la isolamento di sottopopolazioni o singoli RNAs e successiva analisi delle proteine interagenti o RNAs. Recentemente, un approccio incentrato sul RNA per l'identificazione del repertorio RBP interagendo con poliadenilazione (poly(A)) RNA5 è diventato sempre più popolare incorporando un passo di reticolazione luce ultravioletto (UV) per stabilizzare la proteina-RNA Priore di interazioni l'isolamento di mRNA, che drammaticamente esteso il catalogo di RBPs in cellule umane6,7,8,9,10,11, Caenorhabditis elegans 12, saccharomyces cerevisiae12,13,14e altri organismi15,16,17,18, 19,20. Tuttavia, la mappatura delle proteine e/o ncRNAs assemblati in particolare trascrizioni è ancora una grande sfida. A tal fine, sono attualmente utilizzati due approcci principali: da un lato, RNA aptamero tag sono fuse al RNA di interesse per consentire la purificazione di affinità. In tal modo, il RNA aptameri legano ad alta affinità di antibiotici aminoglicosidici tra cui tobramicina e streptomicina21,22,23,24, o di proteine, come la proteina del cappotto dalla R17/MS2 batteriofago o batteriche streptavidina S125,26,27,28,29. Anche se questo approccio è stato indicato per essere relativamente robusto e versatile, richiede la clonazione al design il RNA sotto inchiesta e così non può essere utilizzato per catturare i mRNAs naturali. D'altra parte, oligonucleotidi antisenso (ASOs) sono stati utilizzati fin dall'inizio per il recupero e caratterizzazione di altamente espresso nativo RNP30,31 e virali RNA32. Più recentemente, ASOs sono stati applicati per catturare lungo non codificanti RNA33,34,35 e in vitro formata mRNPs36. Uno dei principali fattori limitano con tutti gli approcci incentrati sul RNA è il numero di copia del RNA di interesse, rendendo il recupero dei mRNAs basso-espresso più problematico. Questa limitazione può essere superata mediante upscaling la reazione; Tuttavia, questo può potenzialmente portare ad aumento sullo sfondo.
Qui, descriviamo il nostro tandem ASO-based sviluppata di recente procedura di isolamento del RNA (viaggio) per isolare i complessi di mRNA-proteina nativa con alta selettività37 (Figura 1). Il protocollo prevede due passaggi di purificazione ASO sequenziale, vale a dire l'isolamento di mRNA di poli (a) con oligo commercialmente disponibile accoppiati perline seguite da acquisizione di specifici mRNA utilizzando specificamente progettato biotinilati breve 2'-metossi ASOs RNA. Questa procedura in due fasi aiuta eliminando proteine contaminanti e aggiunge opportunità per aggiustamenti e ottimizzazione. In questo caso, viaggio è stato applicato il mRNA selezionati da lievito, nematodi, e cellule umane per confermare in vivo formano complessi mRNA-proteina.
Protocollo
1. progettare oligonucleotidi antisenso (ASOs)
- Analizzare la struttura secondaria di mRNA o di un frammento della stessa mediante strumenti online disponibili38. Quindi, immettere la sequenza di nucleotidi (nt) nella casella vuota. Nella finestra opzioni di base, selezionare minimo l'energia libera (MFE) e funzione di partizione ed evitare isolato paia di basi. Nella casella Opzioni di output, selezionare grafico di struttura secondaria del RNA interattiva e infine fare clic sul pulsante procedi . Un nuova finestra pop-up che visualizza la struttura secondaria del mRNA di interesse.
- Selezionare almeno tre differenti 21-24 nts lunghe sequenze all'interno di mRNA di interesse, preferenzialmente nelle regioni carenti strutture secondarie rilevabile (ad es., nei cicli non strutturati) e in 3' non tradotta (UTR) regioni.
Nota: Si è osservato che ASOs ricottura alle sequenze delle regioni 3' non tradotta (UTR) risultati migliori. Una possibilità è che i ribosomi associati alla sequenza di codificazione (CDS) occasionalmente possono ostacolare la ricottura di ASOs. ASOs ricottura a 5' UTR non sono stati testati.- Selezionare le regioni con un tasso di guanidina/citosina vicino al 50% e carente tandem del nucleotide ripete per evitare la potenziale formazione di forcine o auto-ricottura.
- Progettazione 2'-metossi modificato manualmente oligonucleotidi da RNA recanti una molecola di biotina al 3', pienamente complementare alle regioni selezionate (punto 1.2) entro il desiderato mRNA di destinazione.
Nota: 2'-metossi modifiche rendono il RNA resistente ai cellulari RNasi e aumenta il duplex temperatura di fusione, che permette lavaggio rigorosa. La frazione di biotina è necessaria per l'acquisizione di ASOs con streptavidina.- Regolare la temperatura di fusione degli ibridi RNA a ~ 60-65 ° C e garantire che ha una complessità di alta sequenza linguistica (> 60%) come determinato con idonei strumenti online39.
- Utilizzare lo strumento di ricerca di allineamento locale base40 per la ricerca di potenziali ASO cross-ibridazione con altri mRNAs in trascrittoma. Selezionare Nucleotide BLAST, inserire le sequenze nella casella vuota e scegliere l'organismo di interesse. Mantenere i parametri rimanenti come predefinito e fare clic su BLAST.
Nota: Anche parziale allineamento continuo di 8-10 nts può portare a croce-ibridazione e recupero di questo mRNA.
2. cultura, irradiazione UV e delle cellule delle cellule Lisi
Nota: In seguito, la procedura è descritta per S.cerevisiae, nematodi c. eleganse cellule embrionali umane del rene (HEK293). Tuttavia, può essere adattato ad altri organismi pure, anche se in modo esplicito, non è stato testato ancora.
- S. cerevisiae
- Crescere le cellule di lievito (ad es., ceppo BY4743 derivato; MATa/α his3Δ1/his3Δ1 leu2Δ0/leu2Δ0 LYS2/lys2Δ0 met15Δ0/MET15 ura3Δ0/ura3Δ0 PFK2:TAP / PFK2) in 500 mL di terreno YPD (Estratto di lievito 1%, 2% di peptone, 2% D-glucosio) a 30 ° C con agitazione costante a 220 giri/min.
- Raccogliere le cellule in fase di Mid-registro (densità ottica a 600 nm (OD)600 ~ 0,6) di filtrazione con filtri di nylon 0,45 µm o mediante centrifugazione a 3.000 × g per 2 minuti a temperatura ambiente (TA). Scartare il flusso continuo o il supernatante, rispettivamente.
- Lavare le cellule tre volte con 25 mL di fisiologica tamponata fosfato (PBS) utilizzando il filtro in nylon da 0,45 µm o raccogliere mediante centrifugazione a 3.000 × g per 2 min a RT e scartare il surnatante. Raccogliere le cellule più velocemente possibile come lo stress imposto poteva avere un impatto sulle interazioni RNA-proteina.
- Risospendere le cellule in 25 mL di PBS e poi versare la sospensione in un 15cm di Petri. Mettere la teglia sul ghiaccio, rimuovere il coperchio ed esporre le cellule tre volte con 400 mJ cm-2 della luce UV di 254 nm in un UV-reticolante con interruzioni di 2 min su ghiaccio tra ogni ciclo di esposizione con miscelazione delicata.
- Trasferire le cellule in una provetta 50 mL e raccogliere le cellule mediante centrifugazione a 3.000 × g per 3 min a 4 ° C. Scartare il surnatante e tenere il pellet.
Nota: Le cellule possono essere snap congelati in azoto liquido e conservato a-80 ° C. - Risospendere le cellule in 4 mL di tampone di lisi refrigerati A (LB-A; 100 mM Tris-HCl, pH 7.5, 500mm LiCl, 10 mM EDTA, 1% Triton X-100, 5 mM DTT, 20 U ml-1 DNasi, 100 U ml-1 RNasin, completo privo di EDTA cocktail di inibitore della proteasi).
- Trasferire le cellule in due provette da 2 mL. Aggiungi max. Volumi ⅔ di refrigerati perle di vetro e frantumare le cellule in un lyser tessuto a 30 Hz per 10 min a 4 ° C.
- Fare un buco nel fondo del tubo con un ago caldo e trasferire il lisato ad un tubo fresco 1,5 mL mediante centrifugazione a 600 × g per 30 s.
- Deselezionare il lisato di tre centrifugazioni sequenziale a 4 ° C a 3.000 × g per 3 minuti, poi 5.000 × g e 10.000 × g per 5 minuti ciascuno.
Nota: Gli estratti possono essere snap-congelati in azoto liquido e conservati a-80 ° C.
- C. elegans
- Vermi di Bristol N2 di cultura a 20 ° C sulle piastre di Nematode crescita medio (NGM) (0,3% NaCl, agar 1,7%, 0,25% di peptone, 1 mM CaCl2, colesterolo di 5 µ g/mL, 1 mM MgSO4, 25mm KPO4 tampone pH 6.0) seminati con il ceppo OP50 Escherichia coli 41 .
- Aggiungere 5 mL di tampone di M941 (0,3% KH2PO4, 0,6% Na2HPO4, 0.5% NaCl, 1 mM MgSO4) per ciascuna piastra, agitare la piastra per risospendere i vermi e trasferire i vermi in una provetta da 15 mL. Mettere 2 µ l della sospensione in una diapositiva e contare i vermi in sospensione con un microscopio. Quindi applicare il fattore per calcolare il numero di vermi al piatto.
- Raccogliere ~ 120.000 vermi mediante centrifugazione a temperatura ambiente (400 × g, 2 min, RT) e scartare il surnatante.
- Lavare i vermi tre volte. Di conseguenza, aggiungere 10 mL di tampone di M9, miscelare capovolgendo e raccogliere i vermi mediante centrifugazione a 400 × g per 2 minuti a TA.
- Aggiungere 15 mL di buffer di M9 i vermi e posizionarlo su una ruota orientabile per 15 minuti a TA.
- Trasferire i vermi piatti NGM (~ 4.000 vermi al piatto) ed esporre alla luce UV (254 nm) a 300 mJ cm-2 in un UV-reticolante.
- Aggiungere 5 mL di tampone di M9 direttamente alla piastra e scuotere la piastra per risospendere i vermi nel buffer. Trasferire la sospensione con una pipetta per un tubo da 15 mL e raccogliere i vermi mediante centrifugazione a 400 × g per 2 minuti a TA.
- Risospendere i vermi in 2 mL di tampone di lisi B (LB-B; 100 mM Tris-HCl, pH 8.0, 150 mM NaCl, 1 mM EDTA, 0,75% IGEPAL, 1 millimetro DTT, 20 U mL-1 DNasi, 100 U mL-1 RNasin, completo privo di EDTA cocktail di inibitore della proteasi).
- Macinare i vermi in un mortaio riempito con azoto liquido. Raccogliere la polvere in un tubo da 50 mL e disgelo a TA.
Nota: Azoto liquido dovrà essere gestito secondo le procedure di sicurezza (ad es., fume hood e sicurezza guanti) - Deselezionare il lisato compreso lo strato di grasso mediante centrifugazione a 14.000 × g per 10 min e successivamente passare il chiarificato lisato attraverso un filtro di 0,45 μm con una siringa.
- Cellule umane coltivate
Nota: La seguente descrizione si basa su trasfezione transiente di cellule HEK293 con pGL3-CDKN1B-3'UTR plasmide del reporter del che esprime il 3′UTR di CDKN1B/27 a valle del firefly luciferase gene42.- Cellule HEK293 cultura dell'Aquila per volta di Dulbecco Medium (DMEM) contenenti glucosio 25 mM e 1 mM sodio piruvato, completati con 100 U-1 mL di penicillina, la streptomicina µ g 100 mL-1 e 10% siero bovino fetale (FBS) e incubare a 37 ° C in un camera umidificata (incubatore) contenente 5% CO2.
- Sementi di ~ 3 × 106 HEK293 cellule in una coltura di tessuti standard 10 cm piatto il giorno prima la transfezione. Contare le celle con un emocitometro.
- Mescolare 2 µ g del gene reporter (ad es., pGL3-p27-3'UTR) con 20 µ l di reagente di transfezione e transfect le cellule al 70% confluency (~ 7 × 106 cellule).
- Posizionare le cellule in un incubatore a 37 ° C per ulteriori 48 h prima della raccolta.
- Rimuovere il supporto con una pipetta sierologica e lavare rapidamente le cellule due volte con 10 mL di PBS pre-riscaldato a 37 ° C. Rimuovere PBS con una pipetta sierologica.
- Aggiungere 6 mL di PBS al piatto, mettere sul ghiaccio e poi esporre le cellule ai raggi UV (254 nm) a 100 mJ cm-2 in un UV-reticolante.
Nota: La piastra deve essere tenuta su ghiaccio durante l'esposizione ai raggi UV. - Raschiare le cellule (in totale ~ 107 cellule) in PBS e trasferirlo in una provetta da 15 mL.
- Rotazione verso il basso le cellule a 250 × g per 10 min a 4 ° C e quindi rimuovere il surnatante con una pipetta.
Nota: Il pellet cellulare può essere snap-congelati in azoto liquido e conservato a-80 ° C fino all'utilizzo. - Risospendere le cellule in 2 mL di tampone di lisi pre-refrigerati (LB-A) pipettando su e giù per 5 - 6 volte, mantenendo il tubo sul ghiaccio.
- Trasferire con una pipetta lisato in una provetta di 5ml posizionata su ghiaccio.
- Sottoporre il lisato a tre giri di sonicazione composto da 20 s raffica a 10 μm ampiezza con 30 s di raffreddamento periodi sul ghiaccio.
Nota: Sonicazione è raccomandato come completa la lisi delle cellule e frammenti del DNA. - Trasferire il lisato per provette da 2 mL e centrifugare a 15.000 × g per 10 min a 4 ° C. Raccogliere il surnatante (= estrazione) e trasferirlo in una nuova provetta. Rimuovere un campione analitico (5-10%) per ulteriori analisi di proteine e RNA.
Nota: Questo passaggio è fondamentale per rimuovere eventuali detriti residui delle cellule e può essere ripetuto.
3. primo passo: Poli (a) RNA isolamento
- Equilibrare 1 mg di oligo25-accoppiato biglie magnetiche in 500 µ l di buffer di lisi rispettivi.
- Combinare 5 mg, 10 mg o 4 mg (proteina) di S. cerevisiae, c. elegans o HEK293 estratti, rispettivamente, con 1 mg di oligo25 -accoppiato biglie magnetiche. Mescolare i campioni vigorosamente per 10 min a 25 ° C in un mixer.
Nota: Le concentrazioni di proteina degli estratti possono essere determinate con analisi di Bradford utilizzando albumina di siero bovino (BSA) come uno standard di riferimento. L'agitazione vigorosa di campioni previene la sedimentazione di perline. Per valutare la specificità dell'isolamento di mRNA, un esperimento di controllo possono essere eseguito in parallelo con l'aggiunta di un eccesso (20 µ g) degli acidi polyadenylic (pA). - Mettete i tubi su un supporto magnetico per 10 s e rimuovere il surnatante. Mantenere il supernatante in ghiaccio per successive fasi di recupero (punto 3.7).
- Aggiungere 500 µ l di lavaggio tampone A (10 mM Tris-HCl, pH 7.5, 600mm LiCl, 1 mM EDTA, 0,1% Triton X-100) per le perline e vortexare per 5 s.
Nota: La concentrazione di LiCl nei buffer di lavaggio possa variare. 600 mM LiCl è consigliato, ma le concentrazioni più basse sono stati anche testate (500 mM per c. elegans, 300 mM per HEK293). - Raccogliere le perle con un magnete e poi lavare le perle due volte con 500 µ l di tampone di lavaggio B (WB; 10 mM Tris-HCl, pH 7.5, 600mm LiCl, 1 mM EDTA).
- Eluire il RNA in 30 µ l di 10 mM Tris-HCl, pH 7.5 a 80 ° C per 2 min con continua agitazione (1.000 giri/min) in un mixer. Immediatamente posizionare il tubo sul supporto magnetico e raccogliere l'eluato dopo 10 s. salvare le perline per turni aggiuntivi di purificazione.
Nota: Raccogliere l'eluato più rapidamente possibile per evitare potenziali riassociazione dei mRNAs per le perline a temperature più basse. - Aggiungere le perline dal passaggio precedente il surnatante raccolto al punto 3.3 e ripetere l'acquisizione, il lavaggio e la eluizione dei mRNAs due volte (passaggi 3.3-3.6). Eluati da ripetuti colpi vengono quindi combinati e possono essere conservati a-80 ° C.
4. secondo passo: l'acquisizione di specifici mRNA con 3'-Biotinylated 2'-metossi oligonucleotidi da RNA antisenso Modified
Nota: Per verificare l'idoneità di ASOs, la seguente procedura può essere eseguita con RNA totale isolato dalle cellule.
- Equilibrare 30 µ l di biglie magnetiche streptavidina-accoppiati in 1 mL di associazione e tampone (buffer B & W; 10 mM Tris-HCl, pH 7.5, 150 mM NaCl, 0,5 mM EDTA, pH 8.0) di lavaggio contenente 0,1 mg mL-1Escherichia coli RNA transfer (tRNA) in un rotatore per 1 h a RT.
- Lavare le perle tre volte con 750 µ l di tampone B & W.
- Risospendere le sfere in 30 µ l di tampone B & W e tenere il ghiaccio fino all'utilizzo.
- Diluire ~ 35 µ g di proteina totale dall'isolamento di poli (a) precedente (Vedi 3) in 100 µ l di tampone B & W in una provetta da 1,5 mL.
Nota: Per testare la specificità di ASOs con RNA totale, aggiungere 600 ng di RNA totale purificato a 100 µ l di tampone B & W. - Aggiungere 200 pmol del rispettivo ASO e incubare a 70 ° C per 5 min.
Nota: La temperatura elevata consente di risolvere strutture secondarie nel RNA facilitando la ricottura dell'ASO. - Rimuovere l'intero blocco di calore dal dispositivo e posizionarlo a temperatura ambiente per 10 min raffreddare lentamente.
- Aggiungere 30 µ l di biglie magnetiche streptavidina-accoppiato equilibrati dal punto 4.1 al campione.
- Incubare la miscela per 30 minuti a 25 ° C con agitazione costante a 950 giri/min in un miscelatore.
Nota: Si consiglia di scorrere i tubi ogni 10 min per evitare la sedimentazione di perline. - Disporre le provette sul supporto magnetico, eliminare il surnatante e lavare le perle tre volte con 750 µ l di tampone di B & W pre-riscaldato a 55 ° C.
Nota: La temperatura di lavaggio ottimale può differire/variare leggermente tra diversi ASOs. Si raccomanda di regolare questa condizione prima del RNA totale non reticolato che effettua l'esperimento con estratti cellulari. - Eluire il RNA in 20 µ l di 10 mM Tris-HCl, pH 7.5 a 90 ° C per 10 minuti con agitazione costante a 950 giri/min in un miscelatore. Posizionare le provette sul supporto magnetico e subito raccogliere l'eluato.
Nota: Per l'analisi della proteina, aggiungere 20 µ l di tampone di Laemmli × 1 ai talloni e incubare a 95 ° C per 5 min. proteine sono monitorati dall'analisi del immunoblot seguendo protocolli di laboratorio standard.
Risultati
Abbiamo sviluppato una strategia di isolamento basati su ASO RNA, chiamata viaggio, per catturare i mRNAs particolari con loro proteine rilegate da tre organismi diversi37. Essenzialmente, complessi RNA-proteina sono stati reticolati in vivo di irradiazione UV delle cellule a 254 nm e poli (a) RNA sono stati recuperati con i branelli magnetici accoppiato commercialmente disponibili oligo (dT), quindi il mRNA di interesse è stato isolato con 3'-biotinylated 2-0'-metossi per volta oligonucleotidi antisenso RNA (Figura 1). Abbiamo quindi progettato diversi 21-24 nts per volta ASOs con pieno-complementarità alle regioni in mRNA selezionati da lievito, c. elegans e umani e testato la loro idoneità per recuperare il mRNA di interesse (un elenco di primer e ASOs è dato in tabella 1). l'efficienza e la specificità dei singoli ASOs in primo luogo sono stati valutati con RNA totale non reticolato isolato dall'organismo rispettivo. In questi esperimenti, RNA ASOs erano accoppiati alla streptavidina coniugata paramagnetici perline e incubati con il RNA totale preparato dall'organismo corrispondente non reticolato. Dopo il rilascio dei mRNAs catturati dalle perle, la presenza di mRNA destinato anche come indipendente controllo mRNA è stata monitorata da trascrizione inversa (RT)-polimerasi reazione a catena (PCR)37. Abbiamo notato che due variabili, la concentrazione di sale e la temperatura di wash buffer, giocato ruoli critici nell'efficienza della cattura di PFK2 mRNA da lievito che è stato testato con tre differenti ASOs (Figura 2A). Abbassando la concentrazione di sale (NaCl) a 25 mM ridotto il recupero del negativo il controllo mRNA (PFK1) a livelli non rilevabili con ASOs tutti, ma inoltre ha ridotto il recupero del desiderato mRNA di destinazione PFK2 (10-15% dell'input). Al contrario, un aumento delle concentrazioni sale a livelli fisiologici (150 mM NaCl) aumentato il recupero dei mRNAs PFK2 fino al 75% con ASO PFK2-2, superiore a quella del controllo PFK1 mRNA da almeno 5 volte (Figura 2A). Di ulteriore nota, la diversa ASOs ha mostrato grande variazione di mRNA efficacies di acquisizione del bersaglio alle sale-concentrazioni fisiologiche, sottolineando la necessità di validazione empirica di ASOs. La dipendenza di recupero mRNA la temperatura del tampone di lavaggio è esemplificata per c. elegans cep-1 e lievito RPS20 mRNA, utilizzando i rispettivi ASOs (tabella 1). Abbiamo osservato che la temperatura di lavaggio ottimale era tra 50 ° C e 55 ° C, come è evidente dal fondo basso con mRNA non correlati e il recupero efficiente dei mRNAs bersaglio (Figura 2B). A questo punto, vogliamo sottolineare la possibilità di cross-ibridazione di ASOs con altri mRNA. Ad esempio, l'ASO DNM1 è pienamente complementare ad una sequenza all'interno di DNM1 sequenza di codificazione, ma anche parzialmente accoppia con ACT1 mRNA. DNM1 ASOs recuperato entrambi mRNAs indipendentemente la temperatura di lavaggio, mostrando forte propensione per cross-ibridazione (Figura 2). Infine, abbiamo usato le prove descritte sopra e ottimizzazioni per selezionare tre ASOs che erano adatti per il recupero di mRNA bersaglio rispettivi da total RNA isolato da S. cerevisiae, c. elegans e cellule umane (Figura 2D ).
Dopo la selezione iniziale di adatto ASOs in vitro, abbiamo effettuato il viaggio con estratti di cellule ottenuti da UV-reticolati organismi/cellule (Figura 1). In particolare, abbiamo testato il recupero di tre differenti mRNA da tre organismi diversi: PFK2 mRNA dal lievito S. cerevisiae, cep-1 dal nematode c. elegans e un reporter mRNA (pGL3-CDKN1B-3'UTR) recanti il 3' UTR sequenza di mRNA umano CDKN1B/p27 fusa ad un reporter di luciferase (luc) per espressione transiente in cellule HEK293 umane. Per controllare la specificità dell'isolamento del RNA, abbiamo monitorato il recupero di alcuni mRNA non correlati, e abbiamo eseguito esperimenti di competizione con l'aggiunta di un eccesso di pA di estratti cellulari, che compete con il legame di mRNA cellulare da oligo25 perline durante la prima fase di purificazione di passaggio. Come precedentemente visto con campioni di RNA totali non reticolato (Figura 2), RT-PCR ha confermato l'arricchimento dei rispettivi mRNA target mRNA durante il viaggio, considerando che alcuni non correlato a controllo mRNA non sono stati arricchiti (Figura 3A). Inoltre, nessuno dei due mRNAs sono stati rilevati su perline senza ASOs, indicando le opportune procedure di bloccare che evitare l'associazione aspecifici. Su questa linea, ASOs estranei/uova strapazzate può anche essere utilizzato come controllo, anche se il potenziale per cross-ibridazione con alcuni mRNA e la cattura dedotta di proteine rilegate deve quindi essere presi in considerazione. Poiché proteine nell'eluito finale potrebbero essere difficilmente fruiti su argento-macchiate di gel di poliacrilammide (dati non mostrati), la presenza di proteine interagenti mRNA precedentemente note più ulteriormente è stata valutata dall'analisi del immunoblot. Questo include Pfk2p da S. cerevisiae, che si lega selettivamente al PFK2 mRNA in un modo indipendente ribosoma12; C. elegans GLD-1, un canonico RBP che associa al 3' sequenze UTR di cep-1 mRNA per regolazione traduzionale43; e HuR, un RBP che regola la stabilità del mRNA e traduzione di p27/CDKN1B mRNA44. Come previsto, tutte queste proteine sono state identificate negli eluati di viaggio dei rispettivi mRNA mediante analisi Western Blot (Figura 3B).
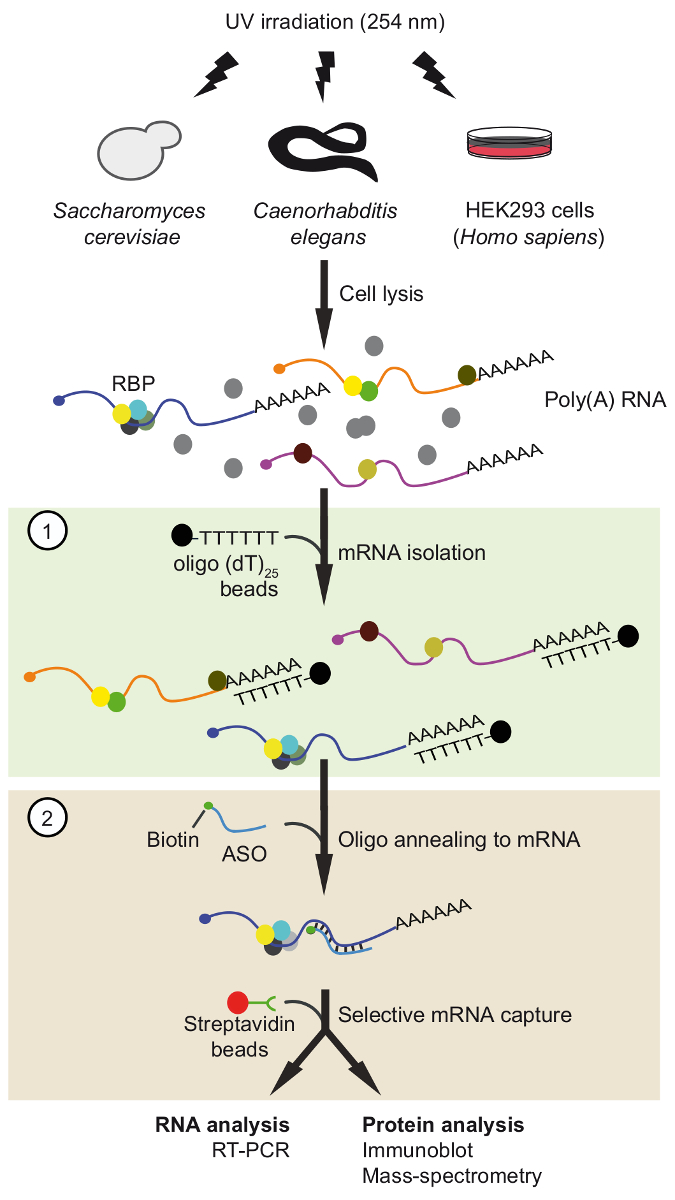
Figura 1. Rappresentazione schematica del viaggio. Le proteine sono reticolate di RNA in vivo di irradiazione UV. Nel primo passaggio (scatola verde chiaro), poli (a) della RNA-proteina complessi vengono recuperati con oligo25 perline applicando condizioni rigorose lavaggio per rimuovere proteine non associati. Nella seconda fase (scatola rosa), la destinazione mRNP è estrarre con oligonucleotidi RNA antisenso biotinilato e streptavidina perline. Il mRNPs purificati vengono poi analizzati mediante RT-PCR e immunoblot /-spettrometria di massa (MS) per identificare RNA e proteine che interagiscono con il mRNA di interesse, rispettivamente. La figura è stata modificata dalla precedente pubblicazione37 con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
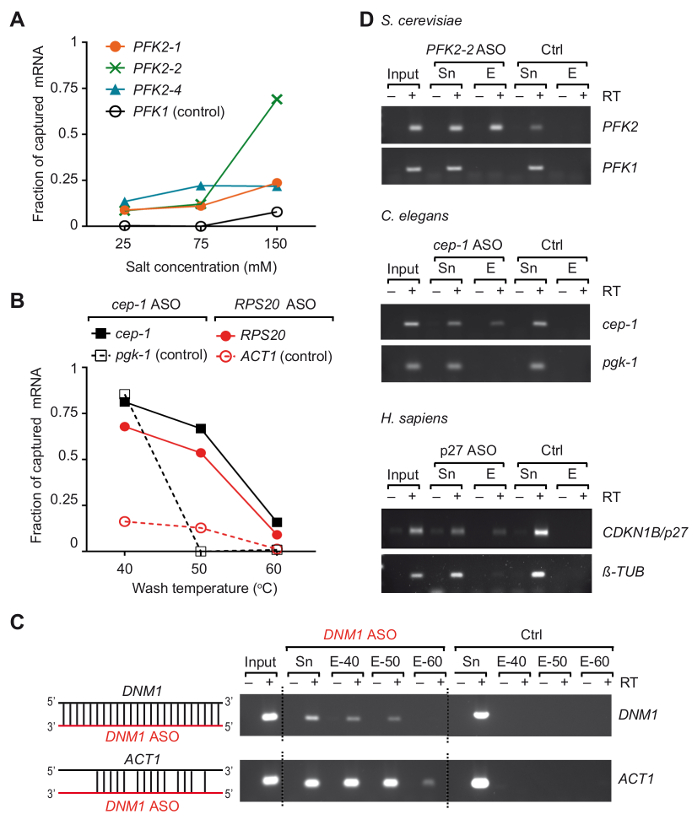
Figura 2. Per l'isolamento dei mRNAs selezionati da RNA totale non reticolato cellule/organismi con volta antisenso RNA cattura sonde. (A) impatto delle concentrazioni saline sull'efficienza di cattura del lievito PFK2 mRNA con ASOs. RNA totale dalle cellule di lievito è stato combinato con il ASOs indicato e lavata con un tampone contenente la specificato concentrazione di sale (NaCl) a 55 ° C. Verde, blue e arancione linee rappresentano PFK2 recupero di mRNA con ASOs differenti come determinato mediante RT-PCR37: PFK2-1 e PFK2 -2 tempri nel 3' UTR, PFK2-4 i CD. PFK1 è un negativo controllo mRNA. PCR è stata eseguita con 32 cicli di amplificazione e quantificati come descritto in precedenza37. (B) frazione di c. eleganscep-1 e lievito RPS20 ASO legata mRNAs alle temperature di lavaggio diverso, rappresentate nelle linee di nere e rosse, rispettivamente. C. eleganspgk-1 e lievito ACT1 mRNA non-bersaglio (controllo). 30 e 32 cicli PCR sono stati applicati per il rilevamento di lievito e mRNA di c. elegans , rispettivamente. (C) rappresentazione schematica dell'ibridazione di DNM1 ASO (rosso) con sequenze in DNM1 mRNA, nonché potenziale cross-ibridazione con ACT1 mRNA è mostrato a sinistra. Risultati prodotti da reazioni di RT-PCR (30 cicli) per l'individuazione dei mRNAs di lievito DNM1 e ACT1 eluito dai branelli del gel dell'agarosi è mostrato a destra. Ingresso, RNA totale; Sn, supernatante dopo incubazione con ASOs; E, eluati da perline lavati a indicato eluizione preventiva temperature (40 ° C, 50 ° C e 60 ° C). Un esperimento di controllo (Ctrl) è stato effettuato in parallelo senza l'aggiunta di ASO. (D) gel dell'agarosi mostrando i prodotti di RT-PCR per l'individuazione dei mRNAs (a destra) catturato da RNA totale di lievito S. cerevisiae, c. eleganse cellule HEK293 umane (H. sapiens). Ingresso, RNA totale; Sn, surnatante; E, eluati dalle perle. PCR è stata effettuata da 30 cicli di amplificazione per lievito mRNA, 32 cicli per i mRNAs di c. elegans , e 28 e 30 cicli per umano tubulina e mRNAs p27 , rispettivamente. La figura è stata modificata dalla precedente pubblicazione37 con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
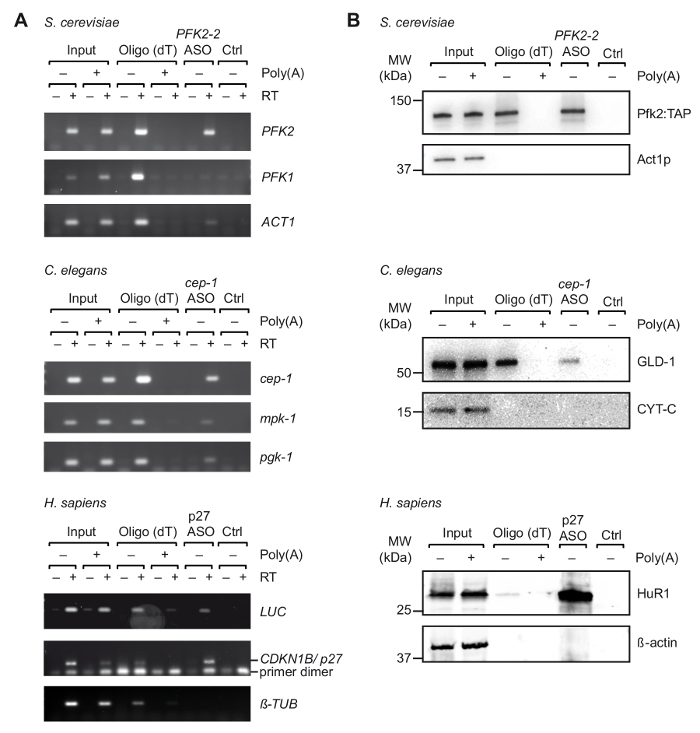
Figura 3. Acquisizione di complessi di specifici mRNA-proteina dagli estratti derivati da cellule reticolati UV con viaggio. Gel di agarosio (A) per l'individuazione dei mRNAs (a destra) con RT-PCR dopo l'acquisizione con oligo e ASOs indicato da S. cerevisiae, c. elegans ed estratti di cellule umani (H. sapiens). Ingresso, RNA totale da reticolati cellule/organismi; CTRL, controllo senza ASO. Poli (a), concorrenza con il poli (a). RT-PCR è stata effettuata come descritto37 con 35 cicli di amplificazione per LUC, 32 cicli per p27, e cicli di 29 per tubulina. (B) l'analisi di Immunoblot di proteine mRNA associato con gli anticorpi indicati (a destra). Caricato le frazioni sono come segue: 0,1%, 2,5% e 1% per il lievito, ingressi del nematode e umani; 10%, 10% e 5% per il lievito, corsie di oligo del nematode e umano; e del 66% per tutte le corsie di ASOs. Peso molecolare (MW) sono indicati in kilodaltons (kDa). Figura ristampata con permesso37. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome di primer | Sequenza | Destinazione | Dimensioni | |
| Pfk2_Fwd | GTGTTAAGGGTTCACATGTCG | PFK2 S. cerevisiae | 133 bp | |
| Pfk2_Rev | CTTCCAACCAAATGGTCAGC | PFK2 S. cerevisiae | 133 bp | |
| Pfk1_Fwd | GGTGATTCTCCAGGTATGAATG | PFK1 S. cerevisiae | 97 bp | |
| Pfk1_Rev | CTTCGTAACCTTCGTAAACAGC | PFK1 S. cerevisiae | 97 bp | |
| Act1_Fwd | GTCTGGATTGGTGGTTCTATC | ACT1 S. cerevisiae | 85 bp | |
| Act1_Rev | GGACCACTTTCGTCGTATTC | ACT1 S. cerevisiae | 85 bp | |
| Dnm1_Fwd | CTGTGTTCGATGCATCAGAC | DNM1 S. cerevisiae | 156 bp | |
| Dnm1_Rev | CGCACTCCAATTCTTCTCTC | DNM1 S. cerevisiae | 156 bp | |
| Rps20_Fwd | CGCTGAACAACACAACTTGG | RPS20 S. cerevisiae | 228 bp | |
| Rps20_Rev | GGAAGCAACAACAACTTCGAC | RPS20 S. cerevisiae | 228 bp | |
| Cep1_Fwd | CGATGAAGAGAAGTCGCTGT | CEP-1 c. elegans | 110 bp | |
| Cep1_Rev | ATCTGGGAACTTTTGCTTCG | CEP-1 c. elegans | 110 bp | |
| Pgk1_Fwd | GCGATATTTATGTCAATGATGCTTTC | PGK-1 c. elegans | 74 bp | |
| Pgk1_Rev | TGAGTGCTCGACTCCAACCA | PGK-1 c. elegans | 74 bp | |
| Mpk1_Fwd | TGCTCAGTAATCGGCCATTG | MPK-1 c. elegans | 74 bp | |
| Mpk1_Rev | TCCAACAACTGCCAAAATCAAA | MPK-1 c. elegans | 74 bp | |
| p27_Fwd | TTTAAAAATACATATCGCTGACTTCATGG | P27 H. sapiens | 212 bp | |
| p27_Rev | CAAAGTTTATGTGCTACATAAAAGGTAAAAA | P27 H. sapiens | 212 bp | |
| Luc_Fwd | AATGGCTCATATCGCTCCTGGAT | Luciferasi p. pγralis | 117 bp | |
| Luc_Rev | TGGACGATGGCCTTGATCTTGTCT | Luciferasi p. pγralis | 117 bp | |
| Β-TUBULIN_Fwd | CTGAACCACCTTGTCTCAGC | Β-tubulina H. sapiens | 136 bp | |
| Β-TUBULIN_Rev | AGCCAGGCATAAAGAAATGG | Β-tubulina H. sapiens | 136 bp | |
| PFK2-1 ASO | AAUAGAAAGUGUAAUAAAAGGUCAU | 3' UTR PFK2 S. cerevisiae | - | |
| PFK2-2 ASO | GUUUCAUGGGGUAGUACUUGU | 3' UTR PFK2 S. cerevisiae | - | |
| ASO PFK2-4 | CUUGAAGAGGAGCGUUCAUA | ORF PFK2 S. cerevisiae | - | |
| DNM1 ASO | UCGGUCAGUGGAGGUUCAGCGUUU | ORF DNM1 S. cerevisiae | - | |
| RPS20 ASO | GUCGGUAAUAGCCUUCUCAUUCUUG | ORF RPS20 S. cerevisiae | - | |
| CEP-1 ASO | GUGAGAAAUGCGGUGCUUUGAAA | 3' UTR cep-1 c. elegans | - | |
| p27 ASO | UCAUACCCCGCUCCACGUCAGUU | 3' UTR p27 H. sapiens | - | |
Tabella 1. Sequenze di oligonucleotidi. Elenco dei PCR primer e ASOs utilizzato in questo lavoro, sequenza primer, gene bersaglio e dimensione del frammento previsto dopo l'amplificazione.
Discussione
VIAGGIO permette l'analisi di proteine legate a specifici mRNA in vivo con mezzi biochimici. Mentre abbiamo utilizzato il metodo per convalidare l'interazione di particolare RBPs con mRNA bersaglio da diverse organismi o cellule, viaggio potrebbe anche essere applicabile per studiare altri tipi di RNA, poli (a) quali citoplasmico ncRNAs lunghi. Inoltre, un'analisi sistematica delle proteine rilegate e/o RNA con sequenziamento MS o RNA potrebbe essere realizzata da un up-scaling della procedura. A questo proposito, i nostri dati preliminari indicano che 1 L di colture di lievito e almeno 100 milioni di cellule HEK293 umane potrebbe fornire sufficiente materiale di partenza per ottenere dati affidabili di MS per ben espresso mRNA (risultati non pubblicati).
Il protocollo descritto di viaggio include l'irradiazione delle cellule con luce UV a 254 nm a interazioni crosslink della RNA-proteina. Questo consente l'implementazione di lavaggio stringente condizioni durante la purificazione. Tuttavia, anche se non esplicitamente testati, desideriamo reticolazione è opzionale e RNP attivo può anche essere recuperati con buffer fisiologico. Tuttavia, in questo caso, il potenziale riassetto delle proteine e/o RNA su RNA dell'obiettivo nei lysates dovrebbe tenere conto. Al contrario, se UV o altre procedure di reticolazione sono applicate (ad es., formaldeide), dovrebbe essere valutata l'integrità del RNA come degradazione del RNA potrebbe portare al recupero diminuita di mRNPs. Inoltre, reticolazione UV è piuttosto inefficiente (~ 5%) e ancor meno per proteine che interagiscono con RNA double-stranded, che potrebbero introdurre bias per la rilevazione di determinate classi di RBPs12. Infine, irradiazione UV può anche indurre le reticolazioni proteina-proteina e proteina-DNA che dovrebbero essere considerate per dati interpretazione45,46. Pertanto, le procedure di reticolazione alternativo, ad esempio con formaldeide, potrebbero anche essere di particolare interesse per catturare grandi assemblee di proteina su mRNA.
Una caratteristica fondamentale del viaggio è la procedura di purificazione basato su ASO per recuperare particolare RNAs, aumentando così la selettività e diminuendo la probabilità di co-purificante contaminanti in due fasi. Per esempio, una preoccupazione riguarda aggiungere biotinilati ASOs direttamente estratti cellulari, che potrebbero portare a co-purificazione di biotina-binding proteins. Questo è aggirato in viaggio implementando un primo round di purificazione di poli (a) RNA mediante legame covalente accoppiato oligo-(dT)25 biglie magnetiche, che permette per condizioni rigorose purificazione come fatto per la cattura di Interactoma della RNA-proteina. Inoltre, selezione di RNA di poli (a) rimuove ncRNAs molto abbondante, come RNA ribosomiale (rRNA) e tRNAs e quindi riduce la complessità del campione e fonti potenziali per cross-ibridazione e contaminazione. Nel secondo passaggio, particolare mRNA vengono recuperati con biotinilato e modificato ASOs che tempri con le regioni il mRNA bersaglio. In alternativa, ASOs modificate potrebbe anche essere direttamente in covalenza collegato a biglie magnetiche tramite un'ammina-linker, riducendo la propensione a catturare le proteine obbligatorie di biotina. Tuttavia, nella nostra esperienza, l'incubazione della frazione di RNA di poli (a) con ASOs prima dell'aggiunta di streptavidina perline recuperato mRNPs più efficientemente di aggiunta diretta di perline ASO-accoppiato. Possibilmente, i oligos gratis sono cineticamente favoriti migliore accesso alle sequenze di RNA strutturato rispetto i oligos immobilizzati. Quindi, l'accoppiamento covalente di ASOs per perline prima l'incubazione con il campione potrebbe essere pregiudizievole per il recupero della destinazione del RNA e deve essere testato empiricamente.
Uno svantaggio con metodi ASO basata è inefficiente recupero di alcuni mRNA bersaglio. Si consiglia pertanto la valutazione di diversi ASOs che tempri a diverse regioni della trascrizione. In particolare, suggeriamo il design di ASOs di 2-3 che tempri con sequenze in 3'-UTR o i CD del mRNA di interesse. Ogni ASO possono esibire una differente efficienza per il recupero del rispettivo target di mRNA e dovrebbe essere empiricamente testati (Figura 2A, B, dati non mostrati). Alla fine, abbiamo trovato che ASOs ricottura per il 3' UTR eseguito meglio rispetto a quelli di ricottura per CD. Questo potrebbe essere a causa di una maggiore accessibilità dei siti di legame in UTR a causa dell'assenza di tradurre i ribosomi, considerando che i ribosomi possono ostruire l'ibridazione efficiente all'interno del CD. Abbiamo anche sperimentato che combinazione di ASOs diversi potrebbe portare a una maggiore contaminazione da abbondanti non bersaglio mRNA e ridotta efficienza di discesa. In ogni caso, la selettività dei oligos selezionate può essere controllata da esperimenti di competizione (ad es., aggiunta di concorrenti i oligos).
Un ulteriore preoccupazione riguarda potenziale cross-ibridazione con RNA non correlati, come abbiamo osservato che anche parziale ibridazione con altri mRNA può portare al recupero di quello mRNA (Figura 2). Per valutare l'idoneità del progettato ASOs, pertanto si consiglia di eseguire iniziale in vitro test con RNA totale per ottimizzare concentrazioni saline e temperatura di lavaggio di buffer. Nelle nostre mani, lavaggio di streptavidina-accoppiato di biglie magnetiche nei buffer contenente 150 mM NaCl a 55 ° C i risultati migliori, ma si consiglia di test indipendenti delle condizioni per ogni ASO progettato. Degno di nota, abbiamo trovato che tutti ASOs selezionato da esperimenti in vitro (Figura 2D) erano appropriati per catturare la rispettiva destinazione mRNA dagli estratti di cellule reticolate (Figura 3), ulteriormente comprovare la validità delle in vitro test. Oltre alla progettazione di ASOs adatto per l'acquisizione di mRNA, altri fattori potrebbero avere un impatto sulla selettività. Pertanto, procedure di bloccare complete con BSA e/o tRNA secondo le specifiche del tipo di perlina possono impedire aspecifici associazione di perline. Per ridurre ulteriormente l'assorbimento non specifico di proteine, si consiglia inoltre l'utilizzo di tubi Lo-bind, specialmente quando si lavora con basse quantità di campioni.
In genere, viaggio possa integrare gli approcci precedentemente stabiliti per catturare RNAs con ASOs. Per esempio, l' RNA non codificanti Xist è stato isolato con proteine rilegate da estratti nucleari derivati dalle cellule di reticolati UV 200 milioni utilizzando una matrice di lungo (90-mer) biotinylated del DNA oligonucleotidi che copriva l'intera trascrizione 34,35. Tuttavia, l'approccio scelto piastrelle poteva diventare un problema alla luce il grande potenziale per cross-ibridazione con RNA estranei, e non può essere utilizzato per selezionare trascrizione specifici, ad es., alternativamente impiombati forme. Recentemente, specificamente progettato dell'acido nucleico bloccato (LNA) o DNA ASO mixmers erano legame covalente di una resina magnetica per recuperare in vitro trascrizione o RNA ribosomiale (rRNA) altamente espressi dagli estratti senza cellula; Tuttavia, non è stato testato per il recupero di in vivo formata mRNA-proteina complessi36. Alla luce il riconoscimento aumentato di controllo genico post-trascrizionale di RBPs e ncRNA, riteniamo che il viaggio è un metodo utile per studiare la composizione e la dinamica dei complessi di RNP all'interno delle cellule al momento segnali intracellulari e ambientale e il suo impatto nella salute e nella malattia.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Siamo grati al Dr. Jonathan Hall e Mauro Zimmermann (ETH Zurigo) per la sintesi di PFK2 e cep-1 ASOs, Dr. Maikel Wouters per la progettazione di p27/CDKN1B ASOs e Dr. Rafal Ciosk (Friedrich Miescher Institute for Biomedical Research, Basilea) per gli anticorpi anti-GLD-1. Questo lavoro è stato sostenuto da biotecnologia e Biological Sciences Research Council (BB/K009303/1) e una Royal Society Wolfson ricerca Merit Award (WM170036) a a.p.g.
Materiali
| Name | Company | Catalog Number | Comments |
| MaxQ 5000 Large Incubated and Refrigerated Orbital Shaker | Thermo Fisher Scientific | SHKE5000-8CE | Floor shaker to grow yeast cells |
| BBD 6220 | Thermo Fisher Scientific | CO2 incubator to grow human cells | |
| Sonicator Soniprep150 | MSE | MSS150.CX4.5 | Sonicator/cell disruptor. Used to shear DNA in human cell lysates |
| Stratalinker 1800 | Stratagene | Stratalinker to expose cells to UV light at 254 nm | |
| Tissue Lyser | Qiagen | RETSCH MM200 | Device to mechanically disrupt yeast cells |
| Refrigerated Centrifuge | Eppendorf | 5810R | Centrifuge to spin down cells, lysates |
| Shaking incubator, Thriller | Peqlab | Thermoshaker for 1.5 mL tubes | |
| Glass beads 0.5mm | Stratech Scientific Limited | 11079105 | Lysis of yeast cells |
| Nylon Filter, 0.41 μm | Millipore | NY4104700 | Collection of yeast cells |
| Millex-HA Filter, 0.45 µm | EMD Millipore | SLHA02510 | Filter to clear nematode lysate |
| Eppendorf LoBind microcentrifuge tubes Protein 1.5 mL | Sigma-Aldrich | 22431081 | Minimise protein loss |
| 2 mL microfuge tube | Ambion | AM12425 | Yeast extract preparation and other applications |
| 5 mL tube | Thermo Fisher Scientific | 129A | Collection of HEK293 cell lysate |
| 15 mL tube | Sarstedt | 62.547.254 | Collection C. elegans |
| 50 mL plastic tube | Sarstedt | 62.554.502 | Collection yeast cells |
| DMEM, high glucose, pyruvate | Thermo Fisher Scientific | 41966 | Media for culturing HEK293 cells |
| Penicillin-Streptomycin | Sigma-Aldrich | P4333 | Used to supplement cell culture media to control bacterial contamination |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F7524 | Used to supplement cell culture media |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| RQ1 RNase-Free DNase | Promega | M6101 | DNAse I to degrade DNA from cell lysate |
| RNasin Plus RNase Inhibitor | Promega | N2611 | RNase inhibitor to avoid RNA degradation |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitor cocktail to avoid protein degradation |
| Dynabeads Oligo (dT)25 | Thermo Fisher Scientific | 61011 | Magnetic beads required for the first step of mRNA-protein complex purification |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | Magnetic beads required for the second step of mRNA-protein complex purification |
| E. coli tRNA | Sigma-Aldrich | 10109550001 | transfer RNA from E. coli used for blocking streptavidin beads and avoid unsepecific interactions |
| Quick Start Bradford 1x Dye Reagent | BioRad | 5000205 | To determine protein concentration within lysates, using BSA as reference |
| Bovine Serum Albumin | Sigma | A7906-100G | Used to perform the standard curve that will be used as reference for protein concentration determination |
| mouse anti-Act1 | MP Biomedicals | 869100 | Antibody for detection of yeast actin in Western blot (1:2,500) |
| mouse anti-Act1 | Sigma | A1978 | Antibody for detection of human actin in Western blot (1:2,000) |
| mouse anti-HuR | Santa Cruz | sc-5261 | Antibody for detection of HuR in Western blot (1:500) |
| mouse anti–CYC-1 | Invitrogen | 456100 | Antibody for detection of Cyc-1 in Western blot (1:1,000) |
| peroxidase anti-peroxidase soluble complex | Sigma | P1291 | Detection of Pfk2:TAP in Western blot (1:5,000) |
| HRP-conjugated sheep anti-mouse IgG | Amersham | NXA931 | HRP-coupled secondary antibody |
Riferimenti
- Mitchell, S. F., Parker, R. Principles and properties of eukaryotic mRNPs. Molecular Cell. 54 (4), 547-558 (2014).
- Iadevaia, V., Gerber, A. P. Combinatorial Control of mRNA Fates by RNA-Binding Proteins and Non-Coding RNAs. Biomolecules. 5 (4), 2207-2222 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Matia-González, A. M., Gerber, A. P., Sesma, A., von der Haar, T. Ch. 14. Fungal RNA Biology. , 347-370 (2014).
- Tsvetanova, N. G., Klass, D. M., Salzman, J., Brown, P. O. Proteome-wide search reveals unexpected RNA-binding proteins in Saccharomyces cerevisiae. PLoS One. 5 (9), (2010).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Baltz, A. G., et al. The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Molecular Cell. 46 (5), 674-690 (2012).
- Castello, A., et al. System-wide identification of RNA-binding proteins by interactome capture. Nature Protocols. 8 (3), 491-500 (2013).
- Liao, Y., et al. The Cardiomyocyte RNA-Binding Proteome: Links to Intermediary Metabolism and Heart Disease. Cell Reports. 16 (5), 1456-1469 (2016).
- Liepelt, A., et al. Identification of RNA-binding Proteins in Macrophages by Interactome Capture. Molecular Cell Proteomics. 15 (8), 2699-2714 (2016).
- Conrad, T., et al. Serial interactome capture of the human cell nucleus. Nature Communications. 7, 11212 (2016).
- Matia-Gonzalez, A. M., Laing, E. E., Gerber, A. P. Conserved mRNA-binding proteomes in eukaryotic organisms. Nature Structural and Molecular Biology. 22 (12), 1027-1033 (2015).
- Mitchell, S. F., Jain, S., She, M., Parker, R. Global analysis of yeast mRNPs. Nature Structural and Molecular Biology. 20 (1), 127-133 (2013).
- Beckmann, B. M., et al. The RNA-binding proteomes from yeast to man harbour conserved enigmRBPs. Nature Communications. 6, 10127 (2015).
- Wessels, H. H., et al. The mRNA-bound proteome of the early fly embryo. Genome Research. 26 (7), 1000-1009 (2016).
- Sysoev, V. O., et al. Global changes of the RNA-bound proteome during the maternal-to-zygotic transition in Drosophila. Nature Communications. 7, 12128 (2016).
- Despic, V., et al. Dynamic RNA-protein interactions underlie the zebrafish maternal-to-zygotic transition. Genome Research. 27 (7), 1184-1194 (2017).
- Zhang, Z., et al. UV crosslinked mRNA-binding proteins captured from leaf mesophyll protoplasts. Plant Methods. 12, 42 (2016).
- Reichel, M., et al. In Planta Determination of the mRNA-Binding Proteome of Arabidopsis Etiolated Seedlings. Plant Cell. 28 (10), 2435-2452 (2016).
- Koster, T., Marondedze, C., Meyer, K., Staiger, D. RNA-Binding Proteins Revisited - The Emerging Arabidopsis mRNA Interactome. Trends Plant Sciences. 22 (6), 512-526 (2017).
- Hamasaki, K., Killian, J., Cho, J., Rando, R. R. Minimal RNA constructs that specifically bind aminoglycoside antibiotics with high affinities. Biochemistry. 37 (2), 656-663 (1998).
- Bachler, M., Schroeder, R., von Ahsen, U. StreptoTag: a novel method for the isolation of RNA-binding proteins. RNA. 5 (11), 1509-1516 (1999).
- Vazquez-Pianzola, P., Urlaub, H., Rivera-Pomar, R. Proteomic analysis of reaper 5' untranslated region-interacting factors isolated by tobramycin affinity-selection reveals a role for La antigen in reaper mRNA translation. Proteomics. 5 (6), 1645-1655 (2005).
- Hartmuth, K., Vornlocher, H. P., Luhrmann, R. Tobramycin affinity tag purification of spliceosomes. Methods Molecular Biology. 257, 47-64 (2004).
- Beach, D. L., Keene, J. D. Ribotrap : targeted purification of RNA-specific RNPs from cell lysates through immunoaffinity precipitation to identify regulatory proteins and RNAs. Methods Molecular Biology. 419, 69-91 (2008).
- Slobodin, B., Gerst, J. E. A novel mRNA affinity purification technique for the identification of interacting proteins and transcripts in ribonucleoprotein complexes. RNA. 16 (11), 2277-2290 (2010).
- Slobodin, B., Gerst, J. E. RaPID: an aptamer-based mRNA affinity purification technique for the identification of RNA and protein factors present in ribonucleoprotein complexes. Methods Molecular Biology. 714, 387-406 (2011).
- Yoon, J. H., Gorospe, M. Identification of mRNA-Interacting Factors by MS2-TRAP (MS2-Tagged RNA Affinity Purification). Methods Molecular Biology. 1421, 15-22 (2016).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Blencowe, B. J., Sproat, B. S., Ryder, U., Barabino, S., Lamond, A. I. Antisense probing of the human U4/U6 snRNP with biotinylated 2'-OMe RNA oligonucleotides. Cell. 59 (3), 531-539 (1989).
- Lingner, J., Cech, T. R. Purification of telomerase from Euplotes aediculatus: requirement of a primer 3' overhang. Proceedings of the National Academy of Sciences U S A. 93 (20), 10712-10717 (1996).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Molecular Cell Proteomics. 12 (6), 1539-1552 (2013).
- Imig, J., et al. miR-CLIP capture of a miRNA targetome uncovers a lincRNA H19-miR-106a interaction. Nature Chemical Biology. 11 (2), 107-114 (2015).
- Chu, C., et al. Systematic discovery of Xist RNA binding proteins. Cell. 161 (2), 404-416 (2015).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Rogell, B., et al. Specific RNP capture with antisense LNA/DNA mixmers. RNA. 23 (8), 1290-1302 (2017).
- Matia-Gonzalez, A. M., Iadevaia, V., Gerber, A. P. A versatile tandem RNA isolation procedure to capture in vivo formed mRNA-protein complexes. Methods. , 93-100 (2017).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic Acids Research. 36 (Web Server issue), W70-W74 (2008).
- Kalendar, R., Khassenov, B., Ramankulov, Y., Samuilova, O., Ivanov, K. I. FastPCR: An in silico tool for fast primer and probe design and advanced sequence analysis. Genomics. 109 (3-4), 312-319 (2017).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic local alignment search tool. Journal of Molecular Biology. 215 (3), 403-410 (1990).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Kedde, M., et al. A Pumilio-induced RNA structure switch in p27-3' UTR controls miR-221 and miR-222 accessibility. Nature Cell Biology. 12 (10), 1014-1020 (2010).
- Schumacher, B., et al. Translational repression of C. elegans p53 by GLD-1 regulates DNA damage-induced apoptosis. Cell. 120 (3), 357-368 (2005).
- Ziegeler, G., et al. Embryonic lethal abnormal vision-like HuR-dependent mRNA stability regulates post-transcriptional expression of cyclin-dependent kinase inhibitor p27Kip1. Journal of Biological Chemistry. 285 (20), 15408-15419 (2010).
- Itri, F., et al. Femtosecond UV-laser pulses to unveil protein-protein interactions in living cells. Cellular and Molecular Life Sciences. 73 (3), 637-648 (2016).
- Zhang, L., Zhang, K., Prandl, R., Schoffl, F. Detecting DNA-binding of proteins in vivo by UV-crosslinking and immunoprecipitation. Biochemical and Biophysical Research Communications. 322 (3), 705-711 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon