Method Article
基于 rna 的人原代成纤维细胞重组成诱导多能干细胞
摘要
在这里, 我们描述了一种临床相关的, 高效率, 无微弱的方法, 重新编程人类的原代成纤维细胞到诱导多能干细胞使用修改 mrna 编码重编程因子和成熟的微 rna-367/302 模拟。还包括评估重新编程效率、扩大克隆 ipsc 菌落和确认多能标记 tra-1-60 的表达的方法。
摘要
诱导多能干细胞 (ipsc) 已被证明是研究人类发育和疾病的宝贵工具。进一步推进 ipsc 作为再生治疗需要一个安全、可靠和方便的重新编程协议。在这里, 我们提出了一个临床相关的分步协议, 使用非集成方法将人类真皮成纤维细胞重新编程到 ipsc 中。该协议的核心是表达多能性因子 (sox2, klf4, cmyc, lin28a, nanog, oct4-myod 融合) 的体外转录信使 rna 合成修饰核苷酸 (修饰 mrna)。重新编程修改 mrna 与成熟的胚胎干细胞特异性微 rna-367 302 仿事一起每48小时转染一次原代成纤维细胞, 为期两周。然后, 可以在无弱的条件下隔离和直接扩展生成的 ipsc 菌落。为了最大限度地提高我们在成纤维细胞样本中重新编程协议的效率和一致性, 我们优化了各种参数, 包括 rna 转染方案、转染时间、培养条件和播种密度。重要的是, 我们的方法从大多数成纤维细胞源生成高质量的 ipsc, 包括难以再治疗的疾病、老化和衰老样品。
引言
将体细胞重新编程为诱导多能干细胞 (ipsc) 需要扩展表达一组核心转录因子, 这些转录因子对维持多能性1,2非常重要。在为临床应用生产 ipsc 时, 在处理过程中必须最大限度地减少输入单元的突变负担, 并且在患者样本中保持相对较高的 ipsc 生成效率。然而, 大多数重新编程方法, 包括无集成协议, 都存在着非常低的重新编程效率, 这限制了这些方法的临床用途3。低重新编程效率也可能促进携带预先存在的突变的细胞的选择性重新编程, 增加产生的 ipsc 的突变负担。此外, 所有基于 dna 的重编程方法, 如基于 lentivir和 iapal-apian 的方法, 都受到安全问题的影响, 即 dna 可能会随机整合到基因组中, 并为有害的插入突变和不需要的物质创造机会 (多能基因在下游组织衍生物4中的表达。
一个很有希望的方法, 以实现高效诱导多能性的体细胞和减少突变负担, 由此产生的 ipsc 是使用合成上限信使 rna 含有改性核酸酶 (修改 mrna) 重新编程5。通过添加胚胎干细胞特异性微 rna (miRNAs)-367/302s 3, 可以进一步提高改进的基于 mrna 的重编程方法的效率, 这些微rna 已被证明可以重新编程, 提高效率 6 ,7。然而, 即使添加了 mirnas-367/302s, 改进后的基于 mrna 的重新编程方法在应用于新分离的患者细胞3的过程中也常常失败。为了解决这种改进的基于 mrna 的方法的不一致问题, 我们最近报告了一种优化的、无集成的策略, 该策略在人类原生成纤维细胞中诱导多能性, 成功率很高, 并同时利用了改进的 mrna 编码重编程因素和成熟的 mirna-367/302 模拟8。在我们的方法中, 重新编程修改 mrna 鸡尾酒包括一个修改版本的 oct4 融合与 myod 转化域 (称为 m3o)9和其他五个重新编程因素 (sox2, klf4, cmyc, lin28a 和 nanog).将改进后的 mrna 编码多能因子与 mirna 模拟物结合起来, 似乎对该协议的重新编程效率有协同作用。还需要对 rna 转染方案、细胞播种和培养条件进行额外的优化, 以提高超高水平8方法的重新编程效率.
与许多其他协议不同, 我们的重新编程方法通常只需要几千种输入成纤维细胞。此外, 许多常用的非整合策略使用星代体质粒、仙台病毒或自复制 rna, 涉及广泛的传代, 以稀释生成的 ipsc 中的重编程载体。相反, 修饰的 mrna 和成熟的 mirna 模拟物的半衰期较短, 并迅速从细胞中消除。综合来看, 在这种方法中, 收集患者样本和生成可用的 ipsc 之间的累积细胞培养时间很小, 从而有效地限制了产生的 ipsc 中的突变积累, 并提高了成本效益。
在这里, 我们提出了详细的分步协议, 以实现高效的重新编程成人成纤维细胞到 ipsc 使用我们的组合修改 mrna/mirna 为基础的方法 8。这种基于 rna 的重新编程协议提供了一种简单、经济高效且可靠的方法, 用于生成用于研究和潜在临床应用的无集成 ipsc。此外, 它还适用于各种成纤维细胞系的重新编程, 包括难以重新编程、疾病相关、老化和衰老成纤维细胞。重新编程人类成纤维细胞的协议示意图如图 1所示。该协议特别描述了一种在6井格式板中重新编程人类成人原生成纤维细胞三口井的方法。两口井通常会产生足够数量的高质量 ipsc 菌落。在许多情况下, 只需要一口井, 第三口井可以用来分析重新编程效率。如果需要, 可以放大井的数量。
研究方案
在无 rnase 条件下工作, 并在可能的情况下使用无菌技术。使用无菌技术在生物安全柜中执行所有与细胞培养相关的操作。遵循机构生物安全标准与人体细胞合作。
1. 准备重新编程启动的试剂和设备
- 准备修改后的 mrna 鸡尾酒编码重新编程因素。
- 遵循先前公布的协议10 , 对每个修改后的 mrna 编码单个重编程因子执行体外转录、封盖和脱磷酸化过程。最后纯化后, 用无核酸酶水洗脱改性的 mrna, 用 1u/μl rnase 抑制剂补充纯化后的 mrna 溶液, 用分光光度计对改性 mrna 进行定量, 并在-80°c 下储存长达6个月。为每个改性的 mrna 准备多个等价物, 以最大限度地减少冻融循环的数量。
注: 其他地方也报告了类似的改性 mrna 生产协议。根据先前公布的协议 10, 可以使用材料表中列出的引物从相应的质粒模板中进行 pcr 扩增, 从而生成体外转录模板.每个重编程因子 (m3o9、sox2、transfection、cmyc、nanog、lin28a) 的质粒模板可从非营利质粒存储库 adgene 获得. - 以 3:1x-1: 1 (m3o2:sox2:klf4:cmo:innog:lin28a) 的摩尔比将所有修改后的 mrna 混合在一起, 并包括10% 的 mwasabi 改性 mrna 作为转染效率的控制。通过加入无核酸酶水, 辅以 1uμl rnase 抑制剂, 将完整改性的 mrna 重编程混合物的浓度调整为最终浓度为 100 ngμl。准备 7个33μl 的完整改性 mrna 鸡尾酒。将混合重编程的等价物存放在-80°c。
注: 每次转染时, 每口井添加 1, 000 纳克改性 mrna 鸡尾酒 (即3 口井共 3, 000 纳克)。每个 33μl aliquot 的尺寸是为了转染6井格式板的3口井, 并包括3微米的多余体积, 以考虑移液误差。准备 7个33μl 等位器足以完成6井格式板中3口井的全成纤维细胞重新编程。
- 遵循先前公布的协议10 , 对每个修改后的 mrna 编码单个重编程因子执行体外转录、封盖和脱磷酸化过程。最后纯化后, 用无核酸酶水洗脱改性的 mrna, 用 1u/μl rnase 抑制剂补充纯化后的 mrna 溶液, 用分光光度计对改性 mrna 进行定量, 并在-80°c 下储存长达6个月。为每个改性的 mrna 准备多个等价物, 以最大限度地减少冻融循环的数量。
- 准备重新编程 mirna 模拟的鸡尾酒。
- 溶解冻干 mirna 仿制品 (syn-has-mir-3a-3p, syn-has-mir-302-3p, syn-has-32c-3p, syn-has-空运-302-3p, Syn-has-miR-302a-3p) 到 5 pmolμμl (5μm) 最终浓度在无核水中补充了 rnase 抑制剂的1une-μl。准备每个 mirna 的多个等价物模拟并存储在-80°c, 以便进行长期储存。
- 将所有 mirna 模拟物混合在 1:1: 1 摩尔比中, 最终浓度为 5 pmolμμμl (5μm)。准备 7个14μl 等位基因的 mirna 模拟混合。将混合等价物存放在-80°c。
注: 每次转染, 每口井添加 20 pmol 的 mirna 模拟混合物 (即3 口井共 60 pmol)。每个 14μl aliquot 的尺寸是为了转染6井格式板的3口井, 并包括2μl 的多余体积, 以考虑移液误差。准备 7个14μl 等位数足以完成6井格式板中3口井的全成纤维细胞重新编程。
- 准备转染缓冲器。
- 预热一个500毫升瓶和一个100毫升的新鲜降-血清中的培养基到室温 (rt) 约2小时。不要使用水浴。
- 将 ph 计传输到生物安全柜。用无核酸水清洗仪表的玻璃电极。根据制造商的说明校准 ph 计。用无核酸水再次清洗电极。
- 将两瓶降低血清培养基转移到生物安全柜。使用500毫升 rt 瓶调整 ph 值。
- 通过将 ph 计的玻璃电极插入缓冲液中, 测量还原血清介质的碱 ph 值。在读取仪表上的 ph 值之前, 请等待1分钟。
- 在500毫升降糖血清中加入3-4 毫升的 naoh, 关闭瓶子, 搅拌均匀。在打开瓶子进行 ph 测量之前, 请等待5分钟。将 ph 计的电极插入缓冲液中, 然后等待 ph 计上的读数稳定。
- 继续加入小容量的 1 m naoh, 直到还原血清培养基的 ph 值达到8.15-8.17。在此过程中多次校准 ph 计。如果在任何时候 ph 值高于 8.18, 则通过从预热的 100 ml 瓶 (步骤 1.3.1) 中添加新鲜的降糖血清培养基来降低 ph 值。
注: 减少血清中导液转染缓冲液的 ph 值至关重要。如果转染缓冲液的 ph 值约为 8.2±0.05, 则重新编程将成功, 但如果 ph 值高于 8.25, 则可能会失败。因此, 建议在缓冲区的 ph 值达到8.15-8.17 时停止添加 naoh。这将确保经缓冲液的最终 ph 值在灭菌后达到约8.2-8.22。 - 过滤器使用0.22μm 真空过滤系统对转染缓冲液进行灭菌。将灭菌缓冲液加入5或15毫升管, 空气空间最小。
- 在4°c 下, 将转染缓冲液的等价物存放3个月。定期测量等价物的 ph 值。如果 ph 值高于 8.25, 请丢弃等价物。仅将脂肪的使用限制在 2次, 因为将转染缓冲液暴露在大气中会增加缓冲液的 ph 值。
- 制备成纤维细胞膨胀介质 (fem): 以10% 的胎牛血清 (fbs)、1x 非必需氨基酸、1x 谷氨酰胺补充剂、55μm 2-甲氨基乙醇、1x pen/streppcrep/fungizone 为补充的最低必要培养基。将有限元存储在4°c。
- 准备重新编程介质: dmm/f12 (无 hepes) 补充20% 的淘汰赛血清置换术 (kosr), 0.5倍非必需氨基酸, 0.5 x 谷氨酰胺补充剂, 55μm 2-硫醇, 50μgml 抗坏血酸, 1x pen/strep/fungizone, 100 ng/ml bfgf和 200 ngml b18r。在没有 bfgf 和 bFGF 的情况下, 在重新编程开始时准备介质, 并将其存储在4°c。不要在准备后1个月以上使用。每次使用前立即添加 bfgf 和 bFGF, 以达到当天使用的大小。
- 在包含 20% kosr 的再编程介质中加入5% 的热失活 (hi) fbs, 从步骤1.5 开始, 在不含 bfgf 和 bFGF 的情况下, 加入5% 的热失活 (hi) fbs, 准备电镀介质。在预定使用的当天准备新鲜的培养基。在重新编程的第0天使用之前立即添加 bfgf 和 bFGF。
- 根据制造商的说明, 使用手持数字 co2 分析仪, 在开始重新编程之前, 对组织培养孵化器进行校准。使用低 o2孵化器重新编程步骤, 以确保成功生成 ipsc。
2. 培养用于重新编程的成纤维细胞
- 在重新编程开始之前准备成纤维细胞 (第2天)。
- 涂上新的10厘米组织培养板, 5 毫升的0.1% 明胶。轻触或旋流板材, 以确保整个表面都被涂覆。在37°c 下孵化15分钟。吸入明胶, 加入10毫升的有限元法. 放在一边。在加入有限元法之前, 不要让板材的涂层表面干燥。
- 从成纤维细胞中仔细吸气所用的培养基。用5毫升的 dpbs 冲洗细胞一次, 去除残留血清。加入3毫升的色氨酸 edta。轻轻摇晃板, 以确保完全覆盖在细胞上。
- 吸入多余的液体, 留下约500μl 的胰蛋白酶。不要过度吸气, 因为这样会干燥并杀死成纤维细胞。在37°c 条件下, 用胰蛋白酶-edta 培养成纤维细胞3分钟。
- 从孵化器中取出盘子, 并用力但轻轻地点击盘子的一侧, 将细胞移开。检查显微镜下的细胞。如果单元格已分离并浮动, 请继续操作。如果细胞仍然附着, 再孵育3分钟。
- 快速冲洗收集分离细胞使用5毫升的有限元法中和色氨酸 edta。将成纤维细胞悬浮液移入15毫升锥形管。确保单元格混合良好。
- 轻轻混合细胞悬浮液, 打破大的细胞团块, 然后将其计算在血细胞计上。将 2.5 x 10 5 细胞转移到步骤2.1.1 准备的涂胶10厘米的糊状物盘中。在 37°c, 5% co2 加湿常规组织培养孵化器中孵育细胞.
注: 细胞密度是实现高重新编程效率的关键, 因为它是重要的是成纤维细胞是健康的, 并迅速划分。在2天后, 大多数成纤维细胞样本的镀存在 2.5 x 10 5个细胞会产生40-60% 的融合。相应地调整患病或衰老细胞的数量, 以达到所需的40-60% 融合48小时后。 - 第二天 (第1天) 将介质替换为10毫升新鲜的 fem。
- 将成纤维细胞进行切片, 以启动重新编程 (第0天)。
- 验证要重新编程的成纤维细胞是否融合 40–60% (图 2, 第0天)。如果细胞是在融合或下融合, 通过成纤维细胞如步骤2.1 中所述, 并相应地调整电镀密度。文化再过2天。
- 将4毫升的 dpbs 转移到一个15毫升的锥形管中。加入100μl 的重组人 (rh) 层压-521。上下移液器进行彻底混合。
- 每井加入1毫升稀释的拉明-521, 放入6井板的3口井中。在37°c 下将涂层板培养2小时。
- 热6毫升的有限元和4毫升的电镀介质至37°c。用 bfgf 将电镀介质补充到最终浓度为 100 ngml 和 bFGF, 最终浓度为 200 ng/ml。
- 从成纤维细胞中仔细吸气所用培养基 (步骤 2.2.1)。用5毫升的 dpbs 冲洗细胞, 加入3毫升的胰蛋白酶-edta。轻轻摇晃板, 用胰蛋白酶 edta 覆盖细胞。
- 吸入多余的液体, 留下 ~ 500μl 的胰蛋白酶, 如步调式2.1.3。在37°c 条件下, 用胰蛋白酶-edta 培养成纤维细胞3分钟。从孵化器中取出盘子, 并用力但轻轻地点击盘子的一侧, 将细胞取出。检查显微镜下的细胞。如果单元格已分离并浮动, 请继续操作。如果细胞仍然附着, 再孵育3分钟。
- Rinse/collect 使用5毫升的有限元法收集分离细胞, 中和胰蛋白酶-edta。将成纤维细胞悬浮液移入15毫升锥形管。确保细胞混合良好, 并将其计数在血细胞计上。从步进2.2.4 将 12, 000个细胞放入4毫升预热电镀介质中。
注: 不要在任何时候对电池进行离心。镀金单元的数量可以根据慢线或快速增长线的需要向上或向下调整。 - 从孵化器中取出涂层板, 从涂布井中吸气稀释的罗明-521。不要让水井表面干燥。在电镀介质中轻轻重新悬浮细胞。移液器1毫升的细胞悬浮液进入每个涂层井 (即每口井 3, 000个细胞)。
- 将镀金细胞放入 o2 设置为 5% (低 o2) 的三气组织培养孵化器中。一旦板被设置下来, 轻轻但彻底分散细胞之间交替向上/向下, 然后左/右的运动。再重复重复重复2次。将细胞连夜孵化。不要旋转盘子混合。
- 移液器4毫升重新编程介质到15毫升锥形管, 并将其放置在低 o2孵化器与一个松动的盖子, 以平衡一夜。直到第二天才添加 bfgf 和 bFGF。
3. 重新编程的启动 (第1天)
注: 一旦重新编程启动, 需要进行大约1个月的日常维护。一定要有相应的计划。所有随后的细胞培养必须在低 o2 条件下进行 37°c, 5% co 2, 5% o2 加湿的三气组织培养孵化器.
- 在转染前至少1小时更换电镀介质。
- 从低 o2 孵化器中取出均衡的再编程介质.将 bfgf 添加到最终浓度为 100 ngml 和 bFGF 的最终浓度为 200 ngml。混合好。
- 一次进行1口井, 使用1毫升移液器去除使用的介质, 并将其替换为1毫升的重新编程介质, 再加上 bfgf 和 bFGF。对每口井重复此操作。不要使用真空抽吸, 真空抽吸会使细胞过度干燥, 造成压力, 并降低重新编程效率。将带有细胞的板移回低 o2孵化器。
- 每1微克改性 mrna 用5μl 的 rnax 转染试剂转染成纤维细胞, 每转染一次, 每 6 pmol 模拟剂每转染一次, 转染转化试剂为1微米升。
注: 当转染持续16–20小时时, 可获得最佳结果。转染试剂稀释 10倍;100 ngμμl 改性 mrna 鸡尾酒稀释 5倍;5 pmolμ-α模拟物混合稀释 8.33 x 与转染缓冲剂在络合前。有关转染设置的摘要, 请参见表 1 。- 在准备转染混合物时, 遵循标准的实验室实践, 最大限度地减少试剂对 rnase 的潜在接触。
- 在 rt 中平衡转染缓冲液 (步骤 1.3) 的一个例外约1小时。不要使用37°c 的水浴或孵化器来加热转染缓冲液。
- 从-80°c 中取出改性 mrna (33μl) 和 mirna 模拟物 (14μl), 将其加热约 3-5分钟, 直到解冻。不要在37°c 下解冻。在微泡中短暂地旋转它们。
- 将转染试剂加热约3-5分钟。请勿使用37°c 的水浴或孵化器。将封闭管反转2-3 次, 以混合试剂。在微泡中短暂旋转。
- 将 rt 转染缓冲液的279μl 转移到无 rnase 的微离心管中。加入31μl 的转染试剂, 通过移液彻底混合, 最终体积为310μl。不要漩涡。在 rt 处为管材提供1分钟的经费。
注: 稀释转染试剂的体积足以使修改后的 mrna 和 mirna 模拟从3.2.3 步骤开始的等价物。 - 在30μl 改性 mrna 中加入132μl 的 rt 转染缓冲液。轻轻的移液器混合: 最终体积为165μl。
- 在 mirna 模拟剂的14μl 像中加入102.6 μl 的 rt 转染缓冲液。轻轻的移液器混合: 最终体积为16.6μl。
- 将稀释后的转染试剂从步骤3.2.5 添加 165μl, 从步骤3.2.6 添加到稀释的改性 mrna 中。混合的移液器: 最终体积为330μl。
- 将稀释后的转染试剂从步骤3.2.5 添加到从步骤3.2.7 混合的稀释 mirna 模拟剂中。搅拌均匀 (最终体积为 233.2μl)。在 rt 中培养 15分钟, 使转染缓冲液与改良的 mrna 和 mirna 模拟复合。
- 用低 o2 孵化器中的细胞取出盘子。在每口井中加入 100μl (1μg) 的复合改性 mrna 转染混合物, 滴至井内。通过轻轻但彻底地搅拌板, 然后左/右的运动, 分散转染配合物。不要旋转盘子混合。
- 添加 66.7μl (20 pmol) 的络合 mirna 模仿转染混合到每口井, 滴至井对面。通过轻轻但彻底地搅拌板, 然后左/右的运动, 分散转染配合物。不要旋转盘子混合。
- 将转染的细胞放入 o2 设置为 5% (低 o2) 的三气孵化器中.一旦板被设置下来, 再次分散转染配合物轻轻但彻底搅拌板向上/向下, 然后左/右的议案。
- 将介质重新编程到15毫升锥形管中, 并将其放置在低 o2孵化器中, 并将其与松盖放在一起, 以平衡一夜。直到第二天才添加 bfgf 和 bFGF。
| 管 1-rnaimax 稀释 (第一次混合) | ||
| 试剂 | 浓度 | 体积 |
| 转染缓冲器 | 279μl | |
| rnax (添加第二个) | 10倍 | 31μl |
| 总计: 310μl | ||
| (室温下孵育 1分钟) | ||
| 管 2-改性 mrna 混合 (第2次混合) | ||
| 试剂 | 浓度 | 体积 |
| 改性 mrna 混合 | 100 ngμl (5倍) | 33μl |
| 转染缓冲器 | 132μl | |
| 总计: 165μl | ||
| (从管1中添加相等的稀释 rnax) | ||
| 管 3-mirna 模拟混合 (第3次混合) | ||
| 试剂 | 浓度 | 体积 |
| mirna 模拟混合 | 5 pmopμl (8.33x) | 14μl |
| 转染缓冲器 | 102.6 μl | |
| 总计: 16.6μl | ||
| (从管1中添加相等的稀释 rnax) | ||
表 1: 转染混合物的制备。
4. 替换转换之间的替换介质 (第2、4、6、8、10和12天)
- 如 3.1.1 3.1.2 中所述, 在转染后16–20小时更换介质。将 bfgf 加入 100 ngml 和 bFGF 的最终浓度, 最终浓度为 200 ngml, 以重新编程中的 aliquots。
- 每天监测 mwasabi 表达, 以确认转染质量使用显微镜配置可视化 transfection。
注: mwasabi 表达应在第2天最低明显, 并增加亮度与每一个额外的转染。
5. 日常转染 (第3、5、7、9、11和13天)
- 按照步骤3.2 中的说明执行转染。不要在转染的日子改变介质。
- 准备一个4毫升的重新编程介质, 并将其放置在低 o2孵化器中, 以平衡第二天的介质变化。直到第二天才添加 bfgf 和 bFGF。
6. 最终转染后要执行的程序
- 执行从第14天到大约第17天的每日介质更改。将介质重新编程到37°c 的温度为7毫升。加入 bfgf 到最终浓度为 100 ngml。
注: b18r 在最终转染后不再需要。在第14天之后, 不再需要在低 o2 孵化器中夜间平衡介质. - 使用血清学移液器从所有井中取出介质。继续避免使用渴望。加入2毫升的重新编程介质, 再加上每口 bfgf。
- 在第17天和第18天, 在倒置或解剖显微镜下分析重新编程的井。如果由于重新编程的高效率而在彼此接近的情况下形成菌落, 并且不能手动隔离, 则通过执行下面步骤7中所述的重新编程井的可选传递来分离菌落。如果菌落稀疏且分离良好, 请根据标准协议, 从重新编程的区域中直接选择克隆体, 并在步骤8和亚培养 ipsc 中进行描述。
7. 可选程序: 使用 edta 从重新编程的井中传代细胞
- 通过稀释 0.5 m edta 库存解决方案, 在 dpbs (edta) 中准备 0.5 mm edta。使用0.22μm 真空过滤系统对 edta 进行灭菌。
- 根据制造商的说明制备无系体多能干细胞 (psc) 培养基 (例如, mtesr1)。预热32毫升的 psc 介质至37°c。
- 按照制造商的说明, 用 hesc 合格的细胞外基质 (ecm) 为两个6孔格式板的所有油井涂上涂层。用石蜡膜密封板, 并在 rt 孵育1小时。
- 从预热板中吸收 ecm 溶液, 并将其替换为每口2毫升的 psc 介质。不要让水井表面干燥。把它们放在一边。
- 在菌落大而清晰的一天 (通常是第18天), 从2口重新编程的井中吸收重新编程介质。用1毫升的 edta 冲洗一次, 吸气。
- 每口添加1毫升 edta。在37°c 下孵化4分钟。
- 轻轻地从孵化器上取下盘子, 放在生物安全柜里。
注: 此时, 细胞可能非常松散粘附, 并且很容易被移出。 - 从两口井仔细吸气 edta。在每种经过 edta 处理的良好处理中加入3毫升预热 psc 培养基。使用刮刀轻轻但彻底地将细胞从两口井中取出。
- 使用血清学移液器轻轻地移液细胞悬浮液, 并打破大团块。在所有单元格团块都消失之前, 不要移液器。保留 ipsc 群集。
注: 电镀大型团块不会影响 ipsc 菌落的生长。如果有过大的细胞团块, 只需避免将它们转移到下一道菜中。 - 使用血清学移液器, 通过将 0.5 ml 的移液液移入 ecm 涂层的6孔板的每一口井, 均匀地将每个 edta 处理良好的细胞均匀地分配。
注: 不要将重新编程的井中的细胞组合在一起 (即,重新编程的井1应均匀地分布在第一6井板中, 而重新编程的井2应均匀地分布在第二6井板中)。 - 将镀金细胞移回低 o2 孵化器 。要均匀地分配细胞, 请来回晃动每个板块。不要旋转。每天更换 psc 介质。
8. 挑选 ipsc 殖民地
- 预热15毫升的 psc 介质至37°c。按照制造商的说明, 用 hesc 合格的 ecm 为单个6井板的所有油井涂上涂层。用石蜡膜密封盘子, 在 rt 孵育1小时。
- 从用于收集 ipsc 的水井中吸收培养基。用1毫升的 edta 冲洗一次, 吸气。每口添加1毫升 edta。37°C, 孵化4分钟。当细胞孵育时, 从预热板中吸气 ecm 溶液, 每口井用2毫升的 psc 培养基代替。把盘子放在一边。
- 轻轻取出从孵化器中取出的带有 ipsc 的盘子, 放在生物安全柜中。
注: 此时, 细胞可能非常松散粘附, 并且很容易被移出。 - 仔细吸气 edta。非常温和地添加3毫升预热 psc 介质, 注意不要驱逐 ipsc 菌落。
- 将板材移动到倒置或解剖范围, 以更好地可视化殖民地。准备一个1毫升的移液器与无菌的尖端。充分按压柱塞, 然后使用移液器尖端轻轻刮伤菌落, 同时慢慢地将液体吸引到柱中收集菌落。在采摘 ipsc 菌落时, 画尽可能少的介质。
- 要转移菌落, 从步骤8.2 开始, 在 ecm 涂层板的单井中上下液器3-4 次。重复, 直到6个菌落被采摘并转移到各个井中。如果执行了步骤7中描述的可选传递, 则从任何一个井中选择不超过2个菌落。
- 使用标准的人体干细胞协议冻结所有剩余的油井。解冻和重新板块冷冻股票, 如果必须选择更多的殖民地后。
9. ipsc 的特性
- 执行 tra-1-60 染色, 以分析重新编程效率 (第18天)。
注: 3 口重新编程的井中有2口被通过, 用于未来的群体采摘。剩余的井可用于 tra-1-60 染色。此过程可在实验室台面上执行。- 用1毫升的 pbs 清洗重新编程的好。用冰凉甲醇将细胞固定在-20°c 下5分钟。
- 吸入甲醇。把井擦干大约 2分钟. 小心不要过度干燥。当细胞变成半透明的颜色时, 它们已经足够干燥了。
- 用1毫升的 pbs 清洗3口, 用轻微的晃动5分钟。在清洗过程中, 在 pbs 中制备3% 过氧化氢的3毫升。
- 吸收 pbs。加入2毫升稀释的过氧化物溶液。在 rt 用温柔的晃动孵化15分钟。制备4毫升的阻滞溶液: 10% 的牛血清白蛋白 (bsa) 在 pbs 中。
- 吸出过氧化物溶液。用1毫升的 pbs 清洗2口井5分钟。
- 吸气 pbs, 并在井中加入2毫升的阻塞溶液。在 rt 孵化1小时。
- 用1毫升的 pbs 清洗3口, 用轻微的晃动5分钟。
- 稀释原代抗 tra-1-60 抗体在 1: 100 在阻断溶液中补充0.05% 的氮化钠。为6孔格式的盘准备1毫升抗体稀释。将抗体稀释添加到井中, 用副回液包裹板, 以避免蒸发。在4°c 下过夜, 轻轻晃动。
注: 如有必要, 在 rt 处, 用原代抗体进行孵育 1小时, 轻轻晃动。主要抗体稀释可重复使用多达5次。 - 用原代抗体孵育后, 用1毫升 pbs 清洗井 3次, 用轻微晃动5分钟。
- 在阻断溶液中以1:200 稀释继发抗小鼠 hrp 共轭抗体。用二次抗体稀释2小时在 rt 用温和的晃动进行井的培养。
- 用1毫升 pbs 吸干二级抗体稀释, 用1毫升的 pbs 清洗 3次, 轻轻晃动5分钟。
- 在第三次清洗过程中, 按照制造商的指示制备基板溶液。最后洗完之后, 吸气 pbs。加入1毫升的基板溶液, 孵育, 直到所需的颜色发展 (约 10分钟)。
- 吸气基板溶液。在轻轻晃动的同时, 用水冲洗5分钟。
- 如果需要的话, 数一数殖民地。重新编程效率 = (菌落数)/(镀金成纤维细胞数) x 100%。
- 长期储存时, 从染色板中吸气, 并在 rt 风干一夜, 用副体密封干燥的盘子, 并在4°c 下存放长达 2年, 以评估以后的重新编程效率。
结果
从开始成纤维细胞重新编程到冷冻 ipsc 的第一瓶, 通常需要大约5-6周的时间 (图 1)。重新编程协议通常可分为两个阶段。第1阶段包括成纤维细胞培养和七种转染, 每48小时进行一次 rna 鸡尾酒的重新编程, 第2阶段包括隔离、扩张和描述 ipsc 菌落。
在启动协议之前, 应确保重新编程的成纤维细胞质量良好。健康的成纤维细胞应出现纺锤形、双极和折射, 加倍时间约为24小时。到第0天, 250, 000个细胞在一天-2 镀金成10厘米的盘子应生长到40-60% 的融合 (图 1, 第0天), 并产生大约 6-10 x10 5 细胞。在较慢的速度增殖的细胞可以通过在第2天和第0天以更高的密度电镀来补偿重新编程。
在将纤维细胞镀到6孔格式的盘子中进行重新编程后, 成纤维细胞应该显得非常稀疏 (图 2, 第1天)。第一次转染后 24小时, 成纤维细胞将失去纺锤形, 并采用更圆润的形态 (图 2, 第2天), 这是通过重新编程的剩余部分保持的。从 mwasabi mWasabi 的绿色荧光应该是最低限度地观察到第2天和稳步增加的亮度是清晰的第4天。检测芥末荧光的能力取决于示波器的灵敏度和设置。细胞密度将在前三次转染 (第1-6天) 中逐渐持续增加, 在第7天和第8天之间发生明显的增殖。到第10天, 细胞应在很大程度上出现融合 (图 2, 第10天)。最早的 ipsc 菌落最早可以出现在第11天 (图 2, 第11天);然而, 殖民地可能要到第18天才能观察到。一般来说, 到15-18 天, 将有大型和明显的 ipsc 菌落, 与周围的未完全重新编程成纤维细胞明显不同 (图 2, 第15天和图 3, 第17天)。可以对多能性标记 tra-1-60 进行免疫染色, 以评估重新编程效率 (图 3, 第17天, tra-1-60)。根据我们的经验, 大多数成纤维细胞系每重新编程良好就会产生数百种菌落 (图 3, 插入 b)。
在我们的协议中, 次优电镀密度是重新编程效率降低的最常见原因, 并且经常与患病、衰老和高通道的成纤维细胞有关。如果电镀密度太低, 在重新编程结束时将有较大的无细胞贫瘠补丁 (图 4c), 并且 ipsc 菌落可能无法形成 (图 4C)。在第14天之前, 重新编程的单元应该非常融合 (将图 4 a和4A与图 2、第14天进行比较)。同样, 如果细胞被镀得太密或增殖过快, 重新编程的效率就会大大降低。
为了保持患者衍生的 ipsc 的同质性, 从单个菌落中扩展细胞系是非常重要的。由于重新编程效率在我们的协议中非常高, 邻近的 ipsc 菌落可能会在彼此接近的情况下形成并相互成长 (图 3, 第15-17 天)。这有时会使机械分离单个群体进行克隆扩张变得困难。我们发现, 首先把重新编程好的通道, 把它稀释到更大的文化区域, 是有帮助的。一个好的通过率包括均匀地分割一个重新编程的6孔格式板很好地跨越整个6井板。
稀释后, ipsc 作为菌落生长, 很容易与成纤维细胞区分开来 (图 5, 第18天)。最初, ipsc 菌落可能是松散包装, 和单个细胞有一个相对较大的细胞质面积。在4-7天的时间里, ipsc 扩散, 形成一个具有定义边缘的典型的紧密拥挤的群体。菌落内的单个细胞有一个大的核部分, 有突出的核仁 (图 5, 第22天)。每口井中应该有许多菌落形成, 只有那些具有经典 ipsc 形态的菌落才应该被选择进行扩张。
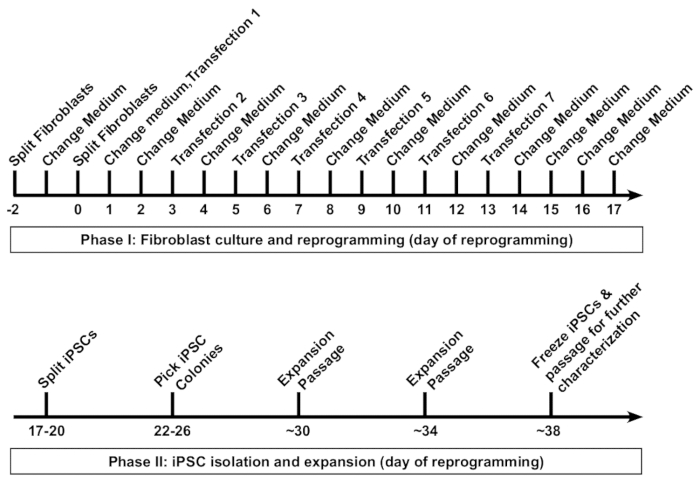
图 1: 将人成纤维细胞重新编程为诱导多能干细胞 (ipsc).提出了一种人成纤维细胞重编程的原理图协议。成纤维细胞首先以低密度被传入6孔格式的盘子中, 然后每隔48小时进行7次转染。每次转染后, 介质被替换为16–20小时。重新编程的 ipsc 在大约第18天首次被传代, 并在第26天之前选择克隆菌落。通常情况下, 纤维细胞衍生的 ipsc 生产线可以在第38天之前冻结以进行长期存储。请点击这里查看此图的较大版本.
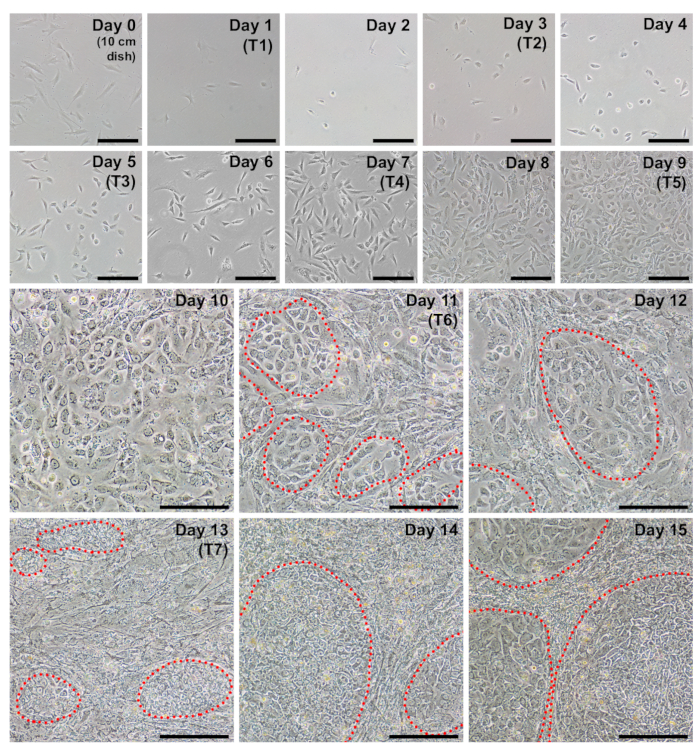
图 2: 在重新编程的每一天中, 每天都有代表性的图像.成纤维细胞在通过时应约为40-60% 的融合, 以启动重新编程 (第0天, 细胞被镀成10厘米的盘子)。第一次转染 (t1) 发生在第1天, 细胞在这一点上应该显得非常稀疏。第二天 (第2天), 更圆润的形态应该会变得明显。在整个协议过程中, 细胞的密度将继续增加, ipsc 集群早在第11天就开始出现 (用红色圈)。到第15天, ipsc 殖民地将是大的与谨慎的边界。刻度栏 = 200μm. 请点击这里查看此图的较大版本.
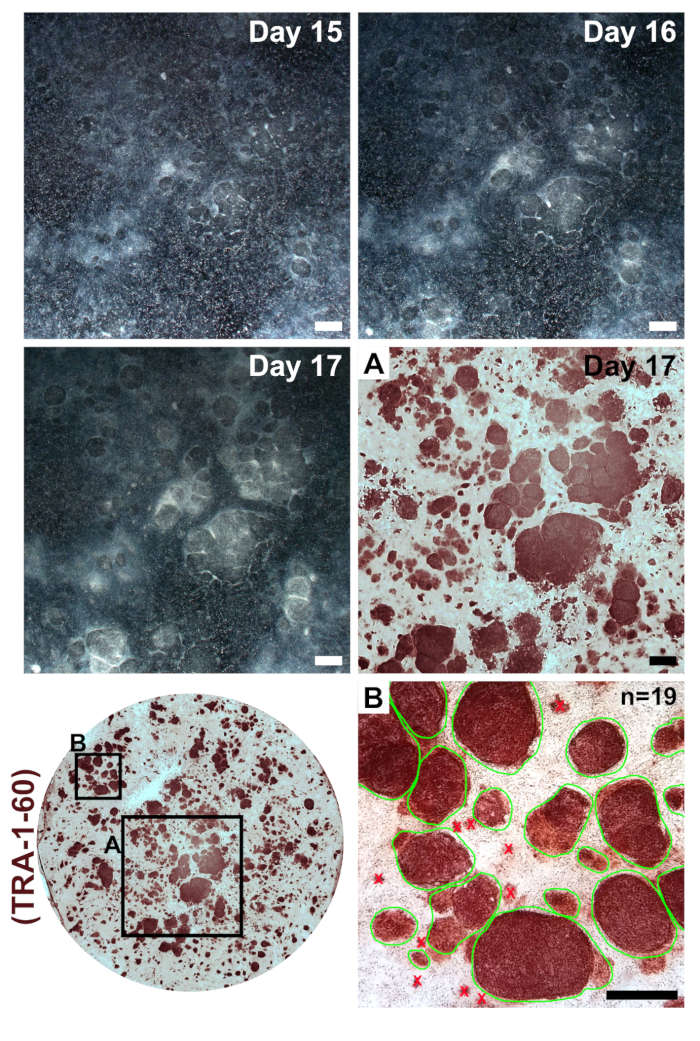
图 3: 通过重新编程修饰的 mrna 和 mirna 模拟, 在转染后形成菌落.在15-17 天拍摄了具有代表性的重新编程时的低放大倍率图像。经过最终转染后, 重新编程的 ipsc 将形成清晰的菌落, 具有明确的边界, 其大小和冷凝程度明显不同于未完全重新编程的成纤维细胞周围。多能性标记 tra-1-60 的免疫染色表明存在 ipsc (插入 a), 可用于计算重新编程效率, 方法是计算单个井内的所有菌落 (插入 b, 绿色圈出的可数菌落的例子)。刻度栏 = 1 毫米. 请点击这里查看此图的较大版本.
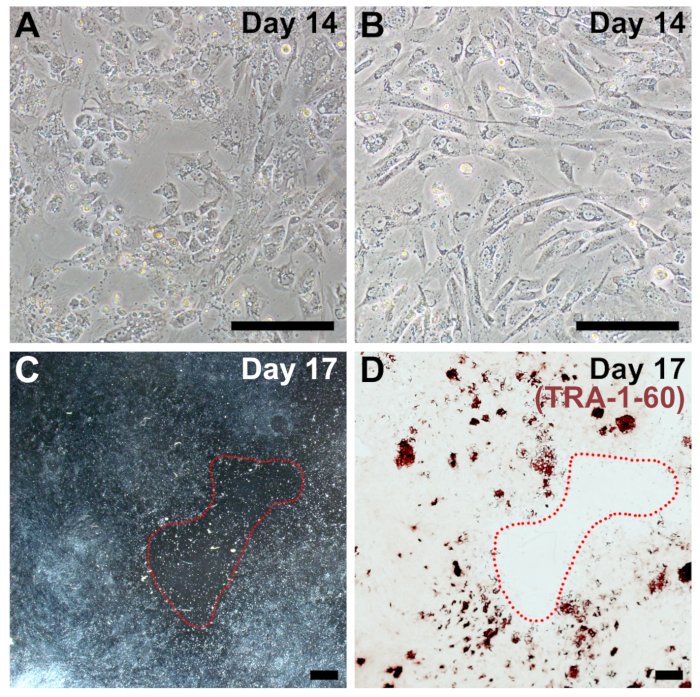
图 4: 具有代表性的子优镀层密度图像, 用于重新编程.(a、b)在重新编程的第14天之前稀疏的成纤维细胞的例子 (与图 2, 第14天相比)。(c) 在重新编程的第17天, 用红色圆圈的大、贫瘠的补丁, 放大倍率较低。(d) tr-1-60 的同一口井被固定和染色, 以确认由于细胞密度低, 重新编程效率总体较差。刻度柱 = 200 微米 (a、b) 和1毫米 (c, d)。请点击这里查看此图的较大版本.
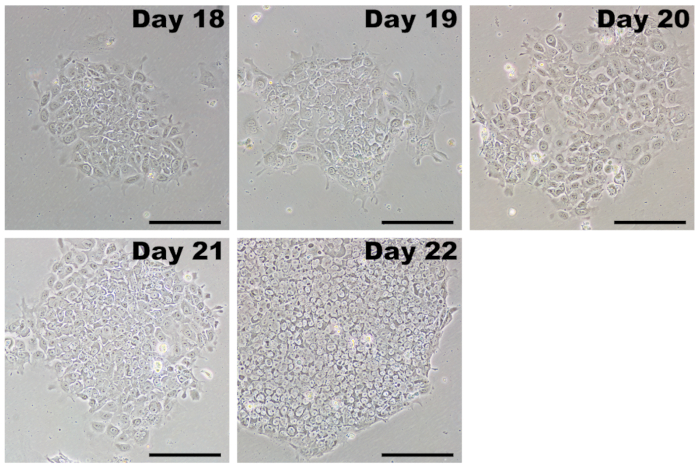
图 5: 初始通过后的 ipsc 的代表性图像.在通过重新编程修改后的 mrna 和 mirna 模拟完成转染后, 重新编程的细胞将被17–20天通过。ipsc 在 psc 介质中具有生长优势, 并迅速超过任何未完全重新编程的成纤维细胞。最初, ipsc 将形成可能在定义不明确的边界下松散的群体。在几天内, 细胞迅速增殖, 并采取紧密包装的细胞具有高核-细胞质比率的特征形态, 紧密聚集到具有明显边界的群体中。刻度栏 = 200μm. 请点击这里查看此图的较大版本.
讨论
该协议描述了一种临床相关的、非集成的、基于 rna 的方法, 该方法允许以超高效率将正常和疾病相关的人成纤维细胞线重新编程到 ipsc 中。到目前为止, 我们尝试使用所述协议重新编程的每一条人成纤维细胞线都为下游应用产生了数量令人满意的高质量 ipsc。由此产生的 ipsc 可以在无弱的培养条件下立即传输和扩展。
用于重新编程的成纤维细胞质量:
重新编程的成功在很大程度上取决于开始成纤维细胞的质量。理想情况下, 应启动重新编程与最低通道成纤维细胞可用, 以实现最高的效率。对于第2–4段的成纤维细胞, 重新编程效率最好。重新编程仍然可以工作的高通道 (通过 5-8), 甚至衰老成纤维细胞, 虽然效率降低。有时低通道成纤维细胞是不可用的或病人样本有一个基因突变, 阻止健康成长。在这种情况下, 可能需要优化初始电镀密度。受损成纤维细胞系的重新编程通常与 rna 转染过程中细胞死亡的增加有关。因此, 重新编程中的细胞在重新编程的10-14天之前会显得稀疏。大的无细胞区域也将在井中可见。如果是这种情况, 重新编程协议将需要重新启动与更高的初始开始数成纤维细胞。在大多数成人成纤维细胞系中, 每井电镀 3, 000个输入细胞, 效果稳定。但是, 将电镀数量增加到 5000–10000 (老化线为 50, 000) 可能有助于改进与疾病相关的样本的重新编程, 正如我们在以前的出版物8中所报告的那样。相反, 过早达到融合的细胞也可以抵抗重新编程。如果通过重新编程 rna 进行转导的细胞增殖过快 (如有时是原发性新生儿成纤维细胞), 则启动重新编程, 每口6孔格式的菜品8每井有500个成纤维细胞.
转染缓冲器的处理:
转染缓冲液 (血清还原培养基调整为 8.2) 的 ph 值对于实现此重新编程协议所需的最佳转染效率至关重要。因此, 建议采取一些有关处理转染缓冲区的预防措施。我们发现, 即使是短暂地暴露在大气中的转染缓冲液也会影响缓冲液的 ph 值。因此, 转染缓冲液应与最小空气空间的螺帽容器 (我们使用5或 15 ml 螺帽锥形管)。为了进一步减少空气暴露, 请使用每个转染缓冲液, 最多两次转染。最后, 由于温度对 ph 值的影响, 将转染缓冲液平衡到 rt 以组装转染配合物是至关重要的。建议不要将转染缓冲液加热到37°c。
isc 的通过:
虽然许多以前发布的协议建议在重新编程结束时选择单个 ipsc 菌落, 但当重新编程的效率非常高或菌落聚集在一起时, 可能很难实现这一点, 我们的情况通常是这样的协议。因此, 如果 ipsc 菌落彼此非常接近, 我们建议首先对细胞进行传代, 以便在手动采摘菌落之前传播重新编程的 ipsc。执行此早期段落步骤有几个优点。分散殖民地给了他们更多的生长空间, 产生了比原来的井更大的采摘群体。这大大提高了从精选克隆建立 ipsc 生产线的成功率。我们还发现, 用成纤维细胞增加培养时间, 尽管比例被稀释, 但似乎能提高被采摘的 ipsc 菌落的平均质量。未完全重新编程的成纤维细胞可能提供支持的旁分泌因子, 这将继续有助于建立 ipsc, 并缓解直接过渡到无弱细胞培养条件。与在 mtesr1 中培养的 ipsc 相比, 成纤维细胞在偶然中具有选择性生长劣势。因此, 污染成纤维细胞群在3-4 通道内迅速稀释到可以忽略不计的数量。
y-276332 等 rock 抑制剂经常用于人体 ipsc 的常规培养。我们发现, 一些使用 y-27632 的 ipsc 线路的频繁和扩展培养会对整体质量产生有害影响。当使用团块传递方法, 如与 edta, y-27632 是不需要保持 ipsc 的生存能力后, 分裂。我们已经完全消除了对 y-27632 的补充介质, 用于所有 ipsc 隔离、扩展或常规培养。
协议限制:
对所描述的基于 rna 的重新编程方法的一个限制是与重新编程试剂的制备相关的初始成本和复杂性。虽然产生 mrna 试剂的准备程序都是常规的, 并已在前面描述过 (pcr, 体外转录, dnase 处理, 封盖, 脱磷酸化, 纯化), 累积 mrna 试剂的生产一个相对漫长和不平凡的过程。该协议的另一个主要挑战是需要每48小时转染一次细胞, 从而提高重新编程协议的劳动强度。如果只需要对少数患者样本进行重新编程, 这些考虑因素可能会令人望而却步。但是, 如果主要考虑的是生成与临床相关的 ipsc 或实现非常高的重新编程效率, 则所描述的基于 rna 的重新编程方法是理想的。
总之, 所描述的基于 rna 的高效重新编程方法取决于优化转染效率的体细胞类型将重新编程, 如我们在以前的出版物8中所述。本研究中提出的 rna 转染协议针对人类原代成纤维细胞进行了高度调整, 但有可能针对其他细胞类型进行定制, 以提高各种体细胞的重新编程效率。
披露声明
i. k. 和 g. b. 是题为 "重新编程细胞的方法和组合" 的专利申请的共同发明者, pct 申请号。PCT/US2016/063258。
致谢
我们感谢国家卫生研究院 (t32ar007411-33) 和科罗拉多大学皮肤疾病研究中心 (p30ar057 212) 的资助。我们还感谢 eb 研究伙伴关系、eb 医学研究基金会、治疗 eb 慈善、营养不良性巨无酸研究协会 (debra)、baudouin 基金会的 vlinderkindje 国王基金和盖茨前沿基金。
材料
| Name | Company | Catalog Number | Comments |
| Plasmid templates for PCR | |||
| pcDNA3.3_KLF4 | Addgene | 26815 | |
| pcDNA3.3_SOX2 | Addgene | 26817 | |
| pcDNA3.3_c-MYC | Addgene | 26818 | |
| pcDNA3.3_LIN28A | Addgene | 26819 | |
| pCR-Blunt_hM3O | Addgene | 112638 | |
| pCR-Blunt_hNANOG | Addgene | 112639 | |
| pCR-Blunt_mWasabi | Addgene | 112640 | |
| Modified mRNA in vitro transcription and miRNA mimics | |||
| Forward Primer | Integrated DNA Technologies | TTGGACCCTCGTACAGAAGC TAATACG | |
| Reverse Primer (Ordered as ultramer, 4nmol scale) | Integrated DNA Technologies | TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT CTTCCTACTCAGGCTTTATTCA AAGACCA | |
| (ARCA Cap) 3´-0-Me-m7G(5')ppp(5')G | New England Biolabs | S1411S | |
| Pfu Ultra II Hotstart 2x Master Mix | Agilent | 600850-51 | |
| 5-Methylcytidine-5'-Triphosphate | Trilink Biotechnologies | N-1014 | |
| Antarctic Phosphatase | New England Biolabs | M0289L | |
| DNase I | NEB | M0303S | |
| MEGAscript T7 Transcription Kit | ThermoFisher Scientific | AM1334 | |
| Pseudouridine-5'-Triphosphate | Trilink Biotechnologies | N-1019 | |
| Riboguard RNase Inhibitor | Lucigen | RG90910K | |
| RNA Clean & Concentrator | ZymoResearch | R1019 | |
| Syn-hsa-miR-302a-3p miScript miRNA Mimic | Qiagen | MSY0000684 | |
| Syn-hsa-miR-302b-3p miScript miRNA Mimic | Qiagen | MSY0000715 | |
| Syn-hsa-miR-302c-3p miScript miRNA Mimic | Qiagen | MSY0000717 | |
| Syn-hsa-miR-302d-3p miScript miRNA Mimic | Qiagen | MSY0000718 | |
| Syn-hsa-miR-367-3p miScript miRNA Mimic | Qiagen | MSY0000719 | |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9937 | Use to dilute modified mRNAs and miRNA mimics |
| Fibroblast culture and reprogramming | |||
| 0.1% Gelatin in H2O | StemCell technologies | #07903 | |
| Stericup-GV Sterile Vacuum Filtration System | EMD Millipore | SCGVU05RE | Use to sterilize the transfection buffer and 0.5 mM EDTA in DPBS |
| 2-Mercaptoethanol | ThermoFisher Scientific | 21985023 | |
| 6-well plates (tissue culture treated) | Corning | 3516 | |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | |
| Fetal Bovine Serum | Fisher | 26-140-079 | |
| FGF Basic | ThermoFisher Scientific | phg0263 | |
| GlutaMax Supplement | ThermoFisher Scientific | 35050061 | Glutamine supplement used for the prepration of media |
| Heat Inactivated FBS | Gibco Technologies | 10438026 | |
| KnockOut Serum Replacement | ThermoFisher Scientific | 10828010 | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher Scientific | 13778500 | The protocol is optimized for the Lipofectamine RNAiMax transfection reagent |
| MEM | ThermoFisher Scientific | 11095080 | |
| MEM Non-essential amino acids | ThermoFisher Scientific | 11140050 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985070 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 500 mL |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985062 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 100 mL |
| 10 M NaOH | Sigma-Aldrich | 72068 | Make a 1 M solution by diluting in nuclease free water and use for pH adjustment |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9932 | Use to dilute NaOH and wash a pH meter |
| Pen/Strep/Fungizone | GE Healthcare | SV30079.01 | |
| rhLaminin-521 | ThermoFisher Scientific | A29248 | Supplied at a concentration of 100 µg/mL, use as a matrix for the reprogramming procedure |
| Dulbecco's phosphate-buffered saline (DPBS) | Life Technologies | 14190144 | |
| Trypsin-EDTA 0.25% Phenol Red | Life Technologies | 2520056 | |
| Vaccinia Virus B18R (CF) | ThermoFisher Scientific | 34-8185-86 | |
| iPSC culture | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| EDTA, 0.5 M stock solution | K&D Medical | RGF-3130 | Dilute to 0.5 mM in DPBS, filter sterilize and use for iPSC passaging |
| mTeSR1 | StemCell Technologies | 85850 | Pluripotent stem cell (PSC) medium, provides growth advantage to iPSCs over fibroblasts |
| Antibodies and Detection | |||
| Rabbit anti Mouse (HRP conjugated) | Abcam | ab97046 | |
| Tra-1-60 (mouse anti human) | Stemgent | 09-0010 | |
| Hydrogen Peroxide (30%) | LabChem | LC154301 | Dilute to 3% with PBS |
| Bovine Serum Albumin (BSA) | Fisher | BP9703100 | |
| Phosphate-buffered salin (PBS) | Hyclone | SH30258.02 | Supplied as 10x, dilute to 1x |
| VECTOR NovaRED Peroxidase Substrate Kit | Vector Laboratories | SK-4800 | |
| Special Equipment | |||
| Description | Notes | ||
| Biological safety cabinet | |||
| Regular humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 37 °C. | ||
| Tri-gas humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 5% O2, 37 °C. Use for the reprogramming procedure. | ||
| pH Meter | Must have resolution to two decimal places. Designate to RNA work if possible. | ||
| Inverted microscope | Microscope configured to visualize EGFP for monitoring transfection efficiency. |
参考文献
- Takahashi, K., Yamanaka, S. Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors. Cell. 126 (4), 663-676 (2006).
- Takahashi, K., et al. Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors. Cell. 131 (5), 861-872 (2007).
- Schlaeger, T. M., et al. A comparison of non-integrating reprogramming methods. Nature Biotechnology. 33 (1), 58-63 (2015).
- Hacein-Bey-Abina, S., et al. LMO2-Associated Clonal T Cell Proliferation in Two Patients after Gene Therapy for SCID-X1. Science. 302 (5644), 415-419 (2003).
- Warren, L., et al. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7 (5), 618-630 (2010).
- Judson, R. L., Babiarz, J. E., Venere, M., Blelloch, R. Embryonic stem cell-specific microRNAs promote induced pluripotency. Nature Biotechnology. 27 (5), 459-461 (2009).
- Anokye-Danso, F., et al. Highly efficient miRNA-mediated reprogramming of mouse and human somatic cells to pluripotency. Cell Stem Cell. 8 (4), 376-388 (2011).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nature Communications. 9 (1), (2018).
- Hirai, H., et al. Radical acceleration of nuclear reprogramming by chromatin remodeling with the transactivation domain of MyoD. Stem Cells. 29 (9), 1349-1361 (2011).
- Mandal, P. K., Rossi, D. J. Reprogramming human fibroblasts to pluripotency using modified mRNA. Nature Protocols. 8 (3), 568-582 (2013).
- Avci-Adali, M., et al. In vitro synthesis of modified mRNA for induction of protein expression in human cells. Journal of Visualized Experiments. , e51943 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。