Method Article
RNA-basierten Reprogrammierung von menschlichen primäre Fibroblasten in induzierte pluripotente Stammzellen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine klinisch relevante, hohem Wirkungsgrad, Feeder-free Methode, um menschliche primäre Fibroblasten in induzierte pluripotente Stammzellen mit modifizierten mRNAs codieren Neuprogrammierung Faktoren neu zu programmieren und Reife MicroRNA-367/302 imitiert. Ebenfalls enthalten sind Methoden zur Beurteilung der Neuprogrammierung Effizienz klonalen iPSC Kolonien zu erweitern, und Ausdruck der Pluripotenz Markierung TRA-1-60 zu bestätigen.
Zusammenfassung
Induzierte pluripotente Stammzellen (iPSCs) haben sich als ein wertvolles Instrument zur menschlichen Entwicklung und Krankheit zu studieren. Weitere Förderung iPSCs als eine regenerative Therapie erfordert eine sichere, robuste und zweckmäßig Neuprogrammierung Protokoll. Hier präsentieren wir Ihnen eine klinisch relevante, Schritt für Schritt Protokoll für die extrem Hocheffizienz-Reprogrammierung von menschlichen dermalen Fibroblasten in iPSCs mit einem Ansatz nicht zu integrieren. Der Kern des Protokolls besteht Pluripotenz Faktoren zum Ausdruck zu bringen (SOX2, KLF4, cMYC, LIN28A, NANOG, OCT4-MyoD Fusion) von in-vitro- transkribierten Messenger RNA synthetisiert mit Nukleotiden (modifizierte mRNAs) geändert. Die Neuprogrammierung modifizierte mRNAs sind für zwei Wochen in primäre Fibroblasten alle 48 h zusammen mit Reifen embryonalen Stammzell-spezifische MicroRNA-367/302 Mimik transfiziert. Die daraus resultierende iPSC-Kolonien können dann isoliert und direkt im Feeder-freien Bedingungen erweitert. Zur Maximierung der Effizienz und Konsistenz unserer Neuprogrammierung Protokoll über Fibroblasten Proben haben wir verschiedene Parameter einschließlich der RNA Transfektion Therapie optimiert Timing Transfektionen, Kulturbedingungen und seeding dichten. Wichtig ist, erzeugt unsere Methode qualitativ hochwertige iPSCs aus den meisten Fibroblasten-Quellen, einschließlich schwer zu programmieren Kranke, Alter und/oder alternde Proben.
Einleitung
Reprogrammierung von somatischen Zellen in induzierte pluripotente Stammzellen (iPSCs) erfordert erweiterte Ausdruck der einen Kernsatz von Transkriptionsfaktoren, die wichtig sind bei der Aufrechterhaltung der Pluripotenz1,2. Bei der Herstellung von iPSCs für klinische Anwendungen ist es wichtig, dass die Mutationszucht Last Eingabezellen während der Verarbeitung minimiert wird und die Effizienz der iPSC-Generation ist in Patientenproben auf relativ hohem Niveau gepflegt. Allerdings leidet die Mehrheit der Neuprogrammierung Methoden, einschließlich der Integration-freie Protokolle von sehr niedrigen Neuprogrammierung Effizienz, welche Grenze klinische Nützlichkeit dieser3nähert. Die geringe Effizienz der Neuprogrammierung kann auch fördern selektiv Reprogrammierung von bereits vorhandenen Mutationen, Erhöhung der Mutationszucht Belastung in daraus resultierenden iPSCs tragen. Darüber hinaus leiden alle DNA-basierte Neuprogrammierung Methoden, z. B. Lentivirus und episomal-basierte Ansätze, die Sicherheitsbedenken, dass DNA kann zufällig in das Genom zu integrieren und die Möglichkeit für schädliche insertional Mutagenese und unerwünschte schaffen ( potenziell onkogene) Expression von Genen der Pluripotenz in nachgeschalteten Gewebe Derivate4.
Ein vielversprechender Ansatz zur effizienten Induktion von Pluripotenz in somatischen Zellen erreichen und mutagenen Belastung in daraus resultierenden iPSCs verringern ist mit synthetischen angeschnittene Ärmel Messenger-RNAs mit Nucleobasen (modifizierte mRNAs) Umprogrammierung5geändert. Die Effizienz der modifizierte mRNA-basierte Neuprogrammierung Ansätze durch Zugabe von embryonalen Stammzellen (ESC) weiter ausgebaut werden kann-spezifischen MicroRNAs (MiRNAs)-367/302s3, die erwiesenermaßen reprogram Körperzellen mit Effizienzsteigerung6 ,7. Jedoch sogar mit dem Zusatz von MiRNAs-367/302s scheitert die modifizierte mRNA-Neuprogrammierung Ansatz oft während der Anwendung frisch isolierte Patientenzellen3. Zu Adresse Inkonsistenzen dieses modifizierte mRNA-basierten Ansatzes haben wir vor kurzem eine optimierte, Integration-freie Strategie berichtet, die induziert Pluripotenz in menschlichen primäre Fibroblasten mit einer hohen Erfolgsquote und nutzt beide veränderten mRNAs codieren Reprogrammierung Faktoren und Reife MiRNA-367/302 imitiert8. In unserer Methode umfasst die Neuprogrammierung modifizierte mRNA cocktail eine modifizierte Version des OCT4 mit MyoD werden Domäne (genannt M3O)9 und fünf andere Neuprogrammierung Faktoren (SOX2, KLF4 cMYC, LIN28A und NANOG) verschmolzen. Kombiniert die modifizierte mRNA Kodierung der Pluripotenz Faktoren mit der MiRNA schien imitiert einen synergistischen Effekt auf Effizienz in diesem Protokoll Umprogrammierung zu haben. Weitere Optimierungen der RNA Transfektion Regime cell seeding und Kultivierung Bedingungen waren auch nötig, um die Neuprogrammierung Effizienzsteigerung des Ansatzes für eine ultra-high Level8.
Im Gegensatz zu vielen anderen Protokollen erfordert unserer Neuprogrammierung Ansatz in der Regel nur ein paar tausend Eingabe Fibroblasten. Darüber hinaus beinhalten viele gemeinsame nicht-Integration von Strategien mit episomal Plasmide, Sendai-Virus oder selbstreplizierende RNA umfangreiche Passagierung um die Neuprogrammierung Vektor in generierten iPSCs verdünnen. Andererseits veränderte mRNA und Reife MiRNA Mimik haben eine kurze Halbwertszeit und sind schnell aus Zellen eliminiert. Zusammengenommen ist die Menge an kumulative Zelle Kulturzeit zwischen Verwertungsgesellschaften Patientenproben und die Generierung von nutzbaren iPSCs minimal in diesem Ansatz, Mutation Akkumulation im resultierenden iPSCs effektiv zu begrenzen und Wirtschaftlichkeit steigern.
Hier präsentieren wir Ihnen die ausführliche Schritt für Schritt Protokoll zur Erreichung Hocheffizienz Reprogrammierung von Erwachsenen menschlichen Fibroblasten in iPSCs mit unseren kombinatorische modifizierte mRNA/MiRNA-basierten Ansatz8. Diese RNA-basierten Neuprogrammierung Protokoll bietet eine robuste, einfache und kostengünstige Methode zur Erzeugung von Integration-freie iPSCs für klinische Anwendungen in Forschung und Potenzial. Darüber hinaus gilt es für die Reprogrammierung von eine Vielzahl von Fibroblasten Linien schwer zu programmieren, krankheitsassoziierten, einschließlich Alter und alternde Fibroblasten. Abbildung 1zeigt eine schematische Darstellung des Protokolls für eine Anpassung der menschliche Fibroblasten. Das Protokoll beschreibt insbesondere ein Verfahren zur Reprogrammierung drei Brunnen von menschlichen Erwachsenen primäre Fibroblasten in einer 6-Well-Format-Platte. Zwei Brunnen liefern in der Regel eine ausreichende Anzahl von qualitativ hochwertigen iPSC Kolonien. In vielen Fällen einzige nun notwendig ist, und die dritte gut einsetzbar für die Analyse der Neuprogrammierung Effizienz. Bei Bedarf kann die Anzahl der Vertiefungen hochskaliert.
Protokoll
RNase-freie Bedingungen arbeiten Sie und aseptische Techniken wenn möglich. Führen Sie alle Zelle kulturbezogene Manipulationen in einer biologischen Sicherheitsschrank mit aseptischen Techniken. Folgen Sie institutionelle Biosafety Standards für die Arbeit mit menschlichen Zellen.
1. Reagenzien und Geräte zur Vorbereitung der Neuprogrammierung Einleitung
- Bereiten Sie die geändert-mRNA cocktail Codierung Neuprogrammierung Faktoren.
- Folgen Sie die bisher veröffentlichten Protokoll10 um in-vitro- Transkription, Begrenzung, und dephosphorylation Verfahren für jede veränderte mRNA Kodierung Neuprogrammierung Faktor. Nach der letzten Aufbereitungsschritt eluieren die modifizierte mRNA mit Nuklease-freies Wasser, ergänzen die gereinigten modifizierte mRNA-Lösung mit 1 U/µL RNase-Inhibitor quantitate die modifizierte mRNAs mit einem Spektralphotometer und bei-80 ° C für bis zu 6 Monate zu speichern. Bereiten Sie mehrere Aliquote jede veränderte mRNA zur Minimierung der Anzahl der Frost-Tau-wechseln.
Hinweis: Ähnliche Protokolle für modifizierte mRNA Produktion wurden berichtet an anderer Stelle5,8,11. Vorlagen für in-vitro- Transkription können durch PCR-Amplifikation von entsprechenden Plasmid-Vorlagen mit Primer in der Tabelle der Materialien gemäß den zuvor veröffentlichten Protokoll10aufgeführten generiert werden. Plasmid-Vorlagen für jeden Neuprogrammierung Faktor (M3O9, SOX2, KLF4, cMYC, NANOG, LIN28A) und mWasabi (Kontrolle zur Transfektion) von Addgene, ein Non-Profit-Plasmid-Repository verfügbar sind. - Mischen Sie die modifizierte mRNAs in einem molaren Verhältnis von 3:1:1:1:1:1 (M3O: SOX2: KLF4: cMYC: NANOG: LIN28A) und beinhalten 10 % mWasabi modifizierte mRNA als ein Steuerelement für die Transfektion Effizienz. Passen Sie die Konzentration der komplette modifizierte mRNA Umprogrammierung mischen, um eine Endkonzentration von 100 ng/µL indem Nuklease-freie Wasser mit 1 U/µL RNase-Inhibitor ergänzt. Bereiten Sie sieben 33 µL-Aliquots der kompletten geändert mRNA-Cocktail. Speichern der gemischten Neuprogrammierung Aliquote bei-80 ° C.
Hinweis: für jede Transfektion 1.000 ng der modifizierte mRNA-Cocktail wird pro Bohrloch hinzugefügt (d. h. insgesamt 3.000 ng für 3 Brunnen). Jede 33 µL aliquoten ist bemessen, um 3 Brunnen einer 6-Well-Format-Platte transfizieren und umfasst 3 µL überschüssige Menge zu pipettieren Fehler ausmachen. Vorbereitung sieben 33 µL-Aliquots ist ausreichend, füllen Sie eine volle Fibroblasten Neuprogrammierung des 3 Brunnen in einer 6-Well-Format-Platte.
- Folgen Sie die bisher veröffentlichten Protokoll10 um in-vitro- Transkription, Begrenzung, und dephosphorylation Verfahren für jede veränderte mRNA Kodierung Neuprogrammierung Faktor. Nach der letzten Aufbereitungsschritt eluieren die modifizierte mRNA mit Nuklease-freies Wasser, ergänzen die gereinigten modifizierte mRNA-Lösung mit 1 U/µL RNase-Inhibitor quantitate die modifizierte mRNAs mit einem Spektralphotometer und bei-80 ° C für bis zu 6 Monate zu speichern. Bereiten Sie mehrere Aliquote jede veränderte mRNA zur Minimierung der Anzahl der Frost-Tau-wechseln.
- Bereiten Sie den Cocktail der Neuprogrammierung MiRNA imitiert.
- Auflösen, lyophilisiert MiRNA Mimik (Syn-hat-MiR-302a - 3p, Syn-hat-MiR-302b - 3p, Syn-hat-MiR - 302c - 3p, Syn-hat-MiR-302d-3 p, Syn-hat-MiR-367-3 p), eine Endkonzentration von 5 Pmol/µL (5 µM) in Nuklease-freies Wasser mit 1 U/µL RNase-Inhibitor ergänzt. Bereiten Sie mehrere Aliquote jede MiRNA Mimik und bei-80 ° C für langfristige Lagerung Lagern.
- Mischen Sie alle MiRNA Mimik in einem Molverhältnis von 1:1:1:1:1, eine Endkonzentration von 5 Pmol/µL (5 µM). Bereiten Sie sieben 14 µL-Aliquots von der MiRNA imitiert Mischung. Speichern der gemischten Aliquote bei-80 ° C.
Hinweis: Für jede Transfektion wird 20 Pmol des MiRNA-Mimik-Mix pro Bohrloch (d. h. insgesamt 60 Pmol für 3 Brunnen) hinzugefügt. Jedes 14 µL aliquoten ist bemessen, um 3 Brunnen einer 6-Well-Format-Platte transfizieren und beinhaltet 2 µL überschüssige Menge zu pipettieren Fehler ausmachen. Vorbereitung sieben 14 µL-Aliquots ist ausreichend, füllen Sie eine volle Fibroblasten Neuprogrammierung des 3 Brunnen in einer 6-Well-Format-Platte.
- Bereiten Sie die Transfektion Puffer.
- Vorwärmen einer 500 mL-Flasche und eine 100 mL Flasche frisch reduziert Serum Mittel-bis ca. 2 h bei Raumtemperatur (RT). Verwenden Sie kein Wasserbad.
- Übertragen Sie einen pH-Meter auf eine biologische Schrank. Waschen Sie das Messgerät Glaselektrode mit Nuklease-freies Wasser. Kalibrieren Sie das pH-Meter gemäß den Anweisungen des Herstellers. Waschen Sie die Elektrode wieder mit Nuklease-freies Wasser.
- Übertragen Sie beide Flaschen reduziert Serum Medium in der Biosicherheit Schrank. Verwenden Sie zur Einstellung des pH-Wertes der RT 500 mL Flasche.
- Messen Sie den Basen pH-Wert des Mediums reduziert Serum durch Einfügen des pH-Messgerätes Glaselektrode in den Puffer. Warten Sie bis zu 1 min vor dem Lesen des pH-Werts auf dem Zähler.
- Fügen Sie 3 – 4 mL 1 M NaOH in 500 mL reduziert Serum Medium, verschließen Sie die Flasche und mischen Sie gut. Warten Sie bis zu 5 min, vor dem Öffnen der Flasche für pH-Messung. Legen Sie das pH-Meter-Elektrode in den Puffer und warten Sie, bis die Lesung am pH-Meter stabilisiert.
- Weitere kleine Volumen von 1 M NaOH hinzu, bis der pH-Wert des Mediums reduziert Serum 8.15 – 8.17 erreicht. Kalibrieren Sie das pH-Meter mehrmals während des Prozesses. Wenn der pH-Wert zu jedem Zeitpunkt höher als 8,18 wird, reduzieren sie durch Zugabe von frischen reduziert Serum Medium aus der vorgewärmten 100 mL-Flasche (Schritt 1.3.1).
Hinweis: Der pH-Wert des Puffers reduziert Serum Medium-basierte Transfektion ist wichtig. Umprogrammierung wird erfolgreich sein, wenn der pH-Wert des Puffers Transfektion etwa 8,2 ± 0,05 ist aber fehlschlagen kann, wenn der pH-Wert höher als 8,25 ist. Daher ist es ratsam zu stoppen, NaOH hinzufügen, sobald der Puffer pH 8,15-8.17 erreicht. Dadurch wird sichergestellt, dass der endgültige pH-Wert des Puffers Transfektion ca. 8,2 – 8,22 nach der Sterilisation. - Filter zu sterilisieren den Transfektion Puffer mit einem 0,22 µm Vakuumfiltration System. Aliquot der sterilisierten Puffer in 5 oder 15 mL Röhrchen mit minimalen Luftraum.
- Lagern Sie Aliquote des Puffers Transfektion für bis zu 3 Monate bei 4 ° c Messen Sie den pH-Wert der Aliquote in regelmäßigen Abständen. Die Aliquote zu verwerfen, wenn der pH-Wert höher als 8,25. Begrenzen Sie aliquoten Nutzung, 2 Transfektionen, nur da Exposition des Puffers Transfektion, atmosphärische Luft den pH-Wert des Puffers erhöht.
- Bereiten Sie die Fibroblasten Erweiterung Medium (FEM): minimale wesentliche Medium mit 10 % fetalen bovine Serum (FBS), 1 x 1 X Glutamin ergänzen und nicht-essentiellen Aminosäuren, 55 µM 2-Mercaptoethanol, 1 X Pen/Strep/Fungizone ergänzt. Speichern der FEM bei 4 ° C.
- Vorbereiten der Neuprogrammierung Medium: DMEM/F12 (keine HEPES) mit 20 % Ko Serum Ersatz (KOSR), 0,5 x nicht-essentiellen Aminosäuren, 0,5 X Glutamin ergänzen, 55 µM 2-Mercaptoethanol, 50 µg/mL Ascorbinsäure, 1 X Stift/Strep/Fungizone, 100 ng/mL bFGF ergänzt , und 200 ng/mL B18R. Bereiten Sie das Medium bei Einleitung der Reprogrammierung ohne bFGF und B18R, und bei 4 ° c lagern Nicht nach 1 Monat nach Vorbereitung verwenden. Fügen Sie bFGF und B18R unmittelbar vor jedem Gebrauch eine aliquote Größe für diesen Tag nutzen hinzu.
- Bereiten Sie die Beschichtung Medium durch Zugabe von 5 % Hitze-inaktivierten (HI) FBS in der Neuprogrammierung Medium mit 20 % KOSR ohne bFGF und B18R aus Schritt 1.5. Am Tag der beabsichtigten Verwendung der mittlere frisch zubereiten. Fügen Sie bFGF und B18R unmittelbar vor der Anwendung am Tag 0 der Neuprogrammierung.
- Kalibrieren der Gewebekultur Inkubatoren vor dem Beginn der Reprogrammierung nach den Anweisungen des Herstellers mit einem handheld digital CO2 -Analysator. Verwenden Sie einen Low-O2 -Inkubator für die Neuprogrammierung Schritte zur erfolgreichen iPSC Generation sorgen.
2. Kultivierung Fibroblasten Umprogrammierung
- Bereiten Sie Fibroblasten vor Einleitung einer Neuprogrammierung (Tag -2).
- Eine neue 10 cm Gewebekultur Platte mit 5 mL 0,1 % Gelatine zu beschichten. Tippen Sie auf oder schwenken Sie die Platte, um sicherzustellen, dass die gesamte Oberfläche beschichtet ist. 15 min bei 37 ° c inkubieren Aspirieren Sie Gelatine und 10 mL FEM Set beiseite. Lassen Sie sich nicht die beschichtete Oberfläche der Platte zum Trocknen vor der Zugabe FEM.
- Aspirieren Sie sorgfältig das abgebrannte Medium aus der Fibroblasten. Spülen Sie die Zellen einmal mit 5 mL des DPBS, das restliche Serum zu entfernen. 3 mL Trypsin-EDTA hinzugeben. Schütteln Sie die Platte, um vollständige Abdeckung über der Zellen zu gewährleisten.
- Aspirieren Sie überschüssigen Flüssigkeit, ~ 500 µL von Trypsin zu verlassen. Nicht übermäßig abzusaugen, da kann das Trocknen und die Fibroblasten zu töten. Inkubieren Sie die Fibroblasten mit Trypsin-EDTA für 3 min bei 37 ° c
- Entfernen Sie die Platte aus dem Inkubator und fest aber vorsichtig auf die Seite der Platte, die Zellen zu verdrängen. Überprüfen Sie die Zellen unter dem Mikroskop. Wenn die Zellen abgetrennt und schwebend sind, fahren Sie fort. Wenn die Zellen noch hängen, für weitere 3 min inkubieren.
- Schnell spülen/sammeln der freistehenden Zellen mittels FEM 5 mL Trypsin-EDTA neutralisieren. Bewegen Sie die Fibroblasten-Suspension in einem 15 mL konische Röhrchen. Stellen Sie sicher, dass die Zellen gut gemischt sind.
- Mischen Sie vorsichtig die Zellsuspension um große Klumpen von Zellen zu brechen, dann zählen sie auf eine Hemocytometer. Transfer 2,5 x 105 Zellen in die Gelatine beschichtet 10-cm-Schüssel in Schritt 2.1.1 vorbereitet. Inkubieren Sie die Zellen über Nacht bei 37 ° C, 5 % CO2 befeuchtete regelmäßige Gewebekultur Inkubator.
Hinweis: Die Zelldichte ist entscheidend für hohe Neuprogrammierung Effizienz zu erreichen, da es wichtig ist, dass die Fibroblasten gesund und schnell teilenden sind. Beschichtung 2,5 x 105 Zellen ergibt sich eine 40-60 % Konfluenz 2 Tage später für die meisten Fibroblasten Proben. Passen Sie die Menge entsprechend für kranke oder alternde Zellen, die gewünschte 40-60 % Konfluenz 48 h später zu erreichen. - Ersetzen Sie das Medium mit 10 mL frisches FEM am Folgetag (Tag-1).
- Platte die Fibroblasten zur Neuprogrammierung (Tag 0) zu initiieren.
- Überprüfen Sie die Fibroblasten umprogrammiert werden bei 40 – 60 % Konfluenz (Abbildung 2, Tag 0). Wenn die Zellen über- oder unter Zusammenfluss sind, Durchgang der Fibroblasten, wie unter Punkt 2.1 beschrieben und die Dichte Beschichtung entsprechend anzupassen. Kultur für weitere 2 Tage.
- Überführen Sie 4 mL des DPBS in ein 15 mL konische Röhrchen. Fügen Sie 100 µL der rekombinanten menschlichen (Rh) Laminin-521. Pipette rauf und runter, um gründlich zu mischen.
- Fügen Sie 1 ml pro Bohrloch des verdünnten RhLaminin-521 in 3 Brunnen von einem 6-Well-Platte. Inkubieren Sie die beschichtete Platte bei 37 ° C für 2 h.
- 6 mL FEM und 4 mL Beschichtung Mittel-bis 37 ° c warm Ergänzen Sie die Beschichtung Medium mit bFGF, eine Endkonzentration von 100 ng/mL und B18R, eine Endkonzentration von 200 ng/mL.
- Aspirieren Sie sorgfältig das abgebrannte Medium von Fibroblasten (Schritt 2.2.1). Spülen Sie die Zellen mit 5 mL des DPBS und 3 mL Trypsin-EDTA. Schütteln Sie die Platte, um die Zellen mit Trypsin-EDTA zu decken.
- Aspirieren Sie überschüssigen Flüssigkeit, ~ 500 µL von Trypsin, verlassen, wie in Schritt 2.1.3 getan. Inkubieren Sie die Fibroblasten mit Trypsin-EDTA für 3 min bei 37 ° c Entfernen Sie die Platte aus dem Inkubator und fest aber vorsichtig auf die Seite der Platte, Zellen zu verdrängen. Überprüfen Sie die Zellen unter dem Mikroskop. Wenn die Zellen abgetrennt und schwebend sind, fahren Sie fort. Wenn die Zellen noch hängen, für weitere 3 min inkubieren.
- Spülen/sammeln der freistehenden Zellen mittels FEM 5 mL Trypsin-EDTA neutralisieren. Bewegen Sie die Fibroblasten-Suspension in einem 15 mL konische Röhrchen. Stellen Sie sicher, dass die Zellen gut gemischt sind und auf eine Hemocytometer zählen. Pipette 12.000 Zellen in 4 mL Medium vorgewärmte Überzug aus Schritt 2.2.4.
Hinweis: Zentrifugieren Sie die Zellen an jedem beliebigen Punkt nicht. Die Anzahl der vergoldeten Zellen einstellbar nach oben oder unten je nach Bedarf für langsam oder schnell-wachsende Linien, beziehungsweise. - Entfernen Sie die beschichtete Platte aus dem Inkubator und aspirieren Sie verdünnte RhLaminin-521 aus den beschichteten Vertiefungen. Lassen Sie nicht die Oberfläche der Brunnen trocken. Aufschwemmen Sie sanft Zellen Mittel-und Beschichtung. 1 mL Zellsuspension in jedem beschichtet Pipette gut (d. h. 3.000 Zellen pro Well).
- Legen Sie die vergoldeten Zellen in ein Trigas-Gewebekultur Inkubator mit O2 bis 5 % (Low-O2) festgelegt. Sobald die Platte nach unten festgelegt ist, Dispergieren Sie sanft aber gründlich Zellen durch den Wechsel zwischen einer nach oben/unten und links/rechts Bewegung. Wiederholen Sie die Anträge 2 weitere Male. Inkubieren Sie die Zellen über Nacht. Schwenken Sie die Platte zu mischen nicht.
- Reprogrammierung Mittel-bis eine 15 mL konische Rohr 4 mL Pipette und legen Sie sie in den Low-O2 Inkubator mit einer gelockerten Kappe, über Nacht equilibrate. Fügen Sie bFGF und B18R nicht bis zum nächsten Tag.
3. Einleitung des Reprogramming (Tag 1)
Hinweis: Sobald die Reprogrammierung initiiert wird, ist tägliche Wartung erforderlich für ca. 1 Monat. Achten Sie darauf, entsprechend zu planen. Alle nachfolgenden Zelle Inkubationen müssen unter Low-O2 Bedingungen im 37 ° C, 5 % CO2, 5 % O2 befeuchtete Trigas Gewebekultur Brutkasten durchgeführt werden.
- Ersetzen Sie das Überzug Medium mindestens 1 h vor Transfektion.
- Entfernen Sie die äquilibriert Neuprogrammierung Mittel aus dem Low-O2 -Inkubator. Fügen Sie bFGF hinzu, eine Endkonzentration von 100 ng/mL und B18R, eine Endkonzentration von 200 ng/mL. Mischen Sie gut.
- Verfahren mit 1 gut zu einem Zeitpunkt, eine 1-mL-Pipette verwenden Sie, um das verbrauchte Medium zu entfernen und ersetzen Sie es mit 1 mL der Neuprogrammierung Medium mit bFGF und B18R ergänzt. Wiederholen Sie diesen Vorgang für jedes gut. Verwenden Sie keine Vakuum Aspiration, die übermäßig trocknet die Zellen, verursacht Stress und Neuprogrammierung Effizienz reduziert. Die Platte mit Zellen in den Low-O2 Inkubator zurück.
- Transfizieren Fibroblasten mit 5 µL RNAiMax Transfection Reagens pro 1 µg der modifizierte mRNA, und 1 µL des Transfection Reagens pro 6 Pmol von MiRNA imitiert pro Transfektion.
Hinweis: Optimale Ergebnisse werden erzielt, wenn Transfektionen für 16-20 h gehen. Die Transfektion Reagenz ist verdünnter 10 X; die 100 ng/µL modifizierte mRNA cocktail ist verdünnt 5 X; und die 5 Pmol/µL MiRNA imitiert, die Mischung ist verdünnt 8,33 x mit der Transfektion Puffer vor der Komplexierung. Eine Zusammenfassung der Transfektion Setup finden Sie unter Tabelle 1 .- Während der Vorbereitung der Transfektion mischt, um zu minimieren potentielle Reagenzien RNase indem Sie die folgenden standard-Labor-Praxis.
- Equilibrate ein Aliquot des Puffers Transfektion (Schritt 1.3) für ca. 1 h bei RT Verwenden Sie eine 37 ° C Wasserbad oder Inkubator nicht zum Aufwärmen des Transfektion Puffers.
- Entfernen einer einzigen aliquoten modifizierte mRNAs (33 µL) und MiRNA imitiert (14 µL) von-80 ° C und wärmen sie auf RT für ca. 3-5 min bis aufgetaut. Nicht Auftauen der Aliquote bei 37 ° C. Drehen sie sich kurz in ein Microfuge.
- Wärmen Sie die Transfektion Reagenz RT ca. 3-5 Minuten. Verwenden Sie keine 37 ° C Wasserbad oder Inkubator. Invertieren der geschlossenen Rohr 2 – 3 Mal um das Reagenz zu mischen. Drehen Sie es nach unten kurz in ein Microfuge.
- Übertragen Sie 279 µL des Puffers RT Transfektion in eine RNase-freie Microcentrifuge Schlauch. Fügen Sie 31 µL des Transfection Reagens zu einem Endvolumen von 310 µL. Mischung gründlich durch pipettieren. Tun Sie nicht Wirbel. Inkubieren Sie das Rohr bei RT für 1 min.
Anmerkung: Dieses Volumen des verdünnten Transfection Reagens reicht bis hin zu komplexen modifizierte mRNA und MiRNA imitiert Aliquote aus Schritt 3.2.3. - Die 33 µL aliquoten modifizierte mRNA 132 µL des Puffers RT Transfektion hinzufügen. Pipette vorsichtig mischen: Endvolumen ist 165 µL.
- 14 µL aliquoten von MiRNA Fließbildern 102,6 µL des Puffers RT Transfektion hinzufügen. Pipette vorsichtig mischen: Endvolumen ist 116.6 µL.
- Fügen Sie 165 µL des verdünnten Transfection Reagens aus Schritt 3.2.5 zu den verdünnten mRNA aus Schritt 3.2.6 geändert. Pipette zum Mischen: Endvolumen ist 330 µL.
- Die verdünnte MiRNA Mimik Mischung aus Schritt 3.2.7 116.6 µL des verdünnten Transfection Reagens aus Schritt 3.2.5 hinzufügen. Gut mischen (Endvolumen ist 233.2 µL). 15 min bei RT erlauben die Transfektion Puffer bis hin zu komplexen mit der modifizierten mRNAs inkubieren und MiRNA imitiert.
- Entfernen Sie die Platte mit Zellen aus dem Low-O2 -Inkubator. Fügen Sie 100 µL (1 µg) die komplexiert mRNA Transfektion Mischung in jede Vertiefung tropfenweise die gut rüber geändert. Die Transfektion komplexe durch sanft aber gründlich rühren die Platte mit einer nach oben/unten und links/rechts Bewegung zu zerstreuen. Schwenken Sie die Platte zu mischen nicht.
- Fügen Sie 66,7 µL (20 Pmol) komplexiert MiRNA Mimik Transfektion Mix in jede Vertiefung tropfenweise das gut rüber. Transfektion komplexe durch sanft aber gründlich rühren die Platte mit einer nach oben/unten und links/rechts Bewegung zu zerstreuen. Schwenken Sie die Platte zu mischen nicht.
- Setzen Sie die transfizierten Zellen Trigas-Brutkasten mit O2 bis 5 % (Low-O2) festgelegt. Sobald die Platte nach unten festgelegt ist, verteilen Sie sich die Transfektion komplexe wieder sanft aber gründlich rühren die Platte mit einer nach oben/unten und links/rechts Bewegung.
- Reprogrammierung Medium in einer 15 mL konische Rohr 4 mL Pipette und legen Sie sie in den Low-O2 Inkubator mit gelösten Deckel über Nacht equilibrate. Fügen Sie bFGF und B18R nicht bis zum nächsten Tag.
| Rohr 1 - RNAiMax-Verdünnung (1. Mix) | ||
| Reagenz | Konzentration | Volumen |
| Transfektion Puffer | 279 ΜL | |
| RNAiMax (add 2) | 10 x | 31 ΜL |
| Gesamt: 310 µL | ||
| (inkubieren Sie 1 min bei Raumtemperatur) | ||
| Rohr 2 - modifizierte mRNA mix (2. Mischung) | ||
| Reagenz | Konzentration | Volumen |
| modifizierte mRNA-Mischung | 100 ng/µL (5 X) | 33 ΜL |
| Transfektion Puffer | 132 ΜL | |
| Gesamt: 165 µL | ||
| (gleiches Volumen des verdünnten RNAiMax aus Rohr 1 hinzufügen) | ||
| Rohr 3 - MiRNA Mimik mix (3. Mischung) | ||
| Reagenz | Konzentration | Volumen |
| MiRNA imitiert Mischung | 5 Pmol/µL (8,33 X) | 14 ΜL |
| Transfektion Puffer | 102,6 ΜL | |
| Gesamt: 116.6 µL | ||
| (gleiches Volumen des verdünnten RNAiMax aus Rohr 1 hinzufügen) | ||
Tabelle 1: Vorbereitung der Transfektion Mischung.
4. ersetzen Reprogramming Medium zwischen Transfektionen (2, 4, 6, 8, 10 und 12 Tage)
- Die mittleren 16-20 h nach Transfektion zu ersetzen, wie in 3.1.1–3.1.2 beschrieben. Hinzu kommt eine Endkonzentration von 100 ng/mL und B18R, eine Endkonzentration von 200 ng/mL in Neuprogrammierung mittlere Aliquote bFGF.
- Überwachen Sie mWasabi Ausdruck täglich Transfektion Qualität unter Verwendung eines Mikroskops, so konfiguriert, dass EGFP visualisieren zu bestätigen.
Hinweis: mWasabi Ausdruck sollte minimal ersichtlich sein, am 2. Tag und Erhöhung der Helligkeit mit jeder zusätzlichen Transfektion.
(5) jeden zweiten Tag Transfektionen (3, 5, 7, 9, 11 und 13 Tage)
- Führen Sie die Transfektion, wie in Schritt 3.2 beschrieben. Ändern Sie das Medium nicht auf Tage der Transfektion.
- Bereiten Sie eine 4 mL Aliquot der Neuprogrammierung Medium und legen Sie sie in den Low-O2 Inkubator für die mittlere Änderung am Folgetag equilibrate. Fügen Sie bFGF und B18R nicht bis zum nächsten Tag.
6. Verfahren nach endgültigen Transfection durchgeführt werden
- Führen tägliche mittlere Änderungen vom 14. Tag bis ca. 17. Tag. 7 mL der Neuprogrammierung Mittel-bis 37 ° c warm Fügen Sie bFGF hinzu, eine Endkonzentration von 100 ng/mL.
Hinweis: B18R entfällt nach der endgültigen Transfection. Über Tag 14 ist es nicht mehr notwendig, das Medium über Nacht im Brutschrank Low-O2 equilibrate. - Entfernen Sie das Medium aus allen Brunnen mit einer serologischen Pipette. Weiterhin vermeiden Sie die Verwendung von Aspiration. Hinzugeben Sie 2 mL Medium ergänzt mit bFGF pro Bohrloch Umprogrammierung.
- Analysieren Sie an den Tagen, 17 und 18 die umprogrammierten Brunnen unter einem invertierten oder sezierenden Mikroskop. Wenn Kolonien sich in der Nähe zueinander durch hohen Wirkungsgrad der Neuprogrammierung bilden und nicht manuell isoliert werden kann, trennen Sie die Kolonien durch eine optionale Passagierung umprogrammierten Brunnen in Schritt 7 unten beschrieben durchführen. Wenn Kolonien spärlich und gut getrennt sind, manuell wählen Sie die Klone direkt aus der umprogrammierten nun, wie in Schritt 8 und Subkultur iPSCs nach Standardprotokollen beschrieben aus.
7. Optional Verfahren: Passagierung Zellen aus umprogrammiert Brunnen mit EDTA
- Bereiten Sie 0,5 mM EDTA in DPBS (EDTA vor) durch Verdünnung 0,5 M EDTA Vorratslösung. Filter zu sterilisieren EDTA mit einem 0,22 µm Vakuumfiltration System.
- Bereiten Sie Feeder-freie pluripotenten Stammzellen (PSC) Medium (z. B. mTeSR1) entsprechend den Anweisungen des Herstellers. 32 mL PSC Mittel-bis 37 ° c Vorwärmen
- Alle Brunnen aus zwei 6-Well-Format-Platten mit hESC-qualifizierte extrazelluläre Matrix (ECM) nach den Anweisungen des Herstellers zu beschichten. Dichtplatten mit Paraffin zu Filmen und inkubieren sie für 1 h bei RT
- Aspirieren der ECM-Lösung von den vorgewärmten Tellern und ersetzen Sie es mit 2 mL PSC Medium pro Bohrloch. Lassen Sie nicht die Oberfläche der Brunnen trocken. Legen Sie sie beiseite.
- Aspirieren der Neuprogrammierung Medium aus 2 umprogrammierten Brunnen an einem Tag, wenn die Kolonien groß und klar geformte sind (in der Regel Tag 18). Spülen Sie einmal mit 1 mL EDTA und Absaugen.
- Fügen Sie 1 mL EDTA pro Bohrloch. 4 min bei 37 ° c inkubieren
- Schonend entfernen Sie die Platte aus dem Inkubator und legen Sie sie in die Biosicherheit Kabinett.
Hinweis: an dieser Stelle können Zellen sehr locker eingehalten und leicht verdrängt werden. - Aspirieren Sie sorgfältig EDTA aus beiden Brunnen. Fügen Sie 3 mL vorgewärmten PSC Medium zu jedem gut behandelt mit EDTA. Verwenden Sie einen Zelle-Schaber, um sanft aber gründlich Zellen aus den beiden Brunnen zu verdrängen.
- Verwenden einer serologischen Pipette vorsichtig pipette die Zellsuspension und unterteilen Sie große Klumpen. Nicht pipette, bis die Zelle Klumpen verschwunden sind. Erhalten Sie iPSC-Cluster.
Hinweis: Große Klumpen Plating iPSC Kolonie Auswuchs wirkt nicht. Wenn es übermäßig große Zelle Klumpen gibt, vermeiden Sie einfach in das nächste Gericht zu übertragen. - Mit einer serologischen Pipette, gleichmäßig verteilen Sie Zellen aus jedem Brunnen EDTA behandelt durch Pipettieren 0,5 mL in jede Vertiefung der ECM-beschichteten 6-Well-Platte.
Hinweis: Zellen aus den umprogrammierten Brunnen nicht kombinieren (z.B. umprogrammiert gut 1 sollten gleichmäßig verteilt werden, in den ersten 6-Well-Platte und umprogrammierten gut 2 sollten gleichmäßig verteilt werden, in der zweiten 6-Well-Platte). - Die vergoldeten Zellen in den Low-O2 Inkubator zurück. Um Zellen gleichmäßig zu verteilen, schütteln Sie jede Platte hin- und Herbewegung und von Seite zu Seite. Nicht schwenken. Ersetzen Sie täglich das PSC-Medium.
8. Entnahme iPSC Kolonien
- 15 mL PSC Mittel-bis 37 ° c Vorwärmen Bestreichen Sie alle Brunnen von einem einzigen 6-Well-Platte mit hESC-qualifizierte ECM nach den Anweisungen des Herstellers. Versiegeln Sie die Platten mit Paraffin Film zu und inkubieren sie für 1 h bei RT
- Aspirieren Sie das Kulturmedium aus Brunnen iPSCs sammeln verwendet wird. Spülen Sie einmal mit 1 mL EDTA und Absaugen. Fügen Sie 1 mL EDTA pro Bohrloch. 4 min bei 37° c inkubieren Während die Zellen Inkubation sind, Aspirieren der ECM-Lösung von den vorgewärmten Tellern und mit 2 mL PSC Medium pro Bohrloch zu ersetzen. Die Teller beiseite stellen.
- Entfernen Sie vorsichtig die Platte mit iPSCs abgeholt aus dem Inkubator und legen Sie sie in die Biosicherheit Kabinett.
Hinweis: an dieser Stelle können Zellen sehr locker eingehalten und leicht verdrängt werden. - Aspirieren Sie sorgfältig EDTA. Fügen Sie sehr sanft 3 mL vorgewärmten PSC Medium, kümmert sich nicht um iPSC Kolonien zu vertreiben.
- Bewegen Sie die Platte an einen umgekehrten oder sezierenden Bereich besser zu visualisieren Kolonien. Bereiten Sie eine 1-mL-Pipette mit einer sterilen Spitze. Drücken Sie den Kolben vollständig, dann verwenden Sie die Pipettenspitze vorsichtig kratzen eine Kolonie während des Zeichnens langsam Flüssigkeit in die Spitze, die Kolonie zu sammeln. Zeichnen Sie als wenig Mittel wie möglich beim Pflücken der iPSC-Kolonie.
- Um die Kolonie zu übertragen, pipette 3 – 4 Mal rauf und runter in einem einzigen Brunnen der ECM-beschichtete Platte aus Schritt 8.2. Wiederholen Sie bis 6 Kolonien abgeholt und in einzelnen Wells überführt wurden. Wählen Sie no more als 2 Kolonien aus jedem einzelnen Brunnen, wenn eine optionale Passagierung in Schritt 7 beschrieben durchgeführt wurde.
- Verwenden Sie ein standard humanen Stammzellen-Protokoll, um unten alle verbleibenden Brunnen einfrieren. Auftauen lassen und neu Platte gefrorenen Vorräte, wenn weitere Kolonien später abgeholt werden müssen.
(9) Charakterisierung der iPSCs
- Durchführen Sie TRA-1-60 Färbung für die Analyse der Effizienz (Tag 18) Neuprogrammierung.
Hinweis: 2 von 3 umprogrammiert Brunnen sind für die zukünftige Kolonie Kommissionierung passagiert. Die restlichen gut einsetzbar für TRA-1-60 zu beflecken. Dieses Verfahren kann auf die Labor-Benchtop durchgeführt werden.- Waschen Sie die umprogrammierten gut mit 1 mL PBS. Zellen mit eiskalten Methanol bei-20 ° C für 5 min zu beheben.
- Aspirieren Sie Methanol. Trocken gut für ca. 2 min. Seien Sie vorsichtig, nicht übermäßig trocken. Die Zellen sind genug, wenn sie eine transluzente/Matte Farbe werden getrocknet.
- Waschen Sie die gut 3 Mal mit 1 mL PBS für 5 min mit sanft schütteln. Während der Waschgänge bereiten Sie 3 mL 3 % Wasserstoffperoxid in PBS.
- Aspirieren Sie die PBS. 2 mL verdünnte Wasserstoffperoxid-Lösung zugeben. 15 min bei RT mit sanft schütteln inkubieren. 4 mL der blockierenden Lösung vorbereiten: 10 % Bovine Serum Albumin (BSA) in PBS.
- Aspirieren der Peroxid-Lösung. Waschen Sie nun 2 Mal mit 1 mL PBS für 5 Minuten.
- Aspirieren Sie PBS und 2 mL Lösung in den Brunnen zu blockieren. 1 h bei RT inkubieren
- Waschen Sie die gut 3 Mal mit 1 mL PBS für 5 min mit sanft schütteln.
- Verdünnen Sie Primärantikörper Anti-TRA-1-60 in 1: 100 in der blockierenden Lösung ergänzt mit 0,05 % Natriumazid. 1 mL der Antikörper Verdünnung für 1 auch ein 6-Well-Format Gericht vorzubereiten. Der Brunnen der Antikörper-Verdünnung hinzu und wickeln Sie die Platte mit Parafilm um Verdunstung zu vermeiden. Inkubation über Nacht bei 4 ° C mit sanft schütteln.
Hinweis: Bei Bedarf kann die Inkubation mit dem primären Antikörper für 1 h bei RT mit sanft schütteln erfolgen. Die Primärantikörper Verdünnung kann bis zu 5 mal wiederverwendet werden. - Waschen Sie nach der Inkubation mit dem primären Antikörper auch 3 Mal mit 1 mL PBS für 5 min mit sanft schütteln.
- Verdünnen Sie die Anti-Maus HRP-konjugierten Sekundärantikörper bei 1: 200 in der blockierenden Lösung. Inkubieren Sie den Brunnen mit der Sekundärantikörper Verdünnung für 2 h bei RT mit sanft schütteln.
- Aspirieren der Sekundärantikörper Verdünnung und waschen Sie auch 3 Mal mit 1 mL PBS für 5 min mit sanft schütteln.
- Bereiten Sie beim dritten waschen die Substratlösung nach Anweisungen des Herstellers. Aspirieren Sie nach der letzten Wäsche PBS. Fügen Sie 1 mL der Substratlösung hinzu und inkubieren Sie bis die gewünschte Farbe (ca. 10 min) entwickelt.
- Aspirieren Sie die Substratlösung. Spülen Sie den Brunnen mit Wasser für 5 min unter sanft schütteln.
- Zählen der Kolonien, falls gewünscht. Reprogrammierung Effizienz = (Anzahl der Kolonien) / (Anzahl der vergoldeten Fibroblasten) x 100 %.
- Aspirieren Sie für die langfristige Lagerung Wasser aus den bunten Teller und an der Luft trocknen über Nacht bei RT Seal der getrocknete Platte mit Parafilm und Lagerung bei 4 ° C für bis zu 2 Jahre später Neuprogrammierung Effizienz bewerten.
Ergebnisse
Normalerweise dauert es ca. 5 – 6 Wochen nach der Einleitung des Fibroblasten Umprogrammierung zu Einfrieren von der ersten Fläschchen iPSCs (Abbildung 1). Die Neuprogrammierung Protokoll kann in der Regel in zwei Phasen eingeteilt werden. Phase 1 umfasst die Fibroblasten-Kultur und sieben Transfektionen mit der Neuprogrammierung RNA cocktail durchgeführt alle 48 h. Phase 2 beinhaltet, Isolierung, Ausbau und Charakterisierung der iPSC Kolonien.
Vor der Einleitung des Protokolls, sollte sichergestellt werden, dass die Fibroblasten umprogrammiert werden von guter Qualität sind. Gesunde Fibroblasten sollte erscheinen, spindelförmig, bipolar, und strahlenbrechende mit einer Verdoppelung Zeit von ca. 24 h. Am Tag 0 250.000 Zellen überzogen in ein 10 cm Teller am Tag-2 sollte bis zu 40 – 60 % Konfluenz (Abbildung 1, Tag 0) und Ertrag ca. 6 – 10 x 105 Zellen. In einem langsameren Tempo proliferierenden Zellen können durch Beschichtung mit einer höheren Dichte an Tag-2 und am Tag 0 Umprogrammierung kompensiert werden.
Fibroblasten erscheint sehr spärlich folgenden Beschichtung in einen Brunnen ein 6-Well-Format Gericht für Neuprogrammierung (Abbildung 2, Tag 1). Vierundzwanzig Stunden nach der ersten Transfection Fibroblasten verlieren ihre Spindel Form und übernehmen eine rundere Morphologie (Abbildung 2, Tag 2), die durch den Rest der Neuprogrammierung beibehalten wird. Grüne Fluoreszenz von mWasabi mRNA sollte minimal zu beobachten am 2. Tag und stetig Anstieg der Helligkeit von Tag 4 klar ersichtlich sein. MWasabi Fluoreszenz erkennen kann abhängig von Umfang Empfindlichkeit und Setup. Zelldichte erhöht schrittweise und konsequent in den ersten drei Transfektionen (Tage 1-6), mit einer scheinbaren platzen in Verbreitung zwischen Tag 7 und 8. Die Zellen sollten tagsüber 10 (Abbildung 2, 10. Tag) größtenteils konfluierende erscheinen. Die ersten iPSC-Kolonien kommen früh Tag 11 (Abbildung 2, Tag 11); jedoch möglicherweise Kolonien bis spät 18. Tag nicht beobachtbar. In der Regel bis zum Tage 15 – 18, werden groß und offensichtlich iPSC Kolonien, die deutlich von den umliegenden, unvollständig umprogrammierten Fibroblasten (Tag 15,Abbildung 2und Abbildung 3, 17. Tag). Immunostaining für die Pluripotenz Marker TRA-1-60 kann durchgeführt werden, um die Beurteilung der Neuprogrammierung Effizienz (Abbildung 3, Tag 17, TRA-1-60). Nach unserer Erfahrung die meisten Fibroblasten Linien führen Hunderte von Kolonien pro auch umprogrammiert (Abbildung 3, inset B).
Suboptimale Beschichtung Dichte ist der häufigste Grund für die geringere Effizienz der Umprogrammierung in unserem Protokoll und ist häufig verbunden mit Fibroblasten, die Kranken, alternde und/oder High-Passage. Wenn Dichte Beschichtung zu niedrig ist, werden große azelluläre kahle Flecken am Ende der Neuprogrammierung (Abbildung 4), und iPSC Kolonien nicht bilden können (Abbildung 4). Reprogrammierte Zellen sollten bis zum 14. Tag sehr konfluierende sein (Vergleiche Abbildung 4A und 4 b in Abbildung 2, Tag 14). Ebenso, wenn Zellen viel zu dicht überzogen sind oder zu schnell vermehren, ist Umprogrammierung Effizienz drastisch reduziert.
Um Homogenität des Patienten abgeleitet iPSCs zu erhalten, ist es wichtig, die Zell-Linien aus einer einzigen Kolonie zu erweitern. Da Neuprogrammierung Effizienz in unserem Protokoll sehr hoch ist, kann benachbarten iPSC Kolonien in unmittelbarer Nähe zu bilden und in jeder weitere (Abbildung 3, 15 – 17 Tage) wachsen. Dies macht es manchmal schwierig, eine einzelne Kolonie für klonale Expansion mechanisch zu trennen. Wir haben festgestellt, dass es hilfreich zum ersten Durchgang einer umprogrammierten gut und verdünnen Sie es über einen größeren Bereich der Kultur. Eine guten Durchgang Verhältnis besteht aus eine umprogrammierten 6-Well-Format-Platte gut auf eine gesamte 6-Well-Platte gleichmäßig aufteilen.
Nach der Verdünnung Passage iPSCs wachsen als Kolonien und sind leicht zu unterscheiden von Fibroblasten (Abbildung 5, 18. Tag). Zunächst iPSC Kolonien können lose verpackt, und einzelne Zellen haben eine relativ große cytoplasmatische Fläche. Im Laufe von 4 – 7 Tage die iPSCs vermehren sich und bilden eine charakteristische dicht gedrängten Kolonie mit definierten Kanten. Einzelne Zellen innerhalb der Kolonie haben einen großen nuklearen Teil mit prominenten Nukleolen (Abbildung 5, Tag 22). Es sollte viele Kolonien, die in jede Vertiefung bilden, und nur diejenigen mit klassischen iPSC Morphologie sollte für Expansion abgeholt werden.
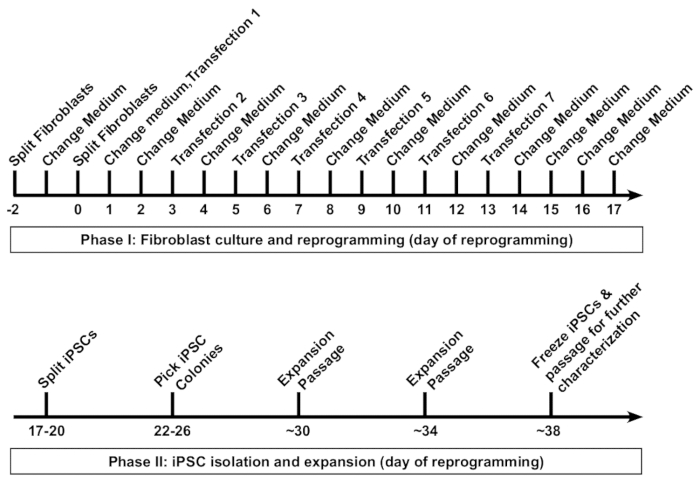
Abbildung 1 : Neuprogrammierung des menschlichen Fibroblasten in induzierte pluripotente Stammzellen (iPSCs). Eine schematische Protokoll für die Reprogrammierung von menschlichen Fibroblasten wird vorgestellt. Fibroblasten sind zunächst mit einer niedrigen Dichte in einen Brunnen ein 6-Well-Format-Gericht, gefolgt von sieben Transfektionen in 48 h Abständen durchgeführt passagiert. Medium ist nach jeder Transfektion ersetzt 16 – 20 Uhr. Umprogrammierten iPSCs sind zuerst am Tag ca. 18 und klonalen Kolonien passagiert werden tagsüber 26 gepflückt. In der Regel können Fibroblasten abgeleitet iPSC Linien für die Langzeitspeicherung von 38. Tag eingefroren werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
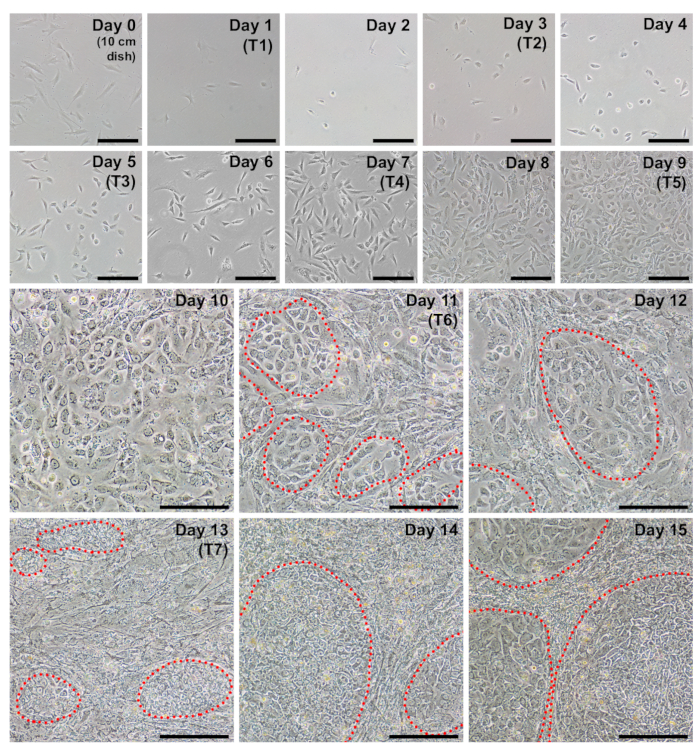
Abbildung 2 : Repräsentative täglich Bilder während jedes Tages der Neuprogrammierung. Fibroblasten sollten etwa 40 – 60 % Zusammenfluss am Übergang zur Reprogrammierung zu initiieren (Tag 0, Zellen sind in einer 10 cm Petrischale beschichtet). Die ersten Transfektion (T1) tritt am 1. Tag, und die Zellen zu diesem Zeitpunkt sehr spärlich erscheinen soll. Am nächsten Tag (Tag 2) sollte eine rundere Morphologie sichtbar. Zellen werden weiterhin Zunahme der Dichte im gesamten Protokoll mit iPSC beginnen zu früh Tag 11 (rot eingekreist) erscheinen. Tag 15 werden iPSC Kolonien groß mit dezenten Grenzen. Maßstabsleiste = 200 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
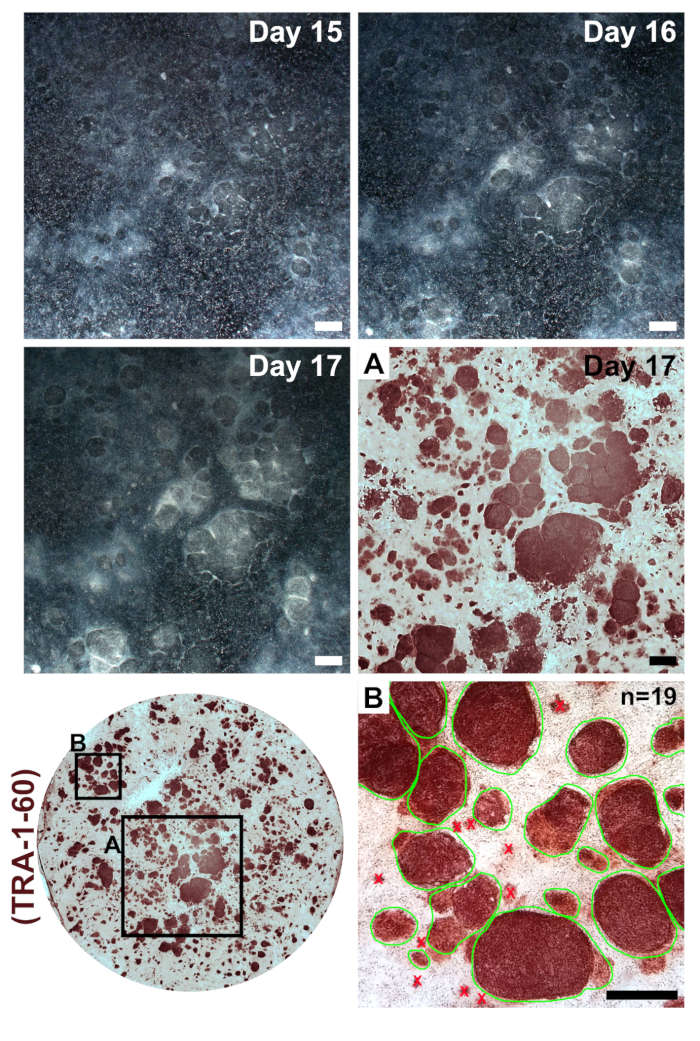
Abbildung 3 : Bildung der Kolonie nach Transfektionen mit Umprogrammierung geändert mRNAs und MiRNA imitiert. Low-Vergrößerung Bilder wurden eines Vertreters Neuprogrammierung an Tagen 15 – 17. Im Anschluss an die letzte Transfektion wird umprogrammiert iPSCs klarere Kolonien mit festgelegten Grenzen bilden, in der Größe zu erweitern und klar von unvollständig umprogrammierten umliegenden Fibroblasten zu kondensieren. Immunostaining für die Pluripotenz Marker TRA-1-60 zeigt die Anwesenheit von iPSCs (kleines Foto A) und kann für die Berechnung der Umprogrammierung Effizienz durch zählen alle Kolonien innerhalb einer einzigen Brunnen (kleines Foto B, Beispiele der zählbaren Kolonien in grün eingekreist) verwendet werden. Maßstabsleiste = 1 mm. Bitte klicken Sie hier für eine größere Version dieser Figur.
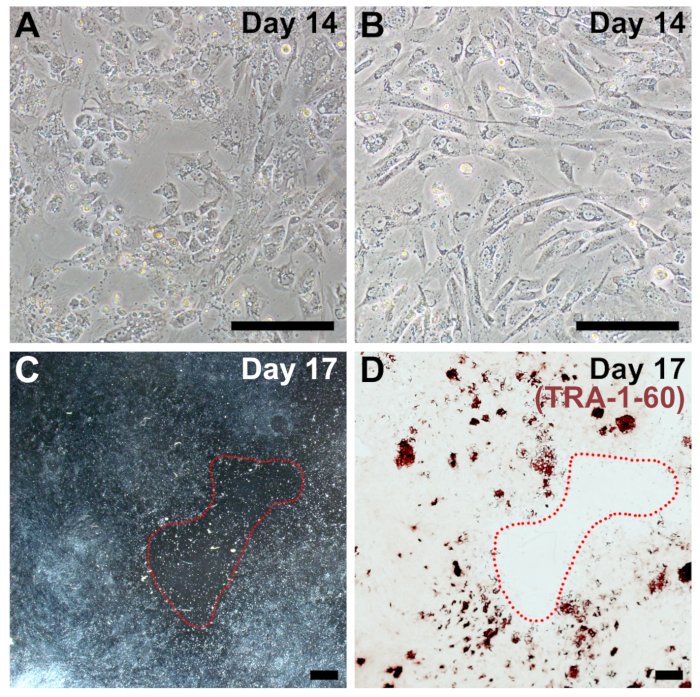
Abbildung 4 : Repräsentative Bilder von Sub-optimale Beschichtung Dichte Umprogrammierung. (A, B) Beispiele von Fibroblasten, die bis zum Tag 14 der Umprogrammierung zu spärlich sind (im Vergleich zu Abbildung 2, Tag 14). (C) geringer Vergrößerung Bild am Tag 17 der Neuprogrammierung mit einem großen, kahlen Flecken rot eingekreist. (D) den gleichen Brunnen wurde behoben und gebeizt für TRA-1-60 Insgesamte Armen bestätigen Umprogrammierung Effizienz aufgrund geringer Zelldichte. Skalieren von Balken = 200 µm (A, B) und 1 mm (C, D). Bitte klicken Sie hier für eine größere Version dieser Figur.
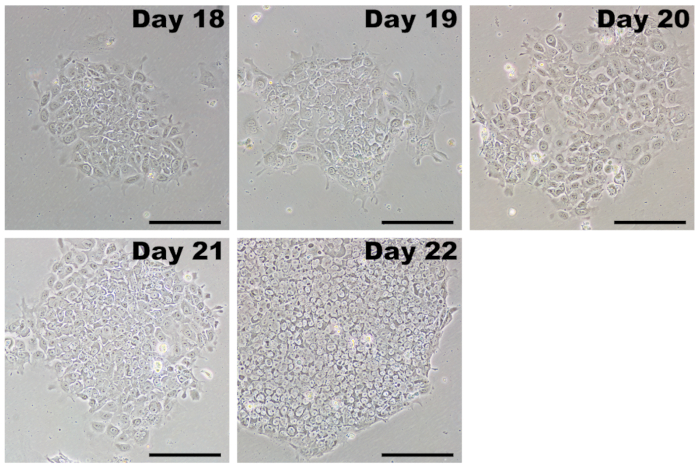
Abbildung 5 : Repräsentative Bilder von iPSCs nach dem ersten Durchgang. Nach Abschluss der Transfektionen mit Umprogrammierung modifizierte mRNAs und MiRNA Mimik, reprogrammierten Zellen sind von 17-20 Tage passagiert. iPSCs haben einen PSC Mittel- und schnell überholen alle Fibroblasten, die nicht vollständig neu programmiert wurden. Zunächst werden iPSCs Kolonien bilden, die locker mit schlecht definierten Grenzen auftreten können. Innerhalb weniger Tage die Zellen rasch vermehren und nehmen auf die charakteristische Morphologie der dicht gepackten Zellen mit einem hohen Anteil der Kern-Zytoplasma, dicht clustering in Kolonien mit deutlichen Grenzen. Maßstabsleiste = 200 µm. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Dieses Protokoll beschreibt eine klinisch relevante, nicht-Integration, RNA-basierten Methode, die für die Reprogrammierung von normalen und krankheitsassoziierten menschlichen Fibroblasten Linien in iPSCs auf einen extrem hohen Wirkungsgrad ermöglicht. Bis heute hat jeder menschlichen Fibroblasten-Linie, die wir versucht haben, mit dem beschriebenen Protokoll reprogram eine ausreichende Anzahl von qualitativ hochwertigen iPSCs für downstream-Anwendungen ergeben. Daraus resultierende iPSCs kann sofort übertragen und im Feeder-freie Kulturbedingungen erweitert werden.
Qualität der Fibroblasten für Re-Programmierung:
Reprogrammierung Erfolg hängt stark von der Qualität der Fibroblasten ab. Im Idealfall sollte mit der niedrigsten Stelle Fibroblasten zur Verfügung, um höchste Effizienz zu erreichen Umprogrammierung begonnen werden. Reprogrammierung Effizienz ist am besten mit Fibroblasten Passage 2 – 4. Umprogrammierung kann noch mit hoch-Passage (Durchgang 5 – 8), auch alternde Fibroblasten, allerdings mit einem reduzierten Wirkungsgrad arbeiten. Manchmal niedrig-Passage Fibroblasten sind nicht verfügbar oder Patientenproben haben eine genetische Mutation, die gesundes Wachstum verhindert. In diesem Fall möglicherweise Optimierung der ersten Beschichtung Dichte erforderlich. Die Umprogrammierung der kompromittierten Fibroblasten Linien ist in der Regel verbunden mit erhöhten Zelltod während RNA Transfektionen. Infolgedessen erscheint die Zellen in der Umprogrammierung auch von 10 – 14 Tagen der Umprogrammierung zu spärlich. Azelluläre Großflächen werden auch in der gut sichtbar sein. Wenn dies der Fall ist, müssen die Neuprogrammierung Protokoll neu initiierten mit eine höhere anfängliche Anfangszahl von Fibroblasten werden. Plattieren 3.000 Eingabezellen pro Bohrloch einer Schüssel 6-Well-Format arbeitet konsequent für die meisten Erwachsenen Fibroblasten-Linien. Jedoch kann die Beschichtung Zahl auf 5.000 – 10.000 (50.000 für alternde Linien) helfen um zu verbessern Reprogrammierung von krankheitsassoziierten Proben, wie in unserer vorherigen Veröffentlichung8berichtet wurde. Umgekehrt können auch Zellen erreichen Konfluenz zu früh Umprogrammierung sein. Wenn Zellen Transfektionen mit Umprogrammierung RNAs zu schnell vermehren (wie manchmal der Fall mit primären neonatale Fibroblasten), initiieren Neuprogrammierung mit 500 Fibroblasten pro Bohrloch eines 6-Well-Format Gericht8.
Handhabung des Puffers Transfektion:
Der pH-Wert des Puffers Transfektion (reduzierte Serum Medium angepasst, um den pH-Wert von 8,2) ist ausschlaggebend für die optimale Transfektion Wirkungsgrade, die hierfür erforderlichen Umprogrammierung Protokoll zu erreichen. Aus diesem Grund sind einige Vorsichtsmaßnahmen für den Umgang mit der Transfektion Puffer empfohlen. Wir haben festgestellt, dass auch kurze Exposition des Puffers Transfektion, atmosphärische Luft wirkt sich den pH-Wert des Puffers. Die Transfektion Puffer sollte regelmÄÑig in einen Schraubverschluss Behälter mit minimalen Luftraum daher (wir verwenden entweder 5 oder 15 mL Schraubverschluss konischen Rohren). Um weitere Luft minimieren, nutzen Sie jede Transfektion Puffer aliquoten für maximal zwei Transfektionen. Schließlich seit Temperatur Auswirkungen pH-Wert ist es wichtig, dass die Transfektion Puffer auf RT für die Montage der Transfektion komplexe equilibriert ist. Es wird empfohlen, nicht die Transfektion Puffer auf 37 ° c warm
Passagierung der iPSCs:
Während viele Protokolle empfehlen Kommissionierung individuelle iPSC Kolonien am Ende der Neuprogrammierung zuvor veröffentlicht, dies kann schwierig sein zu erreichen, wenn die Effizienz der Neuprogrammierung sehr hoch ist oder wenn die Kolonien zusammen, cluster, wie oft der Fall in unserer Protokoll. Daher, wenn iPSC Kolonien in unmittelbarer Nähe zueinander befinden, empfehlen wir, zuerst die Zellen umprogrammiert iPSCs verteilt, bevor manuell aufnehmen Kolonien Passagierung. Es gibt mehrere Vorteile bei diesem ersten Durchgang Schritt. Breitet sich die Kolonien gibt ihnen mehr Platz zum wachsen, mit viel größeren Kolonien für die Kommissionierung als sonst in den ursprünglichen Brunnen erreicht werden konnte. Dies verbessert die Erfolgsquote bei der Errichtung einer iPSC-Linie aus einem ausgewählten Klon. Wir finden auch, dass die zusätzliche Kulturzeit mit Fibroblasten, wenn auch mit einer verdünnten Verhältnis scheint die durchschnittliche Verbesserung der gepflückten iPSC Kolonien. Unvollständig umprogrammierten Fibroblasten vorsehen stützenden parakrine Faktoren, die nach wie vor die iPSCs zu etablieren und den direkten Übergang in den Feeder-freie Zelle Kulturbedingungen zu lindern zu helfen. Fibroblasten haben zufällig einen selektives Wachstum Nachteil im Vergleich zu iPSCs wenn in mTeSR1 kultiviert. Daher wird die kontaminierenden Fibroblasten-Zell-Population schnell zu vernachlässigbaren Mengen innerhalb von 3 – 4 Passagen verdünnt.
ROCK-Hemmer wie Y-27632 werden häufig für routinemäßige Kultur der menschlichen iPSCs eingesetzt. Wir haben festgestellt, dass häufige und/oder längere Kultur einiger iPSC-Zeilen mit Y-27632 kann schädliche Auswirkungen auf die Gesamtqualität. Wenn ein Büschel Passagierung Methode verwenden, ist z. B. mit EDTA, Y-27632 nicht notwendig, nach der Trennung iPSC Lebensfähigkeit zu erhalten. Wir haben ergänzende Medien mit Y-27632 für alle iPSC Isolierung, Erweiterung oder Routine Kultur vollständig beseitigt.
Protokoll-Einschränkungen:
Eine Einschränkung der beschriebenen RNA-basierten Neuprogrammierung Ansatz ist die anfänglichen Kosten und Komplexität im Zusammenhang mit der Ausarbeitung der Neuprogrammierung Reagenzien. Obwohl vorbereitenden Verfahren um mRNA zu generieren, Reagenzien sind alle Routine und wurden bisher (PCR, in-vitro- Transkription, DNase-Behandlung, Deckelung, Dephosphorylation, Reinigung) beschrieben, ist kumulativ die Produktion von mRNA Reagenzien ein relativ langwieriger und nicht-trivialen Prozess. Die andere große Herausforderung dieses Protokolls ist die Notwendigkeit, alle 48 h, Erhöhung der Arbeitsintensität des Protokolls Neuprogrammierung Zellen zu transfizieren. Diese Überlegungen möglicherweise unerschwinglich, wenn Reprogrammierung von nur wenigen Patientenproben gewünscht wird. Wenn der vorrangig zu berücksichtigen die Erzeugung von klinisch relevanten iPSCs oder Neuprogrammierung sehr hohen Wirkungsgrad zu erreichen ist, ist die beschriebene RNA-basierten Neuprogrammierung Vorgehensweise jedoch ideal.
Zusammenfassend lässt sich sagen hängt die beschriebene Hocheffizienz RNA-basierten Neuprogrammierung Methode Effizienz optimierte Transfektion der somatischen Zelle Art neu programmiert werden, wie in unserer vorherigen Veröffentlichung8beschrieben. Die RNA Transfektion Protokoll in dieser Studie vorgestellt ist hoch für menschliche primäre Fibroblasten abgestimmt aber möglicherweise auf andere Zelltypen zur Verbesserung der Neuprogrammierung Effizienz verschiedener somatischer Zellen zugeschnitten werden kann.
Offenlegungen
I.K und G.B sind Co-Erfinder zu einer Patentanmeldung mit dem Titel "Methoden und Kompositionen für die Umprogrammierung Zellen", PCT Antrag Nr. PCT/US2016/063258.
Danksagungen
Wir sind dankbar für finanzielle Unterstützung von den National Institutes of Health (T32AR007411-33) und der University of Colorado Haut Krankheiten Kern Forschungszentrum (P30AR057212). Wir danken auch der Epidermolysis Bullosa (EB) Research Partnership, EB Medical Research Foundation, Heilung EB Charity, dystrophischen Epidermolysis Bullosa Research Association (DEBRA) International, König-Baudouin-Stiftung Vlinderkindje Fonds und Tore Grenzen-Fonds.
Materialien
| Name | Company | Catalog Number | Comments |
| Plasmid templates for PCR | |||
| pcDNA3.3_KLF4 | Addgene | 26815 | |
| pcDNA3.3_SOX2 | Addgene | 26817 | |
| pcDNA3.3_c-MYC | Addgene | 26818 | |
| pcDNA3.3_LIN28A | Addgene | 26819 | |
| pCR-Blunt_hM3O | Addgene | 112638 | |
| pCR-Blunt_hNANOG | Addgene | 112639 | |
| pCR-Blunt_mWasabi | Addgene | 112640 | |
| Modified mRNA in vitro transcription and miRNA mimics | |||
| Forward Primer | Integrated DNA Technologies | TTGGACCCTCGTACAGAAGC TAATACG | |
| Reverse Primer (Ordered as ultramer, 4nmol scale) | Integrated DNA Technologies | TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT CTTCCTACTCAGGCTTTATTCA AAGACCA | |
| (ARCA Cap) 3´-0-Me-m7G(5')ppp(5')G | New England Biolabs | S1411S | |
| Pfu Ultra II Hotstart 2x Master Mix | Agilent | 600850-51 | |
| 5-Methylcytidine-5'-Triphosphate | Trilink Biotechnologies | N-1014 | |
| Antarctic Phosphatase | New England Biolabs | M0289L | |
| DNase I | NEB | M0303S | |
| MEGAscript T7 Transcription Kit | ThermoFisher Scientific | AM1334 | |
| Pseudouridine-5'-Triphosphate | Trilink Biotechnologies | N-1019 | |
| Riboguard RNase Inhibitor | Lucigen | RG90910K | |
| RNA Clean & Concentrator | ZymoResearch | R1019 | |
| Syn-hsa-miR-302a-3p miScript miRNA Mimic | Qiagen | MSY0000684 | |
| Syn-hsa-miR-302b-3p miScript miRNA Mimic | Qiagen | MSY0000715 | |
| Syn-hsa-miR-302c-3p miScript miRNA Mimic | Qiagen | MSY0000717 | |
| Syn-hsa-miR-302d-3p miScript miRNA Mimic | Qiagen | MSY0000718 | |
| Syn-hsa-miR-367-3p miScript miRNA Mimic | Qiagen | MSY0000719 | |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9937 | Use to dilute modified mRNAs and miRNA mimics |
| Fibroblast culture and reprogramming | |||
| 0.1% Gelatin in H2O | StemCell technologies | #07903 | |
| Stericup-GV Sterile Vacuum Filtration System | EMD Millipore | SCGVU05RE | Use to sterilize the transfection buffer and 0.5 mM EDTA in DPBS |
| 2-Mercaptoethanol | ThermoFisher Scientific | 21985023 | |
| 6-well plates (tissue culture treated) | Corning | 3516 | |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | |
| Fetal Bovine Serum | Fisher | 26-140-079 | |
| FGF Basic | ThermoFisher Scientific | phg0263 | |
| GlutaMax Supplement | ThermoFisher Scientific | 35050061 | Glutamine supplement used for the prepration of media |
| Heat Inactivated FBS | Gibco Technologies | 10438026 | |
| KnockOut Serum Replacement | ThermoFisher Scientific | 10828010 | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher Scientific | 13778500 | The protocol is optimized for the Lipofectamine RNAiMax transfection reagent |
| MEM | ThermoFisher Scientific | 11095080 | |
| MEM Non-essential amino acids | ThermoFisher Scientific | 11140050 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985070 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 500 mL |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985062 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 100 mL |
| 10 M NaOH | Sigma-Aldrich | 72068 | Make a 1 M solution by diluting in nuclease free water and use for pH adjustment |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9932 | Use to dilute NaOH and wash a pH meter |
| Pen/Strep/Fungizone | GE Healthcare | SV30079.01 | |
| rhLaminin-521 | ThermoFisher Scientific | A29248 | Supplied at a concentration of 100 µg/mL, use as a matrix for the reprogramming procedure |
| Dulbecco's phosphate-buffered saline (DPBS) | Life Technologies | 14190144 | |
| Trypsin-EDTA 0.25% Phenol Red | Life Technologies | 2520056 | |
| Vaccinia Virus B18R (CF) | ThermoFisher Scientific | 34-8185-86 | |
| iPSC culture | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| EDTA, 0.5 M stock solution | K&D Medical | RGF-3130 | Dilute to 0.5 mM in DPBS, filter sterilize and use for iPSC passaging |
| mTeSR1 | StemCell Technologies | 85850 | Pluripotent stem cell (PSC) medium, provides growth advantage to iPSCs over fibroblasts |
| Antibodies and Detection | |||
| Rabbit anti Mouse (HRP conjugated) | Abcam | ab97046 | |
| Tra-1-60 (mouse anti human) | Stemgent | 09-0010 | |
| Hydrogen Peroxide (30%) | LabChem | LC154301 | Dilute to 3% with PBS |
| Bovine Serum Albumin (BSA) | Fisher | BP9703100 | |
| Phosphate-buffered salin (PBS) | Hyclone | SH30258.02 | Supplied as 10x, dilute to 1x |
| VECTOR NovaRED Peroxidase Substrate Kit | Vector Laboratories | SK-4800 | |
| Special Equipment | |||
| Description | Notes | ||
| Biological safety cabinet | |||
| Regular humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 37 °C. | ||
| Tri-gas humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 5% O2, 37 °C. Use for the reprogramming procedure. | ||
| pH Meter | Must have resolution to two decimal places. Designate to RNA work if possible. | ||
| Inverted microscope | Microscope configured to visualize EGFP for monitoring transfection efficiency. |
Referenzen
- Takahashi, K., Yamanaka, S. Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors. Cell. 126 (4), 663-676 (2006).
- Takahashi, K., et al. Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors. Cell. 131 (5), 861-872 (2007).
- Schlaeger, T. M., et al. A comparison of non-integrating reprogramming methods. Nature Biotechnology. 33 (1), 58-63 (2015).
- Hacein-Bey-Abina, S., et al. LMO2-Associated Clonal T Cell Proliferation in Two Patients after Gene Therapy for SCID-X1. Science. 302 (5644), 415-419 (2003).
- Warren, L., et al. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7 (5), 618-630 (2010).
- Judson, R. L., Babiarz, J. E., Venere, M., Blelloch, R. Embryonic stem cell-specific microRNAs promote induced pluripotency. Nature Biotechnology. 27 (5), 459-461 (2009).
- Anokye-Danso, F., et al. Highly efficient miRNA-mediated reprogramming of mouse and human somatic cells to pluripotency. Cell Stem Cell. 8 (4), 376-388 (2011).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nature Communications. 9 (1), (2018).
- Hirai, H., et al. Radical acceleration of nuclear reprogramming by chromatin remodeling with the transactivation domain of MyoD. Stem Cells. 29 (9), 1349-1361 (2011).
- Mandal, P. K., Rossi, D. J. Reprogramming human fibroblasts to pluripotency using modified mRNA. Nature Protocols. 8 (3), 568-582 (2013).
- Avci-Adali, M., et al. In vitro synthesis of modified mRNA for induction of protein expression in human cells. Journal of Visualized Experiments. , e51943 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten