Method Article
rRNA 2'-O-甲基化的脱氧核糖核酸依赖性分析
摘要
在这里,我们提出了RNADNA依赖性裂解的协议。这使得RNA2'-O-甲基化能够进行快速和位点依赖性分析。这种方法可用于对打用RNA活性的初步或主要评估。
摘要
导箱C/D小核核核RNA(斯诺RNA)催化核糖体和小核RNA的甲基化2'-O-甲基化。然而,高真核生物中的大量打声RNA可能混杂地识别其他RNA物种和2'-O-甲基化多靶点。在这里,我们使用一种采用短DNA寡核苷酸(DNA)的成熟方法,对站点特异性2'-O-甲基化进行快速且非昂贵的分析,提供分步指南。这些DNA片段含有催化序列,在特定的共识位置将RNA裂断,以及将DNA定向到其RNA目标的可变同源性武器。脱氧核糖核酸活性被RNA中靠近裂解位点的核苷酸的2-O甲基化抑制。因此,DNA酶仅受被分块序列的共识限制,是快速分析介导的RNA 2'-O-甲基化的完美工具。我们分析了snoRNA snR13-和snR47引导2'-O-甲基化25S核糖体RNA在糖霉菌中,以证明该技术的简单性,并为脱氧核糖核酸酶依赖测定提供详细的方案。
引言
RNA修饰在基因表达的调节中起着重要的作用。RNA 2'-O-甲基化和伪尿化,分别由盒C/D和盒H/ACA小核核RNA(snoRNA)引导,保护RNA免受降解并稳定其高阶结构1,2,3.鼻RNA靶点主要在核糖体RNA(rRNA)和小型核RNA(snRNA)中确定。然而,在较高的真核生物中,可能有数百种没有分配函数的snoRNA,其中一些可能识别多个RNA1,4,5,6,7。因此,能够识别和分析打手酸的抗性修饰的方法是发现控制细胞过程的机制的重要工具。
盒 C/D snoRNA 引导的假定2'-O-甲基化位点可以通过许多技术进行生物信息识别,并通过许多技术进行实验确认,包括RNase H定向裂解,或特定位点和全基因组方法,采用逆转录在低核苷酸(dNTPs)浓度接近8,9,10,11。这些技术非常敏感,但也费力和昂贵,因此,可能不适合初始或快速测试。识别2'-O-甲基化位点的最简单和低成本的方法之一是DNA酶依赖性RNA裂解12。脱氧核糖核酸是短的、单链的和催化活性的DNA分子,能够在特定位置对RNA进行内源性裂解。它们由保存和催化活性核心序列和 5' 和 3' 结合臂组成,由通过 Watson-Crick 碱基配对与RNA靶点杂交的可变序列组成(图1)。因此,5' 和 3' 臂将催化序列输送到特定的 RNA 位点。DNA依赖性裂解被位于裂解部位12、13正上游的核苷酸的2'-O-甲基化所抑制。这使得DNAzymes非常实用的工具,用于分析假定或已知的RNA 2'-O甲基化位点。
两种类型的脱核糖核酸用于RNA修饰分析12。10-23 DNAzyme 的活性序列(图 1A) 由 15 个核苷酸 (5'RGGCCTACACGA3') 组成,这些核苷酸围绕靶向RNA purine-pyrimidine (RY) dinleotide 形成一个循环,并催化这两个核苷酸之间的裂解。RNA 紫红色 (R) 与脱氧核糖核酸不基配,在 DNA 酶上呈现的 2'-O 甲基化抑制裂解。10-23 DNAzyme 的结合臂通常为 10-15 核苷酸长。第二类脱核糖核酸(8-17 DNA酶(图1B)含有14核苷酸催化序列(5'TCCGAGCCGGACGA3')。核苷酸C2、C3和G4对与C9 G10和G11形成短干环结构。8-17 脱核糖核酸将RNA在无瑕疵胺活性序列中与第一胸腺素不完全配对的任何瓜氨酸上游分离。瓜宁上游的RNA核苷酸与脱氧核糖核酸不基配,其2'-O-甲基化会损害裂解。8-17 脱核糖核酸需要大约20个核苷酸的较长同源臂,才能将脱核糖核酸定向到其特定序列。
在这里,我们提供了一个分步协议,用于使用10-23和8-17 DNA酶依赖方法12,13(图1C)分析糖精菌中2'-O-甲基化。该协议可以很容易地适应其他生物体和RNA物种,并用于对位点特异性RNA 2'-O-甲基化进行快速、初步或主要分析。
研究方案
1. 菌株、媒体和缓冲食谱
- 准备酵母 (S. cerevisiae) 介质如下: YP (1% w/v 酵母提取物, 2% w/v 细菌肽), 葡萄糖和乳糖库存在 20% w/v.
- 准备醋酸钠(NaAc)-EDTA(AE)缓冲液,详见此处:50 mM NaAc pH 5.3 和 10 mM EDTA。
- 准备 10-23 DNAzyme 4x 孵育缓冲液,详见此处:24 mM Tris pH 8.0、60 mM NaCl 和 10-23 DNAzyme 4x 反应缓冲液:200 mM Tris pH 8.0 和 600 mM NaCl。
- 准备 8-17 DNAzyme 2x 反应缓冲液,如此处详细: 200 mM KCl, 800 mM NaCl, 100 mM HEPES pH 7.0, 15 mM MgCl2,和 15 mM MnCl2.
- 准备10x MOPS缓冲液,详见:200m MOPS,50 mM NaAc,1 mM EDTA;pH 7.0 和 1.5x 样品变性缓冲液:50% v/v 形式酰胺,20% v/v 甲醛,1.5x MOPS 缓冲液。
- 获得S.cerevisae菌株,BY4741(MATa他3_1 leu2_0满足15_0 ura3_0);GAL1::SNR13(如BY4741,但GAL1::SNR13:KANMX);GAL1::SNR47(如 BY4741,但GAL1::SNR47:HIS3mX)。任何其他酵母菌株可用于此分析。
2. 脱核系统设计
- 使用适当的数据库查找感兴趣的RNA序列或假定甲基化位点。对于S. cerevisae snoRNA 靶点,使用酵母打样RNA 数据库:http://people.biochem.umass.edu/fournierlab/snornadb/mastertable.php14
- 要查找感兴趣的甲基化位点(例如,snR13 依赖位点,请选择"snR13"并记下修饰的核苷酸的位置(例如,snR13 引导 A2281在 25S rRNA 中)。
- 使用适当的数据库查找修改后的核苷酸的上游和下游序列。对于S.cerevisae,使用糖霉菌基因组数据库:https://www.yeastgenome.org/
- 搜索目标基因名称,例如 RDN25(编码 25S rRNA)。
- 从"序列"选项卡中,在甲基化位点使用 10-23 DNA 酶测定和 20 个核苷酸上游(5' 臂)和下游(3'臂)甲基化位点时,选择甲基化位点上游(5'臂)和下游(3'臂)的10-15个核苷酸(3'臂),以进行8-17个脱氧核糖位。
- 创建 5' 和 3' 臂的补充序列。
- Flank 10-23 或 8-17 DNA催化序列,具有 5' 和 3' 臂的互补序列。
- 从供应商处订购脱氧核糖核酸作为正常DNA寡核苷酸。
3. S. 采尔维西亚增长条件
注:使用了S.cerevisae BY4741菌株衍生物,其中SNR13或SNR47snoRNA的表达从诱导的GAL1启动子驱动。为了诱导或抑制其合成,在含有加糖酶(GAL1依赖转录)或葡萄糖(GAL1依赖转录关闭)的培养基上生长细胞。作为对照,使用生长在糖酸或葡萄糖上的野生型菌株(BY4741)。
- 在适当的介质和条件下生长酵母菌株。分析GAL1::SNR13和GAL1::SNR47菌株以及同源性野生型菌株,在50mL的YP介质中生长细胞,在30°C至中指数相时,用2%葡萄糖(YPD)或角质糖(YPGal)生长细胞。
- 在1,000 x g下离心细胞,在4°C下3分钟。
- 丢弃上清液并保留颗粒。
- 在液氮中冷冻细胞颗粒并将其储存在-80°C。
警告:液氮可能导致严重的低温烧伤。务必穿防护服,并注意安全。
注:细胞颗粒可储存在-80°C至1个月。如果需要,可以在此处暂停该协议。
4. RNA分离15
注:使用最合适的方法分离RNA。对于酵母S.cerevisae,可以使用热酚RNA提取。
- 加入1 mL的冰冷水,重新悬浮颗粒,并将重新悬浮的细胞转移到1.5 mL微管中。
- 在 20,000 x g下在 4°C 下 10 s 离心,并去除上清液。
- 加入400μL的AE缓冲液,然后重新悬浮细胞。
注:除非另有说明,否则步骤 4.4-4.15 在室温下执行。 - 加入40μL的10%SDS和400μL的酸酚(pH 4.5)。
警告:苯酚是有毒的,应在烟罩下处理。使用苯酚时,务必佩戴实验室外套、防护手套和眼镜。按照制度规定处理废物。 - 通过涡旋20s混合良好。
- 在65°C孵育10分钟。每 2 分钟,轻轻打开和关闭管以释放压力,翻转管 2-3 次以混合相位。
- 将管转移到-80°C,孵育10分钟。
- 在室温下,将台和离心机的管子除霜在20,000 x g下,5分钟。
- 将上相转移到含有400μL酸酚:氯仿:二醇(25:24:1)的新管中。不要破坏相间。
警告:氯仿是有毒的,应在烟罩下处理。使用氯仿时,务必佩戴实验室外套、防护手套和眼镜。按照制度规定处理废物。 - 在室温下,在20,000 x g下涡旋30秒,将离心机混合10分钟。
- 将上相 (+400 μL) 转移到含有 400 μL 氯仿的新管中。
- 在室温下,在20,000 x g下涡旋30秒,将离心机混合5分钟。
- 将上相 (+300-350 μL) 转移到含有 1 mL EtOH 和 40 μL 7.5 M 醋酸铵 (NH4AC) 的新管中。通过翻转管子几次进行混合。
- 在-80°C孵育2小时或在-20°C下孵育2小时或过夜。
注:可以在此处暂停该过程。 - 在20,000 x g下离心10分钟,在4°C下。一个小的,白色的RNA颗粒将在管的底部变得可见。
- 通过移液去除 EtOH,以避免干扰颗粒。
- 在室温下,在20,000 x g下加入1 mL 70%EtOH和离心机5分钟。
- 通过移液去除 70% EtOH。
- 在 20,000 x g下离心 15 秒,用 2-20 μL 移液器去除剩余的 EtOH。
- 将管子放在长凳上打开 5 分钟,以干燥 RNA 颗粒。
注:RNA颗粒在干燥时将其颜色从白色变为透明。 - 在30μL的RNase/无DNase H2O中重新悬浮RNA颗粒,立即在冰上转移管,并在微分光光度计上测量RNA浓度。
- 将样品冷冻在-20°C。
注:RNA可储存在-20°C至1个月和-80°C至1年。该过程可以在此处暂停,也可以直接进行下一步。
5. 脱核酶消化
-
10-23 脱核酶消化
- 在1.5 mL管中,通过组合5μgRNA、200pmol的10-23 DNA酶(2μL的100mM库存溶液)和2.5μL的4x 10-23孵育缓冲液,在总体积为10μL中制备培养液。
- 将管转移到 95°C 的干燥热块中,孵育 3 分钟。
- 立即在冰上转移管子,孵育5分钟。
- 短暂地旋转,把管子放回冰上。
- 加入20 U的RNase抑制剂(例如,0.5 μL RiboLock RNase抑制剂)。
- 将管子放入25°C的干热块中,孵育10分钟。
- 同时,将5μL的4x 10-23反应缓冲液与4μL的300 mM MgCl2和1 μL H2O结合在1.5μL管中制备反应混合物。将管置于设置为37°C的干块中。
- 将孵育混合物转移到37°C的干热块中,并加入10μL的预预热反应混合物。
- 在37°C下孵育反应1小时。
- 转移冰上的管子,然后继续执行步骤 5.3.1。
-
8-17 脱核酶消化
- 在总体积为6μL时,制备一个1.5 mL微管,其中5μgRNA。将管放在冰上。
- 准备一个1.5 mL微管,400 pmol 8-17脱胶(100 mM库存中4μL)。把管子放在冰上。
- 将管转移到干燥热块组,温度为 95°C,孵育 2 分钟。
- 在冰上移动RNA样本。
- 用脱核酶将管子向下旋转 5 秒,并在 25°C 孵育 10 分钟。
- 同时,制备1.5 mL管,10μL2x 8-17反应缓冲液,在25°C孵育。
- 在用脱核酶将10μL的预加热2x反应缓冲液加入管中,制备反应混合物。
- 将14μL的反应混合物与RNA一起转移到管中,并加入20U的RN酶抑制剂。
- 在25°C下孵育反应2小时。
- 在冰上转移管,然后进行RNA纯化(步骤5.3.1)。
-
RNA纯化
- 将350μL水和400μL氯仿加入反应管,通过涡旋30秒混合,在室温下以20,000 x g离心5分钟。
- 将上相 (+300-350 μL) 转移到含有 1 mL EtOH、40 μL 7.5 M NH4交流和 1 μL 糖原 (10 μg/μL) 的新管中。通过翻转管子几次进行混合。
- 在-80°C孵育2小时或在-20°C下孵育2小时或过夜。
注:可以在此处暂停该过程。 - 重复从 4.15 到 4.21 的步骤。
- 将RNA颗粒重新悬浮在10μL的RNase/无DNaseH2O中,并立即在冰上转移管。
- 将样品冷冻在-20°C。
注:RNA可储存在-20°C至一个月和-80°C至1年。该过程可以在这里暂停或进行RNA电泳。
6. RNA电泳
- 用 1% SDS 喷洒电泳设备(罐、托盘、梳子),离开 15 分钟,用大量的 ddH2O 冲洗。
- 在微波炉中加热,将1.5克甘蔗在127.5 mL ddH2O中溶解。
- 在角胶溶液中加入15 mL的10x MOPS和7.5 mL的37%甲醛(总体积为150 mL)。
警告:甲醛是有毒的,应在烟罩下处理。使用甲醛时,务必佩戴实验室外套、防护手套和眼镜。按照制度规定处理废物。 - 在腺胶溶液中加入适量的凝胶染色(例如,15 μL 的 SYBR 安全 DNA 凝胶染色)。混合好,将蔗灰片倒入托盘。
- 立即将梳子插入凝胶中。
- 在烟罩下保留 45 分钟。使用感光凝胶染色时,用铝箔盖住托盘。
- 准备 600 mL 的 1x MOPS 缓冲液。
-
RNA样品制备
- 在1.5 mL管中,将10μL的消化和纯化RNA样品、5μL的样品变性缓冲液和0.5μL的6x加载染料混合。
警告:甲酰胺是有毒的,应在烟罩下处理。使用甲酰胺时,务必佩戴实验室外套、防护手套和眼镜。按照制度规定处理废物。 - 在70°C孵育RNA样品5分钟,在冰上转移样品。孵育5分钟。
- 在装入凝胶之前,先先向下旋转。
- 在1.5 mL管中,将10μL的消化和纯化RNA样品、5μL的样品变性缓冲液和0.5μL的6x加载染料混合。
- 将凝胶放入电泳罐中,并填充 1x MOPS 缓冲液。将每个样品(15 μL)的整个体积加载到凝胶上。以80V运行,直到溴酚蓝达到凝胶长度的2/3。
- 使用适当的成像器对凝胶进行成像,以检测所选凝胶染色(例如,紫外线透射器)。
结果
脱口授依赖性裂解在rRNA修饰分析中的效用最近已在snoRNA成熟13的上下文中得到证明。脱氧核糖核酸依赖测定用于表明,缺乏5'端前snoRNA处理影响2'-O甲基化水平25S和18SrRNA在S.cerevisiae13。
在这里,我们使用一个诱导的打用RNA转录系统来演示该技术的有效性和简单性。方框 C/D snR13 在 25S rRNA 中的两个位置引导甲基化,包括阿地宁 2281 (图 2A)。此核苷酸之后是尿素,它构成由 10-23 DNA 酶可夹紧的共识二核苷酸 (RY)。盒 C/D snR47 还指导 25S rRNA 中两个核苷酸的甲基化(图 2B)。位置 2220 中的腺苷跟跟一个瓜氨酸残留物,这种二核苷酸可以由 8-17 DNA 酶来裂解。为了诱导或抑制snR13或snR47 snoRNA的合成,我们插入了SNR13或SNR47基因上游的诱导GAL1启动子,并在含有加糖酶(GAL1-依赖性)的培养细胞中插入转录)或葡萄糖(GAL1-依赖转录关闭)。接下来,从GAL1::SNR13细胞分离的RNA用10-23 DNA酶孵育,设计在snR13依赖位点,在核苷酸2281和2282之间(图2C)之间分离25S rRNA。来自GAL1::SNR47菌株的RNA在核苷酸2220和2221之间用8-17 DNA酶靶向snR47依赖位点进行治疗(图2D)。作为对照,来自野生型BY4741菌株的RNA在半乳糖或葡萄糖上生长,用DNA酶孵育。脱发酶处理RNA的电泳表明,从GAL1::SNR13和GAL1::SNR47菌株中提取的25S rRNA保持原状(图3A,B;车道 3)。相反,从GAL1:SNR13和GAL1:SNR47细胞中分离的RNA被各自的DNA酶(图3A,B;通道4)消化。在这两种情况下,25S rRNA带减少,并观察到5'和3'的截断裂解产物(A和B)。这表明,在GAL1::SNR13和GAL1:SNR47菌株中,25S rRNA在snR13-或snR47引导位点被甲基化,当半乳糖用作碳源,这些鼻能酶被表达。当snR13或snR47表达在葡萄糖上关闭时,缺乏25S rRNA甲基化,允许DNA酶依赖性裂解。未观察到野生型样品的RNA消化(图3A,B;通道1和2),因为snR13和snR47的表达是这种菌株中无糖/葡萄糖无关的。因此,rRNA通常是甲基化的,因此对脱氧核糖核酸活性具有抗药性。
总体而言,我们的实验表明,10-23(图3A)和8-17(图3B)脱发酶的裂解活性与缺少盒C/D snR13或snR47相关,清楚地表明这些鼻状态RNA负责25S rRNA2'-O-甲基化在特定地点。
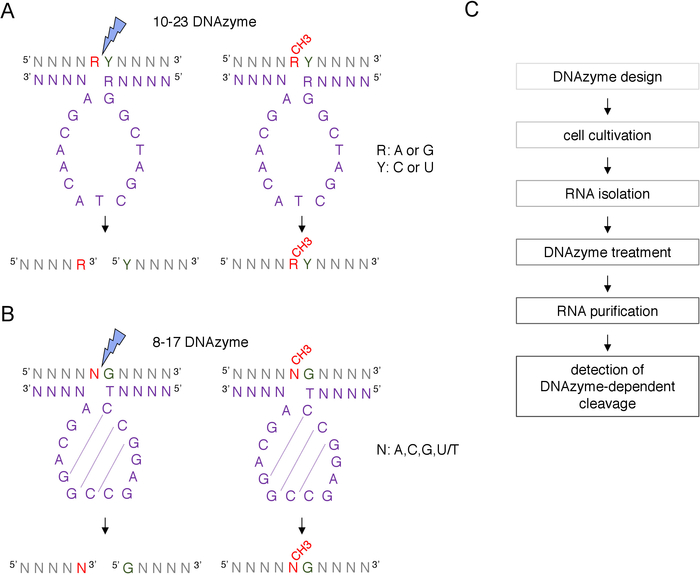
图1:脱核糖核酸及其RNA基质。(A) 10-23 DNA 酶切下一个紫皮球 (RY) RNA 二核苷酸。RNA中的R不与脱核糖核酸子群配对,而Y与脱核糖核酸酶中的R基具有补充性。RNA中紫皮(R)的甲基化抑制脱氧核糖核酸酶依赖性裂解。(B) 8-17 DNA 酶在瓜氨酸上游的RNA,与脱发DNA催化序列中的第一个胸腺素不完全配对。鸟宁前面的核苷酸未配对,其甲基化可防止脱氧核糖核酸依赖性裂解。RNA以灰色显示(除了甲基化位点),脱氧核糖核酸以紫色显示。N = 任何核苷酸,R = 紫菜:阿丁宁或瓜宁,Y = 丙酰胺:细胞氨酸或尿素;CH3- 表示RNA甲基化。DNAzyme 活动序列中的基线配对用虚线标记。蓝色闪电标记裂解部位。(C) 显示脱发酶相关分析步骤的流程图.请点击此处查看此图的较大版本。

图2:在25S rRNA中针对snR13-和snR47依赖甲基化位点进行DNA酶。(A, B)浏览器屏幕截图显示 snR13 依赖性 (A) 和 snR47 依赖性 (B) 甲基化位点在 25S rRNA (C) 25S rRNA 序列围绕 snR13 依赖甲基化位点 (A2281)和 10-23 DNAzyme (以紫色显示)设计用于在A2281和U2282之间将RNA分十。2281不与脱发糖配对,而 U2282与来自脱发糖活性序列的第一个核苷酸形成对(用蓝线标记)。蓝色闪电标记裂解。(D) 围绕 snR47 依赖性甲基化位点 (A2220)和 8-17 DNAzyme(以紫色显示)的 25S rRNA 序列,旨在将 RNA 在 A2220和 G2221之间进行分块。2220未与 DNAzyme 杂交,而 G2221与胸腺素不完全配对(用虚线表示)。蓝色闪电标记裂解部位。请点击此处查看此图的较大版本。

图3:使用10-23和8-17 DNA依赖性测定法分析25S rRNA的位点特异性2'-O-甲基化。(A) 使用 10-23 DNA 酶分析 snR13 依赖性 25S rRNA 甲基化。(B) 使用 8-17 DNA 酶分析 snR47 依赖性 25S rRNA 甲基化。RNA在变性的甘露胶中可视化染色。裂解产品 A 和 B 用红色箭头标记。WT = 野生型菌株;GAL = 乳糖,GLC = 葡萄糖。请点击此处查看此图的较大版本。
讨论
脱氧核糖核酸依赖性消化可用作分析位点特异性RNA 2'-O-甲基化12,13的简单而快速的方法。如果裂解部位上游的核苷酸未甲基化,DNA裂解RNA。与其他方法相反,包括RNase H定向消化、碱性降解或低核苷酸浓度逆转录,然后是定量PCR或测序8、10、11 ,16, DNAzyme 方法需要一个简单的 DNA 寡核苷酸和基本试剂,存在于任何分子生物学实验室.此外,脱毛核糖核酸可用于以类似方式分析由盒H/ACAsnoRNA12介导的RNA伪尿化,这使得它们成为研究斯诺RNA靶点的多功能工具。
脱口无音依赖性的方法仅受裂解部位共识序列17的限制。10-23 DNAzymes 仅用于分析 RY 二核苷酸位置 R 的 2'-O 甲基化,而 8-17 DNA 酶可识别位于瓜氨酸上游的核苷酸的修饰。因此,无法分析二核苷苷-二丁二烯(GA)、二丁胺-二丁胺(AA)、苯丙胺-二丁胺(YA)和苯丙胺-苯丙胺(YY)中第一核苷酸的2'-O甲基化等修饰。此外,还应考虑脱口核子依赖性裂解12的低效率。虽然一些脱发酶几乎完全地将RNA分十四(图3B),但许多脱发酶只部分消化其目标(图3B)。效率可能取决于裂解部位周围的顺序。例如,具有相同核苷酸延伸的RNA区域可能会影响脱核糖核酸活性序列的正确定位。此外,形成强二次结构的RNA区域可以重新杂交并抑制DNA酶结合到靶序列。为了克服这些问题,10-23 DNAzyme及其RNA基质的加热和冷却周期可以应用18。
我们使用脱氧核糖核酸方法研究rRNA的2'-O-甲基化。也可以使用这种技术来分析其他RNA修饰,如N6-甲基腺苷19。核糖体RNA,由于其丰度,可以通过电泳进行分析,裂解产物可以在紫外线下可视化。然而,这不适用于较丰富的RNA,如RNA聚合酶II生成的编码RNA(mRNA)和非编码RNA(ncRNA)。这些RNA通常不能通过在糖或聚丙烯酰胺凝胶中的RNA染色直接检测。在这种情况下,DNA依赖性裂解可以通过北方印迹可视化,通过PCR/定量PCR间接检测,或通过定量PCR分析,聚合酶(例如,KlenTaq DNA聚合酶),能够鉴别2+-O甲基化RNA无甲基化RNA20,21。
披露声明
作者没有什么可透露的。
致谢
我们感谢玛雅·威尔逊和阿妮卡·莱尼对手稿的批判性解读。这项工作得到了由韦康信托基金和皇家学会共同资助的亨利·戴尔爵士奖学金的支持(200473/Z/16/Z)。
材料
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Acid phenol | SIGMA | P4682 | |
| Agarose | VWR | A2114 | |
| Ammonium acetate | SIGMA | A1542 | |
| Chlorophorm | Fisher scientific | 10293850 | |
| DNase/RNase free water | Fischer Scientific | 10526945 | |
| DNAzyme | Integrated DNA Technology | Custom oligo DNA | |
| EDTA | SIGMA | E9884 | |
| Ethanol Absolute | Fisher scientific | 10437341 | |
| Formaldehyde | Sigma | F8775 | |
| Formamide | sigma | F9037 | |
| Galactose | SIGMA | G0750 | |
| Gel Loading Dye | Thermo Fisher Scientific | R0611 | |
| Glucose | SIGMA | G7021 | |
| Glycogen | Thermo Fisher Scientific | R0561 | |
| HEPES | SIGMA | H3375 | |
| Isoamyl | SIGMA | W205702 | |
| KCl | SIGMA | P9333 | |
| MgCl2 | SIGMA | M8266 | |
| MnCl2 | SIGMA | 244589 | |
| MOPS | SIGMA | M1254 | |
| NaCl | SIGMA | S7653 | |
| Oxoid Peptone Bacteriological | Thermo Fisher Scientific | LP0037 | |
| Oxoid Yeast Extract Powder | Thermo Fisher Scientific | LP0021 | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher Scientific | EO0382 | |
| SDS | SIGMA | 74255 | |
| Sodium acetate trihydrate | SIGMA | S8625 | |
| SYBR Safe DNA Gel Stain | Thermo Fisher Scientific | S33102 | |
| Tris base | SIGMA | TRIS-RO | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 1.5 mL microtubes | Sarstedt | ||
| 152VR5C01M -80°C freezer | Thermo Fisher Scientific | ||
| 250 mL Erlenmeyer flasks | Cole-Parmer | ||
| 50 mL conical tubes | Sarstedt | ||
| Combicup VX200 vortex | Appleton Woods | ||
| DS-11 microspectrophotometer | Denovix | ||
| Electrophoresis chamber (20 cm tray) | SIGMA | ||
| FiveEasy F20 pH meter | Appleton Woods | ||
| Gel documentation system | Syngene | ||
| Heraeus Fresco 21 micro centrifuge | Fisher Scientific | ||
| Megafuge 8R centrifuge with rotator suitable for 50 mL conical tubes | Fisher Scientific | ||
| Mini Fuge Plus mini centrifuge | Starlab | ||
| Mixer HC thermal block | Starlab | ||
| OLS26 Shaking Water Bath | Grant | ||
| PowerPac power supplier | BioRad |
参考文献
- Dieci, G., Preti, M., Montanini, B. Eukaryotic snoRNAs: a paradigm for gene expression flexibility. Genomics. 94 (2), 83-88 (2009).
- Watkins, N. J., Bohnsack, M. T. The box C/D and H/ACA snoRNPs: key players in the modification, processing and the dynamic folding of ribosomal RNA. Wiley Interdisciplinary Review RNA. 3 (3), 397-414 (2012).
- Kufel, J., Grzechnik, P. Small Nucleolar RNAs Tell a Different Tale. Trends in Genetics. , (2018).
- Li, T., Zhou, X., Wang, X., Zhu, D., Zhang, Y. Identification and characterization of human snoRNA core promoters. Genomics. 96 (1), 50-56 (2010).
- Jorjani, H., et al. An updated human snoRNAome. Nucleic Acids Research. 44 (11), 5068-5082 (2016).
- Hubbard, T. J., et al. Ensembl 2009. Nucleic Acids Research. 37 (Database issue), D690-D697 (2009).
- Makarova, J. A., Kramerov, D. A. SNOntology: Myriads of novel snoRNAs or just a mirage?. BMC Genomics. 12, 543 (2011).
- Yu, Y. T., Shu, M. D., Steitz, J. A. A new method for detecting sites of 2'-O-methylation in RNA molecules. RNA. 3 (3), 324-331 (1997).
- Decatur, W. A., Liang, X. H., Piekna-Przybylska, D., Fournier, M. J. Identifying effects of snoRNA-guided modifications on the synthesis and function of the yeast ribosome. Methods in Enzymology. 425, 283-316 (2007).
- Dong, Z. W., et al. RTL-P: a sensitive approach for detecting sites of 2'-O-methylation in RNA molecules. Nucleic Acids Research. 40 (20), e157 (2012).
- Birkedal, U., et al. Profiling of ribose methylations in RNA by high-throughput sequencing. Angewandte Chemie International Edition, England. 54 (2), 451-455 (2015).
- Buchhaupt, M., Peifer, C., Entian, K. D. Analysis of 2'-O-methylated nucleosides and pseudouridines in ribosomal RNAs using DNAzymes. Analytical Biochemistry. 361 (1), 102-108 (2007).
- Grzechnik, P., et al. Nuclear fate of yeast snoRNA is determined by co-transcriptional Rnt1 cleavage. Nature Communication. 9 (1), 1783 (2018).
- Piekna-Przybylska, D., Decatur, W. A., Fournier, M. J. New bioinformatic tools for analysis of nucleotide modifications in eukaryotic rRNA. RNA. 13 (3), 305-312 (2007).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18 (10), 3091-3092 (1990).
- Maden, B. E. Mapping 2'-O-methyl groups in ribosomal RNA. Methods. 25 (3), 374-382 (2001).
- Santoro, S. W., Joyce, G. F. A general purpose RNA-cleaving DNA enzyme. Proceedings of the National Academy of Science, U. S. A. 94 (9), 4262-4266 (1997).
- Hengesbach, M., Meusburger, M., Lyko, F., Helm, M. Use of DNAzymes for site-specific analysis of ribonucleotide modifications. RNA. 14 (1), 180-187 (2008).
- Sednev, M. V., et al. N(6) -Methyladenosine-Sensitive RNA-Cleaving Deoxyribozymes. Angewandte Chemie International Edition, England. 57 (6), 15117-15121 (2018).
- Aschenbrenner, J., Marx, A. Direct and site-specific quantification of RNA 2'-O-methylation by PCR with an engineered DNA polymerase. Nucleic Acids Research. 44 (8), 3495-3502 (2016).
- Lee, K. W., Bogenhagen, D. F. Assignment of 2'-O-methyltransferases to modification sites on the mammalian mitochondrial large subunit 16 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (36), 24936-24942 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。