Method Article
rRNA 2'-O-メチル化のDNAzyme依存性分析
要約
ここでは、RNAのDNAzyme依存性切断に関するプロトコルを提示する。これにより、RNA 2'-O-メチル化の迅速かつ部位依存性の分析が可能になります。このアプローチは、snoRNA活性の予備的または主要な評価に使用することができる。
要約
ガイドボックスC/D小型核RNA(snoRNA)は、リボソームおよび小核RNAの2'-O-メチル化を触媒する。しかしながら、高い真核生物における多数のsnoRNAは、他のRNA種および2'-O-メチル酸複数の標的を無差別に認識してもよい。ここでは、DNAzymesと呼ばれる短いDNAオリゴヌクレオチドを用いた確立された方法を用いたサイト特異的2'-O-メチル化の高速かつ非高価な分析のためのステップバイステップガイドを提供する。これらのDNA断片には、特定のコンセンサス位置でRNAを切断する触媒配列と、そのRNA標的にDNAzymeを導く可変相同性アームが含まれています。DNAzyme活性は、RNAにおける切断部位に隣接するヌクレオチドの2-O-メチル化によって阻害される。したがって、DNAzymesは、切断された配列のコンセンサスによってのみ制限され、snoRNA媒介RNA 2'-O-メチル化の迅速な分析のための完璧なツールです。サッカロマイセスセレビシエにおける25SリボソームRNAのsnR13誘導2'-O-メチル化を分析し、この技術の簡略性を実証し、DNAzyme依存性アッセイの詳細なプロトコルを提供した。
概要
RNA修飾は、遺伝子発現の調節において重要な役割を果たす。RNA 2'-O-メチル化および擬似性酸化は、それぞれボックスC/DおよびボックスH/ACA小型核RNA(snoRNA)によって導かれ、RNAを分解から保護し、より高次構造1、2、3を安定化させる.SnoRNA標的は、主にリボソームRNA(rRNA)および小型核RNA(snRNA)で同定されている。しかし、より高い真核生物では、割り当てられた機能を持たない何百ものsnoRNAが存在する可能性があり、そのうちのいくつかは複数のRNA1、4、5、6、7を認識する可能性があります。したがって、snoRNA誘導修飾の同定と分析を可能にする方法は、細胞プロセスを支配するメカニズムを明らかにする上で重要なツールである。
ボックスC/D snoRNA誘導性2'-O-メチル化部位は、RNase H指向切断、または逆転転写を採用する部位特異的およびゲノム全体の方法を含む多くの技術によって生物学的に生物学的に同定され、実験的に確認することができる。低ヌクレオチド(dNTP)濃度アプローチ8、9、10、11で。これらの技術は非常に敏感であるが、また、手間がかかり、高価であるため、初期または迅速なテストには適していない可能性があります。2'-O-メチル化部位を同定する最も簡単で低コストな方法の1つは、DNAzyme依存性RNA切断12である。DNAzymesは、特定の位置でRNAの内核溶解切断が可能な短い、一本鎖および触媒活性DNA分子です。それらは、保存され、触媒的に活性なコア配列と、ワトソン・クリックベース対化によってRNA標的にハイブリダイズするように設計された可変配列からなる5'および3'結合アームで構成される(図1)。したがって、5'および3'腕は、特定のRNA部位に触媒配列を提供する。DNAzyme依存性切断は、切断部位12、13の直接上流に位置するヌクレオチドの2'-O-メチル化によって阻害される。これにより、DNAzymesは、置換または既知のRNA 2'-O-メチル化部位の分析のための非常に実用的なツールになります。
RNA修飾解析には2種類のDNAzymesが使用されています12.10-23 DNAzyme(図1A)の活性配列は、標的RNAプリンピリミジン(RY)ジヌクレオチドの周りにループを形成し、これら2つのヌクレオチド間の切断を触媒する15ヌクレオチド(5'RGGCTAGCTACAACGA3')から構成される。RNAプリン(R)はDNAzymeと塩基対化されず、DNAzyme上の2'-O-メチル化は切断を阻害する。10-23 DNAzymesの結合アームは、通常、長い10〜15ヌクレオチドである。第2のDNAzymeクラス、8-17 DNAzymes(図1B)は、14ヌクレオチド触媒配列(5'TCCGAGCCGGACGA3')を含む。ヌクレオチドC2、C3およびG4対C9G10およびG11を伴い、短いステムループ構造を形成する。 8-17 DNAzymesは、DNAzyme活性配列からの最初のチミンと不完全に組み合わされた任意のグアニンの上流のRNAを切り開く。グアニンの上流のRNAヌクレオチドはDNAzymeと塩基対化されず、その2'-O-メチル化は切断を損なう。8-17 DNAzymesは、その特定の配列にDNAzymeを導くために約20ヌクレオチドのより長い相同性腕を必要とする。
ここでは、10-23および8-17 DNAzyme依存アプローチ12、13(図1C)を用いてサッカロマイセスセレビシエにおけるrRNAの2'-O-メチル化の分析のためのステップバイステッププロトコルを提供する。このプロトコルは、他の生物およびRNA種に容易に適合し、部位特異的RNA 2'-O-メチル化の高速、予備または主要な分析のために使用することができる。
プロトコル
1. 株、メディア、およびバッファレシピ
- ここで詳しく説明されているように酵母(S.セレビシエ)メディアを調用する:YP(1%w/v酵母エキス、2%w/v細菌性ペプトン)、およびグルコースおよびガラクトースストックは20%w/vである。
- ここで詳しく説明されているように酢酸ナトリウム(NaAc)-EDTA(AE)バッファーを調製する:50 mM NaAc pH 5.3および10mM EDTA。
- ここで詳しく説明されているように10-23 DNAzyme 4xインキュベーションバッファを準備する:24 mMトリスpH 8.0、60 mM NaClおよび10-23 DNAzyme 4x反応バッファー:200 mMトリスpH 8.0および600 mM NaCl。
- ここで詳しく説明されているように8-17 DNAzyme 2x反応バッファーを準備する:200 mM KCl、800 mM NaCl、100 mM HEPES pH 7.0、15 mM MgCl2、および15 mM MnCl2.
- ここで詳しく説明されているように10x MOPSバッファを準備する:200 mM MOPS、50 mM NaAc、1 mM EDTA;pH 7.0および1.5xサンプル脱電バッファー:50%v/vホルムアミド、20%v/vホルムアルデヒド、1.5x MOPSバッファー。
- S. セレビシエ株を得る, BY4741(MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0);GAL1::SNR13(BY4741としてしかしGAL1::SNR13:KANmX)GAL1::SNR47(BY4741として、GAL1::SNR47:HIS3mX)。他の酵母株は、この分析に使用することができます。
2. DNAzymeデザイン
- 適切なデータベースを使用して、関心のあるRNA配列または推定メチル化部位を見つけます。S.セレビシエ・スノールナ標的の場合、酵母snoRNAデータベースを使用する:http://people.biochem.umass.edu/fournierlab/snornadb/mastertable.php 14
- 目的のメチル化部位を見つけるには、例えば、snR13依存部位を選択し、「snR13」を選択し、修飾されたヌクレオチドの位置をメモする(例えば、snR13誘導A2281を25S rRNAで)。
- 適切なデータベースを使用して、改変されたヌクレオチドの上流および下流の配列を検索します。S.セレビシエの場合は、サッカロマイセスゲノムデータベースを使用https://www.yeastgenome.org/
- 標的遺伝子名例、RDN25(コード25S rRNA)を検索する。
- 「シーケンス」タブから、10-23 DNAzymeアッセイを使用する場合は、メチル化部位の上流10-15ヌクレオチド(5'腕)と下流(3'アーム)を選択し、8-17Nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnynnnnnnnnnnnnnnynnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnsの10-23 DNAzymeアッセイ
- 5'と3'腕の補完的なシーケンスを作成します。
- フランク10-23または8-17 DNAzyme触媒配列は、5'および3'腕の相補配列を持つ。
- サプライヤーから通常のDNAオリゴヌクレオチドとしてDNAzymeを注文します。
3. S. セレビシエ成長条件
注:S.セレビシエBY4741株誘導体が使用され、SNR13またはSNR47 snoRNAのいずれかの発現が誘導性GAL1プロモーターから駆動される。それらの合成を誘導または阻害するために、ガラクトース(GAL1-依存転写オン)またはグルコース(GAL1-依存転写オフ)を含む培地上で細胞を増殖させる。対照として、ガラクトースまたはブドウ糖のいずれかで成長した野生型株(BY4741)を使用する。
- 適切な培地および条件で酵母株を成長させる。GAL1::SNR13およびGAL1:SNR47株および同位種野生型株を分析するには、2%グルコース(YPD)またはガラクトース(YPGal)のいずれかでYP培地の50 mLで細胞を成長させ、30°Cから中間指数相に。
- 遠心分離球は1,000 x gで、4°Cで3分間。
- 上清を捨て、ペレットを保管してください。
- 液体窒素中の細胞ペレットを凍結し、-80°Cで保存します。
注意:液体窒素は重度の低温熱傷を引き起こすことがある。常に防護服を着用し、安全対策を講じてください。
注:細胞ペレットは-80°Cで1ヶ月まで保存することができます。プロトコルは、必要に応じてここで一時停止できます。
4. RNA分離15
注:RNA を分離するには、最も適切な方法を使用します。酵母S.セレビシエの場合、ホットフェノールRNA抽出が使用できます。
- 1 mLの氷冷水を加え、ペレットを再懸濁し、再懸濁細胞を1.5mLマイクロチューブに移す。
- 4°Cで10sの20,000 x gで遠心分離機を取り除き、上清を取り除きます。
- AE バッファーの 400 μL を追加し、セルを再中断します。
注:手順4.4-4.15は、特に明記されていない限り、室温で行われます。 - 10% SDS の 40 μL と 400 μL の酸性フェノール (pH 4.5) を追加します。
注意:フェノールは有毒であり、ヒュームフードの下で処理する必要があります。フェノールを使用する場合は、必ずラボコート、保護手袋、眼鏡を着用してください。制度上の規制に従って廃棄物を処分する。 - 20sの渦でよく混ぜます。
- 65°Cで10分間インキュベートします。2分ごとに、穏やかにチューブを開閉して圧力を解放し、フェーズを混合するためにチューブを2〜3回反転させます。
- チューブを-80 °Cに移し、10分間インキュベートします。
- ベンチの管を解凍し、室温で5分間、20,000 x gで遠心分離機を解凍します。
- 上相を400μL酸フェノールを含む新しいチューブに移す:クロロホルム:イソアミルアルコール(25:24:1)。フェーズ間を中断しないでください。
注意:クロロホルムは有毒であり、ヒュームフードの下で処理する必要があります。クロロホルムを使用する場合は、必ずラボコート、保護手袋、眼鏡を着用してください。制度上の規制に従って廃棄物を処分する。 - 室温で10分間20,000 x gで30sと遠心分離機の渦でよく混ぜます。
- 上相(約400μL)を400μLクロロホルムを含む新しいチューブに移します。
- 室温で5分間20,000 x gで30sと遠心分離機の渦でよく混ぜます。
- 上相(~300~350 μL)をEtOHの1mLと7.5M酢酸アンモニウム(NH4 AC)の40μLを含む新しいチューブに移します。チューブを数回反転して混ぜます。
- -80 °Cで-20°Cで2時間または一晩-20°Cでインキュベートします。
注:手順はここで一時停止できます。 - 20,000 x gで遠心分離機、4 °Cで10分間。小さい、白いRNAペレットは管の底に見えるようになる。
- ペレットの邪魔にならないように、ピペットでEtOHを取り外します。
- 室温で5分間20,000 x gで70%EtOHと遠心分離機の1 mLを追加します。
- ピペッティングで70%EtOHを取り外します。
- 15秒で20,000 x gで遠心分離し、残りのEtOHを2-20 μLピペットで取り除きます。
- チューブをベンチで5分間開けて乾燥させて、RNAペレットを乾燥させます。
注:RNAペレットは、乾燥時に白から透明に色を変更します。 - RNase/DNaseフリーH2Oの30μLでRNAペレットを再ステーペンし、すぐに氷上にチューブを移し、マイクロ分光光度計でRNA濃度を測定します。
- サンプルを-20 °Cで凍結します。
注:RNAは-20°Cで1ヶ月まで、-80°Cで1年まで保存できます。手順はここで一時停止するか、次の手順に直接進むことができます。
5. DNAzyme 消化
-
10-23 DNAzyme 消化
- 1.5 mLチューブでは、5 μgのRNA、10-23 DNAzymeの200 pmol(100mMストック溶液の2μL)、4x 10-23インキュベーションバッファーの2.5 μLを合計体積10μLの水量で組み合わせてインキュベーションミックスを調製します。
- チューブを95°Cに設定された乾燥したヒートブロックに移し、3分間インキュベートします。
- チューブを氷の上に直ちに移し、5分間インキュベートします。
- 短時間スピンダウンし、チューブを氷の上に戻します。
- RNase阻害剤の20Uを添加する(例えば、0.5 μLリボロックRNase阻害剤)。
- チューブを25°Cに設定した乾燥したヒートブロックに入れ、10分間インキュベートします。
- その間、4x 10-23反応バッファーの5 μLを300mM MgCl2および1 μL H2Oの4μLと組み合わせて1.5μLチューブ内の反応混合物を調製し、チューブを37°Cに設定した乾燥ブロックに入れます。
- インキュベーションミックスを37°Cに設定した乾燥ヒートブロックに移し、予め温めた反応ミックスの10 μLを加えます。
- 反応を37°Cで1時間インキュベートする。
- 氷の上にチューブを移し、ステップ5.3.1に進みます。
-
8-17 DNAzyme 消化
- 総体積6μLのRNAを5μgで1.5 mLのマイクロチューブを準備します。
- 8-17 DNAzyme(100 mMストックのうち4μL)の400 pmolで1.5 mLマイクロチューブを準備します。チューブを氷の上に置いてください。
- チューブを95°Cに設定した乾燥したヒートブロックに移し、2分間インキュベートします。
- RNA サンプルを氷の上に移動します。
- チューブをDNAzymeで5sでスピンダウンし、25°Cで10分間インキュベートします。
- 同時に、2x 8-17反応バッファーの10 μLの1.5 mLチューブを調製し、25°Cでインキュベートします。
- DNAzymeを用いてチューブに予め温められた2x反応バッファーの10 μLを加えて反応混合物を調製する。
- RNAを使用してチューブに反応ミックスの14μLを転送し、RNase阻害剤の20Uを追加します。
- 反応を25°Cで2時間インキュベートする。
- チューブを氷の上に移し、RNA精製に進みます(ステップ5.3.1)。
-
RNA精製
- 反応管に350μLの水と400μLのクロロホルムを加え、室温で5分間20,000 x gで30sと遠心分離機を渦でよく混ぜます。
- 上相(~300~350 μL)をEtOHの1mL、7.5M NH4ACの40 μL、グリコーゲンの1μL(10 μg/μL)を含む新しいチューブに移します。チューブを数回反転して混ぜます。
- -80 °Cで-20°Cで2時間または一晩-20°Cでインキュベートします。
注:手順はここで一時停止できます。 - 4.15 から 4.21 までの手順を繰り返します。
- RnaペレットをRNase/DNaseフリーH2Oの10μLで再ステーペンし、すぐに氷の上にチューブを移します。
- サンプルを-20 °Cで凍結します。
注:RNAは-20°Cで1ヶ月まで、-80°Cで1年まで保存できます。手順はここで一時停止するか、RNA電気泳動に進むことができます。
6. RNA電気泳動
- 1%SDSで電気泳動装置(タンク、トレイ、櫛)をスプレーし、15分間放置し、ddH2Oをたっぷり使ってすすいで下さい。
- 電子レンジで加熱してddH2 Oの127.5mLにアガロース1.5gを溶かします。
- アガロース溶液に10x MOPSの15 mLと37%ホルムアルデヒドの7.5 mLを加えます(総容積は150mL)。
注意:ホルムアルデヒドは有毒であり、ヒュームフードの下で処理する必要があります。ホルムアルデヒドを使用する場合は、必ずラボコート、保護手袋、眼鏡を着用してください。制度上の規制に従って廃棄物を処分する。 - アガロース溶液に適量のゲル染色を加えます(例えば、SYBRセーフDNAゲル染色の15μL)。よく混ぜ、アガロースをトレイに注ぎます。
- すぐにゲルに櫛を挿入します。
- ヒュームフードの下に45分間放置します。敏感なゲル汚れを使用する場合は、アルミホイルでトレイを覆います。
- 1x MOPS バッファの 600 mL を準備します。
-
RNAサンプル調製
- 1.5 mLチューブで、消化および精製されたRNAサンプルの10 μL、サンプル難化バッファーの5μL、6xローディング色素の0.5 μLを組み合わせます。
注意:フォルマミドは有毒であり、ヒュームフードの下で処理する必要があります。フォルマミドを使用する場合は、必ずラボコート、保護手袋、眼鏡を着用してください。制度上の規制に従って廃棄物を処分する。 - 70°CでRNAサンプルを5分間インキュベートし、氷上にサンプルを移します。5分間インキュベートします。
- ゲルにロードする前に、短時間スピンダウンします。
- 1.5 mLチューブで、消化および精製されたRNAサンプルの10 μL、サンプル難化バッファーの5μL、6xローディング色素の0.5 μLを組み合わせます。
- ゲルを電気泳動タンクに入れ、1x MOPSバッファーで満たします。各サンプルの全容積(15μL)をゲルにロードします。ブロモフェノールブルーがゲル長の2/3に達するまで80Vで走る。
- 選択したゲル汚れを検出するのに適したイメージャー(例えば、UVトランジルミネーション)を用いてゲルを画像化する。
結果
rRNA修飾の分析におけるDNAzyme依存性切断の有用性は、snoRNA成熟13の文脈で最近示されている。DNAzyme依存性アッセイは、5'末端のプレsnoRNA処理の欠如がS.セレビシエ13における25Sおよび18S rRNAの2'-O-メチル化レベルに影響を及ぼすことを示すために使用された。
ここでは、誘導性のsnoRNA転写システムを用いて、この技術の有効性と簡易性を実証した。ボックスC/D snR13は、アデニン2281(図2A)を含む25S rRNAの2つの位置でメチル化をガイドします。このヌクレオチドは、10-23 DNAzymeによって硬化可能なコンセンサスジヌクレオチド(RY)を構成するウラシルが続く。ボックスC/D snR47はまた25S rRNAの2つのヌクレオチドのメチル化を導く(図2B)。位置2220のアデニンはグアニン残渣が続き、このジヌクレオチドは8-17 DNAzymeによって切り分けることができる。snR13またはsnR47 snoRNAのいずれかの合成を誘導または阻害するために、我々はSNR13またはSNR47遺伝子のいずれかの上流に誘導性GAL1プロモーターを挿入し、ガラクトースを含有する培地中に培養細胞(GAL1-依存体)上の転写)またはグルコース(GAL1-依存転写オフ)。次に、GAL1:SNR13細胞から単離されたRNAを、snR13依存部位で25S rRNAを切断するように設計された10-23 DNAzymeを用いてインキュベートし、ヌクレオチド2281と2282(図2C)の間にした。GAL1::SNR47株からのRNAを、ヌクレオチド2220〜2221(図2D)の間に8-17 DNAzyme標的snR47依存部位で処理した。対照として、ガラクトースまたはグルコースのいずれかで成長する野生型BY4741株からのRNAを、両方のDNAzymesでインキュベートした。DNAzyme処理RNAの電気泳動は、GAL1::SNR13およびGAL1:SNR47株から抽出された25S rRNAがそのまま残っていることを明らかにした(図3A、B;レーン 3)。対照的に、GAL1:SNR13およびGAL1:SNR47細胞から単離されたRNAは、グルコース(GLC)上で増殖し、それぞれのDNAzymesによって消化された(図3A,B;レーン4)。いずれの場合も、25S rRNAバンドは減少し、5'および3'カットオフ切断産物(AおよびB)が観察された。これは、GAL1:SNR13およびGAL1:SNR47株において、ガラクトースが炭素源として使用され、これらのsnoRNAが発現された場合、snR13-またはsnR47誘導部位で25S rRNAが2'-O-メチル化されたことを示す。snR13またはsnR47発現時の25S rRNAメチル化の欠如は、DNAzyme依存性切断のために許容されるグルコース上で遮断された。野生型試料に対するRNA消化は認められなかった(図3A、B;レーン1および2)。したがって、rRNAは通常メチル化され、DNAzymes活性に対して耐性を有する。
全体として、我々の実験は、10-23(図3A)および8-17(図3B)DNAzymesの切断活性がボックスC/D snR13またはsnR47の不在と相関していることを示し、これらのsnoRNAが25S rRNAに関与していることを明確に示す。2'-O-メチル化は、特定の部位で。
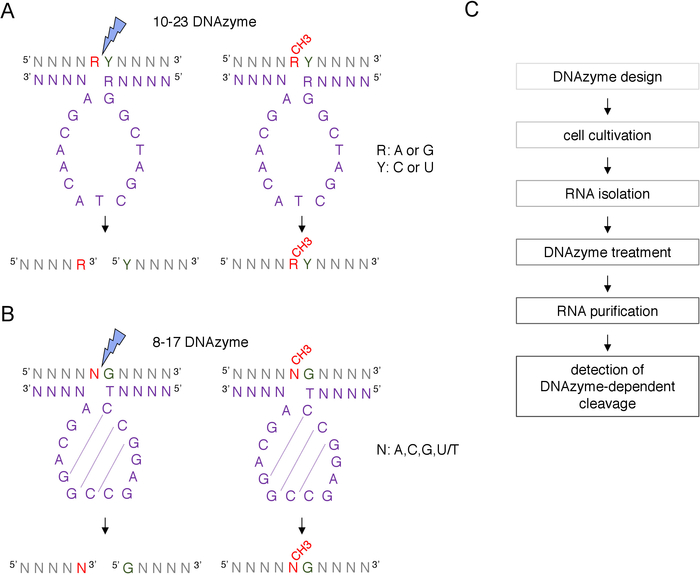
図1:DNAzymes及びそのRNA基板。(A)10-23 DNAzymesは、プリン・ピリミジン(RY)RNAジヌクレオチドをcleavemes.RNA中のRはDNAzymeと対ペアではなく、YはDNAzymeのR塩基に相補的である。RNAにおけるプリン(R)のメチル化は、DNAzyme依存性切断を抑制する。(B)8-17 DNAzymesは、DNAzyme触媒配列の最初のチミンと不完全に組み合わされたグアニンの上流のRNAを切り開く。グアニンの前のヌクレオチドは対化されず、そのメチル化はDNAzyme依存性切断から保護する。RNAは灰色で示され(メチル化部位を除く)、DNAzymeは紫色で示される。N = 任意のヌクレオチド, R = プリン: アデニンまたはグアニン, Y = ピリミジン: シトシンまたはウラシル;CH3-はRNAメチル化を示す。DNAzyme アクティブシーケンス内の基本ペアリングは、点線でマークされます。青い稲妻が切断部位を示します。(C)DNAzyme 依存解析のステップを示すフローチャート。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図2:25S rRNAにおけるsnR13およびsnR47依存性メチル化部位を標的とするDNAzymes。(A, B)snR13依存性(A)およびsnR47依存性(B)25S rRNA(C)snR13依存性メチル化部位(A2281)および10-23 DNAzyme(紫色で示す)を取り巻く25S rRNA配列を示すブラウザスクリーンショットA2281とU2282の間にRNAをcリーブするように設計されています。2281はDNAzymeと対ペアではなく、U2282はDNAzyme活性配列(青い線でマークされた)からの最初のヌクレオチドとの対を形成する。青い稲妻が切断を示す。(D)snR47依存性メチル化部位(A2220)および8-17 DNAzyme(紫色で示す)を取り巻く25S rRNA配列は、A2220とG2221の間でRNAを切断するように設計されている。G 2221はチミンと不完全に組み合わされている間、2220 は DNAzyme とハイブリダイズされません (破線で示されます)。青い稲妻が切断部位を示します。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図3:10-23および8-17 DNAzyme依存性アッセイを用いて25S rRNAの部位特異的2'-O-メチル化の分析。(A)10-23 DNAzymeを用いてsnR13依存性25S rRNAメチル化の解析(B)8-17 DNAzymeを用いてsnR47依存性25S rRNAメチル化の解析RNAは、脱電性アガロースゲル中の染色を可視化した。切断産物AおよびBは赤い矢印で示される。WT = 野生型株;GAL=ガラクトース、GLC=グルコース。この図のより大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
DNAzyme依存性消化は、サイト特異的RNA2'-O-メチル化12、13を分析するための簡単かつ迅速な方法として使用することができる。DNAzymesは、切断部位の上流にあるヌクレオチドがメチル化されていない場合にRNAを切断する。RNase H指向消化を含む他のアプローチとは対照的に、低ヌクレオチド濃度におけるアルカリ分解または逆転転写、定量的PCRまたはシーケンシング8、10、11を含む,16, DNAzyme アプローチは、任意の分子生物学研究室に存在する単純なDNAオリゴヌクレオチドおよび塩基試薬を必要とします。さらに、DNAzymesは、ボックスH/ACA snoRNA12によって媒介されるRNA疑似化を分析する同様の方法で使用することができ、これはsnoRNA標的を研究する上で汎用性の高いツールとなる。
DNAzyme依存的アプローチは、切断部位コンセンサス配列17によってのみ制限される。10-23 DNAzymesは、RYジヌクレオチドの位置Rでのみ2'-O-メチル化を分析するために使用することができ、一方、8-17 DNAzymesはグアニンの上流に位置するヌクレオチドの修飾を認識する。その結果、ジヌクレオチド中の第1ヌクレオチドの2'-O-メチル化のような修飾は、グアニンアデニン(GA)、アデニン-アデニン(AA)、ピリミジンアデニン(YA)およびピリミジン-ピリミジン(YY)を分析することができない。また、DNAzyme依存性切断12の低効率を考慮すべきである。一部のDNAzymesはRNAをほぼ完全に包むが(図3B)、多くのDNAzymesは標的を部分的にしか消化しない(図3B)。効率は、切断部位を取り巻く配列に依存してもよい。例えば、同じヌクレオチドの伸びを有するRNA領域は、DNAzyme活性配列の正しい位置決まりに影響を与えうる。さらに、強い二次構造を形成するRNA領域は、標的配列へのDNAzyme結合を再ハイブリダイズおよび抑制してもよい。これらの問題を克服するために、10-23 DNAzymeおよびそのRNA基板の加熱および冷却のサイクルを18に適用することができる。
我々は、DNAzymeアプローチを用して、rRNAの2'-O-メチル化を調べた。1つは、N6-メチルラデノシン19などの他のRNA修飾を分析するために、この技術を使用することができます。リボソームRNAは、その豊富さのために、電気泳動によって分析することができ、切断産物は、紫外線の下で可視化することができます。しかし、これはRNAポリメラーゼII生成コードRNA(mRNA)および非コードRNA(ncRNA)のようなあまり豊富なRNAには適用されません。これらのRNAは、通常、アガロースまたはポリアクリルアミドゲル中のRNA染色によって直接検出することはできません。このような場合、DNAzyme依存性切断は、ノーザンブロッティングによって可視化することができ、PCR/定量PCRによって間接的に検出されるか、またはポリメラーゼ(例えば、クレンタクDNAポリメラーゼ)を用いて定量的PCRによって分析され、2'-O-メチル化RNAを判別することができる。非メチル化RNA20,21.
開示事項
著者は何も開示していない。
謝辞
マヤ・ウィルソンとアネイカ・レニーに原稿を批判的に読んでくれたことに感謝します。この作品は、ウェルカム・トラストとロイヤル・ソサエティ(200473/Z/16/Z)が共同出資するヘンリー・デール・フェローシップによって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Acid phenol | SIGMA | P4682 | |
| Agarose | VWR | A2114 | |
| Ammonium acetate | SIGMA | A1542 | |
| Chlorophorm | Fisher scientific | 10293850 | |
| DNase/RNase free water | Fischer Scientific | 10526945 | |
| DNAzyme | Integrated DNA Technology | Custom oligo DNA | |
| EDTA | SIGMA | E9884 | |
| Ethanol Absolute | Fisher scientific | 10437341 | |
| Formaldehyde | Sigma | F8775 | |
| Formamide | sigma | F9037 | |
| Galactose | SIGMA | G0750 | |
| Gel Loading Dye | Thermo Fisher Scientific | R0611 | |
| Glucose | SIGMA | G7021 | |
| Glycogen | Thermo Fisher Scientific | R0561 | |
| HEPES | SIGMA | H3375 | |
| Isoamyl | SIGMA | W205702 | |
| KCl | SIGMA | P9333 | |
| MgCl2 | SIGMA | M8266 | |
| MnCl2 | SIGMA | 244589 | |
| MOPS | SIGMA | M1254 | |
| NaCl | SIGMA | S7653 | |
| Oxoid Peptone Bacteriological | Thermo Fisher Scientific | LP0037 | |
| Oxoid Yeast Extract Powder | Thermo Fisher Scientific | LP0021 | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher Scientific | EO0382 | |
| SDS | SIGMA | 74255 | |
| Sodium acetate trihydrate | SIGMA | S8625 | |
| SYBR Safe DNA Gel Stain | Thermo Fisher Scientific | S33102 | |
| Tris base | SIGMA | TRIS-RO | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 1.5 mL microtubes | Sarstedt | ||
| 152VR5C01M -80°C freezer | Thermo Fisher Scientific | ||
| 250 mL Erlenmeyer flasks | Cole-Parmer | ||
| 50 mL conical tubes | Sarstedt | ||
| Combicup VX200 vortex | Appleton Woods | ||
| DS-11 microspectrophotometer | Denovix | ||
| Electrophoresis chamber (20 cm tray) | SIGMA | ||
| FiveEasy F20 pH meter | Appleton Woods | ||
| Gel documentation system | Syngene | ||
| Heraeus Fresco 21 micro centrifuge | Fisher Scientific | ||
| Megafuge 8R centrifuge with rotator suitable for 50 mL conical tubes | Fisher Scientific | ||
| Mini Fuge Plus mini centrifuge | Starlab | ||
| Mixer HC thermal block | Starlab | ||
| OLS26 Shaking Water Bath | Grant | ||
| PowerPac power supplier | BioRad |
参考文献
- Dieci, G., Preti, M., Montanini, B. Eukaryotic snoRNAs: a paradigm for gene expression flexibility. Genomics. 94 (2), 83-88 (2009).
- Watkins, N. J., Bohnsack, M. T. The box C/D and H/ACA snoRNPs: key players in the modification, processing and the dynamic folding of ribosomal RNA. Wiley Interdisciplinary Review RNA. 3 (3), 397-414 (2012).
- Kufel, J., Grzechnik, P. Small Nucleolar RNAs Tell a Different Tale. Trends in Genetics. , (2018).
- Li, T., Zhou, X., Wang, X., Zhu, D., Zhang, Y. Identification and characterization of human snoRNA core promoters. Genomics. 96 (1), 50-56 (2010).
- Jorjani, H., et al. An updated human snoRNAome. Nucleic Acids Research. 44 (11), 5068-5082 (2016).
- Hubbard, T. J., et al. Ensembl 2009. Nucleic Acids Research. 37 (Database issue), D690-D697 (2009).
- Makarova, J. A., Kramerov, D. A. SNOntology: Myriads of novel snoRNAs or just a mirage?. BMC Genomics. 12, 543 (2011).
- Yu, Y. T., Shu, M. D., Steitz, J. A. A new method for detecting sites of 2'-O-methylation in RNA molecules. RNA. 3 (3), 324-331 (1997).
- Decatur, W. A., Liang, X. H., Piekna-Przybylska, D., Fournier, M. J. Identifying effects of snoRNA-guided modifications on the synthesis and function of the yeast ribosome. Methods in Enzymology. 425, 283-316 (2007).
- Dong, Z. W., et al. RTL-P: a sensitive approach for detecting sites of 2'-O-methylation in RNA molecules. Nucleic Acids Research. 40 (20), e157 (2012).
- Birkedal, U., et al. Profiling of ribose methylations in RNA by high-throughput sequencing. Angewandte Chemie International Edition, England. 54 (2), 451-455 (2015).
- Buchhaupt, M., Peifer, C., Entian, K. D. Analysis of 2'-O-methylated nucleosides and pseudouridines in ribosomal RNAs using DNAzymes. Analytical Biochemistry. 361 (1), 102-108 (2007).
- Grzechnik, P., et al. Nuclear fate of yeast snoRNA is determined by co-transcriptional Rnt1 cleavage. Nature Communication. 9 (1), 1783 (2018).
- Piekna-Przybylska, D., Decatur, W. A., Fournier, M. J. New bioinformatic tools for analysis of nucleotide modifications in eukaryotic rRNA. RNA. 13 (3), 305-312 (2007).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18 (10), 3091-3092 (1990).
- Maden, B. E. Mapping 2'-O-methyl groups in ribosomal RNA. Methods. 25 (3), 374-382 (2001).
- Santoro, S. W., Joyce, G. F. A general purpose RNA-cleaving DNA enzyme. Proceedings of the National Academy of Science, U. S. A. 94 (9), 4262-4266 (1997).
- Hengesbach, M., Meusburger, M., Lyko, F., Helm, M. Use of DNAzymes for site-specific analysis of ribonucleotide modifications. RNA. 14 (1), 180-187 (2008).
- Sednev, M. V., et al. N(6) -Methyladenosine-Sensitive RNA-Cleaving Deoxyribozymes. Angewandte Chemie International Edition, England. 57 (6), 15117-15121 (2018).
- Aschenbrenner, J., Marx, A. Direct and site-specific quantification of RNA 2'-O-methylation by PCR with an engineered DNA polymerase. Nucleic Acids Research. 44 (8), 3495-3502 (2016).
- Lee, K. W., Bogenhagen, D. F. Assignment of 2'-O-methyltransferases to modification sites on the mammalian mitochondrial large subunit 16 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (36), 24936-24942 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved