Method Article
Analisi dipendente da DNAzyme del rRNA 2'-O-Metilazione
In questo articolo
Riepilogo
Qui presentiamo un protocollo per la scissione dell'RNA dipendente da DNAzyme. Ciò consente un'analisi rapida e dipendente dal sito dell'RNA 2'-O-metilazione. Questo approccio può essere utilizzato per la valutazione preliminare o importante dell'attività dello snoRNA.
Abstract
Riquadro guida C/D piccoli RNA nucleolari (snoRNA) catalizzano 2'-O-metilazione dell'RNA ribosomal e nucleare piccolo. Tuttavia, un gran numero di snoRNA in eucarioti superiori può riconoscere promiscuamente altre specie di RNA e 2'-O-metilato più bersagli. Qui, forniamo una guida passo-passo per l'analisi rapida e non costosa della metilazione 2'O-o-metilazione specifica del sito utilizzando un metodo ben consolidato che impiega oligonucleotidi di DNA corti chiamato DNAzymes. Questi frammenti di DNA contengono sequenze catalitiche che fendeno l'RNA in posizioni di consenso specifiche, così come bracci di omologia variabile che indirizzano il Dna ai suoi bersagli di RNA. L'attività del DNAzyme è inibita da 2-'O-metilazione del nucleotide adiacente al sito di scissione nell'RNA. Così, i DNAzymes, limitati solo dal consenso della sequenza scissionda, sono strumenti perfetti per la rapida analisi dell'RNA 2'-O-metilazione mediata dallo snoRNA. Abbiamo analizzato snoRNA snR13- e snR47-guidato 2'-O-metilazione di 25S ribosomico IN Saccharomyces cerevisiae per dimostrare la semplicità della tecnica e per fornire un protocollo dettagliato per il saggio dipendente da DNAzyme.
Introduzione
Le modifiche dell'RNA svolgono un ruolo importante nella regolazione dell'espressione genica. RNA 2'-O-metilazione e pseudouridilazione, che sono guidati da scatola C/D e scatola H/ACA piccoli RNA nucleolari (snoRNA) rispettivamente, proteggere l'RNA dalla degradazione e stabilizzare le loro strutture di ordine superiore1,2,3 . Gli obiettivi di SnoRNA sono stati identificati principalmente negli RNA ribosomici (rRNA) e nei piccoli RNA nucleari (snRNA). Tuttavia, negli eucarioti superiori, ci sono potenzialmente centinaia di snoRNA senza funzioni assegnate e alcuni di loro possono riconoscere più RNA1,4,5,6,7. Pertanto, i metodi che consentono l'identificazione e l'analisi delle modifiche guidate dallo snoRNA sono strumenti importanti per scoprire i meccanismi che regolano i processi cellulari.
Un sito di putazione 2'-O-metilazione a cimpianto guidato dallo snoRNA C/D può essere identificato bioinformaticamente e confermato sperimentalmente da molte tecniche, tra cui la scissione diretta da RNase H, o metodi site-specific e a livello di genoma, che impiegano la trascrizione inversa nei nucleotidi bassi (dNTP) l'approccio alla concentrazione8,9,10,11. Queste tecniche sono molto sensibili, ma anche laboriose e costose, quindi, potrebbero non essere adatte per il test iniziale o rapido. Uno dei metodi più semplici e a basso costo per identificare i siti di metilazione 2'-O-o-metilazione è la cleavazione dell'RNA dipendente dal DNAzyme12. I DNA sono molecole di DNA corte, a filamento singolo e catalicamente attive in grado di scissione endonucleolitica di RNA in posizioni specifiche. Sono costituiti da una sequenza di base conservata e cataliticamente attiva e bracci di legame 5 e 3' composti da sequenze variabili progettate per ibridare da Watson-Crick base-pairing al bersaglio RNA (Figura 1). Così, le braccia 5' e 3 ' forniscono la sequenza catalitica al sito specifico dell'RNA. La scissione dipendente dal DNAzyme è inibita dalla metilazione 2'-O-metilazione del nucleotide posizionato direttamente a monte del sito di scissione12,13. Questo rende i DNAzymes strumenti molto pratici per l'analisi dei siti putativi o noti di RNA 2'-O-metilazione.
Due tipi di DNAzymes sono utilizzati per le modifiche dell'RNA analisi12. La sequenza attiva di 10-23 DNAzyme (Figura 1A) è costituita da 15 nucleotidi (5'RGGCTAGCTACACGA3') che formano un ciclo intorno alla dinucleotide RNA purina-pirimidina (RY) mirata e catalizzano la scissione tra questi due nucleotidi. La purina dell'RNA (R) non è accoppiata alla base con il DNAzyme e la metilazione 2'-O-metilazione presente sul DNAzyme inibisce la scissione. I bracci di legame di 10-23 DNAzymes sono di solito lunghi 10-15 nucleotidi. La seconda classe DNAzyme, 8-17 DNAzymes (Figura 1B) contiene una sequenza catalitica a 14 nucleotidi (5'TCCCCGGACGA3'). Nucleotides C2, C3 e G4 coppia con C9 G10 e G11 formando una struttura breve stelo-loop. 8-17 DNAzymes fende l'RNA a monte di qualsiasi guanino che sia perfettamente abbinato alla prima timina della sequenza attiva DNAzyme. Il nucleotide dell'RNA a monte della guanina non è accoppiato alla base con il DNAzyme e la sua metilazione 2'-O-metilazione compromette la scissione. 8-17 DNAzymes richiedono bracci omologia più lunghi di circa 20 nucleotidi per indirizzare il DNAzyme alla sua sequenza specifica.
Qui, forniamo un protocollo passo-passo per l'analisi di 2'-O-metilazione di rRNA in Saccharomyces cerevisiae utilizzando 10-23 e 8-17 approcci dipendenti da DNAzyme12,13 (Figura 1C). Questo protocollo può essere facilmente adattato ad altri organismi e specie di RNA e impiegato per le analisi rapide, preliminari o principali dell'RNA 2'-O-metilazione site-specific.
Protocollo
1. Sceppamenti, supporti e ricette buffer
- Preparare i supporti per il lievito (S. cerevisiae) come descritto qui: YP (1% w/v di estratto di lievito, 2% w/v peptone batteriologico), e gli stock di glucosio e galactose al 20% w/v.
- Preparare il buffer di acetato di sodio (NaAc)-EDTA (AE) come descritto qui: 50 mM NaAc pH 5.3 e 10 mM EDTA.
- Preparare 10-23 DNAzyme 4x Incubation Buffer come descritto qui: 24 mM Tris pH 8.0, 60 mM NaCl e 10-23 DNAzyme 4x Buffer di reazione: 200 mM Tris pH 8.0 e 600 mM NaCl.
- Preparare 8-17 DNAzyme 2x Reaction Buffer come descritto qui: 200 mM KCl, 800 mM NaCl, 100 mM HEPES pH 7.0, 15 mM MgCl2e 15 mM MnCl2.
- Preparare 10 volte MOPS Buffer come descritto qui: 200 mM MOPS, 50 mM NaAc, 1 mM EDTA; pH 7.0 e 1,5x Buffer di Denaturing del campione: 50% v/v formamide, 20% v/v formaldeide, buffer MOPS 1.5x.
- Ottenere ceppi S. cerevisiae, BY4741 (MATa his3-1 leu20 0 met150 - ura3 - 0); Elenco indirizzi globale1::SNR13 (come BY4741 ma GAL1::SNR13:KANmX); GAL1::SNR47 (come BY4741 ma GAL1::SNR47:HIS3mX). Qualsiasi altro ceppo di lievito può essere utilizzato per questa analisi.
2. DNAzyme Design
- Trova la sequenza di RNA di interesse o il sito di metilazione putativa utilizzando un database appropriato. Per i bersagli snoRNA S. cerevisiae, utilizzare il database dello snoRNA del lievito: http://people.biochem.umass.edu/fournierlab/snornadb/mastertable.php14
- Per trovare il sito di metilazione di interesse, ad esempio, sito dipendente da snR13, selezionare "snR13" e prendere nota della posizione del nucleotide modificato (ad esempio, A2281 guidato da snR13 in 25S rRNA).
- Trova le sequenze a monte e a valle del nucleotide modificato utilizzando un database appropriato. Per S. cerevisiae, utilizzare il database del genoma di Saccharomyces: https://www.yeastgenome.org/
- Cercare il nome del gene bersaglio, ad esempio RDN25 (codificando 25S rRNA).
- Dalla scheda "sequenza", selezionare 10-15 nucleotidi a monte (5" braccio) e a valle (3 'braccio) del sito di metilazione quando si utilizza un saggio 10-23 DNAzyme e 20 nucleotidi a monte (5' braccio) e a valle (3'braccio) del sito di metilazione per un 8-17 DNAzyme.
- Creare sequenze complementari di braccia da 5 e 3'.
- Affianco 10-23 o 8-17 Sequenza catalitica DNAzyme con sequenze complementari di braccia 5' e 3'.
- Ordina DNAzyme come un normale oligonucleotide del DNA dal fornitore.
3. Condizioni di crescita S. cerevisiae
NOT: Sono stati utilizzati derivati della deformazione S. cerevisiae BY4741, in cui l'espressione dello snoRNA SNR13 o SNR47 è scacciata dal promotore inducibile di GAL1. Per indurre o inibire la loro sintesi, far crescere le cellule su un mezzo contenente galactosi (trascrizione dipendente daGAL1)o glucosio ( trascrizione dipendente daGAL1). Come controllo, utilizzare il ceppo di tipo selvatico (BY4741) cresciuto sia su galactose o glucosio.
- Coltivare ceppi di lievito in un mezzo e condizioni appropriate. Per analizzare i ceppi GAL1::SNR13 e GAL1::SNR47 e il ceppo isogenico di tipo selvaggio, far crescere le cellule in 50 mL di supporti YP con un glucosio del 2% (YPD) o un galactose (YPGal) a 30 gradi centigradi alla fase esponenziale centrale.
- Centrifuga cellule a 1.000 x g, per 3 min a 4 gradi centigradi.
- Scartare il supernatante e tenere i pellet.
- Congelare i pellet cellulari in azoto liquido e conservarli a -80 gradi centigradi.
ATTENZIONE: L'azoto liquido può causare gravi ustioni criogeniche. Indossare sempre indumenti protettivi e precauzioni di sicurezza per l'esercizio fisico.
NOT: I pellet cellulari possono essere conservati a -80 gradi centigradi fino a 1 mese. Il protocollo può essere sospeso qui se necessario.
4. Isolamento RNA15
NOT: Utilizzare il metodo più appropriato per isolare l'RNA. Per il lievito S. cerevisiae, può essere utilizzata l'estrazione di RNA a caldo-fenolo.
- Aggiungere 1 mL di acqua ghiacciata, sospendere i pellet e trasferire le cellule risospese a microtubi da 1,5 mL.
- Centrifuga a 20.000 x g per 10 s a 4 gradi centigradi e rimuovere il supernatante.
- Aggiungere 400 l di buffer AE e sospendere nuovamente le celle.
NOT: I passaggi 4.4-4.15 vengono eseguiti a temperatura ambiente, salvo diversa indicazione. - Aggiungere 40 -L di 10% SDS e 400 l of acid phenol (pH 4.5).
ATTENZIONE: Il fenolo è tossico e deve essere maneggiato sotto un cappuccio fumatorio. Indossare sempre un camice da laboratorio, guanti protettivi e occhiali quando si lavora con il fenolo. Smaltire i rifiuti secondo le normative istituzionali. - Mescolare bene vorticendo per 20 s.
- Incubare a 65 gradi centigradi per 10 min. Ogni 2 min, aprire delicatamente e chiudere il tubo per rilasciare la pressione e capovolgere il tubo 2-3 volte per mescolare le fasi.
- Trasferire i tubi a -80 gradi centigradi e incubare per 10 min.
- Scongelare i tubi sulla panca e centrifugare a 20.000 x g, per 5 min a temperatura ambiente.
- Trasferire la fase superiore in un nuovo tubo contenente 400 -L di fenolo acido:cloroformato:alcool isoamyl (25:24:1). Non interrompere l'interfase.
ATTENZIONE: Il cloroformio è tossico e deve essere maneggiato sotto un cofano di fumi. Indossare sempre un camice da laboratorio, guanti protettivi e occhiali quando si lavora con cloroformio. Smaltire i rifiuti secondo le normative istituzionali. - Mescolare bene vorticendo per 30 s e centrifugare a 20.000 x g per 10 min a temperatura ambiente.
- Trasferire la fase superiore (400 dollari l) in un nuovo tubo contenente 400 cloroformi ll.
- Mescolare bene vorticendo per 30 s e centrifugare a 20.000 x g per 5 min a temperatura ambiente.
- Trasferire la fase superiore (300-350 l) in un nuovo tubo contenente 1 mL di EtOH e 40 -L di 7,5 M di acetato di ammonio (NH4AC). Mescolare capovolgendo il tubo un paio di volte.
- Incubare a -80 gradi per 2 h o peruna notte a -20 gradi centigradi.
NOT: La procedura può essere messa in pausa qui. - Centrifuga a 20.000 x g, per 10 min a 4 gradi centigradi. Un piccolo pellet di RNA bianco diventerà visibile sul fondo del tubo.
- Rimuovere EtOH pipetting per evitare di disturbare il pellet.
- Aggiungere 1 mL di 70% EtOH e centrifugare a 20.000 x g per 5 min a temperatura ambiente.
- Rimuovere il 70% di EtOH pipettando.
- Centrifuga a 20.000 x g per 15 s e rimuovere la restante EtOH con 2-20 pipetta ll.
- Lasciare il tubo aperto sulla panca per 5 min per asciugare il pellet di RNA.
NOT: RNA pellet cambia colore dal bianco al trasparente quando è asciutto. - Risospendere il pellet di RNA in 30 gradi l di RNase/DNase-free H2O, trasferire il tubo immediatamente sul ghiaccio e misurare la concentrazione di RNA sul microspettrofotometro.
- Congelare i campioni a -20 gradi centigradi.
NOT: L'RNA può essere immagazzinato a -20 gradi centigradi fino a 1 mese e a -80 gradi centigradi fino a 1 anno. La procedura può essere messa in pausa qui o ha proceduto direttamente al passaggio successivo.
5. Digestione di DNAzyme
-
10-23 digestione DNAzyme
- In 1,5 mL i tubi preparano un mix di incubazione combinando 5 g di RNA, 200 pmol di 10-23 DNAzyme (2 -L di 100 mM di soluzione di riserva) e 2,5 luna di 4x 10-23 Buffer di incubazione in un volume totale di 10.L. Mantenere i tubi sul ghiaccio.
- Trasferire i tubi in un blocco di calore asciutto a 95 gradi centigradi e incubare per 3 min.
- Trasferire immediatamente i tubi sul ghiaccio e incubare per 5 min.
- Girare giù brevemente e mettere i tubi di nuovo sul ghiaccio.
- Aggiungere 20 U di inibitore di RNase (ad esempio, 0,5 -L L RiboLock RNase inibitore).
- Mettere i tubi in un blocco di calore asciutto per 25 gradi centigradi e incubare per 10 minuti.
- Nel frattempo, preparate una miscela di reazione in un tubo da 1,5 ll combinando 5 -L di 4x 10-23 Reaction Buffer con 4 XL di 300 mM MgCl2 e 1 -L H2O. Collocare il tubo in un blocco secco impostato su 37 .
- Trasferire il mix di incubazione in un blocco di calore asciutto per 37 gradi centigradi e aggiungere 10 L di miscela di reazione preriscaldata.
- Incubare la reazione a 37 gradi centigradi per 1 h.
- Trasferire i tubi sul ghiaccio e procedere al passaggio 5.3.1.
-
8-17 digestione dNAzyme
- Preparare un microtubo da 1,5 mL con 5 g di RNA in un volume totale di 6.L. Tenere il tubo sul ghiaccio.
- Preparare un microtubo da 1,5 mL con 400 pmol di 8-17 DNAzyme (4 - L su 100 mM di magazzino). Tieni il tubo sul ghiaccio.
- Trasferire i tubi in un blocco di calore asciutto per 95 gradi centigradi e incubare per 2 min.
- Spostare il campione di RNA sul ghiaccio.
- Girare verso il basso il tubo con DNAzyme per 5 s e incubare a 25 gradi centigradi per 10 min.
- Allo stesso tempo, preparare un tubo da 1,5 mL con 10 gradi l di 2x 8-17 Reaction Buffer e incubare a 25 gradi centigradi.
- Preparare una miscela di reazione aggiungendo 10 l di buffer di reazione preriscaldato 2x al tubo con DNAzyme.
- Trasferire 14 - L della miscela di reazione al tubo con RNA e aggiungere 20 U di inibitore di RNase.
- Incubare la reazione a 25 gradi centigradi per 2 h.
- Trasferire il tubo sul ghiaccio e procedere alla purificazione dell'RNA (passaggio 5.3.1).
-
Purificazione dell'RNA
- Aggiungere 350 gradi d'acqua e 400 l' di cloroformio al tubo di reazione, mescolare bene vorticendo per 30 s e centrifugare a 20.000 g per 5 min a temperatura ambiente.
- Trasferire la fase superiore (300-350 l) in un nuovo tubo contenente 1 mL di EtOH, 40 -L di 7,5 M NH4AC e 1 -L di glicogeno (10 g/l). Mescolare capovolgendo il tubo un paio di volte.
- Incubare a -80 gradi per 2 h o peruna notte a -20 gradi centigradi.
NOT: La procedura può essere messa in pausa qui. - Ripetere i passaggi da 4.15 a 4.21.
- Risospendere il pellet di RNA in 10 gradi l di RNase/DNase-free H2O e trasferire immediatamente i tubi sul ghiaccio.
- Congelare i campioni a -20 gradi centigradi.
NOT: L'RNA può essere immagazzinato a -20 gradi centigradi fino a un mese e a -80 gradi centigradi fino a 1 anno. La procedura può essere messa in pausa qui o procedere all'elettroforesi dell'RNA.
6. RNA Elettroforesi
- Spruzzare l'apparecchiatura di elettroforesi (serbatoio, vassoio, pettine) con 1% SDS, lasciare per 15 min e risciacquare con abbondante ddH2O.
- Sciogliere 1,5 g di agarose in 127,5 mL di ddH2O riscaldandolo nel microonde.
- Aggiungere 15 mL di 10x MOPS e 7,5 mL del 37% di formaldeide alla soluzione agarose (il volume totale è 150 mL).
ATTENZIONE: La formaldeide è tossica e deve essere maneggiata sotto un cofano di fumi. Indossare sempre un camice da laboratorio, guanti protettivi e occhiali quando si lavora con formaldeide. Smaltire i rifiuti secondo le normative istituzionali. - Aggiungere un'adeguata quantità di una macchia di gel preferita alla soluzione agarose (ad esempio, 15 -L di macchia di gel di DNA sicuro SYBR). Mescolare bene e versare l'agarose al vassoio.
- Inserire immediatamente un pettine nel gel.
- Lasciare per 45 min sotto il cofano fuma. Coprire il vassoio con un foglio di alluminio quando si utilizza una macchia di gel sensibile alla luce.
- Preparare 600 mL di 1x buffer MOPS.
-
Preparazione del campione di RNA
- In un tubo da 1,5 mL, unire 10 l del campione di RNA digerito e purificato, 5 l of Sample Denaturing Buffer e 0,5 ll di 6x Loading Dye.
ATTENZIONE: Formamide è tossico e deve essere maneggiato sotto un cappuccio fuma. Indossare sempre un camice da laboratorio, guanti protettivi e occhiali quando si lavora con formamide. Smaltire i rifiuti secondo le normative istituzionali. - Incubare campioni di RNA a 70 gradi centigradi per 5 min. Incubare per 5 min.
- Girare brevemente verso il basso prima di caricare sul gel.
- In un tubo da 1,5 mL, unire 10 l del campione di RNA digerito e purificato, 5 l of Sample Denaturing Buffer e 0,5 ll di 6x Loading Dye.
- Mettere il gel nel serbatoio dell'elettroforesi e riempire con 1x tampone MOPS. Caricare l'intero volume di ogni campione (15 o L) sul gel. Eseguire a 80 V fino a quando il blu bromofenolo raggiunge 2/3 della lunghezza del gel.
- Immagina il gel usando un imager appropriato per rilevare la macchia di gel scelta (ad esempio, transilluminatore UV).
Risultati
L'utilità della scissione dipendente dal DNAzyme nell'analisi delle modifiche al rRNA è stata dimostrata di recente nel contesto della maturazione degli snoRNA13. Il saggio dipendente dal DNAzyme è stato usato per dimostrare che la mancanza di elaborazione pre-snoRNA a 5'end influisce sui livelli di 2'O-metilazione di 25S e 18S rRNA in S. cerevisiae13.
Qui, abbiamo usato un sistema di trascrizione del snoRNA inducibile per dimostrare l'efficacia e la semplicità della tecnica. Box C/D snR13 guida la metilazione in due posizioni in 25S rRNA, tra cui l'adenina 2281 (Figura 2A). Questo nucleotide è seguito da uracilo, che costituisce il dinucleotide di consenso (RY) scissione da un 10-23 DNAzyme. Box C/D snR47 guida anche la metilazione di due nucleotidi in rRNA 25S (Figura 2B). L'adenina in posizione 2220 è seguita da un residuo di guanina e questo dinucleotide può essere scisso da un 8-17 DNAzyme. Al fine di indurre o inibire la sintesi di snR13 o snR47 snoRNA, abbiamo inserito il promotore inducibile GAL1 a monte dei geni SNR13 o SNR47 e delle cellule coltivate in media contenenti galactose(GAL1-dipendenti da GALactose ( GAL1 -dipendenti da GALactose trascrizione) o glucosio(trascrizionedipendente da GAL1 disattivata). Successivamente, l'RNA isolato dalle cellule GAL1::SNR13 è stato incubato con 10-23 DNAzyme progettato per fendere 25S rRNA in un sito dipendente da snR13, tra i nucleotidi 2281 e 2282 (Figura 2C). L'RNA proveniente da GAL1::SNR47 è stato trattato con 8-17 dNAzyme destinati al sito dipendente da snR47 tra i nucleotidi 2220 e 2221 (Figura 2D). Come controllo, l'RNA del ceppo BY4741 di tipo selvatico che cresce su galactose o glucosio è stato incubato con entrambi i DNAzymes. L'elettroforesi dell'RNA trattato con DNAzyme ha rivelato che 25S rRNA estratto da GAL1::SNR13 e GAL1::SNR47 che crescono sulla galactose (GAL) è rimasto intatto(Figura 3A,B; corsie 3). Al contrario, l'RNA isolato da GAL1::SNR13 e GAL1::SNR47 cellule che crescono sul glucosio (GLC) è stato digerito dai rispettivi DNAzymes(Figura 3A, B; corsie 4). In entrambi i casi, la banda di rRNA 25S è diminuita e sono stati osservati prodotti di scissione tagliati a 5 e 3' (A e B). Ciò indica che nei ceppi GAL1::SNR13 e GAL1::SNR47, 25S rRNA era 2'-O-metilato nei siti snR13 o snR47-guided quando galactose è stato utilizzato come fonte di carbonio e questi snoRNA sono stati espressi. La mancanza di metilazione rRNA 25S quando l'espressione snR13 o snR47 è stata chiusa sul glucosio consentita per la scissione dipendente dal DNAzyme. Non è stata osservata alcuna digestione dell'RNA per campioni di tipo selvatico (Figura 3A,B; corsie 1 e 2), poiché l'espressione di snR13 e snR47 è indipendente dal galactose/glucoseino in questo ceppo. Pertanto, l'rRNA era normalmente metilato e così resistente all'attività dei DNAzymes.
Nel complesso, il nostro esperimento mostra che l'attività di scissione di 10-23 (Figura 3A) e 8-17 (Figura 3B) DNAzymes correlava con l'assenza di scatola C/D snR13 o snR47, indicando chiaramente che questi snoRNA sono responsabili di 25S rRNA 2'-O-metilazione in siti particolari.
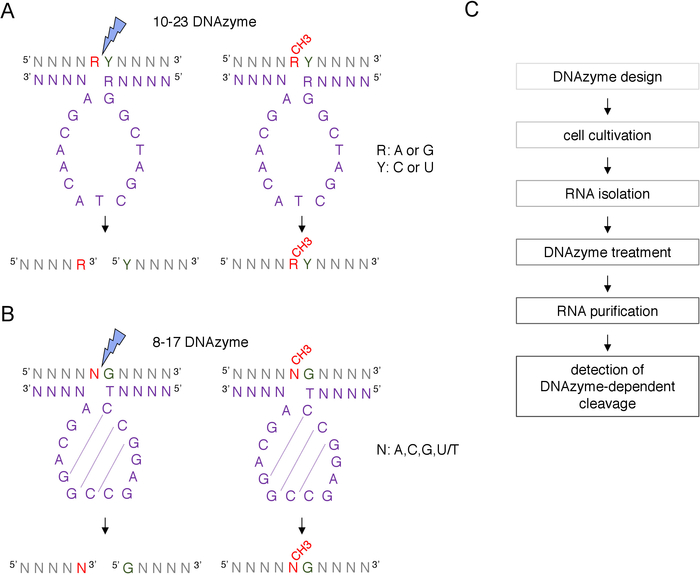
Figura 1: DNAzymes e i loro substrati RNA. (A) 10-23 DNAzymes sciolsono una dinucleotide di purina-pirimidina (RY). R nell'RNA non è accoppiato con DNAzyme, mentre Y è complementare alla base R nel DNAzyme. La metilazione della purina (R) nell'RNA sopprime la scissione dipendente dal DNAzyme. (B) 8-17 DNAzymes spaccano l'RNA a monte della guanina che è in abbinamento imperfetto con la prima timina nella sequenza catalitica DNAzyme. Il nucleotide che precede il guanino non è accoppiato e la sua metilazione protegge dalla scissione dipendente dal DNAzyme. L'RNA è mostrato in grigio (a parte il sito di metilazione), il DNAzyme è mostrato in viola. N - qualsiasi nucleotide, R - purina: adenina o guanina, Y : pirimidina: citosmina o uracil; CH3- denota la metilazione dell'RNA. L'associazione di base all'interno delle sequenze attive DNAzyme è contrassegnata da linee tratteggiate. Un fulmine blu segna il sito di scissione. (C) Un diagramma di flusso che mostra i passaggi di un'analisi dipendente da DNAzyme. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: DNAzymes destinati a siti di metilazione dipendenti da snR13 e snR47 in rRNA 25S. (A, B) Schermate del browser che mostrano i siti di metilazione dipendenti da snR13(A)e snR47(B)nei siti di metilazione 25S rRNA (C) 25S che circondano il sito di metilazione dipendente da snR13 (A2281) e 10-23 DNAzyme (mostrato in viola) progettato per fendere l'RNA tra A2281 e U2282. Un2281 non è accoppiato con il DNAzyme mentre U2282 forma una coppia con il primo nucleotide dalla sequenza attiva DNAzyme (contrassegnato da una linea blu). Un fulmine blu segna la scissione. (D) Sequenza di rRNA 25S che circonda il sito di metilazione dipendente da snR47 (A2220) e 8-17 DNAzyme (mostrato in viola) progettato per fendere l'RNA tra A2220 e G2221. Un2220 non è ibridato con il DNAzyme mentre G2221 è perfettamente accoppiato con la timina (denotato con una linea tratteggiata). Un fulmine blu segna il sito di scissione. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Analisi del modello 2'-O-metilazione 25S specifico del sito di rRNA utilizzando 10-23 e 8-17 analisi dipendenti da DNAzyme. (A) Analisi della metilazione rRNA 25S dipendente da snR13 utilizzando 10-23 DNAzyme. (B) Analisi della metilazione rRNA 25S dipendente da snR47 utilizzando 8-17 DNAzyme. L'RNA è stato visualizzato colorazione in un gel di agarose dente. I prodotti Cleavage A e B sono contrassegnati da frecce rosse. WT - ceppo di tipo selvaggio; L'elenco indirizzi globale è un galactose, un GLC e un glucosio. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La digestione dipendente da DNAzyme può essere utilizzata come metodo semplice e rapido per analizzare l'RNA 2'-O-metilatura specifico del sito12,13. I DNAzymes freno l'RNA se il nucleotide a monte del sito di scissione non è metilato. A differenza di altri approcci, tra cui la digestione diretta da RNase H, la degradazione alcalina o la trascrizione inversa nella bassa concentrazione di nucleotidi seguita da PCR quantitativo o sequenziamento8,10,11 ,16, approccio DNAzyme richiede un semplice oligonucleotide del DNA e reagenti di base che sono presenti in qualsiasi laboratorio di biologia molecolare. Inoltre, i DNAzymes possono essere utilizzati in modo simile per analizzare la pseudouridilazione dell'RNA mediata dalla scatola H/ACA snoRNA12, che li rende strumenti versatili nello studio dei bersagli dello snoRNA.
Gli approcci dipendenti da DNAzyme sono limitati solo dalle sequenze di consenso del sito di scissione17. 10-23 DNAzymes possono essere utilizzati per analizzare 2'-O-metilazione solo nella posizione R del dinucleotide RY, mentre 8-17 DNAzymes riconoscono la modifica del nucleotide situato a monte della guanina. Di conseguenza, non è possibile analizzare modifiche come la metilazione 2'-O-metilazione del primo nucleotide nei dinucleotidi guanine-adenina (GA), adenina (AA), pirinesina (YA) e pirimidina (YY). Inoltre, la bassa efficienza della scissione dipendente dal DNAzyme12 dovrebbe essere considerata. Sebbene alcuni DNAzymes frenino l'RNA quasi completamente (Figura 3B), molti DNAzimes digeriscono solo parzialmente i loro obiettivi (Figura 3B). L'efficienza può dipendere dalla sequenza che circonda il sito di scissione. Ad esempio, le regioni dell'RNA con distese dello stesso nucleotide possono influenzare il corretto posizionamento della sequenza attiva DNAzyme. Inoltre, le regioni dell'RNA che formano una forte struttura secondaria possono re-ibridare e sopprimere il legame di DNAzyme alla sequenza bersaglio. Per superare questi problemi, cicli di riscaldamento e raffreddamento del 10-23 DNAzyme e del suo substrato di RNA possono essere applicati18.
Abbiamo usato l'approccio DNAzyme per studiare la metilazione 2'-O-metilazione del rRNA. Si può anche usare questa tecnica per analizzare altre modifiche dell'RNA, come N6-methyladenosine19. L'RNA ribosomico, grazie alla sua abbondanza, può essere analizzato mediante elettroforesi e i prodotti di scissione possono essere visualizzati sotto la luce UV. Tuttavia, questo non è applicabile per gli RNA meno abbondanti come gli RNA di codifica generati da RNA Polymerase II (mRNA) e gli RNA non codificanti (ncRNA). Questi RNA di solito non possono essere rilevati direttamente dalla colorazione dell'RNA nei gel di agarose o poliacrilammide. In questi casi, la scissione dipendente dalla DNAzyme può essere visualizzata mediante gonfiore settentrionale, rilevata indirettamente da PCR/PCR quantitativa o analizzata da PCR quantitativa con polimerasi (ad esempio, KlenTaq DNA Polymerase) in grado di discriminare RNA non metilato20,21.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Maya Wilson e Aneika Leney per la lettura critica del manoscritto. Questo lavoro è stato sostenuto da una Sir Henry Dale Fellowship finanziata congiuntamente dalla Wellcome Trust e dalla Royal Society (200473 / 16 / . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Acid phenol | SIGMA | P4682 | |
| Agarose | VWR | A2114 | |
| Ammonium acetate | SIGMA | A1542 | |
| Chlorophorm | Fisher scientific | 10293850 | |
| DNase/RNase free water | Fischer Scientific | 10526945 | |
| DNAzyme | Integrated DNA Technology | Custom oligo DNA | |
| EDTA | SIGMA | E9884 | |
| Ethanol Absolute | Fisher scientific | 10437341 | |
| Formaldehyde | Sigma | F8775 | |
| Formamide | sigma | F9037 | |
| Galactose | SIGMA | G0750 | |
| Gel Loading Dye | Thermo Fisher Scientific | R0611 | |
| Glucose | SIGMA | G7021 | |
| Glycogen | Thermo Fisher Scientific | R0561 | |
| HEPES | SIGMA | H3375 | |
| Isoamyl | SIGMA | W205702 | |
| KCl | SIGMA | P9333 | |
| MgCl2 | SIGMA | M8266 | |
| MnCl2 | SIGMA | 244589 | |
| MOPS | SIGMA | M1254 | |
| NaCl | SIGMA | S7653 | |
| Oxoid Peptone Bacteriological | Thermo Fisher Scientific | LP0037 | |
| Oxoid Yeast Extract Powder | Thermo Fisher Scientific | LP0021 | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher Scientific | EO0382 | |
| SDS | SIGMA | 74255 | |
| Sodium acetate trihydrate | SIGMA | S8625 | |
| SYBR Safe DNA Gel Stain | Thermo Fisher Scientific | S33102 | |
| Tris base | SIGMA | TRIS-RO | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 1.5 mL microtubes | Sarstedt | ||
| 152VR5C01M -80°C freezer | Thermo Fisher Scientific | ||
| 250 mL Erlenmeyer flasks | Cole-Parmer | ||
| 50 mL conical tubes | Sarstedt | ||
| Combicup VX200 vortex | Appleton Woods | ||
| DS-11 microspectrophotometer | Denovix | ||
| Electrophoresis chamber (20 cm tray) | SIGMA | ||
| FiveEasy F20 pH meter | Appleton Woods | ||
| Gel documentation system | Syngene | ||
| Heraeus Fresco 21 micro centrifuge | Fisher Scientific | ||
| Megafuge 8R centrifuge with rotator suitable for 50 mL conical tubes | Fisher Scientific | ||
| Mini Fuge Plus mini centrifuge | Starlab | ||
| Mixer HC thermal block | Starlab | ||
| OLS26 Shaking Water Bath | Grant | ||
| PowerPac power supplier | BioRad |
Riferimenti
- Dieci, G., Preti, M., Montanini, B. Eukaryotic snoRNAs: a paradigm for gene expression flexibility. Genomics. 94 (2), 83-88 (2009).
- Watkins, N. J., Bohnsack, M. T. The box C/D and H/ACA snoRNPs: key players in the modification, processing and the dynamic folding of ribosomal RNA. Wiley Interdisciplinary Review RNA. 3 (3), 397-414 (2012).
- Kufel, J., Grzechnik, P. Small Nucleolar RNAs Tell a Different Tale. Trends in Genetics. , (2018).
- Li, T., Zhou, X., Wang, X., Zhu, D., Zhang, Y. Identification and characterization of human snoRNA core promoters. Genomics. 96 (1), 50-56 (2010).
- Jorjani, H., et al. An updated human snoRNAome. Nucleic Acids Research. 44 (11), 5068-5082 (2016).
- Hubbard, T. J., et al. Ensembl 2009. Nucleic Acids Research. 37 (Database issue), D690-D697 (2009).
- Makarova, J. A., Kramerov, D. A. SNOntology: Myriads of novel snoRNAs or just a mirage?. BMC Genomics. 12, 543 (2011).
- Yu, Y. T., Shu, M. D., Steitz, J. A. A new method for detecting sites of 2'-O-methylation in RNA molecules. RNA. 3 (3), 324-331 (1997).
- Decatur, W. A., Liang, X. H., Piekna-Przybylska, D., Fournier, M. J. Identifying effects of snoRNA-guided modifications on the synthesis and function of the yeast ribosome. Methods in Enzymology. 425, 283-316 (2007).
- Dong, Z. W., et al. RTL-P: a sensitive approach for detecting sites of 2'-O-methylation in RNA molecules. Nucleic Acids Research. 40 (20), e157 (2012).
- Birkedal, U., et al. Profiling of ribose methylations in RNA by high-throughput sequencing. Angewandte Chemie International Edition, England. 54 (2), 451-455 (2015).
- Buchhaupt, M., Peifer, C., Entian, K. D. Analysis of 2'-O-methylated nucleosides and pseudouridines in ribosomal RNAs using DNAzymes. Analytical Biochemistry. 361 (1), 102-108 (2007).
- Grzechnik, P., et al. Nuclear fate of yeast snoRNA is determined by co-transcriptional Rnt1 cleavage. Nature Communication. 9 (1), 1783 (2018).
- Piekna-Przybylska, D., Decatur, W. A., Fournier, M. J. New bioinformatic tools for analysis of nucleotide modifications in eukaryotic rRNA. RNA. 13 (3), 305-312 (2007).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18 (10), 3091-3092 (1990).
- Maden, B. E. Mapping 2'-O-methyl groups in ribosomal RNA. Methods. 25 (3), 374-382 (2001).
- Santoro, S. W., Joyce, G. F. A general purpose RNA-cleaving DNA enzyme. Proceedings of the National Academy of Science, U. S. A. 94 (9), 4262-4266 (1997).
- Hengesbach, M., Meusburger, M., Lyko, F., Helm, M. Use of DNAzymes for site-specific analysis of ribonucleotide modifications. RNA. 14 (1), 180-187 (2008).
- Sednev, M. V., et al. N(6) -Methyladenosine-Sensitive RNA-Cleaving Deoxyribozymes. Angewandte Chemie International Edition, England. 57 (6), 15117-15121 (2018).
- Aschenbrenner, J., Marx, A. Direct and site-specific quantification of RNA 2'-O-methylation by PCR with an engineered DNA polymerase. Nucleic Acids Research. 44 (8), 3495-3502 (2016).
- Lee, K. W., Bogenhagen, D. F. Assignment of 2'-O-methyltransferases to modification sites on the mammalian mitochondrial large subunit 16 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (36), 24936-24942 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon