Method Article
RRNA 2'-O-메틸화의 DNAzyme 의존분석
요약
여기에서 우리는 RNA의 DNAzyme 의존적인 절단을 위한 프로토콜을 제시합니다. 이를 통해 RNA 2'-O-메틸화의 신속하고 현장 의존적인 분석을 가능하게 합니다. 이러한 접근법은 스노RNA 활성의 예비 또는 주요 평가를 위해 사용될 수 있다.
초록
가이드 박스 C/D 작은 뉴클레올라 RNA (snoRNAs) 촉매 2'-O-메틸화 리보좀 및 작은 핵 RNA. 그러나, 더 높은 진핵생물에 있는 snoRNA의 다수는 무차별적으로 그밖 RNA 종 및 2'-O-메틸레이트 다중 표적을 인식할 수 있습니다. 여기서, 우리는 DNAzymes에게 불린 짧은 DNA 올리고뉴클레오티드를 채택하는 잘 확립된 방법을 사용하여 사이트 특이적인 2'-O-메틸화의 빠르고 비고가 있는 분석을 위한 단계별 가이드를 제공합니다. 이 DNA 단편은 특정 합의 위치에서 RNA를 갈라지는 촉매 서열, 뿐만 아니라 그것의 RNA 표적에 DNAzyme를 지시하는 가변 상동성 무기를 포함합니다. DNAzyme 활성은 RNA에서 절단 부위에 인접한 뉴클레오티드의 2-O-메틸화에 의해 억제된다. 따라서, DNAzymes, 절단 된 서열의 합의에 의해서만 제한, 스노RNA 매개 RNA 2'-O-메틸화의 빠른 분석을위한 완벽한 도구입니다. 우리는 saccharomyces 세레비시아에서 25S 리보소말 RNA의 snoRNA snR13- 및 snR47 유도 2'-O-메틸화를 분석하여 기술의 단순성을 입증하고 DNAzyme 의존적 분석법에 대한 상세한 프로토콜을 제공했습니다.
서문
RNA 수정은 유전자 발현의 조절에 중요한 역할을 한다. 각각 상자 C/D 및 상자 H/ACA 작은 뉴클레오르 RNA (snoRNAs)에 의해 유도되는 RNA 2'-O-메틸화 및 슈두리딜레이션은 RNA를 분해로부터 보호하고 고차 구조를 안정화시다1,2,3 . SnoRNA 표적은 리보소좀 RNA (rRNA) 및 작은 핵 RNA (snRNAs)에서 주로 확인되었습니다. 그러나, 더 높은 진핵생물에서는, 할당된 기능이 없는 snoRNA의 잠재적으로 수백이 있고 그들 중 일부는 다중 RNA1,4,5,6및7을인식할 수 있습니다 . 따라서, snoRNA 유도한 수정의 확인 그리고 분석을 허용하는 방법은 세포 프로세스를 지배하는 기계장치를 폭로하는 중요한 공구입니다.
상자 C/D snoRNA 유도 putative 2'-O-메틸화 사이트는 RNase H 지시절단, 또는 역전사를 채택하는 사이트 특정 및 게놈 넓은 방법을 포함하여 많은 기술에 의해 생물학적으로 확인되고 실험적으로 확인될 수 있습니다 낮은 뉴클레오티드 (dNTPs) 농도 접근8,9,10,11. 이러한 기술은 매우 민감하지만 힘들고 비용이 많이 들기 때문에 초기 또는 빠른 테스트에는 적합하지 않을 수 있습니다. 2'-O-메틸화 부위를 식별하는 가장 간단하고 저렴한 방법 중 하나는 DNAzyme 의존성 RNA절단(12)이다. DNAzymes는 특정 위치에서 RNA의 내핵성 절단을 할 수 있는 짧은, 단 하나 좌초 및 촉매 활성 DNA 분자입니다. 이들은 보존되고 촉매활성 코어 서열과 5' 및 3' 결합 암으로 구성되며, 이는 RNA 표적에 대한 왓슨 크릭 염기 페어링에 의해 혼성화되도록 설계된 가변 서열로 구성된다(도1). 따라서, 5' 및 3' 암은 특정 RNA 부위에 촉매 서열을 전달한다. DNAzyme 의존성 절단은 절단부위(12,13)의상류에 위치된 뉴클레오티드의 2'-O-메틸화에 의해 억제된다. 이것은 DNAzymes 가설 또는 알려진 RNA 2'-O-메틸화 사이트의 분석을 위한 아주 실용적인 공구를 만듭니다.
DNAzymes의 2가지 의 모형은 RNA 수정 분석12를위해 사용됩니다. 10-23 DNAzyme(그림 1A)의활성 서열은 표적 RNA 푸린 피리미딘 (RY) 디뉴클레오티드 주위에 루프를 형성하고 이 두 뉴클레오티드 사이의 절단을 촉매하는 15 개의 뉴클레오티드 (5'RGGCTAGCTACACACGA3')로 구성됩니다. RNA 퓨린(R)은 DNAzyme와 염기-페어링되지 않으며, DNAzyme상의 2'-O-메틸화 는 분열을 억제한다. 10-23 DNAzymes의 결합 무기는 일반적으로 10-15 뉴클레오티드 길이입니다. 제2 DNAzyme 클래스, 8-17 DNAzymes(도 1B)14-뉴클레오티드 촉매 서열(5'TCCGAGCCACGA3')을 함유한다. 뉴클레오티드C2,C3 및G4 쌍과 C9 G10 및 G11은 짧은 줄기 루프 구조를 형성한다. 8-17 DNAzymes는 DNAzyme 활성 서열로부터의 첫 번째 티민과 불완전하게 짝을 이루는 모든 구아닌의 상류에 RNA를 갈라줍니다. 구아닌의 상류에 있는 RNA 뉴클레오티드는 DNAzyme와 염기 결합되지 않으며 그것의 2'-O-메틸화는 분열을 손상시다. 8-17 DNAzymes는 DNAzyme을 특정 서열로 유도하기 위해 약 20 개의 뉴클레오티드의 더 긴 상동성 무기를 필요로합니다.
여기서, 우리는 10-23 및 8-17 DNAzyme 의존적 접근법을 사용하여 사카로미세세세리에서 rRNA의 2'-O-메틸화의 분석을 위한 단계별프로토콜을 제공한다(도13(도 1C). 이 프로토콜은 다른 유기체 및 RNA 종에 쉽게 적응할 수 있으며 사이트 별 RNA 2'-O-메틸화의 빠른, 예비 또는 주요 분석을 위해 채택될 수 있습니다.
프로토콜
1. 균주, 미디어 및 버퍼 레시피
- 여기에 자세히 설명된 효모(S. cerevisiae)미디어를 준비하십시오 : YP (효모 추출물 1 % / v, 2 % w / v 세균 펩톤), 포도당 및 갈락토제 주식 20 % w / v.
- 여기에 자세히 설명된 바와 같이 아세테이트 나트륨(NaAc)-EDTA(AE) 완충재를 준비합니다: 50 mM NaAc pH 5.3 및 10 mM EDTA.
- 여기에 자세히 설명된 대로 10-23 DNAzyme 4x 배양 버퍼를 준비하십시오: 24 mM Tris pH 8.0, 60 mM NaCl 및 10-23 DNAzyme 4x 반응 버퍼: 200 mM Tris pH 8.0 및 600 mM NaCl.
- 여기에 자세히 설명 된 대로 8-17 DNAzyme 2x 반응 버퍼를 준비하십시오 : 200 mM KCl, 800 mM NaCl, 100 mM HEPES pH 7.0, 15 mM MgCl2,및 15 mM MnCl2.
- 여기에 자세히 설명 된 대로 10x MOPS 버퍼를 준비하십시오 : 200 mM MOPS, 50 mM NaAc, 1 mM EDTA; pH 7.0 및 1.5x 샘플 변성 버퍼: 50% v/v 포르마미드, 20% v/v 포름알데히드, 1.5x MOPS 버퍼.
- 취득 S. 세레비시아균주, BY4741(MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0); GAL1::SNR13 (BY4741로 하지만 GAL1::SNR13:KANmX); GAL1::SNR47 (BY4741로 하지만 GAL1::SNR47:HIS3mX). 다른 효모 변형은 이 해석에 사용할 수 있습니다.
2. DNAzyme 디자인
- 적절한 데이터베이스를 사용하여 관심 있는 RNA 서열 또는 메틸화 부위를 찾습니다. S. 세레비시아 스노RNA 표적의 경우 효모 스노RNA 데이터베이스를 사용하십시오: http://people.biochem.umass.edu/fournierlab/snornadb/mastertable.php14
- 관심 있는 메틸화 부위를 찾으려면, 예를 들어, snR13 의존 부위, "snR13"을 선택하고 변형된 뉴클레오티드의 위치를 기록한다(예를 들어, snR13-유도 A2281 에서 25S rRNA).
- 적절한 데이터베이스를 사용하여 변형된 뉴클레오티드의 상류 및 하류 서열을 찾습니다. S. 세레비시아에 대 한, 사카로 마이세스 게놈 데이터베이스를 사용 하 여: https://www.yeastgenome.org/
- 표적 유전자 이름 예를 들어, RDN25(코딩 25S rRNA)를 검색한다.
- "서열" 탭에서, 8-17 DNAzyme에 대한 메틸화 부위의 10-23 DNAzyme 분석 및 20 개의 뉴클레오티드 상류(5' 암) 및 다운스트림(3'arm)을 사용할 때 메틸화 부위의 상류(5' 암) 및 하류(3'arm)를 선택한다.
- 5' 및 3' 팔의 상호 보완적인 시퀀스를 만듭니다.
- 측면 10-23 또는 8-17 DNAzyme 촉매 시퀀스 5' 및 3' 팔의 상보적 서열.
- 공급 업체로부터 정상적인 DNA 올리고 뉴클레오티드로 DNAzyme을 주문하십시오.
3. S. 세레비시아 성장 조건
참고: S. 세레비시아 BY4741 스트레인 유도체가 사용되었고, 이 때 SNR13 또는 SNR47 스노RNA의 발현은 유도성 GAL1 프로모터로부터 구동된다. 그들의 합성을 유도또는 억제하기 위해, 갈락토오스(GAL1-의존성 전사) 또는 포도당(GAL1-의존적 전사 떨어져 있는)을 함유하는 배지상에서 세포를 성장시다. 대조군으로, 갈락토오스 또는 포도당에서 자란 야생형 균주(BY4741)를 사용한다.
- 적절한 배지 및 조건에서 효모 균주를 성장시다. GAL1::SNR13 및 GAL1:SNR47 균주뿐만 아니라 이원성 야생형 균주들을 분석하려면, 30°C에서 중간 지수상에 2% 포도당(YPD) 또는 갈락토제(YPGal)를 가진 YP 매체의 50 mL에서 세포를 성장시다.
- 1,000 x g에서원심 분리세포, 4°C에서 3분.
- 상급을 버리고 펠릿을 유지하십시오.
- 액체 질소에 세포 펠릿을 동결하고 -80 °C에서 저장합니다.
주의 사항: 액체 질소는 심한 극저온 화상을 일으킬 수 있습니다. 항상 보호복을 착용하고 안전 예방 조치를 취해야 합니다.
참고: 세포 펠릿은 -80°C에서 최대 1개월까지 보관할 수 있다. 필요한 경우 프로토콜을 일시 중지할 수 있습니다.
4. RNA 격리15
참고: RNA를 분리하는 가장 적절한 방법을 사용하십시오. 효모 S. 세레비시아에대해 핫 페놀 RNA 추출을 사용할 수 있습니다.
- 얼음 차가운 물 1 mL을 추가하고 펠릿을 다시 일시 중단하고 다시 일시 중단 된 세포를 1.5 mL 마이크로 튜브로 옮김하십시오.
- 20,000 x g에서 4°C에서 10s에 원심분리기를 제거하고 상급체를 제거한다.
- AE 버퍼 400 μL을 추가하고 셀을 다시 일시 중단합니다.
참고: 단계 4.4-4.15는 달리 명시되지 않는 한 실온에서 수행됩니다. - 40 μL의 10% SDS와 400 μL의 산 페놀 (pH 4.5)을 추가합니다.
주의 사항: 페놀은 독성이 있으며 연기 후드에서 처리해야합니다. 페놀로 작업할 때는 항상 실험실 코트, 보호 장갑 및 안경을 착용하십시오. 기관 규정에 따라 폐기물을 처리합니다. - 20초 동안 소용돌이를 잘 섞으세요.
- 65 °C에서 10 분 동안 배양하십시오. 2분마다 튜브를 부드럽게 열고 닫아 압력을 풀어내고 튜브를 2-3회 뒤집어 위상을 혼합합니다.
- 튜브를 -80°C로 옮기고 10분 동안 배양합니다.
- 실온에서 5 분 동안 20,000 x g에서벤치와 원심 분리기에 튜브를 해동하십시오.
- 상부를 400 μL 산 페놀을 함유한 새로운 튜브로 옮김:클로로포름:이소아밀 알코올(25:24:1). 상호 단계를 방해하지 마십시오.
주의 사항: 클로로포름은 독성이 있으며 연기 후드에서 처리해야 합니다. 클로로폼으로 작업할 때는 항상 실험실 코트, 보호 장갑 및 안경을 착용하십시오. 기관 규정에 따라 폐기물을 처리합니다. - 실온에서 10 분 동안 20,000 x g에서 30 초및 원심 분리기를 소용돌이치면서 잘 섞습니다.
- 상부 상(~400 μL)을 400 μL 클로로포름을 함유하는 새로운 튜브로 옮니다.
- 실온에서 5분 동안 20,000 x g에서 30초와 원심분리기를 휘저음으로써 잘 섞으세요.
- 상층부(~300-350 μL)를 EtOH 1 mL 및 7.5 m 암모늄 아세테이트(NH4AC)의 40 μL을 포함하는 새로운 튜브로 옮니다. 튜브를 몇 번 뒤집어 섞으세요.
- -80°C에서 2시간 동안 또는 -20°C에서 하룻밤 동안 배양한다.
참고: 절차는 여기에서 일시 중지할 수 있습니다. - 20,000 x g에서원심 분리기, 4 °C에서 10 분 동안. 작은, 백색 RNA 펠릿은 관의 바닥에 보일 것입니다.
- 펠릿을 방해하지 않도록 파이펫팅하여 EtOH를 제거하십시오.
- 실온에서 5분 동안 20,000 x g에 70% EtOH 및 원심분리기 1mL를 추가합니다.
- 파이펫팅으로 70% EtOH를 제거합니다.
- 15s에 대해 20,000 x g의 원심분리기를 하고 2-20 μL 파이펫으로 나머지 EtOH를 제거합니다.
- 튜브를 벤치에 놓고 5 분 동안 열어 RNA 펠릿을 건조시면됩니다.
참고: RNA 펠릿은 건조할 때 백색에서 투명으로 색상을 변경합니다. - RNase/DNase-freeH2 O의30 μL에서 RNA 펠릿을 다시 중단하고, 얼음 위에 튜브를 즉시 옮기고 미세 스펙트로 광도계에서 RNA 농도를 측정합니다.
- 샘플을 -20°C에서 동결합니다.
참고: RNA는 -20°C에서 최대 1개월, -80°C에서 최대 1년까지 보관할 수 있다. 프로시저는 여기에서 일시 중지하거나 다음 단계로 직접 진행할 수 있습니다.
5. DNAzyme 소화
-
10-23 DNAzyme 소화
- 1.5 mL 튜브에서 5 μg의 RNA, 10-23 DNAzyme의 200 pmol (100 mM 스톡 용액의 2 μL) 및 4 x 10-23 인큐베이션 버퍼의 2.5 μL을 총 부피10 μL의 양으로 결합하여 인큐베이션 혼합을 준비합니다.
- 튜브를 95°C로 설정된 건조한 열 블록으로 옮기고 3분 동안 배양합니다.
- 튜브를 얼음에 즉시 옮기고 5 분 동안 배양하십시오.
- 잠시 아래로 회전하고 다시 얼음에 튜브를 넣어.
- RNase 억제제 (예를 들어, 0.5 μL 리보록 RNase 억제제)의 20 U를 추가합니다.
- 튜브를 25°C로 설정된 마른 열 블록에 넣고 10분 동안 배양합니다.
- 한편, 반응 혼합물을 4x 10-23 반응 완충액 5 μL과 300 mM MgCl2 및 1 μL H2O의 4 μL과 결합하여 1.5 μL 튜브에 반응 혼합물을 37°C로 설정된 건조 블록에 놓는다.
- 인큐베이션 믹스를 37°C세트의 건조 히트 블록으로 옮기고 10 μL의 미리 온난반응 믹스를 첨가한다.
- 1 시간 동안 37 °C에서 반응을 배양합니다.
- 얼음에 튜브를 전송하고 단계 5.3.1로 진행합니다.
-
8-17 DNAzyme 소화
- 총 부피 6 μL의 RNA 5 μg로 1.5 mL 마이크로튜브를 준비합니다.
- 8-17 DNAzyme의 400 pmol (100 mM 재고 중 4 μL)으로 1.5 mL 마이크로 튜브를 준비하십시오. 튜브를 얼음 위에 두십시오.
- 튜브를 95°C에 설정된 건조한 열 블록으로 옮기고 2분 동안 배양합니다.
- 얼음에 RNA 샘플을 이동합니다.
- DNAzyme로 튜브를 5 초 동안 회전시키고 25 °C에서 10 분 동안 배양하십시오.
- 동시에, 2x 8-17 반응 완충제의 10 μL로 1.5 mL 튜브를 준비하고 25°C에서 배양한다.
- DNAzyme와 튜브에 미리 온화 된 2x 반응 버퍼의 10 μL을 추가하여 반응 혼합물을 준비합니다.
- RNA를 사용하여 튜브에 반응 혼합물의 14 μL을 전달하고 RNase 억제제의 20 U를 추가합니다.
- 25°C에서 2시간 동안 반응을 배양한다.
- 얼음에 튜브를 전송하고 RNA 정제를 진행 (단계 5.3.1).
-
RNA 정제
- 350 μL의 물과 400 μL의 클로로포름을 반응 튜브에 넣고 실온에서 5 분 동안 20,000 x g에서 30 s와 원심 분리기를 소용돌이치면서 잘 섞습니다.
- 상부 단계(~300-350 μL)를 EtOH 1 mL, 7.5 M NH4AC의 40 μL 및 1 μL의 글리코겐(10 μg/μL)을 포함하는 새로운 튜브로 옮긴다. 튜브를 몇 번 뒤집어 섞으세요.
- -80°C에서 2시간 동안 또는 -20°C에서 하룻밤 동안 배양한다.
참고: 절차는 여기에서 일시 중지할 수 있습니다. - 4.15에서 4.21까지 단계를 반복합니다.
- RNase/DNase 프리H2O의 10 μL에서 RNA 펠릿을 다시 중단하고 튜브를 얼음 에 즉시 옮김을 옮김을 옮김.
- 샘플을 -20°C에서 동결합니다.
참고: RNA는 -20°C에서 최대 1개월까지, -80°C에서 최대 1년까지 보관할 수 있다. 절차는 여기에서 일시 중지되거나 RNA 전기 동으로 진행할 수 있습니다.
6. RNA 전기 동아
- 전기 동공 장비 (탱크, 트레이, 빗)에 1 % SDS를 뿌리고 15 분 동안 방치하고 ddH2O를 충분히 헹구십시오.
- 127.5 mL의 ddH2O에 아가로즈 1.5 g을 전자레인지에 가열하여 용해시면 됩니다.
- 아가로즈 용액에 15 mL의 10x MOPS와 7.5 mL의 37 % 포름알데히드를 추가하십시오 (총 부피는 150 mL입니다).
주의 사항: 포름알데히드는 독성이 있으며 연기 후드에서 처리해야합니다. 포름알데히드와 함께 작업할 때는 항상 실험실 코트, 보호 장갑 및 안경을 착용하십시오. 기관 규정에 따라 폐기물을 처리합니다. - 아가로즈 용액에 선택의 겔 얼룩의 적당량을 첨가한다(예를 들어, 15 μL의 SYBR Safe DNA 겔 얼룩). 잘 섞어서 아가로즈를 트레이에 붓습니다.
- 즉시 젤에 빗을 삽입합니다.
- 연기 후드 아래에 45 분 동안 둡니다. 빛에 민감한 젤 얼룩을 사용할 때 는 알루미늄 호일로 트레이를 덮습니다.
- 1x MOPS 버퍼의 600mL를 준비합니다.
-
RNA 샘플 준비
- 1.5 mL 튜브에서 소화 및 정제 RNA 샘플 10 μL, 시료 변성 완충제 5 μL 및 6x 로딩 염료0.5 μL을 결합합니다.
주의 사항: 포르마미드는 독성이 있으며 연기 후드에서 처리해야 합니다. 포름아미드로 작업할 때는 항상 실험실 코트, 보호 장갑 및 안경을 착용하십시오. 기관 규정에 따라 폐기물을 처리합니다. - 70 °C에서 5 분 동안 인큐베이션 RNA 샘플을 얼음에 옮기는다. 5 분 동안 배양하십시오.
- 젤에 적재하기 전에 잠시 아래로 돌입니다.
- 1.5 mL 튜브에서 소화 및 정제 RNA 샘플 10 μL, 시료 변성 완충제 5 μL 및 6x 로딩 염료0.5 μL을 결합합니다.
- 젤을 전기 동공 탱크에 넣고 1x MOPS 버퍼로 채웁니다. 각 샘플(15 μL)의 전체 부피를 겔에 로드합니다. 브로모페놀 블루가 젤 길이의 2/3에 도달할 때까지 80V에서 실행합니다.
- 선택한 겔 얼룩(예를 들어, UV 트랜스칠미네이터)을 검출하기에 적합한 이미저를 사용하여 겔을 이미지화한다.
결과
rRNA 변형의 분석에 DNAzyme 의존적 절단의 유용성은 snoRNAs성숙도 13의맥락에서 최근에 나타났다. DNAzyme 의존성 분석은 5'-말단 pre-snoRNA 처리의 부족이 S. cerevisiae13에서25S 및 18S rRNA의 2'-O-메틸화 수준에 영향을 미친다는 것을 보여주기 위하여 사용되었다.
여기에서, 우리는 기술의 효과 그리고 단순성을 설명하기 위하여 유도성 snoRNA 전사 시스템을 이용했습니다. 박스 C/D snR13은 아데닌 2281(도2A)을포함하여 25S rRNA에서 두 위치에서 메틸화를 유도한다. 이 뉴클레오티드는 우라실에 이어, 이는 10-23 DNAzyme에 의해 절제가능한 컨센서스 디뉴클레오티드(RY)를 구성한다. 박스 C/D snR47은 또한 25S rRNA에 있는 2개의 뉴클레오티드의 메틸화를 안내합니다(그림 2B). 위치(2220)의 아데닌은 구아닌 잔류물이 뒤따르고 이 디뉴클레오티드는 8-17 DNAzyme에 의해 갈라질 수 있다. snR13 또는 snR47 snoRNA의 합성을 유도하거나 억제하기 위해, 우리는 SNR13 또는 SNR47 유전자의 유도성 GAL1 프로모터를 상류에 삽입하고 갈락토오스를 함유하는 배지에서 배양된세포(GAL1-dependent 전사) 또는 포도당(GAL1-의존적 전사 떨어져 있음). 다음으로, GAL1::SNR13 세포로부터 분리된 RNA는 뉴클레오티드 2281과 2282(도2C)사이에 snR13 의존 부위에서 25S rRNA를 갈라도록 설계된 10-23 DNAzyme로 배양하였다. GAL1::SNR47 균주로부터의 RNA는 뉴클레오티드 2220 및 2221 사이에 8-17 DNAzyme 표적화 snR47 의존부위로 처리하였다(도2D). 대조군으로서, 야생형 BY4741 균주로부터의 RNA는 갈락토오스 또는 포도당에서 자라는 둘 다 DNAzymes로 배양하였다. DNAzyme 처리 RNA의 전기 영동은 GAL1::SNR13 및 GAL1::SNR47 균주에서 자라는 25S rRNA가 손상되지 않은 상태로 유지된 것으로 나타났습니다(그림3A,B; 차선 3). 대조적으로, RNA는 GAL1:SNR13 및 GAL1::SNR47 세포에서 포도당(GLC)에 성장하여 각각의 DNAzymes에 의해 소화되었다(도3A, B;레인 4). 두 경우 모두, 25S rRNA 대역이 감소하고 5' 및 3' 절단 절단 제품(A 및 B)이 관찰되었다. 이는 GAL1:SNR13 및 GAL1:SNR47 균주에서, 25S rRNA가 2'-O-메틸화된 snR13- 또는 snR47 유도 부위에서 갈락토오스가 탄소 공급원으로 사용되었고 이들 스노RNA가 발현되었다는 것을 나타낸다. snR13 또는 snR47 발현시 25S rRNA 메틸화의 부족은 DNAzyme 의존성 절단을 허용한 포도당상에서 차단되었다. 야생형 시료에 대해 RNA 소화가 관찰되지않았다(그림 3A,B; 레인 1 및 2), snR13 및 snR47의 발현이 이 균주에서 갈락토제/포도당 독립적이기 때문이다. 따라서, rRNA는 일반적으로 메틸화되었고 DNAzymes 활성에 저항하였다.
전반적으로, 우리의 실험은 10-23(그림 3A)및 8-17(그림 3B)DNAzymes 상자 C/D snR13 또는 snR47의 부재와 상관관계가 있는 절단 활동이 25S rRNA에 대한 책임이 있음을 명확하게 나타낸다는 것을 보여줍니다. 특정 사이트에서 2'-O-메틸화.
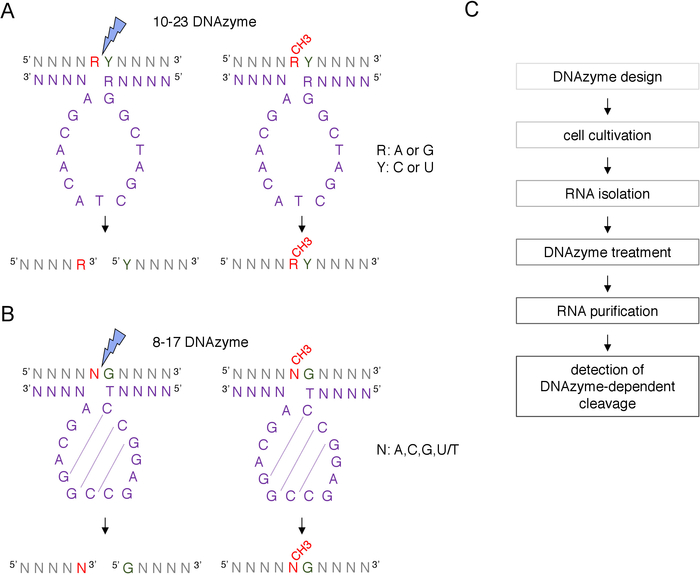
그림 1: DNAzymes 및 그들의 RNA 기판. (A)10-23 DNAzymes는 푸린 피리미딘 (RY) RNA 디뉴클레오티드를 갈라. RNA에서의 R은 DNAzyme와 짝을 이루지 않으며, Y는 DNAzyme의 R 염기와 상보적이다. RNA에서 퓨린 (R)의 메틸화는 DNAzyme 의존적 절단을 억제합니다. (B)8-17 DNAzymes는 DNAzyme 촉매 서열의 첫 번째 티민과 불완전하게 쌍을 이루는 구아닌의 상류에 RNA를 갈라줍니다. 선행 뉴클레오티드는 페어링되지 않으며 메틸화는 DNAzyme 의존적 분열로부터 보호합니다. RNA는 회색으로 도시된다 (메틸화 부위 제외), DNAzyme는 보라색으로 도시된다. N= 임의의 뉴클레오티드, R=푸린: 아데닌 또는 구아닌, Y=피리미딘: 시토신 또는 우라실; CH3-RNA 메틸화를 나타낸다. DNAzyme 활성 시퀀스 내의 기본 페어링은 점선으로 표시됩니다. 파란색 번개 볼트는 분열 부위를 표시합니다. (C)DNAzyme 종속 분석의 단계를 보여주는 순서도입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

도 2: 25S rRNA에서 snR13 및 snR47 의존성 메틸화 부위를 표적으로 하는 DNAzymes. (A, B) snR13 의존성(A)및 snR47 의존성(B)25S rRNA(C)25S rRNA 서열을 둘러싼 snR13 의존메틸화 부위 (A2281)및 10-23 DNAzyme (보라색으로 표시) A2281과 U2282사이의 RNA를 갈라서 설계 . 2281은 DNAzyme와 쌍을 이루지 못하고 U2282는 DNAzyme 활성 서열로부터의 제1 뉴클레오티드와 쌍을 형성한다(파란색 선으로 표시됨). 파란색 번개 볼트는 분열을 표시합니다. (D)snR47 의존성 메틸화 부위를 둘러싼 25S rRNA 서열(A2220)및 8-17 DNAzyme (보라색으로 표시됨) A2220과 G2221사이의 RNA를 절단하도록 설계되었다. 2220은 DNAzyme와 혼성화되지 않으며 G2221은 티민과 불완전하게 쌍을 이룹니다(파선으로 표시됨). 파란색 번개 볼트는 분열 부위를 표시합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

도 3: 10-23 및 8-17 DNAzyme 의존적 분석을 사용하여 25S rRNA의 부위 별 2'-O-메틸화의 분석. (A)10-23 DNAzyme를 사용하여 snR13 의존25S rRNA 메틸화의 분석. (B)8-17 DNAzyme를 사용하여 snR47 의존25S rRNA 메틸화의 분석. RNA는 변성아가로즈 겔에서 염색을 가시화시켰다. 분열 제품 A와 B는 빨간색 화살표로 표시됩니다. WT = 야생형 변형률; GAL = 갈락토오스, GLC = 포도당. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
DNAzyme 의존성 소화는 사이트 별 RNA 2'-O-메틸화12,13을분석하는 간단하고 빠른 방법으로 사용될 수 있다. DNAzymes 는 분열 부위의 상류에 뉴클레오티드가 메틸화되지 않은 경우 RNA를 절단한다. RNase H 지시 소화를 포함 하 여 다른 접근, 알칼리 분해 또는 낮은 뉴클레오티드 농도에서 역 전사 정량 PCR 또는 시퀀싱8,10,11 ,16, DNAzyme 접근법은 임의의 분자 생물학 실험실에 존재하는 간단한 DNA 올리고뉴클레오티드 및 기본 시약을 필요로 한다. 더욱이, DNAzymes는 상자 H/ACA snoRNA12에의해 매개된 RNA pseudouridylation를 분석하기 위하여 유사한 방법으로 이용될 수 있습니다, 이는 그(것)들을 snoRNA 표적을 공부에 있는 다재다능한 공구를 만듭니다.
DNAzyme 의존적 접근법은 분열 부위 합의 서열17에의해서만 제한된다. 10-23 DNAzymes는 RY 디뉴클레오티드의 위치 R에서만 2'-O-메틸화를 분석하는 데 사용될 수 있으며, 8-17 DNAzymes는 구아닌의 상류에 위치한 뉴클레오티드의 변형을 인식한다. 그 결과, 디뉴클레오티드 구아닌 아데닌(GA), 아데닌-아데닌(AA), 피리미딘-아데닌(YA) 및 피리미딘-피리미딘(YY)에서 제1 뉴클레오티드의 2'-O-메틸화와 같은 변형은 분석할 수 없다. 더욱이, DNAzyme 의존적절단(12)의 낮은 효율을 고려해야 한다. 일부 DNAzymes는 RNA를 거의 완전히 갈라지지만(그림 3B),많은 DNAzymes는 부분적으로 그들의 표적을 소화합니다(그림 3B). 효율은 절단 부위를 둘러싼 서열에 의존할 수 있다. 예를 들어, 동일한 뉴클레오티드의 스트레치가 있는 RNA 영역은 DNAzyme 활성 서열의 정확한 위치 형성에 영향을 미칠 수 있다. 더욱이, 강한 이차 구조를 형성하는 RNA 영역은 표적 서열에 결합하는 DNAzyme을 재혼화하고 억제할 수 있다. 이러한 문제를 극복하기 위해 10-23 DNAzyme 및 RNA 기판의 가열 및 냉각 주기를18에적용 할 수 있습니다.
우리는 rRNA의 2'-O-메틸화를 조사하기 위하여 DNAzyme 접근을 이용했습니다. 하나는 또한 N 6-메틸라데노신19와같은 다른 RNA 변형을 분석하기 위해 이 기술을 사용할 수 있다. 리보소말 RNA는 풍부하기 때문에 전기 동극으로 분석 할 수 있으며 절단 제품은 자외선 아래에서 시각화 할 수 있습니다. 그러나, 이것은 RNA 폴리머라제 II 생성 코딩 RNA(mRNA) 및 비코딩 RNA(ncRNA)와 같은 덜 풍부한 RNA에 적용되지 않는다. 이 RNA는 일반적으로 아가로스 또는 폴리아크릴아미드 젤에 있는 RNA 염색에 의해 직접 검출될 수 없습니다. 이러한 경우, DNAzyme 의존적 절단은 북부 블로팅에 의해 시각화 될 수있다, PCR / 정량 적 PCR에 의해 간접적으로 검출 또는 중합체와 정적 PCR에 의해 분석 (예를 들어, KlenTaq DNA 폴리머라제) 에서 2′-O-메틸화 RNA를 차별 할 수 unmethylated RNA20,21.
공개
저자는 공개 할 것이 없다.
감사의 말
마야 윌슨과 아네이카 레니가 원고를 비판적으로 읽어 준 것에 대해 감사드립니다. 이 작품은 웰컴 트러스트와 왕립 학회 (200473 / Z / 16 / Z)가 공동으로 후원한 헨리 데일 경 펠로우십에 의해 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Acid phenol | SIGMA | P4682 | |
| Agarose | VWR | A2114 | |
| Ammonium acetate | SIGMA | A1542 | |
| Chlorophorm | Fisher scientific | 10293850 | |
| DNase/RNase free water | Fischer Scientific | 10526945 | |
| DNAzyme | Integrated DNA Technology | Custom oligo DNA | |
| EDTA | SIGMA | E9884 | |
| Ethanol Absolute | Fisher scientific | 10437341 | |
| Formaldehyde | Sigma | F8775 | |
| Formamide | sigma | F9037 | |
| Galactose | SIGMA | G0750 | |
| Gel Loading Dye | Thermo Fisher Scientific | R0611 | |
| Glucose | SIGMA | G7021 | |
| Glycogen | Thermo Fisher Scientific | R0561 | |
| HEPES | SIGMA | H3375 | |
| Isoamyl | SIGMA | W205702 | |
| KCl | SIGMA | P9333 | |
| MgCl2 | SIGMA | M8266 | |
| MnCl2 | SIGMA | 244589 | |
| MOPS | SIGMA | M1254 | |
| NaCl | SIGMA | S7653 | |
| Oxoid Peptone Bacteriological | Thermo Fisher Scientific | LP0037 | |
| Oxoid Yeast Extract Powder | Thermo Fisher Scientific | LP0021 | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher Scientific | EO0382 | |
| SDS | SIGMA | 74255 | |
| Sodium acetate trihydrate | SIGMA | S8625 | |
| SYBR Safe DNA Gel Stain | Thermo Fisher Scientific | S33102 | |
| Tris base | SIGMA | TRIS-RO | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 1.5 mL microtubes | Sarstedt | ||
| 152VR5C01M -80°C freezer | Thermo Fisher Scientific | ||
| 250 mL Erlenmeyer flasks | Cole-Parmer | ||
| 50 mL conical tubes | Sarstedt | ||
| Combicup VX200 vortex | Appleton Woods | ||
| DS-11 microspectrophotometer | Denovix | ||
| Electrophoresis chamber (20 cm tray) | SIGMA | ||
| FiveEasy F20 pH meter | Appleton Woods | ||
| Gel documentation system | Syngene | ||
| Heraeus Fresco 21 micro centrifuge | Fisher Scientific | ||
| Megafuge 8R centrifuge with rotator suitable for 50 mL conical tubes | Fisher Scientific | ||
| Mini Fuge Plus mini centrifuge | Starlab | ||
| Mixer HC thermal block | Starlab | ||
| OLS26 Shaking Water Bath | Grant | ||
| PowerPac power supplier | BioRad |
참고문헌
- Dieci, G., Preti, M., Montanini, B. Eukaryotic snoRNAs: a paradigm for gene expression flexibility. Genomics. 94 (2), 83-88 (2009).
- Watkins, N. J., Bohnsack, M. T. The box C/D and H/ACA snoRNPs: key players in the modification, processing and the dynamic folding of ribosomal RNA. Wiley Interdisciplinary Review RNA. 3 (3), 397-414 (2012).
- Kufel, J., Grzechnik, P. Small Nucleolar RNAs Tell a Different Tale. Trends in Genetics. , (2018).
- Li, T., Zhou, X., Wang, X., Zhu, D., Zhang, Y. Identification and characterization of human snoRNA core promoters. Genomics. 96 (1), 50-56 (2010).
- Jorjani, H., et al. An updated human snoRNAome. Nucleic Acids Research. 44 (11), 5068-5082 (2016).
- Hubbard, T. J., et al. Ensembl 2009. Nucleic Acids Research. 37 (Database issue), D690-D697 (2009).
- Makarova, J. A., Kramerov, D. A. SNOntology: Myriads of novel snoRNAs or just a mirage?. BMC Genomics. 12, 543 (2011).
- Yu, Y. T., Shu, M. D., Steitz, J. A. A new method for detecting sites of 2'-O-methylation in RNA molecules. RNA. 3 (3), 324-331 (1997).
- Decatur, W. A., Liang, X. H., Piekna-Przybylska, D., Fournier, M. J. Identifying effects of snoRNA-guided modifications on the synthesis and function of the yeast ribosome. Methods in Enzymology. 425, 283-316 (2007).
- Dong, Z. W., et al. RTL-P: a sensitive approach for detecting sites of 2'-O-methylation in RNA molecules. Nucleic Acids Research. 40 (20), e157 (2012).
- Birkedal, U., et al. Profiling of ribose methylations in RNA by high-throughput sequencing. Angewandte Chemie International Edition, England. 54 (2), 451-455 (2015).
- Buchhaupt, M., Peifer, C., Entian, K. D. Analysis of 2'-O-methylated nucleosides and pseudouridines in ribosomal RNAs using DNAzymes. Analytical Biochemistry. 361 (1), 102-108 (2007).
- Grzechnik, P., et al. Nuclear fate of yeast snoRNA is determined by co-transcriptional Rnt1 cleavage. Nature Communication. 9 (1), 1783 (2018).
- Piekna-Przybylska, D., Decatur, W. A., Fournier, M. J. New bioinformatic tools for analysis of nucleotide modifications in eukaryotic rRNA. RNA. 13 (3), 305-312 (2007).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18 (10), 3091-3092 (1990).
- Maden, B. E. Mapping 2'-O-methyl groups in ribosomal RNA. Methods. 25 (3), 374-382 (2001).
- Santoro, S. W., Joyce, G. F. A general purpose RNA-cleaving DNA enzyme. Proceedings of the National Academy of Science, U. S. A. 94 (9), 4262-4266 (1997).
- Hengesbach, M., Meusburger, M., Lyko, F., Helm, M. Use of DNAzymes for site-specific analysis of ribonucleotide modifications. RNA. 14 (1), 180-187 (2008).
- Sednev, M. V., et al. N(6) -Methyladenosine-Sensitive RNA-Cleaving Deoxyribozymes. Angewandte Chemie International Edition, England. 57 (6), 15117-15121 (2018).
- Aschenbrenner, J., Marx, A. Direct and site-specific quantification of RNA 2'-O-methylation by PCR with an engineered DNA polymerase. Nucleic Acids Research. 44 (8), 3495-3502 (2016).
- Lee, K. W., Bogenhagen, D. F. Assignment of 2'-O-methyltransferases to modification sites on the mammalian mitochondrial large subunit 16 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (36), 24936-24942 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유