Method Article
使用χCRAC以高时间分辨率监测体内蛋白质-RNA相互作用动力学
Erratum Notice
摘要
cDNA的动力学交联和分析是一种允许以高时间分辨率研究活细胞中蛋白质-RNA相互作用动力学的方法。这里详细描述了该方案,包括酵母细胞的生长、UV交联、收获、蛋白质纯化和下一代测序文库的制备步骤。
摘要
RNA结合蛋白(RBP)与其RNA底物之间的相互作用表现出流动性和复杂性。在其生命周期内,单个RNA可以被许多不同的RBP结合,这些RBP将调节其产生,稳定性,活性和降解。因此,已经做了很多工作来理解这两种分子之间存在的动力学。一个特别重要的突破是"cross-l inink and immunoprecipitation"(CLIP)的出现。该技术允许严格研究哪些RNA与特定的RBP结合。简而言之,将感兴趣的蛋白质在体内与其RNA底物进行紫外交联,在高度严格的条件下纯化,然后将与蛋白质共价交联的RNA转化为cDNA文库并进行测序。自其概念以来,已经开发了许多衍生技术,以使CLIP适合特定的研究领域。然而,众所周知,使用紫外线进行交联效率低下。这导致暴露时间延长,使得RBP-RNA相互作用的时间研究变得不可能。为了克服这个问题,我们最近设计并制造了大大改进的紫外线照射和细胞收集设备。使用这些新工具,我们开发了一种以高时间分辨率对活细胞中的RBP-RNA相互作用进行时间分辨分析的方案:动力学CRoss-link和cDNA的Nalysis(χCRAC)。我们最近使用这种技术来研究酵母RBP在营养应激适应中的作用。本手稿详细介绍了χCRAC方法,并介绍了使用Nrd1 RBP获得的最新结果。
引言
RNA通常依靠RBP来发挥其功能,这引起了人们对理解这些分子之间动力学的极大兴趣。已在多种生物体中鉴定出许多RBP。然而,在体内研究RBP-RNA相互作用一直是出了名的困难。研究这种相互作用的一个重大突破是CLIP1的出现。该方法利用紫外(UV,254 nm)照射诱导RBP与其直接结合的RNA之间的共价键(即零距离交联)。随后,在严格的条件下对感兴趣的RBP进行免疫纯化,以确保仅鉴定与蛋白质共价交联的RNA。结合的RNA然后用RNA酶部分消化,随后转化为cDNA文库进行测序。高纯化严格性非常重要,因为它大大提高了蛋白质和RNA回收率的特异性,通过交联核糖核蛋白(RNP)复合物的SDS-PAGE纯化也进一步增强了特异性。CLIP和相关方法还提供了对蛋白质结合位点的核苷酸分辨率洞察,因为在测序文库的制备过程中,与RNA交联的氨基酸经常终止逆转录酶或导致酶在该位点引入突变1,2,3。
自推出以来,最初的CLIP协议已经产生了各种各样的衍生方法。一个特别重要的突破来自HITS-CLIP(或CLIP-seq)的开发,它将高通量测序与CLIP方法3相结合。此后,所有基于CLIP的方法都采用了这种方法。iCLIP引入了RNase介导的修剪和衔接连接技术的改进,有助于更准确地映射RBP结合位点4。PAR-CLIP 将 4 硫代尿苷/尿嘧啶标记与 365 nm 的交联相结合,使得通过分析 T-C 取代物5 来绘制交联位点成为可能。CRAC、尿素-iCLIP、dCLIP和uvCLAP引入了变性条件和双重亲和纯化步骤,进一步减少了与亲和树脂的背景结合,并进一步提高了蛋白质捕获的特异性2,6,7,8,9。此外,CRAC、uvCLAP 和 dCLIP 引入了用亲和标签标记感兴趣的 RBP,从而克服了产生特异性抗体的需要。
还进行了一些优化以加快 CLIP 方法。最初的CLIP协议利用捕获的RNA的放射性标记,以便在SDS-PAGE之后可视化RBP-RNA复合物。然而,对于没有为此类工作设立的实验室来说,使用放射性可能会有问题。irCLIP包含一个荧光团偶联接头,可通过红外成像10 进行可视化,sCLIP利用捕获的RNA的生物素化,以便通过链霉亲和素偶联的HRP11对其进行可视化。此外,eCLIP完全放弃了RNA标记;相反,仅根据其已知的大小12切除蛋白质。基于链霉亲和素的纯化也已用于加速FAST-iCLIP中的文库制备过程,其中生物素化的3'接头连接到RNA上,并用于在逆转录和环化后实现纯化13。iCLIP协议的其他增强功能也大大增加了库的复杂性4.
最后,对CLIP进行了修改,以便能够从不同的细胞亚区室14,15捕获RBP,使用可光活化核糖核苷5,16,17的脉冲诱导可视化新转录的RNA,捕获甲基化RNA18,19,20,检查RNA-RNA相互作用21,22,并映射3'末端23,24.
尽管基于CLIP的技术在帮助我们理解RBP和RNA之间的相互作用方面做出了巨大贡献,但它一直受到UV交联效率低下的限制。尽管在单层中生长的培养细胞通常相对容易交联,但这在组织或溶液中的细胞中更具挑战性。组织可能需要多轮紫外线照射才能渗透到所需的细胞层,而微生物细胞通常在含有芳香族紫外线吸收化合物的丰富培养基中生长25。事实上,长达 30 分钟的紫外线照射时间已被用于在此类样品的 RBP 与其结合的 RNA 之间产生足够的交联26,27,28。这种延长的紫外线照射会在细胞内诱导应激反应,例如紫外线诱导的DNA损伤,这可能会污染某些应用中的最终数据。
大多数CLIP研究都集中在生成细胞中特定蛋白质 - RNA相互作用的单个"快照"。然而,蛋白质-RNA相互作用本质上是动态的,特别是当细胞受到环境变化的影响时。这可能包括必需营养素的可用性突然减少或温度的快速变化。因此,要真正了解RBP在压力下的作用,最好进行时间分辨分析,因为它们可以捕获压力期间RBP目标的全部范围,并确定所选RBP在压力反应的哪个阶段处于活动状态。特别是,对酵母的研究表明,适应的最初几分钟对生存绝对至关重要,细菌中的RNA半衰期可以从几分钟到几秒钟不等29,30,31,32,33。因此,理想情况下,这种时间分辨分析应该在高时间分辨率下进行。然而,较长的交联时间使得早期适应性反应的研究特别具有挑战性。
为了克服这些问题,我们最近开发了一种改进的方法,能够在长达一分钟的时间尺度上交联和收获细胞。我们的χCRAC方法允许以以前未见过的分辨率定量测量RBP-RNA相互作用的动态变化。该方法的关键是开发了一种新型紫外线照射装置32 ,该装置将酵母和溶液中细菌中所需的交联时间减少了约10倍,有效地瞬间冷冻RBP-RNA相互作用。此外,为了在紫外线照射后快速收获细胞,我们开发了一种真空过滤装置,可以在大约 30 秒32 的 0.5 L 培养物中收获指数生长的酵母。这些技术创新允许以分钟级分辨率研究RBP-RNA动力学。此外,我们还对原始CRAC协议2 进行了一些优化,以提高其实用性。
使用χCRAC,我们最近研究了酵母核RBP,Nab3的靶组,以响应葡萄糖剥夺。在 酿酒酵母中,Nab3可以与Nrd1,RBP和RNA解旋酶Sen1形成复合物,形成NNS复合物。NNS与RNA聚合酶和新生转录本结合可以触发转录终止34。这种复合物主要参与去除神秘的非编码RNA转录本,但也已被证明可以调节蛋白质编码基因的表达。该研究显示,Nab3在仅一分钟的应激后就对非编码和编码转录本的靶向不同32。我们证明了Nab3的共转录终止导致逆转录转座子基因的非常短暂的脉冲样表达,这很难使用传统的基于CLIP的方法检测到。此外,我们的紫外交联剂中较短的紫外照射时间也显著提高了短寿命非编码RNA的回收率32。χCRAC可能是一个关键工具,不仅可以阐明RBP如何在即时时间尺度上塑造对压力的反应,还可以阐明它们在响应的整个生命周期中不断变化的角色。这份手稿详细概述了χCRAC协议中的所有步骤。出于说明目的,该方法用于研究参与转录终止和RNA衰变35,36的酵母Nrd1蛋白及其RNA靶蛋白在多个时间点响应葡萄糖剥夺。最后,我们还证明,我们的紫外线照射单元可以快速将RBP与HeLa细胞中的RNA交联,从而可以在贴壁细胞中进行高分辨率的时间分辨分析。
研究方案
| TN150 |
| 50 毫米三分 pH 7.8 |
| 150毫米氯化钠 |
| 0.1% NP-40 |
| 1X蛋白酶抑制剂 |
| TN1000 |
| 50 毫米三分 pH 7.8 |
| 1M 氯化钠 |
| 0.1% NP-40 |
| NP-PNK |
| 50 mM 三盐酸 pH 7.8 |
| 10 毫米氯化镁2 |
| 0.1% NP-40 |
| 5 mM β-巯基乙醇 |
| 5 x PNK |
| 250 mM 三盐酸 pH 7.8 |
| 50 毫米氯化镁2 |
| 50 mM β-巯基乙醇 |
| 世界银行 I |
| 50 mM 三盐酸 pH 7.8 |
| 300 毫米氯化钠 |
| 10 mM咪唑 |
| 6M盐酸胍 |
| 0.1% NP-40 |
| 5 mM β-巯基乙醇 |
| 世界银行二 |
| 50 mM 三盐酸 pH 7.8 |
| 50毫米氯化钠 |
| 10 mM咪唑 |
| 0.1% NP-40 |
| 5 mM β-巯基乙醇 |
| 洗脱缓冲液 |
| 50 毫米三分 pH 7.8 |
| 50毫米氯化钠 |
| 250 mM咪唑 |
| 0.1% NP-40 |
| 5 mM β-巯基乙醇 |
| 蛋白酶K缓冲液 |
| 50 毫米三重奏 |
| 0.1% NP-40 |
| 5 mM β-巯基乙醇 |
| 1% 安全数据表(SDS) |
| 5 毫米乙二胺四乙酸 |
| 50 毫米氯化钠2 |
| 哺乳动物裂解缓冲液 |
| 50 mM 三盐酸 pH 8 |
| 100 毫米氯化钠 |
| 0.5% v/v 海卫一 X-100 |
| 0.25% w/v 脱氧胆酸钠 |
| 0.1% w/v SDS |
| 5 毫米乙二胺四乙酸 |
| 1 毫米桌面转换器(新鲜添加) |
| 1X蛋白酶抑制剂 |
表 1:χCRAC 所需的缓冲液及其组成。
1. UV交联和裂解液生产
- 溶液中的微生物
- 用过夜培养物中的酵母接种3.5L所需培养基至起始OD600 为0.05。在30°C下生长,以180rpm的速度连续振荡。
- 在生长过程中,准备其他必要的材料。
- 准备一个液氮容器。
- 准备3L应激诱导培养基,并在水浴中加热至30°C。
- 设置过滤装置,打开交联剂(图2A)并标记50 mL锥形管,每个时间点一个。
- 一旦细胞达到所需的OD 600,将500 mL细胞直接倒入交联剂中,并用250 mJ的254 nm UV进行紫外线照射。有关使用交联剂的详细信息,请参见图2A和图3A。
注意:必须针对每种感兴趣的蛋白质仔细优化紫外线照射能量。有关更多详细信息,请参阅讨论。 - 交联后,使用真空过滤装置之一过滤细胞(图2B,C)。用过滤的细胞卷起膜,放入t = 0(时间零)50 mL锥形管中,并在液氮中快速冷冻。
- 通过六个不同的过滤器过滤剩余的单元格。通过将膜放入培养基中并与条带剧烈混合50秒,将收集的细胞重悬于3L先前加热的应激诱导培养基中。50 秒后,准备取 t = 1 样品。
- 1 分钟后,交联 500 mL 细胞并按步骤 1.1.3–1.1.4 过滤收获。在 2、4、8、14 和 20 分钟后重复,或根据需要在不同的时间点重复。
- 将含有细胞的锥形管储存在-80°C。 将磷酸盐缓冲盐水(PBS)在4°C下放置过夜。
- 第二天,取每个含有交联样品的锥形管,并通过剧烈摇动将细胞重悬于25mL冷PBS中。
- 将细胞悬液转移到新的锥形管中,并在4°C下以4,600× g旋转5分钟。
- 倒出PBS,再次快速旋转以收集残留的PBS,然后用移液管倒出剩余的液体。
- 通过将颗粒与空管进行比较来计算管中颗粒的重量。
- 加入两个沉淀体积的冰冷 TN150、60 μL DNase 1 和 10 μL RNase 抑制剂。在冰上孵育30分钟。
- 例如,对于 400 mg 细胞,加入 800 μL 冰冷的 TN150。
- DNase的添加对于大多数可溶性蛋白质不是必需的,但在研究染色质结合蛋白(如RNA聚合酶)时非常重要。此外,它还降低了细菌裂解物的粘度。使用恰好两个沉淀体积的裂解缓冲液非常重要,否则裂解效率会降低。
- 向细胞悬液中加入三个沉淀体积(以mL为单位)的氧化锆珠。对于酵母,使用直径为0.5毫米的珠子,对于细菌,使用0.1毫米。
- 例如,对于 400 mg 细胞,在 1.5 mL 管中测量出 1.2 mL 氧化锆珠,并将它们添加到重悬于裂解缓冲液中的细胞中。
- 涡旋细胞悬液1分钟,然后放在冰上1分钟。总共重复 5 次。
- 加入两个沉淀体积的TN150缓冲液并剧烈涡旋混合。
- 在台式离心机中以4,600g离心锥形管中的悬浮液在4°C下离心20分钟。
- 离心后,取 50 μL 上清液样品进行将来的蛋白质印迹分析,以检查全细胞蛋白表达。
- 将上清液转移到1.5mL管中,并在微量离心机中以20,000× g 在4°C下旋转裂解物20分钟。
- 或者,如果使用 5 mL 管,则以 13,000 x g 离心 20 分钟。
- 离心后,取 50 μL 上清液样品进行未来的蛋白质印迹分析,以检查蛋白质的可溶性表达。
- 继续执行 RBP 捕获(第 2 节)。
- 培养的贴壁细胞
- 在紫外线交联前24小时在培养皿中接种足够的贴壁细胞,以便它们在第二天达到80%汇合。在37°C,5%CO2的细胞培养箱中的所需培养基中生长过夜。
注意:如果使用石英培养皿,则在接种前2.5小时用聚-D-赖氨酸(70,000-140,000重量)和胎牛血清(FCS)处理培养器皿,促进细胞粘附是有益的。加入足够的聚-D-赖氨酸以覆盖整个生长表面,并在室温(RT)下孵育5分钟。接下来,应用水彻底冲洗石英培养皿并在细胞培养箱中干燥2小时或直到完全干燥。之后,加入足够的FCS以完全覆盖生长表面并放置在培养箱中至少30分钟。在接种细胞之前,应完全去除食品接触物质。 - 一旦细胞达到80%汇合,取出培养基并用15mL冰冷的PBS洗涤。接下来,完全去除所有剩余的液体,并立即进行下一步。
- 将培养皿转移到贴壁细胞的托盘上(图3B),并用300mJ的254nm紫外线照射紫外线。有关使用交联剂的详细信息,请参见图2A和图3B。
注意:必须针对每种感兴趣的蛋白质仔细优化紫外线照射能量。有关更多详细信息,请参阅讨论。 - 交联后立即将培养皿放在冰上,加入 10 mL 冰冷的 PBS。通过刮擦收集细胞并转移到 15 mL 锥形管中。通过在4°C下以300× g 离心5分钟沉淀。
- 取出 PBS 并将细胞沉淀重悬于 1 mL 冰冷的 PBS 中,并转移到 1.5 mL 微量离心管中。通过在4°C下以300× g 离心5分钟再次沉淀细胞。
- 取出PBS并在干冰上快速冷冻细胞沉淀。将细胞沉淀储存在-80°C直至需要。
- 对每个时间点重复步骤 1.2.3–1.2.6。
- 将细胞沉淀重悬于 1 mL 裂解缓冲液中,并转移到 15 mL 锥形管中。之后,加入 1 mL 裂解缓冲液,总共 2 mL。
- 加入 5 μL 哺乳动物 RNase 抑制剂。
- 在冰上以 10 安培的电流超声处理 5 次 10 秒。在超声轮之间等待 30 秒。
- 计算每个样品的蛋白质浓度并归一化到最低浓度。
- 将 1.98 mL 裂解物转移到 2 mL 管中。
- 加入 10 μL DNase I,并在 37 °C 下以 1,200 rpm 振荡孵育 5 分钟。
- 将裂解物在4°C下以16,000× g 离心20分钟。
- 离心后,取 50 μL 上清液样品进行将来的蛋白质印迹分析,以检查蛋白质的可溶性表达。
- 继续执行 RBP 捕获(第 2 节)。
- 在紫外线交联前24小时在培养皿中接种足够的贴壁细胞,以便它们在第二天达到80%汇合。在37°C,5%CO2的细胞培养箱中的所需培养基中生长过夜。
2. RBP 捕获
- 用 5 mL TN150 洗涤磁性抗 FLAG(每个样品 75 μL 浆液)或 IgG 琼脂糖(每个样品 500 μL 浆液)珠子 3 倍。重悬于终体积为 700 μL 的 TN150 中,并将 100 μL 洗涤过的珠子加入七个 15 mL 锥形管中。
- 存放在冰上,直到需要为止。
- 裂解物澄清后,将上清液加入含有抗FLAG/IgG磁珠的管中。
- 在4°C下动动2小时。
注意:一些方案描述了与磁珠的过夜孵育,但不建议这样做,因为较长的孵育时间会大大降低交联RNA的回收率。
3.清洗标签的珠子和TEV裂解
- 收获珠子并去除裂解物。
- 取 50 μL 上清液样品进行将来的蛋白质印迹分析,以检查未捕获的蛋白质。
- 将磁珠重悬于冰冷的TN1000中,并转移到1.5 mL管中。洗涤10分钟,4°C,章动。重复总共三次洗涤。
- 如果使用 IgG 琼脂糖珠,请用 5 mL TN1000 洗涤。如果使用抗 FLAG 磁珠,请使用 2 mL。
- 接下来,用TN150洗涤珠子3次,体积与上述相同。
- 第三次洗涤后,将珠子重悬于 600 μL TN150 中。
- 向磁珠悬浮液中加入30U自制GST-TEV蛋白酶,并在室温下旋转2小时。
注意:重组GST-TEV蛋白酶现在也可以市售,但尚未使用此协议进行测试。- 在酶解过程中,为每个样品设置三个 1.5 mL 管的色谱柱(即对于 7 个样品,有三行 7 个色谱柱),为后续步骤做准备。
- 向最后一排试管中加入0.4g盐酸胍,27μL5M氯化钠和3μL2.5M咪唑(pH = 8)。请注意,咪唑的pH值必须为8。这对于保持RNA的完整性至关重要。
- 此外,在WB I 3x中洗涤所需体积的镍珠。每个样品使用 100 μL 浆液。最后一次洗涤后,将珠子重悬于相同原始体积的WB I中并存放在冰上。
- TEV消化完成后,使用用于抗FLAG磁珠的磁性架或离心IgG珠收集上清液,并转移到先前设置的试管的第一排。
- 取 50 μL TEV 洗脱液样品进行蛋白质印迹分析。
- 将热块培养箱设置为37°C。 向第二排试管中加入 1 μL RNase 混合物(1:50 稀释)。
- 从第一排试管中取出 550 μL TEV 洗脱液,并添加到第二排(含有 RNase 混合物)中。用力移液以确保混合。
- 完成第一个样品的此操作后,立即将试管放入热块并启动计时器。转到后续示例,以便每个示例交错进行。
- 孵育正好5分钟。完成后,从热块中取出第一个样品并将溶液转移到第三排管(含有盐酸胍粉末)中。
注意:用1:50稀释的RNase混合物孵育5分钟通常适用于大多数蛋白质,但此步骤需要仔细优化,每种蛋白质有不同的孵育时间或浓度,以确保交联RNA具有正确的大小(30-100 nt)。 - 立即全速涡旋几秒钟以溶解胍粉,然后继续下一个样品。
- 将所有样品转移到锍粉末中后,再次涡旋以确保所有粉末完全溶解。
- 加入100μL洗涤过的镍珠,并在4°C下旋转过夜。 该孵育可以缩短至2小时。
4. 珠上碱性磷酸酶处理
- 将热块设置为37°C。
- 将纯化离心柱置于 2 mL 管中,每个样品一个。将镍珠转移到色谱柱上,让上清液排出。之后,通过用WB I冲洗并涂在色谱柱上,确保从1.5 mL管中除去所有镍珠。
- 设置 2 mL 管,每个样品 6 个(每次洗涤一个收集)。保持色谱柱外部干燥以保持流动。用 500 μL WB I 洗涤磁珠 3 次,然后用 500 μL NP-PNK 洗涤 3 次。
- 关闭离心柱的盖子并短暂旋转珠以除去多余的缓冲液。
- 将塞子放在色谱柱上,将色谱柱放入1.5 mL管中,并加入60 μL反应混合物,如表2所示。
| 元件 | 1 倍 | 7.5 倍 |
| 5 x PNK 缓冲器 | 12 | 90 |
| 碱性磷酸酶 | 4 | 30 |
| 核糖核酸酶抑制剂 | 2 | 15 |
| H2O | 42 | 315 |
| 最终卷 | 60 微升 | 450 微升 |
表2:碱性磷酸酶反应混合物。
- 将珠子在37°C孵育1小时。
- 用 500 μL WB I 洗涤磁珠 1 次以灭活碱性磷酸酶,然后用 500 μL NP-PNK 缓冲液洗涤 3 次。确保用NP-PNK缓冲液彻底冲洗色谱柱内部,以去除任何痕量的胍。
5. 将 App-PE 接头珠子连接到 RNA 的 3' 端
- 离心出剩余的缓冲液,向色谱柱中加入60 μL表3中指定的混合物(有关App-PE序列,请参见表4)。将反应在25°C孵育6小时。
| 元件 | 1 倍 | 7.5 倍 |
| 5 x PNK 缓冲器 | 12 | 90 |
| 应用-PE 适配器 (100 μM) | 0.6 | 4.5 |
| T4 RNA 连接酶 2 截短 K227Q | 3 | 22.5 |
| 核糖核酸酶抑制剂 | 1.5 | 11.25 |
| 50% 聚乙二醇 8000 | 12 | 90 |
| H2O | 30.9 | 231.75 |
| 最终卷 | 60 微升 | 450 微升 |
表3:App-PE接头连接反应混合物。
| 寡核苷酸名称 | 序列 (5'-3') | |||
| L5Aa | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrUrNrNrUrArGrCrN-OH | |||
| L5Ab | invddT-ACACrGrArCrGrCrCrCrUrCrCrGrArUrCrNrNrArUrUrArGrCrN-OH | |||
| L5交流 | invddT-ACACrGrArCrGrCrCrUrCrUrCrCrGrArUrCrNrNrGrCrGrCrGrCrArGrCrN-OH | |||
| L5广告 | invddT-ACACrGrArCrGrCrCrUrCrUrCrGrArUrCrUrNrNrCrGrCrUrUrArGrCrN-OH | |||
| L5Ba | invddT-ACACrGrArCrGrCrCrUrCrUrCrGrArUrCrNrNrArGrGrCrN-OH | |||
| L5Bb | invddT-ACACrGrArCrGrCrCrUrCrUrCrGrArUrCrUrNrNrGrUrGrGrCrN-OH | |||
| L5Bc | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrNrNrCrArCrUrArGrCrN-OH | |||
| L5Bd | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrCrGrArUrCrNrNrUrCrUrCrUrArGrCrN-OH | |||
| L5Ca | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrUrNrNrNrCrUrArGrCrN-OH | |||
| L5Cb | invddT-ACACrGrArCrGrCrCrUrCrUrCrGrArUrCrUrNrNrUrGrGrArCrNrUrGrArGrCrN-OH | |||
| L5Cc | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrCrGrArUrCrNrNrArCrUrCrArGrGrCrN-OH | |||
| L5镉 | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrCrGrArUrCrNrNrGrArCrUrUrArGrCrN-OH | |||
| L5Da | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrNrNrNrCrGrUrGrUrGrArUrN-OH | |||
| L5分贝 | invddT-ACACrGrArCrGrCrGrCrUrCrGrArUrCrNrNrGrCrArCrArCrArN-OH | |||
| L5直流 | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrNrNrUrArGrUrGrCrN-OH | |||
| L5Dd | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrCrGrArUrCrNrNrArUrCrArCrArCrGrN-OH | |||
| L5Ea | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrNrNrNrCrArCrUrGrUrGrUrN-OH | |||
| L5Eb | invddT-ACACrGrArCrGrCrCrCrUrCrCrGrArUrCrNrNrGrUrGrArCrArCrArRNrGrUrGrArCrArN-OH | |||
| L5Ec | invddT-ACACrGrArCrGrCrGrCrUrCrUrCrGrArUrCrNrNrUrGrUrCrArUrCrArCrN-OH | |||
| L5Ed | invddT-ACACrGrArCrGrCrCrUrCrUrCrGrArUrCrNrNrArCrArGrUrGrUrGrN-OH | |||
| App_PE | App-NAGATCGGAAGAGCACACGTCTG-ddC | |||
表4:连接到捕获RNA的5'和3'末端所需的DNA和RNA接头序列。 这些通过无RNase的HPLC纯化。
- 用 500 μL WB I 洗涤 1 次,用 500 μL NP-PNK 缓冲液洗涤 3 次。将色谱柱放入新管中并离心剩余的缓冲液。
6. RNA的5'末端的磁珠磷酸化
- 向色谱柱中加入80 μL表5中指定的混合物。将反应在37°C孵育40分钟。
注意:样品现在将具有高放射性。因此,所有后续工作都应在保护屏风后面进行,废物应根据当地的健康和安全规则进行处理。
| 元件 | 1 倍 | 7.5 倍 |
| 5 x PNK 缓冲器 | 16 | 120 |
| 32P-ɣATP (10 μCi/μL) | 3 | 22.5 |
| T4 PNK | 3 | 22.5 |
| H2O | 58 | 435 |
| 最终卷 | 80 微升 | 600 微升 |
表5:磷酸化反应混合物。
- 加入 1 μL 100 mM ATP,让反应再进行 20 分钟。这将确保几乎所有的5'末端都有磷酸盐,以促进5'接头的连接。
- 设置 2 mL 管,每个样品 5 个。
- 用 500 μL WB I 洗涤 1 次,用 500 μL NP-PNK 缓冲液洗涤 3 次。请注意,这些洗脱物具有很强的放射性,因此应妥善处理。
- 将色谱柱移至最终试管并离心剩余的缓冲液。
7. 5'接头的珠子连接
注意:5'接头包含一个RNA条形码,用于测序后识别每个样品。因此,注意哪个接头用于哪个样品是绝对关键的。
- 向色谱柱中加入78 μL表6中所述的混合物。向每个试管中加入 2 μL 5' 适配器(100 μM;参见表 4),并在 18 °C 下孵育过夜。
| 元件 | 1 倍 | 7.5 倍 |
| 5 x PNK 缓冲器 | 16 | 120 |
| 三胺类兴奋剂 (10 毫米) | 8 | 60 |
| 核糖核酸酶抑制剂 | 2 | 15 |
| T4 核糖核酸连接酶 | 4 | 30 |
| H2O | 48 | 360 |
| 最终卷 | 78 微升 | 585 微升 |
表6:5'接头连接反应混合物。
- 第二天,用 500 μL WB I 洗涤 1 次,用 500 μL WB II 洗涤 3 次,并将色谱柱转移到新的 2 mL 管中。
8. 洗脱、SDS-PAGE 和 RNA 提取
- 将离心机设置为4°C。 每个样品准备两排 1.5 mL 管进行洗脱。
- 用镍珠快速旋转出柱子的空隙体积。将色谱柱置于洗脱管的第一排中,加入 200 μL 洗脱缓冲液。等待2分钟,然后快速旋转强制缓冲液通过色谱柱。
- 将列移动到第二排管上并重复步骤8.2。现在,每个样品总共有 400 μL 洗脱液,分成两个 1.5 mL 管。
- 取所有洗脱液并将它们一起转移到 5 mL 管中。加入 2 μL 的 20 mg/mL 糖原。因此,如果使用七个样品,则5 mL管中现在将有2.8 mL混合洗脱液。
- 向 5 mL 管中加入每个样品 100 μL 三氯乙酸 (TCA) [例如,7 个样品的 700 μL TCA(2.8 mL 混合洗脱液)],并涡旋孔 30 秒。
- 在冰上孵育20分钟。
- 在台式离心机中以17,000× g,4°C离心30分钟。
- 小心地从锥形管中取出上清液,用盖革计数器检查移液器,以确保沉淀没有被意外去除。如果有,将上清液返回到管中并再离心10分钟。
注意:上清液可能仍具有高放射性。确保使用适当的屏蔽。 - 将沉淀完全重悬于 2 mL 冰冷的丙酮中。
- 在17,000× g,4°C下离心15分钟。
- 用P1000移液器尽可能多地去除丙酮。之后,短暂旋转试管以收集小液滴丙酮,然后用 P10 移液管取出。在通风橱中干燥2分钟。
注意:丙酮上清液仍具有放射性。确保使用适当的屏蔽。 - 将样品重悬于 30 μL 1x 蛋白质上样缓冲液中。为确保沉淀正确重悬,通过在P200移液管中取出溶液并使用盖革计数器测量残留在1.5 mL管中的活性,检查绝大多数放射性现在是否存在于上样缓冲液中,而不是留在1.5 mL管中。
- 将样品在65°C下加热10分钟。 在 1 mm、4–12% 预制 Bis-Tris 凝胶上上样,并在 MOPS 缓冲液中以 125 V 运行 1.5 小时。
- 凝胶电泳完成后,打开凝胶盒。凝胶应保留在底板上。处理掉顶部。
- 用保鲜膜包裹凝胶,然后用胶带将其固定在光密盒的内部。确保磁带具有放大屏幕以改善信号。
- 将放射自显影胶片暴露在凝胶中,并在曝光期间将暗盒储存在-80°C。具有不同交联效率的蛋白质之间的暴露时间会有所不同。
- 放置胶片时,必须有一种方法将其重新对齐到盒式磁带上,以便在后续步骤中剪掉感兴趣的带。为确保这一点,请使用荧光尺,并确保凝胶位于盒的一角,然后将其覆盖,也放置在最角落的薄膜。
注意:根据经验,洗脱液在上样缓冲液中洗脱,当显示到盖革计数器时,读数至少为~250 cps,可提供足够的信号进行3小时的曝光。否则,进行过夜暴露。
- 放置胶片时,必须有一种方法将其重新对齐到盒式磁带上,以便在后续步骤中剪掉感兴趣的带。为确保这一点,请使用荧光尺,并确保凝胶位于盒的一角,然后将其覆盖,也放置在最角落的薄膜。
- 开发电影。切掉覆盖凝胶的保鲜膜,但不要移动凝胶。否则,图像将从凝胶上偏移。
注意:凝胶可能具有高放射性。切出凝胶片时,请确保使用适当的屏蔽。 - 将薄膜放在凝胶上并切除感兴趣的条带。将凝胶切片放入 2 mL 管中。
- 使用 P1000 移液器吸头粉碎凝胶切片,并加入 600 μL 蛋白酶 K 缓冲液和 200 μg 蛋白酶 K(本协议使用 10 μL 的 20 mg/mL 蛋白酶 K 溶液)。在55°C下剧烈摇动孵育2小时。
- 之后,用干净的手术刀切下 P1000 尖端的末端,并将上清液和凝胶片转移到放置在 2 mL 管中的离心柱中。
- 在室温下以17,000× g 旋转色谱柱1分钟。 收集含有放射性分离RNA的流通物。
- 进行苯酚:氯仿萃取。
- 加入 50 μL 3 M 乙酸钠,pH = 5.2,和 500 μL 苯酚:氯仿并涡旋良好。以 17,000 x g 旋转 5 分钟。去除表层水溶液,放入新的 1.5 mL 试管中。
- 加入 500 μL 氯仿并剧烈涡旋 10–15 秒。在室温下以 17,000 x g 旋转 5 分钟。 去除水层并放入新的 1.5 mL 管中。
- 加入 1 μL 20 mg/mL 糖原和 1 mL 冰冷的 96% 乙醇。在-80°C下沉淀30分钟或在-20°C下沉淀过夜。
- 在4°C,17,000× g下离心30分钟。除去上清液,加入500μL70%乙醇,在17,000× g下离心5分钟,4°C。取出所有乙醇,快速旋转以收集残留物,并用P10移液器去除多余的乙醇。
- 将颗粒在通风橱中干燥~3分钟。重悬于 20 μL 经 DEPC 处理的水中。
- 将RNA在-80°C下储存过夜或立即进行逆转录步骤。
9. 逆转录
- 向 20 μL RNA 中加入 2 μL 10 μM RT 寡核苷酸(PE_reverse;参见 表 7)和 4 μL 5 mM dNTP。
| 寡核苷酸名称 | 序列 (5'-3') | |||
| P5 向前 | AATGATACGGCGACCACCGAGATCTACTACTACTTCCCTACACGCTCTTCCGATCT | |||
| 公元前1 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前3年 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前4年 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前5年 | CAAGCAGAAGACGGCATACGAGATCACTGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前7年 | CAAGCAGAAGACGGCATACGAGATCAGATCGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前8年 | CAAGCAGAAGACGGCATACGAGATTAGCTTGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前9年 | CAAGCAGAAGACGGCATACGAGATGATCAGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| 公元前10年 | CAAGCAGAAGACGGCATACGAGATATCACGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| PE_reverse | CAGACGTGTGCTCTTCCGATCT | |||
表7:PCR引物(包括条形码序列)和逆转录引物。
- 转移到85°C的预热热块中3分钟,然后在冰上快速冷却5分钟。通过短暂离心收集管中的内容物,然后加入 8 μL 5x 逆转录酶缓冲液、2 μL 100 mM DTT 和 2 μL RNase 抑制剂。
- 将混合物在50°C孵育3分钟,然后加入2μL逆转录酶并在50°C下孵育1小时。
- 通过在65°C孵育15分钟来灭活逆转录酶。
- 将试管转移到37°C的预热热块中,静置3分钟以适应环境。
- 加入2μLRNase H并在37°C下孵育30分钟。
- 使用 SPRI 磁珠分离 cDNA。
- 加入两体积的 84 μL 磁珠。孵育15分钟。将珠子放在磁性架上,静置1分钟以收获珠子。
- 取出并处理上清液,加入 200 μL 70% 乙醇。请勿从磁性架上取下磁珠。将珠子与乙醇孵育30秒。
- 取出乙醇并重复洗涤步骤。使用 P10 尖端去除所有残留的乙醇。
- 将珠子放入通风橱中2分钟以干燥。从架子上取下磁珠,将其重悬于12 μL水中,然后将珠子放回架子上。除去 11 μL 上清液。
- 将cDNA冷冻在-20°C或立即进行PCR步骤。
10. qPCR反应
- 在对cDNA进行最终PCR扩增之前,进行定量聚合酶链反应(qPCR)以确定扩增cDNA的最佳循环次数,以防止文库过度扩增。
- 根据表8在冰上建立qPCR反应。所有引物见表7。
| 元件 | 1 倍 |
| 2x qPCR 反应预混液 | 5 |
| 0.1 μM P5 引物(正向) | 0.8 |
| 0.1 μM BC 引物(反向) | 0.8 |
| cDNA(或水作为阴性对照) | 1 |
| H2O | 2.4 |
| 最终卷 | 10 微升 |
表8:qPCR反应混合物。
- 为了正确定量扩增所需的循环,请使用cDNA的三个技术重复和三个阴性(即水)对照。
- 用光学透明薄膜密封板,并根据试剂盒制造商的说明运行qPCR。
- 通过绝对定量方法分析样品,以确定达到指数增长拐点的周期数(n)(示例见 图4C )。然后将该循环次数用于其余cDNA的最终扩增。
11. PCR反应和凝胶提取
- 根据表9在冰上设置PCR反应。所有引物见表7。
注意:仅使用 5 μL 的 cDNA 文库。
| 元件 | 1 倍 |
| 10 x 校对聚合酶缓冲液 | 5 |
| 10 μM P5 引物(正向) | 1 |
| 10 μM BC 引物(反向) | 1 |
| 5 mM dNTP | 2.5 |
| 证明读数聚合酶 | 1 |
| cDNA | 5 |
| H2O | 34.5 |
| 最终卷 | 50 微升 |
表9:PCR反应混合物。
- 按如下方式运行PCR:95°C2分钟;n 循环 98 °C 持续 20 秒、52 °C 持续 30 秒和 72 °C 循环 1 分钟;和72°C5分钟。扩增χCRAC文库的循环次数(n)由第10节中描述的qPCR确定。
- 加入1μL核酸外切酶I,并在37°C下孵育60分钟。
- 如上所述,使用两个体积的磁珠(即 100 μL)纯化扩增的 cDNA。在 11 μL 中洗脱。
- 加入 3 μL 6x 上样染料,并在 1x TBE 缓冲液中以 100 V 在预制的 6% TBE 凝胶上运行 1 小时。使用适合定量短DNA片段的分子量标准。
- 完成后,从凝胶盒中取出凝胶,并放入合适的液密容器中,该容器具有足够的1x TBE以覆盖凝胶(例如~50 mL)。添加适量的 SYBR 安全染料(例如,对于 50 mL,使用 5 μL 的 10,000x 染料)
- 让凝胶在室温下轻轻混合15分钟,沥干含SYBR的1x TBE,并用干净的1x TBE代替。在室温下轻轻摇动凝胶10分钟。
- 排干1x TBE并将凝胶放入透明文件夹中。将文件夹剪切到适当的大小。
- 通过适当的方式(例如磷光成像仪)对凝胶进行成像。切除 ~175 bp 和 ~400 bp 之间的 DNA 片段。将凝胶切片放入 1.5 mL 管中。
- 使用P1000尖端彻底粉碎凝胶切片,并加入400μLH2O.在37°C下在热块中振荡孵育1小时。
- 将样品在干冰上冷冻10分钟,然后放回37°C的热块中,振荡1小时。
- 通过取一个过滤柱并在里面插入两个玻璃超细纤维过滤器来创建过滤单元。将装置放入 1.5 mL 管中。
- 用干净的手术刀切掉P1000尖端的末端,吸收粉碎的TBE凝胶悬浮液,然后分配到步骤11.12中创建的过滤单元中。以 17,000 x g 旋转 30 秒。
- 向上清液中加入 1 μL 糖原,以及 40 μL 乙酸钠,pH = 5.2 和 1 mL 96% 乙醇。在-80°C孵育30分钟。
- 在17,000× g,4°C下离心30分钟。 弃去上清液并用500μL70%乙醇洗涤。
- 旋转5分钟,完全除去乙醇,然后在通风橱中干燥沉淀3分钟。
- 重悬于10μL的H2O中并测量DNA浓度。
结果
为了证明χCRAC方法的有效性,对表达HTP标记Nrd1蛋白的酵母菌株进行了时程实验。 图1提供了描述该方法工作原理的详细原理图。与Nab3一样,Nrd1参与多种RNA转录本的核RNA衰变37。Corden实验室先前的工作表明,当细胞遭受葡萄糖饥饿时,Nrd1与其RNA靶标的结合会发生显着变化28,38。因此,在含有葡萄糖(SD-TRP)的培养基中呈指数生长的细胞在一段时间内转移到不含葡萄糖(S-TRP)的同一培养基上,以监测Nrd1-RNA相互作用的动态变化。在移位前和1、2、4、8、14和20分钟后,在Vari-X-接头室(图3A)中采集样品并交联。用于细胞生长的培养基故意缺乏色氨酸,以减少这种芳香族氨基酸对紫外线的吸收。请注意,最好使用经过过滤灭菌的合成培养基,因为高压灭菌培养基会导致糖的焦糖化。这会降低交联效率。
图4A 显示了χCRAC实验的代表性放射自显影。请注意,在此示例中,样本未合并在一起。相反,每个都在凝胶上单独运行。建议将其用于初始实验测试,以证明蛋白质在所有测试时间点都有效地与RNA交联。在RBP的预期分子量下观察到特别强烈的信号,表示蛋白质与非常短的放射性标记RNA结合,不适合测序。因此,分离出该条带上方的涂抹信号,即与较长RNA片段交联的蛋白质。该片段是从蛋白质条带上方加上约30 kDa切割的。 图4B 显示了切除后的放射自显影,蛋白质与留在凝胶中的短RNA交联,现在切除了先前的涂片信号。
逆转录后,必须使用PCR扩增cDNA文库。但是,必须避免文库的过度扩增,因为这会偏向由聚合酶优先扩增的序列并产生PCR伪影。过度放大的库还包含大量重复序列,这些序列会浪费序列器上的读取。为了计算扩增最终文库的理想PCR循环次数,使用P5和BC寡核苷酸通过qPCR扩增cDNA的等分试样。选择文库达到峰值荧光的第一个循环作为PCR循环计数。 图4C 给出了来自典型cDNA文库的qPCR示例,其峰循环计数为16。然后将该值用于最终的χCRAC PCR。为了处理测序的数据,我们使用实验室先前开发的软件(pyCRAC)和相应的管道来分析动力学CRAC数据(Nues等人,2017; https://git.ecdf.ed.ac.uk/sgrannem/pycrac,https://bitbucket.org/sgrann/kinetic_crac_pipeline/src/default/)。这些开源软件工具能够对数据进行解复用和修剪,去除PCR重复,鉴定具有统计学意义的峰,将读数聚类为连续序列,并识别结合基序39。有关这些工具如何运作的更多详细信息,请参阅其各自的网页。
我们还开始为哺乳动物细胞开发χCRAC协议。大多数哺乳动物细胞系都是单层生长的,我们带有紫外线渗透袋的交联剂中的托盘不适合贴壁细胞实验。为了克服这个问题,我们开发了一个载物台,用户可以用贴壁细胞对1-2个培养皿(直径150毫米,深度25毫米)进行紫外线照射(图3B)。作为第一个测试,通过使用抗GFP抗体和传统的基于CLIP的纯化交联和捕获稳定标记的GFP-RBM7来测量哺乳动物细胞交联剂的效率。如图 5A所示,交联剂能够使用254nm紫外线照射从单层生长的哺乳动物细胞中回收蛋白质-RNA复合物,其效率与广泛使用的紫外线照射装置相当。然而,通常用于紫外交联实验的标准细胞培养塑料器皿对254nm紫外是不可穿透的。因此,在我们的交联剂中,细胞只会接受来自紫外线灯上岸的照射。为了克服这个问题,我们开发了一种用于细胞生长和交联的紫外线渗透石英培养皿。使用石英培养器皿在短短 2 秒的紫外线照射下显示出蛋白质-RNA 复合物的稳健回收率(图 5B)。当与哺乳动物细胞的RBP捕获方法(如CLIP技术)结合使用时,这些较短的交联时间适合于时间进程,以响应遗传毒性应激或蛋白质因子的快速消耗,或与转录或细胞周期同步并行恢复RBP的时空RNA结合谱。
图 6 显示了 χCRAC 管道处理的 Nrd1 数据的几个示例。该图是使用管道生成的bedgraph文件和python GenomeBrowser软件包(https://pypi.org/project/GenomeBrowser/1.6.3/)准备的,我们旨在简化数据的出版质量基因组浏览器图像的制作。灰色矩形表示表达非编码RNA的基因组区域,例如隐秘不稳定转录本(CUTE),稳定未表征转录本(SUTs)40和Xrn1敏感不稳定转录本(XUT)41。 图6 中的数据表明,Nrd1与许多这些非编码RNA转录本结合,这与该蛋白质参与这类转录本降解的观点一致42。 图6A 显示了IV染色体上的~15 kb区域。在这里,Nrd1与编码高亲和力葡萄糖转运蛋白HXT6和HXT7的转录本的结合显着增加,这两者都在葡萄糖饥饿期间上调。NNS复合物的转录终止可能会影响葡萄糖饥饿期间这些基因的诱导动力学。 图6B 显示了Nrd1与Imd3转录本交联的示例,已知Imd3转录本受Nab343调节。在这种情况下,数据显示葡萄糖饥饿的结合显着减少。先前的工作显示,在葡萄糖饥饿期间,Nab3与Tye7转录本的结合减少44。与这一观察结果一致,χCRAC 数据表明,Nrd1 的结合在葡萄糖饥饿期间降低,Nrd1 与 Tye7 的交联在胁迫 8 分钟后处于最低水平(图 4C)。然而,这种效应似乎只是短暂的,因为在葡萄糖饥饿14分钟后,Nrd1结合回到起始水平。
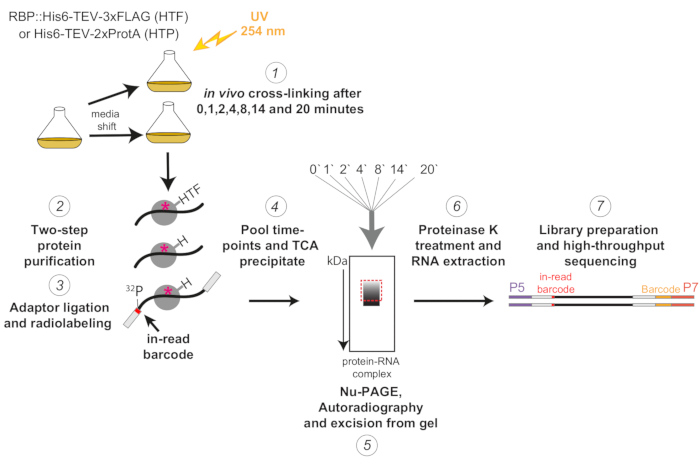
图1: χCRAC 协议的示意图。将标记的菌株生长至所需密度。RBP 表示 RNA 结合蛋白。之后,取参考样品并用254nm紫外光交联。剩余的细胞通过过滤收获,然后迅速转移到应激诱导培养基中。对于此处描述的χCRAC实验,取样并在移位后1,2,4,8,14和20分钟交联(1)。然后使用高度严格的两步亲和纯化纯化目标RBP(2)。接下来,捕获的交联RNA用RNA酶部分消化,在5'端进行放射性标记,并将接头连接到它们上(3)。5'适配器包含独特的"读取中"条形码序列,因此可以在测序后通过生物信息学分离单个样品。然后将RBP-RNA复合物洗脱、汇集并沉淀在一起(4),通过SDS-PAGE分离并通过放射自显影可视化(5)。随后,从凝胶(5)上切下含有主条带正上方放射性信号的单个凝胶切片,在放射自显影图像中用虚线的红色框表示。用蛋白酶K处理凝胶切片,随后提取RNA(6),转化为cDNA,并通过PCR扩增(7)。PCR步骤引入了额外的条形码(P7寡核苷酸引入的黄色块),因此可以将许多文库多路复用到单个泳道中。 请点击此处查看此图的大图。

图 2:交联和真空过滤。 (A)交联剂。将细胞悬液倒入位于机器右上角的漏斗中(特写另见 图3A ),并保存在位于中间托盘的紫外线透明袋中。该袋子的两侧有两个百叶窗,在用户指示机器开始照射步骤之前,它们一直保持关闭状态。细胞用来自上方和下方托盘的紫外线照射。该机器配有 254 和 365 nm 紫外灯,后者适用于 PAR-CLIP 实验。机器通过位于右上角的触摸屏面板进行操作,该面板允许控制紫外线剂量或曝光时间。(B)交联后,细胞从机器的左侧排出。细胞悬浮液通过真空回收并排入玻璃烧瓶中,随后可以将其倒入真空过滤装置中进行收获。(三)真空过滤装置。它们通过夹子打开和关闭,并在它之间插入一个过滤器。四个过滤装置在非常短的时间序列中并行使用,以免因更换过滤器而损失任何时间。(D)过滤后,将培养基上清液排入烧瓶中进行后续处置。阀门安装在真空过滤装置下方,以在移除过滤器时保持系统中的真空。 请点击此处查看此图的大图。

图3:交联悬浮细胞与贴壁细胞。 (A)用于悬浮细胞的带有Vari-X接头室的交联剂。将细胞培养物倒入位于托盘右上角的样品入口(漏斗)中。(B)可以容纳塑料或石英培养皿的托盘,用于交联贴壁细胞或少量悬浮细胞。 请点击此处查看此图的大图。

图 4:文库制备。 (A)来自Nrd1-HTP χCRAC实验的放射自显影示例。强而集中的信号表示与非常短的RNA交联的蛋白质,而上面的涂片表示与足够长度的RNA交联的蛋白质以进行测序。(B)涂片被切除,如凝胶切除后采集的放射自显影所示。(C)来自χCRAC cDNA文库的代表性qPCR。在本例中,cDNA的最大扩增在16个循环中达到。因此,使用16个循环进行最终扩增。误差条表示三个技术qPCR重复的标准偏差。(D)来自6%TBE凝胶上的cDNA文库的磷图像示例。(E)来自芯片的毛细管电泳的cDNA长度和质量分析。 请点击此处查看此图的大图。
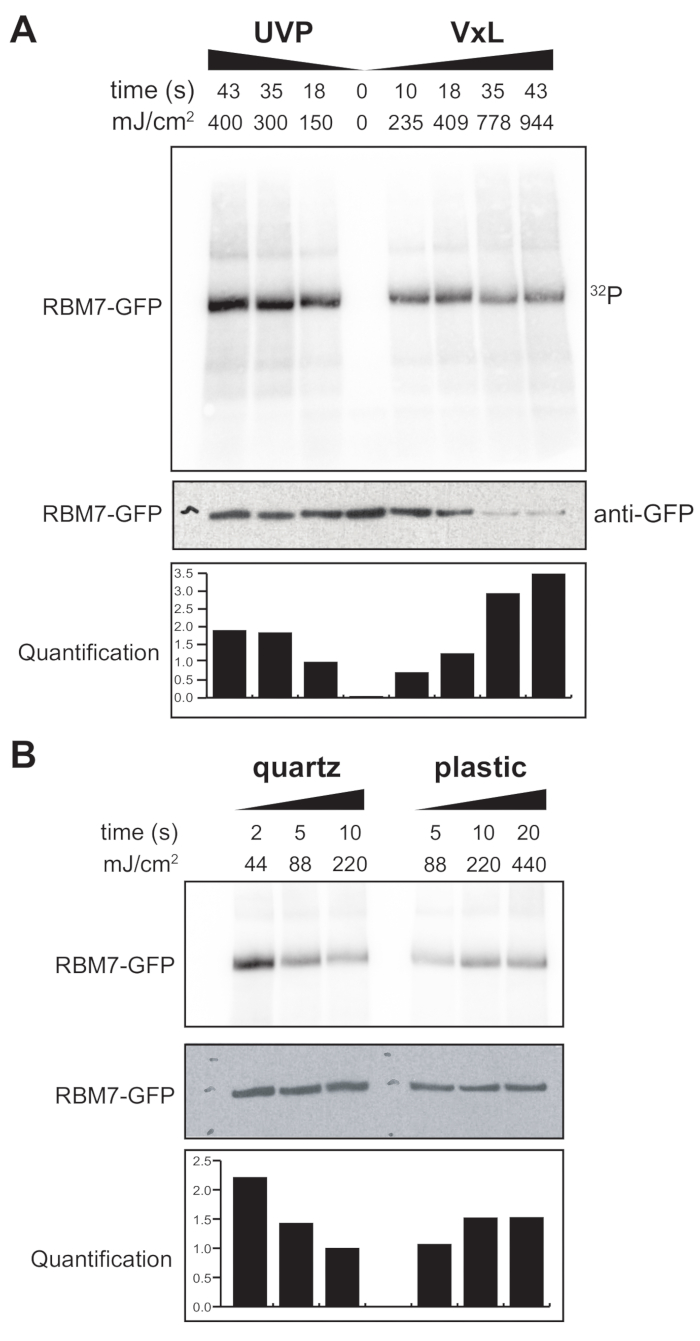
图 5: 高 RNase 测试 iCLIP 实验以测试哺乳动物细胞中的交联。 图中显示了来自GFP-RBM7 iCLIP实验的放射自体图,该实验测试了各种交联能量下RNP回收的效率。使用抗GFP抗体与稳定表达GFP-RBM7的交联细胞上的磁珠偶联进行免疫沉淀。将免疫沉淀物与高浓度的RNase I一起孵育,以便将相关RNA修剪成短而均匀的长度。RNP通过 32P标记和SDS-PAGE可视化,并作为定义的条带迁移,接近非交联蛋白的迁移。定量表明放射性标记的RBM7-RNA信号的密度分析结果归一化为抗GFP蛋白质印迹信号。(A)常用的UVP交联剂与我们的交联剂(Vari-X-linker;(B)我们的交联剂在石英(左)和塑料(右)培养器上的交联时间过程。 请点击此处查看此图的大图。
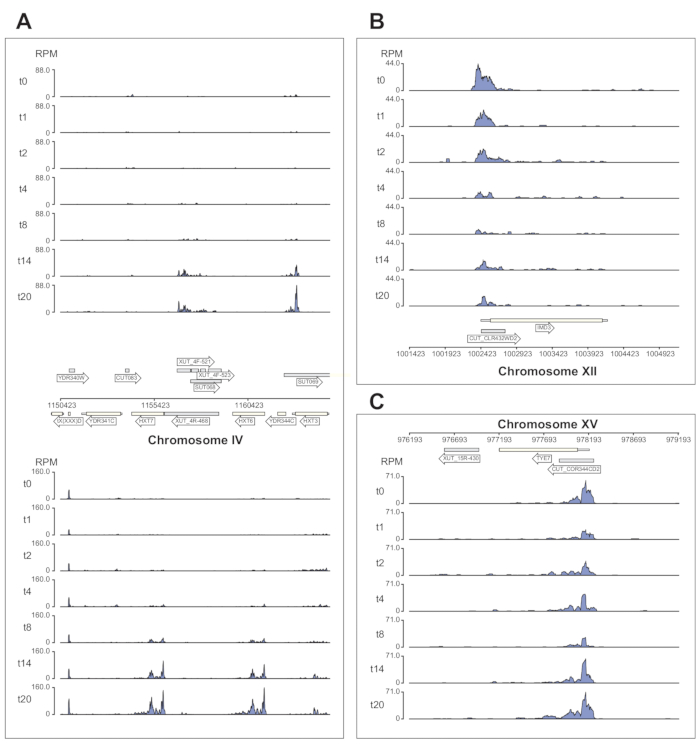
图 6:示例基因组浏览器图显示了 χCRAC 显示 Nrd1 与其靶标的差异、时间结合的能力。每个框显示单个基因组区域的图。箭头表示基因在哪条链上编码(左箭头=减链;右箭头=加链)。时间点(分钟)在每个子图的 y 轴上由 t0、t1、t2 等表示。显示了指示染色体和坐标的罗马数字。(A)在葡萄糖剥夺时,Nrd1结合两种高亲和力葡萄糖转运蛋白HXT6和HXT7,它们在这种情况下都上调。(B)观察到Nrd1与Imd3结合,Imd3是Nab344已经验证的靶标,在葡萄糖饥饿后强度降低。(C)Tye7的Nrd1结合表现出动态和短暂的性质,在葡萄糖饥饿后在胁迫8分钟后降至最小。然而,结合随后在14分钟后恢复到基础水平。读取被标准化为"每百万读取数"(RPM;y 轴)。灰色框表示编码非编码RNA的区域。请点击此处查看此图的大图。
讨论
χCRAC方法与新的交联和细胞收获装置相结合,具有巨大的潜力,因为它适用于广泛的模式生物,因此应该引起RNA领域的普遍兴趣。χCRAC 可用于许多领域。例如,该方法可用于测量蛋白质成大分子复合物的分层组装,例如剪接体和核糖体,这通常涉及蛋白质和RNA分子之间的动态相互作用。我们现在还经常使用它来监测当细胞受到各种压力时RNA衰变因子与其底物之间的相互作用。这使我们能够确定这些因素在适应性反应的哪个阶段最活跃,它们与哪些底物结合,以及这些相互作用的动态性。这些数据应使研究人员能够确定每个因素在适应环境变化方面的相对贡献。
χCRAC 使用双亲和纯化标签(HTF 或 HTP)在高度严格和变性的条件下纯化蛋白质。这确保了共纯化的RNA对于与目标蛋白共价交联的RNA具有高度富集性。但是,依赖亲和力标记有缺点。例如,标签可能会干扰蛋白质功能,这可能会使其RNA结合相互作用组的读数失真。此外,对于某些模式生物,可能并不总是能够利用标签,因为尚未获得将DNA片段整合到基因组中或转化表达质粒的遗传工具。然而,改变χCRAC方案的某些部分以使其与依赖抗体纯化RBP的基于CLIP的方案兼容是很简单的。事实上,这项研究表明,可以将基于iCLIP的纯化与我们的交联剂相结合。我们现在正在开发CLIP协议,以研究人类RNA结合蛋白与新生RNA转录本的时间关联。
当对新蛋白质进行χCRAC时,必须优化紫外线照射以诱导最大的交联。这一点很重要,因为高紫外线照射会降低纯化步骤中RNA的回收率。表达重组RBP的细胞暴露于各种紫外线剂量,100 mJ / cm 2,250 mJ / cm 2,500 mJ / cm 2和1 J/ cm 2。然后捕获RNP,并对RNA片段化和放射性标记。之后,通过SDS-PAGE解析RNP,并进行放射自显影,以推断哪种暴露产生最强烈的信号(即最大交联)。
一旦实验条件得到优化,建议在进行χCRAC时进行几个对照实验。首先,可以使用紫外线照射的未标记样品来监测与纯化微球的背景结合。其次,当在转移实验期间应用χCRAC时,细胞移回原始培养基的第二个时间序列可以研究细胞本身的过滤是否诱导RNA水平或蛋白质 - RNA相互作用的变化。
如引言所述,最近发表的许多论文提出了对CLIP协议的一些优化。这包括使用荧光标记的适配器通过红外扫描10 检测蛋白质-RNA复合物,以及对各种核酸纯化和大小选择步骤的优化,以增加所得文库的复杂性12,45。我们目前正在实施其中一些改进,以进一步完善χCRAC协议。这里介绍的协议已经包含对原始CRAC和χCRAC协议的许多改进,这些改进增加了数据的复杂性。例如,以前,在分离SDS-PAGE凝胶上的交联放射性蛋白质-RNA复合物后,将它们转移到硝酸纤维素膜上,并从印迹中分离交联RNA。然而,RNP的转移和随后的RNA提取可能非常低效,特别是在处理大RBP如RNA聚合酶亚基时。这可能导致交联RNA的回收率显着降低。在当前方案中,交联RNA直接从SDS-PAGE凝胶切片中提取,如图 1所示。这增加了交联RNA的回收率。此外,在cDNA的PCR扩增后,产物最初在3%的低熔解温度琼脂糖凝胶上分离,然后从凝胶中提取175-300 bp PCR产物。然而,这些凝胶很容易过载,导致DNA分离非常差。用预制TBE凝胶代替琼脂糖凝胶可实现更一致的尺寸分离和更好的PCR产物回收率。
披露声明
A. Langford和W. Worboys隶属于商业公司UVO3。他们在研究设计、数据收集和解释或提交作品发表的决定中没有任何作用。
致谢
这项工作得到了威康信托基金会(091549给S.G.和109093/Z/15 / A给S.M.),威康信托基金会细胞生物学中心核心资助(092076)和医学研究委员会非临床高级研究奖学金(MR / R008205 / 1到S.G.),欧洲分子生物学组织的长期博士后奖学金(ALTF 1070-2017到R.A.C.)的资助, 以及丹麦独立研究基金(T.H.J)。
材料
| Name | Company | Catalog Number | Comments |
| 1,4-dithioreitol | Merck | 10708984001 | Buffer component in mammalian cell lysis |
| 1.5 mL tubes | Eppendorf | 0030 120.086 | General reaction tube |
| 2 mL tubes | Eppendorf | 0030 123.344 | For holding columns and collection of waste |
| 32P-yATP | Perkin Elmer | NEG502Z-250 | For radiolabelling the 5' end of the RNA |
| 4-12% Bis-Tris gel | Invitrogen | NP0321BOX | SDS-PAGE gel |
| 4X loading buffer | Novex | NP0008 | Protein loading dye concentrate |
| 50 bp ladder | New England Biolabs | N3236 | Reference ladder for excising region of interest from the amplified cDNA library |
| 50% PEG | NEB | B100045 | For the L5 linker ligation |
| 6% TBE gel | Invitrogen | EC6265BOX | For separation and purification of the cDNA library |
| Acetone | ACROS Organics | 423245000 | Washing of TCA-precipitated proteins |
| anti-FLAG beads | Sigma Aldrich | M8823-1ML | For purifcation of FLAG-tagged RBPs |
| ATP (100 mM) | Thermo Fisher Scientific | R0441 | For ligation of the L5 linker onto the 5' end of captured RNAs |
| Beta-mercaptoethanol | Sigma Aldrich | M3148-100ML | Buffer component |
| Biomax MS intensifying screen | Sigma Aldrich | Z363162-1EA | For intensifying the autoradiogram signal |
| Chloroform | Thermo Fisher Scientific | 1010219 | For phenol-chloroform extraction following RNA purification |
| cOmplete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | For inhibition of cellular proteases after lysis |
| Complete supplement mixture -TRP | Formedium | DCS0149 | For preparation of synthetic defined medium |
| Costar Spin-X 0.22 µm filters | Sigma Aldrich | CLS8160 | For isolating the excised cDNAs following gel extraction |
| DNase RQ1 | Promega | M6101 | For DNA digest following cell lysis |
| dNTPs (10 mM) | Sigma Aldrich | 4638956001 | For reverse transcription and PCR |
| Ethanol | Thermo Fisher Scientific | 10041814 | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Ethylenediaminetetraacetic acid | Invitrogen | AM9261 | For protease K buffer |
| Exonuclease I | New England Biolabs | M0293 | For degradation of primers following PCR |
| Glass microfiber filters | Whatman | 1823-010 | For isolating the excised cDNAs following gel extraction |
| Glucose | Formedium | GLU03 | For preparation of glucose-containing, synthetic defined medium |
| Glycogen (20 mg/mL) | Roche | 10901393001 | Precipitation of proteins, RNA and DNA |
| GST-TEV | Homemade | Construct and purification protocol is available upon request | |
| Guanidium hydrochloroide | Thermo Fisher Scientific | 10071503 | Required for pulldown denaturing conditions and washing buffer |
| IgG beads | GE Healthcare | 17-0969-01 | For purification of protein A-tagged RBPs |
| Imidazole | Sigma Aldrich | I2399-100G | For elution of captured proteins from Nickel beads |
| Isoamyl alcohol | Thermo Fisher Scientific | A393-500 | For phenol-chloroform extraction following RNA purification |
| Luna Universal One-Step RT-qPCR | NEB | E3005S | For qPCR of the cDNA in order to calculate required number of PCR cycles |
| Magnesium chloride | Fluka Analytical | 63020-1L | For PNK buffer |
| Membrane filters | Millipore | AAWP09000 for yeast or HAWP09000 for bacteria | For vacuum filtration of cells |
| Micro bio-spin columns | Biorad | 732-6204 | For collecting eluate after gel extraction |
| Ni-NTA beads | Qiagen | 30210 | For secondary protein capture |
| NP-40 | Sigma Aldrich | I8896-100ML | Buffer component |
| Pfu polymerase | Promega | M7741 | For amplification of the cDNA library |
| Phenol | Sigma Aldrich | P4682-400ML | For phenol-chloroform extraction following RNA purification |
| Pierce spin columns | Thermo Fisher Scientific | 69725 | For on-column enzymatic reactions |
| Protease K | Roche | 3115887001 | For degradation of the RBP following gel extraction |
| Quartz Petri dish | UVO3 | N/A | For cross-linking of adherent cells. Available from https://www.vari-x-link.com for 400 GBP |
| Radiography films | Amersham | 28906843 | For autoradiography visualisation |
| RNAClean XP beads | Beckmann | A63987 | SPRI beads for clean up of RNAs and cDNAs |
| RNase H | New England Biolabs | M0297 | For degradation of RNAs following reverse transcription |
| RNase-It | Agilent | 400720 | For RNA digestion |
| rRNasin | Promega | N2511 | For inhibition of any contaminating RNases during enzymatic reaction |
| Sodium acetate | Sigma Aldrich | S2889-1KG | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Sodium chloride | Thermo Fisher Scientific | 7647-14-5 | Buffer component |
| Sodium deoxycholate | Sigma Aldrich | D6750-100G | Buffer component in mammalian cell lysis |
| Sodium dodecylsulfate | Sigma Aldrich | L3771-1KG | For protease K buffer |
| SUPERase-In | Invitrogen | AM2694 | For inhibition of cellular RNases after lysis |
| SuperScript IV | Thermo Fisher Scientific | 18090010 | For reverse transcription |
| T4 PNK | New England Biolabs | M0201 | For radiolabelling the 5' end of the RNA |
| T4 RNA ligase 1 | New England Biolabs | M0204 | For ligation of the L5 adaptor onto the RNA 5' end |
| T4 RNase ligase 2, truncated K222Q | NEB | M0351S | For ligation of the App_PE linker onto the 3' end of captured RNAs |
| TBE buffer (10X) | Invitrogen | 15581-028 | For running TBE gels |
| TEV protease | Homemade | For eluting captured proteins following FLAG capture | |
| Thermosensitive alkaline phosphatase | Promega | M9910 | For 5' and 3' dephosphorylation of RNAs |
| Trichloroacetic acid (100%) | Sigma Aldrich | T0699-100ML | For precipitation of RBP-RNA complexes |
| Tris hydrochloride | Invitrogen | 15504-020 | Buffer component |
| Triton X-100 | Sigma Aldrich | T8787-100ML | Buffer component in mammalian cell lysis |
| Vari Filter | UVO3 | N/A | Device for vacuum harvesting cells. Available from https://www.vari-x-link.com for 100 GBP |
| Vari-X-Linker | UVO3 | N/A | Cross-linker for cross-linking cells. Available from https://www.vari-x-link.com for 16,000 GBP |
| Yeast nitrogen base | Formedium | CYN0410 | For preparation of synthetic defined medium |
| Zirconia beads | Thistle | 11079105Z for yeast or 11079101Z for bacteria | For cell lysis via bead beating |
参考文献
- Ule, J., et al. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Granneman, S., Kudla, G., Petfalski, E., Tollervey, D. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proceedings of the National Academy of Sciences. 106 (24), 9613-9618 (2009).
- Licatalosi, D. D., et al. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-469 (2008).
- König, J., et al. iCLIP reveals the function of hnRNP particles in splicing at individual nucleotide resolution. Nature Structural & Molecular Biology. 17 (7), 909-915 (2010).
- Hafner, M., et al. Transcriptome-wide Identification of RNA-Binding Protein and MicroRNA Target Sites by PAR-CLIP. Cell. 141 (1), 129-141 (2010).
- Aktaş, T., et al. DHX9 suppresses RNA processing defects originating from the Alu invasion of the human genome. Nature. 544 (7648), 115-119 (2017).
- Huppertz, I., et al. iCLIP: Protein–RNA interactions at nucleotide resolution. Methods. 65 (3), 274-287 (2014).
- Li, X., et al. Comprehensive in vivo RNA-binding site analyses reveal a role of Prp8 in spliceosomal assembly. Nucleic Acids Research. 41 (6), 3805-3818 (2013).
- Rosenberg, M., et al. Denaturing CLIP, dCLIP, Pipeline Identifies Discrete RNA Footprints on Chromatin-Associated Proteins and Reveals that CBX7 Targets 3′ UTRs to Regulate mRNA Expression. Cell Systems. 5 (4), 368-385 (2017).
- Zarnegar, B. J., et al. irCLIP platform for efficient characterization of protein–RNA interactions. Nature Methods. 13 (6), 489-492 (2016).
- Kargapolova, Y., Levin, M., Lackner, K., Danckwardt, S. sCLIP—an integrated platform to study RNA–protein interactomes in biomedical research: identification of CSTF2tau in alternative processing of small nuclear RNAs. Nucleic Acids Research. 45 (10), 6074-6086 (2017).
- Van Nostrand, E. L., et al. Robust transcriptome-wide discovery of RNA-binding protein binding sites with enhanced CLIP (eCLIP). Nature Methods. 13 (6), 508-514 (2016).
- Flynn, R. A., et al. Dissecting noncoding and pathogen RNA–protein interactomes. RNA. 21 (1), 135-143 (2015).
- Brugiolo, M., Botti, V., Liu, N., Müller-McNicoll, M., Neugebauer, K. M. Fractionation iCLIP detects persistent SR protein binding to conserved, retained introns in chromatin, nucleoplasm and cytoplasm. Nucleic Acids Research. 45 (18), 10452-10465 (2017).
- Sanford, J. R., et al. Identification of Nuclear and Cytoplasmic mRNA Targets for the Shuttling Protein SF2/ASF. PLOS ONE. 3 (10), e3369(2008).
- Garzia, A., Meyer, C., Morozov, P., Sajek, M., Tuschl, T. Optimization of PAR-CLIP for transcriptome-wide identification of binding sites of RNA-binding proteins. Methods. 118-119, 24-40 (2017).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22 (10), 2031-2042 (2012).
- Chen, K., et al. High-Resolution N6-Methyladenosine (m6A) Map Using Photo-Crosslinking-Assisted m6A Sequencing. Angewandte Chemie International Edition. 54 (5), 1587-1590 (2015).
- Ke, S., et al. A majority of m6A residues are in the last exons, allowing the potential for 3′ UTR regulation. Genes & Development. 29 (19), 2037-2053 (2015).
- Linder, B., et al. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nature Methods. 12 (8), 767-772 (2015).
- Kudla, G., Granneman, S., Hahn, D., Beggs, J. D., Tollervey, D. Cross-linking, ligation, and sequencing of hybrids reveals RNA–RNA interactions in yeast. Proceedings of the National Academy of Sciences. 108 (24), 10010-10015 (2011).
- Sugimoto, Y., et al. hiCLIP reveals the in vivo atlas of mRNA secondary structures recognized by Staufen 1. Nature. 519 (7544), 491-494 (2015).
- Hwang, H. W., et al. cTag-PAPERCLIP Reveals Alternative Polyadenylation Promotes Cell-Type Specific Protein Diversity and Shifts Araf Isoforms with Microglia Activation. Neuron. 95 (6), 1334-1349 (2017).
- Hwang, H. W., et al. PAPERCLIP Identifies MicroRNA Targets and a Role of CstF64/64tau in Promoting Non-canonical poly(A) Site Usage. Cell Reports. 15 (2), 423-435 (2016).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Beckmann, B. M. RNA interactome capture in yeast. Methods. 118-119, 82-92 (2017).
- Granneman, S., Petfalski, E., Tollervey, D. A cluster of ribosome synthesis factors regulate pre-rRNA folding and 5.8S rRNA maturation by the Rat1 exonuclease. The EMBO Journal. 30 (19), 4006-4019 (2011).
- Schaughency, P., Merran, J., Corden, J. L. Genome-Wide Mapping of Yeast RNA Polymerase II Termination. PLOS Genetics. 10 (10), e1004632(2014).
- Bernstein, J. A., Khodursky, A. B., Lin, P. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Kresnowati, M. T. A. P., et al. When transcriptome meets metabolome: fast cellular responses of yeast to sudden relief of glucose limitation. Molecular Systems Biology. 2, 49(2006).
- Marguerat, S., Lawler, K., Brazma, A., Bähler, J. Contributions of transcription and mRNA decay to gene expression dynamics of fission yeast in response to oxidative stress. RNA Biology. 11 (6), 702-714 (2014).
- van Nues, R., et al. Kinetic CRAC uncovers a role for Nab3 in determining gene expression profiles during stress. Nature Communications. 8 (1), 12(2017).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Tudek, A., Candelli, T., Libri, D. Non-coding transcription by RNA polymerase II in yeast: Hasard or nécessité? Biochimie. 117, 28-36 (2015).
- Lingaraju, M., et al. The MTR4 helicase recruits nuclear adaptors of the human RNA exosome using distinct arch-interacting motifs. Nature Communications. 10 (1), 1-11 (2019).
- Lubas, M., et al. Interaction Profiling Identifies the Human Nuclear Exosome Targeting Complex. Molecular Cell. 43 (4), 624-637 (2011).
- Conrad, N. K., et al. A yeast heterogeneous nuclear ribonucleoprotein complex associated with RNA polymerase II. Genetics. 154 (2), 557-571 (2000).
- Darby, M. M., Serebreni, L., Pan, X., Boeke, J. D., Corden, J. L. The Saccharomyces cerevisiae Nrd1-Nab3 Transcription Termination Pathway Acts in Opposition to Ras Signaling and Mediates Response to Nutrient Depletion. Molecular and Cellular Biology. 32 (10), 1762-1775 (2012).
- Webb, S., Hector, R. D., Kudla, G., Granneman, S. PAR-CLIP data indicate that Nrd1-Nab3-dependent transcription termination regulates expression of hundreds of protein coding genes in yeast. Genome Biology. 15 (1), R8(2014).
- Jensen, T. H., Jacquier, A., Libri, D. Dealing with Pervasive Transcription. Molecular Cell. 52 (4), 473-484 (2013).
- van Dijk, E. L., et al. XUTs are a class of Xrn1-sensitive antisense regulatory non-coding RNA in yeast. Nature. 475 (7354), 114-117 (2011).
- Thiebaut, M., et al. Futile Cycle of Transcription Initiation and Termination Modulates the Response to Nucleotide Shortage in S. cerevisiae. Molecular Cell. 31 (5), 671-682 (2008).
- Merran, J., Corden, J. L. Yeast RNA-Binding Protein Nab3 Regulates Genes Involved in Nitrogen Metabolism. Molecular and Cellular Biology. 37 (18), e00154-e00117 (2017).
- Bresson, S., Tuck, A., Staneva, D., Tollervey, D. Nuclear RNA Decay Pathways Aid Rapid Remodeling of Gene Expression in Yeast. Molecular Cell. 65 (5), 787-800 (2017).
- Buchbender, A., et al. Improved library preparation with the new iCLIP2 protocol. Methods. , (2019).
Erratum
Formal Correction: Erratum: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC
Posted by JoVE Editors on 2/17/2023. Citeable Link.
An erratum was issued for: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC. The Authors section was updated from:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Synthetic and Systems Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
to:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Mehak Chauhan1
Niki Christopoulou1
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Engineering Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。