Method Article
χCRAC Kullanarak Yüksek Zamansal Çözünürlükte Protein-RNA Etkileşim Dinamiğinin İn Vivo İzlenmesi
Bu Makalede
Erratum Notice
Özet
cDNA'nın kinetik çapraz bağlanması ve analizi, canlı hücrelerdeki protein-RNA etkileşimlerinin dinamiklerinin yüksek zamansal çözünürlükte araştırılmasını sağlayan bir yöntemdir. Burada protokol, maya hücrelerinin büyümesi, UV çapraz bağlama, hasat, protein saflaştırma ve yeni nesil dizileme kütüphanesi hazırlama adımları dahil olmak üzere ayrıntılı olarak açıklanmaktadır.
Özet
RNA bağlayıcı proteinler (RBP'ler) ve RNA substratları arasındaki etkileşim, akışkanlık ve karmaşıklık gösterir. Yaşam süresi boyunca, tek bir RNA, üretimini, kararlılığını, aktivitesini ve bozulmasını düzenleyecek birçok farklı RBP tarafından bağlanabilir. Bu nedenle, bu iki molekül türü arasında var olan dinamikleri anlamak için çok şey yapılmıştır. Özellikle önemli bir atılım, 'cross-l mürekkepleme ve immunoprecipitation' (CLIP) ortaya çıkmasıyla geldi. Bu teknik, hangi RNA'ların belirli bir RBP tarafından bağlandığına dair sıkı bir araştırmaya izin verdi. Kısacası, ilgilenilen protein, RNA substratlarına in vivo olarak çapraz bağlanmış, oldukça katı koşullar altında saflaştırılmış UV'dir ve daha sonra proteine kovalent olarak çapraz bağlanmış RNA'lar cDNA kütüphanelerine dönüştürülür ve sıralanır. Kuruluşundan bu yana, CLIP'yi belirli çalışma alanlarına uygun hale getirmek için birçok türev teknik geliştirilmiştir. Bununla birlikte, ultraviyole ışık kullanarak çapraz bağlama, bilinen bir şekilde verimsizdir. Bu, RBP-RNA etkileşimlerinin zamansal çalışmasını imkansız kılan uzun maruz kalma süreleri ile sonuçlanır. Bu sorunun üstesinden gelmek için, yakın zamanda çok daha gelişmiş UV ışınlama ve hücre toplama cihazları tasarladık ve ürettik. Bu yeni araçları kullanarak, canlı hücrelerdeki RBP-RNA etkileşimlerinin yüksek zamansal çözünürlükte zamana bağlı analizleri için bir protokol geliştirdik: Kinetik CRoss bağlantısı ve cDNA'larının (χCRAC) birnalizi. Son zamanlarda bu tekniği, maya RBP'lerinin besin stresi adaptasyonundaki rolünü incelemek için kullandık. Bu makalede χCRAC yöntemine ayrıntılı bir genel bakış sunulmakta ve Nrd1 RBP ile elde edilen son sonuçlar sunulmaktadır.
Giriş
RNA'lar genellikle işlevlerini yerine getirmek için RBP'lere güvenirler, bu da bu moleküller arasındaki dinamikleri anlamaya büyük ilgi duymuştur. Birçok RBP, çok çeşitli organizmalarda tanımlanmıştır. Bununla birlikte, RBP-RNA etkileşimlerini in vivo olarak incelemek her zaman zor olmuştur. Bu tür etkileşimlerin incelenmesinde büyük bir atılım, CLIP1'in ortaya çıkmasıyla geldi. Bu yöntem, RBP'ler ve doğrudan bağlı RNA'ları arasındaki kovalent bağları (yani, sıfır mesafeli çapraz bağlama) indüklemek için ultraviyole (UV, 254 nm) ışınlamasını kullanır. Daha sonra, ilgilenilen RBP, yalnızca proteinlere kovalent olarak çapraz bağlı RNA'ların tanımlanmasını sağlamak için sıkı koşullar altında immünosaflaştırılır. Bağlı RNA'lar daha sonra RNazlarla kısmen sindirilir ve daha sonra dizileme için cDNA kütüphanelerine dönüştürülür. Yüksek saflaştırma sıkılığı, çapraz bağlı ribonükleoprotein (RNP) kompleksinin SDS-PAGE saflaştırılmasıyla daha da geliştirilen protein ve RNA geri kazanımının özgüllüğünü büyük ölçüde arttırdığı için önemlidir. CLIP ve ilgili yöntemler ayrıca protein bağlanma bölgesine nükleotid çözünürlüğü anlayışı sağlar, çünkü dizileme kütüphanesinin hazırlanması sırasında, RNA'ya çapraz bağlanan amino asitler sıklıkla ters transkriptazı sonlandırır veya enzimin bu bölgede mutasyonlar oluşturmasına neden olur 1,2,3.
Tanıtılmasından bu yana, orijinal CLIP protokolü dikkate değer çeşitlilikte türev metodolojileri üretmiştir. Özellikle önemli bir atılım, yüksek verimli sıralamayı CLIP yaklaşım3 ile birleştiren HITS-CLIP'in (veya CLIP-seq) geliştirilmesiyle geldi. Bu, o zamandan beri tüm CLIP tabanlı metodolojiler tarafından benimsenmiştir. iCLIP, RBP bağlama bölgelerinin daha doğru haritalanmasını kolaylaştıran RNase aracılı kırpma ve adaptör ligasyon tekniklerinde iyileştirmeler getirmiştir4. PAR-CLIP, 4tiyo-idrar / urasil etiketlemeyi 365 nm'de çapraz bağlama ile birleştirerek, T-C ikamelerini analiz ederek çapraz bağlama bölgelerini haritalamayı mümkün kıldı5. CRAC, üre-iCLIP, dCLIP ve uvCLAP, afinite reçinesine arka plan bağlanmasını daha da azaltan ve protein yakalama 2,6,7,8,9'un özgüllüğünü daha da artıran denatüre etme koşulları ve çift afinite saflaştırma adımları getirmiştir. Ek olarak, CRAC, uvCLAP ve dCLIP, ilgilenilen RBP'yi bir afinite etiketi ile etiketlemeyi tanıttı, böylece spesifik antikorlar üretme ihtiyacının üstesinden geldi.
CLIP metodolojisini hızlandırmak için çeşitli optimizasyonlar da yapılmıştır. Orijinal CLIP protokolü, SDS-PAGE'den sonra RBP-RNA komplekslerini görselleştirmek için yakalanan RNA'ların radyoetiketlemesini kullandı. Bununla birlikte, radyoaktivite kullanımı, bu tür çalışmalar için kurulmamış laboratuvarlar için sorunlu olabilir. irCLIP, kızılötesi görüntüleme10 yoluyla görselleştirmeyi kolaylaştıran florofor kuplajlı bir adaptör içerir ve sCLIP, streptavidin konjuge HRP11 aracılığıyla görselleştirmek için yakalanan RNA'ların biyotinilasyonunu kullanır. Ayrıca, eCLIP RNA etiketlemesinden tamamen vazgeçer; bunun yerine, protein sadece bilinen büyüklüğüne göre eksize edilir12. Streptavidin bazlı saflaştırma, biyotinillenmiş bir 3' adaptörün RNA'lara bağlandığı ve ters transkripsiyon ve daireselleştirme13'ten sonra saflaştırmayı sağlamak için kullanıldığı FAST-iCLIP'de kütüphane hazırlama sürecini hızlandırmak için de kullanılmıştır. iCLIP protokolündeki ek geliştirmeler de kütüphanelerin karmaşıklığını büyük ölçüde artırdı4.
Son olarak, CLIP, RBP'lerin farklı hücresel alt bölmelerden 14,15'ten yakalanmasını sağlamak, fotoaktive edilebilir ribonükleozitlerindarbeli indüksiyonu kullanılarak yeni transkribe edilmiş RNA'ları görselleştirmek için 5,16,17, metillenmiş RNA'ları yakalamak için18,19,20, RNA-RNA etkileşimlerini incelemek21,22 ve 3' uçlarını haritalamak için modifiye edilmiştir 23,24.
RBP'ler ve RNA'lar arasındaki etkileşimleri anlamamıza yardımcı olan CLIP tabanlı tekniklerin büyük katkılarına rağmen, UV çapraz bağlamanın verimsizliği ile sınırlandırılmıştır. Tek katmanlı bir tabakada yetiştirilen kültür hücrelerinin çapraz bağlanması genellikle nispeten kolay olsa da, bu dokularda veya çözeltideki hücrelerde önemli ölçüde daha zordur. Dokular, gerekli hücre katmanlarına nüfuz etmek için birden fazla UV maruziyeti turu gerektirebilirken, mikrobiyal hücreler genellikle aromatik, UV emici bileşikler içeren zengin ortamlarda yetiştirilir25. Gerçekten de, bu tür örnekler için RBP'ler ve bağlı RNA'ları arasında yeterli çapraz bağlantı oluşturmak için 30 dakikaya kadar UV ışınlama süreleri kullanılmıştır26,27,28. Bu genişletilmiş UV maruziyeti, hücre içinde, bazı uygulamalarda nihai verileri kirletebilecek UV kaynaklı DNA hasarı gibi stres tepkilerini indükler.
CLIP çalışmalarının çoğu, bir hücredeki spesifik protein-RNA etkileşimlerinin tek bir "anlık görüntüsünü" üretmeye odaklanmıştır. Bununla birlikte, protein-RNA etkileşimleri, özellikle hücreler çevrelerinde değişikliklere maruz kaldıklarında doğal olarak dinamiktir. Bu, temel besin maddelerinin mevcudiyetinde ani bir azalma veya sıcaklıktaki hızlı değişiklikleri içerebilir. Bu nedenle, stres sırasında bir RBP'nin rolünü gerçekten anlamak için, zamana bağlı analizler yapmak en iyisidir, çünkü stres sırasında RBP hedeflerinin tüm spektrumunu yakalayabilir ve seçilen RBP'nin stres tepkisinin hangi aşamasında aktif olduğunu belirleyebilirler. Özellikle, mayadaki çalışmalar, adaptasyonun ilk birkaç dakikasının hayatta kalmak için kesinlikle çok önemli olduğunu ve bakterilerdeki RNA yarı ömrünün dakikalardan saniyelere kadar değişebileceğini göstermiştir 29,30,31,32,33. Bu nedenle, bu tür zaman çözümlü analizler ideal olarak yüksek zamansal çözünürlükte yapılmalıdır. Bununla birlikte, uzun çapraz bağlama süreleri, erken aşama adaptif yanıtların incelenmesini özellikle zorlaştırmaktadır.
Bu sorunların üstesinden gelmek için, yakın zamanda hücreleri dakikalarca süren zaman dilimlerinde çapraz bağlayabilen ve toplayabilen gelişmiş bir yöntem geliştirdik. χCRAC yöntemimiz, RBP-RNA etkileşimlerindeki dinamik değişikliklerin daha önce tanık olunmamış çözünürlükte nicel olarak ölçülmesini sağlar. Bu yöntem için çok önemli olan, çözeltideki maya ve bakterilerde gerekli çapraz bağlanma süresini yaklaşık 10 kat azaltan ve RBP-RNA etkileşimlerini anında etkili bir şekilde donduran yeni bir UV ışınlama cihazı32'nin geliştirilmesiydi. Ek olarak, UV ışınlamasından sonra hücreleri hızlı bir şekilde toplamak için, yaklaşık 30 s32'de 0.5 L'lik bir kültürde katlanarak büyüyen mayayı hasat edebilen bir vakum filtrasyon cihazı geliştirdik. Bu teknolojik yenilikler, RBP-RNA dinamiklerinin dakika ölçeğinde çözünürlükte incelenmesine izin verir. Ek olarak, pratikliğini artırmak için orijinal CRAC protokolü2'ye çeşitli optimizasyonlar da getirdik.
χCRAC kullanarak, yakın zamanda glikoz yoksunluğuna yanıt olarak bir maya nükleer RBP'si olan Nab3'ün hedefini inceledik. Saccharomyces cerevisiae'de Nab3, NNS kompleksini oluşturmak için Nrd1, bir RBP ve RNA helikaz Sen1 ile bir kompleks oluşturabilir. RNA polimerazına ve yeni ortaya çıkan transkripte NNS'nin bağlanması, transkripsiyonel sonlandırmayı tetikleyebilir34. Bu kompleks çoğunlukla şifreli kodlamayan RNA transkriptlerinin çıkarılmasında rol oynar, ancak protein kodlayan genlerin ekspresyonunu düzenlediği de gösterilmiştir. Çalışma, Nab3'ün sadece bir dakikalık stresten sonra kodlamayan ve kodlamayan transkriptlere diferansiyel hedeflemesini gösterdi32. Nab3 ile ko-transkripsiyonel sonlandırmanın, retrotranspozon genlerinin çok geçici, nabız benzeri bir ekspresyonu ile sonuçlandığını ve geleneksel CLIP tabanlı yaklaşımlar kullanılarak tespit edilmesinin zor olacağını gösterdik. Ek olarak, UV çapraz bağlayıcımızdaki kısa UV ışınlama süreleri, kısa ömürlü kodlamayan RNA'ların geri kazanımını da önemli ölçüde artırdı32. χCRAC muhtemelen RBP'lerin strese verilen tepkiyi anlık zaman ölçeklerinde nasıl şekillendirdiğini değil, aynı zamanda bir yanıtın tüm yaşam döngüsü boyunca değişen rollerini de aydınlatmada çok önemli bir araç olacaktır. Bu makale, χCRAC protokolündeki tüm adımlara ayrıntılı bir genel bakış sunmaktadır. Açıklayıcı amaçlar için, yöntem, transkripsiyonel sonlandırma ve RNA bozunumunda35,36 ile ilgili olan maya Nrd1 proteinini ve çok sayıda zaman noktasında glikoz yoksunluğuna yanıt olarak RNA hedefomunu incelemek için kullanıldı. Son olarak, UV ışınlama ünitemizin RBP'leri HeLa hücrelerinde RNA'ya hızlı bir şekilde çapraz bağlayabildiğini ve yapışkan hücrelerde yüksek çözünürlüklü zaman çözümlü analizler yapmayı mümkün kıldığını da gösteriyoruz.
Protokol
| TN150 |
| 50 mM Tris pH 7,8 |
| 150 mM NaCl |
| %0.1 NP-40 |
| 1X proteaz inhibitörü |
| TN1000 |
| 50 mM Tris pH 7,8 |
| 1M NaCl |
| %0.1 NP-40 |
| NP-PNK |
| 50 mM Tris-HCl pH 7,8 |
| 10 mM MgCl2 |
| %0.1 NP-40 |
| 5 mM beta-merkaptoetanol |
| 5 x PNK |
| 250 mM Tris-HCl pH 7,8 |
| 50 mM MgCl2 |
| 50 mM beta-merkaptoetanol |
| Dünya Bankası I |
| 50 mM Tris-HCl pH 7,8 |
| 300 mM NaCl |
| 10 mM imidazol |
| 6M guanidin-HCl |
| %0.1 NP-40 |
| 5 mM beta-merkaptoetanol |
| Dünya Bankası II |
| 50 mM Tris-HCl pH 7,8 |
| 50 mM NaCl |
| 10 mM imidazol |
| %0.1 NP-40 |
| 5 mM beta-merkaptoetanol |
| Elüsyon tamponu |
| 50 mM Tris pH 7,8 |
| 50 mM NaCl |
| 250 mM imidazol |
| %0.1 NP-40 |
| 5 mM beta-merkaptoetanol |
| Proteaz K tamponu |
| 50 mM Tris |
| %0.1 NP-40 |
| 5 mM β-merkaptoetanol |
| %1 SDS |
| 5 mM EDTA |
| 50 mM NaCl2 |
| Memeli lizis tamponu |
| 50 mM Tris-HCl pH 8 |
| 100 mM NaCl |
| %0,5 v/d Triton X-100 |
| Na-deoksikolat ile %0,25 |
| SDS olmadan %0,1 |
| 5 mM EDTA |
| 1 mM DTT (taze eklenmiş) |
| 1X proteaz inhibitörü |
Tablo 1: χCRAC ve bileşimleri için gerekli tamponlar.
1. UV çapraz bağlama ve lizat üretimi
- Çözeltideki mikroorganizmalar
- İstenilen ortamın 3.5 L'sini maya ile bir gecelik kültürden 0.05'lik bir başlangıç OD600'üne aşılayın. 180 rpm'de sürekli çalkalayarak 30 °C'de büyüyün.
- Büyüme sırasında, diğer gerekli malzemeleri hazırlayın.
- Bir sıvı azot kabı hazırlayın.
- Bir su banyosunda 3 L strese neden olan ortam hazırlayın ve 30 ° C'ye kadar ısıtın.
- Filtre aparatını kurun, çapraz bağlayıcıyı açın (Şekil 2A) ve her zaman noktası için bir tane olmak üzere 50 mL konik tüpleri etiketleyin.
- Hücreler istediğiniz OD 600'e ulaştığında,500 mL hücreleri doğrudan çapraz bağlayıcıya dökün ve 250 mJ 254 nm UV ile UV ışınlayın. Çapraz bağlayıcının kullanımıyla ilgili ayrıntılar için Şekil 2A ve Şekil 3A'ya bakın.
NOT: UV ışınlama enerjisi, ilgilenilen her protein için dikkatlice optimize edilmelidir. Daha fazla ayrıntı için Tartışma'ya bakın. - Çapraz bağladıktan sonra, vakum filtreleme cihazlarından birini kullanarak hücreleri filtreleyin (Şekil 2B, C). Membranı filtrelenmiş hücrelerle yuvarlayın, t = 0 (zaman sıfır) 50 mL konik tüpe yerleştirin ve sıvı azotta flaş dondurun.
- Kalan hücreleri altı farklı filtre üzerinden filtreleyin. Toplanan hücreleri, daha önce ısıtılmış strese neden olan ortamın 3 L'sinde, membranları ortama düşürerek ve 50 s boyunca bir stripetle kuvvetlice karıştırarak yeniden askıya alın. 50 s'den sonra, t = 1 örneğini almaya hazırlanın.
- 1 dakika sonra, 500 mL hücreleri çapraz bağlayın ve 1.1.3–1.1.4 adımlarında olduğu gibi filtreleme yoluyla hasat edin. 2, 4, 8, 14 ve 20 dakika sonra veya gerektiğinde farklı zaman noktalarında tekrarlayın.
- Hücreleri içeren konik tüpleri -80 °C'de saklayın. Fosfat tamponlu salini (PBS) gece boyunca 4 ° C'ye ayarlayın.
- Ertesi gün, çapraz bağlı bir numune içeren her konik tüpü alın ve hücreleri kuvvetlice sallayarak 25 mL soğuk PBS'de yeniden askıya alın.
- Hücre süspansiyonlarını yeni konik tüplere aktarın ve 4.600 x g'de, 4 ° C'de 5 dakikada döndürün.
- PBS'yi dökün, kalan PBS'yi toplamak için hızlıca tekrar döndürün ve ardından kalan sıvıyı bir pipetle boşaltın.
- Peletin tüpteki ağırlığını boş bir tüple karşılaştırarak hesaplayın.
- İki pelet hacminde buz gibi soğuk TN150, 60 μL DNaz 1 ve 10 μL RNaz inhibitörü ekleyin. 30 dakika boyunca buz üzerinde inkübe edin.
- Örneğin, 400 mg hücre için 800 μL buz gibi soğuk TN150 ekleyin.
- DNaz ilavesi, çoğu çözünür protein için gerekli değildir, ancak RNA polimeraz gibi kromatine bağlı proteinleri incelerken çok önemlidir. Ek olarak, bakteriyel lizatların viskozitesini azaltır. Lizis tamponunun tam olarak iki pelet hacminin kullanılması çok önemlidir, aksi takdirde lizis verimliliği düşebilir.
- Hücre süspansiyonuna üç pelet hacmi (mL cinsinden) zirkonya boncuğu ekleyin. Maya için 0,5 mm çapında boncuklar ve bakteriler için 0,1 mm kullanın.
- Örneğin, 400 mg hücre için, 1.5 mL'lik bir tüpte 1.2 mL zirkonya boncuklarını ölçün ve bunları lizis tamponunda yeniden askıya alınan hücrelere ekleyin.
- Hücre süspansiyonlarını 1 dakika boyunca vorteksleyin, ardından 1 dakika boyunca buzun üzerine yerleştirin. Toplam 5 kat tekrarlayın.
- İki pelet hacimli TN150 tamponu ekleyin ve karıştırmak için kuvvetlice vorteks ekleyin.
- Konik borudaki süspansiyonu 4.600 g'da 4 °C'de 20 dakika boyunca bir tezgah üstü santrifüjde santrifüjleyin.
- Santrifüjlemeden sonra, tüm hücre protein ekspresyonunu incelemek için gelecekteki Western blot analizi için süpernatantın 50 μL'lik bir örneğini alın.
- Süpernatantları 1.5 mL tüplere aktarın ve lizatı 20 dakika boyunca 4 ° C'de 20.000 x g'de bir mikrofüj içinde döndürün.
- Alternatif olarak, 5 mL tüp kullanıyorsanız, 20 dakika boyunca 13.000 x g'de santrifüj yapın.
- Santrifüjlemeyi takiben, proteinin çözünür ekspresyonunu incelemek için gelecekteki Western blot analizi için süpernatantın 50 μL'lik bir örneğini alın.
- RBP yakalamaya geçin (bölüm 2).
- Kültürlenmiş yapışkan hücreler
- UV çapraz bağlamadan 24 saat önce bir Petri kabına yeterince yapışkan hücre tohumlayın, böylece ertesi gün% 80 akıcılığa ulaşabilirler. İstenilen ortamda bir hücre kültürü inkübatöründe 37 ° C,% 5 CO2'de bir gecede büyütün.
NOT: Kuvars Petri kapları kullanılıyorsa, tohumlamadan 2.5 saat önce kültür kaplarının poli-D-lizin (70.000-140.000 ağırlık) ve fetal buzağı serumu (FCS) ile muamele edilmesi yoluyla hücre yapışmasını teşvik etmek faydalıdır. Tüm büyüme yüzeyini kaplayacak kadar poli-D-lizin ekleyin ve 5 dakika boyunca oda sıcaklığında (RT) inkübe edin. Daha sonra, kuvars Petri kabı su ile iyice durulanmalı ve hücre kültürü inkübatöründe 2 saat veya tamamen kuruyana kadar kurutulmalıdır. Daha sonra, büyüme yüzeyini tamamen kaplayacak kadar FCS ekleyin ve inkübatöre en az 30 dakika boyunca yerleştirin. FCS, hücreleri tohumlamadan önce tamamen çıkarılmalıdır. - Hücreler% 80 akıcılığa ulaştığında, ortamı çıkarın ve 15 mL buz gibi soğuk PBS ile yıkayın. Ardından, kalan tüm sıvıyı tamamen çıkarın ve hemen bir sonraki adıma geçin.
- Petri kabını yapışkan hücreler için tepsiye aktarın (Şekil 3B) ve 300 mJ 254 nm UV ile UV ışınlayın. Çapraz bağlayıcının kullanımıyla ilgili ayrıntılar için Şekil 2A ve Şekil 3B'ye bakın.
NOT: UV ışınlama enerjisi, ilgilenilen her protein için dikkatlice optimize edilmelidir. Daha fazla ayrıntı için Tartışma'ya bakın. - Çapraz bağlamadan hemen sonra, Petri kabını buzun üzerine yerleştirin ve 10 mL buz gibi soğuk PBS ekleyin. Hücreleri kazıyarak toplayın ve 15 mL'lik bir konik tüpe aktarın. 4 °C'de 5 dakika boyunca 300 x g'de santrifüjleme yoluyla pelet.
- PBS'yi çıkarın ve hücre peletini 1 mL buz gibi soğuk PBS'de yeniden askıya alın ve 1,5 mL'lik bir mikrosantrifüj tüpüne aktarın. Pelet hücreleri 4 °C'de 300 x g'de 5 dakika santrifüjleme ile tekrar santrifüjlenir.
- PBS'yi çıkarın ve hücre topaklarını kuru buz üzerinde dondurun. Hücre peletlerini gerekli olana kadar -80 ° C'de saklayın.
- Her zaman noktası için 1.2.3–1.2.6 arasındaki adımları yineleyin.
- Hücre peletlerini 1 mL lizis tamponunda tekrar askıya alın ve 15 mL'lik bir konik tüpe aktarın. Daha sonra, toplam 2 mL için 1 mL lizis tamponu ekleyin.
- 5 μL memeli RNaz inhibitörü ekleyin.
- 5 amperde buz üzerinde 10 s için 10x sonikasyon. Sonikasyon turları arasında 30 s bekleyin.
- Her numunenin protein konsantrasyonunu hesaplayın ve en düşük konsantrasyona normalleştirin.
- 1.98 mL lizatı 2 mL'lik bir tüpe aktarın.
- 10 μL DNaz I ekleyin ve 1.200 rpm'de çalkalayarak 5 dakika boyunca 37 ° C'de inkübe edin.
- Lizatı 16.000 x g'de 4 ° C'de 20 dakika boyunca santrifüj edin.
- Santrifüjlemeden sonra, proteinin çözünür ekspresyonunu incelemek için gelecekteki Western blot analizi için süpernatantın 50 μL'lik bir örneğini alın.
- RBP yakalamaya geçin (bölüm 2).
- UV çapraz bağlamadan 24 saat önce bir Petri kabına yeterince yapışkan hücre tohumlayın, böylece ertesi gün% 80 akıcılığa ulaşabilirler. İstenilen ortamda bir hücre kültürü inkübatöründe 37 ° C,% 5 CO2'de bir gecede büyütün.
2. RBP yakalama
- Manyetik anti-FLAG (numune başına 75 μL bulamaç) veya IgG agaroz (numune başına 500 μL bulamaç) boncuklarını 5 mL TN150 ile 3 kat yıkayın. 700 μL TN150'nin son hacminde tekrar askıya alın ve yedi adet 15 mL konik tüpe 100 μL yıkanmış boncuk ekleyin.
- Gerekli olana kadar buz üzerinde saklayın.
- Lizatlar netleştirildikten sonra, süpernatantı anti-FLAG / IgG boncuklarını içeren tüpe ekleyin.
- 2 saat boyunca 4 ° C'de nutat.
NOT: Bazı protokoller boncuklarla gece boyunca inkübasyonları tanımlar, ancak bu önerilmez, çünkü uzun inkübasyon süreleri çapraz bağlı RNA'ların geri kazanımını önemli ölçüde azaltabilir.
3. Boncukların yıkanması ve etiketlerin TEV bölünmesi
- Boncukları toplayın ve lizatı çıkarın.
- Yakalanmamış proteini incelemek için gelecekteki Western leke analizi için süpernatantın 50 μL'lik bir örneğini alın.
- Boncukları buz gibi soğuk TN1000'de tekrar askıya alın ve 1,5 mL'lik bir tüpe aktarın. Somunlama ile 10 dakika, 4 °C yıkayın. Toplam üç yıkama için tekrarlayın.
- IgG agaroz boncukları kullanıyorsanız, 5 mL TN1000 ile yıkayın. Anti-FLAG boncuk kullanıyorsanız, 2 mL kullanın.
- Ardından, boncukları TN150 ile 3x yıkayın, yukarıdakiyle aynı hacimde.
- Üçüncü yıkamadan sonra, boncukları 600 μL TN150'de tekrar askıya alın.
- Boncuk süspansiyonuna 30 U ev yapımı GST-TEV proteaz ekleyin ve RT'de 2 saat döndürün.
NOT: Rekombinant GST-TEV proteaz artık ticari olarak da temin edilebilir, ancak bu protokolle test edilmemiştir.- Sindirim sırasında, her numune için üç adet 1,5 mL tüpten oluşan sütunlar ayarlayarak sonraki adımlara hazırlanın (yani yedi numune için, yedi sütundan oluşan üç sıra bulunmalıdır).
- Son tüp sırasına, 0.4 g guanidyum hidroklorür, 27 μL 5M sodyum klorür ve 3 μL 2.5 M imidazol (pH = 8) ekleyin. İmidazolün pH'ının 8 olması gerektiğini unutmayın. Bu, RNA bütünlüğünü korumak için kritik öneme sahiptir.
- Ek olarak, gerekli miktarda nikel boncuğu WB I 3x'te yıkayın. Numune başına 100 μL bulamaç kullanın. Son yıkamadan sonra, boncukları WB I'in aynı orijinal hacminde tekrar askıya alın ve buz üzerinde saklayın.
- TEV sindirimi tamamlandıktan sonra, anti-FLAG boncukları için manyetik bir raf veya IgG boncukları için santrifüjleme kullanarak süpernatantı toplayın ve daha önce kurulmuş tüplerin ilk sırasına aktarın.
- Western blot analizi için 50 μL'lik bir TEV elüat örneği alın.
- Bir termoblok inkübatörü 37 ° C'ye ayarlayın. İkinci tüp sırasına 1 μL RNaz kokteyli ekleyin (1:50 seyreltme).
- İlk tüp sırasından 550 μL TEV elüatını alın ve ikinci sıraya ekleyin (RNase kokteylini içeren). Karıştırmayı sağlamak için kuvvetli pipet.
- İlk numune için bunu tamamladıktan sonra, tüpü hemen termobloğa yerleştirin ve bir zamanlayıcı başlatın. Her biri kademelendirilecek şekilde sonraki örneklere geçin.
- Tam olarak 5 dakika boyunca inkübe edin. Tamamlandığında, ilk numuneyi termobloktan çıkarın ve çözeltiyi üçüncü tüp sırasına (guanidium hidroklorür tozu içeren) aktarın.
NOT: RNaz kokteylinin 1:50 seyreltilmesi ile 5 dakikalık bir inkübasyon genellikle çoğu protein için uygundur, ancak çapraz bağlı RNA'ların doğru boyutta (30-100 nt) olduğundan emin olmak için bu adımın her protein için farklı inkübasyon süreleri veya konsantrasyonları ile dikkatlice optimize edilmesi gerekecektir. - Guanidyum tozunu çözmek için hemen birkaç saniye boyunca tam hızda vorteks yapın ve ardından bir sonraki numuneye geçin.
- Tüm numuneler guanidium tozuna aktarıldıktan sonra, tüm tozun tamamen çözünmesini sağlamak için tekrar girdap.
- 100 μL yıkanmış nikel boncuk ekleyin ve gece boyunca 4 ° C'de döndürün. Bu inkübasyon 2 saate kadar kısaltılabilir.
4. Boncuk üzerinde alkalen fosfataz tedavisi
- Bir termobloğu 37 °C'ye ayarlayın.
- Her numune için bir tane olmak üzere 2 mL'lik bir tüpe bir saflaştırma spin sütunu yerleştirin. Nikel boncuklarını sütunlara aktarın ve süpernatantın akmasına izin verin. Daha sonra, WB I ile durulayarak ve kolona uygulayarak tüm nikel boncuklarının 1,5 mL tüpten çıkarıldığından emin olun.
- Numune başına altı tane olmak üzere 2 mL tüp ayarlayın (her yıkamayı toplamak için bir tane). Akışı korumak için kolonların dışını kuru tutun. Boncukları 3 kez 500 μL WB I ile ve ardından 3 kez 500 μL NP-PNK ile yıkayın.
- Sıkma sütununun kapağını kapatın ve fazla tamponu çıkarmak için boncukları kısaca döndürün.
- Durdurucuyu kolonun üzerine koyun, sütunları 1,5 mL'lik tüplere koyun ve Tablo 2'de görülen reaksiyon karışımının 60 μL'sini ekleyin.
| Parça | 1 adet | 7,5 kat |
| 5 x PNK tampon belleği | 12 | 90 |
| Alkalen fosfataz | 4 | 30 |
| RNaz inhibitörü | 2 | 15 |
| H2O | 42 | 315 |
| Son cilt | 60 μL | 450 μL |
Tablo 2: Alkalen fosfataz reaksiyon karışımı.
- Boncukları 37 ° C'de 1 saat inkübe edin.
- Alkalen fosfatazı etkisiz hale getirmek için boncukları 1x 500 μL WB I ile ve daha sonra 3x'i 500 μL NP-PNK tamponu ile yıkayın. Guanidyum izlerini gidermek için kolonun içini NP-PNK tamponu ile iyice duruladığınızdan emin olun.
5. App-PE bağlayıcısının RNA'nın 3' ucuna boncuk üzerinde bağlanması
- Kalan arabelleği döndürün ve sütunlara Tablo 3'te belirtilen karışımın 60 μL'sini ekleyin (App-PE dizisi için Tablo 4'e bakın). Reaksiyonu 25 ° C'de 6 saat boyunca inkübe edin.
| Parça | 1 adet | 7,5 kat |
| 5 x PNK tampon belleği | 12 | 90 |
| App-PE adaptörü (100 μM) | 0.6 | 4.5 |
| T4 RNA ligaz 2 kesilmiş K227Q | 3 | 22.5 |
| RNaz inhibitörü | 1.5 | 11.25 |
| %50 PEG 8000 | 12 | 90 |
| H2O | 30.9 | 231.75 |
| Son cilt | 60 μL | 450 μL |
Tablo 3: App-PE bağlayıcı ligasyon reaksiyonu karışımı.
| Oligonükleotid adı | Sıra (5'-3') | |||
| L5Aa | invddT-ACACrGrArCrGrCrUrUrCrCrCrGrArUrUrNrNrNrUrArGrCrN-OH | |||
| L5Ab | invddT-ACACrGrArCrGrCrCrUrUrCrCrGrArUrUrNrNrNrArUrArGrCrN-OH | |||
| L5Ac | invddT-ACACrGrArCrGrCrUrUrCrCrCrGrArUrUrNrNrGrCrGrGrCrArGrCrNcrN-OH | |||
| L5Ad | invddT-ACACrGrArCrGrCrCrUrUrCrCrGrArUrUrNrNrNrCrGrCrUrUrArGrCrN-OH | |||
| L5Ba | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrNrArGrArGrGrCrN-OH | |||
| L5Bb | invddT-ACACrGrArCrGrCrUrCrUrCrCrArUrUrNrNrGrUrGrArGrGrNr-OH | |||
| L5Bc | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrArUrUrNrNrNrCrArCrUrArGrCrN-OH | |||
| L5Bd | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrArUrUrNrNrNrUrCrUrGrCrN-OH | |||
| L5Ca | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrNrCrUrArGrCrN-OH | |||
| L5Cb | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrNrUrGrGrArGrCrN-OH | |||
| L5CC | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrArUrUrNrNrNrArCrUrCrArGrCrN-OH | |||
| L5Cd | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrArUrUrNrNrGrArCrUrUrArGrCrN-OH | |||
| L5Da | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrNrCrGrUrGrArUrN-OH | |||
| L5Db | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrGrCrArCrCrUrArN-OH | |||
| L5Dc | invddT-ACACrGrArCrGrCrUrUrCrCrCrGrArUrUrNrNrNrUrArGrUrGrCrN-OH | |||
| L5Dd | invddT-ACACrGrArCrGrCrUrUrUrCrCrGrArUrUrNrNrNrArUrCrArCrArCrGrN-OH | |||
| L5Ea | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrNrCrArCrUrGrUrN-OH | |||
| L5Eb | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrGrUrGrArCrArN-OH | |||
| L5Ec | invddT-ACACrGrArCrGrCrCrUrUrCrCrCrArUrUrNrNrNrUrGrUrCrArCrN-OH | |||
| L5Ed | invddT-ACACrGrArCrGrCrUrCrUrCrCrGrArUrUrNrNrNrArCrArGrUrGrN-OH | |||
| App_PE | App-NAGATCGGAAGAGCACACGTCTG-ddC | |||
Tablo 4: Yakalanan RNA'ların 5' ve 3' uçlarına bağlanma için gerekli DNA ve RNA adaptörlerinin dizileri. Bunlar RNase içermeyen HPLC ile saflaştırıldı.
- Boncukları 1x'i 500 μL WB I ve 3x'i 500 μL NP-PNK tamponu ile yıkayın. Kolonu yeni bir tüpe koyun ve kalan tamponu döndürün.
6. RNA'nın 5' uçlarının boncuk üzerinde fosforilasyonu
- Tablolara Tablo 5'te belirtilen karışımın 80 μL'sini ekleyin. Reaksiyonu 37 ° C'de 40 dakika boyunca inkübe edin.
NOT: Numuneler artık oldukça radyoaktif olacaktır. Bu nedenle, sonraki tüm çalışmalar koruyucu bir ekranın arkasında yapılmalı ve atıklar yerel sağlık ve güvenlik kurallarına göre bertaraf edilmelidir.
| Parça | 1 adet | 7,5 kat |
| 5 x PNK tampon belleği | 16 | 120 |
| 32P-ɣATP (10 μCi/μL) | 3 | 22.5 |
| T4 PNK | 3 | 22.5 |
| H2O | 58 | 435 |
| Son cilt | 80 μL | 600 μL |
Tablo 5: Fosforilasyon reaksiyonu karışımı.
- 1 μL 100 mM ATP ekleyin ve reaksiyonun 20 dakika daha devam etmesine izin verin. Bu, 5' bağlayıcının bağlanmasını kolaylaştırmak için neredeyse tüm 5' uçlarının fosfatlara sahip olmasını sağlayacaktır.
- Numune başına beş tane olmak üzere 2 mL tüp ayarlayın.
- Boncukları 1x'i 500 μL WB I ve 3x'i 500 μL NP-PNK tamponu ile yıkayın. Bu salınımların çok radyoaktif olacağını ve bu nedenle uygun şekilde bertaraf edilmesi gerektiğini unutmayın.
- Kolonu son tüpe taşıyın ve kalan arabelleği döndürün.
7. 5' bağlayıcının boncuk üzerinde bağlanması
NOT: 5' bağlayıcılar, dizilemeden sonra her bir numunenin tanımlanması için kullanılan bir RNA barkodu içerir. Bu nedenle, hangi bağlayıcının hangi numune için kullanıldığını not etmek kesinlikle çok önemlidir.
- Sütunlara Tablo 6'da açıklanan karışımın 78 μL'sini ekleyin. Her tüpe 2 μL 5' adaptör (100 μM; bakınız Tablo 4) ekleyin ve gece boyunca 18 °C'de inkübe edin.
| Parça | 1 adet | 7,5 kat |
| 5 x PNK tampon belleği | 16 | 120 |
| ATP (10 mM) | 8 | 60 |
| RNaz inhibitörü | 2 | 15 |
| T4 RNA ligaz | 4 | 30 |
| H2O | 48 | 360 |
| Son cilt | 78 μL | 585 μL |
Tablo 6: 5' bağlayıcı ligasyon reaksiyonu karışımı.
- Ertesi gün, boncukları 1x'i 500 μL WB I ve 3x'i 500 μL WB II ile yıkayın ve kolonları yeni bir 2 mL tüpe aktarın.
8. Elüsyon, SDS-PAGE ve RNA ekstraksiyonu
- Santrifüjü 4 °C'ye ayarlayın. Elüsyon için numune başına iki sıra 1,5 mL tüp hazırlayın.
- Sütunların boşluk hacmini nikel boncuklarla hızlı bir şekilde döndürün. Sütunları elüsyon tüplerinin ilk sırasına yerleştirin ve 200 μL elüsyon tamponu ekleyin. 2 dakika bekleyin, ardından tamponu hızlı bir dönüşle sütundan geçirin.
- Sütunları ikinci tüp sırasına taşıyın ve adım 8.2'yi yineleyin. Her numune şimdi iki adet 1,5 mL tüpe bölünmüş olarak toplamda 400 μL elüata sahip olacaktır.
- Tüm elüatları alın ve birlikte 5 mL'lik bir tüpe aktarın. 2 μL 20 mg/mL glikojen ekleyin. Böylece, yedi numune kullanılıyorsa, şimdi 5 mL tüpte 2.8 mL havuzlanmış elüat olacaktır.
- 5 mL tüpe numune başına 100 μL trikloroasetik asit (TCA) ekleyin [örneğin, 7 numune için 700 μL TCA (2,8 mL havuzlanmış elüat)] ve 30 s boyunca vorteks kuyusu.
- 20 dakika boyunca buz üzerinde inkübe edin.
- Bir tezgah üstü santrifüjde 17.000 x g, 4 °C'de 30 dakika boyunca santrifüj.
- Peletin yanlışlıkla çıkarılmadığından emin olmak için pipeti bir Geiger sayacı ile kontrol ederek süpernatantı konik tüpten dikkatlice çıkarın. Varsa, süpernatantı tüpe geri döndürün ve 10 dakika daha santrifüj yapın.
NOT: Süpernatant hala yüksek oranda radyoaktif olabilir. Doğru ekranlama kullandığınızdan emin olun. - Peletleri 2 mL buz gibi soğuk asetonda tamamen askıya alın.
- 17.000 x g, 4 °C'de 15 dakika boyunca santrifüj.
- Asetonun mümkün olduğunca çoğunu P1000 pipetle çıkarın. Daha sonra, küçük aseton damlacıkları toplamak için tüpü kısaca döndürün ve ardından bir P10 pipet ile çıkarın. Bir duman davlumbazında 2 dakika kurutun.
NOT: Aseton süpernatant hala radyoaktif olabilir. Doğru ekranlama kullandığınızdan emin olun. - Numuneyi 30 μL 1x protein yükleme tamponunda tekrar askıya alın. Peletin düzgün bir şekilde yeniden askıya alındığından emin olmak için, bir P200 pipetindeki çözeltiyi çıkararak ve bir Geiger sayacı kullanarak 1,5 mL tüpte kalan aktiviteyi ölçerek, radyoaktivitenin büyük çoğunluğunun yükleme tamponunda mevcut olup olmadığını ve 1,5 mL tüpte bırakılmadığını kontrol edin.
- Numuneyi 65 °C'de 10 dakika ısıtın. 1 mm, %4-12 prekast Bis-Tris jel üzerine yükleyin ve MOPS tamponunda 125 V'ta 1,5 saat çalıştırın.
- Jel çalışmayı bitirdikten sonra, jel kasetini açın. Jel alt plakada tutulmalıdır. Üstünü atın.
- Jeli streç filme sarın ve ardından bant kullanarak hafif geçirmez bir kasetin içine sabitleyin. Sinyali iyileştirmek için kasetin yükseltici bir ekrana sahip olduğundan emin olun.
- Otoradyografik bir filmi jele maruz bırakın ve pozlama sırasında kaseti -80 ° C'de saklayın. Maruz kalma süresi, farklı çapraz bağlanma verimliliklerine sahip proteinler arasında değişecektir.
- Filmi yerleştirirken, bir sonraki adımda ilgilenilen bandı kesmek için kasete yeniden hizalamanın bir yolu olmalıdır. Bunu sağlamak için, bir floresan cetvel kullanın ve ayrıca jelin kasetin bir köşesinde olduğundan emin olun, bu da en üst köşeye yerleştirilen film tarafından kaplanır.
NOT: Genel bir kural olarak, bir Geiger sayacında görüntülendiğinde en az ~ 250 cps'lik bir okuma veren yükleme tamponundaki elüeler, 3 saatlik bir pozlama için yeterli sinyal verir. Aksi takdirde, gece boyunca maruz kalma gerçekleştirilir.
- Filmi yerleştirirken, bir sonraki adımda ilgilenilen bandı kesmek için kasete yeniden hizalamanın bir yolu olmalıdır. Bunu sağlamak için, bir floresan cetvel kullanın ve ayrıca jelin kasetin bir köşesinde olduğundan emin olun, bu da en üst köşeye yerleştirilen film tarafından kaplanır.
- Filmi geliştirin. Jeli kaplayan streç filmi kesin, ancak jeli hareket ettirmeyin. Aksi takdirde, görüntü jelden dengelenecektir.
NOT: Jel muhtemelen oldukça radyoaktif olacaktır. Jel dilimini keserken uygun ekranlama kullandığınızdan emin olun. - Filmi jelin üzerine yerleştirin ve ilgilendiğiniz bandı tüketin. Jel dilimini 2 mL'lik bir tüpe koyun.
- Bir P1000 pipet ucu kullanarak jel dilimini parçalayın ve 600 μL proteinaz K tamponu artı 200 μg proteinaz K ekleyin (bu protokol 20 mg / mL proteinaz K çözeltisinin 10 μL'sini kullanır). Kuvvetli çalkalama ile 55 ° C'de 2 saat inkübe edin.
- Daha sonra, bir P1000 ucunun ucunu temiz bir neşterle kesin ve süpernatant ve jel parçalarını 2 mL'lik bir tüpe yerleştirilmiş bir spin sütununa aktarın.
- Sütunu RT'de 17.000 x g'de 1 dakika döndürün. radyoaktif, izole RNA'ları içeren akışı toplayın.
- Bir fenol:kloroform ekstraksiyonu gerçekleştirin.
- 50 μL 3 M sodyum asetat, pH = 5.2 ve 500 μL fenol: kloroform ve vorteks iyice ekleyin. 17.000 x g'de 5 dakika döndürün. Sulu üst tabakayı çıkarın ve yeni bir 1,5 mL tüpe yerleştirin.
- 10-15 s boyunca kuvvetli bir şekilde 500 μL kloroform ve vorteks ekleyin. RT'de 17.000 x g'de 5 dakika döndürün. sulu tabakayı çıkarın ve yeni bir 1.5 mL tüpe yerleştirin.
- 1 μL 20 mg / mL glikojen ve 1 mL buz gibi soğuk,% 96 etanol ekleyin. -80 °C'de 30 dakika veya -20 °C'de gece boyunca çökeltin.
- 4 °C'de 30 dakika boyunca santrifüj, 17.000 x g. Süpernatanı çıkarın, 500 μL% 70 etanol ekleyin ve 5 dakika, 17.000 x g'de 4 ° C'de santrifüj yapın. Tüm etanolleri çıkarın, kalıntı toplamak için hızlı bir dönüş yapın ve fazlalığı bir P10 pipet ile giderin.
- Peletleri bir duman davlumbazında ~ 3 dakika kurutun. 20 μL DEPC ile arıtılmış suda tekrar askıya alın.
- RNA'yı gece boyunca -80 ° C'de saklayın veya hemen ters transkripsiyon adımına geçin.
9. Ters transkripsiyon
- 20 μL RNA'ya 2 μL 10 μM RT oligo (PE_reverse; bakınız Tablo 7) ve 4 μL 5 mM dNTP ekleyin.
| Oligonükleotid adı | Sıra (5'-3') | |||
| P5 ileri | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT | |||
| MÖ1 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| M.Ö. 3 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| MÖ4 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| MÖ5 | CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| MÖ7 | CAAGCAGAAGACGGCATACGAGATCAGATCGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| MÖ8 | CAAGCAGAAGACGGCATACGAGATTAGCTTGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| MÖ9 | CAAGCAGAAGACGGCATACGAGATGATCAGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| MÖ10 | CAAGCAGAAGACGGCATACGAGATATCACGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| PE_reverse | CAGACGTGTGCTCTTCCGATCT | |||
Tablo 7: PCR primerleri (barkod dizileri dahil) ve ters transkripsiyon astarı.
- 3 dakika boyunca 85 ° C'de önceden ısıtılmış bir termobloğa aktarın, ardından 5 dakika boyunca buz üzerinde soğutun. Tüpün içeriğini kısa santrifüjleme ile toplayın ve ardından 8 μL 5x ters transkriptaz tamponu, 2 μL 100 mM DTT ve 2 μL RNaz inhibitörü ekleyin.
- Karışımı 50 ° C'de 3 dakika boyunca inkübe edin ve ardından 2 μL ters transkriptaz ekleyin ve 50 ° C'de 1 saat inkübe edin.
- Ters transkriptazı 15 dakika boyunca 65 ° C'de inkübasyonla inaktive edin.
- Tüpleri 37 ° C'de önceden ısıtılmış bir termobloğa aktarın ve iklimlendirmek için 3 dakika bekletin.
- 2 μL RNase H ekleyin ve 37 ° C'de 30 dakika inkübe edin.
- SPRI boncukları kullanarak cDNA'yı izole edin.
- İki hacim 84 μL boncuk ekleyin. 15 dakika boyunca inkübe edin. Boncukları manyetik bir rafa koyun ve boncukları hasat etmek için 1 dakika bekletin.
- Süpernatantı çıkarın ve atın ve 200 μL% 70 etanol ekleyin. Boncukları manyetik raftan çıkarmayın. Boncukları 30 s boyunca etanol ile inkübe edin.
- Etanolün çıkarılması ve yıkama adımının tekrarlanması gerekir. Bir P10 ucu kullanarak tüm artık etanolleri çıkarın.
- Boncukları kurutmak için 2 dakika boyunca bir duman davlumbazına koyun. Boncukları raftan çıkarın, 12 μL suda tekrar askıya alın ve ardından boncukları tekrar rafa koyun. 11 μL süpernatanı çıkarın.
- cDNA'yı -20 ° C'de dondurun veya hemen PCR adımına geçin.
10. qPCR reaksiyonu
- cDNA'ların amplifikasyonu için son PCR'den önce, kütüphanenin aşırı amplifikasyonunu önlemek için cDNA'ları yükseltmek için en uygun döngü sayısını belirlemek için kantitatif bir polimeraz zincir reaksiyonu (qPCR) gerçekleştirilir.
- Tablo 8'e göre buz üzerinde bir qPCR reaksiyonu ayarlayın. Tüm astarlar için Tablo 7'ye bakınız.
| Parça | 1 adet |
| 2x qPCR reaksiyon mastermix | 5 |
| 0,1 μM P5 astar (ileri) | 0.8 |
| 0,1 μM BC astar (ters) | 0.8 |
| cDNA (veya negatif kontrol olarak su) | 1 |
| H2O | 2.4 |
| Son cilt | 10 μL |
Tablo 8: qPCR reaksiyon karışımı.
- Amplifikasyon için gerekli döngülerin doğru bir şekilde ölçülmesi için, cDNA için üç teknik kopya ve üç negatif (yani su) kontrol kullanın.
- Plakaları optik olarak şeffaf filmle kapatın ve qPCR'yi kit üreticisinin talimatlarına göre çalıştırın.
- Üstel büyümenin dizine ulaşılan döngü sayısını (n) belirlemek için numuneleri mutlak bir niceleme yöntemiyle analiz edin (bir örnek için bkz. Şekil 4C ). Bu döngü sayısı daha sonra cDNA'nın geri kalanının son amplifikasyonu için kullanılır.
11. PCR reaksiyonu ve jel ekstraksiyonu
- PCR reaksiyonunu buz üzerinde Tablo 9'a göre ayarlayın. Tüm astarlar için Tablo 7'ye bakınız.
NOT: cDNA kütüphanesinin sadece 5 μL'si kullanılır.
| Parça | 1 adet |
| 10 x prova okuma polimeraz tamponu | 5 |
| 10 μM P5 astar (ileri) | 1 |
| 10 μM BC astar (ters) | 1 |
| 5 mM dNTP | 2.5 |
| Prova okuma polimeraz enzimi | 1 |
| cDNA | 5 |
| H2O | 34.5 |
| Son cilt | 50 μL |
Tablo 9: PCR reaksiyon karışımı.
- PCR'yi aşağıdaki gibi çalıştırın: 2 dakika boyunca 95 ° C; 20 s için 98 °C, 30 s için 52 °C ve 1 dakika boyunca 72 °C n döngü; ve 5 dakika boyunca 72 ° C. χCRAC kütüphanesini yükseltmek için döngülerin sayısı (n), bölüm 10'da açıklanan qPCR tarafından belirlenir.
- 1 μL ekzonükleaz I ekleyin ve 60 dakika boyunca 37 ° C'de inkübe edin.
- Yükseltilmiş cDNA'yı, yukarıda açıklandığı gibi SPRI boncuklarını kullanarak iki hacim boncuk (yani 100 μL) kullanarak temizleyin. 11 μL'de elute.
- 3 μL 6x yükleme boyası ekleyin ve 1x TBE tamponunda 1 saat boyunca 100 V'ta prekast %6 TBE jel üzerinde çalıştırın. Kısa DNA parçalarının nicelleştirilmesi için uygun bir merdiven kullanın.
- İşiniz bittiğinde, jeli kasetten çıkarın ve jeli örtmek için yeterli 1x TBE içeren uygun, sıvı geçirmez bir kaba koyun (örneğin ~ 50 mL). Uygun miktarda SYBR güvenli boya ekleyin (örneğin, 50 mL için, 10.000x boyadan 5 μL kullanın)
- Jelin RT'de 15 dakika boyunca nazik karıştırma yoluyla lekelenmesine izin verin. SYBR içeren 1x TBE'yi boşaltın ve temiz 1x TBE ile değiştirin. Jeli RT'de hafifçe çalkalayarak 10 dakika yıkayın.
- 1x TBE'yi boşaltın ve jeli şeffaf bir klasöre yerleştirin. Klasörü uygun bir boyuta kesin.
- Jeli fosforgörüntüleyici gibi uygun bir yolla görüntüleyin. DNA parçalarını ~175 bp ile ~400 bp arasında tüketin. Jel dilimini 1,5 mL'lik bir tüpe koyun.
- Bir P1000 ucu kullanarak jel dilimini iyice ezin ve 400 μLH2O ekleyin Bir termoblokta 1 saat çalkalayarak 37 ° C'de inkübe edin.
- Numuneyi kuru buz üzerinde 10 dakika dondurun, ardından termobloğa 37 ° C'de 1 saat çalkalayarak tekrar yerleştirin.
- Bir filtre sütunu alarak ve içine iki cam mikrofiber filtre yerleştirerek bir filtre ünitesi oluşturun. Üniteyi 1,5 mL'lik bir tüpe yerleştirin.
- Bir P1000 ucunun ucunu temiz bir neşterle kesin ve parçalanmış TBE jel süspansiyonunu alın, ardından adım 11.12'de oluşturulan filtre ünitesine dağıtın. 30 s boyunca 17.000 x g'de döndürün.
- Süpernatana 1 μL glikojen, 40 μL sodyum asetat, pH = 5.2 ve 1 mL% 96 etanol ekleyin. -80 ° C'de 30 dakika boyunca inkübe edin.
- 17.000 x g, 4 °C'de 30 dakika boyunca santrifüj. Süpernatantı atın ve 500 μL% 70 etanol ile yıkayın.
- 5 dakika döndürün, etanolün tamamen çıkarın ve ardından peleti 3 dakika boyunca bir duman davlumbazında kurutun.
- 10 μLH2O içinde tekrar askıya alın ve DNA konsantrasyonunu ölçün.
Sonuçlar
χCRAC yönteminin etkinliğini göstermek için, HTP etiketli bir Nrd1 proteinini eksprese eden maya suşları ile bir zaman kursu deneyi yapıldı. Yöntemin nasıl çalıştığını açıklayan ayrıntılı bir şematik gösterim Şekil 1'de verilmiştir. Nab3 gibi, Nrd1 de çeşitli RNA transkriptlerinin nükleer RNA bozunumunda rol oynar37. Corden laboratuvarından yapılan önceki çalışmalar, RNA hedeflerine Nrd1 bağlanmasının, hücreler glikoz açlığına maruz kaldığında önemli ölçüde değiştiğini öne sürdü28,38. Bu nedenle, glikoz içeren ortamda (SD-TRP) katlanarak büyüyen hücreler, Nrd1-RNA etkileşimlerindeki dinamik değişiklikleri izlemek için bir zaman boyunca glikoz (S-TRP) olmadan aynı ortama kaydırıldı. Numuneler vardiyadan önce Vari-X-bağlayıcı odasında (Şekil 3A) alındı ve çapraz bağlandı ve daha sonra 1, 2, 4, 8, 14 ve 20 dakika sonra bağlandı. Hücre büyümesi için kullanılan ortam, bu aromatik amino asit tarafından UV emilimini azaltmak için triptofanda kasıtlı olarak eksikti. Filtreyle sterilize edilmiş sentetik ortamın kullanılmasının en iyisi olduğunu unutmayın, çünkü ortamın otoklavlanması şekerlerin karamelizasyonuna yol açabilir. Bu daha sonra çapraz bağlama verimliliğini azaltır.
Şekil 4A , χCRAC deneyinden temsili bir otoradyografi göstermektedir. Bu örnekte, örneklerin birlikte havuza alınmadığını unutmayın. Bunun yerine, her biri jel üzerinde ayrı ayrı çalıştırıldı. Bu, proteinin test edilen tüm zaman noktalarında RNA'ya etkili bir şekilde çapraz bağlandığını göstermek için ilk deneysel testler için önerilir. RBP'nin beklenen moleküler ağırlığında, dizileme için uygun olmayan çok kısa, radyoaktif işaretli RNA'lara bağlı proteini temsil eden özellikle yoğun bir sinyal gözlendi. Bu nedenle, daha uzun RNA fragmanlarına çapraz bağlanmış protein olan bu bandın üzerindeki yayma sinyali izole edildi. Parça, protein bandının hemen üstünden artı yaklaşık 30 kDa'dan kesildi. Şekil 4B , eksizyonu takiben bir otoradyogramı göstermektedir, protein jelde kalan kısa RNA'lara çapraz bağlanmıştır ve daha önce yayılmış olan sinyal şimdi eksize edilmiştir.
Ters transkripsiyondan sonra, cDNA kütüphanesi PCR kullanılarak güçlendirilmelidir. Bununla birlikte, kütüphanenin aşırı amplifikasyonundan kaçınılmalıdır, çünkü bu, polimeraz tarafından tercihen çoğaltılan dizilere karşı önyargı oluşturabilir ve PCR artefaktları üretebilir. Aşırı güçlendirilmiş kütüphaneler ayrıca sıralayıcıda okunan çok sayıda yinelenen dizi içerir. Son kütüphanenin amplifikasyonu için ideal PCR döngüsü sayısını hesaplamak için, cDNA'nın bir alikotu P5 ve BC oligonükleotidleri kullanılarak qPCR yoluyla büyütüldü. Kütüphanenin en yüksek floresana ulaştığı ilk döngü PCR döngü sayısı olarak seçildi. Şekil 4C , tipik bir cDNA kütüphanesinden 16'lık bir tepe döngüsü sayısı veren bir qPCR örneği vermektedir. Bu değer daha sonra son χCRAC PCR için kullanıldı. Sıralanan verileri işlemek için, daha önce laboratuvarımızda geliştirilen yazılımı (pyCRAC) ve kinetik CRAC verilerinin analizi için ilgili boru hattını kullandık (Nues ve ark., 2017; https://git.ecdf.ed.ac.uk/sgrannem/pycrac, https://bitbucket.org/sgrann/kinetic_crac_pipeline/src/default/). Bu açık kaynaklı yazılım araçları, verilerin çoğullanmasının çözülmesini ve kırpılmasını, PCR kopyalarının kaldırılmasını, istatistiksel olarak anlamlı tepe noktalarının tanımlanmasını, kümenin bitişik dizilere okunmasını ve bağlanma motiflerinin tanımlanmasını sağlar39. Bu araçların nasıl çalıştığına dair daha fazla ayrıntı ilgili web sayfalarında bulunabilir.
Ayrıca memeli hücreleri için bir χCRAC protokolü geliştirmeye başladık. Memeli hücre hatlarının çoğunluğu tek katmanlı olarak yetiştirilir ve UV geçirgen torba ile çapraz bağlayıcımızdaki tepsi, yapışkan hücrelerle yapılan deneyler için uygun değildir. Bu sorunun üstesinden gelmek için, kullanıcıların 1-2 Petri kabını (150 mm çapında ve 25 mm derinliğinde) yapışkan hücrelerle UV ışınlayabilecekleri bir aşama geliştirdik (Şekil 3B). İlk test olarak, memeli hücreleri için çapraz bağlayıcının etkinliği, anti-GFP antikorları ve geleneksel bir CLIP tabanlı saflaştırma kullanılarak çapraz bağlanma ve kararlı şekilde etiketlenmiş GFP-RBM7'nin yakalanması yoluyla ölçülmüştür. Şekil 5A'da gösterildiği gibi, çapraz bağlayıcı, yaygın olarak kullanılan bir UV ışınlama cihazıyla karşılaştırılabilir verimliliklerde 254 nm UV ışınlaması kullanarak tek katmanlı olarak yetiştirilen memeli hücrelerinden protein-RNA komplekslerini geri kazanabildi. Bununla birlikte, normalde UV çapraz bağlama deneyleri için kullanılan standart hücre kültürü plastik eşyaları 254 nm UV'ye kadar geçilemez. Bu nedenle, çapraz bağlayıcımızda hücreler sadece UV lambalarının üst bankasından ışınlama alacaktır. Bunun üstesinden gelmek için, hücre büyümesi ve çapraz bağlanma için UV geçirgen bir kuvars Petri kabı geliştirdik. Kuvars kültür kaplarının kullanımı, 2 s kadar az UV ışınlaması ile protein-RNA komplekslerinin sağlam bir şekilde geri kazanıldığını göstermiştir (Şekil 5B). CLIP teknolojileri gibi memeli hücreleri için RBP yakalama yöntemleriyle birleştirildiğinde, bu kısa çapraz bağlama süreleri, genotoksik streslere veya protein faktörlerinin hızlı tükenmesine yanıt olarak veya transkripsiyonel veya hücre döngüsü senkronizasyonuna paralel olarak RBP'lerin mekansal zamansal RNA bağlanma profillerini geri kazanmak için zaman dilimleriyle uyumludur.
Şekil 6'da , χCRAC işlem hattı tarafından işlenen Nrd1 verilerinin birkaç örneği gösterilmektedir. Bu şekil, işlem hattı tarafından oluşturulan yatak grafiği dosyaları ve verilerin yayın kalitesinde genom tarayıcı görüntülerini oluşturmayı basitleştirmek için tasarladığımız python GenomeBrowser paketi (https://pypi.org/project/GenomeBrowser/1.6.3/) kullanılarak hazırlanmıştır. Gri dikdörtgenler, şifreli kararsız transkript (CUT'lar), kararlı karakterize edilmemiş transkriptler (SUT'ler)40 ve Xrn1'e duyarlı kararsız transkriptler (XUT'lar)41 gibi kodlamayan RNA'ları ifade eden genomik bölgeleri temsil eder. Şekil 6'daki veriler, Nrd1'in bu kodlamayan RNA transkriptlerinin çoğuna bağlandığını ve bu proteinin bu transkript sınıfının42 sınıfının bozulmasında rol oynadığı fikriyle tutarlı olduğunu göstermektedir. Şekil 6A , kromozom IV üzerinde ~ 15 kb'lik bir bölgeyi göstermektedir. Burada, Nrd1'in, her ikisi de glikoz açlığı sırasında yukarı regüle edilen yüksek afiniteli glikoz taşıyıcıları HXT6 ve HXT7'yi kodlayan transkriptlere bağlanmasında önemli bir artış olmuştur. NNS kompleksi tarafından transkripsiyon sonlanmasının, glikoz açlığı sırasında bu genlerin indüksiyon kinetiğini etkilemesi muhtemeldir. Şekil 6B , Nab343 tarafından düzenlendiği bilinen Imd3 transkriptine Nrd1 çapraz bağlama örneğini göstermektedir. Bu durumda, veriler glikoz açlığına bağlanmada önemli bir azalma olduğunu göstermiştir. Önceki çalışmalar, glukoz açlığı44 sırasında Nab3'ün Tye7 transkriptine bağlanmasının azaldığını göstermiştir. Bu gözlemle tutarlı olarak, χCRAC verileri, glikoz açlığı sırasında Nrd1'in bağlanmasının azaldığını ve Nrd1'in Tye7'ye çapraz bağlanmasının 8 dakikalık stresten sonra en düşük seviyede olduğunu göstermektedir (Şekil 4C). Bununla birlikte, bu etkinin sadece geçici olduğu görülmektedir, çünkü 14 dakikalık glikoz açlığından sonra, Nrd1 bağlanması başlangıç seviyelerine geri dönmüştür.
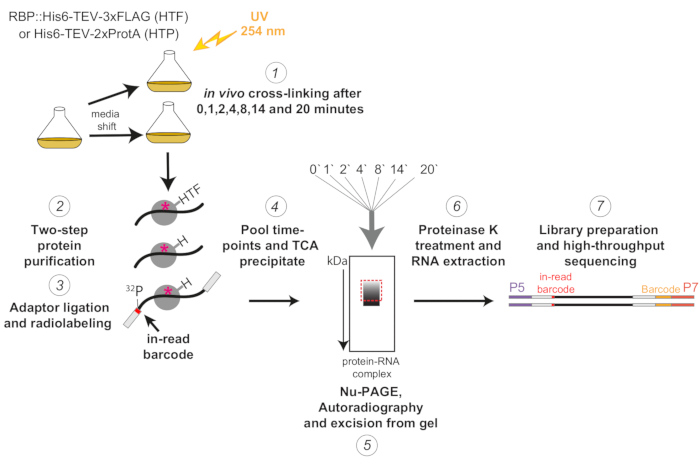
Şekil 1: χCRAC protokolünün şematik gösterimi. Etiketli suşlar istenen yoğunluğa kadar yetiştirildi. RBP, RNA bağlayıcı proteini gösterir. Daha sonra bir referans numune alındı ve 254 nm UV ışığı ile çapraz bağlandı. Kalan hücreler filtrasyon yoluyla toplandı ve daha sonra hızla strese neden olan ortama kaydırıldı. Burada açıklanan χCRAC deneyi için, örnekler alındı ve vardiyadan 1, 2, 4, 8, 14 ve 20 dakika sonra çapraz bağlandı (1). İlgilenilen RBP daha sonra son derece katı iki adımlı bir afinite saflaştırması kullanılarak saflaştırıldı (2). Daha sonra, yakalanan çapraz bağlı RNA'lar kısmen RNazlarla sindirildi, 5' ucunda radyo-etiketli ve adaptörler üzerlerine bağlandı (3). 5' adaptörler benzersiz "okunan" barkod dizileri içeriyordu, böylece tek tek numuneler sıralamadan sonra biyoinformatik olarak ayrılabiliyordu. RBP-RNA kompleksleri daha sonra salınım, havuzlanmış ve birlikte çökeltilmiş (4), SDS-PAGE ile çözülmüş ve otoradyografi ile görselleştirilmiştir (5). Daha sonra, otoradyografi görüntüsünde kesikli kırmızı kutu ile gösterilen, ana bandın hemen üzerindeki radyoaktif sinyali içeren tek bir jel dilimi jelden kesildi (5). Jel dilimleri proteaz K ile muamele edildi ve RNA daha sonra ekstrakte edildi (6), cDNA'lara dönüştürüldü ve PCR (7) ile güçlendirildi. PCR adımı, birçok kütüphanenin tek bir şeride çoğaltılabilmesi için ek barkodlar (P7 oligo tarafından tanıtılan sarı blok) getirdi. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.

Şekil 2: Çapraz bağlama ve vakum filtreleme. (A) Çapraz bağlayıcı. Hücre süspansiyonu, makinenin sağ üst köşesinde bulunan bir huniye dökülür (yakın çekim için ayrıca Şekil 3A'ya bakınız) ve orta tepside bulunan UV şeffaf bir torbada tutulur. Bu torba, kullanıcı makineye ışınlama adımını başlatması talimatını verene kadar kapalı kalan iki panjurla çevrilidir. Hücreler hem yukarıdaki hem de aşağıdaki tepsilerden UV ışığı ile ışınlanır. Makine 254 ve 365 nm UV lambalarla birlikte gelir, ikincisi PAR-CLIP deneyleri için geçerlidir. Makine, sağ üstte bulunan ve UV dozajını veya maruz kalma süresini kontrol etmeyi sağlayan dokunmatik bir panelden çalıştırılır. (B) Çapraz bağlamayı takiben, hücreler makinenin sol tarafından boşaltılır. Hücre süspansiyonları vakum yoluyla geri kazanılır ve daha sonra hasat için bir vakum filtreleme cihazına dökülebilecekleri bir cam şişeye boşaltılır. (C) Vakum filtrasyon cihazları. Bunlar bir klips aracılığıyla açılıp kapatılır ve arasına bir filtre yerleştirilir. Filtrelerin değiştirilmesi sonucu hiç zaman kaybetmemek için çok kısa süreli seriler için dört filtreleme cihazı paralel olarak kullanılmıştır. (D) Filtrelemeyi takiben, ortam süpernatantı daha sonra imha edilmek üzere şişelere boşaltıldı. Filtre çıkarıldığında sistemdeki vakumu korumak için vakum filtrasyon cihazlarının altına vanalar monte edilmiştir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.

Şekil 3: Çapraz bağlama askıya alınmış ve yapışkan hücreler. (A) Süspansiyon hücreleri için Vari-X-bağlayıcı hazneli çapraz bağlayıcı. Hücre kültürü, tepsinin sağ üst köşesinde bulunan numune girişine (huni) dökülür. (B) Yapışkan hücreleri veya küçük hacimli süspansiyon hücrelerini çapraz bağlamak için plastik veya kuvars Petri kaplarını tutabilen tepsi. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.

Şekil 4: Kütüphane hazırlığı. (A) Nrd1-HTP χCRAC deneyinden bir otoradyogram örneği. Güçlü, konsantre sinyal, çok kısa RNA'lara çapraz bağlanmış proteini temsil ederken, yukarıdaki yayma, dizileme için yeterli uzunluktaki RNA'lara çapraz bağlanmış proteini temsil eder. (B) Yayma, jel eksizyonundan sonra alınan bir otoradyogramda gösterildiği gibi eksize edildi. (C) χCRAC cDNA kütüphanesinden temsili bir qPCR. Bu örnekte, cDNA'nın maksimum amplifikasyonuna 16 döngüde ulaşılmıştır. Böylece, son amplifikasyon için 16 döngü kullanıldı. Hata çubuğu, üç teknik qPCR çoğaltmasının standart sapmasını temsil eder. (D) %6 TBE jeli üzerindeki bir cDNA kütüphanesinden fosfor görüntüsü örneği. (E) Çip bazlı kılcal elektroforezden cDNA uzunluğu ve kalite analizi. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
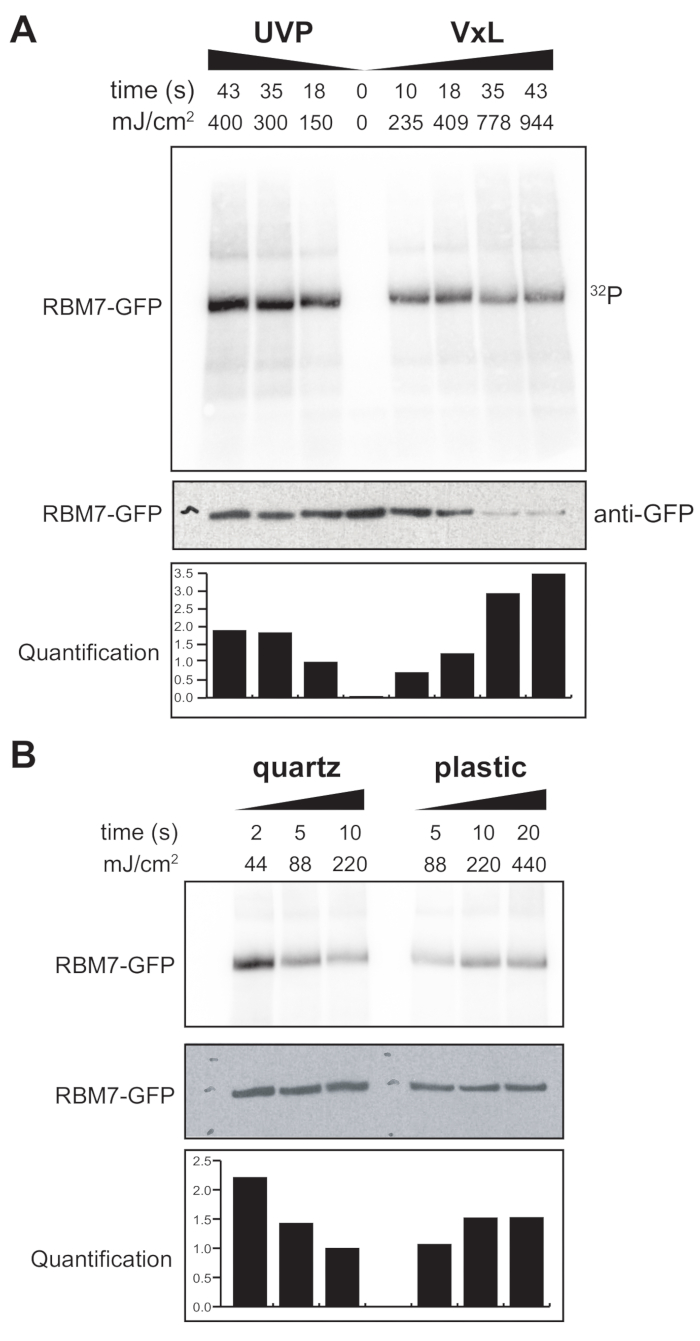
Şekil 5: Memeli hücrelerinde çapraz bağlanmayı test etmek için yüksek RNaz testi iCLIP deneyi. GFP-RBM7 iCLIP deneylerinden elde edilen otoradyogramlar, çeşitli çapraz bağlama enerjilerinde RNP geri kazanımının verimliliğini test etmiştir. İmmünopresipitasyonlar, GFP-RBM7'yi kararlı bir şekilde eksprese eden çapraz bağlı hücreler üzerindeki manyetik boncuklara bağlı anti-GFP antikorları kullanılarak gerçekleştirildi. İmmünoçökeltiler, ilişkili RNA'ları kısa, düzgün uzunluklara kırpmak için yüksek konsantrasyonlarda RNaz I ile inkübe edildi. RNP'ler 32P etiketleme ve SDS-PAGE ile görselleştirildi ve çapraz bağlı olmayan proteinin göçüne yakın, tanımlanmış bir bant olarak göç etti. Niceliklendirme, anti-GFP batı leke sinyaline normalleştirilmiş radyoaktif işaretli RBM7-RNA sinyalinin densitometrik analizlerinin sonuçlarını gösterir. (A) Yaygın olarak kullanılan UVP çapraz bağlayıcısının çapraz bağlayıcı ile çapraz bağlayıcı zaman seyri (Vari-X-bağlayıcı; VxL). (B) Kuvars (solda) ve plastik (sağda) kültür gereçleri üzerindeki çapraz bağlayıcımızın çapraz bağlama zaman seyri. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
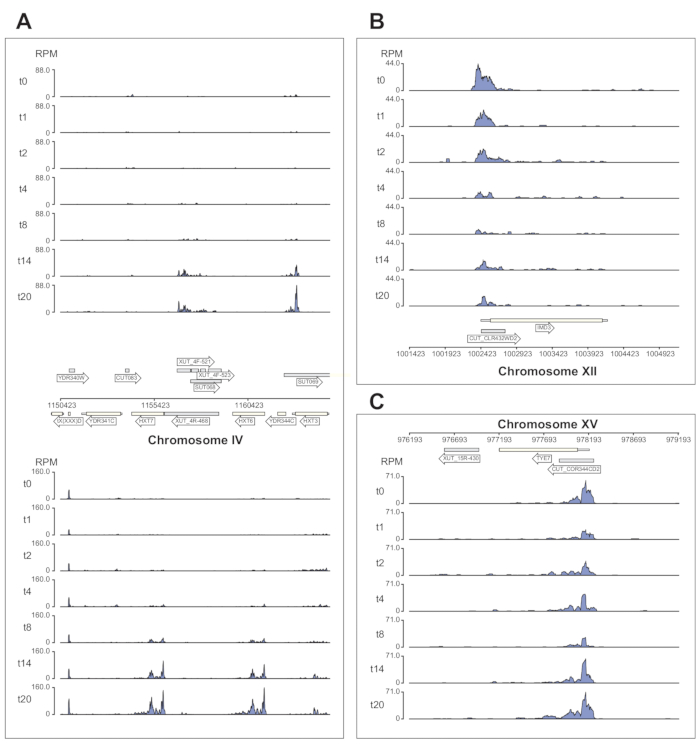
Şekil 6: χCRAC'ın Nrd1'in hedeflerine diferansiyel, zamansal bağlanmasını gösterme gücünü gösteren örnek genom tarayıcı grafikleri. Her kutu ayrı genomik bölgeler için grafikler gösterir. Oklar, genlerin hangi iplikçikte kodlandığını gösterir (sola işaret eden ok = eksi iplikçik; sağa işaret eden ok = artı iplikçik). Zaman noktaları (dk), her bir alt grafiğin y eksenlerinde t0, t1, t2 vb. İle gösterilir. Kromozomları ve koordinatları gösteren Romen rakamları gösterilir. (A) Glikoz yoksunluğu üzerine, Nrd1, her ikisi de bu durumda yukarı regüle edilen iki yüksek afiniteli glikoz taşıyıcısını, HXT6 ve HXT7'yi bağlar. (B) Nrd1'in, Nab344'ün zaten doğrulanmış bir hedefi olan Imd3'e bağlandığı ve glikoz açlığını takiben yoğunluğun azaldığı gözlenmiştir. (C) Tye7'nin Nrd1 bağlanması dinamik ve geçici bir nitelik sergiler, glikoz açlığından sonra 8 dakikalık stresten sonra minimuma iner. Bununla birlikte, bağlanma daha sonra 14 dakika sonra bazal seviyelere geri döner. Okumalar "milyonda okuma" (RPM; y ekseni) olarak normalleştirildi. Gri kutular, kodlamayan RNA'ları kodlayan bölgeleri gösterir. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
Tartışmalar
Yeni çapraz bağlama ve hücre toplama cihazlarıyla birleştirilen χCRAC yöntemi, çok çeşitli model organizmalara uygulanabilir olduğu ve bu nedenle RNA alanına genel ilgi göstermesi gerektiği için büyük bir potansiyele sahiptir. χCRAC'ın kullanılabileceği birçok alan vardır. Örneğin, yöntem, proteinlerin hiyerarşik montajını, genellikle proteinler ve RNA molekülleri arasındaki dinamik etkileşimleri içeren splisozom ve ribozom gibi büyük makromoleküler komplekslere ölçmek için kullanılabilir. Ayrıca, hücreler çeşitli streslere maruz kaldıklarında RNA bozunma faktörleri ve substratları arasındaki etkileşimleri izlemek için rutin olarak kullanıyoruz. Bu, adaptif yanıtın hangi aşamasında bu faktörlerin en aktif olduğunu, hangi substratlara bağlandıklarını ve bu etkileşimlerin ne kadar dinamik olduğunu belirlememizi sağlar. Bu tür veriler, araştırmacıların çevresel değişikliklere adaptasyonda her bir faktörün göreceli katkısını belirlemelerini sağlamalıdır.
χCRAC, proteini son derece sıkı ve denatüre edici koşullar altında saflaştırmak için çift afiniteli saflaştırma etiketleri (HTF veya HTP) kullanır. Bu, kosafifiye RNA'nın, ilgilenilen proteine kovalent olarak çapraz bağlanmış RNA'lar için yüksek oranda zenginleştirilmesini sağlar. Ancak, benzeşim etiketlerine güvenmenin dezavantajları vardır. Örneğin, etiket protein fonksiyonuna müdahale edebilir, bu da RNA bağlayıcı interaktomunun çarpık bir okumasını verebilir. Ek olarak, bazı model organizmalar için etiketleri kullanmak her zaman mümkün olmayabilir, çünkü DNA parçalarını genoma entegre etmek veya ekspresyon plazmidlerini dönüştürmek için genetik araçlar henüz mevcut değildir. Bununla birlikte, χCRAC protokolünün bazı bölümlerini, RBP'nin saflaştırılması için antikorlara dayanan CLIP tabanlı protokollerle uyumlu hale getirmek için değiştirmek kolaydır. Gerçekten de, bu çalışma iCLIP bazlı saflaştırmaları çapraz bağlayıcımızla birleştirmenin mümkün olduğunu göstermiştir. Şimdi, insan RNA bağlayıcı proteinlerinin yeni ortaya çıkan RNA transkriptleriyle zamansal ilişkisini incelemek için CLIP protokolleri geliştirme sürecindeyiz.
Yeni bir protein üzerinde χCRAC gerçekleştirirken, maksimum çapraz bağlanmayı indüklemek için UV maruziyeti optimize edilmelidir. Bu önemlidir, çünkü yüksek UV maruziyeti, saflaştırma adımı sırasında RNA'nın geri kazanımını azaltabilir. Rekombinant RBP'yi eksprese eden hücreler çeşitli UV dozlarına, 100 mJ / cm2, 250 mJ / cm2, 500 mJ / cm2 ve 1 J /cm2'ye maruz bırakıldı. RNP'ler daha sonra yakalandı ve RNA'lar parçalandı ve radyo-etiketlendi. Daha sonra, RNP'ler SDS-PAGE tarafından çözüldü ve hangi maruziyetin en yoğun sinyali verdiğini (yani maksimum çapraz bağlama) çıkarmak için bir otoradyogram alındı.
Deney koşulları optimize edildikten sonra, χCRAC gerçekleştirilirken birkaç kontrol deneyi önerilir. İlk olarak, saflaştırma boncuklarına arka plan bağlanmasını izlemek için UV ışınlanmış, etiketlenmemiş bir numune kullanılabilir. İkincisi, bir vardiya deneyi sırasında χCRAC uygulanırken, hücrelerin orijinal ortama geri kaydırıldığı ikinci bir zaman serisi, hücrelerin filtrasyonunun kendisinin RNA seviyelerinde veya protein-RNA etkileşimlerinde değişikliklere neden olup olmadığının araştırılmasını sağlar.
Giriş'te belirtildiği gibi, yakın zamanda yayınlanmış çok sayıda makale, CLIP protokolünde bir dizi optimizasyon önermektedir. Bu, kızılötesi tarama10 yoluyla protein-RNA kompleksini tespit etmek için floresan olarak etiketlenmiş adaptörlerin kullanımını ve ayrıca ortaya çıkan kütüphanelerin karmaşıklığını arttırdığı gösterilen çeşitli nükleik asit saflaştırma ve boyut seçim adımlarına optimizasyonları içerir12,45. Şu anda χCRAC protokolünü daha da hassaslaştırmak için bu iyileştirmelerden bazılarını uyguluyoruz. Burada sunulan protokol, verilerin karmaşıklığını artıran orijinal CRAC ve χCRAC protokollerinde bir dizi iyileştirme içermektedir. Örneğin, daha önce, SDS-PAGE jelleri üzerindeki çapraz bağlı, radyoaktif protein-RNA komplekslerini çözdükten sonra, bir nitroselüloz membranına aktarıldı ve çapraz bağlı RNA lekeden izole edildi. Bununla birlikte, RNP'nin transferi ve ardından RNA ekstraksiyonu, özellikle RNA polimeraz alt birimleri gibi büyük RBP'lerle uğraşırken çok verimsiz olabilir. Bu, çapraz bağlı RNA'nın geri kazanımında önemli bir azalmaya neden olabilir. Mevcut protokolde, çapraz bağlı RNA, Şekil 1'de gösterildiği gibi doğrudan SDS-PAGE jel dilimlerinden ekstrakte edilir. Bu, çapraz bağlı RNA'ların geri kazanımını arttırdı. Ek olarak, cDNA'ların PCR amplifikasyonundan sonra, ürün başlangıçta% 3, düşük erime sıcaklıklı agaroz jelleri üzerinde çözüldü ve daha sonra jelden 175-300 bp PCR ürünleri ekstrakte edildi. Bununla birlikte, bu jeller kolayca aşırı yüklenebilir ve bu da DNA'nın çok zayıf bir şekilde ayrılmasına neden olur. Agaroz jellerinin prekast TBE jellerle değiştirilmesi, daha tutarlı boyut ayrımı ve PCR ürünlerinin daha iyi geri kazanılması ile sonuçlandı.
Açıklamalar
A. Langford ve W. Worboys, ticari bir şirket olan UVO3'e bağlıdır. Çalışma tasarımında, veri toplama ve yorumlamada veya çalışmayı yayınlanmak üzere gönderme kararında hiçbir rolleri yoktu.
Teşekkürler
Bu çalışma, Wellcome Trust (091549 S.G.'ye ve 109093/Z/15/A'dan S.M.'ye), Wellcome Trust Centre for Cell Biology çekirdek hibesi (092076) ve Tıbbi Araştırma Konseyi Klinik Olmayan Kıdemli Araştırma Bursu (MR/R008205/1'den S.G.'ye), Avrupa Moleküler Biyoloji Örgütü'nden uzun süreli doktora sonrası burs (ALTF 1070-2017'den R.A.C.'ye) tarafından desteklenmiştir. ve Danimarka Bağımsız Araştırma Fonu (T.H.J).
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1,4-dithioreitol | Merck | 10708984001 | Buffer component in mammalian cell lysis |
| 1.5 mL tubes | Eppendorf | 0030 120.086 | General reaction tube |
| 2 mL tubes | Eppendorf | 0030 123.344 | For holding columns and collection of waste |
| 32P-yATP | Perkin Elmer | NEG502Z-250 | For radiolabelling the 5' end of the RNA |
| 4-12% Bis-Tris gel | Invitrogen | NP0321BOX | SDS-PAGE gel |
| 4X loading buffer | Novex | NP0008 | Protein loading dye concentrate |
| 50 bp ladder | New England Biolabs | N3236 | Reference ladder for excising region of interest from the amplified cDNA library |
| 50% PEG | NEB | B100045 | For the L5 linker ligation |
| 6% TBE gel | Invitrogen | EC6265BOX | For separation and purification of the cDNA library |
| Acetone | ACROS Organics | 423245000 | Washing of TCA-precipitated proteins |
| anti-FLAG beads | Sigma Aldrich | M8823-1ML | For purifcation of FLAG-tagged RBPs |
| ATP (100 mM) | Thermo Fisher Scientific | R0441 | For ligation of the L5 linker onto the 5' end of captured RNAs |
| Beta-mercaptoethanol | Sigma Aldrich | M3148-100ML | Buffer component |
| Biomax MS intensifying screen | Sigma Aldrich | Z363162-1EA | For intensifying the autoradiogram signal |
| Chloroform | Thermo Fisher Scientific | 1010219 | For phenol-chloroform extraction following RNA purification |
| cOmplete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | For inhibition of cellular proteases after lysis |
| Complete supplement mixture -TRP | Formedium | DCS0149 | For preparation of synthetic defined medium |
| Costar Spin-X 0.22 µm filters | Sigma Aldrich | CLS8160 | For isolating the excised cDNAs following gel extraction |
| DNase RQ1 | Promega | M6101 | For DNA digest following cell lysis |
| dNTPs (10 mM) | Sigma Aldrich | 4638956001 | For reverse transcription and PCR |
| Ethanol | Thermo Fisher Scientific | 10041814 | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Ethylenediaminetetraacetic acid | Invitrogen | AM9261 | For protease K buffer |
| Exonuclease I | New England Biolabs | M0293 | For degradation of primers following PCR |
| Glass microfiber filters | Whatman | 1823-010 | For isolating the excised cDNAs following gel extraction |
| Glucose | Formedium | GLU03 | For preparation of glucose-containing, synthetic defined medium |
| Glycogen (20 mg/mL) | Roche | 10901393001 | Precipitation of proteins, RNA and DNA |
| GST-TEV | Homemade | Construct and purification protocol is available upon request | |
| Guanidium hydrochloroide | Thermo Fisher Scientific | 10071503 | Required for pulldown denaturing conditions and washing buffer |
| IgG beads | GE Healthcare | 17-0969-01 | For purification of protein A-tagged RBPs |
| Imidazole | Sigma Aldrich | I2399-100G | For elution of captured proteins from Nickel beads |
| Isoamyl alcohol | Thermo Fisher Scientific | A393-500 | For phenol-chloroform extraction following RNA purification |
| Luna Universal One-Step RT-qPCR | NEB | E3005S | For qPCR of the cDNA in order to calculate required number of PCR cycles |
| Magnesium chloride | Fluka Analytical | 63020-1L | For PNK buffer |
| Membrane filters | Millipore | AAWP09000 for yeast or HAWP09000 for bacteria | For vacuum filtration of cells |
| Micro bio-spin columns | Biorad | 732-6204 | For collecting eluate after gel extraction |
| Ni-NTA beads | Qiagen | 30210 | For secondary protein capture |
| NP-40 | Sigma Aldrich | I8896-100ML | Buffer component |
| Pfu polymerase | Promega | M7741 | For amplification of the cDNA library |
| Phenol | Sigma Aldrich | P4682-400ML | For phenol-chloroform extraction following RNA purification |
| Pierce spin columns | Thermo Fisher Scientific | 69725 | For on-column enzymatic reactions |
| Protease K | Roche | 3115887001 | For degradation of the RBP following gel extraction |
| Quartz Petri dish | UVO3 | N/A | For cross-linking of adherent cells. Available from https://www.vari-x-link.com for 400 GBP |
| Radiography films | Amersham | 28906843 | For autoradiography visualisation |
| RNAClean XP beads | Beckmann | A63987 | SPRI beads for clean up of RNAs and cDNAs |
| RNase H | New England Biolabs | M0297 | For degradation of RNAs following reverse transcription |
| RNase-It | Agilent | 400720 | For RNA digestion |
| rRNasin | Promega | N2511 | For inhibition of any contaminating RNases during enzymatic reaction |
| Sodium acetate | Sigma Aldrich | S2889-1KG | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Sodium chloride | Thermo Fisher Scientific | 7647-14-5 | Buffer component |
| Sodium deoxycholate | Sigma Aldrich | D6750-100G | Buffer component in mammalian cell lysis |
| Sodium dodecylsulfate | Sigma Aldrich | L3771-1KG | For protease K buffer |
| SUPERase-In | Invitrogen | AM2694 | For inhibition of cellular RNases after lysis |
| SuperScript IV | Thermo Fisher Scientific | 18090010 | For reverse transcription |
| T4 PNK | New England Biolabs | M0201 | For radiolabelling the 5' end of the RNA |
| T4 RNA ligase 1 | New England Biolabs | M0204 | For ligation of the L5 adaptor onto the RNA 5' end |
| T4 RNase ligase 2, truncated K222Q | NEB | M0351S | For ligation of the App_PE linker onto the 3' end of captured RNAs |
| TBE buffer (10X) | Invitrogen | 15581-028 | For running TBE gels |
| TEV protease | Homemade | For eluting captured proteins following FLAG capture | |
| Thermosensitive alkaline phosphatase | Promega | M9910 | For 5' and 3' dephosphorylation of RNAs |
| Trichloroacetic acid (100%) | Sigma Aldrich | T0699-100ML | For precipitation of RBP-RNA complexes |
| Tris hydrochloride | Invitrogen | 15504-020 | Buffer component |
| Triton X-100 | Sigma Aldrich | T8787-100ML | Buffer component in mammalian cell lysis |
| Vari Filter | UVO3 | N/A | Device for vacuum harvesting cells. Available from https://www.vari-x-link.com for 100 GBP |
| Vari-X-Linker | UVO3 | N/A | Cross-linker for cross-linking cells. Available from https://www.vari-x-link.com for 16,000 GBP |
| Yeast nitrogen base | Formedium | CYN0410 | For preparation of synthetic defined medium |
| Zirconia beads | Thistle | 11079105Z for yeast or 11079101Z for bacteria | For cell lysis via bead beating |
Referanslar
- Ule, J., et al. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Granneman, S., Kudla, G., Petfalski, E., Tollervey, D. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proceedings of the National Academy of Sciences. 106 (24), 9613-9618 (2009).
- Licatalosi, D. D., et al. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-469 (2008).
- König, J., et al. iCLIP reveals the function of hnRNP particles in splicing at individual nucleotide resolution. Nature Structural & Molecular Biology. 17 (7), 909-915 (2010).
- Hafner, M., et al. Transcriptome-wide Identification of RNA-Binding Protein and MicroRNA Target Sites by PAR-CLIP. Cell. 141 (1), 129-141 (2010).
- Aktaş, T., et al. DHX9 suppresses RNA processing defects originating from the Alu invasion of the human genome. Nature. 544 (7648), 115-119 (2017).
- Huppertz, I., et al. iCLIP: Protein–RNA interactions at nucleotide resolution. Methods. 65 (3), 274-287 (2014).
- Li, X., et al. Comprehensive in vivo RNA-binding site analyses reveal a role of Prp8 in spliceosomal assembly. Nucleic Acids Research. 41 (6), 3805-3818 (2013).
- Rosenberg, M., et al. Denaturing CLIP, dCLIP, Pipeline Identifies Discrete RNA Footprints on Chromatin-Associated Proteins and Reveals that CBX7 Targets 3′ UTRs to Regulate mRNA Expression. Cell Systems. 5 (4), 368-385 (2017).
- Zarnegar, B. J., et al. irCLIP platform for efficient characterization of protein–RNA interactions. Nature Methods. 13 (6), 489-492 (2016).
- Kargapolova, Y., Levin, M., Lackner, K., Danckwardt, S. sCLIP—an integrated platform to study RNA–protein interactomes in biomedical research: identification of CSTF2tau in alternative processing of small nuclear RNAs. Nucleic Acids Research. 45 (10), 6074-6086 (2017).
- Van Nostrand, E. L., et al. Robust transcriptome-wide discovery of RNA-binding protein binding sites with enhanced CLIP (eCLIP). Nature Methods. 13 (6), 508-514 (2016).
- Flynn, R. A., et al. Dissecting noncoding and pathogen RNA–protein interactomes. RNA. 21 (1), 135-143 (2015).
- Brugiolo, M., Botti, V., Liu, N., Müller-McNicoll, M., Neugebauer, K. M. Fractionation iCLIP detects persistent SR protein binding to conserved, retained introns in chromatin, nucleoplasm and cytoplasm. Nucleic Acids Research. 45 (18), 10452-10465 (2017).
- Sanford, J. R., et al. Identification of Nuclear and Cytoplasmic mRNA Targets for the Shuttling Protein SF2/ASF. PLOS ONE. 3 (10), e3369(2008).
- Garzia, A., Meyer, C., Morozov, P., Sajek, M., Tuschl, T. Optimization of PAR-CLIP for transcriptome-wide identification of binding sites of RNA-binding proteins. Methods. 118-119, 24-40 (2017).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22 (10), 2031-2042 (2012).
- Chen, K., et al. High-Resolution N6-Methyladenosine (m6A) Map Using Photo-Crosslinking-Assisted m6A Sequencing. Angewandte Chemie International Edition. 54 (5), 1587-1590 (2015).
- Ke, S., et al. A majority of m6A residues are in the last exons, allowing the potential for 3′ UTR regulation. Genes & Development. 29 (19), 2037-2053 (2015).
- Linder, B., et al. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nature Methods. 12 (8), 767-772 (2015).
- Kudla, G., Granneman, S., Hahn, D., Beggs, J. D., Tollervey, D. Cross-linking, ligation, and sequencing of hybrids reveals RNA–RNA interactions in yeast. Proceedings of the National Academy of Sciences. 108 (24), 10010-10015 (2011).
- Sugimoto, Y., et al. hiCLIP reveals the in vivo atlas of mRNA secondary structures recognized by Staufen 1. Nature. 519 (7544), 491-494 (2015).
- Hwang, H. W., et al. cTag-PAPERCLIP Reveals Alternative Polyadenylation Promotes Cell-Type Specific Protein Diversity and Shifts Araf Isoforms with Microglia Activation. Neuron. 95 (6), 1334-1349 (2017).
- Hwang, H. W., et al. PAPERCLIP Identifies MicroRNA Targets and a Role of CstF64/64tau in Promoting Non-canonical poly(A) Site Usage. Cell Reports. 15 (2), 423-435 (2016).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Beckmann, B. M. RNA interactome capture in yeast. Methods. 118-119, 82-92 (2017).
- Granneman, S., Petfalski, E., Tollervey, D. A cluster of ribosome synthesis factors regulate pre-rRNA folding and 5.8S rRNA maturation by the Rat1 exonuclease. The EMBO Journal. 30 (19), 4006-4019 (2011).
- Schaughency, P., Merran, J., Corden, J. L. Genome-Wide Mapping of Yeast RNA Polymerase II Termination. PLOS Genetics. 10 (10), e1004632(2014).
- Bernstein, J. A., Khodursky, A. B., Lin, P. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Kresnowati, M. T. A. P., et al. When transcriptome meets metabolome: fast cellular responses of yeast to sudden relief of glucose limitation. Molecular Systems Biology. 2, 49(2006).
- Marguerat, S., Lawler, K., Brazma, A., Bähler, J. Contributions of transcription and mRNA decay to gene expression dynamics of fission yeast in response to oxidative stress. RNA Biology. 11 (6), 702-714 (2014).
- van Nues, R., et al. Kinetic CRAC uncovers a role for Nab3 in determining gene expression profiles during stress. Nature Communications. 8 (1), 12(2017).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Tudek, A., Candelli, T., Libri, D. Non-coding transcription by RNA polymerase II in yeast: Hasard or nécessité? Biochimie. 117, 28-36 (2015).
- Lingaraju, M., et al. The MTR4 helicase recruits nuclear adaptors of the human RNA exosome using distinct arch-interacting motifs. Nature Communications. 10 (1), 1-11 (2019).
- Lubas, M., et al. Interaction Profiling Identifies the Human Nuclear Exosome Targeting Complex. Molecular Cell. 43 (4), 624-637 (2011).
- Conrad, N. K., et al. A yeast heterogeneous nuclear ribonucleoprotein complex associated with RNA polymerase II. Genetics. 154 (2), 557-571 (2000).
- Darby, M. M., Serebreni, L., Pan, X., Boeke, J. D., Corden, J. L. The Saccharomyces cerevisiae Nrd1-Nab3 Transcription Termination Pathway Acts in Opposition to Ras Signaling and Mediates Response to Nutrient Depletion. Molecular and Cellular Biology. 32 (10), 1762-1775 (2012).
- Webb, S., Hector, R. D., Kudla, G., Granneman, S. PAR-CLIP data indicate that Nrd1-Nab3-dependent transcription termination regulates expression of hundreds of protein coding genes in yeast. Genome Biology. 15 (1), R8(2014).
- Jensen, T. H., Jacquier, A., Libri, D. Dealing with Pervasive Transcription. Molecular Cell. 52 (4), 473-484 (2013).
- van Dijk, E. L., et al. XUTs are a class of Xrn1-sensitive antisense regulatory non-coding RNA in yeast. Nature. 475 (7354), 114-117 (2011).
- Thiebaut, M., et al. Futile Cycle of Transcription Initiation and Termination Modulates the Response to Nucleotide Shortage in S. cerevisiae. Molecular Cell. 31 (5), 671-682 (2008).
- Merran, J., Corden, J. L. Yeast RNA-Binding Protein Nab3 Regulates Genes Involved in Nitrogen Metabolism. Molecular and Cellular Biology. 37 (18), e00154-e00117 (2017).
- Bresson, S., Tuck, A., Staneva, D., Tollervey, D. Nuclear RNA Decay Pathways Aid Rapid Remodeling of Gene Expression in Yeast. Molecular Cell. 65 (5), 787-800 (2017).
- Buchbender, A., et al. Improved library preparation with the new iCLIP2 protocol. Methods. , (2019).
Erratum
Formal Correction: Erratum: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC
Posted by JoVE Editors on 2/17/2023. Citeable Link.
An erratum was issued for: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC. The Authors section was updated from:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Synthetic and Systems Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
to:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Mehak Chauhan1
Niki Christopoulou1
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Engineering Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır