Method Article
从感染的沙门氏菌培养物中去除噬菌体
摘要
噬菌体在地球上无处不在且种类繁多,在细菌宿主内感染和复制,在微生物生态系统中发挥着至关重要的作用。尽管它们很重要,但它们的存在可能会扰乱工业流程。我们开发了一种使用细菌脂多糖从 沙门氏菌 培养物中消除噬菌体的方法。
摘要
噬菌体,或简称噬菌体,在微生物环境中起着至关重要的作用,影响细菌种群并塑造它们的进化和相互作用。这些生物体是感染细菌宿主并在细菌宿主内复制的病毒。噬菌体在地球上无处不在,高度多样化且非常丰富。虽然噬菌体在不同环境中具有重要作用,并且是微生物学和生态学的关键研究领域,但它们在某些工业过程或产品中可能是不可取的。考虑到地球上噬菌体的丰富性和普遍性,从细菌培养物中去除噬菌体的程序设计在各种实验室和工业应用中至关重要,以保持培养物的完整性并确保准确的实验结果或产品质量。在这里,我们微调了一个方案,以消除受感染的 沙门氏菌肠道 培养物中的噬菌体,使用基于位于革兰氏阴性菌外膜中的脂多糖 (LPS) 的策略。细菌 LPS 在噬菌体识别宿主中起着重要作用,我们利用这一特性设计了一种有效的程序来去除沙 门氏菌 培养物中使用 LPS 作为受体的噬菌体。
引言
微生物种群在自然环境中面临多重挑战,尤其严重的威胁是感染噬菌体(感染细菌的病毒)的可能性 1。这些病毒在地球上广泛分布,表现出极大的多样性和丰富性 2,3,4,5。尽管噬菌体的大小、形态和基因组组织各不相同,但都具有相同的结构:DNA 或 RNA 基因组被噬菌体编码蛋白形成的衣壳包裹6。细菌已经进化出多种防御机制来对抗它们7。噬菌体感染的一个关键方面是尾纤维上存在的受体结合域,这与表征和检测有关。噬菌体表面有称为受体结合蛋白或尾纤维的蛋白质,用于识别和结合细菌细胞表面的特定受体位点。在革兰氏阴性菌的情况下,表面结构的识别,如脂多糖 (LPS)、外膜蛋白、菌毛和/或鞭毛,参与噬菌体-细菌相互作用8。噬菌体和细菌之间的这种相互作用是高度特异性的,主要取决于它们附着在宿主表面的能力。脂多糖的 O 抗原是一种常用的受体9。
噬菌体-细菌相互作用的研究不仅从生物学的角度来看令人着迷,而且在噬菌体疗法和生物技术等领域也有实际应用。虽然噬菌体在各种环境中都发挥着有价值的作用,例如,改变微生物种群10,但它们的存在在某些工业过程中可能是不可取的。在制药、生物技术和食品生产中,噬菌体的存在会影响最终产品的质量和安全,因此去除噬菌体对于满足质量标准至关重要。在生物加工和生物制造中,细菌培养物用于生产各种化合物(例如蛋白质、酶或抗生素),噬菌体的存在会导致生产过程中断,因为它们能够平衡每个共享环境中的细菌种群。噬菌体偶尔会把工业微生物学家的职业生涯变成一场噩梦11。设计有效的去除噬菌体程序对于确保一致和可靠的生产、提高工艺效率至关重要。除了这些工业方面之外,在精确度和可重复性至关重要的研究实验室环境中,消除噬菌体对于获得准确可靠的结果至关重要。此外,噬菌体的去除也可用于模拟不同的环境以测试不同的假设12。去除噬菌体在研究环境中也非常有用,因为许多基于噬菌体的研究,例如在噬菌体施用后对细菌进行计数,将受益于去除噬菌体的步骤,以产生更可靠的活细胞计数。
基于菌落分离的噬菌体去除需要几天时间才能确保菌落无噬菌体,而此处描述的程序允许在数小时内生成无噬菌体培养物。该协议允许我们跟踪细菌培养物的进化,而不会阻止它们分离菌落。从这个意义上说,可以模拟波动的环境(噬菌体的存在和/或不存在)来测试不同的假设。此外,该方案允许对细菌培养物中噬菌体的存在进行定性和定量分析。
总之,设计具有成本效益的噬菌体去除程序对于保持各个行业的产品质量、安全性和工艺效率以及基础和应用研究的进步至关重要。在这里,我们描述了一种基于使用 LPS 从受感染的 沙门氏菌 培养物中去除噬菌体的高效方案,这些噬菌体使用 LPS 作为受体,这既省时又需要最少的设备。
研究方案
注:在执行噬菌体消除程序之前,我们描述了用 9NA 噬菌体感染的 沙门氏菌 培养物的制备。 在图 1 中,说明了细菌培养物中噬菌体去除完整程序的一般表示。
1. 噬菌体感染的 沙门氏菌 培养物的制备
- 细菌培养物制备
注:在本研究中,使用了肠道沙门氏菌亚种鼠伤寒血清型菌株 ATCC 14028 opvAB::lacZ (SV8011)13。- 将含有感兴趣的 肠溶葡萄球菌 菌株的 -80 °C 中取出的小瓶放在冰上,慢慢解冻。
- 将肠 肠葡萄球菌 菌株从解冻的二甲基亚砜原液中划线到 Lennox 肉汤 (LB) 板(10 g/L 胰蛋白胨、5 g/L NaCl、5 g/L 酵母提取物和 15 g/L 琼脂,参见 材料表)。将板在 37 °C 孵育 24 小时。
- 使用接种针,从 S. enterica 板转移菌落,并将其接种到玻璃管中的 5 mL 液体 LB 中。在 37 °C 下孵育 24 小时,并以 200 rpm 摇动曝气。
- 噬菌体制备
注意:本方案中使用了属于 Siphoviridae 科(Caudovirales)14 的噬菌体 9NA。值得注意的是,可以使用其他使用脂多糖的 O 抗原作为受体的肠道沙 门氏菌 噬菌体。- 通过将 0.1 mL 浓缩噬菌体裂解物添加到 100 mL 营养肉汤(注;5 g/L 蛋白胨和 3 g/L 酵母提取物)、2 mL 50x E 盐(38.78 mM MgSO47 H2O、520.5 mM 柠檬酸 H2O、2.87 M K2HPO4 无水和 829 mM NaNH4HPO44 H2O)和 0.4 mL 50% 的混合物中制备噬菌体肉汤葡萄糖。倒置充分混合。
- 噬菌体浓度的一个典型例子可能是 109 PFU/mL(噬菌斑形成单位/mL),但可以使用较低的浓度以获得类似的结果(参见 材料表)。按照步骤 1.2.3 估计噬菌体浓度。
- 按照以下步骤制备裂解物。
- 在 5 mL LB(10 g/L 胰蛋白胨、5 g/L NaCl 和 5 g/L 酵母提取物)中制备 肠链球 菌接种物。在 37 °C 和 200 rpm 下孵育 24 小时。
- 将 4 mL 噬菌体肉汤与 1 mL 肠道链球菌过夜培养物混合。将混合物在 37 °C 和 200 rpm 下孵育 8 小时。
- 为了区分细菌和噬菌体,将混合物以 2,377 x g 离心 20 分钟。在玻璃管中回收上清液,加入 800 μL 氯仿(参见 材料表),然后涡旋。确保仅在使用与氯仿的混合物时转移上层相。
注意:防止残留氯仿的转移很重要,这可能会影响程序的效率。氯仿步骤在分离和繁殖方案中都很常见,作为最大化释放噬菌体的一种方式。然而,氯仿可能会使某些噬菌体失活或破坏其传染性15。因此,此步骤仅适用于对氯仿耐药的噬菌体。 - 将裂解物在室温下保持 2 小时,然后将它们储存在 4 °C。
- 滴定裂解物以定量制备的裂解物中存在的每 mL 噬菌体数量/PFU 数量。为此,请执行下面描述的叠加技术。
- 为 S. enterica 准备 5 mL LB 的接种物。在 37 °C 下孵育 24 小时,并以 200 rpm 摇动曝气。
- 将裂解物连续稀释到 LB 中,以达到可计数的 PFU/mL。为了确定必要的稀释因子,请确定浓缩的初始噬菌体裂解物。对于噬菌体浓度约为 109 PFU/mL 的代表性示例,导致可计数噬菌斑数量的稀释因子在 106-10 10 之间。
- 将 100 μL 适当的裂解物稀释液和 60 μL 前一天制备的 肠溶链球菌 过夜培养物添加到 5 mL LB 软琼脂中,通过以 1:1 的比例混合液体 LB 和 LB 琼脂并保持在 56 °C 以避免凝固。小心混合,避免气泡。
- 将每种混合物倒入 LB 板的顶部,并保持在工作台上,直到培养基凝固。
- 将板在 37 °C 下孵育 24 小时。 计算每个盘子上的斑块数量。要确定 PFU/mL 的数量,请将每个板的噬菌斑数量乘以稀释因子 (FD)。为了确保噬菌体的有效感染,需要 109-10 12 PFU/mL。
PFU/mL= 否。板中的斑块 x FD
- 通过将 0.1 mL 浓缩噬菌体裂解物添加到 100 mL 营养肉汤(注;5 g/L 蛋白胨和 3 g/L 酵母提取物)、2 mL 50x E 盐(38.78 mM MgSO47 H2O、520.5 mM 柠檬酸 H2O、2.87 M K2HPO4 无水和 829 mM NaNH4HPO44 H2O)和 0.4 mL 50% 的混合物中制备噬菌体肉汤葡萄糖。倒置充分混合。
- 噬菌体感染 沙门氏菌 培养物
- 将 肠溶链球菌 过夜培养物稀释至 1:100,终体积为 5 mL LB,并加入 0.1 mL 裂解物(浓度为 10、9-10、12 PFU/mL)。在 37 °C 和 200 rpm 下孵育 24 小时。
- 在平板测定中测定噬菌体滴度作为 PFU
- 为了量化感染培养物中噬菌体的数量,进行平板测定以确定噬菌体滴度为 PFU/mL,如下所述。
- 将 1 mL 培养物转移到试管中。以 17,115 x g 离心 2 分钟,然后将上清液倒入新标记的小瓶中。
- 向样品瓶中加入 100 μL 氯仿并涡旋以去除溶液中的细菌碎片。按照步骤 1.2.3 进行操作。
- 为了量化感染培养物中噬菌体的数量,进行平板测定以确定噬菌体滴度为 PFU/mL,如下所述。
2. 从感染的 肠道沙门氏菌 培养物中去除噬菌体(见 图 2)
注:为了监测噬菌体的去除过程,在整个清洁方案中采用不同的等分试样进行滴定。总共有 8 个等分试样,用于确认 PFU/mL 的数量在整个清洁过程中不断减少,直到完全消除。
- 去除悬浮液中的噬菌体
- 在 50 mL 锥形离心管中,在 5 mL 0.22 μm 过滤的 LB 中将含有噬菌体的细菌培养物稀释至 1:100。滴定点 1:这是用于滴定的第一个等分试样(起始感染 的沙门氏菌 培养物)。
- 将悬浮液以 2,377 x g 离心 10 分钟。小心去除上清液。不要使用移液器;倒入试管。尝试在锥形离心管底部留出一些体积,以确保细胞的存在。
- 用 10 mL 的 0.22 μm 过滤 LB 培养基洗涤细菌细胞。重复离心和洗涤步骤 3 次。将沉淀重悬于 5 mL 的 0.22 μm 过滤 LB 中。滴定点 2:这是用于滴定的第二个等分试样。
- 使用真空过滤系统将 50 μL 上述细菌悬浮液通过无菌 0.45 μm 过滤器(参见 材料表)转移。用 100 mL 的 0.22 μm 过滤 LB 冲洗过滤器。
注:噬菌体 9NA 的头部尺寸为 0.067 μm。相比之下, 沙门氏菌 长 2-5 μm,宽 0.5-1.5 μm。因此,如果使用 0.45 μm 的膜,大多数细菌不会通过,因为它们大于 0.45 μm,而大多数噬菌体会通过 0.45 μm 的滤孔。
- 去除 沙门氏菌 细胞内的噬菌体
- 为了促进噬菌体从细菌细胞中释放,将含有细胞的 0.45 μm 过滤器在培养箱内于 37 °C 孵育 2 小时,而无需拆卸系统。
注:该孵育步骤是促进感染细菌裂解和细胞内噬菌体释放到过滤器中所必需的。 - 使用真空过滤系统清洗 0.45 μm 过滤器,用 100 mL 0.22 μm 过滤的 LB 冲洗过滤器。
- 在 2 mL 的 2x LB(20 g/L 胰蛋白胨、10 g/L NaCl、10 g/L 酵母提取物;参见 材料表)中从过滤器中回收细菌细胞。为此,使用无菌镊子将 0.45 μm 过滤器转移到培养瓶中。然后,在过滤器上加入 2 mL 的 2x LB 并移液数次以将细胞从过滤器中释放出来。
- 以 10,354 x g 的速度离心 1 mL 该悬浮液 2 分钟。弃去上清液,用 1 mL 过滤的 LB 培养基洗涤细菌细胞 3 次。将细胞重悬于 1 mL 的 2x LB 中。滴定点 3:这是滴定的第三等分试样。
注:强烈建议使用浓度更高的 LB (2x LB),因为如下所述,将添加商业脂多糖 (LPS),并且该 LPS 溶于水,从而稀释培养基。
- 为了促进噬菌体从细菌细胞中释放,将含有细胞的 0.45 μm 过滤器在培养箱内于 37 °C 孵育 2 小时,而无需拆卸系统。
- 如下所述,通过用商业脂多糖欺骗细菌细胞释放的噬菌体来避免再次感染。
- 在商业 肠链球菌 脂多糖 (LPS;参见 材料表) 存在下,将 20 μL 细菌悬浮液在 1 mL 0.22 μm 过滤的 LB 中孵育至终浓度为 3.75 mg/mL,在 37 °C 下以 200 rpm 振荡。
注:从细胞释放到培养基中的噬菌体可能与市售肠 溶血球菌 LPS 而不是细菌 LPS 结合。 - 孵育 2 小时后,将 1 mL 培养物转移到微量离心管中,并以 10,354 x g 的速度将悬浮液离心 2 分钟 。 滴定点 4:使用上清液作为第四份等分试样进行滴定。
- 用 1 mL 过滤的 LB 培养基洗涤沉淀 3 次。
- 洗涤后,将沉淀重悬于 1 mL 的 2x LB 中。将 20 μL 该混合物放入 1 mL 0.22 μm 过滤的 LB 和 0.8 mg/mL 商业 LPS 中,在 37 °C 下孵育 2 小时,并以 200 rpm 摇动。滴定点 5:使用剩余的细菌悬浮液进行第 5 次滴定。
- 孵育 2 小时后,将 1 mL 培养物转移到微量离心管中,并以 10,354 x g 的速度离心 2 分钟沉淀。滴定点 6:将上清液倒入新样品瓶中,将上清液作为第六等分试样进行滴定。
- 用过滤的 LB 洗涤细菌细胞 3 次。洗涤后,将收集的细胞重悬于 1 mL 的 0.22 μm 过滤 LB 中。
- 使用真空过滤系统将 1 mL 细菌悬浮液通过无菌 0.45 μm 过滤器。使用 100 mL 的 0.22 μm 过滤 LB 冲洗过滤器。
- 从 2 mL 2x LB 培养基中的 0.45 μm 过滤器中回收细胞。为此,使用无菌镊子将 0.45 μm 过滤器转移到培养瓶中。然后,在过滤器上加入 2 mL 的 2x LB 并移液数次以尝试从过滤器中释放细胞。
- 以 10,354 x g 的离心速度离心 2 分钟,沉淀 1 mL 细胞。用 0.22 μm 过滤的 LB 洗涤 3 次。洗涤后,将收集的细胞重悬于 1 mL 的 2x LB 中。滴定点 7:这是滴定的第七个等分试样。该等分试样将告知细菌培养物中噬菌体去除的效率。
- 在商业 肠链球菌 脂多糖 (LPS;参见 材料表) 存在下,将 20 μL 细菌悬浮液在 1 mL 0.22 μm 过滤的 LB 中孵育至终浓度为 3.75 mg/mL,在 37 °C 下以 200 rpm 振荡。
3. 噬菌体去除后无噬菌体沙 门氏菌 培养物的制备
- 准备无噬菌体接种物。将 100 μL 细菌悬液中的细胞加入 900 μL 含有 0.45 mg/mL LPS 的 LB 中。在 37 °C 下孵育 24 小时,并以 200 rpm 振荡。
- 滴定点 8:准备第 8 份等分试样进行滴定(最终可能未感染的培养物)。这一点是为了确认不存在噬菌体再感染。
结果
肠道沙门氏菌和其他革兰氏阴性菌的外膜含有 LPS。LPS 的 O 抗原是噬菌体 9NA 感染沙门氏菌培养物的常用受体16,17。
鉴于噬菌体对 LPS 的 O 抗原或核心多糖区域的特异性亲和力,我们想检查 肠道沙门氏菌 商业 LPS 是否可以用作排除 9NA 噬菌体的诱饵。为此,我们将已知浓度的商业 LPS 和 9NA 裂解物混合,然后进行滴定。将市售 LPS 和 9NA 噬菌体以总体积为 200 μL 的体积混合,并在 37 °C 下孵育 2 小时而不振动。滴定时,将 100 μL LPS-9NA 混合物和 60 μL 过夜培养物加入 5 mL LB 软琼脂中,倒入 LB 板的顶部。将板在 37 °C 下孵育 24 小时。 如图 3 所示,当肠道 链球菌 商业 LPS 浓度增加时,裂解物滴度成比例降低。这些结果表明,商业 LPS 作为 9NA 噬菌体的诱饵正确发挥作用。
有趣的是,细菌和噬菌体的数量促进了噬菌体 9NA 裂解 沙门氏菌 细胞和释放噬菌体所需的时间(图 4)。为了研究这方面如何影响噬菌体去除方案,我们测试了不同的细菌:噬菌体比例(1:1、1:10、1:100 和 1:1000)。如图 4 所示,在加入 9NA 噬菌体后 1.5-2.5 小时,培养物的 OD600 nm 下降。OD600 nm 值接近零表示细菌培养物正在被裂解18。出于这个原因,该方案中的孵育时间定义为 2 小时,以确保沙 门氏菌 细胞内包含的噬菌体有足够的时间裂解细菌并释放。在执行此方案之前,应估计每个宿主噬菌体系统的此时间。
一旦我们确定商业 LPS 作为诱饵和 9NA 感染细菌的裂解时间,我们就执行了描述的清洁噬菌体感染沙 门氏菌 培养物的方案(图 1 和 图 2)。为了监测方案每个步骤中噬菌体的存在,我们进行了噬菌斑测定,以计算所得培养物在不同点的 100 μL 所得混合物中的感染性(图 5)。我们可以观察到,重复洗涤和过滤不足以从培养物中消除噬菌体(滴定点 1-3);然而,一旦我们采用商业 LPS 孵育步骤(滴定点 4),噬菌体的数量就会减少。完全去除细菌培养物中噬菌体的关键步骤是与市售 LPS(滴定点 6)进行第二次孵育。此步骤对于成功去除 沙门氏菌 培养物中的 9NA 噬菌体至关重要。
该方案的一个有趣方面是了解噬菌体去除步骤后的噬菌体抗性水平。将噬菌体与噬菌体抗性细菌分离的程序没有用处。因此,揭示残留在培养物中的细菌对噬菌体敏感至关重要。为了证明易感噬菌体细胞在手术后仍留在培养物中,我们使用伊文思蓝铀氨酸 (EBU) 平板测定法筛选噬菌体污染19。EBU 板由补充有 10 mL/L K2、HPO4 25%、5 mL/L 葡萄糖 50%、2.5 mL/L 荧光素 1%、1.25 mL/L 伊文思蓝 1% 和 15 g/L 琼脂的 LB 培养基制成。使用 9NA 噬菌体在 EBU 板上交叉划线来区分噬菌体抗性和噬菌体敏感分离株(图 6)。在清洁方案结束时获得的细菌培养物用于获得分离的菌落,检查其是否存在噬菌体污染(图 6B)。我们可以观察到耐药细胞和敏感细胞的存在。该方案不赞成选择噬菌体抗性细胞;它只消除噬菌体。
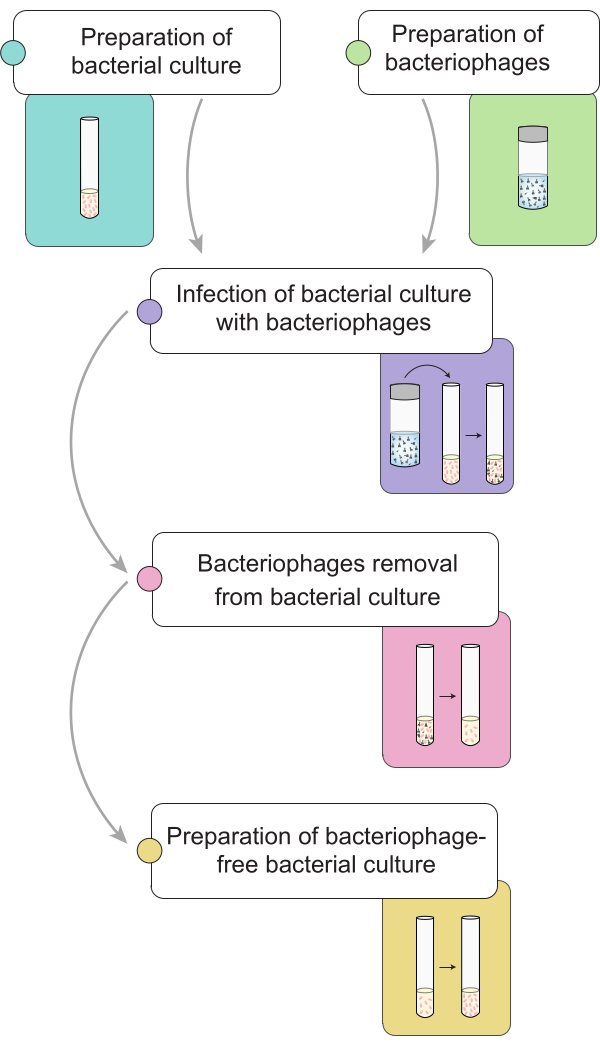
图 1:消除 沙门氏菌 培养物中噬菌体的程序简要概述。 工作流程分为不同的阶段:细菌培养物和裂解物的制备、噬菌体感染细菌培养物、从感染的细菌培养物中去除噬菌体以及制备不含噬菌体的细菌接种物。 请单击此处查看此图的较大版本。
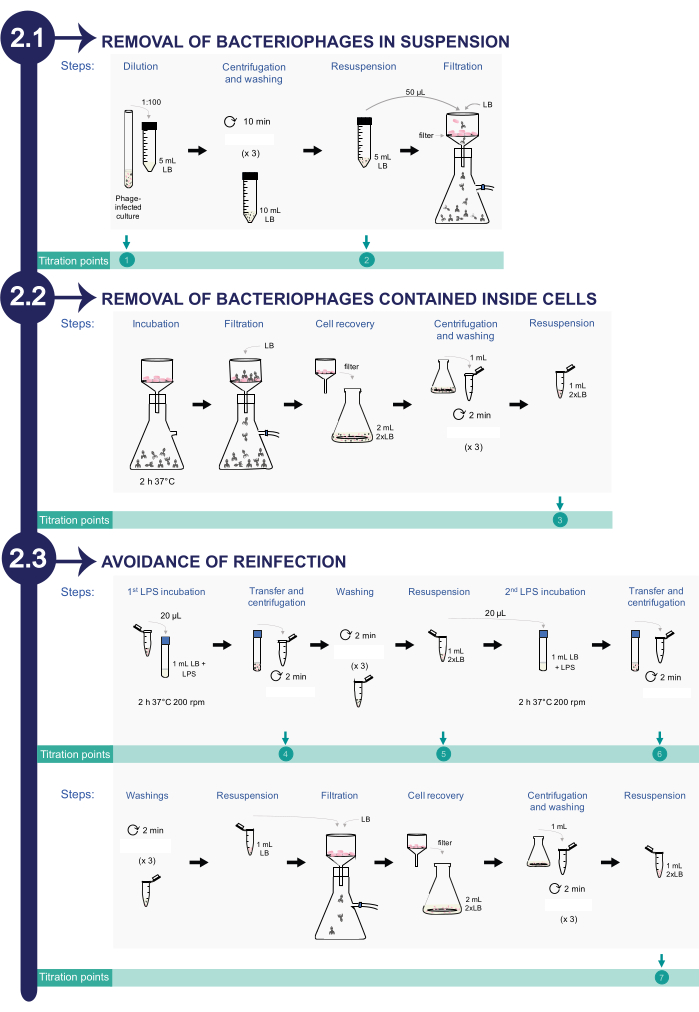
图 2:从受感染的 沙门氏菌中去除噬菌体进入 ica 培养物的程序。 该过程包括三个阶段:1) 去除悬浮液中的噬菌体,2) 去除细菌细胞内的噬菌体,以及 3) 避免再次感染。 请单击此处查看此图的较大版本。
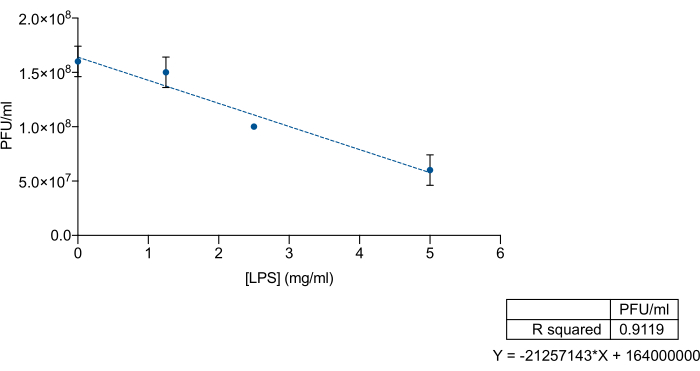
图 3:LPS 诱饵测定法,用于测量 肠道沙门氏菌 商业 LPS 结合噬菌体的效率。 在浓度递增的 肠道沙门氏菌 商业 LPS 下滴定 9NA 裂解物 (PFU/mL)。实验一式三份进行。给出了平均值和标准差。 请单击此处查看此图的较大版本。
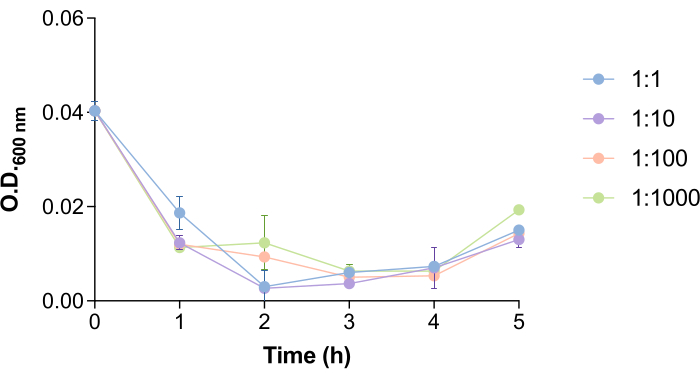
图 4: 沙门氏菌 培养物中的噬菌体 9NA 裂解时间。 在噬菌体 9NA 存在下,肠道 沙门氏菌 培养物的生长曲线,细菌:噬菌体比例为 1:1、1:10、1:100 和 1:1000。实验一式三份进行。给出了平均值和标准差。 请单击此处查看此图的较大版本。
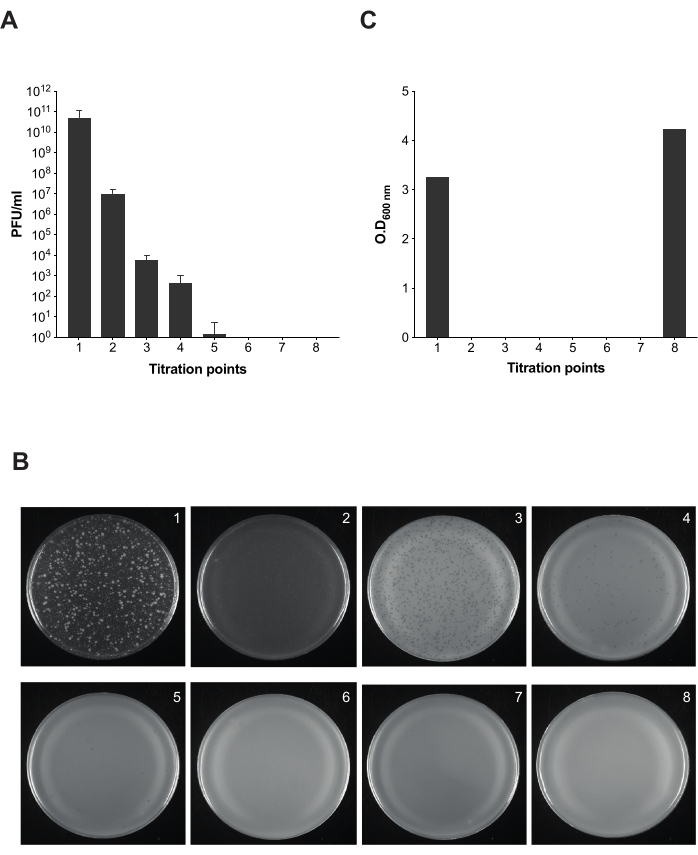
图 5: 沙门氏菌 培养物中噬菌体去除过程中感染性检测的噬菌斑测定。 (A) 在方案的不同点滴定 8 个等分试样(滴定点 1-7 在 图 2 中标记)。本实验采用 肠道沙门氏菌 鼠伤寒血清型菌株 ATCC 14028 opvAB::lacZ (SV8011) 和噬菌体 9NA。实验一式三份进行,并显示平均值和标准差。(B) 采用覆盖技术,使用来自 8 个滴定点的等分试样获得含有 肠沙门氏菌 的软琼脂平板。板从左到右对应于滴定点 1-8。(C) 噬菌体去除方案不同时间细菌培养物在 600 nm 处的光密度。 请单击此处查看此图的较大版本。
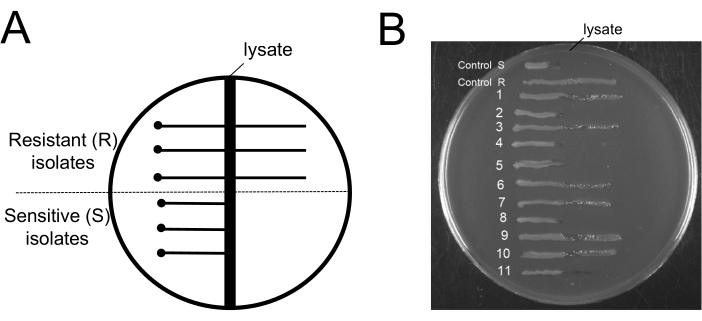
图 6:噬菌体去除程序后噬菌体耐药细菌的检测。(A) 用于交叉条纹琼脂测定的典型 EBU 琼脂板的示意图:中心的垂直深色区域代表 9NA 裂解物的区域。圆点表示在距离裂解物区安全距离处接种测试细胞的位置,水平实线表示在裂解物区生长的噬菌体抗性细胞或未在裂解物区之外生长的噬菌体敏感细胞。(B) 在去除方案结束时获得用于检测 11 个菌落的 EBU 平板测定。对照 R 和 S 分别是噬菌体抗性和噬菌体敏感分离株的例子。请单击此处查看此图的较大版本。
讨论
噬菌体采用多种策略来识别和感染细菌宿主。细菌表面的不同分子结构可以充当噬菌体受体:蛋白质、多糖、脂多糖 (LPS) 和碳水化合物部分20。在革兰氏阴性菌中,LPS 是噬菌体的常见受体。此外,其他受体是外膜蛋白、菌毛和鞭毛21。
在这项工作中,基于识别 LPS8 的噬菌体和细菌之间的特异性相互作用已被用于开发一种高效的方案,用于消除感染细菌培养物中的噬菌体(图 1 和 图 2)。我们的方案不赞成选择噬菌体抗性细胞;它只消除噬菌体(图 6)。在执行此噬菌体去除方案后,噬菌体敏感细胞和噬菌体抗性细胞均保留在细菌培养物中。
发生噬菌体感染时,传统的标准做法是尝试消除所有受污染的材料,然后进行清洁和灭菌22。净化程序包括将细菌培养物暴露在高温等压力条件下,以部分或完全消除细菌细胞。如代表性结果所述,该方案中的关键步骤是将噬菌体感染的细菌培养物与商业 LPS(一种对细菌培养物无害的物质)一起孵育。这有助于保持细菌培养物的活力,并为发酵罐和生物反应器中的工业应用提供显著优势。
该方案中的孵育时间为 2 小时,以确保有足够的时间进行细菌细胞的噬菌体裂解。如果要使用不同的细菌菌株和噬菌体,则应考虑并由用户定义此参数。在这种情况下,应在实验前进行类似于 图 4 中描述的分析。
有趣的是,这种清洁方案的功效也可以通过采用监测给定样品噬菌体含量的测定法来分析。从这个意义上说,表观遗传生物传感器是噬菌体检测的新型工具23。一种能够检测使用 LPS 作为受体的 coliphage 的著名噬菌体生物传感器是 opvAB::gfp 系统 13,18,23,24。该噬菌体生物传感器在存在使用 O 抗原作为受体的噬菌体的情况下检测 OpvABON 亚群的增加。从这个意义上说,我们可以使用 opvAB::gfp 融合来监测该方案的各个步骤和/或不同培养基和条件下的 LPS 结合噬菌体。这些方法对于确定可能需要有效方案的时间和地点可能很有价值。
虽然 LPS 识别很常见,但噬菌体还可以利用细菌细胞上的各种其他表面受体进行附着和感染。在这里,我们使用革兰氏阴性沙门氏菌作为代表性肠杆菌和使用 LPS 作为受体和基因组射出触发器的噬菌体 9NA。其他肠杆菌噬菌体(例如,大肠杆菌 T5)与 LPS 松散结合,需要外膜蛋白进行基因组注射。所描述的方案适用于识别并需要 LPS 的 O 抗原以成功感染的噬菌体,例如 9NA、Det7 和 P22 13,25,26,27。因此,成功实施该方案对细菌培养物进行噬菌体去污涉及确定噬菌体感染的来源是否需要识别宿主中的 LPS。
总之,尽管该方案存在潜在局限性,但我们的代表性结果清楚地表明,该方法是清洁使用 LPS 作为受体和基因组射出触发器的噬菌体的细菌 沙门氏菌 培养物的有力工具。
披露声明
作者没有什么可披露的。
致谢
我们感谢 Carmen R. Beuzón 博士和 Rocío Carvajal-Holguera 的有益讨论和建议。这项工作得到了 MICIU/AEI/10.13039/5011100011033 资助的 PID2020-116995RB-I00 和塞维利亚大学的 VI Plan Propio de Investigación y Transferencia 的支持。
材料
| Name | Company | Catalog Number | Comments |
| 20 mL syringe | BD Discardit II | 300296 | No special requirements |
| 50 mL conical tubes | Avantor | 525-0610 | No special requirements |
| 90 x 14 mm Petri dishes | Deltalab | 200209 | No special requirements |
| Agar | Sigma-Aldrich | A1296 | No special requirements |
| Bacteriophage lysate | Minimal concentration: 109 PFU/mL | ||
| Centrifuge | Eppendorf | No special requirements | |
| Chloroform | Panreac | 131252 | No special requirements |
| Citric acid · H2O | Merck | 1.00247 | |
| Colony counter | No special requirements | ||
| Evans Blue | Sigma-Aldrich | E-2129 | |
| Flasks | No special requirements | ||
| Fluorescein sodium salt | Sigma-Aldrich | F-6377 | |
| Forceps | No special requirements | ||
| Glass tubes | No special requirements | ||
| Glass tubes for lysate | No special requirements | ||
| Glucose | Sigma-Aldrich | G7021 | |
| K2HPO4 | Merck | 1.05104.1000 | |
| K2HPO4 anhydrous | Merck | 1.05104 | |
| Lipopolysaccharide from Salmonella enterica serotype Typhimurium | Sigma-Aldrich | L6511-25 mg | Dissolved in sterile water |
| Membrane 0.45 µm | MF-Millipore | HAWP02500 | No special requirements |
| MgSO4 · 7 H2O | Merck | 1.05886 | |
| NaCl | Sigma-Aldrich | S9888 | No special requirements |
| NaNH4HPO4 · 4 H2O | Sigma-Aldrich | S9506 | |
| Peptone | iNtRON | Ba2001 | No special requirements |
| Syringe Filter 0.22 µm | Millex | SLGSR33SB | No special requirements |
| Toothpicks | No special requirements | ||
| Tryptone | Panreac | 403682.1210 | No special requirements |
| Vacuum pump | Thermo Scientific | No special requirements | |
| Yeast extract | iNtRON | 48045 | No special requirements |
参考文献
- Kasman, L. M., Porter, L. D. . Bacteriophages. , (2024).
- Lawrence, D., Baldridge, M. T., Handley, S. A. Phages and human health: More than idle hitchhikers. Viruses. 11 (7), 587 (2019).
- Beller, L., Matthijnssens, J. What is (not) known about the dynamics of the human gut virome in health and disease. Curr Opin Virol. 37, 52-57 (2019).
- Roux, S., Hallam, S. J., Woyke, T., Sullivan, M. B. Viral dark matter and virus-host interactions resolved from publicly available microbial genomes. eLife. 4, 08490 (2015).
- Wommack, K. E., Colwell, R. R. Virioplankton: Viruses in aquatic ecosystems. Microbiol Mol Biol Rev. 64 (1), 69-114 (2000).
- Louten, J. Virus structure and classification. Essentl Human Virol. , 19-29 (2016).
- Egido, J. E., Costa, A. R., Aparicio-Maldonado, C., Haas, P. J., Brouns, S. J. J. Mechanisms and clinical importance of bacteriophage resistance. FEMS Microbiol Rev. 46 (1), 048 (2022).
- Rakhuba, D. V., Kolomiets, E. I., Dey, E. S., Novik, G. I. Bacteriophage receptors, mechanisms of phage adsorption and penetration into host cell. Polish J Microbiol. 59 (3), 145-155 (2010).
- Lindberg, A. A. Bacteriophage receptors. Ann Rev Microbiol. 27 (1), 205-241 (1973).
- Koskella, B., Meaden, S. Understanding bacteriophage specificity in natural microbial communities. Viruses. 5 (3), 806-823 (2013).
- Marcó, M. B., Moineau, S., Quiberoni, A. Bacteriophages and dairy fermentations. Bacteriophage. 2 (3), 149-158 (2012).
- Fernández-Fernández, R., et al. Evolution of a bistable genetic system in fluctuating and non-fluctuating environments. bioRxiv. , (2024).
- Cota, I., Blanc-Potard, A. B., Casadesús, J. STM2209-STM2208 (opvAB): a phase variation locus of Salmonella enterica involved in control of O-antigen chain length. PloS one. 7 (5), e36863 (2012).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods Mol Biol. 501, 69-76 (2009).
- Ackermann, H. W. Tailed bacteriophages: The order Caudovirales. Adv Virus Res. 51, 135-201 (1998).
- Wilkinson, R. G., Gemski, P., Stocker, B. A. D. Non-smooth mutants of Salmonella typhimurium: Differentiation by phage sensitivity and genetic mapping. J Gen Microbiol. 70 (3), 527-554 (1972).
- Casjens, S. R., Leavitt, J. C., Hatfull, G. F., Hendrix, R. W. Genome sequence of Salmonella Phage 9NA. Genome Announc. 2 (4), e00531 (2014).
- Cota, I., et al. Epigenetic control of Salmonella enterica O-antigen chain length: A tradeoff between virulence and bacteriophage resistance. PLoS Genet. 11 (11), e1005667 (2015).
- Chan, K., Botstein, D., Watanabe, T., Ogata, Y. Specialized transduction of tetracycline resistance by phage P22 in Salmonella typhimurium. Virology. 50, 883-898 (1972).
- Bertozzi Silva, J., Storms, Z., Sauvageau, D. Host receptors for bacteriophage adsorption. FEMS Microbiol Lett. 363 (4), 002 (2016).
- Sørensen, M. C. H., et al. Bacteriophage F336 recognizes the capsular phosphoramidate modification of Campylobacter jejuni NCTC11168. J Bacteriol. 193 (23), 6742-6749 (2011).
- Ogata, S., Hongo, M. Bacteriophages of the Genus Clostridium. Adv Appl Microbiol. 25, 241-273 (1979).
- Olivenza, D. R., Casadesús, J., Ansaldi, M. Epigenetic biosensors for bacteriophage detection and phage receptor discrimination. Environ Microbiol. 22 (8), 3126-3142 (2020).
- Olivenza, D. R., et al. A portable epigenetic switch for bistable gene expression in bacteria. Sci Rep. 9 (1), 862 (2019).
- Walter, M., et al. Structure of the receptor-binding protein of bacteriophage Det7: a Podoviral tail spike in a Myovirus. J Virol. 82 (5), 2265-2273 (2008).
- Davies, M. R., Broadbent, S. E., Harris, S. R., Thomson, N. R., vander Woude, M. W. Horizontally acquired Glycosyltransferase operons drive Salmonellae lipopolysaccharide diversity. PLoS Genet. 9 (6), e1003568 (2013).
- Wahl, A., Battesti, A., Ansaldi, M. Prophages in Salmonella enterica: a driving force in reshaping the genome and physiology of their bacterial host. Mol Microbiol. 111 (2), 303-316 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。