Method Article
감염된 살모넬라균 배양물에서 박테리오파지 제거
요약
지구상에서 어디에나 존재하며 다양한 박테리오파지는 박테리아 숙주 내에서 감염되고 복제되어 미생물 생태계에서 중요한 역할을 합니다. 그들의 중요성에도 불구하고, 그들의 존재는 산업 공정을 혼란에 빠뜨릴 수 있습니다. 우리는 살모넬라 배양에서 박테리오파지를 제거하기 위해 박테리아 지질다당류를 사용하는 방법을 개발했습니다.
초록
박테리오파지(Bacteriophage) 또는 간단히 파지(phage)는 미생물 환경에서 중요한 역할을 하여 박테리아 개체군에 영향을 미치고 박테리아 개체군의 진화와 상호 작용을 형성합니다. 이 유기체는 박테리아 숙주 내에서 감염되고 복제되는 바이러스입니다. 파지는 지구상의 어디에나 존재하며 매우 다양하고 풍부합니다. 박테리오파지는 다양한 환경에서 중요한 역할을 하고 미생물학 및 생태학 연구의 핵심 영역이지만, 특정 산업 공정이나 제품에서는 박테리오파지의 존재가 바람직하지 않을 수 있습니다. 지구상에 박테리오파지가 풍부하고 편재한다는 점을 고려할 때, 박테리아 배양에서 박테리오파지를 제거하기 위한 절차의 설계는 배양물의 무결성을 보존하고 정확한 실험 결과 또는 제품 품질을 보장하기 위해 다양한 실험실 및 산업 응용 분야에서 매우 중요합니다. 여기에서는 그람 음성 박테리아의 외막에 위치한 지질다당류(LPS)를 사용하는 전략을 사용하여 감염된 살모넬라균 배양에서 박테리오파지를 제거하기 위한 프로토콜을 미세 조정했습니다. 박테리아 LPS는 파지에 의한 숙주 인식에 중요한 역할을 하며, 당사는 이 특성을 활용하여 살모넬라균 배양에서 LPS를 수용체로 사용하는 파지를 제거하기 위한 효과적인 절차를 설계합니다.
서문
미생물 개체군은 자연 환경에서 여러 가지 문제에 직면해 있으며, 특히 심각한 위협은 박테리아를 감염시키는 바이러스인 박테리오파지(bacteriophage)에 의한 감염 가능성이다1. 이 바이러스는 지구상에 널리 퍼져 있으며 매우 다양하고 풍부합니다 2,3,4,5. 박테리오파지는 크기, 형태 및 게놈 조직이 다양하지만 모두 동일한 구조를 공유합니다: 파지로 인코딩된 단백질에 의해 형성된 캡시드로 둘러싸인 DNA 또는 RNA 게놈6. 박테리아는 그들에 대한 다양한 방어 기제를 진화시켰다7. 박테리오파지 감염의 핵심 측면은 특성 분석 및 검출과 관련이 있으며, 꼬리 섬유에 존재하는 수용체 결합 도메인입니다. 박테리오파지는 박테리아 세포 표면의 특정 수용체 부위를 인식하고 결합하기 위해 수용체 결합 단백질 또는 꼬리 섬유라고 하는 단백질을 표면에 가지고 있습니다. 그람 음성 세균의 경우, 지질다당류(lipopolysaccharides, LPS), 외막 단백질(outer membrane protein), 필리(pili) 및/또는 편모(flagella)와 같은 표면 구조의 인식이 파지-세균 상호작용에 관여한다8. 박테리오파지와 박테리아 사이의 이러한 상호 작용은 매우 특이적이며 주로 숙주 표면에 부착하는 능력에 달려 있습니다. 지질다당류의 O-항원은 일반적으로 사용되는 수용체(receptor)9이다.
박테리오파지-박테리아 상호 작용에 대한 연구는 생물학적 관점에서 매력적일 뿐만 아니라 파지 요법 및 생명 공학과 같은 분야에서 실용적으로 응용할 수 있습니다. 박테리오파지는 예를 들어, 미생물 개체군을 변화시키는 것과 같은 다양한 상황에서 중요한 역할을 하지만10, 특정 산업 공정에서는 이들의 존재가 바람직하지 않을 수 있다. 제약, 생명 공학 및 식품 생산에서 박테리오파지의 존재는 최종 제품의 품질과 안전성에 영향을 미칠 수 있으므로 품질 표준을 충족하기 위해 박테리오파지를 제거하는 것이 필수적입니다. 박테리아 배양이 다양한 화합물(예: 단백질, 효소 또는 항생제)을 생산하는 데 사용되는 바이오프로세싱 및 바이오 제조에서 박테리오파지의 존재는 모든 공유 환경에서 박테리아 개체군의 균형을 맞추는 능력으로 인해 생산 공정의 중단으로 이어질 수 있습니다. 파지는 때때로 산업 미생물학자의 직업 생활을 악몽으로 바꿀 수 있다11. 파지를 제거하기 위한 효과적인 절차의 설계는 일관되고 신뢰할 수 있는 생산을 보장하여 공정 효율성을 향상시키는 데 매우 중요합니다. 이러한 산업적 측면 외에도 정밀도와 재현성이 중요한 연구 실험실 환경에서 박테리오파지를 제거하는 것은 정확하고 신뢰할 수 있는 결과를 얻는 데 필수적입니다. 또한, 파지의 제거는 다양한 가설을 테스트하기 위해 다양한 환경을 시뮬레이션하는 데 사용될 수도 있습니다12. 파지 도포 후 박테리아 계수와 같은 많은 파지 기반 연구에서 훨씬 더 신뢰할 수 있는 생존 가능한 수를 생성하기 위해 파지를 제거하는 단계가 도움이 될 수 있기 때문에 파지를 제거하는 것도 연구 환경에서 매우 유용할 수 있습니다.
콜로니 분리를 기반으로 한 파지 제거는 콜로니에 파지가 없는지 확인하는 데 며칠이 걸리는 반면, 여기에 설명된 절차를 사용하면 몇 시간 내에 파지가 없는 배양물을 생성할 수 있습니다. 이 프로토콜을 사용하면 박테리아 배양이 콜로니를 고립시키는 것을 막지 않고 박테리아 배양의 진화를 추적할 수 있습니다. 이러한 의미에서 변동하는 환경(파지의 존재 및/또는 부재)을 시뮬레이션하여 다양한 가설을 테스트할 수 있습니다. 또한, 이 프로토콜은 박테리아 배양에서 파지의 존재에 대한 정성적 및 정량적 분석을 가능하게 합니다.
요약하면, 박테리오파지 제거를 위한 비용 효율적인 절차를 설계하는 것은 다양한 산업에서 제품 품질, 안전성 및 공정 효율성을 유지하고 기초 및 응용 연구의 발전에 매우 중요합니다. 여기에서는 감염된 살모넬라 균 배양물에서 LPS를 수용체로 사용하는 박테리오파지를 제거하기 위해 LPS를 사용하는 매우 효과적인 프로토콜에 대해 설명하며, 이는 시간 효율적이고 최소한의 장비만 필요합니다.
프로토콜
참고: 파지 제거 절차를 수행하기 전에 9NA 박테리오파지에 감염된 살모넬라 배양액의 준비에 대해 설명합니다. 그림 1에는 박테리아 배양에서 박테리오파지 제거를 위한 전체 절차의 일반적인 표현이 나와 있습니다.
1. 박테리오파지에 감염된 살모넬라 균 배양물의 제조
- 세균 배양 준비
참고: 이 연구에서는 Salmonella enterica subsp. enterica serovar Typhimurium 균주 ATCC 14028 opvAB::lacZ (SV8011)13 를 사용했습니다.- -80 °C에서 회수한 관심 S. enterica 균주를 함유한 바이알을 얼음 위에 놓아 천천히 해동합니다.
- 해동된 디메틸 설폭사이드 스톡에서 S. enterica 균주를 Lennox 육수(LB) 플레이트(10g/L 트립톤, 5g/L NaCl, 5g/L 효모 추출물 및 15g/L 한천, 재료 표 참조)에 줄무늬를 표시합니다. 플레이트를 37°C에서 24시간 동안 배양합니다.
- 접종 바늘을 사용하여 S. enterica 플레이트에서 콜로니를 옮기고 유리관에 담긴 액체 LB 5mL에 접종합니다. 37 °C에서 24 시간 동안 200 rpm으로 흔들어 폭기 농축합니다.
- 박테리오파지 제제
참고: Siphoviridae(Caudovirales 목)14과에 속하는 박테리오파지 9NA가 이 프로토콜에 사용되었습니다. 특히, 지질다당류의 O-항원을 수용체로 사용하는 다른 살모넬라 엔테리카 박테리오파지를 사용할 수 있습니다.- 영양 육수 100mL(NB; 5g/L 펩톤 및 3g/L 효모 추출물), 50x E 염 2mL(38.78mM MgSO4 7H2O, 520.5mM 구연산H2O, 2.87MK2HPO4 무수 및 829 mM NaNH4HPO44 H2O) 및 0.4mL 50%의 혼합물에 농축 박테리오파지 용해물 0.4mL를 첨가하여 박테리오파지 육수를 준비합니다. 포도당. 반전으로 철저히 섞는다.
- 박테리오파지 농도의 대표적인 예는10,9PFU /mL(Plaque Forming Units/mL)일 수 있지만, 더 낮은 농도를 사용해도 유사한 결과를 얻을 수 있습니다( 재료 표 참조). 단계 1.2.3에 따라 박테리오파지 농도를 추정합니다.
- 아래 설명된 단계에 따라 용해물을 준비합니다.
- LB 5mL(트립톤 10g/L, NaCl 5g/L, 효모 추출물 5g)에 S. enterica 접종물을 준비합니다. 37 °C 및 200 rpm에서 24 시간 동안 배양합니다.
- 박테리오파지 육수 4mL와 S. enterica의 하룻밤 배양액 1mL를 섞습니다. 혼합물을 37°C 및 200rpm에서 8시간 동안 배양합니다.
- 박테리아와 박테리오파지를 구별하기 위해 혼합물을 2,377 x g 에서 20분 동안 원심분리합니다. 유리관에서 상층액을 회수하고 800μL의 클로로포름( 재료 표 참조)을 첨가한 다음 소용돌이칩니다. 클로로포름과의 혼합물을 사용할 때만 상부를 전달해야 합니다.
알림: 절차의 효율성에 영향을 줄 수 있는 잔류 클로로포름의 전달을 방지하는 것이 중요합니다. 클로로포름 단계는 방출된 파지를 최대화하기 위한 방법으로 분리 및 증식 프로토콜 모두에서 일반적입니다. 그러나 클로로포름은 일부 파지를 비활성화시키거나 감염성을 파괴할 수 있다15. 따라서 이 단계는 클로로포름에 내성이 있는 파지에만 적용할 수 있습니다. - 용해물을 실온에서 2시간 동안 유지한 다음 4°C에서 보관합니다.
- 준비된 용해물에 존재하는 박테리오파지의 수/mL당 PFU의 수를 정량화하기 위해 용해물을 적정화합니다. 이렇게 하려면 아래에 설명된 오버레이 기술을 수행합니다.
- S. enterica에 대한 접종물 5mL를 준비합니다. 37 °C에서 24 시간 동안 200 rpm으로 흔들어 폭기 농축합니다.
- 용해물을 LB로 연속 희석하여 가산 가능한 PFU/mL 수에 도달합니다. 필요한 희석 계수를 결정하려면 농축된 초기 박테리오파지 용해물을 식별하십시오. 약 109 PFU/mL에서 박테리오파지 농도의 대표적인 예로, 셀 수 있는 플라크 수를 생성하는 희석 인자는 106-10 10 사이입니다.
- 전날 준비한 적절한 용해물 희석액 100μL와 S . enterica 의 오버나이트 배양액 60μL를 액체 LB와 LB-한천을 1:1 비율로 혼합하고 응고를 피하기 위해 56°C에서 유지하여 제조한 LB 연질 한천 5mL에 첨가합니다. 거품을 피하고 조심스럽게 섞는다.
- 각 혼합물을 LB 플레이트 상단에 붓고 매체가 응고될 때까지 벤치에 두십시오.
- 37 °C에서 24 시간 동안 플레이트를 배양합니다. 각 접시에 있는 플라크의 수를 세십시오. PFU/mL의 수를 측정하려면 각 플레이트의 플라크 수에 희석 계수(FD)를 곱합니다. 효율적인 파지 감염을 위해서는 109-10 12 PFU/mL가 필요합니다.
PFU/mL= 아니요. 플레이트의 플라크 x FD
- 영양 육수 100mL(NB; 5g/L 펩톤 및 3g/L 효모 추출물), 50x E 염 2mL(38.78mM MgSO4 7H2O, 520.5mM 구연산H2O, 2.87MK2HPO4 무수 및 829 mM NaNH4HPO44 H2O) 및 0.4mL 50%의 혼합물에 농축 박테리오파지 용해물 0.4mL를 첨가하여 박테리오파지 육수를 준비합니다. 포도당. 반전으로 철저히 섞는다.
- 박테리오파지에 의한 살모넬라 배양균의 감염
- S. enterica의 야간 배양액을 최종 부피 5mL의 LB에서 1:100으로 희석하고 0.1mL의 용해물(10,9-10,12PFU/mL에서 농축)을 추가합니다. 37 °C 및 200 rpm에서 24 시간 동안 배양합니다.
- 플레이트 분석에서 PFU로서의 파지 역가 측정
- 감염된 배양물에서 박테리오파지의 수를 정량화하려면 아래 설명된 대로 플레이트 분석을 수행하여 파지 역가를 PFU/mL로 측정합니다.
- 배양액 1mL를 튜브에 옮깁니다. 17,115 x g 에서 2분 동안 원심분리하고 상층액을 라벨이 부착된 새 바이알에 붓습니다.
- 바이알과 와류에 클로로포름 100μL를 추가하여 용액에서 박테리아 파편을 제거합니다. 1.2.3단계를 따릅니다.
- 감염된 배양물에서 박테리오파지의 수를 정량화하려면 아래 설명된 대로 플레이트 분석을 수행하여 파지 역가를 PFU/mL로 측정합니다.
2. 감염된 살모넬라균 배양물에서 박테리오파지 제거(그림 2 참조)
참고: 박테리오파지 제거 과정을 모니터링하기 위해 적정을 위한 세척 프로토콜 전반에 걸쳐 다양한 부분 표본을 채취합니다. 전체적으로 PFU/mL의 수가 완전히 제거될 때까지 세척 과정 전반에 걸쳐 감소하고 있음을 확인하기 위한 8개의 부분 표본이 있습니다.
- 현탁액의 박테리오파지 제거
- 50mL 원추형 원심분리 튜브에서 0.22μm 여과된 LB 5mL에서 파지가 포함된 박테리아 배양액을 1:100으로 희석합니다. 적정 포인트 1: 이것은 적정(감염된 살모넬라 배양 시작)을 위한 첫 번째 부분 표본입니다.
- 2,377 x g에서 10분 동안 현탁액을 원심분리합니다. 상층액을 조심스럽게 제거하십시오. 피펫을 사용하지 마십시오. 튜브를 붓습니다. 세포가 있는지 확인하기 위해 원추형 원심분리기 튜브의 바닥에 약간의 부피를 남겨 두십시오.
- 0.22μm 여과된 LB 배지 10mL로 박테리아 세포를 세척합니다. 원심분리 및 세척 단계를 3번 반복합니다. 펠릿을 0.22 μm 여과된 LB 5 mL에 재현탁시킵니다. 적정 포인트 2: 이것은 적정을 위한 두 번째 부분 표본입니다.
- 진공 여과 시스템을 사용하여 위의 박테리아 현탁액 50μL를 멸균 0.45μm 필터( 재료 표 참조)를 통해 전달합니다. 100mL의 0.22μm 여과된 LB로 필터를 헹굽니다.
참고: 박테리오파지 9NA의 헤드 크기는 0.067μm입니다. 대조적으로, 살모넬라균 은 길이 2-5 μm, 너비 0.5-1.5 μm입니다. 따라서 0.45μm 멤브레인을 사용하면 대부분의 박테리아는 0.45μm보다 크기 때문에 통과하지 못하는 반면 대부분의 파지는 0.45μm 필터 기공을 통과합니다.
- 살모넬라균 세포 내에 함유된 박테리오파지 제거
- 박테리아 세포에서 파지 방출을 용이하게 하려면 시스템을 분해하지 않고 인큐베이터 내부에서 37°C에서 2시간 동안 세포가 포함된 0.45μm 필터를 배양합니다.
알림: 이 배양 단계는 감염된 박테리아의 용해와 세포 내 파지가 필터로 방출되는 것을 촉진하는 데 필요합니다. - 진공 여과 시스템을 사용하여 0.45μm 필터를 세척하고 0.22μm 여과된 LB 100mL로 필터를 헹굽니다.
- 필터에서 2xLB(20g/L 트립톤, 10g/L NaCl, 10g/L 효모 추출물, 재료 표 참조)로 박테리아 세포를 회수합니다. 이렇게 하려면 멸균 집게를 사용하여 0.45μm 필터를 플라스크에 옮깁니다. 그런 다음 필터와 피펫에 2mL의 2x LB를 여러 번 추가하여 필터에서 세포를 분리합니다.
- 이 현탁액 1mL를 10,354 x g에서 2분 동안 원심분리합니다. 상층액을 버리고 여과된 LB 배지 1mL로 박테리아 세포를 3배 세척합니다. 2x LB의 1 mL에 세포를 재현탁시킵니다. 적정 포인트 3: 이것은 적정을 위한 세 번째 부분 표본입니다.
참고: 아래에 설명된 대로 상업용 LPS(lipopolysaccharide)가 추가되고 이 LPS가 물에 용해되어 매체가 희석되기 때문에 더 농축된 LB(2x LB)를 사용하는 것이 좋습니다.
- 박테리아 세포에서 파지 방출을 용이하게 하려면 시스템을 분해하지 않고 인큐베이터 내부에서 37°C에서 2시간 동안 세포가 포함된 0.45μm 필터를 배양합니다.
- 세균 세포에서 방출되는 박테리오파지를 아래에 설명된 대로 상업용 지질다당류로 속여 재감염을 피하십시오.
- 상업용 S. enterica lipopolysaccharide(LPS, 재료 표 참조)의 존재 하에 박테리아 현탁액 20μL를 37°C에서 2시간 동안 200rpm에서 진탕하면서 0.22μm 여과된 LB 1mL에 3.75mg/mL의 최종 농도로 배양합니다.
참고: 세포에서 배지로 방출된 파지는 박테리아 LPS 대신 상업용 S. enterica LPS에 결합할 수 있습니다. - 2시간 배양 후 1mL의 배양액을 마이크로 원심분리기 튜브에 옮기고 현탁액을 10,354 x g에서 2분 동안 원심분리합니다 . 적정 포인트 4: 상층액을 적정을 위한 4차 부분 표본으로 사용하십시오.
- 여과된 LB 배지 1mL로 펠릿을 3번 세척합니다.
- 세척 후 펠릿을 1mL의 2x LB에 재현탁시킵니다. 이 혼합물 20μL의 0.22μm 여과 LB 1mL에 0.8mg/mL의 상업용 LPS와 함께 37°C에서 2시간 동안 200rpm에서 진탕하여 배양합니다. 적정 포인트 5: 나머지 박테리아 현탁액을 5차 적정에 사용합니다.
- 2시간 배양 후 10,354 x g에서 2분 동안 원심분리하여 1mL의 배양액을 마이크로 원심분리 튜브와 펠릿에 옮깁니다. 적정 포인트 6: 상층액을 새 바이알에 부어 적정을 위한 6번째 부분 표본으로 상층액을 사용합니다.
- 여과된 LB로 박테리아 세포를 3번 세척합니다. 세척 후 수집된 세포를 0.22μm 여과된 LB 1mL에 재현탁시킵니다.
- 진공 여과 시스템을 사용하여 1mL의 박테리아 현탁액을 제균 0.45μm 필터를 통과시킵니다. 100mL의 0.22μm 여과된 LB를 사용하여 필터를 헹굽니다.
- 2x LB 배지의 2mL에서 0.45μm 필터의 세포를 회수합니다. 이를 위해 멸균 겸자를 사용하여 0.45μm 필터를 플라스크에 옮깁니다. 그런 다음 필터와 피펫에 2mL의 2x LB를 여러 번 추가하여 필터에서 세포를 분리합니다.
- 10,354 x g에서 2분 동안 원심분리하여 1mL의 세포를 펠렛합니다. 0.22μm 필터 LB로 3회 세척합니다. 세척 후 수집된 세포를 1mL의 2x LB에 재현탁시킵니다. 적정 포인트 7: 이것은 적정을 위한 7번째 부분 표본입니다. 이 부분 표본은 박테리아 배양에서 파지 제거의 효율성에 대해 알려줍니다.
- 상업용 S. enterica lipopolysaccharide(LPS, 재료 표 참조)의 존재 하에 박테리아 현탁액 20μL를 37°C에서 2시간 동안 200rpm에서 진탕하면서 0.22μm 여과된 LB 1mL에 3.75mg/mL의 최종 농도로 배양합니다.
3. 박테리오파지 제거 후 박테리오파지가 없는 살모넬라 배양액의 제조
- 파지가 없는 접종물을 준비합니다. 박테리아 현탁액의 세포 100μL를 0.45mg/mL LPS를 포함하는 900μL의 LB에 추가합니다. 37 °C에서 24 시간 동안 200 rpm으로 진탕하여 배양합니다.
- 적정 포인트 8: 적정(최종적으로 잠재적인 비감염 배양)을 위해 여덟 번째 부분 표본을 준비합니다. 이 포인트는 파지 재감염이 없는지 확인하기 위함입니다.
결과
살모넬라균 및 기타 그람 음성 박테리아는 LPS를 포함하는 외막을 가지고 있습니다. LPS의 O-항원은 박테리오파지 9NA가 살모넬라 배양균을 감염시키기 위해 일반적으로 사용하는 수용체입니다16,17.
LPS의 O-항원 또는 코어 다당류 영역에 대한 박테리오파지의 특정 친화력을 감안할 때, 우리는 Salmonella enterica commercial LPS가 9NA 박테리오파지를 배제하기 위한 미끼로 사용될 수 있는지 여부를 조사하고 싶었습니다. 이를 위해 상용 LPS와 9NA 용해물의 알려진 농도를 혼합한 후 적정을 수행했습니다. 상업용 LPS 및 9NA 박테리오파지를 총 200μL의 부피로 혼합하고 37°C에서 2시간 동안 흔들지 않고 배양했습니다. 적정을 위해 100 μL의 LPS-9NA 혼합물과 60 μL의 오버나이트 배양액을 5 mL의 LB 연질 한천에 첨가하고 LB 플레이트의 상단에 부었습니다. 플레이트를 37°C에서 24시간 동안 배양하였다. 그림 3에서 볼 수 있듯이 용해물 역가는 S. enterica commercial LPS의 농도가 증가할 때 비례적으로 감소합니다. 이러한 결과는 상업용 LPS가 9NA 박테리오파지에 대한 미끼로 올바르게 기능한다는 것을 나타냅니다.
흥미롭게도, 파지 9NA가 살모넬라균 세포를 용해하고 파지를 방출하는 데 필요한 시간은 박테리아와 파지의 수에 의해 촉진됩니다(그림 4). 이 측면이 파지 제거 프로토콜에 어떤 영향을 미칠 수 있는지 연구하기 위해 다양한 박테리아, 즉 파지 비율(1:1, 1:10, 1:100 및 1:1000)을 테스트했습니다. 그림 4에서 볼 수 있듯이 9NA 파지를 첨가한 후 1.5-2.5시간 내에 배양액의 OD600nm 가 감소합니다. OD600 nm 값이 0에 가까우면 박테리아 배양이 용해되고 있음을 나타냅니다18. 이러한 이유로, 이 프로토콜의 배양 시간은 살모넬라 세포 내부에 포함된 파지가 박테리아를 용해하고 방출될 수 있는 충분한 시간을 보장하기 위해 2시간으로 정의되었습니다. 이 프로토콜을 수행하기 전에 각 호스트 파지 시스템에 대해 이 시간을 추정해야 합니다.
상업용 LPS가 미끼로 작용하고 9NA에 감염된 박테리아에 대한 용해 시간이 확인되면 박테리오파지의 감염된 살모넬라 배양균을 세척하기 위해 설명된 프로토콜을 수행했습니다(그림 1 및 그림 2). 프로토콜의 각 단계에서 박테리오파지의 존재를 모니터링하기 위해 플라크 분석을 수행하여 서로 다른 지점에서 생성된 혼합물 100μL에서 결과 배양물의 감염성을 계산했습니다(그림 5). 우리는 반복적인 세척 및 여과가 배양물에서 박테리오파지를 제거하기에 충분하지 않다는 것을 관찰할 수 있습니다(적정 포인트 1-3). 그러나 파지의 수는 상업용 LPS(적정 지점 4)로 배양 단계를 사용하자마자 감소합니다. 박테리아 배양에서 박테리오파지를 완전히 제거하기 위한 중요한 단계는 상업용 LPS(적정 포인트 6)를 사용한 두 번째 배양입니다. 이 단계는 살모넬라 균 배양에서 9NA 박테리오파지를 성공적으로 제거하는 데 필수적입니다.
이 프로토콜의 흥미로운 측면은 파지 제거 단계 후 파지 저항성 수준을 아는 것입니다. 파지 저항성 박테리아에서 파지를 분리하는 절차는 유용성이 없습니다. 이러한 이유로 배양액에 남아 있는 박테리아가 파지에 취약하다는 것을 밝히는 것이 중요합니다. 시술 후 감수성 파지 세포가 배양에 남아 있음을 입증하기 위해 Evans Blue Uranine(EBU) 플레이트 분석을 사용하여 파지 오염을 스크리닝했습니다19. EBU 플레이트는 10mL/L K2HPO4 25%, 5mL/L 포도당 50%, 2.5mL/L 플루오레세인 1%, 1.25mL/L Evans Blue 1% 및 15g/L 한천이 보충된 LB 배지로 만들어졌습니다. 9NA 파지가 있는 EBU 플레이트의 교차 줄무늬를 사용하여 파지 저항성 및 파지에 민감한 분리물을 구별했습니다(그림 6). 세척 프로토콜의 끝에서 얻은 박테리아 배양물을 사용하여 분리된 콜로니를 얻었고, 이를 통해 파지 오염을 확인했습니다(그림 6B). 우리는 저항성 세포와 민감한 세포의 존재를 관찰할 수 있습니다. 이 프로토콜은 파지 저항성 세포의 선택을 선호하지 않습니다. 박테리오파지만 제거합니다.
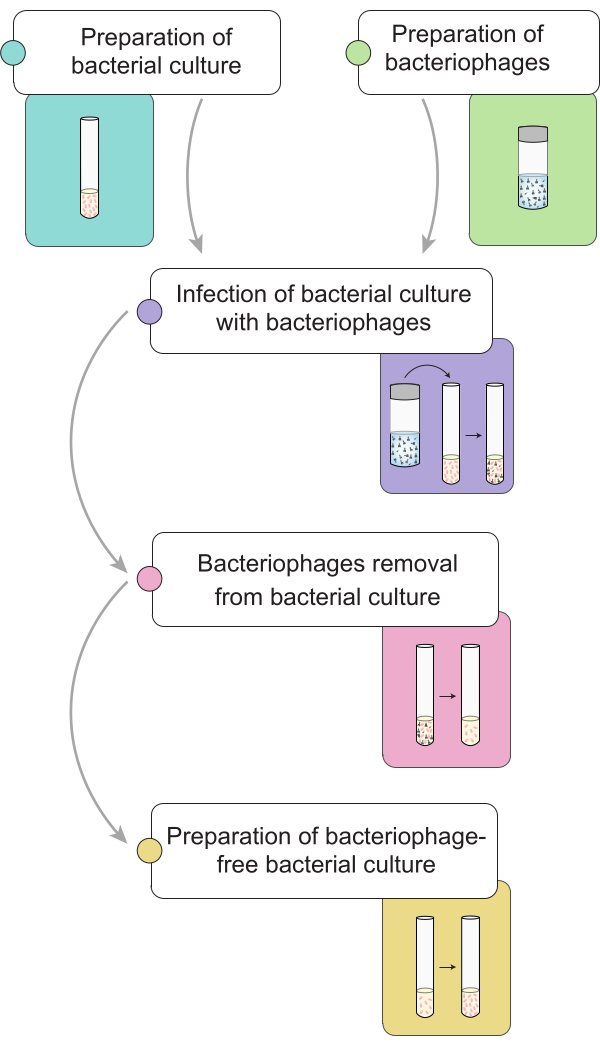
그림 1: 살모넬라 균 배양에서 박테리오파지를 제거하는 절차에 대한 간략한 개요. 워크플로우는 박테리아 배양 및 용해물 준비, 박테리오파지를 통한 세균 배양 감염, 감염된 세균 배양에서 박테리오파지 제거, 파지가 없는 세균 접종물 준비와 같은 여러 단계로 나뉩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
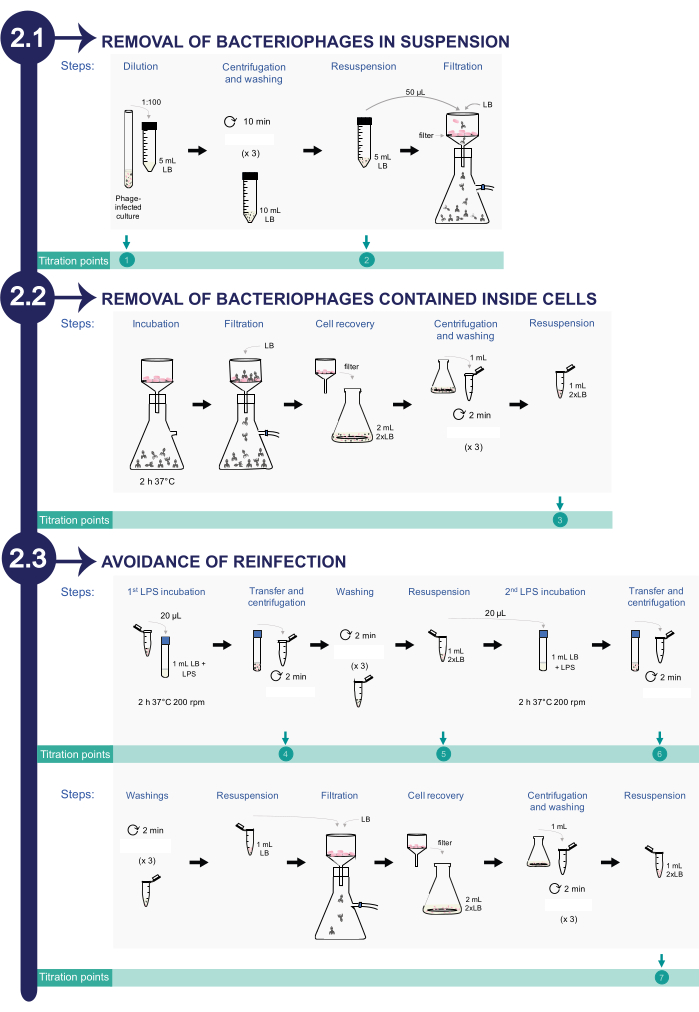
그림 2: 감염된 살모넬라균에서 박테리오파지를 제거하는 절차는 이카 배양에 들어갑니다. 이 과정은 1) 현탁 상태의 박테리오파지 제거, 2) 세균 세포 내에 포함된 파지 제거, 3) 재감염 방지의 3단계로 구성됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
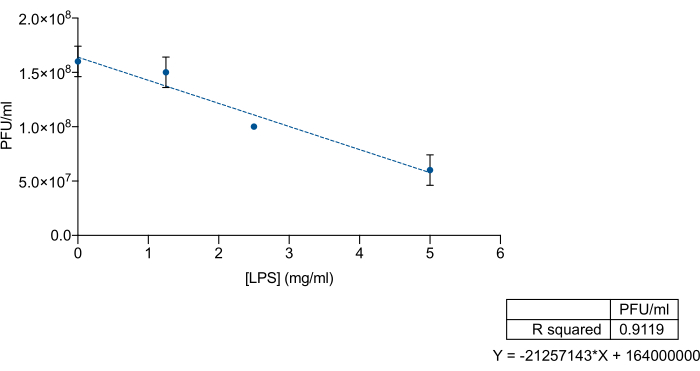
그림 3: 박테리오파지와 결합하기 위한 Salmonella enterica 상업용 LPS의 효율성을 측정하기 위한 LPS-미끼 분석. Salmonella enterica 상업용 LPS의 농도 증가에서 9NA 용해물(PFU/mL)의 적정. 실험은 세 번에 걸쳐 수행되었습니다. 평균 및 표준 편차가 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
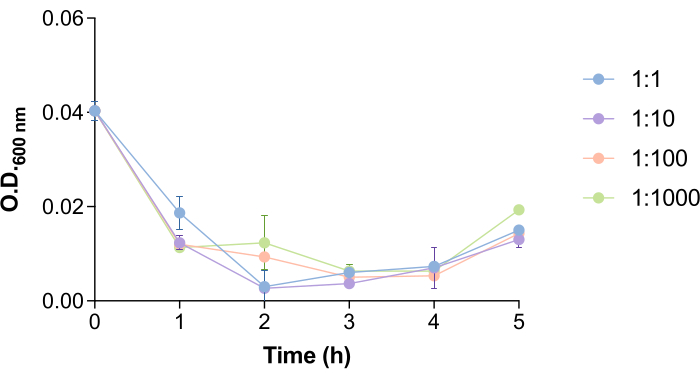
그림 4: 살모넬라 균 배양에서의 박테리오파지 9NA 용해 시간. 박테리아:파지 비율이 1:1, 1:10, 1:100 및 1:1000인 박테리오파지 9NA가 있는 경우 Salmonella enterica 배양의 성장 곡선. 실험은 세 번에 걸쳐 수행되었습니다. 평균 및 표준 편차가 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
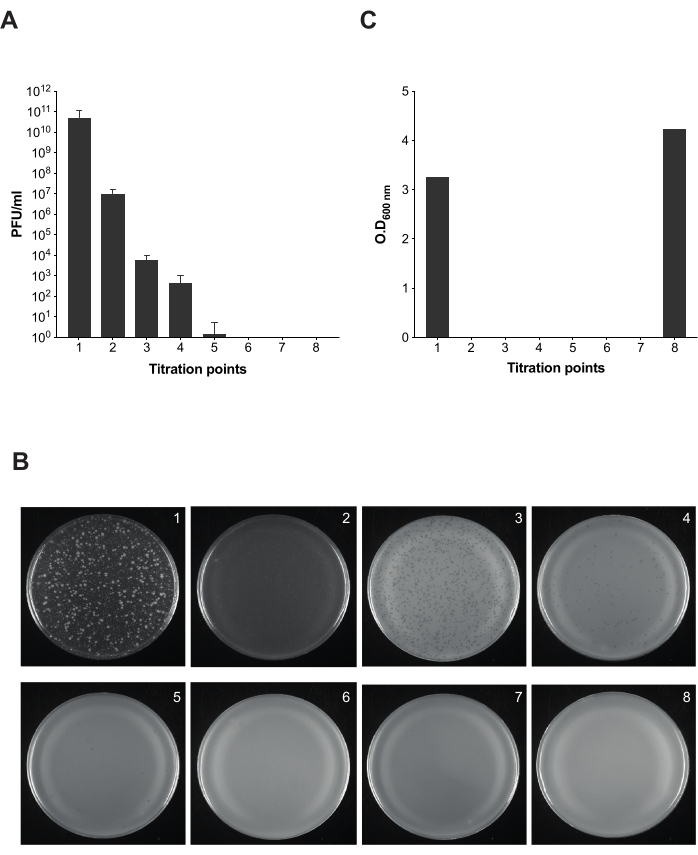
그림 5: 살모넬라균 배양에서 박테리오파지 제거 중 감염성 검사를 위한 플라크 분석. (A) 8개의 부분 표본에 대한 적정은 프로토콜의 서로 다른 지점에서 수행되었습니다(적정 지점 1-7은 그림 2에 표시되어 있음). 이 실험을 위해 Salmonella enterica serovar Typhimurium strain ATCC 14028 opvAB::lacZ (SV8011) 및 박테리오파지 9NA를 사용하였다. 실험은 세 번에 걸쳐 수행되었으며 평균 및 표준 편차가 표시됩니다. (B) Salmonella enterica 가 함유된 연질 한천 플레이트는 8개의 적정 지점에서 부분 표본을 사용하는 오버레이 기법에 따라 획득되었습니다. 플레이트는 적정 포인트 1-8과 함께 왼쪽에서 오른쪽으로 해당합니다. (C) 600 nm의 박테리아 배양에서 phage-remove protocol의 서로 다른 시간에 대한 광학 밀도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
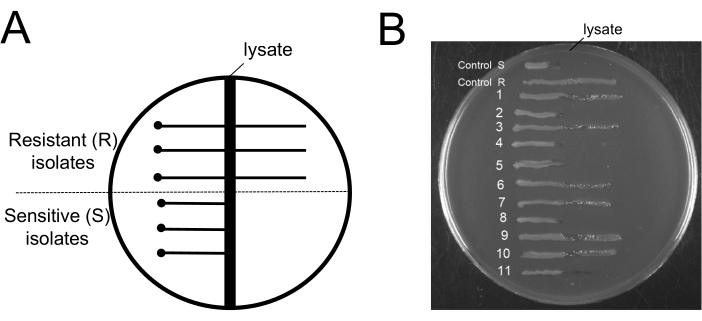
그림 6: 박테리오파지 제거 절차 후 파지 저항성 박테리아 테스트. (A) 교차 줄무늬 한천 분석에 사용되는 일반적인 EBU 한천 플레이트의 개략도: 중앙의 수직 어두운 영역은 9NA 용해물 영역을 나타냅니다. 점은 테스트된 세포가 용해물 영역에서 안전한 거리에서 접종되는 위치를 나타내고, 수평 실선은 용해물 영역을 가로질러 성장하는 파지 저항성 세포 또는 용해물 영역을 넘어 성장하지 않는 파지 민감성 세포를 나타냅니다. (B) 11개의 콜로니를 테스트하기 위한 EBU 플레이트 분석은 제거 프로토콜이 끝날 때 얻어졌습니다. 대조군 R 및 S는 각각 파지 저항성 및 파지 민감성 분리체의 예입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
박테리오파지는 박테리아 숙주를 인식하고 감염시키기 위해 다양한 전략을 사용합니다. 박테리아 표면의 다양한 분자 구조가 파지 수용체로 작용할 수 있습니다: 단백질, 다당류, 지질다당류(LPS) 및 탄수화물 부분20. 그람 음성 박테리아에서 LPS는 파지의 일반적인 수용체입니다. 또한 다른 수용체는 외막 단백질, pili 및 편모21입니다.
LPS8 의 인식을 기반으로 한 박테리오파지와 박테리아 간의 특정 상호 작용은 감염된 박테리아 배양에서 박테리오파지를 제거하기 위한 매우 효율적인 프로토콜의 개발을 위해 이 연구에서 활용되었습니다(그림 1 및 그림 2). 우리의 프로토콜은 파지 저항성 세포의 선택을 선호하지 않습니다. 박테리오파지만 제거합니다(그림 6). 파지 감수성 세포와 파지 저항성 세포는 모두 이 파지 제거 프로토콜을 수행한 후에도 박테리아 배양에 남아 있습니다.
파지 감염이 발생했을 때 전통적인 표준 관행은 오염된 모든 물질을 제거한 후 세척 및 멸균을 시도하는 것이다22. 오염 제거 절차에는 박테리아 세포를 부분적으로 또는 완전히 제거하기 위해 박테리아 배양액을 고온과 같은 스트레스가 많은 조건에 노출시키는 작업이 포함됩니다. 대표적인 결과에서 설명한 바와 같이, 이 프로토콜의 중요한 단계는 박테리아 배양에 무해한 물질인 상업용 LPS로 파지에 감염된 박테리아 배양물을 배양하는 것입니다. 이는 박테리아 배양의 생존력을 보존하는 데 도움이 되며 발효기 및 생물 반응기의 산업 응용 분야에 상당한 이점을 제공합니다.
이 프로토콜의 배양 시간은 박테리아 세포의 파지 용해를 위한 충분한 시간을 보장하기 위해 2시간입니다. 다른 박테리아 균주와 박테리오파지를 사용하는 경우 이 매개변수는 사용자가 고려하고 정의해야 합니다. 이 경우 그림 4 에 설명된 것과 유사한 분석을 실험 전에 수행해야 합니다.
흥미롭게도, 이 세척 프로토콜의 효능은 주어진 샘플의 파지 함량을 모니터링하는 분석을 사용하여 분석할 수도 있습니다. 이러한 의미에서, 후성유전학적 바이오센서는 박테리오파지 검출을 위한 새로운 도구이다23. LPS를 수용체로 사용하는 대장균을 검출할 수 있는 잘 알려진 파지 바이오센서는 opvAB::gfp 시스템(13,18,23,24)이다. 이 파지 바이오센서는 O-항원을 수용체로 사용하는 파지가 있을 때 OpvABON 하위 집단의 증가를 감지합니다. 이러한 의미에서 opvAB::gfp fusion을 사용하여 이 프로토콜의 다양한 단계 및/또는 다양한 매체 및 조건에서 LPS 결합 파지를 모니터링할 수 있습니다. 이러한 접근 방식은 효과적인 프로토콜이 필요할 수 있는 시기와 위치를 결정하는 데 유용할 수 있습니다.
LPS 인식이 일반적이지만, 파지는 부착 및 감염을 위해 박테리아 세포의 다양한 다른 표면 수용체를 활용할 수도 있습니다. 여기에서는 대표적인 장내세균으로 그람 음성 살모넬라균(Gram-negative Salmonella)을 사용하고, LPS를 수용체 및 게놈 방출 인자(genome ejection trigger)로 사용하는 박테리오파지 9NA(bacteriophage 9NA)를 사용하였다. 다른 장내세균 파지(예: Escherichia coli T5)는 LPS에 느슨하게 결합하며 게놈 주입을 위해 외막 단백질을 필요로 합니다. 설명된 프로토콜은 성공적인 감염을 위해 LPS의 O-항원을 인식하고 필요로 하는 박테리오파지, 예를 들어 9NA, Det7 및 P22 13,25,26,27에 적용할 수 있습니다. 따라서, 박테리아 배양의 파지 오염 제거를 위한 이 프로토콜의 성공적인 구현은 파지 감염의 원인이 숙주에서 LPS를 인식해야 하는지 여부를 결정하는 것을 포함한다.
결론적으로, 프로토콜의 잠재적 한계에도 불구하고, 당사의 대표적인 결과는 이 방법이 LPS를 수용체 및 게놈 배출 트리거로 사용하는 박테리오파지의 박테리아 살모넬라 배양을 세척하는 강력한 도구임을 명확하게 보여줍니다.
공개
저자는 밝힐 것이 없습니다.
감사의 말
유용한 토론과 제안을 해주신 Dr. Carmen R. Beuzón과 Rocío Carvajal-Holguera에게 감사드립니다. 이 작업은 MICIU/AEI/10.13039/5011100011033와 Universidad de Sevilla의 VI Plan Propio de Investigación y Transferencia가 자금을 지원하는 보조금 PID2020-116995RB-I00의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 20 mL syringe | BD Discardit II | 300296 | No special requirements |
| 50 mL conical tubes | Avantor | 525-0610 | No special requirements |
| 90 x 14 mm Petri dishes | Deltalab | 200209 | No special requirements |
| Agar | Sigma-Aldrich | A1296 | No special requirements |
| Bacteriophage lysate | Minimal concentration: 109 PFU/mL | ||
| Centrifuge | Eppendorf | No special requirements | |
| Chloroform | Panreac | 131252 | No special requirements |
| Citric acid · H2O | Merck | 1.00247 | |
| Colony counter | No special requirements | ||
| Evans Blue | Sigma-Aldrich | E-2129 | |
| Flasks | No special requirements | ||
| Fluorescein sodium salt | Sigma-Aldrich | F-6377 | |
| Forceps | No special requirements | ||
| Glass tubes | No special requirements | ||
| Glass tubes for lysate | No special requirements | ||
| Glucose | Sigma-Aldrich | G7021 | |
| K2HPO4 | Merck | 1.05104.1000 | |
| K2HPO4 anhydrous | Merck | 1.05104 | |
| Lipopolysaccharide from Salmonella enterica serotype Typhimurium | Sigma-Aldrich | L6511-25 mg | Dissolved in sterile water |
| Membrane 0.45 µm | MF-Millipore | HAWP02500 | No special requirements |
| MgSO4 · 7 H2O | Merck | 1.05886 | |
| NaCl | Sigma-Aldrich | S9888 | No special requirements |
| NaNH4HPO4 · 4 H2O | Sigma-Aldrich | S9506 | |
| Peptone | iNtRON | Ba2001 | No special requirements |
| Syringe Filter 0.22 µm | Millex | SLGSR33SB | No special requirements |
| Toothpicks | No special requirements | ||
| Tryptone | Panreac | 403682.1210 | No special requirements |
| Vacuum pump | Thermo Scientific | No special requirements | |
| Yeast extract | iNtRON | 48045 | No special requirements |
참고문헌
- Kasman, L. M., Porter, L. D. . Bacteriophages. , (2024).
- Lawrence, D., Baldridge, M. T., Handley, S. A. Phages and human health: More than idle hitchhikers. Viruses. 11 (7), 587 (2019).
- Beller, L., Matthijnssens, J. What is (not) known about the dynamics of the human gut virome in health and disease. Curr Opin Virol. 37, 52-57 (2019).
- Roux, S., Hallam, S. J., Woyke, T., Sullivan, M. B. Viral dark matter and virus-host interactions resolved from publicly available microbial genomes. eLife. 4, 08490 (2015).
- Wommack, K. E., Colwell, R. R. Virioplankton: Viruses in aquatic ecosystems. Microbiol Mol Biol Rev. 64 (1), 69-114 (2000).
- Louten, J. Virus structure and classification. Essentl Human Virol. , 19-29 (2016).
- Egido, J. E., Costa, A. R., Aparicio-Maldonado, C., Haas, P. J., Brouns, S. J. J. Mechanisms and clinical importance of bacteriophage resistance. FEMS Microbiol Rev. 46 (1), 048 (2022).
- Rakhuba, D. V., Kolomiets, E. I., Dey, E. S., Novik, G. I. Bacteriophage receptors, mechanisms of phage adsorption and penetration into host cell. Polish J Microbiol. 59 (3), 145-155 (2010).
- Lindberg, A. A. Bacteriophage receptors. Ann Rev Microbiol. 27 (1), 205-241 (1973).
- Koskella, B., Meaden, S. Understanding bacteriophage specificity in natural microbial communities. Viruses. 5 (3), 806-823 (2013).
- Marcó, M. B., Moineau, S., Quiberoni, A. Bacteriophages and dairy fermentations. Bacteriophage. 2 (3), 149-158 (2012).
- Fernández-Fernández, R., et al. Evolution of a bistable genetic system in fluctuating and non-fluctuating environments. bioRxiv. , (2024).
- Cota, I., Blanc-Potard, A. B., Casadesús, J. STM2209-STM2208 (opvAB): a phase variation locus of Salmonella enterica involved in control of O-antigen chain length. PloS one. 7 (5), e36863 (2012).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods Mol Biol. 501, 69-76 (2009).
- Ackermann, H. W. Tailed bacteriophages: The order Caudovirales. Adv Virus Res. 51, 135-201 (1998).
- Wilkinson, R. G., Gemski, P., Stocker, B. A. D. Non-smooth mutants of Salmonella typhimurium: Differentiation by phage sensitivity and genetic mapping. J Gen Microbiol. 70 (3), 527-554 (1972).
- Casjens, S. R., Leavitt, J. C., Hatfull, G. F., Hendrix, R. W. Genome sequence of Salmonella Phage 9NA. Genome Announc. 2 (4), e00531 (2014).
- Cota, I., et al. Epigenetic control of Salmonella enterica O-antigen chain length: A tradeoff between virulence and bacteriophage resistance. PLoS Genet. 11 (11), e1005667 (2015).
- Chan, K., Botstein, D., Watanabe, T., Ogata, Y. Specialized transduction of tetracycline resistance by phage P22 in Salmonella typhimurium. Virology. 50, 883-898 (1972).
- Bertozzi Silva, J., Storms, Z., Sauvageau, D. Host receptors for bacteriophage adsorption. FEMS Microbiol Lett. 363 (4), 002 (2016).
- Sørensen, M. C. H., et al. Bacteriophage F336 recognizes the capsular phosphoramidate modification of Campylobacter jejuni NCTC11168. J Bacteriol. 193 (23), 6742-6749 (2011).
- Ogata, S., Hongo, M. Bacteriophages of the Genus Clostridium. Adv Appl Microbiol. 25, 241-273 (1979).
- Olivenza, D. R., Casadesús, J., Ansaldi, M. Epigenetic biosensors for bacteriophage detection and phage receptor discrimination. Environ Microbiol. 22 (8), 3126-3142 (2020).
- Olivenza, D. R., et al. A portable epigenetic switch for bistable gene expression in bacteria. Sci Rep. 9 (1), 862 (2019).
- Walter, M., et al. Structure of the receptor-binding protein of bacteriophage Det7: a Podoviral tail spike in a Myovirus. J Virol. 82 (5), 2265-2273 (2008).
- Davies, M. R., Broadbent, S. E., Harris, S. R., Thomson, N. R., vander Woude, M. W. Horizontally acquired Glycosyltransferase operons drive Salmonellae lipopolysaccharide diversity. PLoS Genet. 9 (6), e1003568 (2013).
- Wahl, A., Battesti, A., Ansaldi, M. Prophages in Salmonella enterica: a driving force in reshaping the genome and physiology of their bacterial host. Mol Microbiol. 111 (2), 303-316 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유