Method Article
Entfernung von Bakteriophagen aus infizierten Salmonellenkulturen
In diesem Artikel
Zusammenfassung
Bakteriophagen, die auf der Erde allgegenwärtig und vielfältig sind, infizieren und vermehren sich in bakteriellen Wirten und spielen eine entscheidende Rolle in mikrobiellen Ökosystemen. Trotz ihrer Bedeutung kann ihr Vorhandensein industrielle Prozesse stören. Wir haben eine Methode entwickelt, bei der bakterielle Lipopolysaccharide verwendet werden, um Bakteriophagen aus Salmonellenkulturen zu eliminieren.
Zusammenfassung
Bakteriophagen oder einfach Phagen spielen eine wichtige Rolle in mikrobiellen Umgebungen, beeinflussen Bakterienpopulationen und prägen deren Evolution und Interaktionen. Diese Organismen sind Viren, die bakterielle Wirte infizieren und sich darin vermehren. Phagen sind auf der Erde allgegenwärtig, sehr vielfältig und sehr häufig. Während Bakteriophagen in verschiedenen Umgebungen eine wertvolle Rolle spielen und ein Schlüsselgebiet der Forschung in der Mikrobiologie und Ökologie sind, kann ihre Anwesenheit in bestimmten industriellen Prozessen oder Produkten unerwünscht sein. In Anbetracht der Häufigkeit und Allgegenwart von Bakteriophagen auf der Erde ist die Entwicklung von Verfahren zur Entfernung von Bakteriophagen aus Bakterienkulturen in verschiedenen Labor- und Industrieanwendungen von entscheidender Bedeutung, um die Integrität der Kulturen zu erhalten und genaue experimentelle Ergebnisse oder Produktqualität zu gewährleisten. Hier haben wir ein Protokoll zur Eliminierung der Bakteriophagen aus infizierten Salmonella enterica-Kulturen entwickelt, wobei wir eine Strategie verwenden, die auf der Verwendung von Lipopolysacchariden (LPS) basiert, die sich in der äußeren Membran von gramnegativen Bakterien befinden. Bakterielles LPS spielt eine wichtige Rolle bei der Wirtserkennung durch Phagen, und wir machen uns diese Eigenschaft zunutze, um ein effektives Verfahren zur Entfernung von Phagen, die LPS als Rezeptor verwenden, in Salmonellen-Bakterienkulturen zu entwickeln.
Einleitung
Mikrobielle Populationen sind in der natürlichen Umwelt mit zahlreichen Herausforderungen konfrontiert, und eine besonders große Bedrohung ist das Potenzial einer Infektion durch Bakteriophagen, die Viren, die Bakterien infizieren1. Diese Viren sind auf dem Planeten weit verbreitet und weisen eine große Vielfalt und Häufigkeit auf 2,3,4,5. Obwohl Bakteriophagen in Größe, Morphologie und genomischer Organisation unterschiedlich sind, haben sie alle die gleiche Struktur: ein DNA- oder RNA-Genom, das von einem Kapsid umgeben ist, das aus Phagen-kodierten Proteinen gebildet wird6. Bakterien haben eine Vielzahl von Abwehrmechanismen gegen sie entwickelt7. Ein Schlüsselaspekt der Bakteriophageninfektion, der sowohl für die Charakterisierung als auch für den Nachweis relevant ist, sind die Rezeptorbindungsdomänen, die auf den Schwanzfasern vorhanden sind. Bakteriophagen haben Proteine auf ihrer Oberfläche, die als rezeptorbindende Proteine oder Schwanzfasern bezeichnet werden, um spezifische Rezeptorstellen auf der Oberfläche der Bakterienzelle zu erkennen und an diese zu binden. Im Falle von gramnegativen Bakterien ist die Erkennung von Oberflächenstrukturen, wie Lipopolysacchariden (LPS), äußeren Membranproteinen, Pili und/oder Flagellen, an der Phagen-Bakterien-Interaktion beteiligt8. Diese Interaktion zwischen Bakteriophagen und Bakterien ist hochspezifisch und hängt hauptsächlich von ihrer Fähigkeit ab, sich an Wirtsoberflächen zu heften. Das O-Antigen des Lipopolysaccharids ist ein häufig verwendeter Rezeptor9.
Die Untersuchung von Bakteriophagen-Bakterien-Wechselwirkungen ist nicht nur aus biologischer Sicht faszinierend, sondern hat auch praktische Anwendungen in Bereichen wie der Phagentherapie und Biotechnologie. Während Bakteriophagen in verschiedenen Kontexten eine wertvolle Rolle spielen, z. B. bei der Veränderung mikrobieller Populationen10, kann ihre Anwesenheit in bestimmten industriellen Prozessen unerwünscht sein. In der Pharmazie, Biotechnologie und Lebensmittelproduktion kann das Vorhandensein von Bakteriophagen die Qualität und Sicherheit der Endprodukte beeinträchtigen, so dass ihre Entfernung für die Einhaltung von Qualitätsstandards unerlässlich ist. In der Bioprozesstechnik und Bioproduktion, wo Bakterienkulturen zur Herstellung verschiedener Verbindungen (z. B. Proteine, Enzyme oder Antibiotika) verwendet werden, kann das Vorhandensein von Bakteriophagen zu einer Störung der Produktionsprozesse führen, da sie in der Lage sind, die Bakterienpopulation in jeder gemeinsamen Umgebung auszugleichen. Phagen können das Berufsleben des industriellen Mikrobiologen gelegentlich in einen Albtraum verwandeln11. Die Entwicklung wirksamer Verfahren zur Entfernung von Phagen ist entscheidend, um eine konsistente und zuverlässige Produktion zu gewährleisten und die Prozesseffizienz zu steigern. Abgesehen von diesen industriellen Aspekten ist die Eliminierung von Bakteriophagen in einem Forschungslabor, in dem Präzision und Reproduzierbarkeit von entscheidender Bedeutung sind, unerlässlich, um genaue und zuverlässige Ergebnisse zu erhalten. Darüber hinaus kann die Entfernung von Phagen auch verwendet werden, um unterschiedliche Umgebungen zu simulieren und verschiedene Hypothesen zu testen12. Die Entfernung von Phagen kann auch im Forschungsumfeld sehr nützlich sein, da viele Phagen-basierte Studien, wie z. B. die Zählung von Bakterien nach Phagenanwendung, von einem Schritt zur Entfernung von Phagen profitieren würden, um viel zuverlässigere lebensfähige Zählungen zu erzielen.
Die Phagenentfernung auf der Grundlage der Kolonieisolierung würde mehrere Tage dauern, um sicherzustellen, dass die Bienenvölker phagenfrei sind, während das hier beschriebene Verfahren die Erzeugung von phagenfreien Kulturen in Stunden ermöglicht. Dieses Protokoll ermöglicht es uns, die Entwicklung von Bakterienkulturen zu verfolgen, ohne sie daran zu hindern, Kolonien zu isolieren. In diesem Sinne ist es möglich, fluktuierende Umgebungen (Vorhandensein und/oder Fehlen von Phagen) zu simulieren, um verschiedene Hypothesen zu testen. Darüber hinaus ermöglicht dieses Protokoll die qualitative und quantitative Analyse des Vorhandenseins von Phagen in einer Bakterienkultur.
Zusammenfassend lässt sich sagen, dass die Entwicklung kostengünstiger Verfahren zur Entfernung von Bakteriophagen entscheidend für die Aufrechterhaltung der Produktqualität, Sicherheit und Prozesseffizienz in verschiedenen Branchen sowie für Fortschritte in der Grundlagen- und angewandten Forschung ist. Hier beschreiben wir ein hochwirksames Protokoll, das auf der Verwendung von LPS zur Entfernung von Bakteriophagen, die LPS als Rezeptor verwenden, aus infizierten Salmonellenkulturen basiert, das sowohl zeiteffizient ist als auch minimalen Aufwand erfordert.
Protokoll
HINWEIS: Vor der Durchführung des Verfahrens zur Eliminierung von Phagen beschreiben wir die Vorbereitung einer Salmonellenkultur , die mit 9NA-Bakteriophagen infiziert ist. In Abbildung 1 ist eine allgemeine Darstellung des gesamten Verfahrens zur Entfernung von Bakteriophagen in Bakterienkulturen dargestellt.
1. Vorbereitung von Salmonellenkulturen , die mit Bakteriophagen infiziert sind
- Vorbereitung der Bakterienkultur
HINWEIS: In dieser Studie wurde Salmonella enterica subsp. enterica serovar Typhimurium Stamm ATCC 14028 opvAB::lacZ (SV8011)13 verwendet.- Tauen Sie das bei -80 °C entnommene Fläschchen, das den gewünschten S. enterica-Stamm enthält, langsam auf, indem Sie es auf Eis legen.
- Streichen Sie den S. enterica-Stamm aus dem aufgetauten Dimethylsulfoxid-Stamm auf eine Lennox-Bouillonplatte (LB) (10 g/l Trypton, 5 g/l NaCl, 5 g/l Hefeextrakt und 15 g/l Agar, siehe Materialtabelle). Inkubieren Sie die Platte bei 37 °C für 24 h.
- Übertragen Sie mit einer Impfnadel eine Kolonie aus der S. enterica-Platte und impfen Sie sie in 5 ml Flüssigkeit LB in einem Glasröhrchen. 24 h bei 37 °C inkubieren und durch Schütteln bei 200 U/min belüften.
- Zubereitung von Bakteriophagen
HINWEIS: In diesem Protokoll wurde der Bakteriophagen 9NA verwendet, der zur Familie der Siphoviridae (Ordnung Caudovirales)14 gehört. Insbesondere können andere Salmonella enterica Bakteriophagen verwendet werden, die das O-Antigen des Lipopolysaccharids als Rezeptor verwenden.- Bereiten Sie die Bakteriophagenbrühe vor, indem Sie 0,1 mL konzentriertes Bakteriophagenlysat zu einer Mischung aus 100 mL Nährbrühe (NB; 5 g/L Pepton und 3 g/L Hefeextrakt), 2 mL 50x E-Salzen (38,78 mM MgSO47 H2O, 520,5 mM ZitronensäureH2O, 2,87 M K2HPO4 wasserfrei und 829 mM NaNH4HPO44 H2O) und 0,4 mL 50% hinzufügen Traubenzucker. Durch Inversion gründlich mischen.
- Ein repräsentatives Beispiel für die Konzentration von Bakteriophagen könnte 109 PFU/ml (Plaque Forming Units/ml) sein, aber niedrigere Konzentrationen können mit ähnlichen Ergebnissen verwendet werden (siehe Materialtabelle). Schätzen Sie die Bakteriophagenkonzentration nach Schritt 1.2.3.
- Bereiten Sie das Lysat vor, indem Sie die unten beschriebenen Schritte ausführen.
- Bereiten Sie das S. enterica-Inokulum in 5 ml LB (10 g/l Trypton, 5 g/l NaCl und 5 g/l Hefeextrakt) vor. Bei 37 °C und 200 U/min 24 h inkubieren.
- Mischen Sie 4 ml Bakteriophagenbrühe mit 1 ml der Nachtkultur von S. enterica. Die Mischung bei 37 °C und 200 U/min 8 h lang inkubieren.
- Um zwischen Bakterien und Bakteriophagen zu unterscheiden, zentrifugieren Sie die Mischung bei 2.377 x g für 20 Minuten. Den Überstand wird in ein Glasröhrchen gegeben, 800 μl Chloroform (siehe Materialtabelle) zugegeben und verwirbelt. Achten Sie darauf, die obere Phase nur zu übertragen, wenn ein Gemisch mit Chloroform verwendet wird.
HINWEIS: Es ist wichtig, die Übertragung von Chloroformresten zu verhindern, die die Effizienz des Verfahrens beeinträchtigen könnten. Der Chloroform-Schritt ist sowohl in Isolierungs- als auch in Vermehrungsprotokollen üblich, um die freigesetzten Phagen zu maximieren. Chloroform kann jedoch einige Phagen inaktivieren oder ihre Infektiosität zerstören15. Daher ist dieser Schritt nur auf Phagen anwendbar, die gegen Chloroform resistent sind. - Halten Sie die Lysate 2 h bei Raumtemperatur und lagern Sie sie dann bei 4 °C.
- Titrieren Sie das Lysat, um die Anzahl der Bakteriophagen/Anzahl der im vorbereiteten Lysat vorhandenen PFU pro ml zu quantifizieren. Führen Sie dazu die unten beschriebene Überlagerungstechnik aus.
- Bereiten Sie 5 ml LB des Inokulums für S. enterica vor. 24 h bei 37 °C inkubieren und durch Schütteln bei 200 U/min belüften.
- Führen Sie serielle Verdünnungen des Lysats in LB durch, um eine zählbare Anzahl von PFU/ml zu erreichen. Um die notwendigen Verdünnungsfaktoren zu bestimmen, identifizieren Sie das anfänglich konzentrierte Bakteriophagenlysat. Für ein repräsentatives Beispiel für die Bakteriophagenkonzentration bei etwa 109 PFU/ml liegen die Verdünnungsfaktoren, die zu einer zählbaren Anzahl von Plaques führen, zwischen 106 und 1010.
- 100 μl der entsprechenden Lysatverdünnung und 60 μl der am Vortag hergestellten Übernachtkultur von S. enterica werden zu 5 ml LB-Weichagar gegeben, der durch Mischen von flüssigem LB und LB-Agar im Verhältnis 1:1 und bei 56 °C hergestellt wurde, um eine Verfestigung zu vermeiden. Vorsichtig mischen, Blasen vermeiden.
- Gießen Sie jede Mischung in die Oberseite eines LB-Tellers und lassen Sie sie auf der Bank, bis das Medium fest wird.
- Die Platten 24 h bei 37 °C inkubieren. Zähle die Anzahl der Tafeln auf jeder Platte. Um die Anzahl der PFU/ml zu bestimmen, multiplizieren Sie die Anzahl der Plaques jeder Platte mit dem Verdünnungsfaktor (FD). Um eine effiziente Phageninfektion zu gewährleisten, sind 10 9-1012 PFU/ml erforderlich.
PFU/mL= Nein. Plaques in der Platte x FD
- Bereiten Sie die Bakteriophagenbrühe vor, indem Sie 0,1 mL konzentriertes Bakteriophagenlysat zu einer Mischung aus 100 mL Nährbrühe (NB; 5 g/L Pepton und 3 g/L Hefeextrakt), 2 mL 50x E-Salzen (38,78 mM MgSO47 H2O, 520,5 mM ZitronensäureH2O, 2,87 M K2HPO4 wasserfrei und 829 mM NaNH4HPO44 H2O) und 0,4 mL 50% hinzufügen Traubenzucker. Durch Inversion gründlich mischen.
- Infektion von Salmonellenkulturen mit Bakteriophagen
- Eine Übernachtkultur von S. enterica auf 1:100 in einem Endvolumen von 5 ml LB verdünnen und 0,1 ml Lysat (konzentriert bei 10 9-1012 PFU/ml) hinzufügen. Bei 37 °C und 200 U/min 24 h inkubieren.
- Bestimmung des Phagentiters als PFU im Plattenassay
- Um die Anzahl der Bakteriophagen in der infizierten Kultur zu quantifizieren, führen Sie Plattenassays durch, um den Phagentiter als PFU/ml zu bestimmen, wie unten beschrieben.
- Übertragen Sie 1 ml der Kultur in ein Röhrchen. Zentrifugieren Sie 2 Minuten lang bei 17.115 x g und gießen Sie den Überstand in ein neues, beschriftetes Fläschchen.
- Geben Sie 100 μl Chloroform in das Fläschchen und wirbeln Sie es ein, um bakterielle Ablagerungen aus der Lösung zu entfernen. Folgen Sie Schritt 1.2.3.
- Um die Anzahl der Bakteriophagen in der infizierten Kultur zu quantifizieren, führen Sie Plattenassays durch, um den Phagentiter als PFU/ml zu bestimmen, wie unten beschrieben.
2. Entfernung von Bakteriophagen aus infizierten Salmonella enterica-Kulturen (siehe Abbildung 2)
HINWEIS: Um den Prozess der Bakteriophagenentfernung zu überwachen, werden während des gesamten Reinigungsprotokolls verschiedene Aliquots für die Titration entnommen. Insgesamt gibt es acht Aliquots, die bestätigen, dass die Anzahl der PFU/ml während des gesamten Reinigungsprozesses bis zur vollständigen Eliminierung abnimmt.
- Entfernung von Bakteriophagen in Suspension
- Die Bakterienkultur, die Phagen enthält, wird in einem konischen 50-ml-Zentrifugenröhrchen mit 0,22 μm-filtriertem LB auf 1:100 mm verdünnt. Titrationspunkt 1: Dies ist das erste Aliquot für die Titration (die beginnende infizierte Salmonellenkultur ).
- Die Suspension wird 10 min lang bei 2.377 x g zentrifugiert. Entfernen Sie vorsichtig den Überstand. Verwenden Sie keine Pipette; Gießen Sie das Rohr ein. Versuchen Sie, etwas Volumen am Boden des konischen Zentrifugenröhrchens zu lassen, um das Vorhandensein von Zellen sicherzustellen.
- Bakterienzellen werden mit 10 mL 0,22 μm gefiltertem LB-Medium gewaschen. Wiederholen Sie die Zentrifugations- und Waschschritte 3x. Resuspendieren Sie das Pellet in 5 mL 0,22 μm-filtriertem LB. Titrationspunkt 2: Dies ist das zweite Aliquot für die Titration.
- 50 μl der oben genannten Bakteriensuspension werden unter Verwendung eines Vakuumfiltrationssystems durch einen sterilen 0,45-μm-Filter (siehe Materialtabelle) geleitet. Spülen Sie den Filter mit 100 mL 0,22 μm-filtriertem LB.
HINWEIS: Die Kopfgröße des Bakteriophagen 9NA beträgt 0,067 μm. Im Gegensatz dazu sind Salmonellen 2-5 μm lang und 0,5-1,5 μm breit. Wenn also 0,45 μm Membranen verwendet werden, passieren die meisten Bakterien nicht, da sie größer als 0,45 μm sind, während die meisten Phagen die 0,45 μm Filterporen passieren.
- Entfernung von Bakteriophagen, die in Salmonellenzellen enthalten sind
- Um die Phagenfreisetzung aus Bakterienzellen zu erleichtern, inkubieren Sie den 0,45-μm-Filter mit den Zellen 2 Stunden lang bei 37 °C im Inkubator, ohne das System zu zerlegen.
HINWEIS: Dieser Inkubationsschritt ist erforderlich, um die Lyse der infizierten Bakterien und die Freisetzung intrazellulärer Phagen in den Filter zu fördern. - Waschen Sie den 0,45-μm-Filter mit einem Vakuumfiltrationssystem und spülen Sie den Filter mit 100 mL 0,22 μm-gefiltertem LB.
- Entnahme von Bakterienzellen aus dem Filter in 2 mL 2x LB (20 g/L Trypton, 10 g/L NaCl, 10 g/l Hefeextrakt; siehe Materialtabelle). Dazu wird der 0,45 μm Filter mit einer sterilen Pinzette in einen Kolben überführt. Geben Sie dann 2 mL 2x LB über den Filter und pipettieren Sie mehrmals, um Zellen aus dem Filter zu lösen.
- Zentrifugieren Sie 1 mL dieser Suspension für 2 min bei 10.354 x g. Entsorgen Sie den Überstand und waschen Sie die Bakterienzellen 3x mit 1 mL gefiltertem LB-Medium. Resuspendieren Sie die Zellen in 1 mL von 2x LB. Titrationspunkt 3: Dies ist das dritte Aliquot für die Titration.
HINWEIS: Die Verwendung eines konzentrierteren LB (2x LB) wird dringend empfohlen, da, wie unten beschrieben, kommerzielles Lipopolysaccharid (LPS) zugesetzt wird, das sich in Wasser auflöst und so das Medium verdünnt.
- Um die Phagenfreisetzung aus Bakterienzellen zu erleichtern, inkubieren Sie den 0,45-μm-Filter mit den Zellen 2 Stunden lang bei 37 °C im Inkubator, ohne das System zu zerlegen.
- Vermeiden Sie eine erneute Infektion, indem Sie die aus Bakterienzellen freigesetzten Bakteriophagen mit kommerziellem Lipopolysaccharid täuschen, wie unten beschrieben.
- 20 μl der bakteriellen Suspension in Gegenwart eines handelsüblichen S. enterica-Lipopolysaccharids (LPS; siehe Materialtabelle) auf eine Endkonzentration von 3,75 mg/ml in 1 ml 0,22 μm-filtriertem LB für 2 h bei 37 °C und Schütteln bei 200 U/min inkubieren.
HINWEIS: Phagen, die aus Zellen in das Medium freigesetzt werden, können an das kommerzielle S. enterica LPS anstelle von bakteriellem LPS binden. - Nach 2 Stunden Inkubation wird 1 ml Kultur in ein Mikrozentrifugenröhrchen überführt und die Suspension 2 Minuten lang bei 10.354 x g zentrifugiert . Titrationspunkt 4: Verwenden Sie den Überstand als viertes Aliquot für die Titration.
- Waschen Sie das Pellet 3x mit 1 mL gefiltertem LB-Medium.
- Nach dem Waschen resuspendieren Sie das Pellet in 1 mL von 2x LB. Inkubieren Sie 20 μl dieser Mischung in 1 mL von 0,22 μm gefiltertem LB mit 0,8 mg/mL handelsüblichem LPS für 2 h bei 37 °C und Schütteln bei 200 U/min. Titrationspunkt 5: Verwenden Sie den Rest der Bakteriensuspension für die fünfte Titration.
- Nach 2 Stunden Inkubation wird 1 ml Kultur in ein Mikrozentrifugenröhrchen überführt und durch Zentrifugation für 2 Minuten bei 10.354 x g pelletiert. Titrationspunkt 6: Verwenden Sie den Überstand als sechstes Aliquot für die Titration, indem Sie den Überstand in ein neues Fläschchen gießen.
- Bakterienzellen 3x mit gefiltertem LB waschen. Nach dem Waschen werden die gesammelten Zellen in 1 ml 0,22 μm-filtriertem LB resuspendiert.
- 1 ml Bakteriensuspension wird mit einem Vakuumfiltrationssystem durch einen sterilen 0,45-μm-Filter geleitet. Spülen Sie den Filter mit 100 mL 0,22 μm-filtriertem LB.
- Gewinnen Sie Zellen aus dem 0,45 μm-Filter in 2 mL 2x LB-Medium. Zu diesem Zweck wird der 0,45-μm-Filter mit einer sterilen Pinzette in einen Kolben überführt. Geben Sie dann 2 mL 2x LB über den Filter und pipettieren Sie mehrmals, um zu versuchen, Zellen aus dem Filter zu lösen.
- 1 ml Zellen durch Zentrifugation für 2 min zu 10.354 x g pelletieren. 3x mit 0,22 μm-gefiltertem LB waschen. Nach dem Waschen resuspendieren Sie die gesammelten Zellen in 1 ml von 2x LB. Titrationspunkt 7: Dies ist das siebte Aliquot für die Titration. Dieses Aliquot gibt Aufschluss über die Wirksamkeit der Entfernung von Phagen in Bakterienkulturen.
- 20 μl der bakteriellen Suspension in Gegenwart eines handelsüblichen S. enterica-Lipopolysaccharids (LPS; siehe Materialtabelle) auf eine Endkonzentration von 3,75 mg/ml in 1 ml 0,22 μm-filtriertem LB für 2 h bei 37 °C und Schütteln bei 200 U/min inkubieren.
3. Vorbereitung einer bakteriophagenfreien Salmonellenkultur nach Entfernung von Bakteriophagen
- Bereiten Sie das phagenfreie Inokulum vor. Fügen Sie 100 μl Zellen aus der Bakteriensuspension zu 900 μl LB mit 0,45 mg/ml LPS hinzu. Inkubieren Sie bei 37 °C für 24 h und schütteln Sie bei 200 U/min.
- Titrationspunkt 8: Bereiten Sie das achte Aliquot für die Titration vor (die letzte potenziell nicht infizierte Kultur). Dieser Punkt soll bestätigen, dass keine Phagen-Reinfektion vorliegt.
Ergebnisse
Salmonella enterica und andere gramnegative Bakterien haben eine äußere Membran, die LPS enthält. Das O-Antigen von LPS ist ein häufig verwendeter Rezeptor von Bakteriophagen 9NA zur Infektion von Salmonellenkulturen 16,17.
Angesichts der spezifischen Affinität von Bakteriophagen zum O-Antigen oder den Kernpolysaccharidregionen von LPS wollten wir untersuchen, ob Salmonella enterica kommerzielles LPS als Köder zum Ausschluss von 9NA-Bakteriophagen verwendet werden kann. Zu diesem Zweck mischten wir bekannte Konzentrationen des kommerziellen LPS und des 9NA-Lysats, gefolgt von einer Titration. Kommerzielle LPS- und 9NA-Bakteriophagen wurden in einem Gesamtvolumen von 200 μl gemischt und 2 h bei 37 °C ohne Schütteln inkubiert. Für die Titration wurden 100 μl des LPS-9NA-Gemisches und 60 μl einer Übernachtkultur zu 5 ml LB-Weichagar gegeben und auf die Oberseite einer LB-Platte gegossen. Die Platten wurden 24 h bei 37 °C inkubiert. Wie in Abbildung 3 dargestellt, nimmt der Lysattiter proportional ab, wenn die Konzentration von S. enterica commercial LPS zunimmt. Diese Ergebnisse deuten darauf hin, dass das kommerzielle LPS korrekt als Köder für 9NA-Bakteriophagen fungiert.
Interessanterweise wird die Zeit, die Phagen 9NA benötigt, um Salmonellenzellen zu lysieren und Phagen freizusetzen, durch die Anzahl der Bakterien und Phagen erleichtert (Abbildung 4). Um zu untersuchen, wie sich dieser Aspekt auf das Phagenentfernungsprotokoll auswirken könnte, haben wir verschiedene Bakterien-Phagen-Verhältnisse getestet (1:1, 1:10, 1:100 und 1:1000). Wie in Abbildung 4 zu sehen ist, kommt es 1,5-2,5 h nach der Zugabe von 9NA-Phagen zu einem Abfall vonOD 600 nm der Kultur. OD600 nm-Werte nahe Null sind ein Hinweis darauf, dass die Bakterienkultur lysiert wird18. Aus diesem Grund wurden die Inkubationszeiten in diesem Protokoll auf 2 h festgelegt, um sicherzustellen, dass die in Salmonellenzellen enthaltenen Phagen genügend Zeit haben, Bakterien zu lysieren und freizusetzen. Diese Zeit sollte für jedes Wirt-Phagen-System geschätzt werden, bevor dieses Protokoll durchgeführt wird.
Nachdem wir festgestellt hatten, dass kommerzielles LPS als Köder fungiert und die Lysezeit für 9NA-infizierte Bakterien verkürzt war, führten wir das beschriebene Protokoll zur Reinigung infizierter Salmonellenkulturen der Bakteriophagen durch (Abbildung 1 und Abbildung 2). Um das Vorhandensein von Bakteriophagen in jedem Schritt des Protokolls zu überwachen, führten wir Plaque-Assays durch, um die Infektiosität der resultierenden Kulturen in 100 μl der resultierenden Mischung an verschiedenen Stellen zu berechnen (Abbildung 5). Es ist zu beobachten, dass wiederholtes Waschen und Filtern nicht ausreichen, um Bakteriophagen aus Kulturen zu eliminieren (Titrationspunkte 1-3); die Anzahl der Phagen nimmt jedoch ab, sobald wir einen Inkubationsschritt mit kommerziellem LPS (Titrationspunkt 4) anwenden. Der entscheidende Schritt für die vollständige Entfernung von Bakteriophagen in einer Bakterienkultur ist die zweite Inkubation mit kommerziellem LPS (Titration Point 6). Dieser Schritt ist essentiell für die erfolgreiche Entfernung von 9NA-Bakteriophagen in Salmonellenkulturen .
Ein interessanter Aspekt dieses Protokolls wäre es, den Grad der Phagenresistenz nach der Phagenentfernung zu kennen. Ein Verfahren, das Phagen von phagenresistenten Bakterien trennt, ist nutzlos. Aus diesem Grund ist es wichtig aufzuzeigen, dass die in der Kultur verbleibenden Bakterien anfällig für Phagen sind. Um zu zeigen, dass empfindliche Phagenzellen nach dem Eingriff in den Kulturen verbleiben, verwendeten wir den Evans Blue Uranin (EBU) Plattenassay, um auf Phagenkontaminationzu untersuchen 19. Die EBU-Platten bestanden aus LB-Medium, das mit 10 ml/L,K2, HPO4 , 25%, 5 mL/L, Glukose 50%, 2,5 mL/L, Fluorescein 1%, 1,25 mL/L, Evans Blue 1% und 15 g/L Agar ergänzt wurde. Cross-Striping auf EBU-Platten mit 9NA-Phagen wurde verwendet, um Phagen-resistente und Phagen-empfindliche Isolate zu unterscheiden (Abbildung 6). Die am Ende des Reinigungsprotokolls gewonnenen Bakterienkulturen wurden verwendet, um isolierte Kolonien zu erhalten, die auf Phagenkontamination untersucht wurden (Abbildung 6B). Wir können die Existenz sowohl resistenter als auch empfindlicher Zellen beobachten. Dieses Protokoll begünstigt nicht die Selektion von phagenresistenten Zellen; Es eliminiert nur Bakteriophagen.
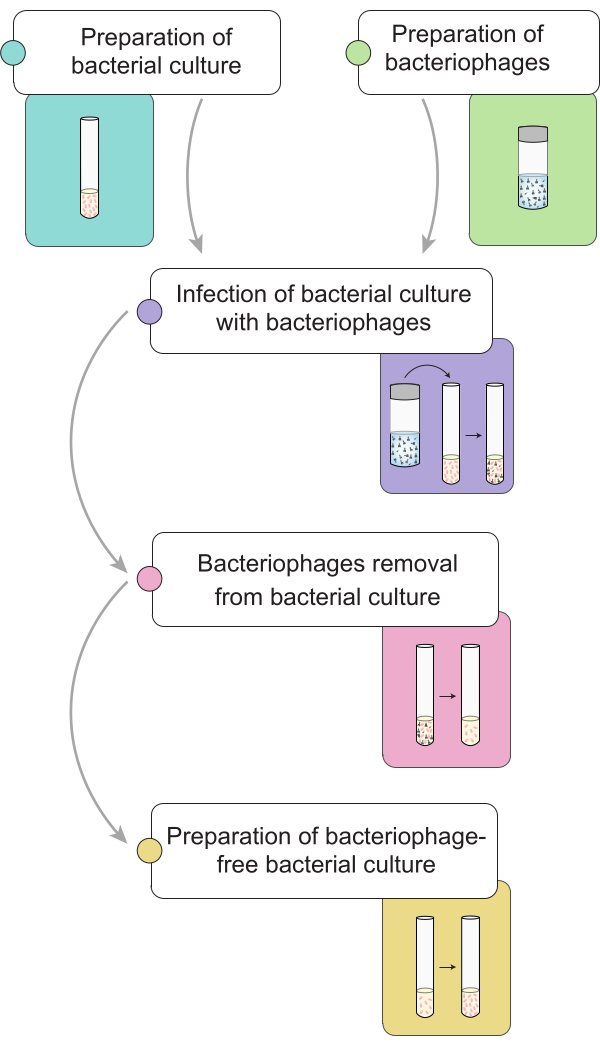
Abbildung 1: Kurzer Abriss des Verfahrens zur Eliminierung von Bakteriophagen in Salmonellenkulturen . Der Arbeitsablauf gliedert sich in verschiedene Phasen: Aufbereitung der Bakterienkultur und des Lysats, Infektion der Bakterienkultur mit Bakteriophagen, Entnahme von Bakteriophagen aus infizierten Bakterienkulturen und Aufbereitung eines phagenfreien Bakterieninokulums. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
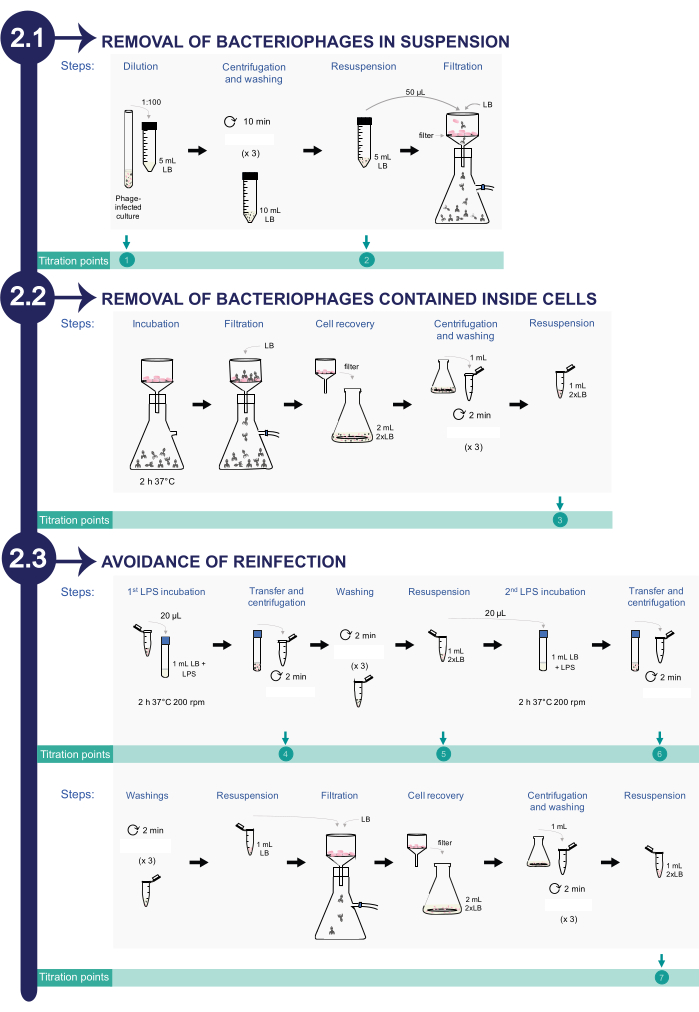
Abbildung 2: Verfahren zur Entfernung von Bakteriophagen aus infizierten Salmonellenkulturen. Der Prozess besteht aus drei Phasen: 1) Entfernung von Bakteriophagen in Suspension, 2) Entfernung von Phagen, die in Bakterienzellen enthalten sind, und 3) Vermeidung einer erneuten Infektion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
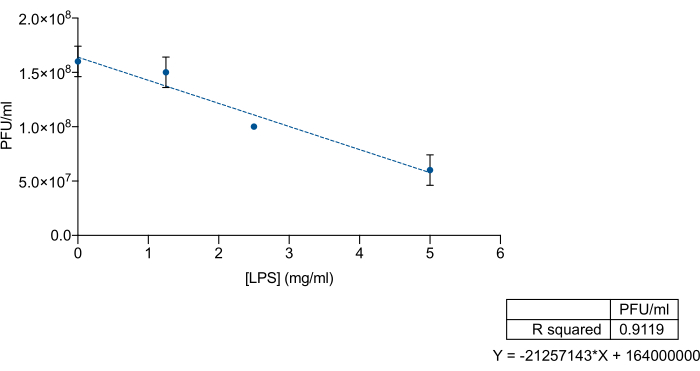
Abbildung 3: LPS-Köder-Assay zur Messung der Wirksamkeit von Salmonella enterica kommerziellem LPS zur Bindung von Bakteriophagen . Titration eines 9NA-Lysats (PFU/ml) bei steigenden Konzentrationen von Salmonella enterica commercial LPS. Das Experiment wurde in dreifacher Ausfertigung durchgeführt. Mittelwert und Standardabweichung werden dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
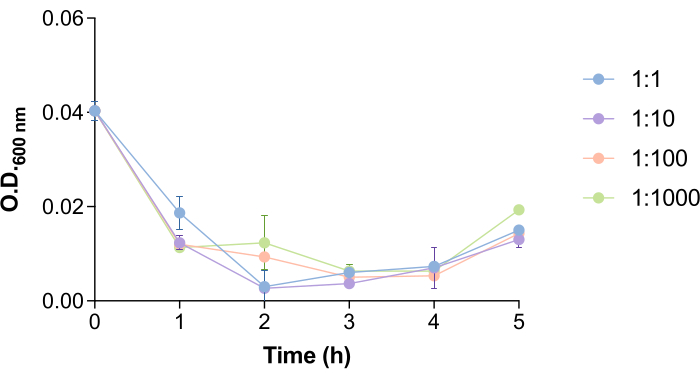
Abbildung 4: Lysezeit von Bakteriophagen 9NA in Salmonellenkulturen . Wachstumskurven der Salmonella enterica-Kulturen in Gegenwart von Bakteriophagen 9NA bei Bakterien:Phagen-Verhältnissen von 1:1, 1:10, 1:100 und 1:1000. Das Experiment wurde in dreifacher Ausfertigung durchgeführt. Mittelwert und Standardabweichung werden dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
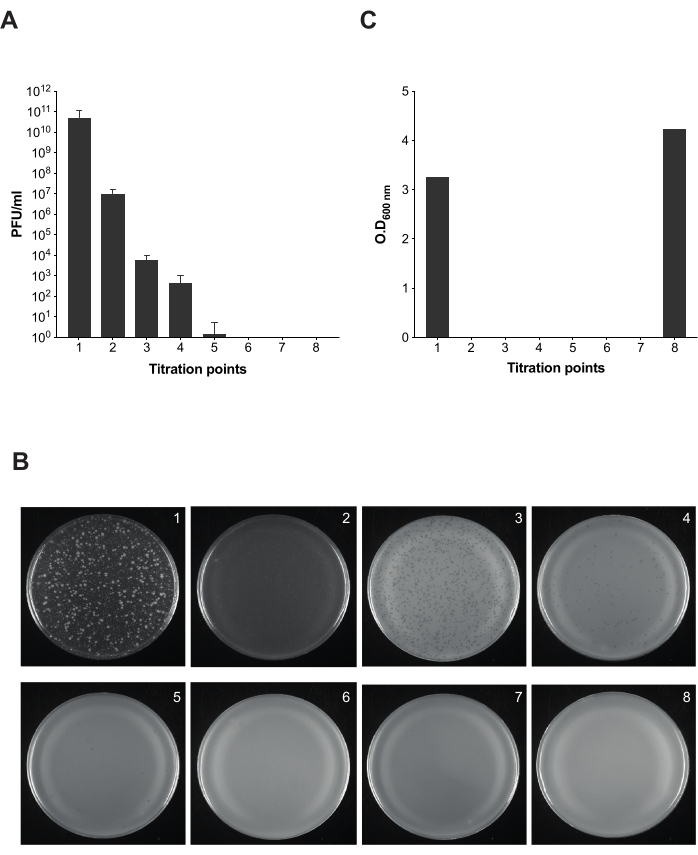
Abbildung 5: Plaque-Assay für die Infektiositätsprüfung bei der Entfernung von Bakteriophagen in Salmonellenkulturen . (A) Die Titration von acht Aliquoten wurde zu verschiedenen Zeitpunkten des Protokolls durchgeführt (die Titrationspunkte 1-7 sind in Abbildung 2 markiert). Für dieses Experiment wurden Salmonella enterica Serovar Typhimurium Stamm ATCC 14028 opvAB::lacZ (SV8011) und Bakteriophagen 9NA verwendet. Die Experimente wurden in dreifacher Ausfertigung durchgeführt, wobei der Mittelwert und die Standardabweichung dargestellt sind. (B) Weiche Agarplatten mit Salmonella enterica wurden nach der Overlay-Technik unter Verwendung von Aliquoten von acht Titrationspunkten erhalten. Die Platten entsprechen von links nach rechts den Titrationspunkten 1-8. (C) Optische Dichte bei 600 nm Bakterienkultur zu verschiedenen Zeitpunkten des Phagenentfernungsprotokolls. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
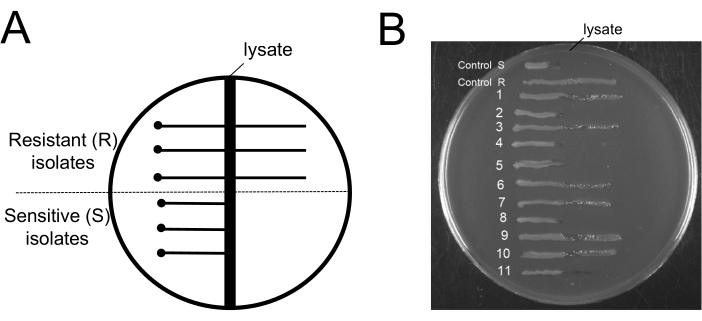
Abbildung 6: Test auf Phagen-resistente Bakterien nach der Entfernung von Bakteriophagen. (A) Schematische Darstellung einer typischen EBU-Agarplatte, die für den Cross-Streak-Agar-Assay verwendet wird: Der vertikale dunkle Bereich in der Mitte stellt die Zone des 9NA-Lysats dar. Der Punkt stellt den Ort dar, an dem die getesteten Zellen in sicherem Abstand von der Lysatzone inokuliert werden, und die horizontalen durchgezogenen Linien stellen entweder die Phagen-resistenten Zellen dar, die über die Lysatzone wachsen, oder die Phagen-empfindlichen Zellen, die nicht über die Lysatzone hinaus wachsen. (B) EBU-Plattenproben zur Untersuchung von 11 Völkern wurden am Ende des Entnahmeprotokolls durchgeführt. Control R und S sind Beispiele für phagenresistente bzw. phagenempfindliche Isolate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Bakteriophagen wenden verschiedene Strategien an, um bakterielle Wirte zu erkennen und zu infizieren. Als Phagenrezeptoren können verschiedene molekulare Strukturen auf der Oberfläche von Bakterien fungieren: Protein, Polysaccharide, Lipopolysaccharide (LPS) und Kohlenhydrate20. In gramnegativen Bakterien ist LPS ein häufiger Rezeptor für Phagen. Darüber hinaus sind weitere Rezeptoren die Proteine der äußeren Membran, Pili und Flagellen21.
Die spezifische Interaktion zwischen Bakteriophagen und Bakterien, die auf der Erkennung von LPS8 basiert, wurde in dieser Arbeit für die Entwicklung eines hocheffizienten Protokolls zur Eliminierung von Bakteriophagen in infizierten Bakterienkulturen genutzt (Abbildung 1 und Abbildung 2). Unser Protokoll begünstigt nicht die Selektion von Phagen-resistenten Zellen; es eliminiert nur Bakteriophagen (Abbildung 6). Sowohl Phagen-empfindliche als auch Phagen-resistente Zellen verbleiben nach Durchführung dieses Protokolls der Phagenentfernung in Bakterienkultur.
Die traditionelle Standardpraxis bei einer Phageninfektion besteht darin, zu versuchen, das gesamte kontaminierte Material zu beseitigen, gefolgt von Reinigung und Sterilisation22. Bei der Dekontamination wird die Bakterienkultur Stressbedingungen wie hohen Temperaturen ausgesetzt, um die Bakterienzellen teilweise oder vollständig zu eliminieren. Wie in repräsentativen Ergebnissen beschrieben, ist der entscheidende Schritt in diesem Protokoll die Inkubation von Phagen-infizierten Bakterienkulturen mit kommerziellem LPS, einer nicht schädlichen Substanz für Bakterienkulturen. Dies trägt dazu bei, die Lebensfähigkeit von Bakterienkulturen zu erhalten und bietet erhebliche Vorteile für industrielle Anwendungen in Fermentern und Bioreaktoren.
Die Inkubationszeit in diesem Protokoll beträgt 2 Stunden, um genügend Zeit für die Phagenlyse von Bakterienzellen zu gewährleisten. Sollen unterschiedliche Bakterienstämme und Bakteriophagen verwendet werden, sollte dieser Parameter berücksichtigt und vom Anwender definiert werden. In diesem Fall sollte vor dem Versuch ein ähnlicher Assay wie in Abbildung 4 beschrieben durchgeführt werden.
Interessanterweise konnte die Wirksamkeit dieses Reinigungsprotokolls auch durch den Einsatz eines Assays analysiert werden, der den Phagengehalt einer bestimmten Probe überwacht. In diesem Sinne sind epigenetische Biosensoren ein neuartiges Werkzeug zum Nachweis von Bakteriophagen23. Ein bekannter Phagen-Biosensor, der in der Lage ist, Coliphagen zu detektieren, die LPS als Rezeptor verwenden, ist das opvAB::gfp-System 13,18,23,24. Dieser Phagen-Biosensor detektiert eine Zunahme der OpvABON-Subpopulation in Gegenwart von Phagen, die das O-Antigen als Rezeptor verwenden. In diesem Sinne könnten wir eine opvAB::gfp-Fusion verwenden, um LPS-bindende Phagen in verschiedenen Schritten dieses Protokolls und/oder in verschiedenen Medien und Bedingungen zu überwachen. Diese Ansätze könnten wertvoll sein, um den Zeitpunkt und die Orte zu bestimmen, an denen ein wirksames Protokoll erforderlich sein könnte.
Während die LPS-Erkennung üblich ist, können Phagen auch eine Vielzahl anderer Oberflächenrezeptoren auf Bakterienzellen zur Anheftung und Infektion nutzen. Hier haben wir die gramnegativen Salmonellen als repräsentative Enterobakterien und den Bakteriophagen 9NA verwendet, der LPS als Rezeptor und Genomausstoßauslöser verwendet. Andere Enterobakterien-Phagen (z. B. Escherichia coli T5) binden lose an LPS und benötigen ein äußeres Membranprotein für die Genominjektion. Das beschriebene Protokoll ist anwendbar für Bakteriophagen, die das O-Antigen von LPS erkennen und für eine erfolgreiche Infektion benötigen, wie z. B. 9NA, Det7 und P22 13,25,26,27. Dementsprechend beinhaltet die erfolgreiche Implementierung dieses Protokolls für die Phagendekontamination von Bakterienkulturen die Bestimmung, ob die Quelle der Phageninfektion die Erkennung von LPS im Wirt erfordert.
Zusammenfassend lässt sich sagen, dass unsere repräsentativen Ergebnisse trotz der potenziellen Einschränkungen des Protokolls eindeutig zeigen, dass diese Methode ein leistungsfähiges Werkzeug zur Reinigung von bakteriellen Salmonellenkulturen von Bakteriophagen ist, die LPS als Rezeptor- und Genomausstoßauslöser verwenden.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Wir danken Dr. Carmen R. Beuzón und Rocío Carvajal-Holguera für hilfreiche Gespräche und Anregungen. Diese Arbeit wurde durch das Stipendium PID2020-116995RB-I00 unterstützt, das von MICIU/AEI/ 10.13039/5011100011033 und dem VI Plan Propio de Investigación y Transferencia der Universidad de Sevilla finanziert wurde.
Materialien
| Name | Company | Catalog Number | Comments |
| 20 mL syringe | BD Discardit II | 300296 | No special requirements |
| 50 mL conical tubes | Avantor | 525-0610 | No special requirements |
| 90 x 14 mm Petri dishes | Deltalab | 200209 | No special requirements |
| Agar | Sigma-Aldrich | A1296 | No special requirements |
| Bacteriophage lysate | Minimal concentration: 109 PFU/mL | ||
| Centrifuge | Eppendorf | No special requirements | |
| Chloroform | Panreac | 131252 | No special requirements |
| Citric acid · H2O | Merck | 1.00247 | |
| Colony counter | No special requirements | ||
| Evans Blue | Sigma-Aldrich | E-2129 | |
| Flasks | No special requirements | ||
| Fluorescein sodium salt | Sigma-Aldrich | F-6377 | |
| Forceps | No special requirements | ||
| Glass tubes | No special requirements | ||
| Glass tubes for lysate | No special requirements | ||
| Glucose | Sigma-Aldrich | G7021 | |
| K2HPO4 | Merck | 1.05104.1000 | |
| K2HPO4 anhydrous | Merck | 1.05104 | |
| Lipopolysaccharide from Salmonella enterica serotype Typhimurium | Sigma-Aldrich | L6511-25 mg | Dissolved in sterile water |
| Membrane 0.45 µm | MF-Millipore | HAWP02500 | No special requirements |
| MgSO4 · 7 H2O | Merck | 1.05886 | |
| NaCl | Sigma-Aldrich | S9888 | No special requirements |
| NaNH4HPO4 · 4 H2O | Sigma-Aldrich | S9506 | |
| Peptone | iNtRON | Ba2001 | No special requirements |
| Syringe Filter 0.22 µm | Millex | SLGSR33SB | No special requirements |
| Toothpicks | No special requirements | ||
| Tryptone | Panreac | 403682.1210 | No special requirements |
| Vacuum pump | Thermo Scientific | No special requirements | |
| Yeast extract | iNtRON | 48045 | No special requirements |
Referenzen
- Kasman, L. M., Porter, L. D. . Bacteriophages. , (2024).
- Lawrence, D., Baldridge, M. T., Handley, S. A. Phages and human health: More than idle hitchhikers. Viruses. 11 (7), 587 (2019).
- Beller, L., Matthijnssens, J. What is (not) known about the dynamics of the human gut virome in health and disease. Curr Opin Virol. 37, 52-57 (2019).
- Roux, S., Hallam, S. J., Woyke, T., Sullivan, M. B. Viral dark matter and virus-host interactions resolved from publicly available microbial genomes. eLife. 4, 08490 (2015).
- Wommack, K. E., Colwell, R. R. Virioplankton: Viruses in aquatic ecosystems. Microbiol Mol Biol Rev. 64 (1), 69-114 (2000).
- Louten, J. Virus structure and classification. Essentl Human Virol. , 19-29 (2016).
- Egido, J. E., Costa, A. R., Aparicio-Maldonado, C., Haas, P. J., Brouns, S. J. J. Mechanisms and clinical importance of bacteriophage resistance. FEMS Microbiol Rev. 46 (1), 048 (2022).
- Rakhuba, D. V., Kolomiets, E. I., Dey, E. S., Novik, G. I. Bacteriophage receptors, mechanisms of phage adsorption and penetration into host cell. Polish J Microbiol. 59 (3), 145-155 (2010).
- Lindberg, A. A. Bacteriophage receptors. Ann Rev Microbiol. 27 (1), 205-241 (1973).
- Koskella, B., Meaden, S. Understanding bacteriophage specificity in natural microbial communities. Viruses. 5 (3), 806-823 (2013).
- Marcó, M. B., Moineau, S., Quiberoni, A. Bacteriophages and dairy fermentations. Bacteriophage. 2 (3), 149-158 (2012).
- Fernández-Fernández, R., et al. Evolution of a bistable genetic system in fluctuating and non-fluctuating environments. bioRxiv. , (2024).
- Cota, I., Blanc-Potard, A. B., Casadesús, J. STM2209-STM2208 (opvAB): a phase variation locus of Salmonella enterica involved in control of O-antigen chain length. PloS one. 7 (5), e36863 (2012).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods Mol Biol. 501, 69-76 (2009).
- Ackermann, H. W. Tailed bacteriophages: The order Caudovirales. Adv Virus Res. 51, 135-201 (1998).
- Wilkinson, R. G., Gemski, P., Stocker, B. A. D. Non-smooth mutants of Salmonella typhimurium: Differentiation by phage sensitivity and genetic mapping. J Gen Microbiol. 70 (3), 527-554 (1972).
- Casjens, S. R., Leavitt, J. C., Hatfull, G. F., Hendrix, R. W. Genome sequence of Salmonella Phage 9NA. Genome Announc. 2 (4), e00531 (2014).
- Cota, I., et al. Epigenetic control of Salmonella enterica O-antigen chain length: A tradeoff between virulence and bacteriophage resistance. PLoS Genet. 11 (11), e1005667 (2015).
- Chan, K., Botstein, D., Watanabe, T., Ogata, Y. Specialized transduction of tetracycline resistance by phage P22 in Salmonella typhimurium. Virology. 50, 883-898 (1972).
- Bertozzi Silva, J., Storms, Z., Sauvageau, D. Host receptors for bacteriophage adsorption. FEMS Microbiol Lett. 363 (4), 002 (2016).
- Sørensen, M. C. H., et al. Bacteriophage F336 recognizes the capsular phosphoramidate modification of Campylobacter jejuni NCTC11168. J Bacteriol. 193 (23), 6742-6749 (2011).
- Ogata, S., Hongo, M. Bacteriophages of the Genus Clostridium. Adv Appl Microbiol. 25, 241-273 (1979).
- Olivenza, D. R., Casadesús, J., Ansaldi, M. Epigenetic biosensors for bacteriophage detection and phage receptor discrimination. Environ Microbiol. 22 (8), 3126-3142 (2020).
- Olivenza, D. R., et al. A portable epigenetic switch for bistable gene expression in bacteria. Sci Rep. 9 (1), 862 (2019).
- Walter, M., et al. Structure of the receptor-binding protein of bacteriophage Det7: a Podoviral tail spike in a Myovirus. J Virol. 82 (5), 2265-2273 (2008).
- Davies, M. R., Broadbent, S. E., Harris, S. R., Thomson, N. R., vander Woude, M. W. Horizontally acquired Glycosyltransferase operons drive Salmonellae lipopolysaccharide diversity. PLoS Genet. 9 (6), e1003568 (2013).
- Wahl, A., Battesti, A., Ansaldi, M. Prophages in Salmonella enterica: a driving force in reshaping the genome and physiology of their bacterial host. Mol Microbiol. 111 (2), 303-316 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten