Method Article
从原子力显微镜衍生的力图中提取天然小鼠肺基底膜的杨氏模量
* 这些作者具有相同的贡献
摘要
该协议可视化了如何制备小鼠肺组织的冷冻切片、进行原子力显微镜力图实验以及分析数据以确定天然小鼠肺基底膜的杨氏模量。
摘要
原子力显微镜 (AFM) 允许以几十纳米的空间分辨率表征样品的机械性能。由于哺乳动物细胞会感知并对其直接微环境的力学做出反应,因此以高空间分辨率表征组织的生物力学特性对于理解各种发育、稳态和病理过程至关重要。基底膜 (BM) 是一种大约 100 - 400 nm 的薄细胞外基质 (ECM) 亚结构,在肿瘤进展和转移形成中起着重要作用。尽管确定如此薄的 ECM 亚结构的杨氏模量具有挑战性,但 BM 的生物力学数据为 BM 如何影响细胞行为提供了基本的新见解,此外,还提供了有价值的诊断潜力。在这里,我们提出了一种可视化方案,用于评估小鼠肺组织中 BM 机制,小鼠肺组织是易发生转移的主要器官之一。我们描述了一种确定 BM 的杨氏模量的有效工作流程,BM 位于肺组织的内皮细胞层和上皮细胞层之间。分步说明包括小鼠肺组织冷冻、冷冻切片和组织切片上的 AFM 力图记录。此外,我们使用 CANTER Processing Toolbox 提供半自动数据分析程序,CANTER Processing Toolbox 是一款内部开发的用户友好型 AFM 数据分析软件。该工具可以自动加载记录的力图,将力与压电拉伸曲线转换为力与压痕曲线,计算杨氏模量,并生成杨氏模量图。最后,它展示了如何通过使用空间过滤工具来确定和分离从肺 BM 得出的杨氏模量值。
引言
AFM 力图已成为一种广泛使用的技术,用于确定各种生物样品的纳米力学特性,具有纳米空间分辨率和皮微孔力灵敏度 1,2,3,4,5,6,7,8,9,10,11,12 .P. Hansma 及其同事对生理条件下生物样品的 AFM 测量应用进行了开创性研究 13,14,15,16,17,18,19,20。随后,采用 AFM 力测量来评估单个分子21、活细胞及其细胞骨架9、10、22、23、24、25、26、组织和组织切片6、8、11、27、28、 29,30,31,以及生物水凝胶 3,4,32。在我们的研究中,我们使用 AFM 来检查生物键和化学键的机械特性18、33、34、35、36、37 并测量单个细胞的纳米力学特性38、39、40。此外,生长板软骨8 的测量表明,胶原蛋白结构和细胞外基质 (ECM) 的杨氏模量指导软骨细胞分裂,从而指导胚胎发育过程中小鼠生长板的生长方向。此外,我们研究了关节软骨软骨变性和骨关节炎相关的变化2、41、42、43、44、45、46、47。
近年来,许多 AFM 研究检查了健康和病理肺组织的机械特性 48,49,50,51,52。然而,这些研究主要检查了整体组织力学,而不是关注特定的组织成分,例如 BM。BM 构成了特殊 ECM 结构的薄层(人类为 100 nm - 400 nm),该结构排列在大多数哺乳动物器官和组织结构(如神经元、肌肉、脂肪细胞组织和血管)上。我们发现小鼠肺 BM 的机械特性在转移形成过程中起着关键作用,较软的 BM 与乳腺癌和肾癌患者较高的生存概率相关53。此外,这项研究表明,分泌的细胞外基质蛋白 netrin-4 通过与层粘连蛋白 γ1 的化学计量 1:1 相互作用降低 BM 的杨氏模量,层粘连蛋白 γ 是 BMs40 内几乎所有层粘连蛋白网络中的关键成分。
在这里,我们展示了我们开发的方案的可视化,以确定 100 - 200 nm 薄小鼠肺 BM 的杨氏模量 (EBM)。BM 的杨氏模量是其刚度的量度,定义为线性弹性变形期间应力(每单位面积的力)与产生的轴向应变(位移)的比率54,在这种情况下,压痕 AFM 针尖对 BM 进行小幅压缩。在 AFM 实验中,力和位移(压痕深度)可以从 AFM 悬臂偏转和悬臂位置获得,杨氏模量(应力-应变-关系)可以通过使用合适的弹性模型从产生的力与压痕曲线中提取,该模型考虑了压痕尖端的几何形状(在本例中为改进的赫兹模型55)。主要步骤如图 1 所示。最初,该协议描述了在 OCT 化合物中制备和包埋小鼠肺组织样品的方法(参见步骤 1),从而促进了随后的冷冻切片程序(步骤 2),从而能够使用 AFM 力图确定 BM 刚度。这里介绍的冷冻切片技术利用单面和双面胶带,无需固定和解冻切片即可切片肺组织。该协议的第三部分概述了收集合适数据以评估 BM 刚度的详细 AFM 程序。在协议的下一节中,我们将解释如何使用内部开发的基于 MATLAB 的软件 CANTER Processing Toolbox56 自动分析产生的力体积(步骤 4.1)。在此步骤中,通过拟合修改后的赫兹模型,从每个记录的力-压痕曲线中提取杨氏模量,该模型针对压头几何形状进行了校正,在本例中为四边金字塔55。最后,我们在步骤 4.2 和 4.3 中描述了如何应用开发的工具箱的空间过滤工具和 R2 过滤工具,从包含肺泡壁所有组织隔室的力图中获得的杨氏模量值中提取特定于基底膜 (BM) 的杨氏模量值。
研究方案
根据丹麦动物福利法,丹麦环境和食品部动物实验委员会批准了处理动物样本的所有程序(许可编号 2017-15-0201-01265)。为了证明该协议,我们使用了 13 周龄的雄性 C57BL/6 小鼠。
注意:在以下方案中,有几个步骤需要处理组织标本。处理生物样品时,始终佩戴足够的手套和实验室外套。
1. 样品制备
- 将新鲜收获的小鼠肺放入一次性 15 x 15 x 5 mm 的底座模具中。使用带有 0.9 mm 针头的 10 mL 注射器,小心地将最佳切割温度 (OCT) 培养基与磷酸盐缓冲盐水 (PBS) 溶液的 1:1(体积-体积)比例注入样品中,同时缩回针头。在室温下进行注射,同时用小型手术镊子牢固地固定样品。当样品肿胀并观察到明显的渗出量时,认为注射完成。
- 将纯 OCT 填充到模具中,直到试样完全浸入。为确保整个肺完全冷冻,请将样品在 -80 °C 的温度下储存至少 4 小时。
注:此时,样品可以在 -80 °C 下储存至少 2 年。 根据制造商的 OCT 规格,包埋组织的储存时间超过 10 年。通常建议将需要储存超过 4 个月的样品包裹在透明薄膜中,以防止水分被样品吸收。
2. 包埋肺标本的冷冻切片
- 将冻存仪腔室的温度调节至 -17 °C,因为这是肺组织样品的最佳切割温度。将切割台装置的温度设置为 -22 °C(如果此功能可用)。这有助于确保在整个切片过程中保持样品表面的最佳切割温度。
- 使用双面胶带准备 40 张显微镜载玻片,小心地将其放在载玻片中间,然后将 15 mL 离心管卷在胶带上,以确保牢固且无气泡的粘附。确保双面胶带的长度大致等于包含样品的 OCT 块的宽度。带有磨砂边缘的显微镜载玻片允许使用包含样品信息和章节编号的铅笔进行标记。
- 将准备好的显微镜载玻片插入载玻片盒中。随后,将装有载玻片的盒子放入冷冻切片机腔室中,以便冷却它们。
注意:冷却载玻片可防止在拾取切片时发生冻融循环,这会损害样品的机械性能。 - 准备多个单面胶带,每个胶带的尺寸与冷冻样品块的大小大致相同。
- 用 OCT 培养基填充样品架的内两个环。将样品放在样品架上的 OCT 培养基中,确保其尽可能水平。随后,将样品架置于冻存室中约 10 分钟,直到 OCT 培养基完全凝固并且样品牢固地固定在支架上。
- 将装有样品的支架安装到低温切片机的切割台装置上。根据设备的用户手册调整冷冻切片机的所有基本组件,例如切割台的倾斜度和刀片的位置,以建立一个平行于 OCT 块表面对齐的切割平面。
- 通过将切片厚度调整到 50 μm 来对样品进行初步修剪,然后开始切割,直到到达样品内所需的位置(通常从其表面在肺标本内部约 500 μm)以收集组织载玻片。将冷冻切片机的切片厚度调整为 15 μm,这与组织切片的所需厚度相对应。
注意: 使用冷冻切片机等切割工具时要小心谨慎。为确保人身安全,请遵守制造商的安全指南,并让手指远离刀片。 - 用拇指用力按压,将一块单面胶带贴在样品上。事先,将拇指靠在冻存机的腔室壁上几秒钟,确保拇指已冷却。
注意:胶带应牢固地贴在样品上,因为任何缺乏粘附都可能导致胶带在切片过程中从组织上脱落。 - 用冷冻切片产生 15 μm 的组织切片。使用刷子引导切片并防止胶带在切割过程中从样品上脱落。为了组织切片的光滑表面,切割速度必须保持缓慢和一致。
- 在上一步中创建的部分现在平放在刀片块上,磁带朝上。使用其中一张带有载玻片盒中的双面胶带的冷冻显微镜载玻片,然后将其用力按压到切片上以将其拾取。然后,将现在携带切片的显微镜载玻片放回载玻片盒中。
注意:处理显微镜载玻片和切片时,请确保始终将它们放在冷冻切片机室内,以防止切片在切割过程中解冻。 - 重复前三个步骤,直到收集到所需数量的部分,即 40 个。
- 完成后,盖上载玻片盒的盖子,将其放在干冰上转移到 -20 °C 的冰箱中。最后,通过对所有用过的物品进行消毒并打开冷冻切片机室中的紫外线灯来清理冻存机。
注意:此时可以暂停协议。将样品储存在 -20 °C 的温度下,直到进行 AFM 测量。切片和 OCT 培养基的推荐储存时间不应超过 6 周。超过此时间范围,切片和 OCT 培养基可能会表现出类似于冷冻灼伤的变化,因为它们的表面积很大并且容易吸收水分。
3. 肺切片的 AFM 力图
注意:对于该协议,JPK NanoWizard 4XP (AFM) 和布鲁克的电动载物台与徕卡的 DMi8(倒置光学显微镜)相结合,用于记录肺切片的力-位移曲线。
- 设置 AFM
- 打开 AFM 设置的所有必需设备(例如,计算机、AFM 控制器、隔振台、光学显微镜等)。
- 启动 AFM 控制软件。在 Choose Experiment overview(选择实验 概述)中,选择 QI Advanced Imaging 模式。此外,指定实验数据和校准文件的存储位置。
注意:如果您没有 QI Advanced Imaging 扩展,请使用 Contact Mode Force Mapping 模式作为替代选项。 - 为正在观察的特定样品类型选择具有合适弹簧常数的悬臂。对于软肺组织样品,建议使用布鲁克的 MLCT 悬臂 F,其尖端为金字塔形,标称弹簧常数为 0.6 N/m。如果芯片包含多个悬臂(例如 MLCT),请在体视显微镜下使用一对镊子从芯片上取下任何更长的悬臂,以确保只有用于测量的悬臂与样品接触。
注意:与样品接触的其他悬臂(用于测量的悬臂除外)可能会导致样品在实验过程中发生意外移动,甚至导致样品与下面的胶带分离。
注意: 避免在当前或后续步骤中使用镊子接触用于测量的悬臂。 - 将芯片安装到悬臂支架上。将支架插入 AFM 头。将 AFM 头放在位于倒置光学显微镜上的样品台上。按照用户手册中的说明对齐光学杆路径。当激光束最初放置在测量悬臂的前端时,即可实现光学杆路径的正确对准,随后,反射的激光束照射到分段光电二极管的中心。
- 使用热噪声方法进行悬臂校准57
注意:悬臂的校准包括确定其倒置光杆灵敏度 (InvOLS) 和弹簧常数。每个使用的悬臂的单独校准都是必要的,因为通常为悬臂提供的标称弹簧常数通常只是一个粗略的近似值。在根据记录的力曲线确定样品机械性能的实验中,例如本协议中描述的曲线,必须通过基于接触的方法在刚性基板上直接确定 InvOLS。确定 InvOLS 的替代方法,例如从悬臂的几何形状中推导出 InvOLS,只能提供粗略的近似值,不适用于此处描述的应用类型。或者,如果在供应商提供精确弹簧常数的情况下使用悬臂,则可以通过 Schiller 等人描述的程序来确定 InvOLS。58.- 将显微镜载玻片放在 AFM 的样品台上。显微镜载玻片用作基于接触测定 InvOLS 的刚性基板。
- 将 AFM 头放在样品台上后,使用步进电机降低头,直到悬臂支架和显微镜载玻片之间的剩余间隙仅约为 1 - 2 mm。使用带有长针头 (0.9 mm x 70 mm) 的 1 mL 注射器,将 PBS 涂抹在悬臂支架的侧面,使其向动并在支架和显微镜载玻片之间的间隙中形成液体弯月面。
注意:确保用于样品力体积测量的相同液体(例如缓冲液或介质)也用于校准过程。 - 要在软件中执行基于接触的校准,请导航到 Acquire Data 页面并选择 Advanced View 选项。使用位于右上角的 burger 菜单按钮访问 Calibration Manager 。在此界面中,选择 Contact-based 作为 Method 并从下拉菜单中选择 MLCT F 悬臂作为悬臂名称。此外,在相应的输入字段中将 Setpoint 设置为 1 V,将 Number of scans 设置为 10。
- 在 Acquire Data 页面的左侧控制面板中,为自动接近程序输入 1 V 的设定值。单击用户界面左上角的蓝色向下箭头按钮以启动自动进近。当方法完成并且悬臂与显微镜载玻片接触时,单击 Calibration Manager 窗口中的 Calibrate 按钮。
注意:然后,通过记录 10 条力曲线以及热功率谱密度 (PSD) 自动确定 InvOLS 和悬臂弹簧常数,PSD 用于通过热噪声法57 确定弹簧常数。自动校准程序完成后,目视检查拟合曲线是否正确描述了记录的悬臂 PSD 的法向模式(第一个峰值),以及确定的弹簧常数是否与悬臂制造商给出的标称弹簧常数具有相同的数量级。如果不是这种情况,则必须手动调整拟合范围,以确保拟合收敛到并代表热功率谱的第一个峰值。 - 校准完成后,关闭 Calibration Manager 窗口。关闭 校准管理器后,在预选目录(参见步骤 3.1.2)中生成一个校准文件(*.tnd ascii 文件),其中包含确定的校准结果,稍后数据分析需要这些结果。记录校准值,以便在数据分析过程中以备将来参考,例如,在实验室笔记本中。
注意:从此步骤开始,请勿更改悬臂上的激光位置、调整 AFM 头中的镜子或重新定位悬臂支架。修改这些元素将影响 InvOLS 并使校准值无效。为了补偿热漂移,请调整 AFM 头的分段光电二极管的位置。
- 分析肺切片上的肺泡壁
- 从冰箱中取出标本载玻片进行评估。让样品在室温下解冻约 1 分钟,直到整个 OCT 培养基变得透明。随后,向肺组织切片中添加一些 PBS 液滴以使其再水化。
- 将携带肺组织切片的显微镜载玻片放在样品台上。将 AFM 头装回样品台上并降低头,直到悬臂位于样品上方约 1 mm 处。使用 1 mL 注射器和 0.9 mm x 70 mm 针头在悬臂支架的侧面添加一些额外的 PBS,直到在组织切片和悬臂支架之间形成液体弯月面。
- 导航到 设置管理器 通过单击用户界面右上角的 带有扳手图标的按钮。在设置管理器中,调整以下常规设置。在 Approach Settings 部分中,将 Target Height 设置为 4 μm。然后,在 Current Mode Settings 部分中,继续 Advanced Feedback Settings 并将 Multiplier 设置为 1。最后,在 Force Settings 子部分(也位于 Current Mode Settings 部分),从“Mode at end”下拉菜单中选择选项 Retracted Piezo。
- 使用倒置光学显微镜识别并导航至完整的肺泡壁。完整的墙壁在明场图像中的特点是边缘暗而光滑。(参见图 2A-B)。
注意:在 AFM 控制软件中导入明场图像(如果可用)可以帮助将悬臂导航到特定的肺泡壁,并准确捕获其顶部的力或 QI 图。在 JPK SPM 软件中,此功能称为 Direct Overlay。 - 根据使用的 AFM、使用的悬臂和观察到的样品,在左侧控制面板中设置力-压痕曲线的参数。对于布鲁克的 NanoWizard 4XP 和 MLCT 悬臂 F,我们建议使用以下参数: 设定值 = 5 nN; z 长度 = 8 μm; z 速度 = 300 μm/s。
注意:推荐的 z 长度 8 μm 相当大。这归因于肺组织的特性。鉴于肺组织通常非常柔软,即使相对适中的设定点为 5 nN,在某些点的压痕深度也可以达到 4 μm。为了保证尖端不与样品接触的基线至少占力-压痕曲线的一半左右,建议将曲线的总长度设置为 8 μm。这将大大简化以后的曲线分析。对于其他生物原子力显微镜,这些参数也应该适用,尽管某些参数的命名法,例如决定最大压痕力的设定点,可能因制造商而异。如果 AFM 系统无法达到此处给出的高垂直速度,则必须根据 AFM 制造商的建议降低 z 速度。
注意:为了确保结果的可比性,必须在所有实验中采用相同的 z 接近速度。 - 设置初始总览力图的位置和大小,确保它包含整个牙槽壁,从一侧空气延伸到另一侧。对于小鼠肺组织样品,20 μm x 20 μm 和 30 μm x 30 μm 之间的尺寸通常就足够了。无论贴图的特定大小如何,都将像素数(对应于记录的力曲线数)设置为 50 x 50。这导致为每个映射生成 2,500 条力-缩进曲线,从而提供了必要的分辨率来识别力映射的斜率通道中的 BM。(参见图 2C-D)。
- 录制总览图后,检查力图的高度和坡度通道。如果高度通道表明牙槽壁完整(见图 2D),并且如果可以将斜率通道中的 BM 区分为位于上皮和内皮两层之间的明确定义的亮线(见图 2C,E),请在 BM 上捕获尺寸为 3 μm x 3 μm 或 4 μm x 4 μm 的更聚焦的力图。将像素保持在 50x 50 条曲线。
注意:此过程导致更高的地图分辨率,并允许记录更多的杨氏模量值,特别是对于 BM(参见图 2E-F)。此外,这使得 BM 和周围组织之间能够更准确地区分,从而提高后续空间过滤过程的精度。如果概览图的通道显示坍塌或破裂的壁,由于悬臂不仅用尖端接触样品而导致的伪影,或者在斜率通道中没有可识别的 BM,请导航到组织切片上的另一壁,直到可以识别出具有无伪影评估的壁 BM。
对同一肺标本的更多切片重复前面的步骤(3.3.6 和 3.3.7),直到评估到所需数量的肺泡壁。建议每个肺标本至少有四个壁,以获得稳健的结果59。 - 测量完成后,清洁所有与样品和 PBS 接触的设备和材料。如果不再需要样品,请将其丢弃在可高压灭菌的废玻璃容器中。保存所有记录的数据,并关闭 AFM 控制软件和计算机。最后,关闭所有使用的电气设备。
注意:此时,可以暂停协议。实验数据被保存,可以在以后的时间点进行分析。
4. 数据分析
- 力-压痕曲线分析
注意:力曲线分析应用程序的详细手册可在此处找到:https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/2a.-Force-Curve-Analysis。- 在 MATLAB 中启动 CANTER ProcessingToolbox 56 并打开 力曲线分析 应用程序。
注意:可以通过以下链接从 GitHub 存储库下载 Toolbox:https://github.com/CANTERhm/CANTER_Processing_Tool。README.md 文件包含在有关如何使用 MATLAB 安装软件和启动软件的综合指南,可在 GitHub 存储库中找到。有关 Toolbox 中包含的每个应用程序的更多信息,请参阅相应的 wiki https://github.com/CANTERhm/CANTER_Processing_Tool/wiki。 - 通过单击“ 选择文件 ”按钮,导航到保存力图的位置,双击它,然后单击应用程序用户界面上的“ 加载数据 ”按钮,加载高分辨率(3 μm x 3 μm 或 4 μm x 4 μm)力图。
- 此时会出现一个弹出窗口,请求为用于记录在上一个协议步骤中选择的力图的悬臂确定的校准值(InvOLS 和弹簧常数)(请参阅步骤 3.2)。在相应的编辑字段中输入校准值。如果在步骤 3.2.4 中未记录这些值,请参阅自动生成的校准文件,其中也包含校准值。对于是否应计算针尖-样品分离的问题,保留默认选项 yes 。随后,单击 提交 按钮继续。
- 出现第二个弹出窗口,允许为压痕样品选择压头几何形状和泊松比。对于使用的 MLCT 悬臂,选择 Four-sided pyramid 作为尖端几何图形,并输入 17.5° 作为到边缘的半角(两者都是默认值)。使用默认的泊松比 0.5,它表示不可压缩的材质。要继续 QI 映射的加载过程,请单击 提交 按钮。
注意:在加载过程中,记录的偏转-位移曲线(AFM 的原始数据)根据提供的校准值转换为力-压痕曲线。有关各个转换步骤和相关方程的全面理解,请参阅 Hartmann 等人的补充信息59。 - 加载过程完成后,力图的初始力曲线将显示在屏幕上。确保选择基线校正模式 Offset + Tilt 。此模式会自动检测力感应曲线的基线,并将倾斜和垂直偏移校正为零。此外,选择 via Hertz model 作为 Contact Point Finder 的算法,并在此选项的指定编辑字段中使用 20% 的值。该算法特别适用于检测软组织样本上记录的力-压痕曲线中的接触点。最后,在相应的编辑字段中将拟合深度设置为 1.5 μm。
注意:为确保显微镜载玻片的刚度不会影响产生的杨氏模量,建议将最大拟合深度设置为不大于组织切片厚度60 的 10% 的值。有关校正算法的更多详细信息,请参阅 Hartmann 等人的补充信息59。
注意:在整个实验中使用相同的拟合深度,以保证结果具有可比性。 - 要将修改后的赫兹模型的拟合应用于QI地图的所有力-压痕曲线,请点击 “保留并应用于全部 ”按钮。将打开一个对话框窗口,询问在分析过程中是否应显示每条力曲线。如果不显示曲线,则处理速度会略微加快。
- 完成最后一次力曲线分析后,将出现一个窗口,询问是否应保存拟合结果。以保存两者中的结果。tsv(制表符分隔值)和 .xlsx (Excel) 文件格式,请单击 Yes 并输入结果文件的名称。此外,还会保存文件扩展名为 *.meta_data 的文本文件,其中包含有关曲线分析的所有详细信息,例如使用的校准值、所选的拟合模型、所选的接触点检测算法、拟合范围和其他相关信息。
- 通过单击主窗口右上角的十字图标关闭 Force Curve Analysis 应用程序。
- 在 MATLAB 中启动 CANTER ProcessingToolbox 56 并打开 力曲线分析 应用程序。
- QI 地图结果的空间筛选
注意:空间过滤步骤可确保在进行任何进一步分析时仅考虑源自 BM 的 QI 图的杨氏模值。结果 筛选工具 的详细手册可在此处找到:https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/Result-Filtering-Tool。- 从 CANTER Processing Toolbox 的应用程序选择窗口的应用程序列表中选择 Result Filtering Tool,然后单击 Start Application 按钮启动应用程序。
- 要加载拟合结果,请点击 Result Filtering Tool 用户界面顶部菜单栏中的 Open。在随后的弹出窗口中,找到位于 JPK Maps 选项卡中的第一个 Set 按钮,然后选择包含力曲线分析结果的 .tsv 文件(请参阅步骤 4.1.6)。单击第二个 Set 按钮,找到与已选择的 .tsv 文件对应的 QI 映射文件。随后,单击 Submit 以加载地图数据和力曲线分析结果。
- 从顶部的 Displayed channel 下拉菜单中选择 Emodul 选项,以将获得的杨氏模数结果显示为映射图像。此外,从直方图下方的 Data channel 下拉菜单中选择 Emodul 选项(如果尚未选择),以可视化 QI 映射的加载杨氏模值的分布。
- 单击位于用户界面中心的 Manipulation flow 箭头按钮,并确保它指向右侧。将 图像过滤器 切换按钮设置为位于直方图轴上方的过滤器面板中的 On 位置。选择 手绘 选项从 过滤器几何 下拉菜单,然后单击 添加 按钮。
- 在顶部通道图像中,通过在按住鼠标左键的同时环绕 BM 来绘制滤镜蒙版。与邻近的较软组织相比,BM 在 Young 模量图中可识别为明亮(高 Young 模量值)结构(参见图 3A)。绘制完蒙版后,松开鼠标左键并双击要应用于地图的蒙版。右侧的直方图现在仅显示以绿色突出显示的地图区域的杨氏模量值(默认颜色;参见图 3C-D)。
- 要创建仅包含掩蔽杨氏模量值的新 .tsv 结果文件,请单击用户界面顶部菜单栏中的 保存>保存直方图 > 保存数据。或者,右键单击直方图绘制轴的空白区域,然后从上下文菜单中选择 Save data 。
- 在弹出窗口中单击 Select All (全选 ) 询问要将哪些结果写入 .tsv 文件。然后,通过单击确认选择 OK 按钮。然后,在保存对话框中输入 .tsv 文件的名称,选择所需的保存位置,最后单击 OK 保存过滤结果。
- 通过单击应用程序右上角的 Cross Icon 关闭 Result Filtering Tool。
- R2 结果筛选
注意:此协议步骤确保仅考虑杨氏模量结果,其中修改后的赫兹模型显示与拟合它的力-压痕曲线具有良好的一致性。R ² 过滤工具 的详细手册可在此处找到:https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/R%C2%B2-Filtering-Tool。- 在 CANTER Processing Toolbox 的应用程序选择窗口的应用程序列表中选择 R² 过滤工具,然后单击 Start Application,启动 R² 过滤工具。
- 导航到用户界面的顶部菜单栏,然后单击 打开 .tsv 文件。在随后的加载文件对话框中,找到包含上一步 4.2.5 中保存的 BM 的杨氏模量结果的 .tsv 文件,然后双击选择它。
- 此时将出现一个对话框,其中显示 .tsv 文件中包含的数据列的列表。选择 Emodul 作为包含 Young 模数数据的列。随后,在请求 R² 数据的单独列表框对话框中,选择 rsquare_fit。 加载过程完成后,加载的杨氏模量数据以直方图的形式显示在屏幕的左上角,而 R² 数据分布则显示在右上角的图表中。
- 在图形用户界面中央的编辑字段中输入 0.96。单击 Filter Data 按钮,使用输入的值应用 R² 过滤器。此作将仅保留 R² 值为 0.96 或更高的结果。杨氏模量结果的过滤分布和相应的 R² 值显示在用户界面的下半部分。
- 从顶部菜单栏中选择 Save filtered data 以创建新的 .tsv 文件,该文件仅包含满足定义的 R² 筛选条件 > 0.96 的结果。保存的数据现在可以显示为直方图,例如,使用 直方图绘图工具,该工具可视化探测的肺 BM 的杨氏模量分布(参见 图 4A)。
结果
在识别和测量完整的肺泡壁后,该方案的主要结果是过滤后的 BM 的杨氏模量值。需要注意的是,不可能在每个组织切片中都找到合适的肺泡壁进行 AFM 测量。在我们的实验观察中,大约 75% 的小鼠肺标本产生的冷冻切片表现出可量化的肺泡壁。为了准确确定肺 BM 中杨氏模量的空间分布,我们建议采用跨度为 3 μm x 3 μm 或 4 μm x 4 μm 的力图。这种方法确保了检查小鼠肺 BM 的足够分辨率,其厚度通常为 100 nm - 200 nm。因此,实验方法涉及为每个力贴图捕获 50 x 50 条力曲线,总共产生 2,500 条力曲线。在力贴图中,力曲线的间距相等。这意味着,除了 BM 的杨氏模量外,即使在小的力图中,也会探测 BM 周围的组织结构。因此,为了准确和选择性地确定肺 BM 的杨氏模量,我们在该方案中实施了两步过滤过程。
依赖于作员的空间过滤的初始过滤步骤选择源自小鼠肺 BM 的力曲线拟合结果,占分析的力图中包含的总结果值的大约 10% 到 25%。在此初始空间过滤之后,将保留大约 250 - 625 个拟合结果以供进一步分析。下一个过滤步骤确保仅包含源自由修改后的赫兹模型充分描述的力曲线的 BM 相关结果。因此,使用修正的 Hertz 模型拟合的决定系数 (R2) 作为过滤准则。我们的实证经验表明,保持 R2 值高于 0.96 的结果适用于通过遵守该协议获得的拟合结果。需要注意的是,R2 值受力曲线基线的质量(平滑度)和长度、接触点的确定以及拟合模型的收敛性有显著影响。在应用这两种过滤程序之后,单个肺泡壁的 BM 通常保留大约 100 到 500 杨氏模量值。
BM 中这些剩余杨氏模量值的分布可以使用直方图进行可视化(图 4A)。需要注意的是,得到的肺 BM 的杨氏模量值服从对数正态分布,这通常适用于只能达到正值的随机变量61。图 4B 中的 QQ 图对此进行了可视化。因此,正态(高斯)分布可以拟合到对数变换的杨氏模量 (E) 值的分布。在这里,自然对数 (ln) 用于变换 E 值。从该拟合中提取分布的峰值位置 (μ) 和标准差 (σ),然后使用以下方程重新转换以获得具有代表性的杨氏模量,表示为 EBM:
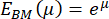
请注意,在重新变换之后,标准差区间不再围绕峰值对称。标准差区间的负 (σ-) 和正 (σ+) 边缘可以使用以下表达式计算:
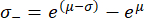
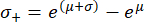
这些方程是指数函数是自然对数62 的反函数这一事实的结果。
重新转换峰值 μ = 9.31 且标准差 σ = 0.18 从 图 4A 中的直方图确定后,代表性杨氏模量值为 EBM = 11.05 kPa,标准偏差区间为 [-1.82 kPa, +2.18 kPa]。或者,可以将对数正态分布拟合到未变换的杨氏模量 (E) 值的直方图,以确定分布的特征参数。
建议每只小鼠至少分析四个壁以获得稳健的代表性结果,正如我们在 Hartmann 等人 59 中详细展示的那样。为了确定小鼠的代表性杨氏模量 EBM ,将所有四个壁的对数变换值绘制在一个组合直方图中,并通过拟合正态分布确定μ和 σ。随后,如前所述重新转换 μ 和 σ 以检索 EBM 和主题小鼠的标准差区间。
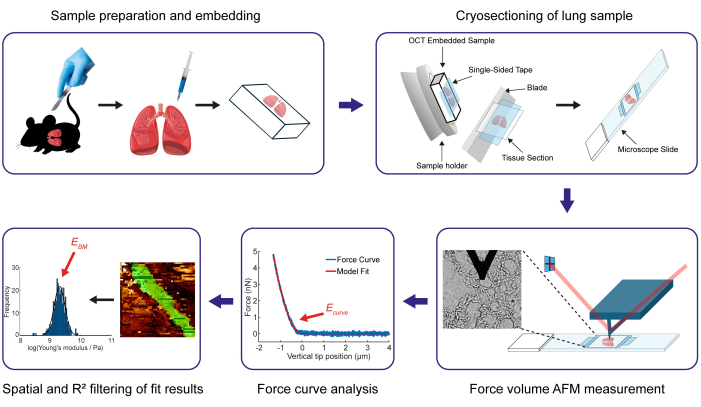
图 1:确定小鼠肺基底膜杨氏模量的主要方案步骤概述。 第一步包括获取和准备鼠肺,然后嵌入它们。因此,向肺注射 OCT 培养基和 PBS 的组合,然后完全浸入 OCT 化合物中。之后,通过使用冷冻切片机冷冻切片获得 15 μm 厚的肺组织切片。在该协议中,我们详细介绍了在切割过程中使用单面和双面胶带将样品固定到位的过程。随后,通过对组织切片的 AFM 力图测量来确定 BM 的杨氏模量值。在完成力曲线分析过程后,应用空间和 R² 滤波来隔离感兴趣结构的杨氏模量结果,特别是 BM(在力图中以绿色显示,并在对数变换的杨氏模量分布中量化)。 请单击此处查看此图的较大版本。

图 2:在小鼠肺组织切片上进行的示例性力图 AFM 实验。 (A) 小鼠肺组织切片的明场显微镜概览图像,能够识别位于两个肺泡之间的合适完整肺泡壁。在图像的右侧,可以看到 AFM 的三角形 MLCT 悬臂 F。(B) 组织切片中选定壁的放大视图。(C) 斜率和 (D) 高度通道 15 x 15 μm 概览图,其中包含牙槽壁的 50 x 50 条力曲线。在斜率通道中,BM 可以识别为一条亮线(白色箭头),表示墙内刚度增加的结构。高度通道确认了墙壁的完整性,没有显示出破裂的迹象。在概览 15 x 15 μm 图中识别 BM 后,记录一个较小的 4 x 4 μm 图(E:斜率通道, F:高度通道),具有 50 x 50 条力曲线。比例尺:(A) 200 μm 和 (BD) 25 μm。所示 AFM 图像的色标范围为 (C) 2.36 - 9.29 nN/μm,(D) 0 - 3.38 μm,(E) 2.36 - 9.29 nN/μm(概览图像)和 2.42 - 8.37 nN/μm(较小图像),(F) 0 - 3.38 μm(概览图像)和 0 - 1.99 μm(较小图像),从暗到亮。所有 AFM 图像都使用线性色标。 请单击此处查看此图的较大版本。
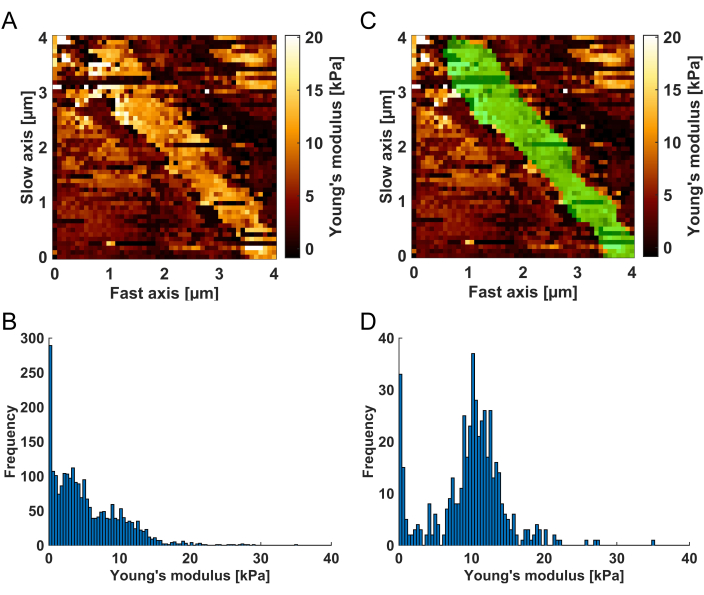
图 3:空间过滤过程的可视化。 (A) 力曲线分析得到的杨氏模量图,如结果过滤工具中所示,用于启用结果的空间过滤。BM 可识别为被较软的组织隔室包围的明亮(高杨氏模量值)条纹。(B) 对应于未过滤映射 (A) 的杨氏模量值的直方图。在 (C) 应用空间滤波器掩码(绿色区域)后,(D) 直方图仅包含来自绿色突出显示区域的杨氏模量值,该区域被标识为 BM。请注意,在直方图左侧观察到的残差软值可归因于收敛不良的拟合,这将在随后的 R² 滤波步骤中消除。 请单击此处查看此图的较大版本。
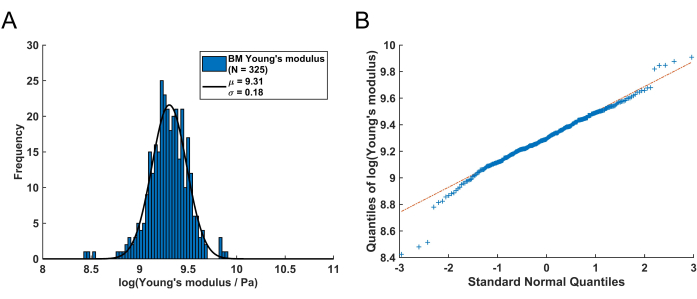
图 4:遵循此方案接收的小鼠肺 BM 的 Young 模量分布。 (A) 所得 BM Young 模量值的对数归一化直方图 (N = 325)。通过拟合正态分布(黑色实线),确定均值 μ = 9.31,标准差为 σ = 0.18。再转化后,这对应于 E BM 的杨氏模量BM = 11.05 kPa,不对称标准差区间为 [-1.82 kPa, +2.18 kPa]。(B) 对数 BM Young 模量值的分位数与标准正态分布的分位数的分位数的 QQ 图。该图表明,除了分布的左尾和右尾中的一些值外,得到的杨氏模量值主要是对数正态分布的。 请单击此处查看此图的较大版本。
讨论
由于 BM 的机械特性对各种细胞功能的影响,例如肿瘤出芽、细胞分化、细胞运动和染色质的可及性,因此引起了相当大的兴趣 53,63,64,65,66,67,68.因此,在以一致和可衡量的方式评估 BM 力学的能力方面存在未填补的差距。该协议旨在使 AFM 用户能够从野生型或转基因小鼠获得的健康或病理小鼠肺样本中确定肺 BM 的杨氏模量。基于用户友好型图形用户界面的 MATLAB 应用程序 CANTER Processing Toolbox56 有助于对 AFM 数据进行下游分析。预计该协议将对其他研究人员进行研究有用,这些研究可能会导致肺部疾病的临床诊断、管理或新治疗策略的进步。
该方案中的关键步骤包括确保在肺标本的冷冻切片过程中,组织切片在任何时候都不会解冻,以防止由于冻融循环而改变机械性能。因此,必须仅使用预冷的显微镜载玻片来收集切片,并仅在冷冻切片机的腔室内处理它们。在记录牙槽壁上的力图时,最关键的步骤是利用生成的斜率图像来确定 BM 的适当测量位置,该图像提供了探测区域刚度分布的定性概述。对获得的杨氏模量值进行空间过滤构成了协议中的关键步骤,也是 CANTER Processing Toolbox 的一个关键功能。该过滤步骤使研究人员能够通过其独特的更硬的足迹来识别位于两个较软细胞层之间的 BM,并提取 BM 的相应杨氏模量值以供进一步分析。
这种先进的分析工作流程表明,与野生型窝鼠相比,netrin-4 敲除小鼠在其肺基底膜中的 Young 模量明显高(约两倍)53。因此,本协议有助于对不同条件或组进行比较分析。
除了 AFM 之外,还有许多方法可用于检查生物材料的机械性能,包括分子水平的光学或磁镊 69,70,71,中等尺度的微压痕技术72,73 和宏观水平的受限或非受限压缩测试74,75。具体来说,压力肌层造影、拉伸试验76,77 和流变学78 已被用于评估血管样品和重组 BM 基质的 BM 刚度。然而,压力肌层造影和拉伸试验等技术只能评估完整组织或整个组织结构(例如整个血管)的机械特性,其中包括各种细胞层和结构元件。因此,当使用这些方法评估 BM 生物力学时,非 BM 的机械性能(例如细胞成分)总是叠加在 BM 结果上,因此很难分离 BM 本身的特定特性。
在该协议中,通过对力图结果进行空间过滤来提取感兴趣的特征 (BM)。这种方法的一个局限性是要求感兴趣的特征在获得的杨氏模量映射或任何其他可用的映射通道中是可识别和识别为独特的模式。例如,肺泡组织中的 BM 仅因其互连结构而被识别,与周围细胞相比,该结构表现出明显更高的杨氏模量。因此,BM 在获得的 Young 模量图中表现为连续的亮条纹。然而,其他生物结构表现出的机械特性与周围组织的机械特性没有足够大的区别,无法在地图结果中进行空间过滤。尽管该方案可以与其他方法结合使用来识别记录的力图中的结构,例如免疫荧光染色,但这种方法可能会产生难以解释的结果。之所以出现这种并发症,是因为染色程序会改变组织的机械性能。因此,只有在没有替代方法可用于识别感兴趣的特征时,才应在 AFM 测量之前对组织进行任何处理或染色。此外,进行适当的对照实验比较染色和未染色样品的生物力学特性也很重要。
该协议引入了一种基于 Young 模量对比识别目标结构的方法。因此,它可以应用于解剖 BM 类似于肺的组织,包括甲状腺、结肠和前列腺。(参见 Hartmann 等人 59 中的扩展数据图)。虽然最初是为分析 BM 力学而开发的,但该协议和 CANTER Processing Toolbox56 的通用性足以检查 AFM 力图中任何机械上不同和连接的区域(请参阅 www.github.com/CANTERhm/CANTER_Processing_Tool/wiki 的说明)。这种广泛的适用性使该工具在整个 AFM 领域都有价值,并支持更广泛的科学界对机械生物学的日益关注。
披露声明
作者声明没有利益冲突。
致谢
BH 和 HCS.感谢巴伐利亚州科学与艺术部通过巴伐利亚研究重点 Herstellung und biophysikalische Charakterisierung von dreidimensionalen Geweben (CANTER) 和巴伐利亚学术论坛 (BayWISS)-博士联盟健康研究提供的资助。数据分析软件 CANTER Processing Toolbox 的开发由德国研究基金会资助,作为研究联盟探索骨关节炎中的关节软骨和软骨下骨变性和再生的子项目 1 (CL 409/4-1/2) 的一部分 - ExCarBon (FOR2407-1/2)。BH 和 H.C.-S.感谢德国研究基金会通过主要仪器活动 GGA-HAW (INST 99/38-1) 提供的资金。这项工作得到了丹麦癌症协会 (R204-A12454 (RR)) 和德国研究基金会 (539446614 to R.R.) 的进一步支持。
材料
| Name | Company | Catalog Number | Comments |
| 1 mL Syringe | B Braun | 9166017V | Injekt-F |
| 10 mL Syringe | B Braun | 4606108V | Injekt Luer Solo |
| 15 mL Falcon tube | Sarstedt | 62.554.002 | screw cap tube |
| Cantilever - MLCT | Bruker AFM Probes | 3444 | AFM cantilever with a pyramidal tip shape |
| Cryotome blades | Leica Biosystems | 14035843496 | Low-profile disposable blades DB80LX |
| Cryotome sample holder | Leica Biosystems | 14047740044 | Specimen disc 30 |
| Cyotome | Leica Biosystems | CM1950 | Leica Cryostat |
| Direct Overlay Extension | Bruker | Software extension for the JPK SPM Software which enables to import the optical image of the inverted microscope into the Data Viewer of the SPM software. | |
| Disposable base mold | Science Services | SA62534-15 | Tissue-Tek Cryomold 15x15x5 mm |
| Double-sided tape | tesa Film | 56661-00002 | Photo Film Tape |
| Fixed-Spring Cantilever Holder | Bruker | ||
| Inverted Microscope - Leica DMi8 | Leica Microsystems | ||
| JPK Motorized Stage | Bruker | ||
| JPK NanoWizard 4XP BioScience | Bruker | ||
| JPK SPM Software | Bruker | ||
| K5 CMOS Microscope Kamera | Leica Microsystems | ||
| MATLAB | Mathworks | Version R2024a or higher | |
| MATLAB - Curve Fitting Toolbox | Mathworks | ||
| MATLAB - Image Processing Toolbox | Mathworks | ||
| MATLAB - Signal Processing Toolbox | Mathworks | ||
| MATLAB - Statistics and Machine Learning Toolbox | Mathworks | ||
| Microscope slides | Carl Roth | H869.1 | Plain microscope slides for cantilever calibration |
| Microscope slides - frosted edge | Carl Roth | H870.1 | Microscope slides with frosted edge for cryosectioning |
| Needle ø0.9 mm × 25 mm | B Braun | 4657500 | OCT injection into the lung sample |
| Needle ø0.9 mm × 70 mm | B Braun | 4665791 | Long needle to apply PBS under the AFM |
| OCT compound | Sakura Finetek | 4583 | Tissue-Tek O.C.T. Compound |
| Phosphate Buffered Saline | Bio&Sell | BS.L1825 | PBS solution without Ca2+, Mg2+, 500 mL |
| QI Advanced Imaging Extension | Bruker | Software extension for the JPK SPM Software which provides for each recorded image pixel the whole underlying force curve. | |
| Scalpel | B Braun | 5518083 | Surgical Disposable Scalpel |
| Scissors | Kaut-Bullinger | M681700 | Precise Scissors 13 cm |
| Single-sided tape | tesa Film | 57330-00000 | crystal clear tape, 33 m x 19 mm |
| Slide box | GWL Storing Systems | K50W | Slidebox for 50 slides |
| Stereo Microscope - Stemi DR1663 | Zeiss | ||
| Tweezers - Vomm SS-SA-ESD | Eleshop | ELE002121 |
参考文献
- Alsteens, D., et al. Atomic force microscopy-based characterization and design of biointerfaces. Nat Rev Mater. 2 (5), 17008 (2017).
- Aro, E., et al. Severe extracellular matrix abnormalities and chondrodysplasia in mice lacking collagen prolyl 4-hydroxylase isoenzyme II in combination with a reduced amount of isoenzyme I. J Biol Chem. 290 (27), 16964-16978 (2015).
- Domke, J., Radmacher, M. Measuring the elastic properties of thin polymer films with the atomic force microscope. Langmuir. 14 (12), 3320-3325 (1998).
- Huth, S., Sindt, S., Selhuber-Unkel, C. Automated analysis of soft hydrogel microindentation: Impact of various indentation parameters on the measurement of Young's modulus. PLoS One. 14 (8), e0220281 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nat Rev Phys. 1 (1), 41-57 (2019).
- Loparic, M., et al. Micro- and nanomechanical analysis of articular cartilage by indentation-type atomic force microscopy: validation with a gel-microfiber composite. Biophys J. 98 (11), 2731-2740 (2010).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nat Nanotechnol. 7 (11), 757-765 (2012).
- Prein, C., et al. Structural and mechanical properties of the proliferative zone of the developing murine growth plate cartilage assessed by atomic force microscopy. Matrix Biol. 50, 1-15 (2016).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophys J. 70 (1), 556-567 (1996).
- Radmacher, M., Tillamnn, R. W., Fritz, M., Gaub, H. E. From molecules to cells: imaging soft samples with the atomic force microscope. Science. 257 (5078), 1900-1905 (1992).
- Stolz, M., et al. Early detection of aging cartilage and osteoarthritis in mice and patient samples using atomic force microscopy. Nat Nanotechnol. 4, 186-192 (2009).
- Stolz, M., et al. Dynamic elastic modulus of porcine articular cartilage determined at two different levels of tissue organization by indentation-type atomic force microscopy. Biophys J. 86 (5), 3269-3283 (2004).
- Florin, E. L., Moy, V. T., Gaub, H. E. Adhesion forces between individual ligand-receptor pairs. Science. 264 (5157), 415-417 (1994).
- Hansma, P. K., et al. Tapping mode atomic force microscopy in liquids. Appl Phys Lett. 64 (13), 1738-1740 (1994).
- Moy, V. T., Florin, E. L., Gaub, H. E. Intermolecular forces and energies between ligands and receptors. Science. 266 (5183), 257-259 (1994).
- Radmacher, M., Fritz, M., Hansma, H. G., Hansma, P. K. Direct observation of enzyme activity with the atomic force microscope. Science. 265 (5178), 1577-1579 (1994).
- Radmacher, M., Fritz, M., Hansma, P. K. Imaging soft samples with the atomic force microscope: gelatin in water and propanol. Biophys J. 69 (1), 264-270 (1995).
- Rief, M., Clausen-Schaumann, H., Gaub, H. E. Sequence-dependent mechanics of single DNA molecules. Nat Struct Biol. 6 (4), 346-349 (1999).
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible unfolding of individual titin immunoglobulin domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Drake, B., et al. Imaging crystals, polymers, and processes in water with the atomic force microscope. Science. 243 (4898), 1586-1589 (1989).
- Clausen-Schaumann, H., Rief, M., Tolksdorf, C., Gaub, H. E. Mechanical stability of single DNA molecules. Biophys J. 78 (4), 1997-2007 (2000).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophys J. 78 (1), 520-535 (2000).
- Lekka, M., et al. Elasticity of normal and cancerous human bladder cells studied by scanning force microscopy. Eur Biophys J. 28 (4), 312-316 (1999).
- Weisenhorn, A. L., Khorsandi, M., Kasas, S., Gotzos, V., Butt, H. J. Deformation and height anomaly of soft surfaces studied with an AFM. Nanotechnology. 4 (2), 106 (1993).
- Rotsch, C., Jacobson, K., Radmacher, M. Dimensional and mechanical dynamics of active and stable edges in motile fibroblasts investigated by using atomic force microscopy. Proc Natl Acad Sci USA. 96 (3), 921-926 (1999).
- Goldmann, W. H., Ezzell, R. M. Viscoelasticity in wild-type and vinculin-deficient (5.51) mouse F9 embryonic carcinoma cells examined by atomic force microscopy and rheology. Exp Cell Res. 226 (1), 234-237 (1996).
- Tschaikowsky, M., et al. Hybrid fluorescence-AFM explores articular surface degeneration in early osteoarthritis across length scales. Acta Biomater. 126, 315-325 (2021).
- Tschaikowsky, M., et al. Proof-of-concept for the detection of early osteoarthritis pathology by clinically applicable endomicroscopy and quantitative AI-supported optical biopsy. Osteoarthr Cartilage. 29 (2), 269-279 (2021).
- Kinney, J. H., Balooch, M., Marshall, S. J., Marshall, G. W., Weihs, T. P. Atomic force microscope measurements of the hardness and elasticity of peritubular and intertubular human dentin. J Biomech Eng. 118 (1), 133-135 (1996).
- Lundkvist, A., et al. . Symposium on Thin Films - Stresses and Mechanical Properties VI,. 1996 MRS Spring Meeting. , 353-358 (1997).
- Tao, N. J., Lindsay, S. M., Lees, S. Measuring the microelastic properties of biological material. Biophys J. 63 (4), 1165-1169 (1992).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Eng Med Biol Mag. 16 (2), 47-57 (1997).
- Becke, T. D., et al. Pilus-1 backbone protein RrgB of Streptococcus pneumoniae binds collagen I in a force-dependent way. ACS Nano. 13 (6), 7155-7165 (2019).
- Becke, T. D., et al. Single molecule force spectroscopy reveals two-domain binding mode of Pilus-1 tip protein RrgA of Streptococcus pneumoniae to fibronectin. ACS Nano. 12 (1), 549-558 (2018).
- Pill, M. F., East, A. L. L., Marx, D., Beyer, M. K., Clausen-Schaumann, H. Mechanical activation drastically accelerates amide bond hydrolysis, matching enzyme activity. Angew Chem Int Ed Engl. 58 (29), 9787-9790 (2019).
- Schmidt, S. W., Filippov, P., Kersch, A., Beyer, M. K., Clausen-Schaumann, H. Single-molecule force-clamp experiments reveal kinetics of mechanically activated silyl ester hydrolysis. ACS Nano. 6 (2), 1314-1321 (2012).
- Docheva, D., et al. Researching into the cellular shape, volume and elasticity of mesenchymal stem cells, osteoblasts and osteosarcoma cells by atomic force microscopy. J Cell Mol Med. 12 (2), 537-552 (2008).
- Docheva, D., Padula, D., Schieker, M., Clausen-Schaumann, H. Effect of collagen I and fibronectin on the adhesion, elasticity and cytoskeletal organization of prostate cancer cells. Biochem Biophys Res Commun. 402 (2), 361-366 (2010).
- Kiderlen, S., et al. Age related changes in cell stiffness of tendon stem/progenitor cells and a rejuvenating effect of ROCK-inhibition. Biochem Biophys Res Commun. 509 (3), 839-844 (2019).
- Reuten, R., et al. Structural decoding of netrin-4 reveals a regulatory function towards mature basement membranes. Nat Commun. 7, 13515 (2016).
- Fleischhauer, L., et al. Nano-scale mechanical properties of the articular cartilage zones in a mouse model of post-traumatic osteoarthritis. Appl Sci. 12 (5), 2596 (2022).
- Alberton, P., et al. Aggrecan hypomorphism compromises articular cartilage biomechanical properties and is associated with increased incidence of spontaneous osteoarthritis. Int J Mol Sci. 20 (5), 1-16 (2019).
- Alberton, P., et al. Aggrecan is critical in maintaining the cartilage matrix biomechanics which in turn influences the correct development of the growth plate. Osteoarthr Cartilage. 27, S178-S178 (2019).
- Gronau, T., et al. Forced exercise-induced osteoarthritis is attenuated in mice lacking the small leucine-rich proteoglycan decorin. Ann Rheum Dis. 76 (2), 442-449 (2017).
- Hartmann, B., et al. Early detection of cartilage degeneration: A comparison of histology, fiber Bragg grating-based micro-indentation, and atomic force microscopy-based nano-indentation. Int J Mol Sci. 21 (19), 7384-7398 (2020).
- Rellmann, Y., et al. ER stress in ERp57 knockout knee joint chondrocytes induces osteoarthritic cartilage degradation and osteophyte formation. Int J Mol Sci. 23 (1), 182 (2021).
- Kamper, M., et al. Early changes in morphology, bone mineral density and matrix composition of vertebrae lead to disc degeneration in aged collagen IX -/- mice. Matrix Biol. 49, 132-143 (2016).
- Akhtar, R., Sherratt, M. J., Cruickshank, J. K., Derby, B. Characterizing the elastic properties of tissues. Mater Today. 14 (3), 96-105 (2011).
- Junior, C., et al. Baseline stiffness modulates the non-linear response to stretch of the extracellular matrix in pulmonary fibrosis. Int J Mol Sci. 22 (23), 12928 (2021).
- Junior, C., et al. Multi-step extracellular matrix remodelling and stiffening in the development of idiopathic pulmonary fibrosis. Int J Mol Sci. 24 (2), 1708 (2023).
- Sicard, D., Fredenburgh, L. E., Tschumperlin, D. J. Measured pulmonary arterial tissue stiffness is highly sensitive to AFM indenter dimensions. J Mech Behav Biomed Mater. 74, 118-127 (2017).
- Zemla, J., et al. AFM-based nanomechanical characterization of bronchoscopic samples in asthma patients. J Mol Recognit. 31 (12), e2752 (2018).
- Reuten, R., et al. Basement membrane stiffness determines metastases formation. Nat Mater. 20 (6), 892-903 (2021).
- Johnstone, A. H. CRC Handbook of Chemistry and Physics. J Chem Technol Biotechnol. 50 (2), 294-295 (1991).
- Rico, F., et al. Probing mechanical properties of living cells by atomic force microscopy with blunted pyramidal cantilever tips. Phys Rev E. 72 (2), 1-10 (2005).
- . . CANTER Processing Toolbox v.5.7.0. , (2022).
- Hutter, J. L. J. B. Calibration of atomic-force microscope tips. Rev Sci Instrum. 64 (7), 1868-1873 (1993).
- Schillers, H., et al. Standardized nanomechanical atomic force microscopy procedure (SNAP) for measuring soft and biological samples. Sci Rep. 7 (1), 5117 (2017).
- Hartmann, B., et al. Profiling native pulmonary basement membrane stiffness using atomic force microscopy. Nat Protoc. 19 (5), 1498-1528 (2024).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophys J. 82 (5), 2798-2810 (2002).
- Jolicoeur, P. . Introduction to Biometry. , (1999).
- Bronshtein, I. N., Semendyayev, K. A., Musiol, G., Mühlig, H. . Handbook of Mathematics. , (2015).
- Fiore, V. F., et al. Mechanics of a multilayer epithelium instruct tumour architecture and function. Nature. 585 (7825), 433-439 (2020).
- Koester, J., et al. Niche stiffening compromises hair follicle stem cell potential during ageing by reducing bivalent promoter accessibility. Nat Cell Biol. 23 (7), 771-781 (2021).
- Bedzhov, I., Zernicka-Goetz, M. Self-organizing properties of mouse pluripotent cells initiate morphogenesis upon implantation. Cell. 156 (5), 1032-1044 (2014).
- Kyprianou, C., et al. Basement membrane remodelling regulates mouse embryogenesis. Nature. 582 (7811), 253-258 (2020).
- Saraswathibhatla, A., Indana, D., Chaudhuri, O. Cell-extracellular matrix mechanotransduction in 3D. Nat Rev Mol Cell Biol. 24 (7), 495-516 (2023).
- Sherwood, D. R. Basement membrane remodeling guides cell migration and cell morphogenesis during development. Curr Opin Cell Biol. 72, 19-27 (2021).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Opt Lett. 11 (5), 288 (1986).
- Marago, O. M., Jones, P. H., Gucciardi, P. G., Volpe, G., Ferrari, A. C. Optical trapping and manipulation of nanostructures. Nat Nanotechnol. 8 (11), 807-819 (2013).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nat Methods. 5 (6), 491-505 (2008).
- Marchi, G., et al. Microindentation sensor system based on an optical fiber Bragg grating for the mechanical characterization of articular cartilage by stress-relaxation. Sens Actuators B. 252, 440-449 (2017).
- Wakitani, S., et al. Repair of large full-thickness articular cartilage defects with allograft articular chondrocytes embedded in a collagen gel. Tissue Eng. 4 (4), 429-444 (1998).
- Moutos, F. T., Freed, L. E., Guilak, F. A biomimetic three-dimensional woven composite scaffold for functional tissue engineering of cartilage. Nat Mater. 6 (2), 162-167 (2007).
- Schwarz, S., et al. Contactless vibrational analysis of transparent hydrogel structures using laser-Doppler vibrometry. Exp Mech. 60 (8), 1067-1078 (2020).
- Bhave, G., Colon, S., Ferrell, N. The sulfilimine cross-link of collagen IV contributes to kidney tubular basement membrane stiffness. Am J Physiol Renal Physiol. 313 (3), F596-F602 (2017).
- Fisher, R. F., Wakely, J. The elastic constants and ultrastructural organization of a basement membrane (lens capsule). Proc R Soc Lond B Biol Sci. 193 (1113), 335-358 (1976).
- Wisdom, K. M., et al. Covalent cross-linking of basement membrane-like matrices physically restricts invasive protrusions in breast cancer cells. Matrix Biol. 85-86, 94-111 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。