Method Article
Extraction du module de Young des membranes basales pulmonaires murines natives à partir de cartes de force dérivées de la microscopie à force atomique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole visualise comment préparer des cryosections de tissu pulmonaire murin, effectuer des expériences de carte de force de microscopie à force atomique et analyser les données pour déterminer le module de Young de la membrane basale pulmonaire murine native.
Résumé
La microscopie à force atomique (AFM) permet de caractériser les propriétés mécaniques d’un échantillon avec une résolution spatiale de plusieurs dizaines de nanomètres. Parce que les cellules de mammifères détectent et réagissent à la mécanique de leur microenvironnement immédiat, la caractérisation des propriétés biomécaniques des tissus avec une haute résolution spatiale est cruciale pour comprendre divers processus de développement, homéostatiques et pathologiques. La membrane basale (BM), une sous-structure à matrice extracellulaire (ECM) mince d’environ 100 à 400 nm, joue un rôle important dans la progression tumorale et la formation de métastases. Bien qu’il soit difficile de déterminer le module de Young d’une sous-structure ECM aussi mince, les données biomécaniques du BM fournissent de nouvelles informations fondamentales sur la façon dont le BM affecte le comportement cellulaire et, en outre, offrent un potentiel de diagnostic précieux. Ici, nous présentons un protocole visualisé pour évaluer la mécanique de la BM dans le tissu pulmonaire murin, qui est l’un des principaux organes sujets aux métastases. Nous décrivons un flux de travail efficace pour déterminer le module de Young du BM, qui est situé entre les couches de cellules endothéliales et épithéliales dans le tissu pulmonaire. Les instructions étape par étape comprennent la congélation de tissus pulmonaires murins, la cryosection et l’enregistrement de la carte de force AFM sur des coupes de tissus. De plus, nous fournissons une procédure d’analyse de données semi-automatique à l’aide de la boîte à outils de traitement CANTER, un logiciel d’analyse de données AFM convivial développé en interne. Cet outil permet le chargement automatique des cartes de force enregistrées, la conversion des courbes de force en fonction de l’extension piézoélectrique en courbes de force en fonction de l’indentation, le calcul des modules de Young et la génération des cartes de module de Young. Enfin, il montre comment déterminer et isoler les valeurs du module de Young dérivées de la BM pulmonaire à l’aide d’un outil de filtrage spatial.
Introduction
Les cartes de force AFM sont devenues une technique largement utilisée pour déterminer les propriétés nanomécaniques d’une gamme variée d’échantillons biologiques avec une résolution spatiale nanométrique et une sensibilité à la force du piconewton 1,2,3,4,5,6,7,8,9,10,11,12 . Des recherches pionnières sur l’application des mesures AFM à des échantillons biologiques dans des conditions physiologiques ont été menées par P. Hansma et ses collègues 13,14,15,16,17,18,19,20. Par la suite, des mesures de force AFM ont été utilisées pour évaluer les propriétés mécaniques des molécules individuelles21, des cellules vivantes et de leur cytosquelette 9,10,22,23,24,25,26, des tissus et des coupes de tissus 6,8,11,27,28, 29,30,31, ainsi que des hydrogels biologiques 3,4,32. Dans nos recherches, nous avons utilisé l’AFM pour examiner les propriétés mécaniques des liaisons biologiques et chimiques 18,33,34,35,36,37 et pour mesurer les caractéristiques nanomécaniques des cellules individuelles 38,39,40. De plus, les mesures du cartilage de la plaque de croissance8 ont démontré que l’architecture du collagène et le module de Young de la matrice extracellulaire (ECM) dirigent la division des chondrocytes et, par conséquent, la direction de croissance de la plaque de croissance murine au cours du développement embryonnaire. De plus, nous avons étudié les changements associés à la dégénérescence du cartilage et à l’arthrose dans le cartilage articulaire 2,41,42,43,44,45,46,47.
Au cours des dernières années, de nombreuses investigations AFM ont examiné les propriétés mécaniques des tissus pulmonaires sains et pathologiques 48,49,50,51,52. Cependant, ces recherches ont principalement porté sur la mécanique tissulaire globale, sans se concentrer sur des composants tissulaires spécifiques tels que la BM. La BM constitue une couche mince (100 nm - 400 nm chez l’homme) d’une structure ECM spécialisée qui tapisse la majorité des organes et des structures tissulaires des mammifères tels que les neurones, les muscles, les tissus adipocytaires et les vaisseaux sanguins. Nous avons découvert que les propriétés mécaniques de la BM pulmonaire murine jouent un rôle central lors de la formation des métastases, de manière à ce qu’une BM plus molle soit corrélée à une probabilité de survie plus élevée chez les patients atteints de cancer du sein et du rein53. De plus, cette étude a révélé que la protéine de matrice extracellulaire nétrine-4 sécrétée diminue le module de Young du BM par une interaction stœchiométrique 1:1 avec la laminine γ1, un composant clé présent dans presque tous les réseaux de laminines à l’intérieur des BM40.
Ici, nous présentons la visualisation de notre protocole développé pour déterminer le module de Young (E BM) de la BM pulmonaire murine mince de 100 à 200 nm. Le module de Young du BM est une mesure de sa rigidité et est défini comme le rapport entre la contrainte (force par unité de surface) et la déformation axiale (déplacement) résultante lors d’une déformation élastique linéaire54, dans ce cas, une petite compression du BM par l’indentation de la pointe AFM. Dans une expérience AFM, la force et le déplacement (profondeur d’indentation) peuvent être obtenus à partir de la déflexion en porte-à-faux AFM et de la position en porte-à-faux, et le module de Young (la relation contrainte-déformation-déformation) peut être extrait de la force résultante en fonction de la courbe d’indentation en utilisant un modèle élastique approprié, qui tient compte de la géométrie de la pointe d’indentation (dans ce cas, un modèle Hertz55 modifié). Les principales étapes sont illustrées à la figure 1. Initialement, ce protocole définit la méthodologie de préparation et d’intégration d’échantillons de tissu pulmonaire murin dans un composé OCT (voir étape 1), facilitant ainsi la procédure de cryosection ultérieure (étape 2) qui permet de déterminer la rigidité de la BM à l’aide des cartes de force AFM. La technique de cryosectionnement présentée ici, qui utilise du ruban adhésif unilatéral et double face, permet de sectionner le tissu pulmonaire sans nécessiter de fixation et sans décongélation de la section. La procédure détaillée de l’AFM pour la collecte de données appropriées pour évaluer la rigidité de la BM est décrite dans la troisième section du protocole. Dans la section suivante du protocole, nous expliquons comment les volumes de force générés peuvent être analysés automatiquement (étape 4.1) à l’aide d’un logiciel basé sur MATLAB développé en interne, le CANTER Processing Toolbox56. Dans cette étape, le module de Young est extrait de chaque courbe force-indentation enregistrée en ajustant un modèle Hertz modifié, qui est corrigé pour la géométrie du pénétrateur, dans ce cas, une pyramide à quatre côtés55. Enfin, nous décrivons aux étapes 4.2 et 4.3 comment appliquer l’outil de filtrage spatial et l’outil de filtrage R2 de la boîte à outils développée pour extraire les valeurs du module de Young spécifiques à la membrane basale (BM) à partir de l’ensemble complet des valeurs du module de Young obtenues dans la carte de force contenant tous les compartiments tissulaires de la paroi alvéolaire.
Protocole
Toutes les procédures de manipulation d’échantillons d’animaux ont été approuvées par le Conseil de l’expérimentation animale du ministère de l’Environnement et de l’Alimentation du Danemark (numéro d’autorisation 2017-15-0201-01265) conformément à la loi danoise sur le bien-être animal. Pour démontrer ce protocole, nous avons utilisé une souris mâle C57BL/6 à l’âge de 13 semaines.
ATTENTION : Dans le protocole suivant, plusieurs étapes nécessitent la manipulation d’échantillons de tissus. Portez toujours des gants adéquats et une blouse de laboratoire lorsque vous manipulez des échantillons biologiques.
1. Préparation de l’échantillon
- Placez un poumon de souris fraîchement récolté dans un moule de base jetable de 15 x 15 x 5 mm. À l’aide d’une seringue de 10 mL munie d’une aiguille de 0,9 mm, injectez soigneusement dans l’échantillon un rapport de 1:1 (volume à volume) entre le milieu à température de coupe optimale (OCT) et la solution saline tamponnée au phosphate (PBS) tout en rétractant simultanément l’aiguille. Effectuez l’injection à température ambiante tout en tenant l’échantillon solidement à l’aide d’une petite pince à épiler chirurgicale. L’injection est considérée comme terminée lorsque l’échantillon devient gonflé et qu’une quantité notable de suintement est observée.
- Remplissez l’OCT pur dans le moule jusqu’à ce que l’échantillon soit complètement immergé. Pour assurer la congélation complète de l’ensemble du poumon, conservez l’échantillon à une température de -80 °C pendant au moins 4 h.
REMARQUE : À ce stade, l’échantillon peut être stocké pendant au moins 2 ans à -80 °C. D’après les spécifications du fabricant pour l’OCT, la durée de conservation des tissus incrustés est de plus de 10 ans. Il est généralement recommandé d’envelopper les échantillons qui doivent être stockés pendant des périodes supérieures à 4 mois dans un film transparent pour éviter que l’humidité ne soit absorbée par l’échantillon.
2. Cryosectionnement d’un échantillon pulmonaire encastré
- Réglez la température de la chambre du cryotome à -17 °C, car il s’agit de la température de coupe optimale pour les échantillons de tissu pulmonaire. Réglez la température de l’unité d’étape de coupe sur -22 °C, si cette fonction est disponible. Cela permet de s’assurer que la température de coupe optimale est maintenue à la surface de l’échantillon tout au long du processus de sectionnement.
- Préparez 40 lames de microscope à l’aide d’un ruban adhésif double face qui est soigneusement placé au milieu de la lame de verre avant de rouler un tube de centrifugation de 15 ml sur le ruban pour assurer une adhérence ferme et sans bulles. Assurez-vous que la longueur du ruban adhésif double face est à peu près équivalente à la largeur du bloc OCT contenant l’échantillon. Les lames de microscope avec un bord givré permettent un étiquetage avec un crayon contenant les informations sur l’échantillon et le numéro de section.
- Insérez les lames de microscope préparées dans une boîte à lames. Par la suite, placez la boîte contenant les lames à l’intérieur de la chambre du cryotome afin de les refroidir.
REMARQUE : Le refroidissement des lames empêche l’apparition d’un cycle de gel-dégel lors du ramassage des sections, ce qui compromettrait les propriétés mécaniques de l’échantillon. - Préparez plusieurs rubans adhésifs simple face, chacun mesurant à peu près la même taille que le bloc d’échantillon congelé.
- Remplissez les deux anneaux intérieurs du porte-échantillon avec un milieu OCT. Placez l’échantillon dans le milieu OCT sur le porte-échantillon, en veillant à ce qu’il soit aussi plat que possible. Ensuite, placez le support d’échantillon pendant environ 10 minutes à l’intérieur de la chambre du cryotome jusqu’à ce que le milieu OCT se soit complètement solidifié et que l’échantillon soit solidement fixé au support.
- Installez le support portant l’échantillon sur l’unité de l’étage de coupe du cryotome. Ajustez tous les composants essentiels du cryotome, tels que l’inclinaison de l’étage de coupe et la position de la lame, selon le manuel d’utilisation de l’appareil pour établir un plan de coupe aligné parallèlement à la surface du bloc OCT.
- Entreprendre le découpage initial de l’échantillon en ajustant l’épaisseur de la coupe à 50 m et commencer la coupe jusqu’à ce qu’il atteigne l’emplacement souhaité dans l’échantillon (généralement à environ 500 m à l’intérieur de l’échantillon pulmonaire à partir de sa surface) pour le prélèvement de lames de tissu. Ajustez l’épaisseur de sectionnement du cryotome à 15 μm, ce qui correspond à l’épaisseur souhaitée des sections de tissu.
ATTENTION : Il est crucial de faire preuve de prudence lors de l’utilisation d’un instrument de coupe, tel qu’un cryotome. Pour assurer votre sécurité personnelle, respectez les consignes de sécurité du fabricant et éloignez les doigts de la lame. - Fixez un morceau de ruban adhésif unilatéral à l’échantillon en appliquant une pression ferme avec le pouce. Au préalable, assurez-vous que le pouce a été refroidi en le maintenant contre la paroi de la chambre du cryotome pendant quelques secondes.
REMARQUE : Le ruban doit être fermement appliqué sur l’échantillon, car tout manque d’adhérence peut entraîner le détachement du ruban du tissu pendant la section. - Produire une coupe de tissu de 15 μm avec le cryotome. Utilisez une brosse pour diriger la section et empêcher le ruban adhésif de se détacher de l’échantillon pendant la procédure de coupe. Il est essentiel pour une surface lisse de la section de tissu que le rythme de coupe reste lent et constant.
- La section créée à l’étape précédente repose maintenant à plat sur le bloc de lames, le ruban vers le haut. Utilisez l’une des lames de microscope réfrigérées avec le ruban adhésif double face de la boîte de lames et appuyez fermement dessus sur la section pour la ramasser. Ensuite, remettez la lame de microscope, qui porte maintenant la section, dans la boîte à lames.
REMARQUE : Lors de la manipulation de la lame de microscope et de la coupe, assurez-vous de les garder à l’intérieur de la chambre du cryotome à tout moment pour éviter que la section ne dégèle pendant la procédure de coupe. - Répétez les trois étapes précédentes jusqu’à ce que le nombre requis de sections, c’est-à-dire 40, ait été rassemblé.
- Lorsque vous avez terminé, mettez le couvercle sur la boîte à glissières et transférez-le sur de la glace carbonique dans un congélateur à -20 °C. Enfin, nettoyez le cryotome en désinfectant tous les articles utilisés et en allumant la lumière UV dans la chambre du cryotome.
REMARQUE : Le protocole peut être mis en pause à ce stade. Stockez les échantillons à une température de -20 °C jusqu’à ce que les mesures AFM soient effectuées. La durée de conservation recommandée pour les coupes et le milieu OCT ne doit pas dépasser 6 semaines. Au-delà de cette période, les sections et le milieu OCT peuvent présenter des altérations ressemblant à des brûlures de congélation en raison de leur surface importante et de leur propension à absorber l’humidité.
3. Cartes de force AFM d’une section pulmonaire
REMARQUE : Pour ce protocole, le JPK NanoWizard 4XP (AFM) et la platine motorisée de Bruker combinés au DMi8 (microscope optique inversé) de Leica ont été utilisés pour enregistrer les courbes force-déplacement des sections pulmonaires.
- Mise en place de l’AFM
- Allumez tous les appareils requis de la configuration AFM (par exemple, ordinateur, contrôleur AFM, table d’isolation des vibrations, microscope optique, etc.).
- Lancez le logiciel de contrôle AFM. Dans la vue d’ensemble Choisir l’expérience , sélectionnez le mode d’imagerie avancée QI . De plus, spécifiez l’emplacement où les données de l’expérience et les fichiers d’étalonnage doivent être stockés.
REMARQUE : Si vous ne possédez pas l’extension QI Advanced Imaging , utilisez le mode Contact Mode Force Mapping comme autre option. - Sélectionnez un porte-à-faux avec une constante de ressort adaptée au type spécifique d’échantillon observé. Pour les échantillons de tissus pulmonaires mous, il est recommandé d’utiliser le MLCT Cantilever F de Bruker avec une pointe pyramidale et une constante de ressort nominale de 0,6 N/m. Si une puce contient plusieurs porte-à-faux (comme le MLCT), retirez tous les porte-à-faux plus longs de la puce à l’aide d’une pince à épiler sous un stéréomicroscope pour vous assurer que seul le porte-à-faux utilisé pour la mesure entre en contact avec l’échantillon.
REMARQUE : D’autres porte-à-faux en contact avec l’échantillon que celui utilisé pour la mesure peuvent entraîner un mouvement indésirable de l’échantillon pendant l’expérience ou même un détachement de l’échantillon de la bande sous-jacente.
ATTENTION : Évitez d’entrer en contact avec le porte-à-faux utilisé pour la mesure avec la pince à épiler à l’étape en cours ou suivante. - Entreprenez l’installation de la puce sur le support en porte-à-faux. Insérez le support dans la tête AFM. Positionnez la tête AFM sur la platine d’échantillonnage, qui se trouve sur le microscope optique inversé. Alignez la trajectoire du levier optique comme indiqué dans le manuel d’utilisation. L’alignement correct de la trajectoire du levier optique est obtenu lorsque le faisceau laser est initialement placé à l’extrémité avant du porte-à-faux de mesure, puis que le faisceau laser réfléchi frappe le centre de la photodiode segmentée.
- Calibrage en porte-à-faux à l’aide de la méthode du bruit thermique57
REMARQUE : L’étalonnage du cantilever comprend la détermination de sa sensibilité au levier optique inversé (InvOLS) et de sa constante de ressort. L’étalonnage individuel de chaque porte-à-faux utilisé est nécessaire, car la constante nominale du ressort généralement fournie pour les porte-à-faux n’est généralement qu’une approximation approximative. Dans les expériences où les propriétés mécaniques d’un échantillon sont déterminées à partir de courbes de force enregistrées, comme celle décrite dans ce protocole, il est essentiel de déterminer directement l’InvOLS par une méthode basée sur le contact sur un substrat rigide. D’autres méthodes pour déterminer l’InvOLS, comme le dériver de la géométrie du porte-à-faux, ne peuvent fournir qu’une approximation approximative et ne conviennent pas au type d’application décrit ici. Alternativement, si des porte-à-faux sont utilisés lorsque le fournisseur fournit la constante de ressort exacte, l’InvOLS peut être déterminé par une procédure décrite par Schiller et al.58.- Placez une lame de microscope sur la platine d’échantillonnage de l’AFM. La lame de microscope sert de substrat rigide pour la détermination par contact de l’InvOLS.
- Après avoir placé la tête AFM sur la platine d’échantillonnage, abaissez la tête à l’aide des moteurs pas à pas jusqu’à ce que l’espace restant entre le support en porte-à-faux et la lame de microscope ne soit plus que d’environ 1 à 2 mm. Appliquez du PBS sur le côté du support en porte-à-faux, en lui permettant de s’écouler vers le bas et de former un ménisque fluide dans l’espace entre le support et la lame de microscope à l’aide d’une seringue de 1 ml avec une longue aiguille (0,9 mm x 70 mm).
REMARQUE : Assurez-vous que le même liquide, tel qu’un tampon ou un support, qui est utilisé pour les mesures de force et de volume de l’échantillon est également utilisé pour le processus d’étalonnage. - Pour effectuer un étalonnage basé sur le contact dans le logiciel, accédez à la page Acquérir des données et sélectionnez l’option Vue avancée. Accédez au gestionnaire d’étalonnage à l’aide du bouton de menu burger situé dans le coin supérieur droit. Dans cette interface, sélectionnez Basée sur le contact comme méthode et choisissez le porte-à-faux MLCT F dans le menu déroulant comme nom du porte-à-faux. De plus, réglez le point de consigne sur 1 V et le nombre de balayages sur 10 dans les champs de saisie correspondants.
- Sur la page Acquérir des données , dans le panneau de commande de gauche, entrez un point de consigne de 1 V pour la procédure d’approche automatique. Cliquez sur le bouton Flèche bleue pointant vers le bas situé dans le coin supérieur gauche de l’interface utilisateur pour démarrer l’approche automatique. Lorsque l’approche est terminée et que le cantilever est en contact avec la lame du microscope, cliquez sur le bouton Calibrer dans la fenêtre du Gestionnaire d’étalonnage .
REMARQUE : L’InvOLS et la constante de ressort en porte-à-faux sont alors toutes deux déterminées automatiquement en enregistrant 10 courbes de force ainsi que la densité spectrale de puissance thermique (PSD), qui est utilisée pour déterminer la constante de ressort avec la méthode du bruit thermique57. Une fois la procédure d’étalonnage automatique terminée, vérifiez visuellement que la courbe ajustée décrit correctement le mode normal (premier pic) de la DSP enregistrée du porte-à-faux et que la constante de ressort déterminée est du même ordre de grandeur que la constante nominale de ressort donnée par le fabricant du porte-à-faux. Si ce n’est pas le cas, la plage de montage doit être ajustée manuellement pour s’assurer que l’ajustement converge vers et représente le premier pic du spectre de puissance thermique. - Une fois l’étalonnage terminé, fermez la fenêtre Gestionnaire d’étalonnage . Après la fermeture du gestionnaire d’étalonnage, un fichier d’étalonnage (fichier *.tnd ascii) dans le répertoire présélectionné (voir étape 3.1.2) est généré, contenant les résultats d’étalonnage déterminés, qui sont ensuite nécessaires pour l’analyse des données. Documentez les valeurs d’étalonnage pour référence future pendant le processus d’analyse des données, par exemple dans le cahier de laboratoire.
ATTENTION : À partir de cette étape, évitez de changer la position du laser sur le cantilever, de régler le miroir dans la tête AFM ou de déplacer le support de cantilever. La modification de ces éléments aura un impact sur l’InvOLS et rendra les valeurs de calibrage invalides. Pour compenser la dérive thermique, ajustez la position de la photodiode segmentée de la tête AFM.
- Analyse d’une paroi alvéolaire sur une section pulmonaire
- Récupérez la lame de l’échantillon pour l’évaluer dans le congélateur. Laisser décongeler l’échantillon à température ambiante pendant environ 1 minute jusqu’à ce que tout le milieu OCT soit devenu clair. Par la suite, ajoutez quelques gouttelettes de PBS dans la section du tissu pulmonaire pour le réhydrater.
- Placez la lame de microscope portant la section de tissu pulmonaire sur la platine de l’échantillon. Réinstallez la tête AFM sur la platine d’échantillonnage et abaissez la tête jusqu’à ce que le porte-à-faux soit positionné à environ 1 mm au-dessus de l’échantillon. Ajoutez un peu de PBS supplémentaire sur le côté du support en porte-à-faux à l’aide d’une seringue de 1 mL combinée à une aiguille de 0,9 mm x 70 mm jusqu’à ce qu’un ménisque liquide se forme entre la section de tissu et le porte-porte-porte-à-faux.
- Accédez au gestionnaire de paramètres en cliquant sur le bouton avec l’icône de la clé dans le coin supérieur droit de l’interface utilisateur. Dans le Gestionnaire de paramètres, ajustez les paramètres généraux suivants. Dans la section Paramètres d’approche , réglez la hauteur cible sur 4 μm. Ensuite, dans la section Paramètres du mode actuel , passez aux paramètres de retour avancés et réglez le multiplicateur sur 1. Enfin, dans la sous-section Paramètres de force , qui se trouve également dans la section Paramètres du mode actuel , sélectionnez l’option Piézo rétracté dans le menu déroulant Mode à la fin .
- Utilisez le microscope optique inversé pour identifier et naviguer vers une paroi alvéolaire intacte. Un mur intact se caractérise dans l’image en champ clair par des bords sombres et lisses. (voir Figure 2A-B).
REMARQUE : L’importation de l’image en fond clair dans le logiciel de contrôle AFM, lorsqu’elle est disponible, peut aider à naviguer le cantilever vers une paroi alvéolaire particulière et à capturer avec précision une carte de force ou de QI au-dessus de celle-ci. Dans le logiciel JPK SPM, cette fonction est appelée Superposition directe. - Définissez les paramètres des courbes force-indentation dans le panneau de commande de gauche en fonction de l’AFM utilisé, du cantilever utilisé et de l’échantillon observé. Pour le NanoWizard 4XP et le MLCT Cantilever F de Bruker, nous recommandons les paramètres suivants : Consigne = 5 nN ; Longueur z = 8 μm ; Vitesse z = 300 μm/s.
REMARQUE : La longueur z recommandée de 8 μm est plutôt grande. Ceci est attribué aux caractéristiques du tissu pulmonaire. Étant donné que le tissu pulmonaire est généralement assez mou, la profondeur d’indentation peut atteindre jusqu’à 4 μm à certains endroits, même avec un point de consigne relativement modeste de 5 nN. Afin de garantir que la ligne de base, où la pointe n’est pas en contact avec l’échantillon, représente au moins environ la moitié de la courbe force-indentation, il est recommandé de fixer la longueur totale de la courbe à 8 μm. Cela simplifiera considérablement l’analyse de la courbe par la suite. Pour d’autres bio-AFM, ces paramètres devraient également s’appliquer, bien que la nomenclature de certains paramètres, tels que le point de consigne, qui détermine la force d’indentation maximale, puisse varier d’un fabricant à l’autre. Si le système AFM n’est pas en mesure d’atteindre la vitesse verticale élevée indiquée ici, il est nécessaire de diminuer la vitesse z selon les recommandations de votre fabricant d’AFM.
ATTENTION : Afin d’assurer la comparabilité des résultats, il est essentiel d’utiliser des vitesses d’approche z identiques dans toutes les expériences. - Définissez l’emplacement et la taille de la carte de force de vue d’ensemble initiale, en vous assurant qu’elle englobe toute la paroi alvéolaire, s’étendant d’un côté de l’air à l’autre. Pour les échantillons de tissu pulmonaire murin, une taille comprise entre 20 μm x 20 μm et 30 μm x 30 μm est généralement suffisante. Quelle que soit la taille spécifique de la carte, réglez le nombre de pixels (qui correspondent au nombre de courbes de force enregistrées) sur 50 x 50. Cela conduit à la génération de 2 500 courbes d’indentation de force pour chaque carte, ce qui fournit la résolution nécessaire pour identifier le BM dans le canal de pente de la carte de force. (voir Figure 2C-D).
- Après avoir enregistré la carte d’ensemble, examinez la hauteur et le canal de pente de la carte de force. Si le canal de hauteur indique que la paroi alvéolaire est intacte (voir Figure 2D) et s’il est possible de distinguer le BM dans le canal de pente sous la forme d’une ligne claire clairement définie (voir Figure 2C,E) située entre deux couches de l’épithélium et de l’endothélium, capturez une carte de force plus ciblée d’une taille de 3 μm x 3 μm ou 4 μm x 4 μm sur le BM. Gardez les pixels à 50 x 50 courbes.
REMARQUE : Cette procédure permet d’obtenir une résolution de carte plus élevée et d’enregistrer un plus grand nombre de valeurs de module de Young, en particulier pour le BM (voir Figure 2E-F). De plus, cela permet une différenciation plus précise entre le BM et les tissus environnants, améliorant ainsi la précision du processus de filtrage spatial ultérieur. Si les canaux de la carte d’ensemble montrent une paroi effondrée ou rompue, des artefacts dus au fait que le porte-à-faux touche l’échantillon non seulement avec la pointe, ou aucun BM identifiable dans le canal de pente, naviguez vers une autre paroi sur la section du tissu jusqu’à ce qu’une paroi avec une évaluation du BM sans artefact puisse être identifiée.
Répétez les étapes précédentes (3.3.6 et 3.3.7) sur d’autres sections du même échantillon pulmonaire jusqu’à ce que le nombre souhaité de parois alvéolaires ait été évalué. Au moins quatre parois sont recommandées pour chaque échantillon pulmonaire afin d’obtenir des résultats robustes59. - Une fois la mesure terminée, nettoyez tout l’équipement et les matériaux qui sont entrés en contact avec l’échantillon et le PBS. Si l’échantillon n’est plus nécessaire, jetez-le dans un conteneur à déchets en verre autoclavable. Sauvegardez toutes les données enregistrées et éteignez le logiciel de contrôle AFM et l’ordinateur. Enfin, éteignez tous les appareils électriques utilisés.
REMARQUE : À ce stade, le protocole peut être mis en pause. Les données expérimentales sont sauvegardées et peuvent être analysées ultérieurement.
4. Analyse des données
- Analyse des courbes force-indentation
REMARQUE : Un manuel détaillé de l’application d’analyse de la courbe de force peut être trouvé ici : https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/2a.-Force-Curve-Analysis.- Lancez la CANTER Processing Toolbox56 dans MATLAB et ouvrez l’application Force Curve Analysis .
REMARQUE : La boîte à outils peut être téléchargée à partir du référentiel GitHub en suivant ce lien : https://github.com/CANTERhm/CANTER_Processing_Tool. Le fichier README.md, qui comprend un guide complet sur l’installation du logiciel et son lancement à l’aide de MATLAB, est disponible dans le référentiel GitHub. De plus amples informations sur chaque application incluse dans la boîte à outils peuvent être trouvées dans le wiki correspondant à l’https://github.com/CANTERhm/CANTER_Processing_Tool/wiki. - Chargez la carte de force haute résolution (3 μm x 3 μm ou 4 μm x 4 μm) en cliquant sur le bouton Sélectionner un fichier , en naviguant jusqu’à l’emplacement où la carte de force est enregistrée, en double-cliquant dessus, puis en cliquant sur le bouton Charger les données dans l’interface utilisateur de l’application.
- Une fenêtre contextuelle s’affiche, demandant les valeurs d’étalonnage (InvOLS et constante de ressort) qui ont été déterminées (voir étape 3.2) pour le porte-à-faux utilisé pour enregistrer la carte de force sélectionnée à l’étape de protocole précédente. Saisissez les valeurs d’étalonnage dans les champs d’édition respectifs. S’ils n’ont pas été documentés à l’étape 3.2.4, reportez-vous au fichier d’étalonnage généré automatiquement, qui contient également les valeurs d’étalonnage. Conservez l’option par défaut oui pour la question de savoir si la séparation pointe-échantillon doit être calculée. Ensuite, cliquez sur le bouton Soumettre pour continuer.
- Une deuxième fenêtre contextuelle apparaît, permettant de sélectionner la géométrie du pénétrateur et le coefficient de Poisson pour l’échantillon en retrait. Pour les porte-à-faux MLCT utilisés, sélectionnez Pyramide à quatre côtés comme géométrie de pointe et entrez 17,5° pour le demi-angle par rapport au bord (les deux sont les valeurs par défaut). Utilisez le coefficient de Poisson par défaut de 0,5, qui représente un matériau incompressible. Pour poursuivre la procédure de chargement de la carte QI, cliquez sur le bouton Soumettre .
REMARQUE : Au cours de la procédure de chargement, les courbes de déflexion-déplacement enregistrées (données brutes de l’AFM) sont converties en courbes de force-indentation basées sur les valeurs d’étalonnage fournies. Pour une compréhension complète des différentes étapes de transformation et des équations associées, reportez-vous aux informations supplémentaires de Hartmann et al.59. - Une fois le processus de chargement terminé, la courbe de force initiale de la carte de force s’affichera à l’écran. Assurez-vous que le mode de correction de la ligne de base Décalage + Inclinaison est sélectionné. Ce mode détecte automatiquement la ligne de base de la courbe d’induction de force et corrige l’inclinaison et le décalage vertical pour qu’ils soient nuls. De plus, sélectionnez via le modèle Hertz comme algorithme pour l’outil de recherche de points de contact et utilisez une valeur de 20 % dans le champ d’édition désigné pour cette option. Cet algorithme est particulièrement bien adapté à la détection du point de contact dans les courbes force-indentation qui ont été enregistrées sur des échantillons de tissus mous. Enfin, réglez la profondeur d’ajustement dans le champ d’édition correspondant sur 1,5 μm.
REMARQUE : Pour s’assurer que la rigidité de la lame de microscope n’a pas d’impact sur le module de Young résultant, il est recommandé de régler la profondeur d’ajustement maximale à une valeur qui ne dépasse pas 10 % de l’épaisseur de la section de tissu60. Pour plus de détails sur les algorithmes de correction, voir les informations supplémentaires de Hartmann et al.59.
ATTENTION : Utilisez la même profondeur d’ajustement tout au long des expériences pour garantir que les résultats sont comparables. - Pour appliquer l’ajustement du modèle Hertz modifié à toutes les courbes force-indentation de la carte QI, cliquez sur le bouton Conserver et appliquer à tous . Une boîte de dialogue s’ouvre pour vous demander si chaque courbe de force doit être affichée pendant l’analyse. En n’affichant pas les courbes, la vitesse de traitement est légèrement accélérée.
- Une fois la dernière analyse de la courbe de force terminée, une fenêtre s’affiche, demandant si les résultats de l’ajustement doivent être enregistrés. Pour enregistrer les résultats dans les deux. tsv (valeurs séparées par des tabulations) et .xlsx (Excel), cliquez sur Oui et entrez un nom pour les fichiers de résultats. De plus, un fichier texte avec l’extension de fichier *.meta_data est enregistré, qui contient tous les détails concernant l’analyse des courbes, tels que les valeurs d’étalonnage utilisées, le modèle d’ajustement choisi, l’algorithme de détection de point de contact sélectionné, la plage d’ajustement et d’autres informations pertinentes.
- Fermez l’application Analyse de la courbe de force en cliquant sur l’icône en forme de croix dans le coin supérieur droit de la fenêtre principale.
- Lancez la CANTER Processing Toolbox56 dans MATLAB et ouvrez l’application Force Curve Analysis .
- Filtrage spatial des résultats de la carte QI
REMARQUE : L’étape de filtrage spatial garantit que seules les valeurs de module de Young des cartes QI provenant du BM sont prises en compte pour toute analyse ultérieure. Un manuel détaillé de l’outil de filtrage des résultats peut être trouvé ici : https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/Result-Filtering-Tool.- Sélectionnez l’outil de filtrage des résultats dans la liste des applications de la fenêtre de sélection d’applications de la boîte à outils de traitement CANTER et démarrez l’application en cliquant sur le bouton Démarrer l’application .
- Pour charger les résultats de l’ajustement, cliquez sur Ouvrir dans la barre de menu supérieure de l’interface utilisateur de l’outil de filtrage des résultats . Dans la fenêtre contextuelle suivante, recherchez le premier bouton Définir situé dans l’onglet JPK Maps et choisissez le fichier .tsv qui contient les résultats de l’analyse de la courbe de force (reportez-vous à l’étape 4.1.6). Cliquez sur le deuxième bouton Set et localisez le fichier de mappage QI qui correspond au fichier .tsv déjà sélectionné. Cliquez ensuite sur Soumettre pour charger les données de la carte et les résultats de l’analyse de la courbe de force.
- Sélectionnez l’option Emodul dans le menu déroulant supérieur du canal affiché pour afficher les résultats obtenus du module de Young sous forme d’image de carte. De plus, choisissez l’option Emodul (si ce n’est pas déjà fait) dans le menu déroulant Canal de données situé sous le graphique de l’histogramme pour visualiser la distribution des valeurs du module de Young chargées de la carte QI.
- Cliquez sur le bouton fléché de flux de manipulation , situé au centre de l’interface utilisateur, et assurez-vous qu’il pointe vers le côté droit. Réglez le bouton bascule du filtre d’image sur la position On dans le panneau de filtre situé au-dessus des axes de l’histogramme. Sélectionnez l’option Main levée dans le menu déroulant Filtrer la géométrie , puis cliquez sur le bouton Ajouter .
- Dans l’image du canal supérieur, dessinez le masque de filtre en encerclant le BM tout en maintenant la touche gauche de la souris enfoncée. Le BM est identifiable dans la carte du module de Young comme une structure brillante (valeurs de module de Young élevées) par rapport aux tissus mous voisins (voir Figure 3A). Lorsque vous avez terminé de dessiner le masque, relâchez la touche gauche de la souris et double-cliquez sur le masque à appliquer à la carte. Le graphique de l’histogramme de droite ne montre plus que les valeurs du module de Young pour les régions de la carte qui sont surlignées en vert (la couleur par défaut ; voir la figure 3C-D).
- Pour créer un nouveau fichier de résultats .tsv contenant uniquement les valeurs de module de Young masquées, cliquez dans la barre de menu supérieure de l’interface utilisateur sur Enregistrer > Enregistrer l’histogramme > Enregistrer les données. Vous pouvez également cliquer avec le bouton droit de la souris sur une zone vide des axes de traçage de l’histogramme et sélectionner Enregistrer les données dans le menu contextuel.
- Cliquez sur Sélectionner tout dans la fenêtre contextuelle demandant quels résultats écrire dans le fichier .tsv. Ensuite, confirmez la sélection en cliquant sur le bouton OK . Ensuite, entrez un nom pour le fichier .tsv dans la boîte de dialogue d’enregistrement, choisissez l’emplacement d’enregistrement souhaité, et enfin cliquez sur OK pour enregistrer les résultats filtrés.
- Fermez l’outil de filtrage des résultats en cliquant sur l’icône en forme de croix dans le coin supérieur droit de l’application.
- R2 filtrage des résultats
REMARQUE : Cette étape du protocole garantit que seuls les résultats du module de Young sont pris en compte lorsque le modèle Hertz modifié a montré une bonne concordance avec la courbe force-indentation à laquelle il a été ajusté. Un manuel détaillé de l’outil de filtrage R² peut être trouvé ici : https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/R%C2%B2-Filtering-Tool.- Démarrez l’outil de filtrage R² en le sélectionnant dans la liste des applications de la fenêtre de sélection d’applications de la boîte à outils de traitement CANTER et en cliquant sur Démarrer l’application.
- Naviguez jusqu’à la barre de menu supérieure de l’interface utilisateur et cliquez sur Ouvrir le fichier .tsv. Dans la boîte de dialogue de chargement de fichier suivante, recherchez le fichier .tsv contenant les résultats du module de Young du BM enregistré à l’étape précédente 4.2.5 et sélectionnez-le en double-cliquant.
- Une boîte de dialogue s’affiche et répertorie les colonnes de données contenues dans le fichier .tsv. Sélectionnez Emodul comme colonne contenant les données de module de Young. Ensuite, dans une boîte de dialogue de liste distincte demandant les données R², choisissez rsquare_fit. Une fois la procédure de chargement terminée, les données de module de Young chargées sont affichées sous la forme d’un histogramme en haut à gauche de l’écran, tandis que la distribution des données R² est représentée dans le graphique en haut à droite.
- Entrez 0,96 dans le champ d’édition situé au centre de l’interface utilisateur graphique. Cliquez sur le bouton Filtrer les données pour appliquer le filtre R² en utilisant la valeur saisie. Cette action ne conservera que les résultats qui ont une valeur R² de 0,96 ou plus. Les distributions filtrées des résultats du module de Young et les valeurs R² correspondantes sont affichées dans la moitié inférieure de l’interface utilisateur.
- Sélectionnez Enregistrer les données filtrées dans la barre de menu supérieure pour créer un nouveau fichier .tsv qui inclut uniquement les résultats qui répondent au critère de filtre R² défini de > 0,96. Les données sauvegardées peuvent maintenant être affichées sous forme d’histogramme, par exemple, à l’aide de l’outil de traçage d’histogramme, qui visualise la distribution du module de Young pour la BM pulmonaire sondée (voir Figure 4A).
Résultats
Après l’identification et la mesure d’une paroi alvéolaire intacte, le résultat principal de ce protocole est les valeurs filtrées du module de Young du BM. Il est important de noter qu’il n’est pas possible de trouver une paroi alvéolaire appropriée pour la mesure de l’AFM dans chaque section de tissu. Dans nos observations expérimentales, environ 75 % des cryosections dérivées d’échantillons de poumons murins présentaient une paroi alvéolaire quantifiable. Pour déterminer avec précision la distribution spatiale du module de Young dans une BM pulmonaire, nous vous conseillons d’utiliser des cartes de force couvrant soit 3 μm x 3 μm ou 4 μm x 4 μm. Cette approche garantit une résolution suffisante pour l’examen des BM pulmonaires de souris, qui mesurent généralement de 100 à 200 nm d’épaisseur. Par conséquent, l’approche expérimentale consiste à capturer des courbes de force de 50 x 50 pour chaque carte de force, ce qui donne un total de 2 500 courbes de force. Dans la carte des forces, les courbes de force sont régulièrement espacées. Cela implique qu’en plus du module de Young du BM, les structures tissulaires entourant le BM sont sondées même dans les petites cartes de force. Par conséquent, afin de déterminer avec précision et sélection le module de Young exclusivement de la BM pulmonaire, nous avons mis en œuvre un processus de filtrage en deux étapes dans ce protocole.
L’étape initiale de filtrage du filtrage spatial dépendant de l’opérateur sélectionne les résultats d’ajustement de la courbe de force provenant de la BM pulmonaire murine, représentant environ 10 % à 25 % des valeurs totales des résultats contenues dans la carte de force analysée. Après ce filtrage spatial initial, environ 250 à 625 résultats d’ajustement sont conservés pour une analyse plus approfondie. L’étape de filtrage suivante permet de s’assurer que seuls les résultats liés à la BM provenant des courbes de force décrites de manière adéquate par le modèle Hertz modifié sont inclus. Par conséquent, le coefficient de détermination (R2) de l’ajustement du modèle Hertz modifié est utilisé comme critère de filtre. Notre expérience empirique suggère que le maintien des résultats avec des valeurs R2 supérieures à 0,96 convient aux résultats d’ajustement obtenus en adhérant à ce protocole. Il est important de noter que la valeur R2 est significativement influencée par la qualité (douceur) et la longueur de la ligne de base de la courbe de force, la détermination du point de contact et la convergence du modèle ajusté. Après l’application des deux procédures de filtrage, il reste généralement environ 100 à 500 valeurs de module de Young pour la BM d’une seule paroi alvéolaire.
La distribution de ces valeurs restantes du module de Young à partir du BM peut être visualisée à l’aide d’un histogramme (Figure 4A). Il est important de noter que les valeurs résultantes du module de Young de la BM pulmonaire suivent une distribution log-normale, qui est couramment observée pour les variables aléatoires qui ne peuvent atteindre que des valeurs positives61. Ceci est visualisé par le graphique QQ de la figure 4B. Par conséquent, une distribution normale (gaussienne) peut être ajustée à la distribution des valeurs du module de Young (E) transformée en logarithme. Ici, le logarithme népérien (ln) a été utilisé pour transformer les valeurs E . La position de crête de la distribution (μ) et de l’écart-type (σ) a été extraite de cet ajustement et ensuite retransformée pour obtenir le module de Young représentatif, noté EBM, à l’aide de l’équation suivante :
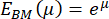
Notez qu’à la suite de la retransformation, l’intervalle de l’écart-type n’est plus symétrique autour de la valeur de crête. Les arêtes négatives (σ-) et positives (σ+) de l’intervalle type peuvent être calculées à l’aide des expressions suivantes :
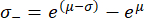
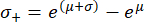
Ces équations sont une conséquence du fait que la fonction exponentielle est la fonction inverse du logarithme népérien62.
Après retransformation de la valeur de crête μ = 9,31 et de l’écart-type σ = 0,18 déterminés à partir de l’histogramme de la figure 4A, la valeur représentative du module de Young est EBM = 11,05 kPa avec un intervalle d’écart-type de [-1,82 kPa, +2,18 kPa]. Alternativement, une distribution log-normale peut être ajustée à l’histogramme des valeurs non transformées du module de Young (E) pour déterminer les paramètres caractéristiques de la distribution.
Il est recommandé d’analyser au moins quatre parois par souris pour obtenir un résultat représentatif robuste, comme nous l’avons montré en détail dans Hartmann et al.59. Pour déterminer le module de Young représentatif EBM pour une souris, les valeurs transformées en logarithme des quatre parois ont été tracées dans un histogramme et une μ combinés et σ ont été déterminées en ajustant une distribution normale. Par la suite, μ et σ sont retransformés comme décrit précédemment pour récupérer EBM et l’intervalle d’écart type pour la souris sujete.
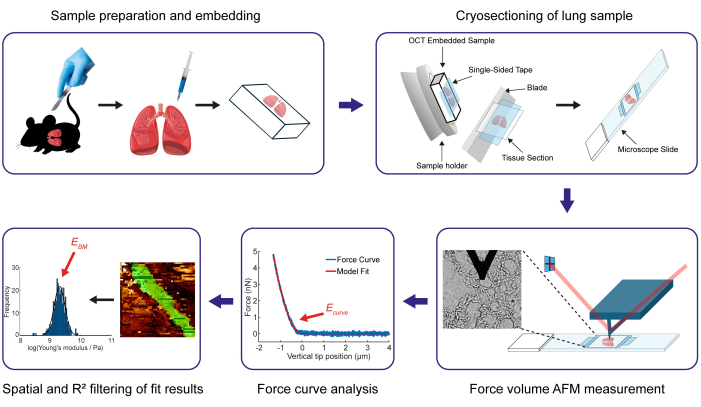
Figure 1 : Vue d’ensemble des principales étapes du protocole pour déterminer le module de Young d’une membrane basale pulmonaire murine. La première étape consiste à obtenir et à préparer des poumons murins, puis à les intégrer. Par conséquent, le poumon est injecté avec une combinaison de milieu OCT et de PBS, puis complètement immergé dans le composé OCT. Ensuite, des coupes de tissu pulmonaire de 15 m d’épaisseur sont obtenues par cryosectionnement avec un cryotome. Dans ce protocole, nous détaillons le processus d’utilisation de rubans adhésifs unilatéraux et bilatéraux pour fixer les échantillons en place pendant le processus de découpe. Par la suite, les valeurs du module de Young du BM sont déterminées par des mesures de la carte de force AFM sur les sections de tissus. Après l’achèvement du processus d’analyse de la courbe de force, un filtrage spatial et R² est appliqué pour isoler les résultats du module de Young de la structure d’intérêt, en particulier le BM (indiqué en vert sur la carte de force et quantifié dans la distribution du module de Young transformée en logarithme). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Une expérience AFM exemplaire de carte de force menée sur une coupe de tissu pulmonaire murin. (A) Image de synthèse en microscopie à fond clair d’une section de tissu pulmonaire murin, permettant d’identifier des parois alvéolaires intactes appropriées situées entre deux alvéoles. Sur le côté droit de l’image, le porte-à-faux F du MLCT de forme triangulaire est visible. (B) Vue agrandie d’une paroi sélectionnée à l’intérieur de la section du tissu. (C) Canal de pente et (D) de hauteur d’une carte d’ensemble de 15 x 15 μm contenant 50 x 50 courbes de force de la paroi alvéolaire. Dans le canal de pente, le BM peut être identifié par une ligne brillante (flèche blanche), indiquant une structure de rigidité accrue à l’intérieur du mur. Le canal en hauteur confirme l’intégrité du mur, ne montrant aucun signe de rupture. Suite à l’identification du BM dans la carte d’ensemble 15 x 15 μm, une carte plus petite de 4 x 4 μm (E : canal de pente, F : canal de hauteur) avec des courbes de force de 50 x 50 est enregistrée. Barres d’échelle : (A) 200 μm et (B-D) 25 μm. Les gammes des échelles de couleurs des images AFM montrées sont les suivantes : (C) 2,36 - 9,29 nN/μm, (D) 0 - 3,38 μm, (E) 2,36 - 9,29 nN/μm (image d’ensemble) et 2,42 - 8,37 nN/μm (image plus petite), (F) 0 - 3,38 μm (image d’ensemble) et 0 - 1,99 μm (image plus petite), de sombre à clair. Des échelles de couleurs linéaires ont été utilisées pour toutes les images AFM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
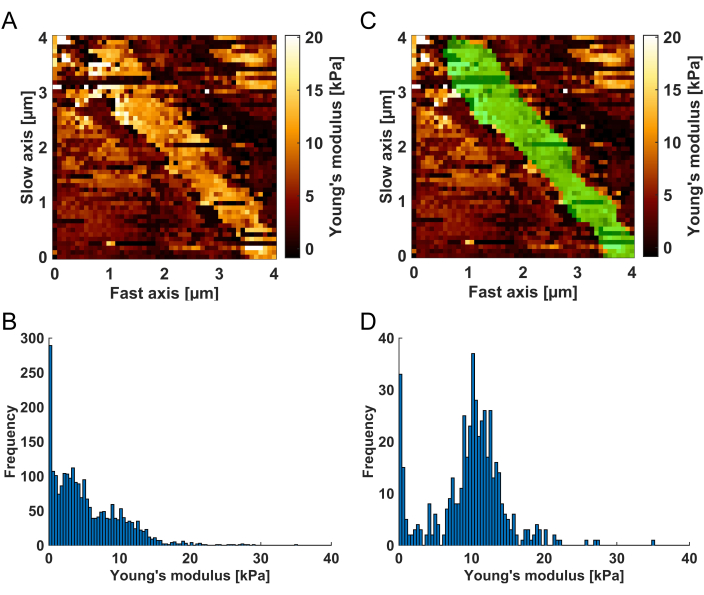
Figure 3 : Visualisation de la procédure de filtrage spatial. (A) La carte de module de Young résultant de l’analyse de la courbe de force, telle qu’elle est montrée dans l’outil de filtrage des résultats pour permettre le filtrage spatial des résultats. Le BM est identifiable par une bande brillante (valeurs de module de Young élevées) entourée de compartiments de tissus plus mous. (B) Histogramme des valeurs du module de Young correspondant à l’application non filtrée (A). Après l’application (C) du masque de filtre spatial (zone verte), (D) l’histogramme comprend exclusivement les valeurs du module de Young de la région en surbrillance verte, qui a été identifiée comme la BM. Notez que les valeurs douces résiduelles observables sur le côté gauche de l’histogramme peuvent être attribuées à des ajustements peu convergents, qui seront éliminés lors de l’étape de filtrage R² suivante. Veuillez cliquer ici pour voir une version agrandie de cette figure.
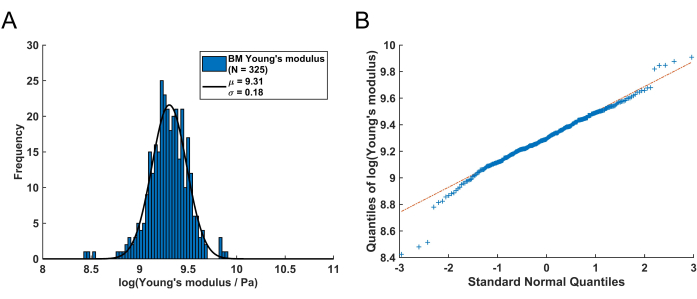
Figure 4 : Distribution du module de Young d’une BM pulmonaire murine reçue en suivant ce protocole. (A) Histogramme normalisé logarithmiquement des valeurs du module de Young de la BM résultante (N = 325). En ajustant une distribution normale (ligne continue noire), la valeur moyenne μ = 9,31 avec un écart type de σ = 0,18 est déterminée. Après retransformation, cela correspond à un module de Young de la BM de EBM = 11,05 kPa avec un intervalle d’écart-type asymétrique de [-1,82 kPa, +2,18 kPa]. (B) QQ Tracé des quantiles des valeurs du module de Young BM logarithmisé en fonction des quantiles d’une distribution normale standard. Ce graphique montre qu’à l’exception de certaines valeurs dans les extrémités gauche et droite de la distribution, les valeurs du module de Young résultantes sont principalement distribuées log-normales. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Les caractéristiques mécaniques des BM ont suscité un intérêt considérable en raison de leur influence sur diverses fonctions cellulaires, telles que le bourgeonnement tumoral, la différenciation cellulaire, le mouvement cellulaire et l’accessibilité de la chromatine 53,63,64,65,66,67,68 . Par conséquent, il existe une lacune non comblée dans la capacité d’évaluer la mécanique de la BM de manière cohérente et mesurable. Ce protocole vise à permettre aux utilisateurs de l’AFM de déterminer le module de Young des BM pulmonaires à partir d’échantillons de poumons murins sains ou pathologiques obtenus sur des souris de type sauvage ou génétiquement modifiées. L’interface utilisateur graphique conviviale basée sur l’application MATLAB CANTER Processing Toolbox56 facilite l’analyse en aval des données AFM. Ce protocole devrait être utile à d’autres chercheurs menant des études qui pourraient mener à des progrès dans le diagnostic clinique, la prise en charge ou de nouvelles stratégies de traitement des troubles pulmonaires.
Les étapes critiques du protocole consistent à s’assurer que pendant la cryosection de l’échantillon pulmonaire, les coupes de tissu ne dégèlent à aucun moment afin d’éviter les altérations des propriétés mécaniques dues aux cycles de gel-dégel. Par conséquent, il est essentiel d’utiliser uniquement des lames de microscope pré-refroidies pour collecter les coupes et de ne les manipuler qu’à l’intérieur de la chambre du cryotome. L’étape la plus cruciale lors de l’enregistrement de cartes de force sur une paroi alvéolaire consiste à utiliser l’image de pente générée, qui fournit un aperçu qualitatif de la distribution de rigidité de la zone sondée, afin d’identifier un emplacement de mesure approprié du BM. Le filtrage spatial des valeurs de module de Young obtenues constitue une étape critique du protocole et constitue une caractéristique clé de la boîte à outils de traitement CANTER. Cette étape de filtrage permet au chercheur d’identifier le BM, qui est situé entre deux couches cellulaires plus molles, grâce à son empreinte plus rigide distinctive, et d’extraire les valeurs correspondantes du module de Young du BM pour une analyse plus approfondie.
Ce flux d’analyse avancé a révélé que les souris knock-out netrin-4 présentaient un module de Young considérablement (environ deux fois) plus élevé dans leur membrane basale pulmonaire par rapport aux souris de litière de type sauvage53. Ainsi, le présent protocole facilite l’analyse comparative de diverses affections ou groupes.
Outre l’AFM, il existe de nombreuses méthodologies pour examiner les propriétés mécaniques des biomatériaux, englobant les pinces optiques ou magnétiques 69,70,71 au niveau moléculaire, les techniques de micro-indentation 72,73 à l’échelle intermédiaire et les essais de compression confinés ou nonconfinés 74,75 au niveau macroscopique. Plus précisément, la myographie de pression, les essais de traction76,77 et la rhéologie78 ont été utilisés pour évaluer la rigidité de la BM d’échantillons vasculaires et de matrices de BM reconstituées. Cependant, des techniques telles que la myographie de pression et les essais de traction ne peuvent évaluer que les caractéristiques mécaniques de tissus complets ou de structures tissulaires entières, telles que des vaisseaux sanguins entiers, qui comprennent diverses couches cellulaires et éléments structurels. Par conséquent, lors de l’utilisation de ces méthodes pour évaluer la biomécanique des BM, les propriétés mécaniques des non-BM, telles que les composants cellulaires, sont toujours superposées aux résultats des BM, ce qui rend difficile l’isolement des propriétés spécifiques de la BM elle-même.
Dans ce protocole, la caractéristique d’intérêt (BM) est extraite par filtrage spatial des résultats de la carte de force. Une limite de cette approche est l’exigence que la caractéristique d’intérêt soit discernable et identifiable comme un motif distinct dans les cartes de module de Young obtenues ou dans tout autre canal de carte disponible. Par exemple, le BM dans le tissu alvéolaire est identifiable uniquement en raison de sa structure interconnectée, qui présente un module de Young nettement plus élevé par rapport aux cellules environnantes. Par conséquent, le BM se manifeste sous la forme d’une bande brillante continue dans les cartes de module de Young obtenues. Néanmoins, d’autres structures biologiques présentent des propriétés mécaniques qui ne sont pas suffisamment distinctes de celles des tissus environnants pour permettre un filtrage spatial dans les résultats de la carte. Bien que le protocole puisse être combiné à d’autres méthodes pour identifier les structures dans les cartes de force enregistrées, telles que la coloration par immunofluorescence, cette approche peut donner des résultats difficiles à interpréter. Cette complication peut survenir parce que les procédures de coloration peuvent altérer les propriétés mécaniques des tissus. Par conséquent, tout traitement ou coloration du tissu avant les mesures AFM ne doit être effectué que lorsqu’aucune autre méthode n’est disponible pour identifier la caractéristique d’intérêt. De plus, il est important d’effectuer des expériences de contrôle appropriées comparant les propriétés biomécaniques des échantillons colorés et non colorés.
Le protocole introduit une méthode d’identification des structures cibles basée sur le contraste de module de Young. Par conséquent, il peut être appliqué sur des tissus ayant une BM anatomique similaire à celle du poumon, y compris la glande thyroïde, le côlon et la prostate. (voir la figure des données étendues dans Hartmann et al.59). Bien qu’initialement développés pour l’analyse de la mécanique BM, ce protocole et la CANTER Processing Toolbox56 sont suffisamment polyvalents pour examiner n’importe quelle région mécaniquement distincte et connectée dans les cartes de force AFM (voir les instructions à la www.github.com/CANTERhm/CANTER_Processing_Tool/wiki). Cette large applicabilité rend cet outil précieux dans le domaine de l’AFM et soutient l’intérêt croissant de la communauté scientifique au sens large pour la mécanobiologie.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
B.H., et H.C.-S. reconnaissance du financement du ministère des Sciences et des Arts de l’État de Bavière par le biais du Bavarian Research Focus Herstellung und biophysikalische Charakterisierung von dreidimensionalen Geweben (CANTER) et du Bavarian Academic Forum (BayWISS)-Doctoral Consortium Health Research. Le développement du logiciel d’analyse de données CANTER Processing Toolbox a été financé par la Fondation allemande pour la recherche dans le cadre du sous-projet 1 (CL 409/4-1/2) du consortium de recherche Exploring articular cartilage and underchondral bone degeneration and regeneration in osteoarthritis - ExCarBon (FOR2407-1/2). B.H. et H.C.-S. reconnaissance du financement de la Fondation allemande pour la recherche dans le cadre de la grande campagne d’instrumentation GGA-HAW (INST 99/38-1). Ce travail a également été soutenu par la Société danoise du cancer (R204-A12454 (R.R.)) et la Fondation allemande pour la recherche (539446614 à R.R.).
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL Syringe | B Braun | 9166017V | Injekt-F |
| 10 mL Syringe | B Braun | 4606108V | Injekt Luer Solo |
| 15 mL Falcon tube | Sarstedt | 62.554.002 | screw cap tube |
| Cantilever - MLCT | Bruker AFM Probes | 3444 | AFM cantilever with a pyramidal tip shape |
| Cryotome blades | Leica Biosystems | 14035843496 | Low-profile disposable blades DB80LX |
| Cryotome sample holder | Leica Biosystems | 14047740044 | Specimen disc 30 |
| Cyotome | Leica Biosystems | CM1950 | Leica Cryostat |
| Direct Overlay Extension | Bruker | Software extension for the JPK SPM Software which enables to import the optical image of the inverted microscope into the Data Viewer of the SPM software. | |
| Disposable base mold | Science Services | SA62534-15 | Tissue-Tek Cryomold 15x15x5 mm |
| Double-sided tape | tesa Film | 56661-00002 | Photo Film Tape |
| Fixed-Spring Cantilever Holder | Bruker | ||
| Inverted Microscope - Leica DMi8 | Leica Microsystems | ||
| JPK Motorized Stage | Bruker | ||
| JPK NanoWizard 4XP BioScience | Bruker | ||
| JPK SPM Software | Bruker | ||
| K5 CMOS Microscope Kamera | Leica Microsystems | ||
| MATLAB | Mathworks | Version R2024a or higher | |
| MATLAB - Curve Fitting Toolbox | Mathworks | ||
| MATLAB - Image Processing Toolbox | Mathworks | ||
| MATLAB - Signal Processing Toolbox | Mathworks | ||
| MATLAB - Statistics and Machine Learning Toolbox | Mathworks | ||
| Microscope slides | Carl Roth | H869.1 | Plain microscope slides for cantilever calibration |
| Microscope slides - frosted edge | Carl Roth | H870.1 | Microscope slides with frosted edge for cryosectioning |
| Needle ø0.9 mm × 25 mm | B Braun | 4657500 | OCT injection into the lung sample |
| Needle ø0.9 mm × 70 mm | B Braun | 4665791 | Long needle to apply PBS under the AFM |
| OCT compound | Sakura Finetek | 4583 | Tissue-Tek O.C.T. Compound |
| Phosphate Buffered Saline | Bio&Sell | BS.L1825 | PBS solution without Ca2+, Mg2+, 500 mL |
| QI Advanced Imaging Extension | Bruker | Software extension for the JPK SPM Software which provides for each recorded image pixel the whole underlying force curve. | |
| Scalpel | B Braun | 5518083 | Surgical Disposable Scalpel |
| Scissors | Kaut-Bullinger | M681700 | Precise Scissors 13 cm |
| Single-sided tape | tesa Film | 57330-00000 | crystal clear tape, 33 m x 19 mm |
| Slide box | GWL Storing Systems | K50W | Slidebox for 50 slides |
| Stereo Microscope - Stemi DR1663 | Zeiss | ||
| Tweezers - Vomm SS-SA-ESD | Eleshop | ELE002121 |
Références
- Alsteens, D., et al. Atomic force microscopy-based characterization and design of biointerfaces. Nat Rev Mater. 2 (5), 17008 (2017).
- Aro, E., et al. Severe extracellular matrix abnormalities and chondrodysplasia in mice lacking collagen prolyl 4-hydroxylase isoenzyme II in combination with a reduced amount of isoenzyme I. J Biol Chem. 290 (27), 16964-16978 (2015).
- Domke, J., Radmacher, M. Measuring the elastic properties of thin polymer films with the atomic force microscope. Langmuir. 14 (12), 3320-3325 (1998).
- Huth, S., Sindt, S., Selhuber-Unkel, C. Automated analysis of soft hydrogel microindentation: Impact of various indentation parameters on the measurement of Young's modulus. PLoS One. 14 (8), e0220281 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nat Rev Phys. 1 (1), 41-57 (2019).
- Loparic, M., et al. Micro- and nanomechanical analysis of articular cartilage by indentation-type atomic force microscopy: validation with a gel-microfiber composite. Biophys J. 98 (11), 2731-2740 (2010).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nat Nanotechnol. 7 (11), 757-765 (2012).
- Prein, C., et al. Structural and mechanical properties of the proliferative zone of the developing murine growth plate cartilage assessed by atomic force microscopy. Matrix Biol. 50, 1-15 (2016).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophys J. 70 (1), 556-567 (1996).
- Radmacher, M., Tillamnn, R. W., Fritz, M., Gaub, H. E. From molecules to cells: imaging soft samples with the atomic force microscope. Science. 257 (5078), 1900-1905 (1992).
- Stolz, M., et al. Early detection of aging cartilage and osteoarthritis in mice and patient samples using atomic force microscopy. Nat Nanotechnol. 4, 186-192 (2009).
- Stolz, M., et al. Dynamic elastic modulus of porcine articular cartilage determined at two different levels of tissue organization by indentation-type atomic force microscopy. Biophys J. 86 (5), 3269-3283 (2004).
- Florin, E. L., Moy, V. T., Gaub, H. E. Adhesion forces between individual ligand-receptor pairs. Science. 264 (5157), 415-417 (1994).
- Hansma, P. K., et al. Tapping mode atomic force microscopy in liquids. Appl Phys Lett. 64 (13), 1738-1740 (1994).
- Moy, V. T., Florin, E. L., Gaub, H. E. Intermolecular forces and energies between ligands and receptors. Science. 266 (5183), 257-259 (1994).
- Radmacher, M., Fritz, M., Hansma, H. G., Hansma, P. K. Direct observation of enzyme activity with the atomic force microscope. Science. 265 (5178), 1577-1579 (1994).
- Radmacher, M., Fritz, M., Hansma, P. K. Imaging soft samples with the atomic force microscope: gelatin in water and propanol. Biophys J. 69 (1), 264-270 (1995).
- Rief, M., Clausen-Schaumann, H., Gaub, H. E. Sequence-dependent mechanics of single DNA molecules. Nat Struct Biol. 6 (4), 346-349 (1999).
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible unfolding of individual titin immunoglobulin domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Drake, B., et al. Imaging crystals, polymers, and processes in water with the atomic force microscope. Science. 243 (4898), 1586-1589 (1989).
- Clausen-Schaumann, H., Rief, M., Tolksdorf, C., Gaub, H. E. Mechanical stability of single DNA molecules. Biophys J. 78 (4), 1997-2007 (2000).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophys J. 78 (1), 520-535 (2000).
- Lekka, M., et al. Elasticity of normal and cancerous human bladder cells studied by scanning force microscopy. Eur Biophys J. 28 (4), 312-316 (1999).
- Weisenhorn, A. L., Khorsandi, M., Kasas, S., Gotzos, V., Butt, H. J. Deformation and height anomaly of soft surfaces studied with an AFM. Nanotechnology. 4 (2), 106 (1993).
- Rotsch, C., Jacobson, K., Radmacher, M. Dimensional and mechanical dynamics of active and stable edges in motile fibroblasts investigated by using atomic force microscopy. Proc Natl Acad Sci USA. 96 (3), 921-926 (1999).
- Goldmann, W. H., Ezzell, R. M. Viscoelasticity in wild-type and vinculin-deficient (5.51) mouse F9 embryonic carcinoma cells examined by atomic force microscopy and rheology. Exp Cell Res. 226 (1), 234-237 (1996).
- Tschaikowsky, M., et al. Hybrid fluorescence-AFM explores articular surface degeneration in early osteoarthritis across length scales. Acta Biomater. 126, 315-325 (2021).
- Tschaikowsky, M., et al. Proof-of-concept for the detection of early osteoarthritis pathology by clinically applicable endomicroscopy and quantitative AI-supported optical biopsy. Osteoarthr Cartilage. 29 (2), 269-279 (2021).
- Kinney, J. H., Balooch, M., Marshall, S. J., Marshall, G. W., Weihs, T. P. Atomic force microscope measurements of the hardness and elasticity of peritubular and intertubular human dentin. J Biomech Eng. 118 (1), 133-135 (1996).
- Lundkvist, A., et al. . Symposium on Thin Films - Stresses and Mechanical Properties VI,. 1996 MRS Spring Meeting. , 353-358 (1997).
- Tao, N. J., Lindsay, S. M., Lees, S. Measuring the microelastic properties of biological material. Biophys J. 63 (4), 1165-1169 (1992).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Eng Med Biol Mag. 16 (2), 47-57 (1997).
- Becke, T. D., et al. Pilus-1 backbone protein RrgB of Streptococcus pneumoniae binds collagen I in a force-dependent way. ACS Nano. 13 (6), 7155-7165 (2019).
- Becke, T. D., et al. Single molecule force spectroscopy reveals two-domain binding mode of Pilus-1 tip protein RrgA of Streptococcus pneumoniae to fibronectin. ACS Nano. 12 (1), 549-558 (2018).
- Pill, M. F., East, A. L. L., Marx, D., Beyer, M. K., Clausen-Schaumann, H. Mechanical activation drastically accelerates amide bond hydrolysis, matching enzyme activity. Angew Chem Int Ed Engl. 58 (29), 9787-9790 (2019).
- Schmidt, S. W., Filippov, P., Kersch, A., Beyer, M. K., Clausen-Schaumann, H. Single-molecule force-clamp experiments reveal kinetics of mechanically activated silyl ester hydrolysis. ACS Nano. 6 (2), 1314-1321 (2012).
- Docheva, D., et al. Researching into the cellular shape, volume and elasticity of mesenchymal stem cells, osteoblasts and osteosarcoma cells by atomic force microscopy. J Cell Mol Med. 12 (2), 537-552 (2008).
- Docheva, D., Padula, D., Schieker, M., Clausen-Schaumann, H. Effect of collagen I and fibronectin on the adhesion, elasticity and cytoskeletal organization of prostate cancer cells. Biochem Biophys Res Commun. 402 (2), 361-366 (2010).
- Kiderlen, S., et al. Age related changes in cell stiffness of tendon stem/progenitor cells and a rejuvenating effect of ROCK-inhibition. Biochem Biophys Res Commun. 509 (3), 839-844 (2019).
- Reuten, R., et al. Structural decoding of netrin-4 reveals a regulatory function towards mature basement membranes. Nat Commun. 7, 13515 (2016).
- Fleischhauer, L., et al. Nano-scale mechanical properties of the articular cartilage zones in a mouse model of post-traumatic osteoarthritis. Appl Sci. 12 (5), 2596 (2022).
- Alberton, P., et al. Aggrecan hypomorphism compromises articular cartilage biomechanical properties and is associated with increased incidence of spontaneous osteoarthritis. Int J Mol Sci. 20 (5), 1-16 (2019).
- Alberton, P., et al. Aggrecan is critical in maintaining the cartilage matrix biomechanics which in turn influences the correct development of the growth plate. Osteoarthr Cartilage. 27, S178-S178 (2019).
- Gronau, T., et al. Forced exercise-induced osteoarthritis is attenuated in mice lacking the small leucine-rich proteoglycan decorin. Ann Rheum Dis. 76 (2), 442-449 (2017).
- Hartmann, B., et al. Early detection of cartilage degeneration: A comparison of histology, fiber Bragg grating-based micro-indentation, and atomic force microscopy-based nano-indentation. Int J Mol Sci. 21 (19), 7384-7398 (2020).
- Rellmann, Y., et al. ER stress in ERp57 knockout knee joint chondrocytes induces osteoarthritic cartilage degradation and osteophyte formation. Int J Mol Sci. 23 (1), 182 (2021).
- Kamper, M., et al. Early changes in morphology, bone mineral density and matrix composition of vertebrae lead to disc degeneration in aged collagen IX -/- mice. Matrix Biol. 49, 132-143 (2016).
- Akhtar, R., Sherratt, M. J., Cruickshank, J. K., Derby, B. Characterizing the elastic properties of tissues. Mater Today. 14 (3), 96-105 (2011).
- Junior, C., et al. Baseline stiffness modulates the non-linear response to stretch of the extracellular matrix in pulmonary fibrosis. Int J Mol Sci. 22 (23), 12928 (2021).
- Junior, C., et al. Multi-step extracellular matrix remodelling and stiffening in the development of idiopathic pulmonary fibrosis. Int J Mol Sci. 24 (2), 1708 (2023).
- Sicard, D., Fredenburgh, L. E., Tschumperlin, D. J. Measured pulmonary arterial tissue stiffness is highly sensitive to AFM indenter dimensions. J Mech Behav Biomed Mater. 74, 118-127 (2017).
- Zemla, J., et al. AFM-based nanomechanical characterization of bronchoscopic samples in asthma patients. J Mol Recognit. 31 (12), e2752 (2018).
- Reuten, R., et al. Basement membrane stiffness determines metastases formation. Nat Mater. 20 (6), 892-903 (2021).
- Johnstone, A. H. CRC Handbook of Chemistry and Physics. J Chem Technol Biotechnol. 50 (2), 294-295 (1991).
- Rico, F., et al. Probing mechanical properties of living cells by atomic force microscopy with blunted pyramidal cantilever tips. Phys Rev E. 72 (2), 1-10 (2005).
- . . CANTER Processing Toolbox v.5.7.0. , (2022).
- Hutter, J. L. J. B. Calibration of atomic-force microscope tips. Rev Sci Instrum. 64 (7), 1868-1873 (1993).
- Schillers, H., et al. Standardized nanomechanical atomic force microscopy procedure (SNAP) for measuring soft and biological samples. Sci Rep. 7 (1), 5117 (2017).
- Hartmann, B., et al. Profiling native pulmonary basement membrane stiffness using atomic force microscopy. Nat Protoc. 19 (5), 1498-1528 (2024).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophys J. 82 (5), 2798-2810 (2002).
- Jolicoeur, P. . Introduction to Biometry. , (1999).
- Bronshtein, I. N., Semendyayev, K. A., Musiol, G., Mühlig, H. . Handbook of Mathematics. , (2015).
- Fiore, V. F., et al. Mechanics of a multilayer epithelium instruct tumour architecture and function. Nature. 585 (7825), 433-439 (2020).
- Koester, J., et al. Niche stiffening compromises hair follicle stem cell potential during ageing by reducing bivalent promoter accessibility. Nat Cell Biol. 23 (7), 771-781 (2021).
- Bedzhov, I., Zernicka-Goetz, M. Self-organizing properties of mouse pluripotent cells initiate morphogenesis upon implantation. Cell. 156 (5), 1032-1044 (2014).
- Kyprianou, C., et al. Basement membrane remodelling regulates mouse embryogenesis. Nature. 582 (7811), 253-258 (2020).
- Saraswathibhatla, A., Indana, D., Chaudhuri, O. Cell-extracellular matrix mechanotransduction in 3D. Nat Rev Mol Cell Biol. 24 (7), 495-516 (2023).
- Sherwood, D. R. Basement membrane remodeling guides cell migration and cell morphogenesis during development. Curr Opin Cell Biol. 72, 19-27 (2021).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Opt Lett. 11 (5), 288 (1986).
- Marago, O. M., Jones, P. H., Gucciardi, P. G., Volpe, G., Ferrari, A. C. Optical trapping and manipulation of nanostructures. Nat Nanotechnol. 8 (11), 807-819 (2013).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nat Methods. 5 (6), 491-505 (2008).
- Marchi, G., et al. Microindentation sensor system based on an optical fiber Bragg grating for the mechanical characterization of articular cartilage by stress-relaxation. Sens Actuators B. 252, 440-449 (2017).
- Wakitani, S., et al. Repair of large full-thickness articular cartilage defects with allograft articular chondrocytes embedded in a collagen gel. Tissue Eng. 4 (4), 429-444 (1998).
- Moutos, F. T., Freed, L. E., Guilak, F. A biomimetic three-dimensional woven composite scaffold for functional tissue engineering of cartilage. Nat Mater. 6 (2), 162-167 (2007).
- Schwarz, S., et al. Contactless vibrational analysis of transparent hydrogel structures using laser-Doppler vibrometry. Exp Mech. 60 (8), 1067-1078 (2020).
- Bhave, G., Colon, S., Ferrell, N. The sulfilimine cross-link of collagen IV contributes to kidney tubular basement membrane stiffness. Am J Physiol Renal Physiol. 313 (3), F596-F602 (2017).
- Fisher, R. F., Wakely, J. The elastic constants and ultrastructural organization of a basement membrane (lens capsule). Proc R Soc Lond B Biol Sci. 193 (1113), 335-358 (1976).
- Wisdom, K. M., et al. Covalent cross-linking of basement membrane-like matrices physically restricts invasive protrusions in breast cancer cells. Matrix Biol. 85-86, 94-111 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon