Method Article
Extracción del módulo de Young de las membranas basales pulmonares murinos nativas de los mapas de fuerza derivados de la microscopía de fuerza atómica
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo visualiza cómo preparar criosecciones de tejido pulmonar murino, realizar experimentos de mapas de fuerza de microscopía de fuerza atómica y analizar los datos para determinar el módulo de Young de la membrana basal pulmonar murina nativa.
Resumen
La microscopía de fuerza atómica (AFM) permite la caracterización de las propiedades mecánicas de una muestra con una resolución espacial de varias decenas de nanómetros. Debido a que las células de mamíferos detectan y reaccionan a la mecánica de su microambiente inmediato, la caracterización de las propiedades biomecánicas de los tejidos con alta resolución espacial es crucial para comprender varios procesos de desarrollo, homeostáticos y patológicos. La membrana basal (MO), una subestructura de matriz extracelular (MEC) de aproximadamente 100 a 400 nm de grosor, desempeña un papel importante en la progresión tumoral y la formación de metástasis. Aunque determinar el módulo de Young de una subestructura de ECM tan delgada es un desafío, los datos biomecánicos del BM proporcionan nuevos conocimientos fundamentales sobre cómo el BM afecta el comportamiento celular y, además, ofrecen un valioso potencial de diagnóstico. Aquí, presentamos un protocolo visualizado para evaluar la mecánica de la MO en el tejido pulmonar murino, que es uno de los principales órganos propensos a la metástasis. Describimos un flujo de trabajo eficiente para determinar el módulo de Young de la MO, que se encuentra entre las capas de células endoteliales y epiteliales en el tejido pulmonar. Las instrucciones paso a paso comprenden la congelación del tejido pulmonar murino, la criosección y el registro del mapa de fuerza de AFM en secciones de tejido. Además, proporcionamos un procedimiento de análisis de datos semiautomático utilizando CANTER Processing Toolbox, un software de análisis de datos AFM fácil de usar desarrollado internamente. Esta herramienta permite la carga automática de mapas de fuerza registrados, la conversión de curvas de fuerza frente a piezoextensión en curvas de fuerza frente a sangría, el cálculo de los módulos de Young y la generación de mapas de módulos de Young. Finalmente, se muestra cómo determinar y aislar los valores del módulo de Young derivados de la MO pulmonar mediante el uso de una herramienta de filtrado espacial.
Introducción
Los mapas de fuerza AFM han surgido como una técnica ampliamente utilizada para determinar las propiedades nanomecánicas de una amplia gama de muestras biológicas con resolución espacial nanométrica y sensibilidad a la fuerza de piconewton 1,2,3,4,5,6,7,8,9,10,11,12 . La investigación pionera sobre la aplicación de las mediciones de AFM a muestras biológicas en condiciones fisiológicas fue realizada por P. Hansma y colaboradores 13,14,15,16,17,18,19,20. Posteriormente, se emplearon mediciones de fuerza AFM para evaluar las propiedades mecánicas de moléculas individuales21, células vivas y su citoesqueleto 9,10,22,23,24,25,26, tejidos y secciones de tejido 6,8,11,27,28, 29,30,31, así como hidrogeles biológicos 3,4,32. En nuestra investigación, utilizamos AFM para examinar las propiedades mecánicas de los enlaces biológicos y químicos 18,33,34,35,36,37 y para medir las características nanomecánicas de las células individuales 38,39,40. Además, las mediciones del cartílago de la placa de crecimiento8 demostraron que la arquitectura del colágeno y el módulo de Young de la matriz extracelular (MEC) dirigen la división de los condrocitos y, en consecuencia, la dirección de crecimiento de la placa de crecimiento murina durante el desarrollo embrionario. Además, investigamos los cambios asociados con la degeneración del cartílago y la osteoartritis en el cartílago articular 2,41,42,43,44,45,46,47.
En los últimos años, numerosas investigaciones sobre la MFA han examinado las propiedades mecánicas del tejido pulmonar tanto sano como patológico 48,49,50,51,52. Sin embargo, estas investigaciones examinaron principalmente la mecánica general de los tejidos, sin centrarse en componentes específicos de los tejidos, como la MO. El BM constituye una capa delgada (100 nm - 400 nm en humanos) de una estructura especializada en ECM que recubre la mayoría de los órganos y estructuras de tejidos de mamíferos, como neuronas, músculos, tejidos de adipocitos y vasos sanguíneos. Descubrimos que las propiedades mecánicas del MO pulmonar murino juegan un papel fundamental durante la formación de metástasis, de manera que un MO más suave se correlaciona con una mayor probabilidad de supervivencia en pacientes con cáncer de mama y riñón53. Además, esta investigación reveló que la proteína de la matriz extracelular secretada netrina-4 disminuye el módulo de Young del BM a través de una interacción estequiométrica 1:1 con la laminina γ1, un componente clave presente en casi todas las redes de laminina dentro de BMs40.
Aquí, presentamos la visualización de nuestro protocolo desarrollado para determinar el módulo de Young (EBM) de la BM pulmonar murina delgada de 100 a 200 nm. El módulo de Young del BM es una medida de su rigidez y se define como la relación entre la tensión (fuerza por unidad de área) frente a la deformación axial resultante (desplazamiento) durante una deformación elástica lineal54, en este caso, una pequeña compresión del BM por la punta AFM de indentación. En un experimento AFM, la fuerza y el desplazamiento (profundidad de indentación) se pueden obtener a partir de la deflexión del voladizo AFM y la posición del voladizo, y el módulo de Young (la relación tensión-deformación) se puede extraer de la curva de fuerza resultante frente a la indentación utilizando un modelo elástico adecuado, que tiene en cuenta la geometría de la punta de indentación (en este caso un modelo de Hertz55 modificado). Los pasos principales se muestran en la Figura 1. Inicialmente, este protocolo delinea la metodología para preparar e incluir muestras de tejido pulmonar murino en el compuesto OCT (ver paso 1), facilitando así el procedimiento de criosección posterior (paso 2) que permite la determinación de la rigidez del BM utilizando mapas de fuerza AFM. La técnica de criosección presentada aquí, que utiliza cinta adhesiva de un lado y de dos caras, permite el corte del tejido pulmonar sin necesidad de fijación y sin descongelación de la sección. El procedimiento detallado de AFM para recopilar datos adecuados para evaluar la rigidez del BM se describe en la tercera sección del protocolo. En la siguiente sección del protocolo, explicamos cómo se pueden analizar automáticamente los volúmenes de fuerza generados (paso 4.1) utilizando un software basado en MATLAB desarrollado internamente, CANTER Processing Toolbox56. En este paso, el módulo de Young se extrae de cada curva de fuerza-indentación registrada ajustando un modelo de Hertz modificado, que se corrige para la geometría del indentador, en este caso, una pirámide de cuatro lados55. Finalmente, describimos en los pasos 4.2 y 4.3 cómo aplicar la Herramienta de Filtrado Espacial y la Herramienta de Filtrado R2 de la caja de herramientas desarrollada para extraer los valores del módulo de Young específicos de la membrana basal (BM) del conjunto completo de valores del módulo de Young obtenidos en el mapa de fuerza que contiene todos los compartimentos de tejido de la pared alveolar.
Protocolo
Todos los procedimientos para el manejo de muestras de animales fueron aprobados por el Consejo de Experimentación Animal del Ministerio de Medio Ambiente y Alimentación de Dinamarca (número de permiso 2017-15-0201-01265) de acuerdo con la Ley Danesa de Bienestar Animal. Para demostrar este protocolo, utilizamos un ratón macho C57BL/6 a las 13 semanas de edad.
PRECAUCIÓN: En el siguiente protocolo, varios pasos requieren el manejo de muestras de tejido. Use siempre guantes adecuados y una bata de laboratorio mientras manipula muestras biológicas.
1. Preparación de la muestra
- Coloque un pulmón de ratón recién cosechado en un molde base desechable de 15 x 15 x 5 mm. Utilizando una jeringa de 10 mL con una aguja de 0,9 mm, inyecte cuidadosamente una relación de 1:1 (volumen a volumen) de medio de temperatura de corte óptima (OCT) a solución salina tamponada con fosfato (PBS) en la muestra mientras retrae simultáneamente la aguja. Realice la inyección a temperatura ambiente mientras sostiene la muestra de forma segura con una pinza quirúrgica pequeña. La inyección se considera completa cuando la muestra se hincha y se observa una cantidad notable de supuración.
- Llene OCT puro en el molde hasta que la muestra esté completamente sumergida. Para garantizar la congelación completa de todo el pulmón, almacene la muestra a una temperatura de -80 °C durante al menos 4 h.
NOTA: En este punto, la muestra puede almacenarse durante al menos 2 años a -80 °C. Según las especificaciones del fabricante para la OCT, el tiempo de almacenamiento del tejido incrustado es de más de 10 años. Por lo general, se recomienda envolver las muestras que deben almacenarse durante períodos superiores a 4 meses en una película transparente para evitar que la humedad sea absorbida por la muestra.
2. Crioseccionamiento de muestras pulmonares incrustadas
- Regule la temperatura de la cámara del criotomo a -17 °C, ya que esta es la temperatura de corte óptima para las muestras de tejido pulmonar. Ajuste la temperatura de la unidad de etapa de corte a -22 °C, si esta función está disponible. Esto sirve para garantizar que se mantenga la temperatura de corte óptima en la superficie de la muestra durante todo el proceso de seccionado.
- Prepare 40 portaobjetos de microscopio con cinta adhesiva de doble cara que se coloca cuidadosamente en el centro del portaobjetos de vidrio antes de enrollar un tubo de centrífuga de 15 ml sobre la cinta para garantizar una adhesión firme y sin burbujas. Asegúrese de que la longitud de la cinta adhesiva de doble cara sea aproximadamente equivalente a la anchura del bloque OCT que contiene la muestra. Los portaobjetos de microscopio con borde esmerilado permiten etiquetar con un lápiz que contiene la información de la muestra y el número de sección.
- Inserte los portaobjetos del microscopio preparados en una caja de portaobjetos. A continuación, coloque la caja que contiene los portaobjetos dentro de la cámara criotomímica para enfriarlos.
NOTA: El enfriamiento de los portaobjetos evita que se produzca un ciclo de congelación-descongelación al recoger las secciones, lo que comprometería las propiedades mecánicas de la muestra. - Prepare varias cintas adhesivas de una sola cara, cada una de las cuales mida aproximadamente el mismo tamaño que el bloque de muestra congelado.
- Llene los dos anillos interiores del portamuestras con medio OCT. Coloque la muestra dentro del medio OCT en el portamuestras, asegurándose de que esté lo más nivelada posible. A continuación, coloque el soporte de muestras durante aproximadamente 10 minutos dentro de la cámara de criotomo hasta que el medio OCT se haya solidificado completamente y la muestra esté firmemente asegurada al soporte.
- Instale el soporte que lleva la muestra en la unidad de etapa de corte del criótomo. Ajuste todos los componentes esenciales del criótomo, como la inclinación de la etapa de corte y la posición de la cuchilla, de acuerdo con el manual del usuario del dispositivo para establecer un plano de corte que se alinee paralelo a la superficie del bloque OCT.
- Realice el recorte inicial de la muestra ajustando el grosor de la sección a 50 μm y comience a cortar hasta alcanzar la ubicación deseada dentro de la muestra (generalmente alrededor de 500 μm dentro de la muestra pulmonar desde su superficie) para la recolección de portaobjetos de tejido. Ajuste el grosor de corte del criotomo a 15 μm, que corresponde al grosor deseado de las secciones de tejido.
PRECAUCIÓN: Es crucial tener cuidado al emplear un instrumento de corte, como un criótomo. Para garantizar la seguridad personal, siga las pautas de seguridad del fabricante y mantenga los dedos alejados de la hoja. - Fije un trozo de cinta adhesiva de un solo lado a la muestra aplicando una presión firme con el pulgar. De antemano, asegúrese de que el pulgar se haya enfriado sosteniéndolo contra la pared de la cámara del criotomo durante unos segundos.
NOTA: La cinta debe aplicarse firmemente a la muestra, ya que cualquier falta de adherencia puede resultar en el desprendimiento de la cinta del tejido durante el corte. - Producir una sección de tejido de 15 μm con el criótomo. Utilice un cepillo para dirigir la sección y evitar que la cinta adhesiva se desprenda de la muestra durante el procedimiento de corte. Es esencial para una superficie lisa de la sección de tejido que el ritmo de corte permanezca lento y constante.
- La sección creada en el paso anterior ahora queda plana sobre el bloque de cuchillas con la cinta hacia arriba. Utilice uno de los portaobjetos de microscopio refrigerados con la cinta adhesiva de doble cara de la caja de portaobjetos y presiónelo firmemente sobre la sección para recogerlo. A continuación, vuelva a colocar el portaobjetos del microscopio, que ahora lleva la sección, en la caja de portaobjetos.
NOTA: Cuando manipule el portaobjetos del microscopio y la sección, asegúrese de mantenerlos dentro de la cámara de criotomo en todo momento para evitar que la sección se descongele durante el procedimiento de corte. - Repita los tres pasos anteriores hasta que se haya reunido el número requerido de secciones, es decir, 40.
- Cuando termine, coloque la tapa en la caja deslizante y transfiérala a un congelador de -20 °C sobre hielo seco. Finalmente, limpie el criotomo desinfectando todos los artículos usados y encendiendo la luz ultravioleta en la cámara del criótomo.
NOTA: El protocolo se puede pausar en este punto. Almacene las muestras a una temperatura de -20 °C hasta que se realicen las mediciones de AFM. El tiempo de almacenamiento recomendado para las secciones y el medio OCT no debe exceder las 6 semanas. Más allá de este período de tiempo, las secciones y el medio OCT pueden exhibir alteraciones que se asemejen a la quemadura por congelación como resultado de su área de superficie sustancial y su propensión a absorber humedad.
3. Mapas de fuerza AFM de una sección pulmonar
NOTA: Para este protocolo, se utilizó el JPK NanoWizard 4XP (AFM) y la platina motorizada de Bruker combinada con el DMi8 (microscopio óptico invertido) de Leica para registrar las curvas de fuerza-desplazamiento de las secciones pulmonares.
- Configuración del AFM
- Encienda todos los dispositivos necesarios de la configuración de AFM (por ejemplo, computadora, controlador de AFM, mesa de aislamiento de vibraciones, microscopio óptico, etc.).
- Inicie el software de control AFM. En la descripción general Elegir experimento , seleccione el modo QI Advanced Imaging . Además, especifique la ubicación donde se deben almacenar los datos del experimento y los archivos de calibración.
NOTA: Si no posee la extensión QI Advanced Imaging , utilice el modo de mapeo de fuerza del modo de contacto como una opción alternativa. - Seleccione un voladizo con una constante de resorte adecuada para el tipo específico de muestra que se está observando. Para las muestras de tejido pulmonar blando, se recomienda el MLCT Cantilever F de Bruker con una punta piramidal y una constante de resorte nominal de 0,6 N/m. Si un chip contiene varios voladizos (como el MLCT), retire los voladizos más largos del chip con un par de pinzas bajo un microscopio estereoscópico para asegurarse de que solo el voladizo que se utiliza para la medición entre en contacto con la muestra.
NOTA: Otros voladizos en contacto con la muestra distintos del utilizado para la medición pueden provocar un movimiento no deseado de la muestra durante el experimento o incluso un desprendimiento de la muestra de la cinta subyacente.
PRECAUCIÓN: Evite entrar en contacto con el voladizo utilizado para la medición con las pinzas en el paso actual o en el subsiguiente. - Realice la instalación del chip en el soporte en voladizo. Inserte el soporte en el cabezal AFM. Coloque el cabezal AFM en la platina de la muestra, que se encuentra en el microscopio óptico invertido. Alinee la trayectoria de la palanca óptica como se indica en el manual del usuario. La alineación adecuada de la trayectoria de la palanca óptica se logra cuando el rayo láser se coloca inicialmente en el extremo frontal del voladizo de medición y, posteriormente, el rayo láser reflejado golpea el centro del fotodiodo segmentado.
- Calibración de voladizos mediante el método de ruido térmico57
NOTA: La calibración del voladizo incluye la determinación de su sensibilidad de palanca óptica invertida (InvOLS) y constante de resorte. La calibración individual de cada voladizo usado es necesaria porque la constante nominal del resorte típicamente proporcionada para los voladizos suele ser solo una aproximación. En experimentos en los que las propiedades mecánicas de una muestra se determinan a partir de curvas de fuerza registradas, como la descrita en este protocolo, es esencial determinar directamente el InvOLS a través de un método basado en contacto sobre un sustrato rígido. Los métodos alternativos que determinan el InvOLS, como derivarlo de la geometría del voladizo, solo pueden proporcionar una aproximación aproximada y no son adecuados para el tipo de aplicación descrito aquí. Alternativamente, si se utilizan voladizos donde el proveedor proporciona la constante de resorte exacta, el InvOLS se puede determinar mediante un procedimiento descrito por Schiller et al.58.- Coloque un portaobjetos de microscopio en la platina de muestra del AFM. El portaobjetos del microscopio sirve como sustrato rígido para la determinación basada en el contacto del InvOLS.
- Después de colocar el cabezal AFM en la platina de muestra, baje el cabezal con los motores paso a paso hasta que el espacio restante entre el soporte en voladizo y el portaobjetos del microscopio sea de aproximadamente 1 a 2 mm. Aplique PBS en el costado del soporte en voladizo, permitiendo que fluya hacia abajo y forme un menisco líquido en el espacio entre el soporte y el portaobjetos del microscopio con una jeringa de 1 ml con una aguja larga (0,9 mm x 70 mm).
NOTA: Asegúrese de que el mismo líquido, como tampón o medio, que se utiliza para las mediciones de fuerza y volumen de la muestra también se emplee para el proceso de calibración. - Para realizar una calibración basada en contacto en el software, vaya a la página Adquirir datos y seleccione la opción Vista avanzada . Acceda al Administrador de calibración utilizando el botón de menú de hamburguesas ubicado en la esquina superior derecha. Dentro de esta interfaz, seleccione Basado en contacto como Método y elija el voladizo MLCT F en el menú desplegable como el nombre del voladizo. Además, establezca el Punto de ajuste en 1 V y el Número de escaneos en 10 en los campos de entrada correspondientes.
- En la página Adquirir datos , en el panel de control de la izquierda, introduzca un punto de ajuste de 1 V para el procedimiento de aproximación automática. Haga clic en el botón de flecha azul que apunta hacia abajo ubicado en la esquina superior izquierda de la interfaz de usuario para iniciar el enfoque automático. Cuando la aproximación haya finalizado y el voladizo esté en contacto con el portaobjetos del microscopio, haga clic en el botón Calibrar en la ventana del Administrador de calibración .
NOTA: El InvOLS y la constante de resorte en voladizo se determinan automáticamente registrando 10 curvas de fuerza, así como la densidad espectral de potencia térmica (PSD), que se utiliza para determinar la constante de resorte con el método de ruido térmico57. Una vez finalizado el procedimiento de calibración automática, compruebe visualmente que la curva ajustada describe correctamente el modo normal (primer pico) de la PSD registrada del voladizo y que la constante de muelle determinada tiene el mismo orden de magnitud que la constante de muelle nominal dada por el fabricante del voladizo. Si este no es el caso, el rango de ajuste debe ajustarse manualmente para garantizar que el ajuste converja y represente el primer pico del espectro de potencia térmica. - Una vez finalizada la calibración, cierre la ventana del Administrador de calibración . Después de cerrar el Administrador de calibración, se genera un archivo de calibración (archivo ascii *.tnd) en el directorio preseleccionado (consulte el paso 3.1.2) que contiene los resultados de calibración determinados, que luego se requieren para el análisis de datos. Documente los valores de calibración para referencia futura durante el proceso de análisis de datos, por ejemplo, en el cuaderno de laboratorio.
ATENCIÓN: A partir de este paso, absténgase de cambiar la posición del láser en el voladizo, ajustar el espejo en el cabezal AFM o reubicar el soporte en voladizo. La modificación de estos elementos afectará al InvOLS y hará que los valores de calibración no sean válidos. Para compensar la deriva térmica, ajuste la posición del fotodiodo segmentado del cabezal AFM.
- Análisis de una pared alveolar en una sección pulmonar
- Recupere el portamuestras para evaluarlo del congelador. Deje que la muestra se descongele a temperatura ambiente durante aproximadamente 1 minuto hasta que todo el medio OCT se haya aclarado. Posteriormente, agregue algunas gotas de PBS a la sección de tejido pulmonar para rehidratarlo.
- Coloque el portaobjetos del microscopio que lleva la sección de tejido pulmonar en la platina de la muestra. Vuelva a instalar el cabezal AFM en la platina de muestra y baje el cabezal hasta que el voladizo se coloque aproximadamente 1 mm por encima de la muestra. Agregue un poco de PBS adicional al costado del soporte en voladizo con una jeringa de 1 ml combinada con una aguja de 0,9 mm x 70 mm hasta que se forme un menisco líquido entre la sección de tejido y el soporte en voladizo.
- Navegue hasta el Administrador de configuración haciendo clic en el botón con el icono de llave inglesa en la esquina superior derecha de la interfaz de usuario. En el Administrador de configuración, ajuste la siguiente configuración general. Dentro de la sección Configuración de aproximación , establezca la Altura objetivo en 4 μm. Luego, dentro de la sección Configuración del modo actual , vaya a la Configuración avanzada de comentarios y establezca el Multiplicador en 1. Por último, dentro de la subsección Configuración de fuerza , que también se encuentra dentro de la sección Configuración del modo actual , seleccione la opción Piezoeléctrico retraído en el menú desplegable Modo al final .
- Utilice el microscopio óptico invertido para identificar y navegar hasta una pared alveolar intacta. Una pared intacta se caracteriza en la imagen de campo claro por bordes oscuros y suaves. (ver Figura 2A-B).
NOTA: La importación de la imagen de campo claro en el software de control AFM, cuando esté disponible, puede ayudar a navegar el voladizo hasta una pared alveolar en particular y capturar con precisión un mapa de fuerza o QI en la parte superior. En el software JPK SPM, esta función se denomina superposición directa. - Establezca los parámetros para las curvas de fuerza-indentación en el panel de control de la izquierda en función del AFM utilizado, el voladizo empleado y la muestra observada. Para el NanoWizard 4XP y el MLCT Cantilever F de Bruker, recomendamos los siguientes parámetros: Consigna = 5 nN; longitud z = 8 μm; Velocidad z = 300 μm/s.
NOTA: La longitud z recomendada de 8 μm es bastante grande. Esto se atribuye a las características del tejido pulmonar. Dado que el tejido pulmonar suele ser bastante blando, la profundidad de indentación puede alcanzar hasta 4 μm en ciertos puntos, incluso con un punto de ajuste relativamente modesto de 5 nN. Con el fin de garantizar que la línea de base, en la que la punta no está en contacto con la muestra, represente al menos aproximadamente la mitad de la curva de fuerza-indentación, se recomienda que la longitud total de la curva se establezca en 8 μm. Esto simplificará significativamente el análisis de la curva más adelante. En el caso de otros bio-AFM, estos parámetros también deberían ser aplicables, aunque la nomenclatura de ciertos parámetros, como el punto de ajuste, que determina la fuerza de indentación máxima, puede variar entre los diferentes fabricantes. Si el sistema AFM no puede alcanzar la alta velocidad vertical que se indica aquí, es necesario disminuir la velocidad z de acuerdo con la recomendación del fabricante de su AFM.
PRECAUCIÓN: Con el fin de garantizar la comparabilidad de los resultados, es esencial emplear velocidades de aproximación z idénticas en todos los experimentos. - Establezca la ubicación y el tamaño del mapa de fuerza de visión general inicial, asegurándose de que abarque toda la pared alveolar, extendiéndose de un lado del aire al otro. Para las muestras de tejido pulmonar murino, un tamaño entre 20 μm x 20 μm y 30 μm x 30 μm suele ser suficiente. Independientemente del tamaño específico del mapa, establezca el número de píxeles (que corresponden al número de curvas de fuerza registradas) en 50 x 50. Esto conduce a la generación de 2.500 curvas de fuerza-indentación para cada mapa, lo que proporciona la resolución necesaria para identificar el BM en el canal de pendiente del mapa de fuerzas. (ver Figura 2C-D).
- Después de registrar el mapa general, examine la altura y el canal de pendiente del mapa de fuerzas. Si el canal de altura indica que la pared alveolar está intacta (ver Figura 2D) y si es posible distinguir el BM en el canal de pendiente como una línea brillante claramente definida (ver Figura 2C, E) situada entre dos capas del epitelio y el endotelio, capture un mapa de fuerza más enfocado con un tamaño de 3 μm x 3 μm o 4 μm x 4 μm en el BM. Mantenga los píxeles en 50 x 50 curvas.
NOTA: Este procedimiento conduce a una mayor resolución del mapa y permite registrar un mayor número de valores del módulo de Young, particularmente para el BM (ver Figura 2E-F). Además, esto permite una diferenciación más precisa entre el BM y los tejidos circundantes, mejorando la precisión del proceso de filtrado espacial posterior. Si los canales del mapa general muestran una pared colapsada o rota, artefactos debidos a que el voladizo tocó la muestra no solo con la punta, o no hay BM identificable en el canal de pendiente, navegue a otra pared en la sección de tejido hasta que se pueda identificar una pared con una evaluación libre de artefactos de la BM.
Repetir los pasos anteriores (3.3.6 y 3.3.7) en secciones posteriores de la misma muestra pulmonar hasta que se haya evaluado el número deseado de paredes alveolares. Se recomiendan al menos cuatro paredes para cada muestra de pulmón para lograr resultados sólidos59. - Una vez completada la medición, limpie todo el equipo y los materiales que hayan entrado en contacto con la muestra y el PBS. Si la muestra ya no es necesaria, deséchela en un contenedor de residuos de vidrio esterilizable en autoclave. Guarde todos los datos registrados y apague el software de control AFM y la computadora. Por último, apague todos los dispositivos eléctricos que se hayan utilizado.
NOTA: En este punto, el protocolo se puede pausar. Los datos experimentales se guardan y se pueden analizar en un momento posterior.
4. Análisis de datos
- Análisis de curvas de fuerza-indentación
NOTA: Un manual detallado de la aplicación Análisis de Curva de Fuerza se puede encontrar aquí: https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/2a.-Force-Curve-Analysis.- Inicie CANTER Processing Toolbox56 en MATLAB y abra la aplicación Force Curve Analysis .
NOTA: La caja de herramientas se puede descargar desde el repositorio de GitHub siguiendo este enlace: https://github.com/CANTERhm/CANTER_Processing_Tool. El archivo README.md, que comprende una guía completa sobre cómo instalar el software e iniciarlo con MATLAB, está disponible en el repositorio de GitHub. Se puede encontrar más información sobre cada aplicación incluida en la caja de herramientas en el wiki correspondiente en https://github.com/CANTERhm/CANTER_Processing_Tool/wiki. - Cargue el mapa de fuerza de alta resolución (3 μm x 3 μm o 4 μm x 4 μm) haciendo clic en el botón Seleccionar archivo , navegando hasta la ubicación donde se guarda el mapa de fuerza, haciendo doble clic en él y, a continuación, haciendo clic en el botón Cargar datos en la interfaz de usuario de la aplicación.
- Aparece una ventana emergente que solicita los valores de calibración (InvOLS y constante de resorte) que se determinaron (ver paso 3.2) para el voladizo utilizado para registrar el mapa de fuerza seleccionado en el paso del protocolo anterior. Introduzca los valores de calibración en los campos de edición respectivos. Si no se han documentado durante el paso 3.2.4, consulte el archivo de calibración generado automáticamente, que también contiene los valores de calibración. Mantenga la opción predeterminada sí para la pregunta de si se debe calcular la separación entre la punta y la muestra. Posteriormente, haga clic en el botón Enviar para continuar.
- Aparece una segunda ventana emergente que permite seleccionar la geometría del indentador y la relación de Poisson para la muestra con sangría. Para los voladizos MLCT utilizados, seleccione Pirámide de cuatro lados como geometría de punta e introduzca 17,5° para el semiángulo con respecto al borde (ambos son los valores predeterminados). Utilice la relación de Poisson predeterminada de 0,5, que representa un material incompresible. Para continuar con el procedimiento de carga del mapa QI, haga clic en el botón Enviar .
NOTA: Durante el procedimiento de carga, las curvas de deflexión-desplazamiento registradas (datos brutos del AFM) se convierten en curvas de fuerza-indentación basadas en los valores de calibración proporcionados. Para una comprensión completa de los pasos de transformación individuales y las ecuaciones asociadas, consulte la información complementaria de Hartmann et al.59. - Al finalizar el proceso de carga, se mostrará en la pantalla la curva de fuerza inicial del mapa de fuerzas. Asegúrese de que esté seleccionado el modo de corrección de línea base Desplazamiento + Inclinación . Este modo detecta automáticamente la línea de base de la curva de inducción de fuerza y corrige la inclinación y el desplazamiento vertical para que sean cero. Además, seleccione el modelo a través de Hertz como algoritmo para el Buscador de puntos de contacto y utilice un valor del 20% en el campo de edición designado para esta opción. Este algoritmo es particularmente adecuado para detectar el punto de contacto en las curvas de fuerza-indentación que se han registrado en muestras de tejidos blandos. Por último, establezca la profundidad de ajuste en el campo de edición correspondiente en 1,5 μm.
NOTA: Para garantizar que la rigidez del portaobjetos del microscopio no afecte al módulo de Young resultante, se recomienda establecer la profundidad de ajuste máxima en un valor que no sea superior al 10% del espesor de la sección de tejido60. Para más detalles sobre los algoritmos de corrección, véase la información complementaria de Hartmann et al.59.
PRECAUCIÓN: Utilice la misma profundidad de ajuste en todos los experimentos para garantizar que los resultados sean comparables. - Para aplicar el ajuste del modelo de Hertz modificado a todas las curvas de fuerza-indentación del mapa QI, haga clic en el botón Mantener y aplicar a todo . Se abre una ventana de diálogo que pregunta si cada curva de fuerza debe mostrarse o no durante el análisis. Al no mostrar las curvas, la velocidad de procesamiento se acelera ligeramente.
- Una vez finalizado el último análisis de la curva de fuerza, aparece una ventana que pregunta si se deben guardar los resultados de ajuste. Para guardar los resultados en ambos. tsv (valores separados por tabulaciones) y .xlsx (Excel), haga clic en Sí e introduzca un nombre para los archivos de resultados. Además, se guarda un archivo de texto con la extensión de archivo *.meta_data, que contiene todos los detalles relacionados con el análisis de las curvas, como los valores de calibración utilizados, el modelo de ajuste elegido, el algoritmo de detección de puntos de contacto seleccionado, el rango de ajuste y otra información relevante.
- Cierre la aplicación Análisis de curva de fuerza haciendo clic en el icono de la cruz en la esquina superior derecha de la ventana principal.
- Inicie CANTER Processing Toolbox56 en MATLAB y abra la aplicación Force Curve Analysis .
- Filtrado espacial de los resultados del mapa QI
NOTA: El paso de filtrado espacial garantiza que solo se tengan en cuenta los valores del módulo de Young de los mapas QI que se originan en el BM para cualquier análisis posterior. Puede encontrar un manual detallado para la herramienta de filtrado de resultados aquí: https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/Result-Filtering-Tool.- Seleccione la herramienta de filtrado de resultados de la lista de aplicaciones en la ventana de selección de aplicaciones de la caja de herramientas de procesamiento de CANTER e inicie la aplicación haciendo clic en el botón Iniciar aplicación .
- Para cargar los resultados de ajuste, haga clic en Abrir en la barra de menú superior de la interfaz de usuario de la herramienta de filtrado de resultados . En la siguiente ventana emergente, localice el primer botón Establecer situado dentro de la pestaña JPK Maps y elija el archivo .tsv que contiene los resultados del análisis de la curva de fuerza (consulte el paso 4.1.6). Haga clic en el segundo botón Establecer y localice el archivo de mapa QI que corresponde al archivo .tsv ya seleccionado. A continuación, haga clic en Enviar para cargar los datos del mapa y los resultados del análisis de la curva de fuerza.
- Seleccione la opción Emodul en el menú desplegable superior Canal mostrado para mostrar los resultados obtenidos del módulo de Young como una imagen de mapa. Además, elija la opción Emódulo (si aún no está seleccionada) en el menú desplegable Canal de datos ubicado debajo del gráfico del histograma para visualizar la distribución de los valores del módulo de Young cargados en el mapa QI.
- Haga clic en el botón de flecha de flujo de manipulación , que se encuentra en el centro de la interfaz de usuario, y asegúrese de que apunte hacia el lado derecho. Establezca el botón de alternancia del filtro de imagen en la posición Activado en el panel de filtro ubicado encima de los ejes del histograma. Seleccione la opción Mano alzada en el menú desplegable Geometría del filtro y luego haga clic en el botón Agregar .
- En la imagen del canal superior, dibuje la máscara de filtro rodeando el BM mientras mantiene presionada la tecla izquierda del mouse. El BM es identificable en el mapa de módulo de Young como una estructura brillante (valores altos del módulo de Young) en comparación con el tejido más blando vecino (ver Figura 3A). Cuando termine de dibujar la máscara, suelte la tecla izquierda del ratón y haga doble clic en la máscara que se aplicará al mapa. El gráfico del histograma de la derecha ahora solo muestra los valores del módulo de Young para las regiones del mapa que están resaltadas en verde (el color predeterminado; consulte la Figura 3C-D).
- Para crear un nuevo archivo de resultados .tsv que solo contenga los valores del módulo de Young enmascarados, haga clic en la barra de menú superior de la interfaz de usuario en Guardar > Guardar histograma > Guardar datos. Como alternativa, haga clic con el botón derecho en un área vacía de los ejes de trazado del histograma y seleccione Guardar datos en el menú contextual.
- Haga clic en Seleccionar todo en la ventana emergente que pregunta qué resultados escribir en el archivo .tsv. A continuación, confirme la selección haciendo clic en el botón Aceptar . A continuación, introduzca un nombre para el archivo .tsv en el cuadro de diálogo de guardado, elija la ubicación de guardado deseada y, por último, haga clic en Aceptar para guardar los resultados filtrados.
- Cierre la herramienta de filtrado de resultados haciendo clic en el icono de la cruz en la esquina superior derecha de la aplicación.
- R2 Filtrado de resultados
NOTA: Este paso del protocolo garantiza que solo se consideren los resultados del módulo de Young donde el modelo de Hertz modificado mostró una buena concordancia con la curva de fuerza-indentación a la que se ajustó. Puede encontrar un manual detallado de la herramienta de filtrado R² aquí: https://github.com/CANTERhm/CANTER_Processing_Tool/wiki/R%C2%B2-Filtering-Tool.- Inicie la herramienta de filtrado R² seleccionándola en la lista de aplicaciones de la ventana de selección de aplicaciones de CANTER Processing Toolbox y haciendo clic en Iniciar aplicación.
- Navegue hasta la barra de menú superior de la interfaz de usuario y haga clic en Abrir archivo .tsv. En el siguiente cuadro de diálogo de carga de ficheros, localice el fichero .tsv que contiene los resultados del módulo de Young del BM guardado durante el paso anterior 4.2.5 y selecciónelo haciendo doble clic.
- Aparece un cuadro de diálogo que presenta una lista de las columnas de datos contenidas en el archivo .tsv. Seleccione Emódulo como la columna que contiene los datos del módulo de Young. A continuación, en un cuadro de diálogo de cuadro de lista separado que solicita los datos de R², seleccione rsquare_fit. Una vez completado el procedimiento de carga, los datos del módulo de Young cargados se muestran en forma de histograma en la parte superior izquierda de la pantalla, mientras que la distribución de los datos R² se muestra en el gráfico de la parte superior derecha.
- Introduzca 0,96 en el campo de edición situado en el centro de la interfaz gráfica de usuario. Haga clic en el botón Filtrar datos para aplicar el filtro R² utilizando el valor introducido. Esta acción conservará solo los resultados que tengan un valor R² de 0,96 o superior. Las distribuciones filtradas de los resultados del módulo de Young y los valores de R² correspondientes se muestran en la mitad inferior de la interfaz de usuario.
- Seleccione Guardar datos filtrados en la barra de menú superior para crear un nuevo archivo .tsv que solo incluya los resultados que cumplan el criterio de filtro R² definido de > 0,96. Los datos guardados ahora se pueden mostrar como un histograma, por ejemplo, utilizando la herramienta de trazado de histograma, que visualiza la distribución del módulo de Young para el BM pulmonar sondeado (consulte la Figura 4A).
Resultados
Después de la identificación y medición de una pared alveolar intacta, el resultado primario de este protocolo son los valores filtrados del módulo de Young de la MO. Es importante tener en cuenta que no es posible encontrar una pared alveolar adecuada para la medición de la MFA en todas las secciones de tejido. En nuestras observaciones experimentales, aproximadamente el 75% de las criosecciones derivadas de especímenes de pulmón murino exhibieron una pared alveolar cuantificable. Para determinar con precisión la distribución espacial del módulo de Young en un BM pulmonar, aconsejamos emplear mapas de fuerza que abarquen 3 μm x 3 μm o 4 μm x 4 μm. Este enfoque garantiza una resolución suficiente para examinar los BM pulmonares de ratón, que suelen medir entre 100 y 200 nm de grosor. Por lo tanto, el enfoque experimental consiste en capturar curvas de fuerza de 50 x 50 para cada mapa de fuerzas, lo que produce un total de 2.500 curvas de fuerza. Dentro del mapa de fuerzas, las curvas de fuerzas están espaciadas uniformemente. Esto implica que, además del módulo de Young de la BM, las estructuras tisulares que rodean la BM se sondean incluso en los mapas de fuerza pequeños. Por lo tanto, para determinar de manera precisa y selectiva el módulo de Young exclusivamente de la MO pulmonar, implementamos un proceso de filtrado de dos pasos en este protocolo.
El paso de filtrado inicial del filtrado espacial dependiente del operador selecciona los resultados de ajuste de la curva de fuerza que se originan en el MO pulmonar murino, que comprende aproximadamente el 10% al 25% de los valores totales de los resultados contenidos en el mapa de fuerza analizado. Después de este filtrado espacial inicial, se conservan aproximadamente entre 250 y 625 resultados de ajuste para su posterior análisis. El siguiente paso de filtrado garantiza que solo se incluyan los resultados relacionados con BM que se originan en las curvas de fuerza adecuadamente descritas por el modelo de Hertz modificado. Por lo tanto, se utiliza como criterio de filtro el coeficiente de determinación (R2) del ajuste del modelo de Hertz modificado. Nuestra experiencia empírica sugiere que mantener resultados con valores de R2 superiores a 0,96 es adecuado para obtener resultados de ajuste mediante la adherencia a este protocolo. Es importante tener en cuenta que el valor de R2 está significativamente influenciado por la calidad (suavidad) y la longitud de la línea base de la curva de fuerzas, la determinación del punto de contacto y la convergencia del modelo ajustado. Después de la aplicación de ambos procedimientos de filtrado, generalmente quedan aproximadamente de 100 a 500 valores del módulo de Young para la MO de una sola pared alveolar.
La distribución de estos valores restantes del módulo de Young desde el BM se puede visualizar utilizando un histograma (Figura 4A). Es importante señalar que los valores del módulo de Young resultantes de la MO pulmonar siguen una distribución logarítmica normal, que se observa comúnmente para variables aleatorias que solo pueden alcanzar valores positivos61. Esto se visualiza mediante el gráfico QQ de la Figura 4B. En consecuencia, se puede ajustar una distribución normal (gaussiana) a la distribución de los valores del módulo de Young (E) transformado logarítmicamente. Aquí, se utilizó el logaritmo natural (ln) para transformar los valores E . De este ajuste se extrajeron la posición máxima de la distribución (μ) y la desviación estándar (σ) y posteriormente se retransformaron para obtener el módulo de Young representativo, denotado como EBM, utilizando la siguiente ecuación:
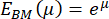
Tenga en cuenta que después de la retransformación, el intervalo de desviación estándar ya no es simétrico alrededor del valor máximo. Los bordes negativos (σ-) y positivos (σ+) del intervalo de desviación estándar se pueden calcular mediante las siguientes expresiones:
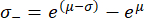
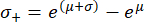
Estas ecuaciones son consecuencia del hecho de que la función exponencial es la función inversa del logaritmo neperiano62.
Después de retransformar el valor máximo μ = 9,31 y la desviación estándar σ = 0,18 determinada a partir del histograma de la Figura 4A, el valor representativo del módulo de Young es EBM = 11,05 kPa con un intervalo de desviación estándar de [-1,82 kPa, +2,18 kPa]. Alternativamente, se puede ajustar una distribución log-normal al histograma de los valores del módulo de Young (E) no transformados para determinar los parámetros característicos de la distribución.
Se recomienda analizar al menos cuatro paredes por ratón para obtener un resultado representativo robusto como hemos demostrado en detalle en Hartmann et al.59. Para determinar el módulo de Young representativo EBM para un ratón, los valores transformados logarítmicamente de las cuatro paredes se trazaron en un histograma combinado y μ y σ se determinaron ajustando una distribución normal. Posteriormente, μ y σ se vuelven a transformar como se describió anteriormente para recuperar EBM y el intervalo de desviación estándar para el ratón en cuestión.
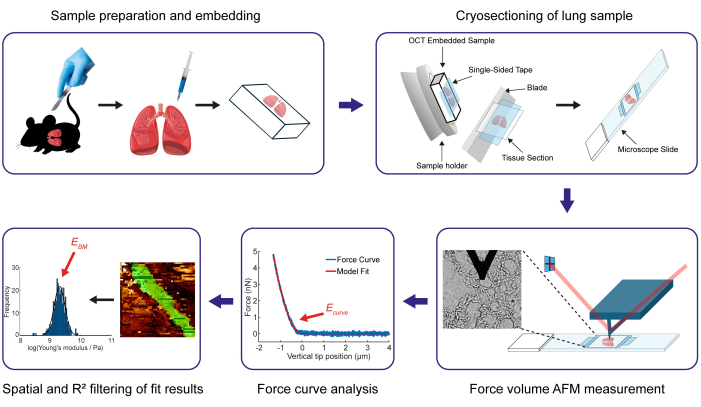
Figura 1: Resumen de los principales pasos del protocolo para determinar el módulo de Young de una membrana basal pulmonar murina. El paso inicial consiste en obtener y preparar pulmones murinos, seguidos de su incrustación. Por lo tanto, el pulmón se inyecta con una combinación de medio OCT y PBS, y luego se sumerge completamente en el compuesto OCT. Posteriormente, se obtienen secciones de tejido pulmonar de 15 μm de espesor mediante criosección con un criótomo. En este protocolo, detallamos el proceso de empleo de cintas adhesivas de una y dos caras para asegurar las muestras en su lugar durante el proceso de corte. Posteriormente, los valores del módulo de Young de la BM se determinan a través de mediciones del mapa de fuerza de AFM en las secciones de tejido. Una vez finalizado el proceso de análisis de la curva de fuerzas, se aplica un filtrado espacial y R² para aislar los resultados del módulo de Young de la estructura de interés, específicamente el BM (mostrado en verde en el mapa de fuerzas y cuantificado en la distribución del módulo de Young transformado logarítmicamente). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Un ejemplo de experimento de AFM con mapa de fuerza realizado en una sección de tejido pulmonar murino. (A) Imagen general de microscopía de campo claro de una sección de tejido pulmonar murino, que permite la identificación de paredes alveolares intactas adecuadas situadas entre dos alvéolos. En el lado derecho de la imagen, se puede ver el voladizo MLCT F de forma triangular del AFM. (B) Vista ampliada de una pared seleccionada dentro de la sección de tejido. (C) Canal de pendiente y (D) altura de un mapa general de 15 x 15 μm que contiene curvas de fuerza de 50 x 50 de la pared alveolar. En el canal de pendiente, el BM se puede identificar como una línea brillante (flecha blanca), lo que indica una estructura de mayor rigidez dentro del muro. El canal de altura confirma la integridad del muro, sin mostrar evidencia de ruptura. Después de la identificación del BM en el mapa general de 15 x 15 μm, se registra un mapa más pequeño de 4 x 4 μm (E: canal de pendiente, F: canal de altura) con curvas de fuerza de 50 x 50. Barras de escala: (A) 200 μm y (B-D) 25 μm. Los rangos de las escalas de color de las imágenes AFM mostradas son (C) 2,36 - 9,29 nN/μm, (D) 0 - 3,38 μm, (E) 2,36 - 9,29 nN/μm (imagen general) y 2,42 - 8,37 nN/μm (imagen más pequeña), (F) 0 - 3,38 μm (imagen general) y 0 - 1,99 μm (imagen más pequeña), de oscuro a brillante. Se utilizaron escalas de color lineales para todas las imágenes AFM. Haga clic aquí para ver una versión más grande de esta figura.
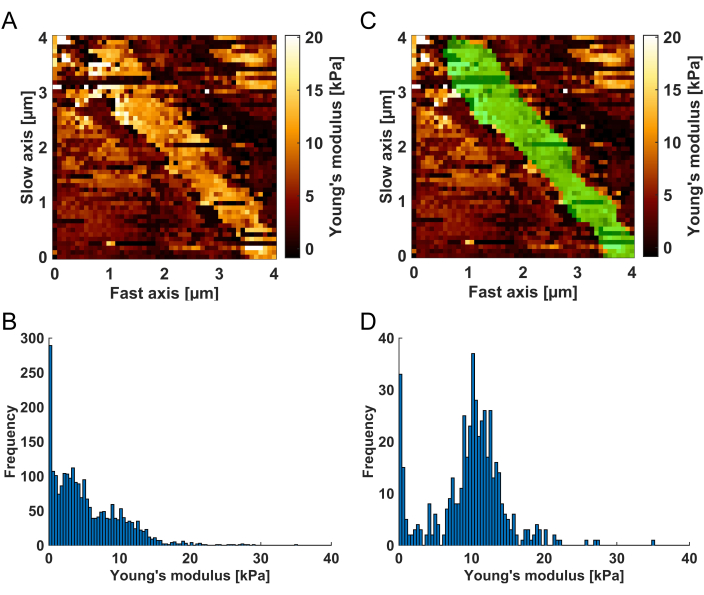
Figura 3: Visualización del procedimiento de filtrado espacial. (A) El mapa de módulo de Young resultante del análisis de la curva de fuerzas, tal como se muestra en la herramienta de filtrado de resultados para permitir el filtrado espacial de los resultados. El BM es identificable como una franja brillante (valores altos del módulo de Young) rodeada de compartimentos de tejido más blandos. (B) Histograma de los valores del módulo de Young correspondientes al mapa sin filtrar (A). Después de la (C) aplicación de la máscara de filtro espacial (área verde), (D) el histograma comprende exclusivamente los valores del módulo de Young de la región resaltada en verde, que se identificó como BM. Tenga en cuenta que los valores suaves residuales observables en el lado izquierdo del histograma se pueden atribuir a ajustes poco convergentes, que se eliminarán en el siguiente paso de filtrado de R². Haga clic aquí para ver una versión más grande de esta figura.
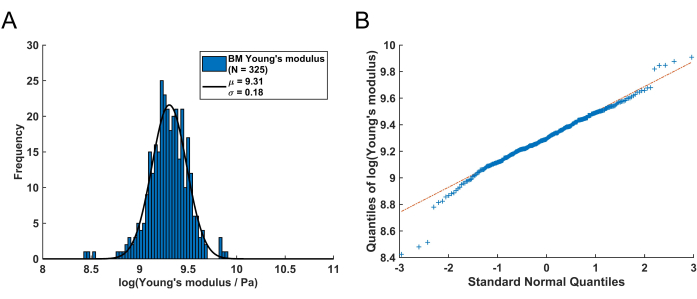
Figura 4: Distribución del módulo de Young de una MO pulmonar murina recibida siguiendo este protocolo. (A) Histograma logarítmico normalizado de los valores del módulo de Young de la MO resultante (N = 325). Ajustando una distribución normal (línea continua negra), se determina el valor medio μ = 9,31 con una desviación estándar de σ = 0,18. Después de la retransformación, esto corresponde a un módulo de Young del BM de EBM = 11,05 kPa con un intervalo de desviación estándar asimétrico de [-1,82 kPa, +2,18 kPa]. (B) QQ: Gráfica de los cuantiles de los valores del módulo de Young de BM logarittizados frente a los cuantiles de una distribución normal estándar. Este gráfico demuestra que, con la excepción de algunos valores en las colas izquierda y derecha de la distribución, los valores del módulo de Young resultantes se distribuyen principalmente log-normal. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Las características mecánicas de los BM han recibido un interés considerable debido a su influencia en diversas funciones celulares, como la gemación tumoral, la diferenciación celular, el movimiento celular y la accesibilidad de la cromatina 53,63,64,65,66,67,68 . En consecuencia, existe una brecha no cubierta en la capacidad de evaluar la mecánica de BM de una manera consistente y medible. Este protocolo tiene como objetivo permitir a los usuarios de AFM determinar el módulo de Young de los BM pulmonares a partir de muestras de pulmón murino sano o patológico obtenidas de ratones de tipo salvaje o modificados genéticamente. La aplicación CANTER Processing Toolbox56, basada en una interfaz gráfica de usuario fácil de usar, facilita el análisis posterior de los datos AFM. Se espera que este protocolo sea útil para otros investigadores que realicen estudios que puedan conducir a avances en el diagnóstico clínico, el manejo o nuevas estrategias de tratamiento para los trastornos pulmonares.
Los pasos críticos del protocolo incluyen garantizar que durante la criosección de la muestra pulmonar, las secciones de tejido no se descongelen en ningún momento para evitar alteraciones de las propiedades mecánicas debido a los ciclos de congelación-descongelación. Por lo tanto, es esencial utilizar solo portaobjetos de microscopio preenfriados para recoger las secciones y manipularlas solo dentro de la cámara del criótomo. El paso más crucial durante el registro de mapas de fuerza en una pared alveolar es utilizar la imagen de pendiente generada, que proporciona una visión general cualitativa de la distribución de la rigidez del área sondeada, para identificar una ubicación de medición adecuada del BM. El filtrado espacial de los valores del módulo de Young obtenidos constituye un paso crítico en el protocolo y es una característica clave de la caja de herramientas de procesamiento CANTER. Este paso de filtrado permite al investigador identificar el BM, que está situado entre dos capas celulares más blandas, a través de su distintiva huella más rígida y extraer los valores correspondientes del módulo de Young del BM para su posterior análisis.
Este flujo de trabajo de análisis avanzado reveló que los ratones knockout de netrina-4 exhibían un módulo de Young sustancialmente (aproximadamente dos veces) más alto en su membrana basal pulmonar en comparación con los ratones de camada de tipo salvaje53. Así, el presente protocolo facilita el análisis comparativo de diversas condiciones o grupos.
Además de la AFM, existen numerosas metodologías para examinar las propiedades mecánicas de los biomateriales, que abarcan las pinzas ópticas o magnéticas 69,70,71 a nivel molecular, las técnicas de microindentación 72,73 a escala intermedia y las pruebas de compresión confinada o no confinada74,75 a nivel macroscópico. En concreto, se han empleado miografías a presión, ensayos de tracción76,77 y reología78 para evaluar la rigidez de la BM de muestras vasculares y matrices de BM reconstituidas. Sin embargo, técnicas como la miografía a presión y las pruebas de tracción solo pueden evaluar las características mecánicas de tejidos completos o estructuras de tejidos completos, como vasos sanguíneos completos, que incluyen varias capas celulares y elementos estructurales. Como resultado, cuando se utilizan estos métodos para evaluar la biomecánica de la BM, las propiedades mecánicas de los no BM, como los componentes celulares, siempre se superponen a los resultados de la BM, lo que dificulta el aislamiento de las propiedades específicas de la propia BM.
En este protocolo, la característica de interés (BM) se extrae a través del filtrado espacial de los resultados del mapa de fuerza. Una limitación de este enfoque es el requisito de que la característica de interés sea discernible e identificable como un patrón distinto en los mapas de módulo de Young obtenidos o en cualquier otro canal de mapa disponible. Por ejemplo, la MO en el tejido alveolar es identificable únicamente debido a su estructura interconectada, que exhibe un módulo de Young significativamente mayor en comparación con las células circundantes. En consecuencia, el BM se manifiesta como una raya brillante continua en los mapas de módulo de Young obtenidos. Sin embargo, otras estructuras biológicas exhiben propiedades mecánicas que no son lo suficientemente distintas de las del tejido circundante para permitir el filtrado espacial en los resultados del mapa. Aunque el protocolo se puede combinar con otros métodos para identificar estructuras en los mapas de fuerza registrados, como la tinción de inmunofluorescencia, este enfoque puede producir resultados difíciles de interpretar. Esta complicación puede ocurrir porque los procedimientos de tinción pueden alterar las propiedades mecánicas de los tejidos. En consecuencia, cualquier procesamiento o tinción del tejido antes de las mediciones de AFM debe realizarse solo cuando no se disponga de un método alternativo para identificar la característica de interés. Además, es importante realizar experimentos de control adecuados comparando las propiedades biomecánicas de las muestras teñidas y no teñidas.
El protocolo introduce un método para identificar las estructuras objetivo basado en el contraste del módulo de Young. En consecuencia, se puede aplicar a tejidos con una MO anatómica similar a la del pulmón, incluyendo la glándula tiroides, el colon y la próstata. (ver Figura de Datos Ampliada en Hartmann et al.59). Si bien se desarrolló inicialmente para analizar la mecánica de BM, este protocolo y la CANTER Processing Toolbox56 son lo suficientemente versátiles como para examinar cualquier región mecánicamente distinta y conectada dentro de los mapas de fuerza de AFM (consulte las instrucciones en www.github.com/CANTERhm/CANTER_Processing_Tool/wiki). Esta amplia aplicabilidad hace que esta herramienta sea valiosa en todo el campo de la AFM y respalda el creciente enfoque en la mecanobiología en la comunidad científica en general.
Divulgaciones
Los autores declaran no tener intereses contrapuestos.
Agradecimientos
B.H., y H.C.-S. reconocer la financiación del Ministerio de Ciencia y Artes del Estado de Baviera a través del Bavarian Research Focus Herstellung und biophysikalische Charakterisierung von dreidimensionalen Geweben (CANTER) y el Foro Académico Bávaro (BayWISS)-Consorcio Doctoral de Investigación en Salud. El desarrollo del software de análisis de datos CANTER Processing Toolbox fue financiado por la Fundación Alemana de Investigación como parte del subproyecto 1 (CL 409/4-1/2) del consorcio de investigación Explorando la degeneración y regeneración del cartílago articular y el hueso subcondral en la osteoartritis - ExCarBon (FOR2407-1/2). B.H. y H.C.-S. reconocer la financiación de la Fundación Alemana de Investigación a través de la gran campaña de instrumentación GGA-HAW (INST 99/38-1). Este trabajo fue apoyado por la Sociedad Danesa del Cáncer (R204-A12454 (R.R.)) y la Fundación Alemana de Investigación (539446614 a R.R.).
Materiales
| Name | Company | Catalog Number | Comments |
| 1 mL Syringe | B Braun | 9166017V | Injekt-F |
| 10 mL Syringe | B Braun | 4606108V | Injekt Luer Solo |
| 15 mL Falcon tube | Sarstedt | 62.554.002 | screw cap tube |
| Cantilever - MLCT | Bruker AFM Probes | 3444 | AFM cantilever with a pyramidal tip shape |
| Cryotome blades | Leica Biosystems | 14035843496 | Low-profile disposable blades DB80LX |
| Cryotome sample holder | Leica Biosystems | 14047740044 | Specimen disc 30 |
| Cyotome | Leica Biosystems | CM1950 | Leica Cryostat |
| Direct Overlay Extension | Bruker | Software extension for the JPK SPM Software which enables to import the optical image of the inverted microscope into the Data Viewer of the SPM software. | |
| Disposable base mold | Science Services | SA62534-15 | Tissue-Tek Cryomold 15x15x5 mm |
| Double-sided tape | tesa Film | 56661-00002 | Photo Film Tape |
| Fixed-Spring Cantilever Holder | Bruker | ||
| Inverted Microscope - Leica DMi8 | Leica Microsystems | ||
| JPK Motorized Stage | Bruker | ||
| JPK NanoWizard 4XP BioScience | Bruker | ||
| JPK SPM Software | Bruker | ||
| K5 CMOS Microscope Kamera | Leica Microsystems | ||
| MATLAB | Mathworks | Version R2024a or higher | |
| MATLAB - Curve Fitting Toolbox | Mathworks | ||
| MATLAB - Image Processing Toolbox | Mathworks | ||
| MATLAB - Signal Processing Toolbox | Mathworks | ||
| MATLAB - Statistics and Machine Learning Toolbox | Mathworks | ||
| Microscope slides | Carl Roth | H869.1 | Plain microscope slides for cantilever calibration |
| Microscope slides - frosted edge | Carl Roth | H870.1 | Microscope slides with frosted edge for cryosectioning |
| Needle ø0.9 mm × 25 mm | B Braun | 4657500 | OCT injection into the lung sample |
| Needle ø0.9 mm × 70 mm | B Braun | 4665791 | Long needle to apply PBS under the AFM |
| OCT compound | Sakura Finetek | 4583 | Tissue-Tek O.C.T. Compound |
| Phosphate Buffered Saline | Bio&Sell | BS.L1825 | PBS solution without Ca2+, Mg2+, 500 mL |
| QI Advanced Imaging Extension | Bruker | Software extension for the JPK SPM Software which provides for each recorded image pixel the whole underlying force curve. | |
| Scalpel | B Braun | 5518083 | Surgical Disposable Scalpel |
| Scissors | Kaut-Bullinger | M681700 | Precise Scissors 13 cm |
| Single-sided tape | tesa Film | 57330-00000 | crystal clear tape, 33 m x 19 mm |
| Slide box | GWL Storing Systems | K50W | Slidebox for 50 slides |
| Stereo Microscope - Stemi DR1663 | Zeiss | ||
| Tweezers - Vomm SS-SA-ESD | Eleshop | ELE002121 |
Referencias
- Alsteens, D., et al. Atomic force microscopy-based characterization and design of biointerfaces. Nat Rev Mater. 2 (5), 17008 (2017).
- Aro, E., et al. Severe extracellular matrix abnormalities and chondrodysplasia in mice lacking collagen prolyl 4-hydroxylase isoenzyme II in combination with a reduced amount of isoenzyme I. J Biol Chem. 290 (27), 16964-16978 (2015).
- Domke, J., Radmacher, M. Measuring the elastic properties of thin polymer films with the atomic force microscope. Langmuir. 14 (12), 3320-3325 (1998).
- Huth, S., Sindt, S., Selhuber-Unkel, C. Automated analysis of soft hydrogel microindentation: Impact of various indentation parameters on the measurement of Young's modulus. PLoS One. 14 (8), e0220281 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nat Rev Phys. 1 (1), 41-57 (2019).
- Loparic, M., et al. Micro- and nanomechanical analysis of articular cartilage by indentation-type atomic force microscopy: validation with a gel-microfiber composite. Biophys J. 98 (11), 2731-2740 (2010).
- Plodinec, M., et al. The nanomechanical signature of breast cancer. Nat Nanotechnol. 7 (11), 757-765 (2012).
- Prein, C., et al. Structural and mechanical properties of the proliferative zone of the developing murine growth plate cartilage assessed by atomic force microscopy. Matrix Biol. 50, 1-15 (2016).
- Radmacher, M., Fritz, M., Kacher, C. M., Cleveland, J. P., Hansma, P. K. Measuring the viscoelastic properties of human platelets with the atomic force microscope. Biophys J. 70 (1), 556-567 (1996).
- Radmacher, M., Tillamnn, R. W., Fritz, M., Gaub, H. E. From molecules to cells: imaging soft samples with the atomic force microscope. Science. 257 (5078), 1900-1905 (1992).
- Stolz, M., et al. Early detection of aging cartilage and osteoarthritis in mice and patient samples using atomic force microscopy. Nat Nanotechnol. 4, 186-192 (2009).
- Stolz, M., et al. Dynamic elastic modulus of porcine articular cartilage determined at two different levels of tissue organization by indentation-type atomic force microscopy. Biophys J. 86 (5), 3269-3283 (2004).
- Florin, E. L., Moy, V. T., Gaub, H. E. Adhesion forces between individual ligand-receptor pairs. Science. 264 (5157), 415-417 (1994).
- Hansma, P. K., et al. Tapping mode atomic force microscopy in liquids. Appl Phys Lett. 64 (13), 1738-1740 (1994).
- Moy, V. T., Florin, E. L., Gaub, H. E. Intermolecular forces and energies between ligands and receptors. Science. 266 (5183), 257-259 (1994).
- Radmacher, M., Fritz, M., Hansma, H. G., Hansma, P. K. Direct observation of enzyme activity with the atomic force microscope. Science. 265 (5178), 1577-1579 (1994).
- Radmacher, M., Fritz, M., Hansma, P. K. Imaging soft samples with the atomic force microscope: gelatin in water and propanol. Biophys J. 69 (1), 264-270 (1995).
- Rief, M., Clausen-Schaumann, H., Gaub, H. E. Sequence-dependent mechanics of single DNA molecules. Nat Struct Biol. 6 (4), 346-349 (1999).
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible unfolding of individual titin immunoglobulin domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Drake, B., et al. Imaging crystals, polymers, and processes in water with the atomic force microscope. Science. 243 (4898), 1586-1589 (1989).
- Clausen-Schaumann, H., Rief, M., Tolksdorf, C., Gaub, H. E. Mechanical stability of single DNA molecules. Biophys J. 78 (4), 1997-2007 (2000).
- Rotsch, C., Radmacher, M. Drug-induced changes of cytoskeletal structure and mechanics in fibroblasts: an atomic force microscopy study. Biophys J. 78 (1), 520-535 (2000).
- Lekka, M., et al. Elasticity of normal and cancerous human bladder cells studied by scanning force microscopy. Eur Biophys J. 28 (4), 312-316 (1999).
- Weisenhorn, A. L., Khorsandi, M., Kasas, S., Gotzos, V., Butt, H. J. Deformation and height anomaly of soft surfaces studied with an AFM. Nanotechnology. 4 (2), 106 (1993).
- Rotsch, C., Jacobson, K., Radmacher, M. Dimensional and mechanical dynamics of active and stable edges in motile fibroblasts investigated by using atomic force microscopy. Proc Natl Acad Sci USA. 96 (3), 921-926 (1999).
- Goldmann, W. H., Ezzell, R. M. Viscoelasticity in wild-type and vinculin-deficient (5.51) mouse F9 embryonic carcinoma cells examined by atomic force microscopy and rheology. Exp Cell Res. 226 (1), 234-237 (1996).
- Tschaikowsky, M., et al. Hybrid fluorescence-AFM explores articular surface degeneration in early osteoarthritis across length scales. Acta Biomater. 126, 315-325 (2021).
- Tschaikowsky, M., et al. Proof-of-concept for the detection of early osteoarthritis pathology by clinically applicable endomicroscopy and quantitative AI-supported optical biopsy. Osteoarthr Cartilage. 29 (2), 269-279 (2021).
- Kinney, J. H., Balooch, M., Marshall, S. J., Marshall, G. W., Weihs, T. P. Atomic force microscope measurements of the hardness and elasticity of peritubular and intertubular human dentin. J Biomech Eng. 118 (1), 133-135 (1996).
- Lundkvist, A., et al. . Symposium on Thin Films - Stresses and Mechanical Properties VI,. 1996 MRS Spring Meeting. , 353-358 (1997).
- Tao, N. J., Lindsay, S. M., Lees, S. Measuring the microelastic properties of biological material. Biophys J. 63 (4), 1165-1169 (1992).
- Radmacher, M. Measuring the elastic properties of biological samples with the AFM. IEEE Eng Med Biol Mag. 16 (2), 47-57 (1997).
- Becke, T. D., et al. Pilus-1 backbone protein RrgB of Streptococcus pneumoniae binds collagen I in a force-dependent way. ACS Nano. 13 (6), 7155-7165 (2019).
- Becke, T. D., et al. Single molecule force spectroscopy reveals two-domain binding mode of Pilus-1 tip protein RrgA of Streptococcus pneumoniae to fibronectin. ACS Nano. 12 (1), 549-558 (2018).
- Pill, M. F., East, A. L. L., Marx, D., Beyer, M. K., Clausen-Schaumann, H. Mechanical activation drastically accelerates amide bond hydrolysis, matching enzyme activity. Angew Chem Int Ed Engl. 58 (29), 9787-9790 (2019).
- Schmidt, S. W., Filippov, P., Kersch, A., Beyer, M. K., Clausen-Schaumann, H. Single-molecule force-clamp experiments reveal kinetics of mechanically activated silyl ester hydrolysis. ACS Nano. 6 (2), 1314-1321 (2012).
- Docheva, D., et al. Researching into the cellular shape, volume and elasticity of mesenchymal stem cells, osteoblasts and osteosarcoma cells by atomic force microscopy. J Cell Mol Med. 12 (2), 537-552 (2008).
- Docheva, D., Padula, D., Schieker, M., Clausen-Schaumann, H. Effect of collagen I and fibronectin on the adhesion, elasticity and cytoskeletal organization of prostate cancer cells. Biochem Biophys Res Commun. 402 (2), 361-366 (2010).
- Kiderlen, S., et al. Age related changes in cell stiffness of tendon stem/progenitor cells and a rejuvenating effect of ROCK-inhibition. Biochem Biophys Res Commun. 509 (3), 839-844 (2019).
- Reuten, R., et al. Structural decoding of netrin-4 reveals a regulatory function towards mature basement membranes. Nat Commun. 7, 13515 (2016).
- Fleischhauer, L., et al. Nano-scale mechanical properties of the articular cartilage zones in a mouse model of post-traumatic osteoarthritis. Appl Sci. 12 (5), 2596 (2022).
- Alberton, P., et al. Aggrecan hypomorphism compromises articular cartilage biomechanical properties and is associated with increased incidence of spontaneous osteoarthritis. Int J Mol Sci. 20 (5), 1-16 (2019).
- Alberton, P., et al. Aggrecan is critical in maintaining the cartilage matrix biomechanics which in turn influences the correct development of the growth plate. Osteoarthr Cartilage. 27, S178-S178 (2019).
- Gronau, T., et al. Forced exercise-induced osteoarthritis is attenuated in mice lacking the small leucine-rich proteoglycan decorin. Ann Rheum Dis. 76 (2), 442-449 (2017).
- Hartmann, B., et al. Early detection of cartilage degeneration: A comparison of histology, fiber Bragg grating-based micro-indentation, and atomic force microscopy-based nano-indentation. Int J Mol Sci. 21 (19), 7384-7398 (2020).
- Rellmann, Y., et al. ER stress in ERp57 knockout knee joint chondrocytes induces osteoarthritic cartilage degradation and osteophyte formation. Int J Mol Sci. 23 (1), 182 (2021).
- Kamper, M., et al. Early changes in morphology, bone mineral density and matrix composition of vertebrae lead to disc degeneration in aged collagen IX -/- mice. Matrix Biol. 49, 132-143 (2016).
- Akhtar, R., Sherratt, M. J., Cruickshank, J. K., Derby, B. Characterizing the elastic properties of tissues. Mater Today. 14 (3), 96-105 (2011).
- Junior, C., et al. Baseline stiffness modulates the non-linear response to stretch of the extracellular matrix in pulmonary fibrosis. Int J Mol Sci. 22 (23), 12928 (2021).
- Junior, C., et al. Multi-step extracellular matrix remodelling and stiffening in the development of idiopathic pulmonary fibrosis. Int J Mol Sci. 24 (2), 1708 (2023).
- Sicard, D., Fredenburgh, L. E., Tschumperlin, D. J. Measured pulmonary arterial tissue stiffness is highly sensitive to AFM indenter dimensions. J Mech Behav Biomed Mater. 74, 118-127 (2017).
- Zemla, J., et al. AFM-based nanomechanical characterization of bronchoscopic samples in asthma patients. J Mol Recognit. 31 (12), e2752 (2018).
- Reuten, R., et al. Basement membrane stiffness determines metastases formation. Nat Mater. 20 (6), 892-903 (2021).
- Johnstone, A. H. CRC Handbook of Chemistry and Physics. J Chem Technol Biotechnol. 50 (2), 294-295 (1991).
- Rico, F., et al. Probing mechanical properties of living cells by atomic force microscopy with blunted pyramidal cantilever tips. Phys Rev E. 72 (2), 1-10 (2005).
- . . CANTER Processing Toolbox v.5.7.0. , (2022).
- Hutter, J. L. J. B. Calibration of atomic-force microscope tips. Rev Sci Instrum. 64 (7), 1868-1873 (1993).
- Schillers, H., et al. Standardized nanomechanical atomic force microscopy procedure (SNAP) for measuring soft and biological samples. Sci Rep. 7 (1), 5117 (2017).
- Hartmann, B., et al. Profiling native pulmonary basement membrane stiffness using atomic force microscopy. Nat Protoc. 19 (5), 1498-1528 (2024).
- Dimitriadis, E. K., Horkay, F., Maresca, J., Kachar, B., Chadwick, R. S. Determination of elastic moduli of thin layers of soft material using the atomic force microscope. Biophys J. 82 (5), 2798-2810 (2002).
- Jolicoeur, P. . Introduction to Biometry. , (1999).
- Bronshtein, I. N., Semendyayev, K. A., Musiol, G., Mühlig, H. . Handbook of Mathematics. , (2015).
- Fiore, V. F., et al. Mechanics of a multilayer epithelium instruct tumour architecture and function. Nature. 585 (7825), 433-439 (2020).
- Koester, J., et al. Niche stiffening compromises hair follicle stem cell potential during ageing by reducing bivalent promoter accessibility. Nat Cell Biol. 23 (7), 771-781 (2021).
- Bedzhov, I., Zernicka-Goetz, M. Self-organizing properties of mouse pluripotent cells initiate morphogenesis upon implantation. Cell. 156 (5), 1032-1044 (2014).
- Kyprianou, C., et al. Basement membrane remodelling regulates mouse embryogenesis. Nature. 582 (7811), 253-258 (2020).
- Saraswathibhatla, A., Indana, D., Chaudhuri, O. Cell-extracellular matrix mechanotransduction in 3D. Nat Rev Mol Cell Biol. 24 (7), 495-516 (2023).
- Sherwood, D. R. Basement membrane remodeling guides cell migration and cell morphogenesis during development. Curr Opin Cell Biol. 72, 19-27 (2021).
- Ashkin, A., Dziedzic, J. M., Bjorkholm, J. E., Chu, S. Observation of a single-beam gradient force optical trap for dielectric particles. Opt Lett. 11 (5), 288 (1986).
- Marago, O. M., Jones, P. H., Gucciardi, P. G., Volpe, G., Ferrari, A. C. Optical trapping and manipulation of nanostructures. Nat Nanotechnol. 8 (11), 807-819 (2013).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nat Methods. 5 (6), 491-505 (2008).
- Marchi, G., et al. Microindentation sensor system based on an optical fiber Bragg grating for the mechanical characterization of articular cartilage by stress-relaxation. Sens Actuators B. 252, 440-449 (2017).
- Wakitani, S., et al. Repair of large full-thickness articular cartilage defects with allograft articular chondrocytes embedded in a collagen gel. Tissue Eng. 4 (4), 429-444 (1998).
- Moutos, F. T., Freed, L. E., Guilak, F. A biomimetic three-dimensional woven composite scaffold for functional tissue engineering of cartilage. Nat Mater. 6 (2), 162-167 (2007).
- Schwarz, S., et al. Contactless vibrational analysis of transparent hydrogel structures using laser-Doppler vibrometry. Exp Mech. 60 (8), 1067-1078 (2020).
- Bhave, G., Colon, S., Ferrell, N. The sulfilimine cross-link of collagen IV contributes to kidney tubular basement membrane stiffness. Am J Physiol Renal Physiol. 313 (3), F596-F602 (2017).
- Fisher, R. F., Wakely, J. The elastic constants and ultrastructural organization of a basement membrane (lens capsule). Proc R Soc Lond B Biol Sci. 193 (1113), 335-358 (1976).
- Wisdom, K. M., et al. Covalent cross-linking of basement membrane-like matrices physically restricts invasive protrusions in breast cancer cells. Matrix Biol. 85-86, 94-111 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados