Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Labeling und Imaging Cells in der Zebrafisch Hinterhirn
In diesem Artikel
Erratum Notice
Zusammenfassung
Schlüssel zum Verständnis der morphogenetischen Prozesse, die der frühe Embryo Form ist die Möglichkeit, Bild-Zellen in hoher Auflösung. Wir beschreiben hier eine Methode zur Kennzeichnung einzelner Zellen oder kleine Gruppen von Zellen in ganzen Zebrafisch-Embryos mit Membran-bezogene Green Fluorescent Protein.
Zusammenfassung
Schlüssel zum Verständnis der morphogenetischen Prozesse, die die frühen Wirbeltierembryo Form ist die Möglichkeit, Bild-Zellen in hoher Auflösung. In Zebrafisch-Embryos, Injektion von Plasmid-DNA führt zu Mosaik-Ausdruck, so dass für die Visualisierung von einzelnen Zellen oder kleine Gruppen von Zellen
Protokoll
1.Microinjection
- Verdünnen Plasmid kodiert Membran gezielte Green Fluorescent Protein (mGFP, mit freundlicher Genehmigung von Richard Harland) zu einer Konzentration von 40 ng / ml. DNA wird durch Linearisierung Plasmid (mGFP/PCS2 +, mit freundlicher Genehmigung von Richard Harland) unter Verwendung des Qiagen Maxi Prep Kit vorbereitet. Die Zugabe von Phenolrot (verwässert 1 / 10 Volumen), um die Lösung bevorzugt, die Lösung zu visualisieren. Keep DNA-Lösung auf Eis.
- Unter einem Stereomikroskop, kalibrieren Sie die Injektionsnadel (90mm Glaskapillaren aus Narishinge Scientific; Kat.-Nr. NA-GD-1), indem Sie die Nadel auf einer abgestuften Rutsche und leichtes Antippen der Teil der Nadel, wo der Durchmesser beträgt 1 &mgr; mit einer Rasierklinge Klinge (ein Tipp von der richtigen Größe zu erhalten).
- Um die Nadel zu kalibrieren, um ein bekanntes Volumen (2nl) spritzen, verbinden Sie die Nadel auf die Mikroinjektion Geräten (PCI 100 Mikroinjektor; Harvard Apparatus) und Front füllen Sie die Nadel mit 0.5μl einer Lösung mit Phenolrot und Wasser auf ein Stück platziert von Paraffin. Die Lösung kann verzichtet mit einem Fußpedal werden. Stellen Sie den Druck bis 7 kPa und variieren Sie die Zeit (10msec 30msec), so dass es 250 Pedale nimmt zu verzichten 0.5μl der Lösung.
- Legen Sie das kalibrierte Nadel auf einem Streifen von Wachs in einem befeuchteten 100 mm Petrischale. Um befeuchten die Schüssel legen nassen Kimwipes an den Rändern (dies verhindert die Verdunstung der Lösung in die Nadel).
- Verwenden einer Mikropipette zu 0,5-1μl der DNA in die Nadel Ende (das Ende, das nicht kalibriert wurde) zu laden.
- Sobald die Lösung der Nadelspitze erreicht hat, verbinden Sie das Ende der Nadel, die für das Laden einer Mikroinjektion Apparat verwendet wurde. Passen Sie die Einstellungen auf dem Mikroinjektor auf ca. 2 nl / Injektion mit einem Fußpedal zu liefern.
- Als nächstes Position Zebrafischembryonen in der Mitte einer Petrischale mit Embryo Medium (1,0 ml Hanks Stock # 1, 0,1 ml Hanks Lager Nr. 2, 1,0 ml Hanks Stock # 4, 95,9 ml ddH 2 O, 1,0 ml Hanks Stock # 5 gefüllt , 1,0 ml frisches Hanks Stock # 6, Verwenden ca. 10 Tropfen 1 M NaOH auf pH 7,2).
- Sobald die Embryonen auf die gewünschte Stufe entwickelt haben, vorsichtig Griff das Chorion mit feinen Pinzetten und situieren die Nadel in den Bereich für die Injektion gezielt. Für breite Palette mGFP Ausdruck, injizieren DNA in den Dotter oder das Zytoplasma der 1CELL Bühne. Um Ausdruck mGFP auf eine kleinere Region beschränken, injizieren DNA an die 4 (oder mehr) Zellen der Bühne in das Zytoplasma einer einzelnen Zelle.
- Drücken Sie das Fußpedal der Mikroinjektion Apparate und liefern ca. 2 nl, sicherzustellen, dass die Lösung (sichtbar durch die Zugabe von Phenolrot) richtig gespritzt wird.
- Entfernen Sie vorsichtig die Nadel aus dem Embryo.
- Wiederholen Sie dies für alle Embryonen in der Petrischale.
- Lassen Embryonen bei 28,5 ° C zu entwickeln, um gewünschte Bühne.
- Dechorionate Embryonen durch leichtes Abziehen des Chorion mit feinen Pinzette (wenn Embryonen jünger als 24hpf sind, dechorionating in einem Glas-Petrischale erhöht die Überlebensrate der Embryonen).
- Entfernen Sie leere Chorion aus der Petrischale mit einer Glaspipette.
2. Imaging mGFP-markierten Vibratom Abschnitte
- Verwenden Sie eine Glaspipette an Embryonen in Fixativ (4% Paraformaldehyd in 1X Phosphat-Pufferlösung (PBS) zu transportieren. Fix Embryonen bei 4 ° C über Nacht.
- Wash Embryonen in 1X PBS 3 mal für jeweils 5 Minuten.
- Legen Sie die Embryonen in Petrischalen und mit feinen Pinzetten, entfernen Sie das Eigelb ohne Beschädigung des Embryos. Entfernen Sie so viel wie möglich Eigelb.
- Bereiten Sie 4% Low-Melt Agarose in H 2 O.
- Legen geschmolzene Agarose in Kunststoff Schneiden Form.
- Verwenden feinen Pinzette zu einem injizierten Embryo aus der Petrischale heben und zu platzieren und zu orientieren in der Agarose. Der Embryo muss zu einem Ende der Form positioniert werden.
- Sobald die Agarose ausgehärtet ist, verwenden Sie eine ebene Fläche Werkzeug, wie einem Spatel, bis die Agarose Block von der Form zu entfernen.
- Schneiden Sie das untere Ende des Agarose-Block mit einer Rasierklinge, so dass es gerade ist.
- Fügen Sie einen Tropfen Superkleber auf die Vibratom Bühne und platzieren Sie die Agarose-Block auf sie (die Seite des Blocks, die aus mit dem Rasiermesser wurde geebnet sollte der Kleber face). Für Schnitte mehrere Embryonen, mount mehrere Agarose eingebettete Proben auf der Bühne.
- Bringen Sie die Vibratom Bühne, um die Vibratom (Vibratom, Inc.).
- Stellen Sie die Parameter des Vibratom (allgemeine Einstellungen: speed-3, Dicke-50 um). Wenn Schnitte mehrere Embryonen, richten Agarose Blöcke auf der Bühne und sicherzustellen, dass die Klinge durch den ganzen Embryo geht aber nicht über die gesamte Breite der Agarose-Blöcke (wie die Abschnitte, die dem gleichen Embryo zusammen bleiben).
- Gießen 1X PBS in der Vibratom Bühne.
- Section.
- Sobald die Embryonen Sectioned, entfernen Sie die Agarose-Block (s) aus dem Vibratom Bühne.
- Sorgfältig trennen Sie die einzelnen Kapitel, indem die Rückseite der Agarose-Block (s).
- Verwenden feinen Pinzette gewünschten Abschnitte und in einen 1X PBS gefüllten Multi-Well-Platte (mit unterschiedlichen Bohrungen für die Abschnitte, die den verschiedenen Embryonen) zu wählen.
- Wiederholen Sie für jeden Embryo.
- Fahren Sie mit Antikörpermarkierung, wenn die Schritte 19 23, zu mGFP Bildgebung (dieser Schritt nicht notwendig sein, wenn Abschnitte innerhalb eines Tages oder zwei der Vorbereitung sind abgebildet) zu verbessern.
- Gönnen Abschnitte für 30 Minuten bei Raumtemperatur mit Blockierungslösung in 1X PBS, um unspezifische Bindung zu blockieren.
- Inkubieren Abschnitte mit primärem Antikörper (Kaninchen a-GFP, Invitrogen Kat. Nr. A11122) verdünnt @ 1:200 in Blocking-Lösung auf einem Schüttler für 5 Stunden bei Raumtemperatur oder über Nacht bei 4 ° C.
- Wash viermal (10 min, 15 min, 30 min und 60 min, jeweils) in 1% BSA/1X PBS / 1% DMSO-Lösung.
- Gönnen Abschnitte mit entsprechenden Sekundär-Antikörper in Blocking-Lösung für 5 Stunden bei Raumtemperatur oder über Nacht bei 4 ° C.
- Wash viermal (10 min, 15 min, 30 min und 60 min, jeweils) in 1% BSA/1X PBS / 1% DMSO-Lösung.
- Behandeln Sie die Abschnitte mit DAPI (0,1% DAPI in 50 ml 1X PBS, Invitrogen Kat. Nr. D1306) für 30 Minuten, um die Kerne zu färben.
- Dreimal in 1X PBS für jeweils 5 Minuten.
- Berg auf dem Objektträger.
- Bild mit Hilfe der konfokalen Mikroskopie.
3. Live-Bildgebung
- Dechorionate leben, injizierten Embryonen 30 Minuten, bevor sie das gewünschte Entwicklungsstadium. Legen Sie die Embryonen in sedative Lösung (0,01% Tricaine in Embryo Medium) für 15 min.
- Geben Sie einen Tropfen 1% Agarose auf einem 35mm Glasboden Kulturschale (MatTek; Art.-Nr. P35G-o-20-C).
- Lassen Agarose verfestigt.
- Hitze Kapillare über Flamme 3 Löcher in Agarose (andere Objekte wie Mikropipettenspitzen verwendet werden, um die Löcher zu machen) zu machen. Stellen Sie sicher, dass es keine Rest-Agarose in das Loch unten.
- Füllen Sie die Petrischale mit Embryo Medium.
- Verwenden Sie eine Glaspipette, den Embryo in das Loch Platz.
- Als nächstes Orientierung des Embryos mit feinen Pinzetten, so dass die Fläche abgebildet werden (dorsal Kopf) ist in Kontakt mit dem Glas.
- Vorsichtig transportieren die Petrischale auf dem Mikroskoptisch (legen Sie den Deckel über der Petrischale, um Verdunstung zu verhindern).
- Während Bildgebung, sicherzustellen, dass die Embryonen bei 28,5 ° C liegen
4. Ergebnisse
Wir beschreiben hier eine einfache Lösung, um Bild Einzelzellen im Zebrafisch Neuralrohr mit Mosaik Ausdruck. Trotz der optischen Transparenz des Zebrafisch-Embryos, haben wir festgestellt, dass die Visualisierung von Nervenzellen stark in Querschnitten unter Verwendung eines Vibratom verbessert. Diese Abschnitte sind dick (50 mm) ermöglicht für die Bildgebung von Zell-Erweiterung aus einer Zelle in verschiedenen Brennebenen mit einem konfokalen Mikroskop. Ergebnisse mit dieser Methode wurden an anderer Stelle 2, veröffentlicht, aber repräsentative Bilder von Zellen in das Neuralrohr eines WT Embryo (Abb. 1) und ein N-Cadherin (N-cad)-Mutante (Abbildung 2) sind vorhanden. Letzteres zeigt, dass Zellen in seitlichen Bereichen der Neuralplatte in N-cad-Mutanten nicht richtig zu orientieren in Richtung der Mittellinie (Abbildung 2). Im Gegensatz zu diesem Mosaik Etikettierung ist Immunfärbung der Neuroepithel mit einer allgemeinen Zelloberflächenmarker wie Beta-Catenin nicht zulassen, dass die Morphologie der einzelnen Zellen visualisiert werden (Abb. 3).
Zeitraffer-Aufnahme von Live-Embryonen ergänzt das Mosaik Kennzeichnung von festen Proben und ermöglicht ein besseres Verständnis der zellulären Dynamik, die während Neurulation. Movie 1 ergab, dass WT-Zellen aktiv an der Mittellinie wandern durch die Ausweitung medial orientierten Membran Vorsprünge.
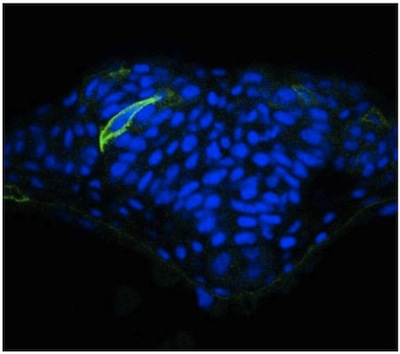
Abbildung 1. MGFP Ausdruck in das Neuralrohr eines WT Zebrafischembryo.

Abbildung 2. MGFP Ausdruck in das Neuralrohr eines N-Cadherin Zebrafischembryo.
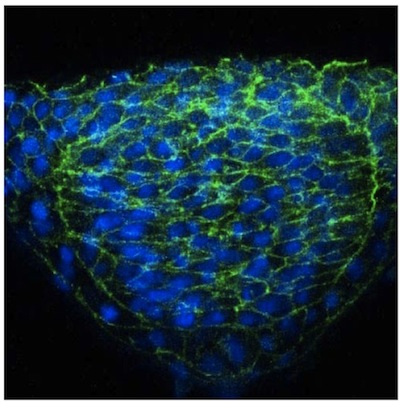
Abbildung 3. B-Catenin Immun-Färbung der Neuroepithel.
Diskussion
Abschließend beschrieb die Kennzeichnung Techniken hier erlauben einzelne Zelle Analyse von morphogenetischen Prozesse im Zebrafisch Embryo. Der primäre Schwerpunkt dieses Protokoll wird auf Methoden zur Darstellung von markierten Zellen in das Neuralrohr mit mGFP unter der Kontrolle eines ubiquitären Promoters. Weitere Anwendungen dieser transiente Expression Assays sollte sich der Leser zu einer neueren Arbeit von Andersen et al verweisen. 3.
Offenlegungen
Danksagungen
Diese Arbeit wurde durch ein NIH Zuschuss für R. Brewster (1R01GM085290-01A1) unterstützt.
Materialien
Lösungen
- Hank Stock # 1: 8,0 g NaCl, 0,4 g KCl in 100 ml dd H 2 O
- Hank Stock # 2: 0,358 g Na 2 HPO 4 Wasserfreies, 0,60 g KH 2 PO 4 in 100 ml ddH 2 O
- Hank Stock # 4: 0,72 g CaCl 2 in 50 ml ddH 2 O
- Hank Stock # 5: 1,23 g MgSO 4 x7H 2 O in 50 ml dd H 2 O
- 1 X-Phosphat-Puffer: 0,8% NaCl, 0,02% KCl, 0,02 M PO4, pH 7,3
- Blocking Solution: 2% normalem Ziegenserum, 1% Rinderserumalbumin (BSA), 1% Dimethylsulfoxid (DMSO)
Referenzen
- Nüsslein-Volhard, C., Dahm, R. . Zebrafish Practical approach series. , (2002).
- Hong, E., Brewster, R. N-cadherin is required for the polarized cell behaviors that drive neurulation in the zebrafish. Development. 133, 3895-3905 (2006).
- Andersen, E., Asuri, N., Clay, M., Halloran, M. Live Imaging of Cell Motility and Actin Cytoskeleton of Individual Neurons and Neural Crest Cells in Zebrafish Embryos. J Vis Exp. , (2010).
Erratum
Formal Correction: Erratum: Labeling and Imaging Cells in the Zebrafish Hindbrain
Posted by JoVE Editors on 9/16/2010. Citeable Link.
A correction was made to Labeling and Imaging Cells in the Zebrafish Hindbrain. There was an error in the authors affiliations. The authors have been corrected to:
Pradeepa Jayachandran1, Elim Hong2, Rachel Brewster1
1Department of Biological Sciences, University of Maryland, Baltimore County
2Center for Neuroscience, Children's National Medical Center
instead of:
Pradeepa Jayachandran1, Elim Hong2, Rachel Brewster2
1Center for Neuroscience, Children's National Medical Center
2Department of Biological Sciences, University of Maryland Baltimore County
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten