Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Etiquetado y las Pilas de imágenes en la parte posterior del cerebro de pez cebra
En este artículo
Erratum Notice
Resumen
La clave para entender los procesos morfogenéticos que forma el embrión temprano es la capacidad de las células de la imagen en alta resolución. Se describe aquí una técnica para el etiquetado de las células individuales o pequeños grupos de células en embriones de pez cebra con toda la membrana dirigidos proteína verde fluorescente.
Resumen
La clave para entender los procesos morfogenéticos que forma el embrión de vertebrados temprana es la capacidad de las células de la imagen en alta resolución. En embriones de pez cebra, la inyección de los resultados de ADN plásmido en la expresión del mosaico, lo que permite la visualización de células individuales o pequeños grupos de células
Protocolo
1.Microinjection
- Diluir plásmido que codifica la membrana dirigidos proteína verde fluorescente (mGFP, cortesía de Richard Harland) a una concentración de 40 ng / ml. El ADN es preparado por linearización del plásmido (mGFP/PCS2 +, cortesía de Richard Harland) purificado utilizando el kit Qiagen Maxi Prep. Además de rojo fenol (diluido volumen 1 / 10 en total) a la solución es preferible a visualizar la solución. Mantener la solución de ADN en el hielo.
- Bajo un microscopio estereoscópico, calibrar la aguja de inyección (90 mm capilares de vidrio de Narishinge científica; Cat. N º NA-GD-1) por la alineación de la aguja sobre un portaobjetos de graduado y golpeando suavemente la parte de la aguja cuando el diámetro es 1μm con una navaja limpia hoja (para obtener una punta del tamaño adecuado).
- Con el fin de calibrar la aguja para inyectar un volumen conocido (2NL), conectar la aguja con el aparato de microinyección (PCI 100 microinyector, Harvard Apparatus) y frente a llenar la aguja con 0.5μl de una solución de rojo fenol y agua colocado en una pieza de la parafina. La solución se puede prescindir de utilizar un pedal. Ajuste la presión a 7 kPa y variar el tiempo (10 ms 30msec) para que tenga 250 pedales para dispensar 0.5μl de la solución.
- Coloque cuidadosamente la aguja calibrada en una tira de cera en un humidificado 100 mm placa de Petri. Para humedecer el plato, coloque Kimwipes húmeda alrededor de los bordes (esto evita la evaporación de la solución en la aguja).
- Use una micropipeta para carga de 0.5 1μl del ADN en el extremo de la aguja (el extremo que no fue calibrado).
- Una vez que la solución ha llegado a la punta de la aguja, conecte el extremo de la aguja que se usó para la carga a un aparato de microinyección. Ajustar la configuración de la microinyector para entregar aproximadamente 2 nl / inyección con un pedal.
- A continuación, los embriones de la posición del pez cebra en el centro de una placa de Petri llena con un medio de embriones (Foto 1.0 ml de Hank # 1, 0,1 ml de Hank Foto # 2, de archivo de 1,0 ml de Hank # 4, 95,9 ml ddH2O, archivo 1,0 ml de Hank # 5 , 1,0 ml de archivo fresco de Hank º 6, el uso de 10 gotas de NaOH 1 M a pH 7,2).
- Una vez que los embriones se han desarrollado hasta el punto deseado, suavemente agarre el corion con unas pinzas finas y situar la aguja en el área destinada a la inyección. Para la expresión de una amplia gama mGFP, inyectar ADN en el citoplasma de la yema de huevo o en la etapa de 1cell. Para limitar la expresión de mGFP a una región más pequeña, se inyecta el ADN en las cuatro (o más) etapa de las células en el citoplasma de una célula.
- Presione el pedal del aparato de microinyección y entregar aproximadamente 2 nl, asegurando que la solución (visible debido a la adición de fenol rojo) está bien inyectada.
- Retire con cuidado la aguja del embrión.
- Repita este procedimiento para todos los embriones en la placa de Petri.
- Permita que los embriones para desarrollar a 28,5 ° C a la etapa deseada.
- Embriones Dechorionate suavemente quitando el corion con unas pinzas finas (si los embriones son más jóvenes que 24hpf, dechorionating en una placa de Petri de vidrio aumenta la tasa de supervivencia de los embriones).
- Quitar corion vacío de la placa de Petri con una pipeta de vidrio.
2. Imágenes mGFP marcado con secciones vibratome
- Use una pipeta de vidrio para el transporte de los embriones en el fijador (4% de paraformaldehído en solución 1X tampón fosfato (PBS). Fix embriones a 4 ° C durante la noche.
- Lavado de los embriones en 1X PBS tres veces durante 5 minutos cada uno.
- Colocar los embriones en una placa Petri y, usando unas pinzas finas, eliminar la yema sin dañar el embrión. Retire como la yema de lo posible.
- Prepare un 4% de agarosa de bajo punto de fusión de H 2 O.
- Lugar agarosa fundida en el molde de plástico de seccionamiento.
- Use unas pinzas finas para levantar un embrión inyectado desde la placa de Petri y colocar y orientar en la agarosa. El embrión debe ser colocado en un extremo del molde.
- Una vez que la agarosa se ha endurecido, usar una herramienta de superficie plana, como una espátula, para quitar el bloqueo de agarosa del molde.
- Corte el extremo inferior del bloque de agarosa con una cuchilla de afeitar para que sea uniforme.
- Añadir una gota de pegamento a la etapa de vibratome y colocar el bloque de agarosa en el mismo (el lado del bloque que se estabilizó con la navaja debe enfrentarse a la cola). Para seccionar varios embriones, montar varios de agarosa incrustados de la etapa.
- Conecte la etapa vibratome a la vibratome (Vibratome, Inc.).
- Establezca los parámetros de la vibratome (conjunto de valores: 3 velocidades, 50 micras de espesor). Si el corte de múltiples embriones, alinear bloques de agarosa en el escenario y asegurarse de que la hoja pasa por todo el embrión, pero no a través de todo el ancho de los bloques de agarosa (por ejemplo, que las secciones correspondientes al embrión mismo estar juntos).
- Vierta 1X PBS en la etapa de vibratome.
- Sección.
- Una vez que los embriones son sectioned, quitar el bloque de agarosa (s) de la etapa vibratome.
- Separe cuidadosamente cada sección mediante la reducción de la parte trasera del bloque de agarosa (s).
- Use unas pinzas finas para seleccionar las secciones que desee y coloque en un 1X PBS lleno de múltiples y plato (pozos de uso diferente para las secciones que corresponden a diferentes embriones).
- Repita este procedimiento para cada embrión.
- Continuar con el etiquetado de anticuerpos, aunque los pasos 19 23, para mejorar la imagen mGFP (este paso puede no ser necesario si las secciones son expuestas en un día o dos de preparación).
- El tratamiento de las secciones durante 30 minutos a temperatura ambiente con solución de bloqueo en 1X PBS para bloquear la unión no específica.
- Secciones se incuban con el anticuerpo primario (un conejo-GFP, Invitrogen Cat. No. A11122) diluido 1:200 @ en la solución de bloqueo en un agitador durante 5 horas a temperatura ambiente o durante la noche a 4 ° C.
- Lavar cuatro veces (10 min, 15 min, 30 min y 60 min, cada uno) en el 1% BSA/1X PBS / 1% de solución de DMSO.
- El tratamiento de las secciones con el anticuerpo adecuado secundaria en solución de bloqueo durante 5 horas a temperatura ambiente o durante la noche a 4 ° C.
- Lavar cuatro veces (10 min, 15 min, 30 min y 60 min, cada uno) en el 1% BSA/1X PBS / 1% de solución de DMSO.
- El tratamiento de las secciones con DAPI (0,1% en 50 ml de DAPI 1X PBS, Invitrogen Cat. No. D1306) durante 30 minutos para teñir los núcleos.
- Lavar tres veces en PBS 1X durante 5 minutos cada uno.
- Montaje de la diapositiva.
- Imagen de microscopía confocal.
3. Imágenes en vivo
- Dechorionate embriones vivos, se inyecta 30 minutos antes de llegar a la etapa de desarrollo deseado. Colocar los embriones en la solución de sedantes (0,01% tricaína en medio de embriones) durante 15 minutos.
- Coloque una gota de agarosa al 1% en una placa de cultivo de vidrio de 35 mm de fondo (MatTek; N º de P35G-o-20-C).
- Permitir agarosa que se solidifique.
- El calor del tubo capilar sobre la llama para hacer tres agujeros en agarosa (otros objetos como puntas de micropipeta se puede utilizar para hacer los agujeros). Asegúrese de que no hay residuos de agarosa en el fondo del agujero.
- Llene la caja de Petri con medio de embriones.
- Use una pipeta de vidrio para colocar el embrión en el agujero.
- A continuación, orientar el embrión utilizando unas pinzas finas para que el área a explorar (la cabeza dorsal) está en contacto con el vidrio.
- Cuidadosamente el transporte de la placa de Petri de la platina del microscopio (coloque la tapa sobre la placa de Petri para evitar la evaporación).
- Mientras que imágenes, asegúrese de que los embriones son de 28,5 ° C.
4. Resultados
Se describe aquí un acercamiento directo a las células de una sola imagen en el tubo neural del pez cebra con la expresión del mosaico. A pesar de la transparencia óptica del embrión de pez cebra, hemos encontrado que la visualización de las células neuronales es mucho mayor en las secciones transversales obtenidas mediante un vibratome. Estas secciones son de espesor (50 mm) que permite obtener imágenes de la extensión celular de una célula en diferentes planos focales con un microscopio confocal. Resultados utilizando esta metodología han sido publicados en otra 2, pero las imágenes representativas de las células en el tubo neural de un embrión de WT (Figura 1) y un N-cadherina (N-cad) mutante (Figura 2) se proporcionan. La última revela que las células en las regiones laterales de la placa neural en la N-cad mutantes no orientan adecuadamente hacia la línea media (Figura 2). En contraste con este documento de mosaico, el neuroepitelio inmunotinción con un marcador de superficie celular en general, tales como beta-catenina no permite la morfología de las células individuales para ser visualizado (Figura 3).
Imagen de lapso de tiempo de embriones vivos complementa el etiquetado mosaico de muestras fijas, lo que permite una mejor comprensión de la dinámica celular que tienen lugar durante la neurulación. Película 1 reveló que las células WT activamente migrar hacia la línea media mediante la ampliación medial orientada protuberancias de la membrana.
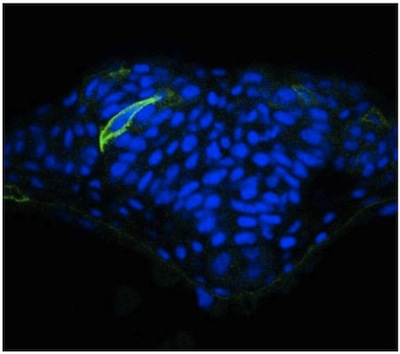
Figura 1. MGFP expresión en el tubo neural de un embrión de pez cebra WT.

Figura 2. MGFP expresión en el tubo neural de un embrión de pez cebra N-cadherina.
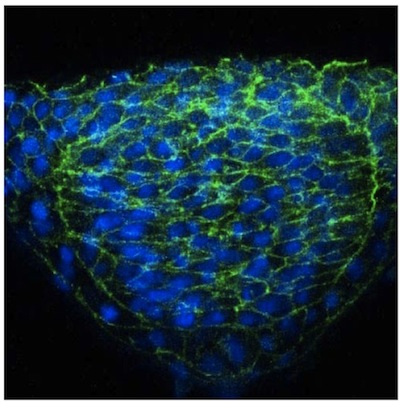
Figura 3. B-catenina inmuno tinción de la neuroepithelium.
Discusión
En conclusión, las técnicas de etiquetado descritos aquí permiten el análisis de células individuales de los procesos morfogenéticos en el embrión de pez cebra. El énfasis principal de este protocolo es sobre los métodos para que las células de imágenes etiquetadas en el tubo neural con mGFP bajo el control de un promotor en todas partes. Para otras aplicaciones de este ensayo de expresión transitoria lectores deben referirse a un documento reciente de Andersen et al. 3.
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por una subvención del NIH otorgado a R. Brewster (1R01GM085290-01A1).
Materiales
Soluciones
- Foto de Hank # 1: 8,0 g de NaCl, 0,4 g de KCl en 100 ml dd H 2 O
- Foto de Hank # 2: 0,358 g de Na 2 HPO 4 anhidro, 0,60 g KH 2 PO 4 en 100 ml ddH2O
- Foto de Hank # 4: 0,72 g de CaCl2 en 50 ml ddH2O
- Foto de Hank # 5: 1,23 g MgSO4 x7H 2 O en 50 ml dd H 2 O
- 1 X tampón fosfato: 0,8% NaCl, KCl 0,02%, 0,02 M PO4, pH 7,3
- Solución de bloqueo: 2% de suero normal de cabra, 1% de albúmina sérica bovina (BSA), dimethysulfoxide 1% (DMSO)
Referencias
- Nüsslein-Volhard, C., Dahm, R. . Zebrafish Practical approach series. , (2002).
- Hong, E., Brewster, R. N-cadherin is required for the polarized cell behaviors that drive neurulation in the zebrafish. Development. 133, 3895-3905 (2006).
- Andersen, E., Asuri, N., Clay, M., Halloran, M. Live Imaging of Cell Motility and Actin Cytoskeleton of Individual Neurons and Neural Crest Cells in Zebrafish Embryos. J Vis Exp. , (2010).
Erratum
Formal Correction: Erratum: Labeling and Imaging Cells in the Zebrafish Hindbrain
Posted by JoVE Editors on 9/16/2010. Citeable Link.
A correction was made to Labeling and Imaging Cells in the Zebrafish Hindbrain. There was an error in the authors affiliations. The authors have been corrected to:
Pradeepa Jayachandran1, Elim Hong2, Rachel Brewster1
1Department of Biological Sciences, University of Maryland, Baltimore County
2Center for Neuroscience, Children's National Medical Center
instead of:
Pradeepa Jayachandran1, Elim Hong2, Rachel Brewster2
1Center for Neuroscience, Children's National Medical Center
2Department of Biological Sciences, University of Maryland Baltimore County
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados