Method Article
Visualisierung der mitochondrialen DNA-Replikation in einzelnen Zellen von Edu Signalverstärkung
In diesem Artikel
Zusammenfassung
Wir entwickelten eine empfindliche Technik zur neu synthetisierten mitochondrialen DNA (mtDNA) in einzelne Zellen markieren, um mtDNA Biogenese zu studieren. Die Technik verbindet die Einbeziehung von EDU zusammen mit einem Tyramid Signalverstärkung (TSA) Protokoll, um mtDNA Replikation innerhalb subzellulären Kompartimenten von Neuronen zu visualisieren.
Zusammenfassung
Mitochondrien sind wichtige Regulatoren der zellulären Energie-und mitochondrialen Biogenese ist ein wesentlicher Bestandteil der Regulierung Mitochondrien Zahlen in gesunden Zellen 1-3. Ein Ansatz für die Überwachung der mitochondrialen Biogenese ist die Rate der mitochondrialen DNA (mtDNA) Replikation 4 zu messen. Wir entwickelten eine empfindliche Technik zur neu synthetisierten mtDNA in einzelne Zellen markieren, um mtDNA Biogenese zu studieren. Die Technik vereint die Aufnahme von 5-Ethinyl-2'-desoxyuridin (EDU) 5-7 mit einem Tyramid Signalverstärkung (TSA) 8 Protokoll zur mtDNA-Replikation in subzellulären Kompartimenten von Neuronen zu visualisieren. Edu ist besser als bei anderen Thymidin-Analoga, wie 5-Bromo-2-desoxyuridin (BrdU), weil die ersten Klick Reaktion auf Etikett EdU 5-7 nicht erforderlich ist die harte Säure-Behandlungen oder Enzym verdaut, dass für das Freilegen der BrdU-Epitop erforderlich sind . Die milderen Kennzeichnung EdU ermöglicht den direkten Vergleich ihrer Gründung mit anderen zellulären Marker 9-10. Die Fähigkeit zu visualisieren und zu quantifizieren mtDNA Biogenese bietet ein wesentliches Instrument für die Untersuchung der Mechanismen zur mitochondrialen Biogenese regulieren und einen Einblick in die Pathogenese mit Medikamententoxizität, Alterung, Krebs und neurodegenerativen Erkrankungen verbundene Dienstleistungen zu erbringen. Unsere Technik ist für sensorische Neuronen sowie andere Zelltypen. Der Einsatz dieser Technik auf mtDNA Biogenese Maßnahme hat erhebliche Auswirkungen in der Förderung des Verständnisses der beiden normalen zellulären Physiologie sowie beeinträchtigt Erkrankungen.
Protokoll
1. Vorbereitung des Neurons
- Spinalganglion (DRG) Neuronen sind sterile (autoklaviert) 12 mm Deckgläschen in einer 24-well Platte gewachsen.
- Die 10 mM Bestand von Edu (in DMSO, Click-iT EdU Microplate Assay Kit) ist in der Regel 1:100 in Kulturmedium verdünnt, um eine 10X EdU-Lösung (100 nM) zu machen und dann 1:10 verdünnt in die Kultur-Wells (z. B. 30 ul der 10X EdU Lösung in insgesamt 300 ul Kulturmedium). DRG-Neuronen sind mit einer Endkonzentration von 10 uM EdU bei 37 ° C und 5% CO 2 zwischen 2-24 Stunden. Die Länge der Zeit abhängt, wie Behandlungen auf die Rate der mtDNA-Synthese.
- In einer Abzugshaube, sind DRG-Neuronen in 2% Paraformaldehyd für 10-15 min bei Raumtemperatur fixiert, zweimal in 1X PBS 2-5 Minuten jede Wäsche und in frischen 1X PBS für bis zu 1 Monat bei 4 ° C.
- Deckgläser mit spitzen Pinzette, eine vorbereitete feuchte Kammer übertragen (Corning BioAssay Gericht mit schwarzen tapeziert unten für Kontrast, eingewickelt in Alufolie, um Licht empfindliche Bauteile und befeuchtet mit Wasser gesättigt Kimwipes zu schützen) und auf ein Blatt Parafilm M, bietet eine hydrophobe Oberfläche, um die Lösungen zu den Deckglas beschränken. Das Protokoll unten ist für 28 Deckgläser konzipiert und Lösungen sind für 32 Deckgläser (ca. 14% mehr Volumen) vorbereitet. Lösungen mit teuren oder beschränkt Komponenten werden so gefertigt, dass 75-80 ul auf jedem Deckglas verwendet werden. Ansonsten sind 200-300 ul für Wasch-und Block-Lösungen verwendet werden, um gründlich auswaschen bisherigen Lösungen.
- Re-gelten 1X PBS, um die Oberfläche jedes Deckglas (200-300 ul) zu decken. Bereiten Sie die Click-iT-Assay und Tyramid Signalverstärkung Kits, wie unten beschrieben und in den Anweisungen des Herstellers.
Vorbereitung der Click-iT EdU Microplate Assay Kit
Die meisten Komponenten aus dem Click-iT EdU Microplate Assay Kit kommen vor und sind bei 4 ° C [2x Click-iT Reaction Buffer (10x Komponente E), CuSO4 (100 mM, Component F), Click-iT EdU Fixativ ( Die Komponente D) und Blocking Buffer (2x Component H)]. Click-iT EdU Buffer Additive (10x Komponente G) wird bei -20 ° C gelagert, um es von sich gelb-braun im Laufe der Zeit zu verhindern. Diese Komponente, wiederholtes Einfrieren und Auftauen.
Die Oregon Green 488 Azid (Komponente B) sollte in kleinen Portionen (10-20 ul) geteilt werden, um Frost-Tau-Zyklen zu minimieren und bei -20 ° C.
Zur Vorbereitung einer Stammlösung von der Anti-Oregon Green HRP-Konjugat (Komponente I), fügen Sie 75 ul Milli Q dH 2 O in das Fläschchen. Mix von sanften Pipettieren oder durch Umdrehen Schaumbildung zu vermeiden und lagern bei 4 ° C. Nicht vortexen.
Vorbereitung der TSA Kit Nr. 12, mit HRP Goat Anti-Rabbit IgG und Alexa Fluor 488 Tyramid Kit
Zur Vorbereitung der Tyramid Stammlösung lösen sich die festen Stoffe (Alexa Fluor 488 Tyramid, Komponente A) in 150 ul DMSO (Komponente B). Drehen Sie die Durchstechflasche einige Male zu lösen jede Tyramid Beschichtung den Seiten des Fläschchens. Lagerung der Stammlösung in kleinen Portionen (10-20 ul) bei ≤ -20 ° C, getrocknet und vor Licht geschützt.
2. Click-iT 5-Ethinyl-2-desoxyuridin (EDU) Labeling
Hinweis: Alle Lösungen sind mit einer Glühbirne Transferpipette mit 200 ul Spitze angebracht, um das Ende zu entfernen sanft Flüssigkeiten ohne Zellen entfernt. Vermeiden Sie die Verwendung eines Vakuums Linie, die in der Regel zu entfernen Lösungen zu heftig. Ein Vollpipette wird verwendet, um sanft Flut Deckgläser mit 200-300 ul der Waschlösungen gründlich auswaschen der bisherigen Lösung.
- Die Zellen werden mit 0,1% Triton-X-100 in 1X PBS-Lösung für 10 Minuten bei Raumtemperatur in einer feuchten Kammer bedeckt permeabilisiert. Verwenden Sie 1% Triton-X-100 Stammlösungen.
- Machen Sie 3000 &mgr; l 0,1% Triton-X-100 durch Zugabe von 300 ul 1% Triton-X-100 Lager zu 2700μL 1X PBS.
- Verwenden 75-80 mL pro Deckglas.
- Entfernen Sie den Triton X-100-Lösung und spülen Sie zweimal mit 1X PBS.
- Endogene Peroxidase-Aktivität wird durch eine 1% H 2 O 2 in 1X PBS-Lösung für 30 Minuten bei Raumtemperatur abgeschreckt. Verdünnen Sie 30% H 2 O 2-Lösung mit 1X PBS. Diese Lösung sollte frisch zubereitet werden, sondern können während der 10 Minuten Permeabilisierung Schritt vor (Schritt 3,1) vorgenommen werden.
- Machen Sie 3000 ul einer 1% igen H 2 O 2 durch Zugabe von 100 ul 30% H 2 O 2 bis 2900 ul 1X PBS.
- Verwenden 75-80 mL pro Deckglas.
- Entfernen Sie die Peroxidase-Lösung und spülen Sie zweimal mit 1X PBS.
- Click-iT EdU Reaction
- Für 32 Deckgläser (32 x 80 ul =2560 ul) insgesamt von 2560 ul erforderlich ist. Die 2x Reaktion ist in 1280 ul, der die Hälfte des Gesamtvolumens der Reaktion ist.
- Frisch bereitet die Click-iT Reaction Cocktail vor dem Start des 5 Minuten post fix (Schritt 3.5).
- Mischen Sie die Cocktail-und Abpipettieren. Nicht vortexen bei der Bereitstellung dieser Reaktion.
2 x Reaction Cocktail
Die Komponenten sind von Click-iT EdU Microplate Assay KitDie Hälfte Volume: 1280 ul Milli Q dH 2 O 1132,8 ul 2x Click-iT Reaction Buffer (10x Komponente E) 100,3 ul Click-iT EdU Buffer Additive (10x Komponente G) 25,6 ul CuSO 4 (100 mM, Component F) 25,6 ul Oregon Green Azid (Komponente A) 6,4 ul Gesamt 1290,7 ul
HINWEIS: Die Volumina eingesetzt oben sind proportional zu den Volumina in den Click-iT EdU Microplate Assay Kit Richtungen aufgeführt. Das Endvolumen der Reaktion Cocktail ist etwas mehr als 1280 ul. - Der 2X Click-iT Reaktion wird mit dem gleichen Volumen von Click-iT EdU Fixativ (Komponente D) unmittelbar vor der Verwendung verdünnt. Das Mischen dieser zusammen macht eine einheitliche Reaktion Lösung für alle Deckgläser.
- Add 1290,7 uL Click-iT EdU Fixativ (Komponente D) auf die 1290,7 ul Reaktion Cocktail. Verwenden 75-80 mL pro Deckglas.
- Post-fix die Neuronen mit Click-iT EdU Fixativ (Komponente D) für 5 Minuten bei Raumtemperatur.
- Entfernen Sie fix und fügen Reaktion Cocktail von oben (Schritt 3,4). Abdeckung zum Schutz vor Licht und Inkubation für 25 Minuten bei Raumtemperatur. Entfernen Sie vorsichtig die Reaction Cocktail. Zweimal waschen mit verdünnter 1X Blocking Buffer (2x Component H).
- Machen Sie 5200 ul 1X Blocking Buffer durch Verdünnung 2600 ul Blocking Buffer (2x Component H) mit dem gleichen Volumen MilliQ d H 2 O (2600 ul). Verwenden 75-80 mL pro Deckglas.
- Im Rahmen eines epi-Fluoreszenz-Mikroskop für Oregon Green Färbung in den Kernen von mitotisch aktiven Kontroll-Zellen zu überprüfen.
3. Tyramid Signalverstärkung (TSA) der EDU Signal
- Add 1% TSA-Block-Lösung in jede Deckglas und Inkubation für 30 Minuten bei Raumtemperatur.
- Machen Sie 6000 ul 1% TSA-Block-Lösung durch Wiegen 0,06 g TSA Blocking Reagent (TSA-Kit Nr. 12, Komponente D) und das Hinzufügen zu 6000 ul 1X PBS. Vortex mischen. Zugabe von 5% Ziegenserum in der 1%-TSA-Block-Lösung wird oft dazu beitragen, unspezifische Bindung zu reduzieren.
- Kurz Spin der Anti-Oregon Green Meerrettichperoxidase konjugierten Antikörper Lager (Komponente I der Click-iT EdU Microplate Assay), hergestellt 2-24 Stunden im Voraus von TSA. Verdünnen Sie den Primärantikörper 1:300 in 1% TSA blockiert und Invertzuckersirup oder Pipette auf und ab sanft zu mischen. Nicht vortexen zu verhindern, dass die HRP-konjugierten Antikörper. Nehmen Sie 1% TSA-Block-Lösung, fügen Sie 75 &mgr; l jedes Deckglas beimpft und über Nacht bei 4 ° C.
- Der verwendete Antikörper können mehr konzentriert werden, wie z. B. 1:150, aber es ist in begrenzten Mengen geliefert, so sollten Sie sie sparsam. Auch hier wird der Zugabe von 5% Ziegenserum in der 1%-TSA-Block-Lösung helfen oft unspezifische Bindung des anti-Oregon Green Meerrettichperoxidase konjugierten Antikörper zu reduzieren.
- Machen Sie 2560 ul einer 1:300-Antikörper-Lösung durch Zugabe von 8,53 ul der Aktie Rabbit anti-Oregon Green-HRP (Click-iT EdU Microplate Assay) bis 2560 ul 1% TSA Blocking. Mix von sanften Pipettieren oder Inversion.
- Am nächsten Tag, entfernen Sie die Antikörper-Lösung und spülen Sie dreimal mit 1X PBS. Inkubieren Sie die Deckgläser in der letzten Wäsche für eine weitere 30-60 Minuten bei Raumtemperatur, um sicherzustellen, dass ungebundene Primärantikörper entfernt wird.
- Bereiten Tyramid Reaktion von 2560 ul (32 Deckgläser x 80 mL).
- Machen Sie 400 ul einer 0,15% H 2 O 2-Lösung (100X) durch Zugabe von 2 ul der 30% H 2 O 2 (TSA-Kit Nr. 12, Component F) bis 398 ul Amplification Buffer (TSA-Kit Nr. 12, Komponente E) . Dies sollte unmittelbar vor erforderlich sein.
- Für ein Endvolumen von 2560 ul kombinieren:
- 25,6 ul Tyramid-488 (TSA-Kit Komponente A)
- 2508,8 uL Amplification Buffer (TSA-Kit Komponente E)
- 25,6 ul 0,15% H 2 O 2 (von oben für eine endgültige 0,0015% H 2 O 2)
- Entfernen 1X PBS und Inkubation mit dem Tyramid Reaktion für 15 Minuten bei Raumtemperatur.
- Entfernen Tyramid Reaktion und spülen dreimal mit 1X PBS, Inkubation in der letzten Wäsche für 30-60 Minuten bei Raumtemperatur wie zuvor.
- Prüfen Sie, ob mtDNA Kennzeichnung unter einem epi-Fluoreszenz-Mikroskop.
- Berg Deckgläser auf Objektträger aus Glas mit einem Antifade Eindeckmedium, wie ProLong Gold mit DAPI. Alternativ kann der EDU Kennzeichnung und Verstärkung durch Standard-Leuchtstofflampen Immunzytochemie zu anderen zellulären Marker Etikett beachten.
4. Repräsentative Ergebnisse: Visualisierung der mitochondrialen DNA-Replikation als Marker für die mitochondrialen Biogenese
Wir sind daran interessiert, wie sensorische Neuronen (Abbildung 1) regelt die Anzahl der Mitochondrien. Dieses Protokoll wird neu synthetisierten mtDNA mit einem fluoreszierenden Marker-Label als eine Möglichkeit zur Messung der neuen Mitochondrien. Die Einbeziehung der synthetische Nukleotid EdU durch die Klick-Chemie der Oregon Green-Azid und nachfolgenden Amplifikation mit den Alexa-Fluor 488 Tyramid Ergebnisse in der Etikettierung von mtDNA mit einem grün fluoreszierenden Signals (Abbildung 2). Wenn es richtig gemacht, ist das verstärkte grüne Signal ausreichend höher als Hintergrund-Fluoreszenz (wie zelluläre Autofluoreszenz oder ein Nebeneffekt der HRP-TSA-Reaktion).
Diese Technik wurde entwickelt, um neu repliziert mtDNA-Label, um zu visualisieren und zu quantifizieren mtDNA Biogenese in subzellulären Kompartimenten von Neuronen (Abbildung 3). Die EDU Kennzeichnung ermöglicht anschließende Fluoreszenz-Immunzytochemie zu anderen zellulären Marker wie Neurofilament (Abbildung 3D) Label.
Die TSA Reaktion kann auch mit anderen fluoreszierenden Alexa Fluor tyramides wie Alexa Fluor 594-Tyramid durchgeführt werden. Daraus ergibt sich ein grün fluoreszierendes nuklearen Signal von der Oregon Green-Azid-Klick-Reaktion in den Zellkern, aber kein grünes Signal in mtDNA, die nachweisbar vor TSA ist. Die Verstärkung mit Alexa Fluor 594-Tyramid intensiviert die nukleare Label und zeigt die eingearbeitet EdU in mtDNA mit einer roten Fluoreszenz-Signal (Abbildung 4). Eine ähnliche Verstärkung Verfahren verwendet wird, um BrdU-Einbau in neu synthetisierte mtDNA (Abbildung 5) sichtbar zu machen, jedoch erfordert diese Methode einen weiteren Schritt auf die BrdU-Epitop entweder durch eine harte Säure (HCl) oder Enzym verdauen, die nicht für EdU notwendig erholen Kennzeichnung.
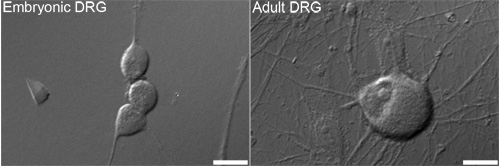
Abbildung 1. Vertreter Differential-Interferenz-Kontrast (DIC) Bilder von embryonalen (links) und Erwachsenen (rechts) Spinalganglien (DRG) Neuronen, die normalerweise für die Analyse. Bars = 10 pm eingesetzt werden.
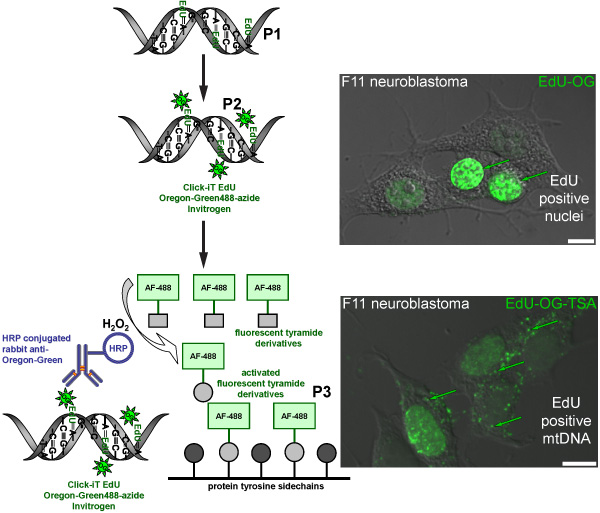
Abbildung 2. Schematische Darstellung, die die Verfahren zur Kennzeichnung EdU in mtDNA mit einem grün fluoreszierenden Signals. Die schematische stellt den Drei-Stufen-Protokoll für die Kennzeichnung EdU in mtDNA mit einem grün fluoreszierenden Signals. Mitotisch aktiven F11 Neuroblastom-Zellen dienen als positive Kontrolle und illustrieren die Kennzeichnung Muster EdU, dass in Kern-und Mitochondrien-DNA eingebaut wurde. Der erste Schritt (P1) ist ein Thymidin-Analogon in neu synthetisierte mtDNA integrieren durch Inkubation von Zellen in Anwesenheit von EDU. Der zweite Schritt (P2) wird auf Klick-Chemie eingebaut EdU mit Oregon Green-Azid-Label basiert. Grün-Signal wird sichtbar in den Kernen der Zellen, ihre Kern-DNA repliziert. Der letzte Schritt (P3) ist mit dem Oregon Gr verstärkeneen-Azid-Signal durch Inkubation mit einem HRP-konjugierten Kaninchen-Antikörper gegen Oregon Green gefolgt von Inkubation mit Alexa Fluor 488 markierten Tyramid in Gegenwart von Wasserstoffperoxid (H 2 O 2) zu grün-Signal in mtDNA zu visualisieren.
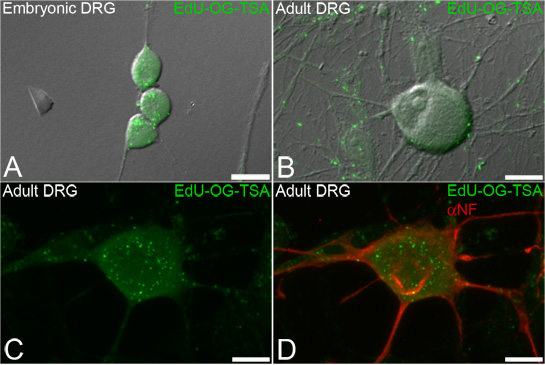
Abbildung 3. EdU Kennzeichnung von mtDNA in Spinalganglien (DRG) Neuronen. Neuronen sind mit der EDU inkubiert und das Signal wird anschließend verstärkt auf mtDNA, die eingearbeitet EdU offenbaren. Vertreter Fluoreszenzbilder auf Durchlicht zeigt grün punktförmige Signale verstärkt EDU (Edu-OG-TSA, grün) in neu synthetisierte mtDNA sowohl embryonale (A) und Erwachsenenbildung (BD) DRG-Neuronen aufgenommen überlagert. Die EDU Kennzeichnung Verfahren ermöglicht die anschließende Immunfluoreszenzfärbung der neuronalen Marker wie Neurofilament (D, αNF, in rot). Maßstabsbalken = 10 pm.
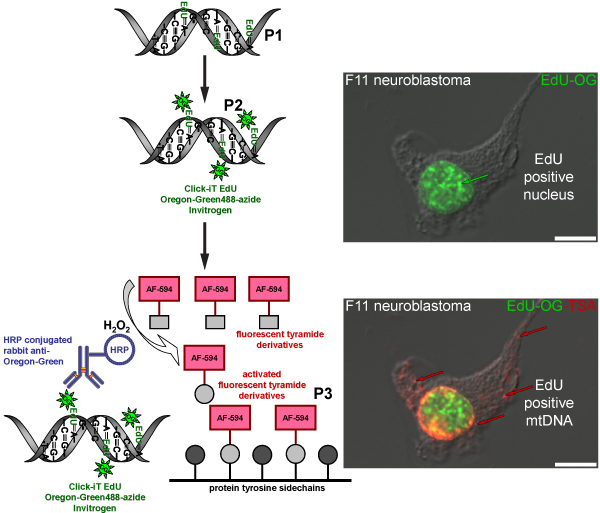
Abbildung 4. Schematische Darstellung, die die Verfahren zur Kennzeichnung EdU in mtDNA mit einer roten Fluoreszenz-Signal. Die schematische stellt den Drei-Stufen-Protokoll für die Kennzeichnung EdU in mtDNA mit einer roten Fluoreszenz-Signal. Mitotisch aktiven F11 Neuroblastom-Zellen dienen als positive Kontrolle und illustrieren die Kennzeichnung Muster EdU, dass in Kern-und Mitochondrien-DNA eingebaut wurde. Der erste Schritt (P1) ist ein Thymidin-Analogon in neu synthetisierte mtDNA integrieren durch Inkubation von Zellen in Anwesenheit von EDU. Der zweite Schritt (P2) wird auf Klick-Chemie eingebaut EdU mit Oregon Green-Azid-Label basiert. Grün-Signal wird sichtbar in den Kernen der Zellen, ihre Kern-DNA repliziert. Der letzte Schritt (P3) ist mit dem Oregon Green-Azid-Signal durch Inkubation mit einem HRP-konjugierten Kaninchen-Antikörper gegen Oregon Green verstärken, gefolgt von Inkubation mit Alexa Fluor 594 markierten Tyramid in Gegenwart von Wasserstoffperoxid (H 2 O 2) zu visualisieren rotes Signal in mtDNA.
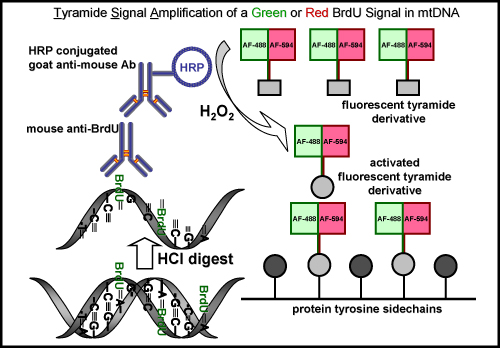
Abbildung 5. BrdU-Markierung und Tyramid Signalverstärkung mit einem grünen oder roten Fluoreszenz-Signal in mtDNA. Die schematische Darstellung stellt das Verfahren für die Kennzeichnung BrdU in neu synthetisierte mtDNA entweder mit einem grünen oder roten Fluoreszenz-Signal. Ein erster Schritt ist erforderlich, um die BrdU-Epitop entweder durch eine Säure (HCl) oder Enzym verdauen, die nicht notwendig ist für EdU Kennzeichnung erholen. Der nächste Schritt ist mit einer Maus primären anti-BrdU-Antikörper inkubiert. Dies wird durch Inkubation mit einem Meerrettichperoxidase (HRP)-konjugiertem Ziege anti-Maus-Antikörper. Schließlich wird das Signal mit einem grünen oder roten fluoreszenzmarkierten Tyramid in Gegenwart von Wasserstoffperoxid (H 2 O 2) zu signalisieren, in mtDNA visualisieren verstärkt.
Diskussion
Wir entwickelten eine sensitive Assay neu synthetisierten mtDNA in einzelnen Zellen mit einem Tyramid Signalverstärkung von EDU-Label. Eines der größten Probleme bei der Optimierung dieses Protokolls war die inkonsistente Ergebnisse zwischen Deckgläsern. Es wurden Änderungen an der Invitrogen Kit-Protokolle, um zu verhindern Mischen kleiner Volumina einzelner Deckgläser und macht Master-Lösungen für alle Deckgläser verwendet werden. Die 75-80 ul für jeden 12 mm runden Deckglas verwendet ist ein ideales Volumen vollständig bedecken die Oberfläche und bietet genügend Lösung, um konsistente Ergebnisse zwischen den Proben zu geben. Inkubationszeiten und Reagenz-Konzentrationen wurden optimiert, sondern Verbesserungen könnten durch die Einstellung von ihnen gesehen zu werden.
Das Protokoll wurde entwickelt, um jeden Prozess einmal durchlaufen. Allerdings haben einige der Schritte, mit neuen Lösungen wiederholt, um Proben, die nach dem ersten Versuch gescheitert zu erholen. Insbesondere haben die EDU klicken Reaktionsschritte (3,4-3,6), ohne einen signifikanten Anstieg in Hintergrundfluoreszenz wiederholt worden. Die Anti-Oregon Green-HRP Antikörper-Inkubation (4.1-4.3) und die Tyramid Verstärkung (4,4-4,5) wurden ebenfalls Schritte wiederholt, aber immer häufiger zu einem schlechten Signal-Rausch-Verhältnis wegen des erhöhten Hintergrundfluoreszenz.
Die empfindlichsten Reagenzien sind die Click-iT EdU Buffer Additive (Komponente G) und dem Kaninchen-Anti-Oregon Green-HRP Antikörper (Komponente I) von der Click-iT EdU Microplate Assay-Kit. Die Click-iT EdU Buffer Additive färbt sich allmählich gelb im Laufe der Zeit bei 4 ° C gelagert und zwischen 6-12 Monaten merklich dunkler,. Die Lagerung der Click-iT EdU Buffer Additive bei -20 ° C wird zur Lösung dieses Problem. Die Kennzeichnung und Tyramid Verstärkung Schritte funktionieren am besten, wenn die Kaninchen-Anti-Oregon Green-HRP-Antikörper innerhalb von 4-6 Monaten Neugründung in MilliQ dH 2 O wird verwendet,
Das milde Kennzeichnung von Edu in mtDNA erlaubt den direkten Vergleich mit weiteren zellulären Marker 9-11 und steigert den Nutzen dieser Technik gegenüber anderen Thymidin-Analoga, wie 5-Bromo-2-desoxyuridin (BrdU), die eine harte Behandlung zur Wiederherstellung erfordert sein Epitop innerhalb der DNA. Unser Labor 11-12 und 13-15 andere haben erfolgreich BrdU zur mtDNA-Label. Die BrdU-Markierung Technik ist ähnlich dem für EdU verwendet, hat aber einige wichtige Unterschiede (Abbildung 5). Nach der Permeabilisierung Schritt oben genannten (3,1-3,2), ist der BrdU-Epitop mit einem Denaturierungsschritt erholt (2 N HCl für 30 min bei 37 ° C durch drei Wäschen in 1X PBS). Die BrdU wird dann mit einem primären Antikörper gegen BrdU markierte (Vector Laboratories 1:50 in 1% Blocking-Lösung aus dem TSA-Kit und inkubiert über Nacht bei 4 ° C). Ein sekundärer Antikörper Schritt ist notwendig, um die BrdU-Signal (Ziege anti-Maus IgG, konjugiert mit HRP von der TSA-Kits # 2 und # 5, 1:100 verdünnt in 1% Blocking-Lösung inkubiert und für 45 min bei Raumtemperatur), bevor verstärken die TSA Schritte (4,3-4,5 oben). Mit zwei Thymidinanaloga, Edu und BrdU, zu beschriften mtDNA ist vorteilhaft für die Durchführung von Dual-Label Experimente, in denen mtDNA nacheinander in Puls-Kennzeichnung Paradigmen 11 bezeichnet werden kann.
Unser Labor ist mit der EDU und BrdU-Markierung der mtDNA, die Regulierung der mitochondrialen Biogenese im Rahmen der diabetischen Neuropathie, eine häufige Komplikation des Diabetes zu untersuchen. Wir haben erfolgreich die Signalverstärkung verwendet, um Änderungen in mtDNA Biogenese in einzelnen Neuronen 11-12 zu messen. Diese Technik wird in anderen Experimenten entwickelt, um die Mechanismen der mtDNA-Replikation und Umsatz oder zur Identifizierung von Drogen, die mtDNA-Synthese zu hemmen erkunden nützlich. Darüber hinaus können die Grundprinzipien der Verstärkung Edu und BrdU-Signale zu anderen Studien angewendet werden, messen die DNA-Replikation oder Reparatur.
Offenlegungen
Danksagungen
Diese Arbeit wurde vom National Institutes of Health Grants NS-38849 und DK-076160, der Juvenile Diabetes Research Foundation Center for the Study of Diabetes Komplikationen in unterstützt, das Programm für Neurologische Forschung und Entdeckung und die A. Alfred Taubman Medical Research Institute an der University of Michigan. Diese Arbeit verwendet die Morphologie und Bildanalyse Kern des Michigan Diabetes Research and Training Center, National Institutes of Health Grants 5P60 DK-20572 vom National Institute of Diabetes and Digestive und Nierenkrankheiten finanziert. Die Autoren bedanken sich Scott T. Clarke von Molecular Probes / Invitrogen für seine wertvollen Ratschläge über die verschiedenen Click-iT EdU Labeling Kits und die großzügige Spende von Reagenzien, die anfängliche Entwicklung des EdU Amplifikationstechnik unterstützen.
Materialien
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | Fisher Scientific | 12-545-82 | |
| 245 mm x 245 mm BioAssay Dish | Corning | 431111 | |
| Parafilm M | Fisher Scientific | PM-996 | |
| paraformaldehye | Sigma-Aldrich | P6148 | |
| Triton X-100 | Sigma-Aldrich | T-8532 | |
| Phosphate buffered saline 10X solution | Fisher Scientific | BP399 | |
| Transfer Pipet | Fisher Scientific | 13-711-7M | |
| Hydrogen peroxide, 30% in water | Fisher Scientific | BP2633 | |
| Click-iT EdU Microplate Assay Kit | Invitrogen | C10214 | |
| TSA Kit #12, with HRP—goat anti-rabbit IgG and Alexa Fluor 488 tyramide | Invitrogen | T20922 | |
| TSA Kit #15, with HRP–goat anti-rabbit IgG and Alexa Fluor 594 tyramide | Invitrogen | T20925 | |
| ProLong Gold antifade reagent with DAPI | Invitrogen | P-36931 | |
| Polyclonal rabbit Neurofilament antibody | Chemicon International | AB1981 | |
| Mouse monoclonal anti-BrdU antibody | Vector Laboratories | VP-B209 | |
| TSA Kit #2, with HRP—goat anti-mouse IgG and Alexa Fluor 488 tyramide | Invitrogen | T20912 | |
| TSA Kit #5, with HRP—goat anti-mouse IgG and Alexa Fluor 594 tyramide | Invitrogen | T20915 |
Referenzen
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu. Rev. Cell Dev. Biol. 22, 79-99 (2006).
- Dimmer, K. S., Scorrano, L. (De)constructing mitochondria: what for. Physiology (Bethesda). 21, 233-241 (2006).
- Suen, D. F., Norris, K. L., Youle, R. J. Mitochondrial dynamics and apoptosis. Genes Dev. 22, 1577-1590 (2008).
- Clay Montier, L. L., Deng, J. J., Bai, Y. Number matters: control of mammalian mitochondrial DNA copy number. J. Genet. Genomics. 36 (3), 125-131 (2009).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc Natl Acad Sci U.S.A. 105, 2415-2420 (2008).
- Buck, S. B., Bradford, J., Gee, K. R., Agnew, B. J., Clarke, S. T., Salic, A. Detection of S-phase cell cycle progression using 5-ethynyl-2'-deoxyuridine incorporation with click chemistry, an alternative to using 5-bromo-2'-deoxyuridine antibodies. Biotechniques. 44, 927-929 (2008).
- Yu, Y., Arora, A., Min, W., Roifman, C. M., Grunebaum, E. EdU incorporation is an alternative non-radioactive assay to [(3)H]thymidine uptake for in vitro measurement of mice T-cell proliferations. J Immunol Methods. 350 (1-2), 29-35 (2009).
- Heusden, J. V. a. n., Jong, P. d. e., Ramaekers, F., Bruwiere, H., Borgers, M., Smets, G. Fluorescein-labeled tyramide strongly enhances the detection of low bromodeoxyuridine incorporation levels. J. Histochem. Cytochem. 45 (2), 315-319 (1997).
- Cappella, P., Gasparri, F., Pulici, M., Moll, J. A novel method based on click chemistry, which overcomes limitations of cell cycle analysis by classical determination of BrdU incorporation, allowing multiplex antibody staining. Cytometry A. 73, 626-636 (2008).
- Kaiser, C. L., Kamien, A. J., Shah, P. A., Chapman, B. J., Cotanche, D. A. 5-Ethynyl-2'-deoxyuridine labeling detects proliferating cells in the regenerating avian cochlea. Laryngoscope. 119, 1770-1775 (2009).
- Lentz, S. I., Edwards, J. L., Backus, C., McLean, L. L., Haines, K. M., Feldman, E. L. Mitochondrial DNA (mtDNA) Biogenesis: Visualization and Duel Incorporation of BrdU and EdU Into Newly Synthesized mtDNA In. Vitro. J Histochem Cytochem. 58 (2), 207-218 (2010).
- Edwards, J. L., Quattrini, A., Lentz, S. I., Figueroa-Romero, C., Cerri, F., Backus, C., Hong, Y., Feldman, E. L. Diabetes regulates mitochondrial biogenesis and fission in mouse neurons. Diabetologia. 53 (1), 160-169 (2010).
- Davis, A. F., Clayton, D. A. In situ localization of mitochondrial DNA replication in intact mammalian cells. J. Cell Biol. 135, 883-893 (1996).
- Magnusson, J., Orth, M., Lestienne, P., Taanman, J. W. Replication of mitochondrial DNA occurs throughout the mitochondria of cultured human cells. Exp. Cell. Res. 289, 133-142 (2003).
- Amiri, M., Hollenbeck, P. J. Mitochondrial biogenesis in the axons of vertebrate peripheral neurons. Dev. Neurobiol. 68, 1348-1361 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten