Method Article
Visualizzazione della replicazione del DNA mitocondriale in singole celle da amplificazione del segnale EdU
In questo articolo
Riepilogo
Abbiamo sviluppato una tecnica sensibile per etichetta di nuova sintesi del DNA mitocondriale (mtDNA) in celle individuali al fine di studiare la biogenesi mitocondriale. La tecnica combina l'incorporazione di EdU insieme ad un tyramide amplificazione del segnale (TSA), protocollo di visualizzare la replicazione del DNA mitocondriale all'interno di compartimenti subcellulari di neuroni.
Abstract
I mitocondri sono regolatori chiave di energia cellulare e la biogenesi mitocondriale è una componente essenziale di regolare numeri mitocondri nelle cellule sane 1-3. Un approccio per il monitoraggio biogenesi mitocondriale è quello di misurare la percentuale di DNA mitocondriale (mtDNA) replica 4. Abbiamo sviluppato una tecnica sensibile per etichettare mtDNA di nuova sintesi in singole cellule per studiare biogenesi mitocondriale. La tecnica combina l'incorporazione di 5-etinil-2'-deossiuridina (EDU) 5-7 con una amplificazione del segnale tyramide (TSA) 8 protocollo di visualizzare la replicazione del DNA mitocondriale all'interno di compartimenti subcellulari di neuroni. EdU è superiore agli analoghi della timidina altri, come il 5-bromo-2-deossiuridina (BrdU), perché la reazione iniziale clicca per EdU etichetta 5-7 non richiede trattamenti acidi forti o enzima digerisce che sono necessarie per esporre l'epitopo BrdU . L'etichettatura più mite di EdU permette la comparazione diretta della sua costituzione con altri marcatori cellulari 9-10. La possibilità di visualizzare e quantificare la biogenesi mitocondriale fornisce uno strumento essenziale per investigare i meccanismi utilizzati per regolare la biogenesi mitocondriale e che consentono di studiare la patogenesi associata a tossicità da farmaci, l'invecchiamento, cancro e malattie neurodegenerative. La nostra tecnica è applicabile ai neuroni sensoriali così come altri tipi di cellule. L'uso di questa tecnica per misurare la biogenesi mitocondriale ha implicazioni importanti nel favorire la comprensione sia normale fisiologia cellulare, così come stati di malattia ridotta.
Protocollo
1. Preparazione dei neuroni
- Ganglio della radice dorsale (DRG) i neuroni sono cresciuti su sterili (autoclave) coprioggetto da 12 mm di vetro in un 24-ben piastra di coltura.
- I 10 magazzino mM di Edu (in DMSO, Click-iT EdU micropiastre Assay Kit) è normalmente diluita 1:100 nel terreno di coltura per ottenere una soluzione 10X EdU (100 mM) e poi diluito 1:10 nella cultura pozzi (per esempio 30 microlitri della soluzione del EdU 10X in un totale di 300 ml di terreno di coltura). Neuroni DRG vengono incubate con una concentrazione finale di 10 EdU mM a 37 ° C e 5% CO 2 tra 2-24 ore. Il periodo di tempo dipende da come trattamenti influenzano il tasso di sintesi del DNA mitocondriale.
- In una cappa aspirante, i neuroni DRG sono fissate nel 2% paraformaldeide per 10-15 minuti a temperatura ambiente, lavate due volte in PBS 1X 2-5 minuti ogni lavaggio e conservati in fresco 1X PBS per fino a 1 mese a 4 ° C.
- Coprioggetto sono trasferiti con punta fine pinze ad una camera preparata umido (BioAssay piatto Corning con fondo nero tappezzata per contrasto, avvolto in un foglio di alluminio per proteggere i componenti sensibili luce e umidificata con Kimwipes acqua satura) e messo su un foglio di Parafilm M che fornisce una superficie idrofoba di confinare le soluzioni per il coprioggetto. Il protocollo che segue è stato progettato per 28 coprioggetto e le soluzioni sono preparate per 32 coprioggetti (circa il 14% di volume in più). Soluzioni con componenti costosi o limitata sono fatti in modo che il 75-80 microlitri vengono utilizzati su ogni vetrino. Altrimenti, 200-300 microlitri vengono utilizzati per soluzioni di lavaggio e blocco di lavare accuratamente le soluzioni precedenti.
- Riapplicare PBS 1X per coprire la superficie di ogni coprioggetto (200-300 mL). Preparare il Click-iT saggio e tyramide kit di amplificazione del segnale come descritto di seguito e nelle istruzioni del produttore.
Preparazione di Click-iT EdU micropiastre Assay Kit
La maggior parte dei componenti del Click-iT Assay Kit EdU Microplate sono pre-made e sono conservati a 4 ° C [2x Click-iT tampone di reazione (E Component 10x), CuSO4 (100 mM, componente F), Click-iT EdU fissativo ( componente D) e tampone di bloccaggio (2x componente H)]. Click-iT Additivo EdU Buffer (10x Component G) è conservato a -20 ° C per evitare che si trasformi giallo-marrone nel tempo. Questo componente tollera ripetuti cicli di gelo-disgelo.
L'Oregon verde 488 azide (componente B) deve essere suddivisa in piccole quantità (10-20 mL) per ridurre al minimo i cicli di gelo-disgelo e conservati a -20 ° C.
Per preparare una soluzione madre della anti-Oregon Verde coniugato HRP (componente I), aggiungere 75 ml di Milli Q dH 2 O al flaconcino. Mescolare pipettando dolce o per inversione per evitare la formazione di schiuma e conservare a 4 ° C. Non vortice.
Preparazione del TSA Kit # 12, con HRP capra anti-IgG e Alexa Fluor 488 Kit Tyramide
Per preparare la soluzione tyramide magazzino, sciogliere il materiale solido (Alexa Fluor 488 tyramide, Componente A) in 150 ml di DMSO (componente B). Capovolgere il flaconcino più volte per sciogliere qualsiasi tipo di rivestimento tyramide lati del flacone. Soluzione negozio in piccole aliquote (10-20 mL) a ≤ -20 ° C, essiccato e protetto dalla luce.
2. Click-iT 5-etinil-2-deossiuridina (EDU) Etichettatura
NOTA: Tutte le soluzioni vengono rimossi con una pipetta a bulbo di trasferimento con una punta di 200 l apposto alla fine per rimuovere delicatamente le cellule liquidi senza perdere. Evitare di utilizzare una linea di vuoto, che di solito rimuovere soluzioni troppo energicamente. Una pipetta bulbo viene utilizzato per inondare delicatamente vetrini con 200-300 ml di soluzioni di lavaggio per lavare accuratamente la soluzione precedente.
- Le cellule sono permeabilizzate con 0,1% Triton X-100 in soluzione PBS 1X per 10 minuti a temperatura ambiente in una camera coperta umida. Utilizzare l'1% Triton-X-100 soluzioni di riserva.
- Fai 3000 ml di 0.1% Triton X-100 con l'aggiunta di 300 ml 1% Triton X-100 stock 2700μL PBS 1X.
- Usa 75-80 microlitri per coprioggetto.
- Rimuovere il Triton X-100 soluzione e lavare due volte con PBS 1X.
- Perossidasi endogena è spento da una riduzione dell'1% H 2 O 2 in soluzione PBS 1X per 30 minuti a temperatura ambiente. Diluire 30% H 2 O 2 soluzione con PBS 1X. Questa soluzione deve essere fresco, ma può essere effettuata durante la fase di permeabilizzazione 10 minuti sopra (punto 3.1).
- Fai 3000 ml di una riduzione dell'1% H 2 O 2 con l'aggiunta di 100 l 30% di H 2 O 2-2900 microlitri di PBS 1X.
- Usa 75-80 microlitri per coprioggetto.
- Rimuovere la soluzione perossidasi e lavare due volte con PBS 1X.
- Click-iT EdU Reazione
- Per 32 coprioggetto (32 x 80 microlitri =2560 mL) per un totale di 2560 microlitri è necessario. La reazione 2x è fatto nel 1280 microlitri, che è la metà del volume totale della reazione.
- Appena preparare il Click-iT Cocktail di reazione prima di iniziare la correzione dopo 5 minuti (punto 3.5 di seguito).
- Mescolate il cocktail pipettando su e giù. Non vortice quando si effettua questa reazione.
2 x reazione Cocktail
I componenti sono da Click-iT EdU micropiastre Assay KitLa metà del volume: 1280 microlitri Milli Q dH 2 O 1132,8 microlitri 2x Click-iT tampone di reazione (E Component 10x) 100,3 microlitri Click-iT EdU Additivo Buffer (G Component 10x) 25,6 microlitri CuSO 4 (100 mm, F Component) 25,6 microlitri Oregon Verde Azide (un componente) 6,4 microlitri Totale 1290,7 microlitri
NOTA: I volumi usato sopra sono proporzionali ai volumi elencati nella Click-iT direzioni Assay kit EdU micropiastre. Il volume finale di Cocktail reazione è poco più di 1280 microlitri. - Il Click-iT 2X reazione viene diluito con uguale volume di Click-iT EdU fissativo (Componente D) a destra prima dell'uso. Mescolando questi insieme rende una soluzione uniforme per tutti reazione coprioggetto.
- Aggiungi 1290,7 microlitri Click-iT EdU fissativo (Componente D) per il cocktail 1290,7 reazione microlitri. Usa 75-80 microlitri per coprioggetto.
- Post-fix i neuroni con Click-iT EdU fissativo (Componente D) per 5 minuti a temperatura ambiente.
- Fissare e rimuovere aggiungere cocktail reazione dall'alto (punto 3.4). Copertura al riparo dalla luce e incubare per 25 minuti a temperatura ambiente. Rimuovere delicatamente il cocktail di reazione. Lavare due volte con tampone diluito 1X blocco (2X Component H).
- Fai 5200 ml di 1X tampone di bloccaggio diluendo 2600 Buffer microlitri di blocco (2x componente H) con un uguale volume di MilliQ d H 2 O (2600 mL). Usa 75-80 microlitri per coprioggetto.
- Sotto un epi-microscopio a fluorescenza, controllare per la colorazione verde Oregon nei nuclei delle cellule di controllo mitoticamente attivo.
3. Tyramide amplificazione del segnale (TSA) di EdU segnale
- Aggiungere 1% di soluzione ad ogni blocco TSA coprioggetto e incubare per 30 minuti a temperatura ambiente.
- Fai microlitri 6000 di 1% soluzione blocco TSA pesando 0,06 g di reagente di blocco TSA (TSA kit # 12, Componente D) e aggiungerla a 6000 microlitri di PBS 1X. Vortex per mescolare. L'aggiunta di siero di capra al 5% nel 1% soluzione blocco TSA spesso contribuire a ridurre legame non specifico.
- In breve rotazione anti-Oregon verde perossidasi coniugato magazzino anticorpi (componente I del Click-iT Assay EdU micropiastre), preparato 2-24 ore prima del TSA. Diluire l'anticorpo primario 1:300 in soluzione% 1 TSA bloccare e invertire o pipetta su e giù delicatamente per mescolare. Non vortice di non perturbare le HRP-coniugato anticorpo. Rimuovere l'1% soluzione blocco TSA, aggiungere 75 microlitri per ogni coprioggetto e incubare una notte a 4 ° C.
- L'anticorpo può essere utilizzato più concentrato, come ad esempio 1:150, ma è disponibile in quantità limitate, in modo da utilizzare con parsimonia. Anche in questo caso, l'aggiunta di siero di capra al 5% nel 1% soluzione blocco TSA spesso contribuire a ridurre legame non specifico della Oregon anti-perossidasi di rafano verde anticorpi coniugati.
- Fai 2560 ml di una soluzione di anticorpo 1:300 con l'aggiunta di 8,53 l di Coniglio magazzino anti-Oregon Green-HRP (Click-iT EdU micropiastre Assay) a 2560 microlitri 1% TSA blocco. Mescolare pipettando dolce o inversione.
- Il giorno seguente, togliere la soluzione di anticorpi e lavare tre volte con PBS 1X. Incubare i coprioggetti nel lavaggio finale, per altri 30-60 minuti a temperatura ambiente per garantire che l'anticorpo non legato primario è stato rimosso.
- Preparare reazione Tyramide di 2560 microlitri (32 coprioggetto x 80 mL).
- Fare 400 ml di una H 0,15% 2 O 2 soluzione (100X) con l'aggiunta di 2 ml di 30% H 2 O 2 (TSA kit # 12, componente F) a 398 Buffer Amplificazione microlitri (TSA kit # 12, E Component) . Questo dovrebbe essere fatto appena prima necessità.
- Per un volume finale di 2560 microlitri combinare:
- 25,6 microlitri Tyramide-488 (TSA componente del kit A)
- 2508,8 Buffer Amplificazione microlitri (TSA E Component kit)
- 25,6 microlitri 0,15% H 2 O 2 (dall'alto per una finale 0,0015% H 2 O 2)
- Rimuovere 1X PBS e incubare con la reazione Tyramide per 15 minuti a temperatura ambiente.
- Rimuovere reazione Tyramide e risciacquare tre volte con PBS 1X, incubando nel lavaggio finale per 30-60 minuti a temperatura ambiente come prima.
- Verificare la presenza di mtDNA etichettatura sotto un epi-microscopio a fluorescenza.
- Coprioggetto montaggio su vetrini da microscopio di vetro con un mezzo Antifade montaggio, come prolungare oro con DAPI. In alternativa, l'etichettatura e EdU amplificazione può essere seguito da standard di immunocitochimica fluorescenti ad etichettare altri marcatori cellulari.
4. Risultati rappresentante: Visualizzazione di replica del DNA mitocondriale come marker per la biogenesi mitocondriale
Siamo interessati a come i neuroni sensoriali (Figura 1) regolano il numero di mitocondri. Questo protocollo verrà etichetta mtDNA di nuova sintesi con un marcatore fluorescente come un modo per misurare nuovi mitocondri. L'incorporazione della EdU sintetico nucleotide seguita dalla chimica clic del amplificazione Oregon Green-azide e la successiva con l'Alexa Fluor-488 risultati tyramide per l'etichettatura di mtDNA con un segnale fluorescente verde (Figura 2). Se fatto correttamente, il segnale amplificato verde è sufficientemente superiore a fluorescenza di fondo (come autofluorescenza cellulare o un effetto collaterale della HRP-reazione TSA).
Questa tecnica è stata progettata per etichetta mtDNA nuova replicato al fine di visualizzare e quantificare la biogenesi mitocondriale all'interno di compartimenti subcellulari dei neuroni (Figura 3). L'etichettatura EdU permette per la successiva immunocitochimica fluorescenti di etichettare gli altri marcatori cellulari come neurofilamenti (Fig. 3D).
La reazione TSA può essere fatto anche con altre fluorescenti tyramides Alexa Fluor come Alexa Fluor 594-tyramide. Il risultato è un segnale fluorescente verde nucleare dalla Green-azide reazione click Oregon nel nucleo, ma nessun segnale verde nel mtDNA, che è rilevabile prima della TSA. L'amplificazione con Alexa Fluor 594-tyramide intensifica l'etichetta nucleare e rivela la EdU incorporato in mtDNA con un segnale rosso fluorescente (Figura 4). Una procedura di amplificazione simile viene utilizzato per visualizzare incorporazione di BrdU in nuova sintesi del DNA mitocondriale (Figura 5), tuttavia, questo metodo richiede un ulteriore passo per recuperare l'epitopo BrdU sia da un acido duro (HCl) o enzimi digerire, che non è necessario per EdU etichettatura.
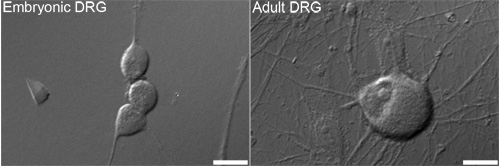
Figura 1. Rappresentante contrasto differenziale di interferenza (DIC) immagini di embrionale (a sinistra) e adulti (a destra) ganglio della radice dorsale (DRG), i neuroni che sono tipicamente utilizzati per l'analisi. Bar = 10 micron.
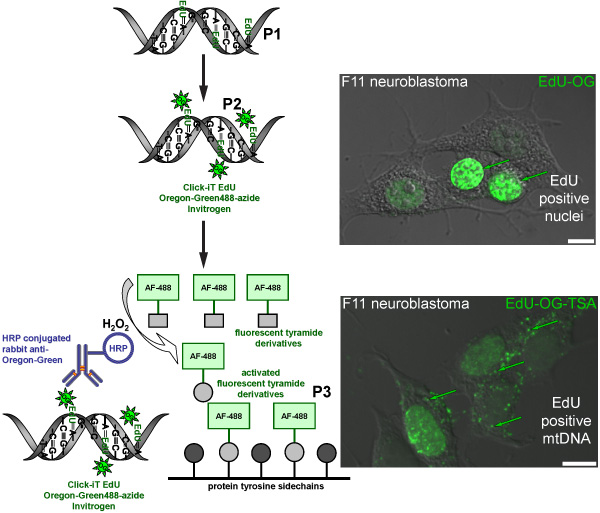
Figura 2. Schema che rappresenta il regime per l'etichettatura EdU nel mtDNA con un segnale verde fluorescente. Lo schema rappresenta le tre fasi del protocollo per l'etichettatura EdU nel mtDNA con un segnale verde fluorescente. Mitoticamente attivo F11 cellule di neuroblastoma servire come controllo positivo e illustrare il modello di etichettatura dei EdU che è stata incorporata nel DNA nucleare e mitocondriale. Il primo passo (P1) è di incorporare un analogo della timidina in nuova sintesi del mtDNA incubando le cellule in presenza di Edu. Il secondo passo (P2) si basa sulla chimica click per etichetta EdU incorporato con una Oregon Green-azide. Segnale verde è visibile nei nuclei delle cellule che replicare il loro DNA nucleare. La fase finale (P3) è quello di amplificare il Gr Oregoneen-azide segnale da incubazione con un anticorpo coniugato HRP-coniglio contro Oregon verde seguita da incubazione con Alexa Fluor 488 tyramide etichettati in presenza di perossido di idrogeno (H 2 O 2) per visualizzare il segnale verde nel mtDNA.
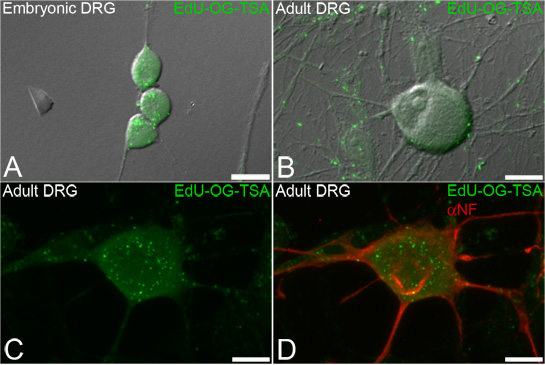
Figura 3. EdU etichettatura del mtDNA nel ganglio della radice dorsale (DRG) neuroni. Neuroni sono incubati con Edu e il segnale viene poi amplificato a rivelare mtDNA che incorporava Edu. Rappresentante immagini di fluorescenza sovrapposti a luce trasmessa mostrando segnali verde puntata di amplificati EdU (EDU-OG-TSA, verde) incorporato nel DNA mitocondriale di nuova sintesi di entrambi embrionale (A) e adulti (BD) neuroni DRG. La procedura di etichettatura EdU permette per la successiva colorazione di immunofluorescenza di marcatori neuronali come neurofilamenti (D, αNF, in rosso). Barre di scala = 10 micron.
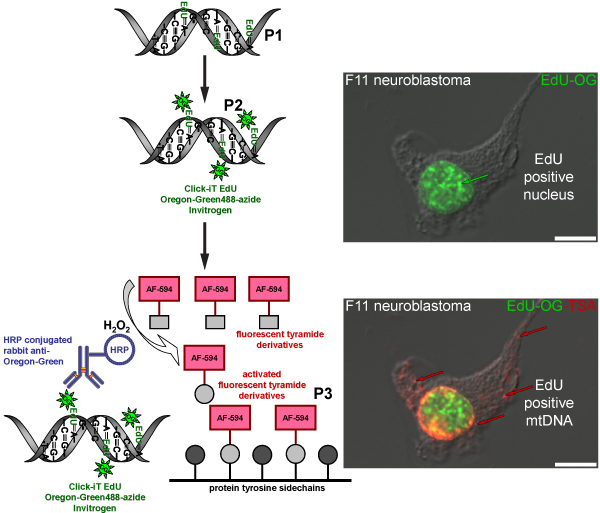
Figura 4. Schema che rappresenta il regime per l'etichettatura EdU nel mtDNA con un segnale rosso fluorescente. Lo schema rappresenta le tre fasi del protocollo per l'etichettatura EdU nel mtDNA con un segnale rosso fluorescente. Mitoticamente attivo F11 cellule di neuroblastoma servire come controllo positivo e illustrare il modello di etichettatura dei EdU che è stata incorporata nel DNA nucleare e mitocondriale. Il primo passo (P1) è di incorporare un analogo della timidina in nuova sintesi del mtDNA incubando le cellule in presenza di Edu. Il secondo passo (P2) si basa sulla chimica click per etichetta EdU incorporato con una Oregon Green-azide. Segnale verde è visibile nei nuclei delle cellule che replicare il loro DNA nucleare. La fase finale (P3) è quello di amplificare il Green-azide Oregon segnale da incubazione con un anticorpo coniugato HRP-coniglio contro Oregon verde seguita da incubazione con Alexa Fluor 594 tyramide etichettati in presenza di perossido di idrogeno (H 2 O 2) per visualizzare segnale rosso nel mtDNA.
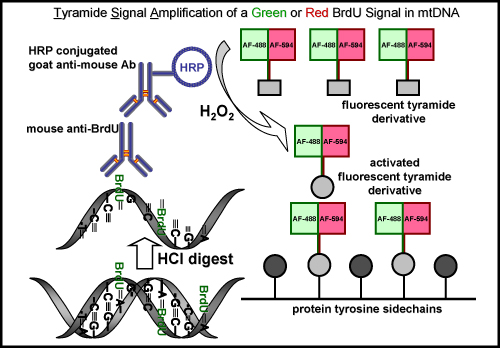
Figura 5. BrdU etichettatura e amplificazione del segnale tyramide con un segnale verde o rosso fluorescente nel mtDNA. Lo schema rappresenta la procedura per l'etichettatura BrdU nel mtDNA di nuova sintesi sia con un segnale verde o rosso fluorescente. Un primo passo è necessario per recuperare il epitopo BrdU sia da un acido (HCl) o enzimi digerire, che non è necessaria per l'etichettatura di Edu. Il passo successivo è l'incubazione con un primario del mouse anticorpi anti-BrdU. Questo è seguito da incubazione con perossidasi di rafano (HRP)-capra coniugato anti-topo. Infine, il segnale viene amplificato con un tyramide verde o rosso fluorescente in presenza di perossido di idrogeno (H 2 O 2) per la visualizzazione del segnale nel mtDNA.
Discussione
Abbiamo sviluppato un dosaggio sensibile per etichettare mtDNA di nuova sintesi in singole cellule utilizzando un amplificazione tyramide segnale di Edu. Uno dei maggiori problemi durante l'ottimizzazione di questo protocollo è stato il risultato incoerenti tra lamelle. Sono state apportate modifiche ai protocolli di kit di Invitrogen per evitare la sovrapposizione di piccoli volumi su vetrini singoli e rendendo le soluzioni maestro da utilizzare per tutte le lamelle. Il 75-80 microlitri utilizzato per ogni bicchiere 12 millimetri vetrino circolare è un volume ideale per coprire completamente la superficie, fornendo una soluzione abbastanza per dare risultati coerenti tra i campioni. I tempi di incubazione e le concentrazioni di reagenti sono stati ottimizzati, ma i miglioramenti possono essere visti con adattamenti per loro.
Il protocollo è progettato per essere eseguito ogni processo una volta. Tuttavia, alcuni dei passaggi sono stati ripetuti con nuove soluzioni per recuperare campioni che non è riuscita dopo il primo tentativo. In particolare, i passi cliccare reazione EdU (3,4-3,6) sono stati ripetuti senza un significativo aumento della fluorescenza di fondo. L'anti-Oregon Green-HRP incubazione degli anticorpi (4,1-4,3) e l'amplificazione tyramide (4,4-4,5) passi sono stati anche ripetuti, ma più spesso si traduce in un segnale di scarsa rumore a causa della fluorescenza di fondo maggiore.
I reagenti più sensibili sono i Click-iT Additivo Buffer EdU (componente G) e il coniglio anti-Oregon Green-HRP anticorpi (componente I) dalla Click-iT Assay kit EdU micropiastre. Il Click-iT Additivo Buffer EdU trasforma gradualmente giallo nel tempo se conservato a 4 ° C e tra 6-12 mesi scurisce notevolmente. Conservazione del Click-iT Additivo Buffer EdU a -20 ° C alleviare questo problema. L'etichettatura e le fasi di amplificazione tyramide funzionano meglio quando il coniglio anti-Oregon Green-HRP anticorpale è utilizzato entro 4-6 mesi di ricostituire in MilliQ dH 2 O.
L'etichettatura mite del EdU nel mtDNA permette di confronti diretti con l'aggiunta di marcatori cellulari 9-11 e migliora l'utilità di questa tecnica su altri analoghi della timidina, come il 5-bromo-2-deossiuridina (BrdU), che richiede un trattamento duro per recuperare il suo epitopo all'interno del DNA. Il nostro laboratorio 11-12 e 13-15 gli altri hanno utilizzato con successo BrdU per etichettare mtDNA. La tecnica di etichettatura BrdU è simile a quello utilizzato per l'istruzione, ma ha alcune importanti differenze (Figura 5). Dopo la fase di permeabilizzazione sopra elencati (3.1-3.2), l'epitopo BrdU viene recuperato con un passo denaturazione (2 N HCl per 30 minuti a 37 ° C seguita da tre lavaggi in PBS 1X). La BrdU viene etichettato con un anticorpo primario contro BrdU (Vector Laboratories diluito 1:50 in soluzione 1% bloccando dal kit TSA e incubate overnight a 4 ° C). Un passo anticorpo secondario è necessario per amplificare il segnale BrdU (capra anti-IgG di topo coniugato con HRP dal kit TSA # 2 e # 5, diluito 1:100 in soluzione 1% di blocco e incubate per 45 minuti a temperatura ambiente) prima i passi TSA (4,3-4,5 sopra). Avere due analoghi della timidina, Edu e BrdU, di mtDNA etichetta è vantaggioso per l'esecuzione di esperimenti di doppia etichetta dove mtDNA possono essere etichettate in sequenza di impulsi di etichettatura paradigmi 11.
Il nostro laboratorio sta usando il EdU BrdU e l'etichettatura di mtDNA per verificare la regolamentazione della biogenesi mitocondriale nel contesto di neuropatia diabetica, una complicanza comune del diabete. Abbiamo utilizzato con successo l'amplificazione del segnale per misurare le variazioni del mtDNA biogenesi in singoli neuroni 11-12. Questa tecnica è utile in altri esperimenti progettati per esplorare i meccanismi di replicazione del DNA mitocondriale e del fatturato o per identificare i farmaci che inibiscono la sintesi del DNA mitocondriale. Inoltre, i principi di base di amplificare segnali Edu e BrdU può essere applicato ad altri studi che misurano la replicazione del DNA o riparazione.
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto dal National Institutes of Grants Salute NS-38849 e DK-076160, la Juvenile Diabetes Research Foundation Centro per lo Studio delle complicanze nel diabete, il Programma di neurologia ricerca e scoperta e la A. Alfred Taubman Medical Research Institute presso l' Università del Michigan. Questo lavoro usato la morfologia e l'analisi Core Image del Centro Michigan Diabetes Research e formazione, finanziato dal National Institutes of Health di Grant 5P60 DK-20572 presso l'Istituto Nazionale del Diabete e digestivi e malattie renali. Gli autori ringraziano T. Scott Clarke da Molecular Probes / Invitrogen per i suoi preziosi consigli sui vari Click-iT EdU etichettatura kit e la donazione generosa di reagenti per sostenere lo sviluppo iniziale della tecnica di amplificazione Edu.
Materiali
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | Fisher Scientific | 12-545-82 | |
| 245 mm x 245 mm BioAssay Dish | Corning | 431111 | |
| Parafilm M | Fisher Scientific | PM-996 | |
| paraformaldehye | Sigma-Aldrich | P6148 | |
| Triton X-100 | Sigma-Aldrich | T-8532 | |
| Phosphate buffered saline 10X solution | Fisher Scientific | BP399 | |
| Transfer Pipet | Fisher Scientific | 13-711-7M | |
| Hydrogen peroxide, 30% in water | Fisher Scientific | BP2633 | |
| Click-iT EdU Microplate Assay Kit | Invitrogen | C10214 | |
| TSA Kit #12, with HRP—goat anti-rabbit IgG and Alexa Fluor 488 tyramide | Invitrogen | T20922 | |
| TSA Kit #15, with HRP–goat anti-rabbit IgG and Alexa Fluor 594 tyramide | Invitrogen | T20925 | |
| ProLong Gold antifade reagent with DAPI | Invitrogen | P-36931 | |
| Polyclonal rabbit Neurofilament antibody | Chemicon International | AB1981 | |
| Mouse monoclonal anti-BrdU antibody | Vector Laboratories | VP-B209 | |
| TSA Kit #2, with HRP—goat anti-mouse IgG and Alexa Fluor 488 tyramide | Invitrogen | T20912 | |
| TSA Kit #5, with HRP—goat anti-mouse IgG and Alexa Fluor 594 tyramide | Invitrogen | T20915 |
Riferimenti
- Chan, D. C. Mitochondrial fusion and fission in mammals. Annu. Rev. Cell Dev. Biol. 22, 79-99 (2006).
- Dimmer, K. S., Scorrano, L. (De)constructing mitochondria: what for. Physiology (Bethesda). 21, 233-241 (2006).
- Suen, D. F., Norris, K. L., Youle, R. J. Mitochondrial dynamics and apoptosis. Genes Dev. 22, 1577-1590 (2008).
- Clay Montier, L. L., Deng, J. J., Bai, Y. Number matters: control of mammalian mitochondrial DNA copy number. J. Genet. Genomics. 36 (3), 125-131 (2009).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proc Natl Acad Sci U.S.A. 105, 2415-2420 (2008).
- Buck, S. B., Bradford, J., Gee, K. R., Agnew, B. J., Clarke, S. T., Salic, A. Detection of S-phase cell cycle progression using 5-ethynyl-2'-deoxyuridine incorporation with click chemistry, an alternative to using 5-bromo-2'-deoxyuridine antibodies. Biotechniques. 44, 927-929 (2008).
- Yu, Y., Arora, A., Min, W., Roifman, C. M., Grunebaum, E. EdU incorporation is an alternative non-radioactive assay to [(3)H]thymidine uptake for in vitro measurement of mice T-cell proliferations. J Immunol Methods. 350 (1-2), 29-35 (2009).
- Heusden, J. V. a. n., Jong, P. d. e., Ramaekers, F., Bruwiere, H., Borgers, M., Smets, G. Fluorescein-labeled tyramide strongly enhances the detection of low bromodeoxyuridine incorporation levels. J. Histochem. Cytochem. 45 (2), 315-319 (1997).
- Cappella, P., Gasparri, F., Pulici, M., Moll, J. A novel method based on click chemistry, which overcomes limitations of cell cycle analysis by classical determination of BrdU incorporation, allowing multiplex antibody staining. Cytometry A. 73, 626-636 (2008).
- Kaiser, C. L., Kamien, A. J., Shah, P. A., Chapman, B. J., Cotanche, D. A. 5-Ethynyl-2'-deoxyuridine labeling detects proliferating cells in the regenerating avian cochlea. Laryngoscope. 119, 1770-1775 (2009).
- Lentz, S. I., Edwards, J. L., Backus, C., McLean, L. L., Haines, K. M., Feldman, E. L. Mitochondrial DNA (mtDNA) Biogenesis: Visualization and Duel Incorporation of BrdU and EdU Into Newly Synthesized mtDNA In. Vitro. J Histochem Cytochem. 58 (2), 207-218 (2010).
- Edwards, J. L., Quattrini, A., Lentz, S. I., Figueroa-Romero, C., Cerri, F., Backus, C., Hong, Y., Feldman, E. L. Diabetes regulates mitochondrial biogenesis and fission in mouse neurons. Diabetologia. 53 (1), 160-169 (2010).
- Davis, A. F., Clayton, D. A. In situ localization of mitochondrial DNA replication in intact mammalian cells. J. Cell Biol. 135, 883-893 (1996).
- Magnusson, J., Orth, M., Lestienne, P., Taanman, J. W. Replication of mitochondrial DNA occurs throughout the mitochondria of cultured human cells. Exp. Cell. Res. 289, 133-142 (2003).
- Amiri, M., Hollenbeck, P. J. Mitochondrial biogenesis in the axons of vertebrate peripheral neurons. Dev. Neurobiol. 68, 1348-1361 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon