Method Article
Erkennung von Histon-Modifikationen in Pflanzenblättern
In diesem Artikel
Zusammenfassung
Eine zuverlässige und nützlicher Ansatz zur Histon-Modifikationen auf spezifische Pflanzen-Gene erkennen beschrieben. Der Ansatz kombiniert Chromatin-Immunopräzipitation (ChIP) und real-time quantitative PCR. Es ermöglicht den Nachweis von Histon-Modifikationen auf bestimmte Gene mit eine Rolle in verschiedenen physiologischen Prozessen.
Zusammenfassung
Chromatin-Struktur ist für die Regulation der Genexpression in Eukaryoten wichtig. In diesem Prozess spielen die Chromatin-Remodeling, DNA-Methylierung und kovalente Modifikationen an der Amino-terminalen Enden der Histone H3 und H4 wesentliche Rollen 1-2. H3 und H4 Histon-Modifikationen zählen die Methylierung von Lysin und Arginin, Acetylierung von Lysin und Phosphorylierung von Serin-Resten 1-2. Diese Änderungen sind entweder mit Genaktivierung, Unterdrückung oder eine grundierte Stand der Gen, das eine schnellere und eine starke Aktivierung des Ausdrucks unterstützt nach Wahrnehmung der entsprechenden Signale (Mikrobe-associated molecular patterns, Licht, Hormone, etc.) 3-7 verbunden.
Hier stellen wir ein Verfahren zur zuverlässigen und empfindlichen Nachweis von spezifischen Chromatin-Modifikationen auf ausgewählte pflanzliche Gene. Die Technik basiert auf der Vernetzung von (modifizierten) Histone und DNA mit Formaldehyd 8,9, Extraktion und Beschallung von Chromatin, Chromatin-Immunopräzipitation (ChIP) mit Modifikation-spezifische Antikörper 9,10 basiert, de-Vernetzung von Histon-DNA-Komplexe und Gen-spezifischen real-time quantitative PCR. Der Ansatz hat sich zum Nachweis von spezifischen Histon-Modifikationen mit C 4 Photosynthese in Mais 5,11 und systemische Immunität in Arabidopsis 3 zugeordnet nützlich.
Protokoll
1. Die Vernetzung von Pflanzenmaterial
- Ernte-1-2g Arabidopsis Blättern (6 bis 10cm Rosette Durchmesser) in einem 50-ml Plastikröhrchen und füllen bis zu 40ml mit Vernetzung Puffer.
- Stellen Sie sicher, Blätter bleiben eingetaucht, zum Beispiel durch Füllung eine maßgeschneiderte Filterschwamm oben rechts Puffer Oberfläche. Dann legen Sie die Röhrchen in einem Exsikkator.
- Vakuum für 10 Minuten zu infiltrieren. Zu jedem Röhrchen add 2,5 ml 2M Glycin, um die Vernetzung zu stoppen und umzukehren Rohre gleich Dispersion von Glycin zu rechtfertigen.
- Vacuum weitere 5min zu infiltrieren und zu verwerfen Flüssigkeit während der Ernte lässt auf ein Sieb.
- Wash Blätter zweimal mit 1 l Wasser in ein Becherglas, dann trockene Blätter gründlich mit Küchenpapier.
- Sammeln Sie Laub in einem frischen Kunststoffrohr und frieren in flüssigem Stickstoff. Shop Blätter bei -80 ° C bis Chromatin isoliert.
2. Isolierung von Chromatin
- Blätter wurden mit Mörtel und Stempel, die in flüssigem Stickstoff bis zur Verwendung aufbewahrt wurden Boden.
- Transfer-Pulver zu einem 50-mL Plastikröhrchen und suspendieren in 30 mL Extraktionspuffer Nr. 1.
- Inkubieren 15min bei 4 ° C auf einem Überkopfschüttler.
- Filtrat Suspension durch vier Schichten Miracloth in ein frisches 50-mL Plastikröhrchen.
- Spin für 20min bei 2.800 xg bei 4 ° C.
- Überstand entfernen und Suspend-Pellet mit einem Pinsel in 1ml Extraktionspuffer # 2. Fügen Sie zunächst ein kleines Volumen von Puffer Nr. 2, dann auszusetzen Pellet mit einem Pinsel, und fügen Sie dann den Rest des Puffers.
- Transfer-Suspension auf eine 1,5 ml Mikrozentrifugenröhrchen und Spin für 10min bei 12.000 xg bei 4 ° C.
- In der Zwischenzeit bereiten 2ml-Mikrozentrifugenröhrchen mit 1,5 ml Extraktionspuffer Nr. 3.
- Entfernen Sie aus dem gesponnen Rohr (Schritt 2,7) Überstand und Pellet in auszusetzen 300μL Extraktionspuffer Nr. 3. Fügen Sie zunächst ein kleines Volumen von Puffer Nr. 3, auszusetzen Pellet mit einem Pinsel, und fügen Sie dann die restlichen Puffer.
- Sorgfältig pipettieren des suspendierten Pellets (Schritt 2,9) auf der Oberseite des 1,5 ml Extraktionspuffer Nr. 3 in Schritt 2,8 zubereitet. Achten Sie darauf, die beiden Phasen nicht zu vermischen. Spin für 1h bei 16.000 xg bei 4 ° C.
- Überstand entfernen und Suspend Chromatin Pellet mit einem Pinsel in 300μL der Beschallung Puffer.
- Ultraschallbad Chromatin mit einer Sonotrode, oder in einem Ultraschallbad, um eine DNA-Größe von ca. 400bp. Als Beschallung, stellen Sie sicher, Chromatin nicht über ca. 30 ° C erhitzt werden, um wärmeempfindlichen Vernetzungen zu schützen. Die Dauer der Beschallung muss experimentell bestimmt werden, da es deutlich variiert zwischen den verschiedenen Ultraschall-Geräten. Zur Orientierung sind die Einstellungen für zwei verschiedene Geräte zur Verfügung:
Für Beschallung mit einem Bandelin Marke Sonotrode aussetzen Chromatin in 0,6 ml Mikrozentrifugenröhrchen fünfmal bis 20 Ausbrüche mit den Einstellungen 25% Leistung, Zyklus = 50. Während dabei kühl Probe nach 20 th platzt in ein Eisbad.
Für Beschallung mit einem Diagenode Bioruptor Ultraschallbad, füllen Bad mit Wasser und Eis und beschallen zehnmal für 30 Sekunden in 1,5 ml Mikrozentrifugenröhrchen mit der Einstellung "hoch". Nach 30 Sekunden, kühlen Proben für weitere 30 Sekunden.
- Spin beschallten Probe für 5 min bei 16.000 g bei 4 ° C zu fällen ungelöstes Material. Der Überstand kann direkt für die Immunpräzipitation eingesetzt werden. Alternativ können die Proben Schock in flüssigem Stickstoff eingefroren und bei -80 ° C bis zur weiteren Verwendung werden.
3. Immunpräzipitation
- Bereiten Sie 2-mL Mikrozentrifugenröhrchen mit 40 ul Protein-A Agarose und 1,8 ml der Antikörper-Bindungspuffer. Add 200 ul des Chromatins Vorbereitung (Schritt 2,13).
- Die Röhrchen für 1 Stunde auf einem Überkopfschüttler.
- Discard-Protein-A Agarose-Beads und die Nutzung für die Immunpräzipitation Überstand.
- Entfernen eines 40 ul-Aliquot auf Chromatin-Konzentration ("Input") zu bestimmen.
- Add 30μL Protein-A-Agarose und die Höhe der Änderung-spezifischen Antikörper, die in der unten stehenden Tabelle der spezifischen Reagenzien und Geräte für 400μL beschallt Chromatin Federung angegeben ist.
- Inkubieren 2.5h oder über Nacht auf einem Überkopfschüttler bei 4 ° C.
- Spin down Perlen für 2min bei 440 x g.
- Wash Bead-Pellet mit 900μL von jedem Waschpuffer (siehe Liste unten). Für jeden Puffer inkubieren Perlen in den Puffer für 10min auf einem Überkopfschüttler bei 4 ° C, dann für 2min Spin bei 440 xg, Überstand verwerfen, und fügen Sie den nächsten Puffer.
- Low Salz Waschpuffer
- Hohe Salz Waschpuffer
- LiCl Waschpuffer
- TE Waschpuffer
- Nach dem letzten Waschen vollständig entfernen Sie alle verbleibenden Puffer.
4. De-Vernetzung von Histonen und DNA, und Aufreinigung von DNA ausgefällt
- Add 100 &mgr; von de-Vernetzung Puffer, um die Agarose Pellet und 40 ul 'Eingang' Probe aus Schritt 3.4, Mix für 5min auf einem Vortex-Mixer, Spin Proben und InkubationDebatte bei 65 ° C über Nacht.
- Purify DNA mit einem kommerziellen DNA oder PCR Purification Kit.
- Typischerweise 5μl einer 1:5-Verdünnung der letzten Spalte Eluat sind ausreichend, um durchzuführen konventionellen Locus-spezifischen real-time quantitative RT-PCR (RT-qPCR).
5. Tabelle der Puffer
| Crosslinking-Puffer | |
| Natriumbutyrat | 10 mM |
| Sucrose | 400mm |
| Tris-HCl, pH 8,0 | 10 mM |
| β-Mercaptoethanol | 5mM |
| Formaldehyd | 3% (v / v) |
| Extraktionspuffer Nr. 1 | |
| Natriumbutyrat | 10 mM |
| Sucrose | 400mm |
| Tris-HCl, pH 8,0 | 10 mM |
| β-Mercaptoethanol | 5mM |
| Komplette Protease Inhibitor | 1x |
| Extraktionspuffer # 2 | |
| Natriumbutyrat | 10 mM |
| Sucrose | 250 mM |
| Tris-HCl, pH 8,0 | 10 mM |
| β-Mercaptoethanol | 5mM |
| MgCl 2 | 10 mM |
| Triton X-100 | 1% (w / v) |
| Komplette Protease Inhibitor | 1x |
| Extraktionspuffer # 3 | |
| Natriumbutyrat | 10 mM |
| Sucrose | 1,7 Mio. |
| Tris-HCl, pH 8,0 | 10 mM |
| β-Mercaptoethanol | 5mM |
| MgCl 2 | 2mM |
| Triton X-100 | 0,15% (w / v) |
| Komplette Protease Inhibitor | 1x |
| Beschallung Puffer | |
| Tris-HCl, pH 8,0 | 25mM |
| EDTA-NaOH, pH 8,0 | 5mM |
| SDS | 0,5% (w / v) |
| Komplette Protease Inhibitor | 1x |
| Antikörper Bindungspuffer | |
| Tris-HCl, pH 8,0 | 50mM |
| EDTA-NaOH, pH 8,0 | 1mM |
| NaCl | 150 mM |
| Triton X-100 | 0,1% (w / v) |
| Low Salz Waschpuffer | |
| NaCl | 150 mM |
| SDS | 0,1% (w / v) |
| Triton X-100 | 1% (w / v) |
| EDTA-NaOH, pH 8,0 | 2mM |
| Tris-HCl, pH 8,0 | 20 mM |
| Hohe Salz Waschpuffer | |
| NaCl | 500 mM |
| SDS | 0,1% (w / v) |
| Triton X-100 | 1% (w / v) |
| EDTA-NaOH, pH 8,0 | 2mM |
| Tris-HCl, pH 8,0 | 20 mM |
| LiCl Waschpuffer | |
| Lithiumchlorid | 250 mM |
| Nonidet-P40 | 1% (v / v) |
| Natriumdesoxycholat | 1% (w / v) |
| EDTA-NaOH, pH 8,0 | 1mM |
| Tris-HCl, pH 8,0 | 20 mM |
| TE Waschpuffer | |
| EDTA-NaOH, pH 8,0 | 10 mM |
| Tris-HCl, pH 8,0 | 1mM |
| Decrosslinking Puffer | |
| Tris-HCl, pH 6,8 | 62.5mm |
| NaCl | 200 mM |
| SDS | 2% (w / v) |
| DTT | 10 mM |
6. Repräsentative Ergebnisse:
Die Expression des Arabidopsis thaliana PR2 Verteidigung Gen für ein β-1,3-Glucanase mit antimikrobieller Wirkung 12, wurde von Benzothiadiazol (BTH), ein synthetisches Analogon des Pflanzenhormon Salicylsäure 3 induziert. Abbildung 1 zeigt, dass die Aktivierung von PR2 Ausdruck ist mit einem Anstieg bei der Acetylierung von Lysin-Resten auf Histon-assoziierten H3 und H4 auf den PR2 Promotor. Da Nukleosomen Dichte nimmt während Genaktivierung 13 wurden Histonacetylierung Werte für das Signal mit einem Antikörper gegen eine Invariante Region von Histon 3 erhaltenen normalisiert.
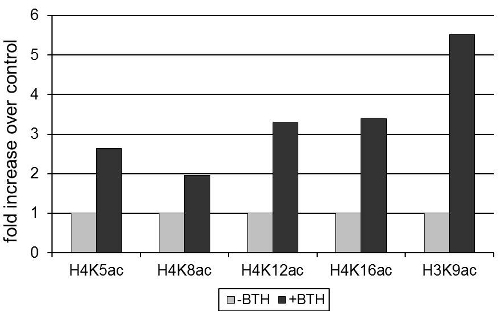
Abbildung 1 BTH induziert Histon-Acetylierung auf die PR2-Promotors in Arabidopsis. Die Pflanzen wurden mit 100 &mgr; BTH oder ein Spritzpulver Kontrolle behandelt. Nach 72h wurden Blätter gesammelt und Chromatin isoliert wurde. Das Signal nach der Fällung mit Acetylierung-spezifischen Antikörpern (wie in der Abbildung angegeben) erhalten wurde, um das Signal durch Fällung mit einem Antikörper erhalten, um eine Invariante Domain von Histon H3 normalisiert. Gefällte Chromatin wurde mittels RT-qPCR quantifiziert. Die Daten werden standardisiert für Histonmodifikationen Ebenen in der Abwesenheit von BTH-Behandlung.
Diskussion
Die meisten kritischen Schritte in dem Protokoll sind die Vernetzung von DNA und Histonen, die Art und Menge der Blattmaterial, Schleifen des Materials und die Beschallung von Chromatin. Für eine detailliertere Erörterung dieser Probleme finden Sie unter refs. 9 und 10.
Beschallung und Vernetzung:
Es ist wichtig, Chromatin während der Beschallung auf Fragmente von ca. 400bp zu stören. Dank intensiver Beschallung wird Chromatin durch Unterbrechung der DNA und Histonen zu zerstören, während unzureichende Beschallung lange Chromatinfragmenten verlassen wird. Diese beeinflussen die Spezifität der Methode, weil Histon-Modifikationen distal von den getesteten locus keine lokalen Änderungen benachteiligt werden. Beschallung Effizienz am besten durch das Sammeln von 20 &mgr; l Chromatin von Proben vor und nach Beschallung, de-Vernetzung DNA und Histonen, Isolierung der DNA und die Bestimmung der Länge der DNA geschert durch Agarosegelelektrophorese überprüft. RNAse sollte, um die Proben vor der Elektrophorese hinzugefügt werden, da das Chromatin Vorbereitung enthält noch große Mengen an RNA in diesem Stadium der Reinigung, die mit der Visualisierung von fragmentierten DNA stören könnten. Die Proben werden für Chromatinimmunpräzipitation nützlich, wenn DNA aus nicht geschert Chromatin ist länger als 10kbp, während DNA aus geschert Chromatin bildet ein Abstrich mit maximaler Signalstärke um 400bp DNA.
Wir verwenden routinemäßig 3% (v / v) Formaldehyd für Chromatin Vernetzung in intakten Blättern, aber 1% (v / v) Formaldehyd könnte in den meisten Fällen ausreichend. In unsere Hände, damit eine niedrige Formaldehyd-Konzentrationen für kürzere Zeiten Beschallung und Hintergrund-Signale in Folge PCR-Reaktionen. Allerdings, unzureichende Mengen an Formaldehyd oft in geringen Reproduzierbarkeit der PCR-Signal zwischen unabhängigen Experimenten führen. Dies könnte durch unzureichende Durchdringung der Blätter mit Formaldehyd bei niedrigen Konzentrationen. Achten Sie auf Ihre Formaldehyd Lager häufig austauschen, wie die Verbindung neigt dazu, sich mit der Zeit polymerisieren. Formaldehyd-Lösungen sind nur für etwa einen Monat stabil, und diese Zeit ist kaum von Methanol Stabilisierung verlängert. Es wird empfohlen, verschiedene Formaldehyd-Konzentrationen Test beim Einrichten experimentellen Bedingungen.
Betrag von Pflanzenmaterial und Schleifen:
Es ist wichtig zu geringe Mengen an Blattmaterial in einem großen Volumen an Puffer zu infiltrieren, um Blätter aneinander kleben zu vermeiden. Blatt kleben oft in unzureichender Infiltration von Formaldehyd. Darüber hinaus wird zu viel Pflanzenmaterial Block Poren der Miracloth Gewebe. Die Mahleffizienz hängt wesentlich mit einem Mörser und Stößel mit einer perfekten Passform. Wir routinemäßig mahlen mit flüssigem Stickstoff gekühlten Mörser in Abwesenheit von zusätzlichen flüssigen Stickstoff dreimal für 50 Sekunden, bis das Material zu einem feinen Pulver gemahlen wird.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von der Deutschen Forschungsgemeinschaft (DFG) und der Exzellenzinitiative der deutschen Bundesregierung und der Länder gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
|---|---|---|---|
| Protein-A-Agarose | Roche | 11 134 515 001 | abhängig von der Antikörper-Klasse |
| Protein-G-Agarose | Roche | 11 243 233 001 | abhängig von der Antikörper-Klasse |
| Anti-hyperacetyl-H4 | Millipore | 06-946 | verwendeten wir 5μL |
| Anti-Acetyl-H4K5 | Millipore | 07-327 | verwendeten wir 5μL |
| Anti-Acetyl-H4K8 | Millipore | 07-328 | verwendeten wir 5μL |
| Anti-Acetyl-H4K12 | Millipore | 07-595 | verwendeten wir 5μL |
| Anti-Acetyl-H4K16 | Millipore | 07-329 | verwendeten wir 5μL |
| Anti-Acetyl-H4K18 | Millipore | 07-354 | verwendeten wir 1μL |
| Anti-Acetyl-H3K9 | Millipore | 07-352 | verwendeten wir 5μL |
| Anti-Acetyl-H3K14 | Millipore | 07-353 | verwendeten wir 1μL |
| Anti-trimethyl-H3K4 | Diagenode | PAB-003 bis 50 | verwendeten wir 5μL |
| Anti-dimethyl-H3K4 | Millipore | 07-030 | verwendeten wir 5μL |
| Anti-Monomethyl-H3K4 | Abcam | ab8895 | 2,5-10 &mgr; l |
| Anti-H3 | Abcam | ab1791 | verwendeten wir 1μL um Histon-Dichte zu überprüfen |
| Bioruptor | Diagenode | UCD-200 an | |
| Sonotrode MS72 | Bandelin | ||
| Miracloth | Calbiochem | 475855 | |
| Komplette Protease Inhibitor | Roche | 11836145001 |
Referenzen
- Eberharter, A., Becker, P. B. Histone acetylation: a switch between repressive and permissive chromatin. EMBO rep. 3, 224-229 (2002).
- Ruthenburg, A. J. Methylation of lysine 4 on histone H3: intricacy of writing and reading a single epigenetic. 25, 15-30 (2007).
- Jaskiewicz, M. Chromatin modification acts as a memory for systemic acquired resistance in the plant stress response. EMBO rep. 12, 50-55 (2011).
- Ng, D. W. Ordered histone modifications are associated with transcriptional poising and activation of the phaseolin promoter. Plant Cell. 18, 119-132 (2006).
- Offermann, S. Illumination is necessary and sufficient to induce histone acetylation independent of transcriptional activity at the C4-specific phosphoenolpyruvate carboxylase promoter in maize. Plant Physiol. 141, 1078-1088 (2006).
- Deng, W. Involvement of the histone acetyltransferase AtHAC1 in the regulation of flowering time via repression of FLOWERING LOCUS C in Arabidopsis. Plant Physiol. 143, 1660-1668 (2007).
- Benhamed, M. Arabidopsis GCN5, HD1, and TAF1/HAF2 interact to regulate histone acetylation required for light-responsive gene expression. Plant Cell. 18, 2893-2903 (2006).
- Bowler, C. Chromatin techniques for plant cells. Plant Journal. 39, 776-789 (2004).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde decrosslinked-chromatin immunoprecipitation. Trends Biochem. Sci. 25, 99-104 (2000).
- Haring, M. Chromatin immunoprecipitation: optimization, quantitative analysis and data normalization. Plant Meth. 3, 11-11 (2007).
- Danker, T. Developmental information but not promoter activity controls the methylation state of histone H3 lysine 4 on two photosynthetic genes in maize. Plant J. 53, 465-474 (2008).
- Uknes, S. Acquired resistance in Arabidopsis. Plant Cell. 4, 645-656 (1992).
- Workman, J. L. Nucleosome displacement in transcription. Genes Develop. 20, 2009-2017 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten