Method Article
Ein PCR-basierte Genotypisierung Methode, zwischen Wildtyp-und Ziersorten Unterscheiden von Imperata cylindrica
In diesem Artikel
Zusammenfassung
Wir bieten eine kostengünstige und schnelle molekulare Genotypisierung Protokoll, das Vielfalt-spezifische PCR-Primer verwendet, die Ziel-DNA-Sequenz Unterschiede innerhalb der Chloroplasten trnL-F Spacer-Region, zwischen Sorten unterscheiden Imperata cylindrica (Cogongrass), die nicht durch Morphologie allein unterschieden werden. Diese Sorten gehören die vom Bund aufgelistet schädliches Unkraut, cogongrass und eng verwandten, weit verbreitete ornamentalen Vielfalt, ich. cylindrica Var. Koenigii (Japanische Blut Gras).
Zusammenfassung
Wild-Typ I. cylindrica (cogongrass) gehört zu den Top Ten der schlimmsten invasiven Pflanzen in der Welt, negative Auswirkungen auf Landwirtschaft und natürliche Ressourcen in 73 verschiedenen Ländern in ganz Afrika, Asien, Europa, Neuseeland, Ozeanien und Amerika 2.1. Cogongrass bildet sich schnell verbreitenden, monodominant Ständen, die eine große Vielfalt an einheimischen Pflanzenarten verdrängen und wiederum bedrohen die heimische Tierwelt, die auf den Vertriebenen einheimischen Pflanzenarten für Futter und Unterkunft ab. Um auf das Problem hinzu, eine ornamentale Vielfalt [I. cylindrica var. koenigii (Retzius)] ist weit verbreitet unter den Namen Imperata cylindrica 'Rubra', Red Baron und Japanisch Blut Gras (JBG) vermarktet. Diese Sorte ist vermeintlich sterilen und nicht-invasive und gilt als eine wünschenswerte Zier für seine rot gefärbten Blättern. Doch unter den richtigen Bedingungen kann JBG produzieren lebensfähige Samen (Carol Holko, 2009 persönliche Mitteilung) und kann zu einem grünen zurückkehren invasive Form, die oft nicht zu unterscheiden ist von cogongrass wie es auf den besonderen Charakteristika des Wildtyp-invasive Vielfalt 4 (Abbildung 1) erfolgt. Dies macht die Identifizierung mit der Morphologie eine schwierige Aufgabe, auch für gut trainierte Pflanzensystematiker. Reversion von JBG zu einem aggressiven grünen Phänotyp ist auch keine Seltenheit. Mit Ablauf der kodierenden und variablen Regionen im kerntechnischen und Chloroplasten-DNA, haben wir bestätigt, dass JBG hat mit dem grünen invasiv in den Staaten Maryland, South Carolina und Missouri zurückgekehrt. JBG ist verkauft worden und in fast jedem Staat gepflanzt in den kontinentalen USA, wo es keine aktive cogongrass Befall. Das Ausmaß des Problems wieder in nicht gut verstanden, weil zurückgekehrt Pflanzen sind nicht dokumentiert und oft zerstört.
Die Anwendung dieser molekularen Protokoll stellt eine Methode zur Identifizierung JBG kehrt und können helfen, diese Sorten von Co-vorkommende einND möglicherweise Hybridisierung. Cogongrass ist ein obligat outcrosser und, wenn mit einem anderen Genotyp überschritten, können lebensfähige Wind verteilt Samen, die über weite Entfernungen cogongrass 7.5 zu verbreiten. JBG hat eine etwas andere Genotypen als cogongrass und kann in der Lage, lebensfähige Hybride mit cogongrass bilden. Um auf das Problem hinzu, ist JBG mehr Kälte und Schatten toleranter als cogongrass 8-10, und Genfluss zwischen diesen beiden Sorten ist wahrscheinlich Hybriden, die aggressiver sind zu generieren, Schatten tolerant, und winterhart als Wildtyp-cogongrass. Während Wildtyp-cogongrass befällt derzeit über 490 Millionen Hektar weltweit, im Südosten der USA befällt es über 500.000 Hektar und ist in der Lage besetzen die meisten von den USA, wie es breitet sich schnell nach Norden wegen seiner breiten Nische und geographische Potenzial 3,7,11. Das Potenzial für eine genetische Kreuzung ist ein ernstes Problem für die USDA-APHIS Federal Noxious Wochenprogramm. Derzeit verbietet die USDA-APHIS JBG in Staaten where gibt es große cogongrass Befall (z. B. Florida, Alabama, Mississippi). Allerdings kann verhindern, dass die beiden Sorten aus der Kombination als schwieriger erweisen als cogongrass JBG und erweitern ihre Distributionen. Ferner ist die Verteilung der JBG wieder derzeit nicht bekannt und ohne die Möglichkeit, diese Sorten durch Morphologie zu identifizieren, einige cogongrass Befall kann das Ergebnis der JBG kehrt sein. Leider aktuellen molekularen Methoden der Identifikation in der Regel verlassen sich auf AFLP (Amplified Fragment Length Polymorphismen) und DNA-Sequenzierung, die beide zeitaufwändig und kostspielig sind. Hier präsentieren wir die erste kostengünstige und zuverlässige PCR-basierter molekularer Genotypisierung Methode, um genau zwischen cogongrass und JBG zurückkehren zu unterscheiden.
Protokoll
1. Probenentnahme und Konservierung
Diese Methode wurde entwickelt und getestet, mit frisch, gefroren, getrocknet und vor kurzem Blattgewebe.
- Identifizieren cogongrass und / oder JBG Gewebe mit Hilfe eines Taxonomen, die im Gras Artbestimmung spezialisiert. Mit seinen leuchtend roten Blättern, ist einfach zu ornamentalen JBG optisch von Wildtyp-und cogongrass JBG unterscheiden zurückgreifen, jedoch sind cogongrass und JBG zurückkehren kaum von einander. Cogongrass und der JBG zurückgekehrt Phänotyp haben grün, Blätter länger, wesentlich größer und länger Wurzelstöcke, und vieles mehr Blattfläche als JBG 12 (Abbildung 1).
- Frische Blattgewebe bietet die am häufigsten vorkommende und höchste Qualität DNA und kann von Feld oder Gewächshaus angebaut I. abgeholt werden cylindrica Pflanzen. Wenn frisches Gewebe verwendet werden soll, Extraktion von DNA innerhalb von 3 Stunden nach der Entnahme, um zu verhindern Abbau. Ansonsten bereiten Gewebe für die Lagerung, keeping das Gewebe kühl und vor direkter Sonneneinstrahlung geschützt.
- Um Gewebe zur DNA-Extraktion zu einem späteren Zeitpunkt zu speichern, ist die optimale Verfahren zum Einfrieren und Speichern des Gewebes bei -80 ° C unmittelbar. Lassen Sie das gefrorene Gewebe zu tauen, bevor die DNA-Extraktion. Übertragen Sie die gefrorenen Gewebe zu flüssigem Stickstoff vor den Schleifen Schritte der DNA-Extraktion zu verhindern Auftauen.
- Wenn ein -80 ° C Gefrierschrank ist nicht verfügbar, sofort trocken das Gewebe. Zur Aufbewahrung, ein Gewebe in einer Papierhülle und speichern den Umschlag in getrocknetem Kieselgel oder andere aktive Trockenmittel bei Raumtemperatur. Eine kleine Menge des Indikators Siliciumdioxid gemischt mit nicht anzeigenden Siliciumdioxid wird sichergestellt, dass Siliciumdioxid vollständig dehydriert und für Trocknen Pflanzengewebe.
- Verwendung von mindestens 10 mal mehr Kieselgel als frische Blattgewebe Gew.. Pflanzengewebe sollte innerhalb von 24 Stunden trocknen lassen. Die Qualität und Quantität der DNA durch trockene Lagerung im Laufe der Zeit reduziert werden (wie im Fall von Herbarium-Exemplare).
2. DNA-Extraktion
Um DNA aus Pflanzengewebe zu extrahieren, folgen Sie dem DNeasy Plant Mini Kit (Qiagen, Valencia, CA; Cat # 69104 oder 69106) den Anweisungen des Herstellers mit einer kleinen Modifikation. Anstelle der Verwendung der vorgeschlagenen weniger als 100 mg frischem Gewebe oder weniger als 20 mg trockenes Gewebe für jede Spalte, schleifen größer als 100 mg, und übertragen Sie dann 100 mg von frischem oder gefrorenem Gewebe (oder> 20 mg von trockenen Geweben) in die entsprechenden Röhrchen für die Extraktion. Kern-und Plastiden-DNA wird gleichzeitig extrahiert.
- Bevor Sie diese Vorgänge, stellen Sie sicher, dass Ethanol wurde auf Puffer AP3 / E und AW aufgenommen.
- Grind> 100 mg von frischem oder gefrorenem Blattgewebe (oder> 20 mg trockenes Blattgewebe) zu einem feinen Pulver mit drei Runden von flüssigem Stickstoff mit Schleifen in einem gekühlten Mörser und Stößel. Zu wenig Störung des Ausgangsmaterials oder unzureichende Lyse kann auch führen zu niedrigeren Ausbeuten an DNA. Vorsichtig schleifen die TISSue und nicht überlasten die Spalten mit zu viel Gewebe.
- Übertragen Sie 100 mg gefrorene Pulver aus frischen oder gefrorenen Gewebe (oder 20 mg Pulver von trockenen Geweben) zu einem 1.5 ml Zentrifugenröhrchen mit 400 ul Puffer AP1 und 4 ul RNase A. Jedes Rohr in einem kleinen Schrank auf einer platziert werden können auszugleichen, um die korrekte Gewicht des Gewebes pro Röhre zu überwachen.
- Schütteln oder Probe (n) zu mischen und Inkubation für 10 min bei 65 ° C, Umdrehen des Röhrchens (en) 2-3 mal während der Inkubation.
- Fügen Sie 130 ul Puffer AP2 zu jeder Probe. Mischen durch Invertieren des Röhrchens (n) mehrmals, und Inkubation für 5 min auf Eis.
- Pipettieren Sie jeweils Lysat in ein separates QIAshredder Mini Spin-Säule, und platzieren Sie jede Spalte in einer 2-ml-Collection-Tube (im Lieferumfang des Kits). Zentrifugen-Spalte (n) für 2 min bei 20.000 xg (~ 14.000 rpm), und transferieren jede Flow-Through-Fraktion in eine neue Röhre (nicht im Kit enthalten) ohne Unterbrechung jedes Pellet gebildet.
- In 1,5 Volumina Puffer AP3 /E, und durch Pipettieren gründlich mischen.
- Übertragen Sie 650 ul der Mischung in eine DNeasy Mini Spin-Säule in ein 2 ml Collection Tube. Zentrifugen-Spalte (n) für 1 min bei 6.000 xg (~ 8.000 Umdrehungen pro Minute), und entsorgen Sie die Flow-Through. Wiederholen Sie diesen Schritt mit der restlichen Mischung für jede Probe.
- Legen Sie die Spin-Säule (n) in ein neues 2 ml Collection Tube (n), und fügen Sie 500 ul Puffer AW auf der Oberseite jeder Spalte. Zentrifugen-Spalte (n) für 1 min bei 6.000 xg (~ 8.000 Umdrehungen pro Minute), und entsorgen Sie Flow-Through.
- Fügen Sie eine weitere 500 ul Puffer AW auf der Oberseite jeder Spalte. Säule für 2 min bei 20.000 × g (~ 14.000 rpm). Dieser Schritt wird die Säule trocknen, so dass jedes restliche Ethanol in den Puffern, die PCR inhibieren kann enthalten.
- Übertragen Sie jede Spin-Säule in ein neues 1,5-ml-Probenröhrchen. Geben Sie 100 ul Puffer AE an die Spitze jeder Säule für die Elution, und inkubieren Spalte (n) für 5 min bei Raumtemperatur. Zentrifuge Spalte (n) für 1 min bei 6.000 × g (~ 8.000 rpm), um die DNA zu sammeln.
- Wiederholen Sie diese Schritte einmal Elution, Elution der DNA in der gleichen 1.5 ml Zentrifugenröhrchen zu 200 ul Probe ergeben. Shop DNA-Proben bei -20 ° C bis zur Verwendung. DNA-Konzentrationen sind abhängig von Gewebetyp und Lagerbedingungen. Optimale Ausbeuten werden erzielt, wenn eluierenden DNA mit einem Gesamtvolumen von 200 ul Puffer AE, jedoch Konzentrationen können erhöht werden, wenn Elutionsvolumen, so wenig wie 50 pl werden reduziert werden.
3. Nachweis der DNA-Qualität und Quantität
- Testen Sie die Qualität und Quantität der extrahierten DNA vor der PCR-Setup mit einem Spektralphotometer oder Fluorimeter und Gelelektrophorese. Dies wird dazu beitragen, den Erfolg der weiteren Schritte.
- Mit einem Spektralphotometer, Test-DNA-Qualität und Quantität. Gute Ausbeuten DNA sollte zwischen 50 und 150 sein ng / ul mit 260/280 und 230/280-Verhältnisse von ca. 2,0. Als Beispiel zeigt Abbildung 2 qualitativ hochwertiger Ergebnisse mit dem NanoDrop Spektralphotometer (ThermoScientific, Wilmington, DE).
- Führen Elektrophorese unter Verwendung eines Standard-1% Agarosegel. Überprüfen Sie das Vorhandensein von relativ großen Bands (> 10 Kb) mit nicht zu kleinen Schlieren von RNA-Kontamination (Abbildung 3).
- Wenn die DNA-Konzentration hoch ist, verdünnter DNA-Proben auf 70 ng / ul für die nachfolgenden Schritte.
4. PCR-Primer
Die PCR-Primer in diesem Protokoll verwendet werden auf Sequenzunterschiede zwischen dem Plastid trnL-F Spacer-Region der cogongrass und JBG Genotypen bezogen. Diese Unterschiede in der Form von SNPs (single nucleotide polymorphisms) und indels (Einfügungen und Löschungen), die die Entwicklung von Vielfalt-spezifische Primer durch Platzierung der Primer an den Standorten der einzigartigen Sequenzen (Abbildung 4) erlaubt zu kommen.
- Die Qualität der Primer können einen erheblichen Einfluss auf die PCR-Ergebnisse. Bestellen Primer von einem seriösen Unternehmen. Wir bestellen unsere Primern aus Oblique Bio, Inc. (:/ / Www.obliquebio.com/web/ "target =" _blank "> http://www.obliquebio.com/web/, Huntsville, AL), Anfordern nur Standard-Verfahren entsalzt.
- trnL-F positive Kontroll-Primer: Primer-Set Dieser verstärkt das plastidäre trnL-F Spacer-Region der meisten Taxa 11-13 Gras und dient als gute positive Kontrolle (was zu einem 890 bp-Bande).
Name Reihenfolge trnF (GAA)-F 5'-ATTTGAACTGGTGACACGAG-3 ' trnL (5'-Exon)-C 5'-CGAAATCGGTAGACGCTACG-3 ' - Wildtyp-Primer cogongrass: Diese Primer sind zu cogongrass und nicht verstärken die trnL-F-Region von JBG Genotypen. Die eingestellten Ergebnisse in einer Band, die 595 bp ist.
Name Sequss trnLF-C-F1 5'-TCCACTTTTTTGAAAAAACAAGTGCAA-3 ' trnLF-C-R1 5'-GCCGATACTCTAATAAATAAAAAAAAAAAAGAAAT-3 ' - JBG und JBG Revert Primer: Diese Primer sind zu JBG Genotyp und nicht verstärken die trnL-F Region cogongrass. Die eingestellten Ergebnisse in einer Band, die 594 bp ist.
Name Reihenfolge trnLF-R-F2 5'-CCAAATCCACTTTTTTGAAAAAACAAGTGGTT-3 ' trnLF-R-R2 5'-CGAGATTCCTTGCCGATACTCTAATAAAA-3 ' - Resuspendieren in jeder Primer ausreichend Volumen der Nuclease-freiem ddH 2 O, um eine 100 mM Stammlösungen, die bei -20 ° C, langen Zeitraum gelagert werden kann erhalten.
- Verdünnen Sie jedes Primer-Lager bis 12 mm vor dem PCR-Setup-Schritte.
5. PCR-Setup
DNA-Extraktionen sind amplifiziert unter Verwendung jeder der obigen Primer-Sets in PCR-Reaktionen. Fügen Sie eine positive Kontrolle, um sicherzustellen, dass alle PCR-Reagenzien arbeiten gut und kann eine Band zu erzeugen. Fügen Sie eine negative Kontrolle, um sicherzustellen, dass keines der Reagenzien mit unerwünschten DNA kontaminiert sind. Die negative Kontrolle enthält kein-Vorlage und sollten in keiner Band Produktion führen.
- Bereiten Sie alle Reaktionen in dünnwandigen PCR-Gefäße zur besseren Wärmeübergang zwischen dem Thermocycler-Block und der Probe zu ermöglichen. Wir empfehlen die Verwendung kommerziell erhältlichen Aerosol-freie Pipettenspitzen zur Vermeidung von Kontamination.
- Für jede DNA-Probe isoliert, bis 50 pl PCR-Reaktionen mit jeder der oben genannten Primer-Sets in 0,2-ml dünnwandigen PCR-Gefäße, indem die folgenden Reagenzien auf Eis in der unten angegebenen Reihenfolge eingestellt. Wenn mehrere Proben hergestellt werden, machen einen Cocktail enthält alle Reagenzien, mit Ausnahmeder DNA-Vorlage, um einheitliche Bedingungen zwischen allen Reaktionen.
PCR Reagenz Volumen der eingesetzten Abschließende Konzentrationsvermögen Nuklease-freiem ddH 2 O 40,5 ul Vorteil 2 10X PCR-Puffer (Clontech, CA) 5,0 ul 10% (v / v) Vorteil UltraPure PCR-dNTP-Mix (jeweils 10 mM, Clonetech, CA) 1,0 ul 0,2 mM Primer 1 (12 &mgr; m in ddH 2 O) 1,0 ul 0,24 pM Primer 2 (12 &mgr; m in ddH 2 O) 1,0 ul 0,24 pM Vorteil 2 Polymerase Mix (Clontech, CA) 0,5 ul 1% (v / v) DNA-Extraktion(70 ng / Reaktion; anpassen ddH 2 O Volumen je nach Bedarf) 1,0 ul 1,4 ng / ul Gesamt: 50,0 ul - Um sicherzustellen, dass alle PCR-Reagenzien sind gut klappt, richten die positive Kontrolle mit Hilfe der positiven Kontroll-Primer. Diese Primer-Set funktioniert genauso gut für cogongrass, JBG, JBG zurückkehren und andere Gräser und werden in einer Band, die 890 bp führen.
- Richten die negative Kontrolle unter Verwendung der gleichen Kontroll-Primer als positive Kontrolle eingestellt, mit ddH 2 O anstelle der DNA-Extraktion. Wenn alle Reagenzien frei von DNA verunreinigt sind, wobei diese Reaktion in keiner Band führen.
- Wenn die DNA-Konzentration niedrig ist, können weitere DNA zu jeder Reaktion gegeben werden, die Anpassung der Menge an Nuklease-freiem ddH 2 O verwendet, um das gesamte Reaktionsvolumen auf 50 ul zu bringen. Verwenden Sie nicht mehr als 5 mu l DNA (10% der das Gesamtvolumen) pro reaction, als mögliche Verunreinigungen in den DNA-Proben enthaltenen PCR-Reaktionen zu inhibieren.
6. PCR-Cycling
- Führen Sie die PCR-Amplifikationen in einem Thermocycler mit beheizbarem Deckel mit folgendem PCR-Cycling-Parameter ausgestattet ist. Wir verwenden die Mastercycle Pro S Thermocycler (Eppendorf, Hauppauge, NY) gesetzt, um mit Standard-Temperaturrampen Bedingungen zu arbeiten. Jede Qualität Thermocycler sollten gute Leistungen.
Zyklus Denaturierung Annealing Polymerisation 1 2 min bei 95 ° C 2 30 Sekunden bei 95 ° C 30 Sekunden bei 61 ° C 90 s lang bei 68 ° C 35 Zyklen 3 5 min bei 68 ° C Halten bei 4 ° C, bis die Probe entnommen - Optimieren der Bedingungen für die PCR (einschließlich Primer Annealing-Temperatur, Verlängerungszeiten, die Anzahl der Zyklen) nach Bedarf in Abhängigkeit von der Qualität der DNA, Primer, verwendet Taq-Polymerase oder Art der Thermocycler. Wir empfehlen unter Verwendung eines Gradienten in der Lage Thermocycler bei der Bestimmung der optimalen Glühtemperaturen.
- Wenn der verwendete Thermocycler keinen beheizbaren Deckel, 1 Tropfen Mineralöl auf der Oberseite jeder Probe der Verdunstung während der PCR-Cycling zu verhindern.
7. Gel-Elektrophorese von PCR-Produkten
Um die Ergebnisse der Analyse visualisieren, getrennte PCR-Produkte auf einem 1% Agarosegel-Elektrophorese unter Verwendung von Standardverfahren.
- Kombinieren Sie 2 ul einer Standard-DNA-Ladepuffer (typischerweise ein 5x oder 6x-Lösung) mit 5 ul jeder amplifizierte PCR-Produkt.
- Laden Proben auf ein 1% Agarosegel, das Ethidiumbromid (Ethidiumbromid zum Anfärben der DNA) mit e gemachtither TAE oder SB (Natriumborat) Puffersysteme 16. Wir verwenden 1 ul einer 10 mg / ml EtBr Stammlösung pro 100 ml 1% Agarose (0,1 ug / ml).
- Führen Proben bei ~ 120 V, bis der Farbstoff vor ¾ des gesamten Länge des Gels.
- Unter UV-Licht (z. B. eine kurzwellige UV-Lichtbox) auch die resultierenden Banden zu sehen, ob eine entsprechende Fragment amplifiziert.
- Dokumentieren Sie das Gel und die daraus resultierenden Bänder mit einer der verfügbaren Foto-Dokumentations-System oder eine Kamera.
8. Repräsentative Ergebnisse
Nach Darstellung der PCR-Produkte, hat eine einzigartige cogongrass Bandenmuster im Vergleich zu der JBG oder zurückgesetzt JBG (5). Für jede DNA-Probe sollte die trnL-F Positivkontrolle Primer-Set in einem einzigen Band mit hoher Intensität bei ~ 890 bp führen. Diese prüft, ob alle PCR-Reagenzien gut funktionieren. Ebenso sollte die negative Kontrolle (kein Template)-Reaktion enthalten keine Banden für jedes Primer-set. Dadurch wird bestätigt, dass keines der Reagenzien wurden kontaminiert.
Wenn die DNA-Probe von Wildtyp-cogongrass, eine PCR-Reaktion mit dem cogongrass-spezifische Primer in einer einzelnen Bande bei ~ 595 bp führen, während die JBG-spezifischen Primern in keiner Band führen abgeleitet. Ebenso, wenn die DNA-Probe von JBG abgeleitet ist oder zurückgesetzt JBG, ein PCR-Reaktion unter Verwendung der JBG-spezifische Primer in einer einzelnen Bande bei ~ 594 bp führen, während die cogongrass-spezifischen Primern in keiner Band führt. Weil JBG und kehrte JBG identisch Nukleinsäuresequenz haben, werden sie daher über identische Bandenmuster. Wenn viele Proben auf einem Gel zur gleichen Zeit verglichen werden sollen, empfehlen wir, alle Proben von jedem Primer gesetzt nebeneinander abgeleitet, wodurch es einfacher, die Proben für die positiven Ergebnisse zu scannen.
Morphologischen Unterschiede zwischen JBG JBG und kehrt ziemlich offensichtlich sind (z. B. rote Farbe der Blätter und kleinere Statur von JB G gegen den Rest der Grünfärbung, größerer Statur und aggressive Wachstum der JBG zurück), so dass während der PCR-Ergebnisse die gleichen sein werden, sind die JBG Sorten leicht zu unterscheiden Verwendung Pflanzenmorphologie.

Abbildung 1. Vergleich von Gewächshaus gezogen Imperata cylindrica var. Koenigii (Japanese Blut Gras), kehrte I. cylindrica var. koenigii (JBG Revert) und I. cylindrica (Wildtyp cogongrass).
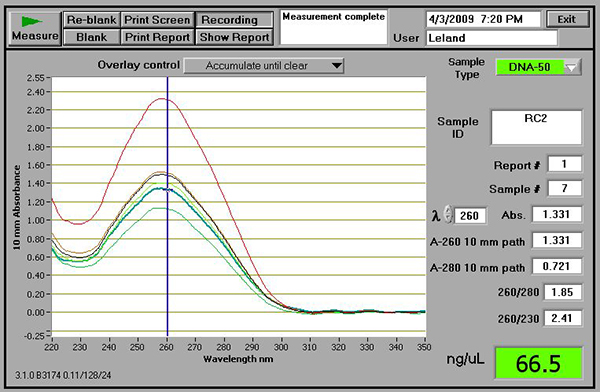
2. Ein Beispiel von DNA-Proben überprüft unter Verwendung eines Spektrophotometers NanoDrop. Beachten Sie, dass, unabhängig von der verwendeten Spektrophotometers, das 260/280 Verhältnis sollte in der Nähe 1,8 sein und die 260/230 Ratio soll bei 2,0 zu schließen.
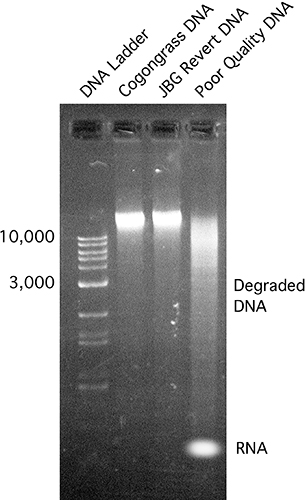
3. DNA-Proben mit Standardmethoden Gel-Elektrophorese auf einem 1% igen Agarosegel. Eine kommerzielle DNA-Marker wurde für Größe Analyse verwendet. Lane # 4 ist ein Beispiel von schlechter Qualität DNA-Probe, zeigt, Verschmieren und einige RNA-Kontamination.
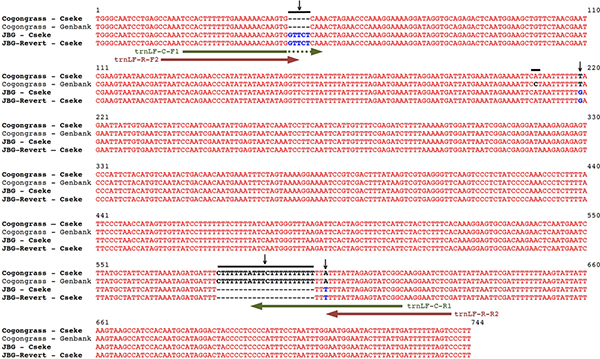
Abbildung 4. Sequenzvergleiche der trnL-F Regionen Imperata cylindrica var. Koenigii (Japanese Blut Gras), kehrte I. cylindrica var. koenigii (JBG Revert) und I. cylindrica (Wildtyp cogongrass). Vertikale schwarze Pfeile zeigen Unterschiede in den Sequenzen, die sich aus SNPs und indels. Horizontal grünen Pfeile zeigen die Positionen der Wildtyp-Primer für cogongrass cogongrass-spezifischen PCR verwendet. Horizontale roten Pfeile zeigen die potungen der JBG und JBG Revert Primer für JBG-spezifische PCR eingesetzt.
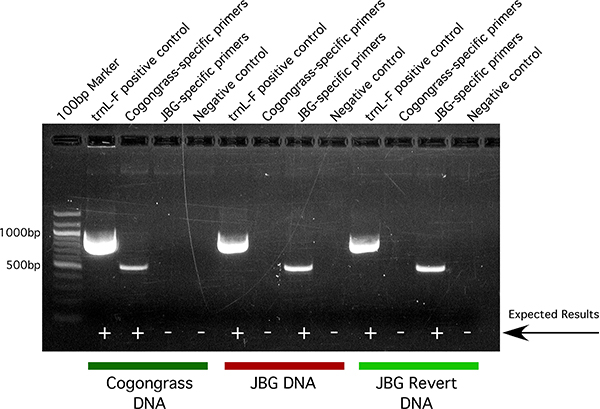
Bild 5. Repräsentative durch Gelelektrophorese von PCR-Produkten aus cogongrass abgeleitet, und JBG JBG wieder DNA-Proben mit cogongrass und JBG-spezifischen Primern sowie die trnL-F positive Kontrolle und eine negative Kontrolle ohne Matrize kombiniert.
Diskussion
Die US-Gärtnerei und Landschaftspflege Industrien leben vom Anbau und Verkauf exotischer und neuartige Pflanzenarten. Diese, mit der zunehmenden Globalisierung des Handels verbunden ist, erhöht die Chancen, dass eine invasive Pflanzenarten geben, zu etablieren, wird verteilt und in den USA die Fähigkeit, staatlich zu regulieren, wie Pflanzen beruht auf Informationen, die oft nicht zur Verfügung steht, einschließlich der Möglichkeit, sich invasiven , korrekte Taxonomie und genetische Verschiedenheit von einheimischen und eingebürgerten Taxa. Da unser Wissen über invasive Pflanzen ist oft begrenzt, haben importierten Pflanzen mit versteckten invasiven Eigenschaften wurden bereitwillig eingeführt, nur um später zu erfahren, dass sie unsere Landwirtschaft und natürliche Ressourcen zu erobern. Dieses Protokoll zielt darauf ab, solche Probleme mit I. verbunden sind, anzugehen cylindrica Sorten durch die Bereitstellung der ersten molekularen vereinfachte Methode, die genau zwischen den Wildtyp-cogongrass und kehrte Form seiner ornamentalen JBG Gegenstück unterscheiden kann.
jove_content "> Für die Entwicklung dieses Protokolls, wurde Wildtyp-cogongrass aus eingebürgerte Populationen an der Pond Creek Forstwirtschaft Einheit in Santa Rosa County in der Nähe von Jay, FL im Juni 2008 gesammelt werden. JBG wurde von einem kommerziellen Kindergarten (Bluebird Nursery Inc beschafft .) im Juni 2008 sowie vom Hausbesitzer die Sammlung in Columbia, kehrt MO JBG wurden aus dem Hof des Campbell Geologisches Museum an der Clemson University, SC erhalten im Juni 2008;. aus University Park in Riverdale, MA im Juni 2009; und aus dem Vorgarten eines Hausbesitzer in Columbia, MO im Jahr 2009 (identifiziert durch Leland Cseke). Alle Pflanzen wurden in einem Gewächshaus an der Universität von Alabama in Huntsville (Huntsville, AL) entfernt gehalten.Genetische Sequenzierung von DNA aus diesen Pflanzen gesammelten enthalten die eingehenden Vergleich von 9 unabhängigen DNA-Regionen allgemein Barcode Pflanzen 2 verwendet. In allen Fällen waren die Sequenzen von JBG eine 100% ige Übereinstimmung mit denen der JBG zurück, was zurüberprüfen, ob JBG in der Tat zu einem grünen, invasive Form zurückkehren. Nur der nuklearen ITS und die Chloroplasten trnL-F Regionen haben Differenzen, die verwendet werden, um genetisch zwischen cogongrass und JBG unterschieden werden kann. Die ITS-Region hat eine Gesamtfläche von 3 SNPs (single nucleotide polymorphisms) zwischen cogongrass und JBG, während die trnL-F-Region verfügt über 2 SNPs und 2 indels (Insertionen und Deletionen). Diese genetischen Unterschiede erlaubt Vielfalt-spezifische PCR-Primer zu entwickeln, welches zwischen Wildtyp-und cogongrass JBG kehrt zu unterscheiden. Die zuverlässigsten Ergebnisse wurden von Primern aus dem Plastid trnL-F abgeleitet Region kommen. Somit ist dieses Protokoll auf der Sequenzunterschiede zwischen den trnL-F Regionen der Chloroplasten-Genom cogongrass, JBG und kehrte JBG (Abbildung 4) basiert.
Damit erzeugen Primer, die eher typisch für die betreffenden Sorten sind und zur Vermeidung von Fehlalarmen aus nahe verwandten Arten, all bekannt trnL-F-Sequenzen von I. cylindrica Sorten wurden mit trnL-F-Sequenzen aus verwandten Grasart (43 unabhängige Sequenzen aus 29 Arten, z. B. Cymbopogon citratus, Sorghastrum incompletum, Coix lacryma-Jobi, Miscanthus sinensis, Saccharum officinarum, Sorghum halepense) verglichen. Obwohl, wir Vielfalt-spezifischen Primer-Sequenzen untersucht, über 29 Arten von Gras, die Spezifität der Vielfalt-spezifische Primer wurde noch nicht umfassend für seine Fähigkeit, DNA aus den meisten anderen Grasarten verstärken untersucht. Folglich sollten Gewebe für eine DNA-Extraktion verwendet sorgfältig wie I. identifiziert werden cylindrica vor Beginn dieses Protokoll. Wenn das Gras nicht als entweder cogongrass oder JBG identifiziert werden, dann empfehlen wir die Sequenzierung des PCR-Produkt, um sicherzustellen, dass Sequenzen, um eine exakte Übereinstimmung cogongrass oder JBG sind. Derzeit ist die genaueste Methode, um die Identität einer bestimmten Probe zu überprüfen Gras PCR bei den T-durchführenRNL-F und seine Regionen, gefolgt von der Sequenz Überprüfung der PCR-Produkte und der Vergleich der Sequenzen zu bekannten Sequenzen von genau identifiziert Taxa. DNA kann verstärkt mit Kontroll-Primer in diesem Protokoll aufgeführt (für die Region trnL-F) oder anderen Primern, dass in anderen Publikationen 13-15 werden. Die Sequenzierung ist viel mehr arbeits-und kostenintensiv als der Einsatz unserer vereinfachten Verfahren.
Die Qualität der Primer für die PCR verwendet wird, ist entscheidend für den Erfolg des Verfahrens. Wir haben die Primer für dieses Verfahren erhältlich von Bio Schrägansicht, Inc. (hergestellt http://www.obliquebio.com/web/ , Huntsville, AL). Der Vorteil für die Bestellung der Primer von Oblique Bio ist, dass sie eine große Anzahl von Aliquots von jedem Primer aus der identischen Probe-Baureihe als die Primer, die wir für die Optimierung in unserem Labor verwendet zu speichern. Folglich Primer nicht nur die gleiche Sequenz, aber they aus exakt den gleichen Produktionscharge, die in diesem Protokoll verwendet wurde, kommen. Unter Verwendung der Primer aus derselben Charge, können zur Vermeidung von externen Variablen in der Prozedur, die von Unterschieden in der Qualität der PCR-Primer zur Folge haben kann. Ebenso wird während andere Taq-Polymerasen sollte aber für PCR, die Qualität der Taq-Polymerase verwendet feine Auswirkungen auf die Qualität der PCR-Ergebnisse. Um eine bessere Konsistenz in PCR-Reagenzien ermöglichen, haben wir das Protokoll unter Verwendung von Reagenzien von Clontech optimiert. Der Vorteil 2 Polymerase (Clontech, Mountain View, CA, Kat. Nr. 639201 oder 639202) ist eine Mischung aus einem robusten, Hot-Start Taq-Polymerase und ein Lektorat Enzym, das eine hohe Spezifität und genauere Verstärkungen sorgen hilft.
Da dieses Protokoll beruht auf Chloroplasten-DNA, die das Muttertier in Gräsern vererbt wird, kann die Hybridisierung zwischen Ereignissen und cogongrass JBG Genotypen nicht mit unseren molekulare Identifizierung Verfahren erfasst werden. In Fällen, in denen hypridization ist & Fahrwerkcted, empfehlen wir die Verwendung nuklearer Regionen, die von beiden Eltern vererbt werden. Die am häufigsten verwendeten nicht-variable Region Plastiden in Pflanzenzellen Genotypisierung Aspekt ist die nukleare ribosomalen ITS-Region 13-15,17. Derzeit machen wir Fortschritte in Richtung Multiplexen der Verstärkung des Chloroplasten trnL-F-Region mit der des nuklearen ITS-Region in der gleichen PCR-Röhrchen. Multiplexen mit Kern-DNA-Regionen Plastiden würde möglicherweise eine Umgehung der Einschränkungen bei der Verwendung entweder allein, allerdings verlangen, dass diese Methoden Optimierung und zusätzliche Auswertung zur Machbarkeit auf einer von Fall zu Fall zu bestimmen. Der Einsatz von quantitativen real-time PCR (qPCR) und neuere Technologien, wie z. B. Molecular Beacons (fluoreszierenden Primer-Sonden), werden auch als Fail-Safe-Anlage und genaue Genotypisierung Methoden ausgewertet.
Die hier vorgestellte Protokoll bietet eine schnelle und zuverlässige Methode zur Unterscheidung der JBG von dem der Wildtyp-cogongrass zurück. Wir ermutigen Siers dieses Protokoll, um Kontakt mit uns auf, um Ergebnisse aus der Verwendung von diesem Protokoll ergeben, zu berichten. Solche gemeinsamen Information wird dazu beitragen, Informationen über die Verteilung der JBG zurückkehrt. Dies wird auch dazu beitragen Regulierungsbehörden auf der USDA fundierte Entscheidungen über Maßnahmen, die werden müssen, kann die Ausbreitung und mögliche Hybridisierung der sehr invasiv cogongrass Sorten zu umgehen.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Wir danken Alan Tasker (USDA-APHIS, Riverdale, MD), Stephen Compton (Clemson), Sherry Aultman (Clemson), Craig Ramsey (USDA-APHIS, Fort Collins, CO), und Betty Marose (UMD) für die Hilfe bei der Beschaffung Proben . Wir bedanken uns bei Studenten Andrew Adrian (UA-Huntsville) und Derek Thacker (UA-Huntsville) für ihre Unterstützung bei der Erprobung dieses Protokoll, und Joseph Herdy für seine Arbeit in den Dreharbeiten zu dem Video. Diese Arbeit wurde vom National Fish and Wildlife Foundation finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| DNeasy Plant Mini Kit | Qiagen, Valencia, CA | 69104 oder 69106 | Jeder seriöse genomischen Pflanzen-DNA Kit, das gute Qualität liefert DNA funktionieren sollte für diese Verfahren in Ordnung. |
| trnF (GAA)-F | Oblique Bio, Inc. | 3-0578 | Vorwärts-Primer positive Kontrolle |
| trnL (5'-Exon)-C | Oblique Bio, Inc. | 3-0579 | Reverse-Primer positive Kontrolle |
| trnLF-C-F1 | Oblique Bio, Inc. | 3-0864 | Vorwärts-Wildtyp-Primer cogongrass |
| trnLF-C-R1 | Oblique Bio, Inc. | 3-0865 | Rückgängig machenWildtyp-Primer cogongrass |
| trnLF-R-F2 | Oblique Bio, Inc. | 3-0866 | Vorwärts und JBG JBG zurückkehren Primer |
| trnLF-R-R2 | Oblique Bio, Inc. | 3-0867 | Reverse JBG JBG und zurück Primer |
| Vorteil UltraPure PCR-dNTP-Mix | Clontech, Mountain View, CA | 639125 | |
| Vorteil 2 Polymerase | Clontech, Mountain View, CA | 639201 oder 639202 | Ein gutes Lektorat, Hot-Start Taq-Polymerase |
Referenzen
- Holm, L. G., Pancho, J. V., Herberger, J. P., Plucknett, D. L. A Geographical Atlas of World Weeds. , Krieger Publishing Company. Malabar, Florida, U.S. (1979).
- CABI, Crop Protection Compendium. Commonwealth Agricultural Bureau International (CABI). , Wallingford, UK. (2007).
- MacDonald, G. E. Cogongrass (Imperata cylindrica) - Biology, ecology, and management. Critical Reviews in Plant Sciences. 23, 367-380 (2004).
- Talley, S. M., Cseke, L. J. Molecular diagnostic technologies for invasive plants. CPHST Fort Collins Laboratory 2009 Annual Report. Zink, R. , USDA-APHIS-PPQ-CPHST. Fort Collins, Colorado. 27-28 (2009).
- Dozier, H., Gaffney, J. F., McDonald, S. -K., Johnson, E. R. R. L., Shilling, D. G. Cogongrass in the United States: History, ecology, impacts, and management. Weed Technology. 12, 737-743 (1998).
- Chikoye, D., Ekeleme, F. Weed flora and soil seedbanks in fields dominated by Imperata cylindrica in the moist savannah of West Africa. Weed Research. 41, 475-490 (2001).
- Weber, E. Invasive Plant Species of the World: A Reference Guide to Environmental Weeds. , CABI Publishing. Wallingford, UK. (2003).
- Patterson, D. T. Shading effects on growth and partitioning of plant biomass in Cogongrass (Imperata cylindrica) from shaded and exposed habitats. Weed Science. 28, 735-740 (1980).
- Cole, J. T., Cole, J. C. Ornamental grass growth response to three shade intensities. Journal of Environmental Horticulture. 18, 18-22 (2000).
- Bryson, C. T., Koger, C. H., Byrd, J. D. Effects of temperature and exposure period to heat on cogongrass (Imperata cylindrica) viability. Weed Technology. 21, 141-144 (2007).
- Capo-chichi, L. J. A., Faircloth, W. H., Williamson, A. G., Patterson, M. G., Miller, J. H., van Santen, E. Invasion Dynamics and Genotypic Diversity of Cogongrass (Imperata cylindrica) at the Point of Introduction in the Southeastern United States. Invasive Plant Science and Management. 1 (2), 133-141 (2008).
- Talley, S. M., Ramsey, C. L. Experimentally assessing the invasive potential of plants. CPHST Laboratory 2009 Annual Report. Zink, R. , USDA-APHIS-PPQ-CPHST. Fort Collins, Colorado. 29-30 (2009).
- Hodkinson, T. R., Chase, M. W., Lledó, M. D., Salamin, N., Renvoize, S. A. Phylogenetics of Miscanthus, Saccharum and related genera (Saccharinae, Andropogoneae, Poaceae) based on DNA sequences from ITS nuclear ribosomal DNA and plastid trnLintron and trnL-F intergenic spacers. J. Plant Res. 115, 381-392 (2002).
- Kress, W. J., Wurdack, K. J., Zimmer, E. A., Weigt, L. A., Janzen, D. H. Use of DNA barcodes to identify flowering plants. Proc. Natl. Acad. Sci. U.S.A. 102 (23), 8369-8374 (2005).
- Roodt-Wilding, R., Spies, J. J. Phylogenetic relationships in southern African chloridoid grasses (Poaceae) based on nuclear and chloroplast sequence data. Systematics and Biodiversity. 4, 401-415 (2006).
- Brody, J. R., Kern, S. E. Sodium boric acid: a Tris-free, cooler conductive medium for DNA electrophoresis. BioTechniques. 36, 214-216 (2004).
- Chou, C. -H., Tsai, C. C. Genetic variation in the intergenic spacer of ribosomal DNA of Imperata cylindrica (L.) Beauv. var. major (Cogongrass) populations in Taiwan. Botanical Bulletin of Academia Sinica (Taipei). 40, 319-332 (1999).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten