Method Article
Une méthode de génotypage basé sur la PCR pour distinguer les variétés de type sauvage et les plantes ornementales de Imperata cylindrica
Dans cet article
Résumé
Nous fournissons un protocole de génotypage coût-efficace et rapide moléculaire qui emploie diverses amorces spécifiques de PCR que les différences cibles séquence d'ADN au sein de la région de l'espaceur chloroplastique trnL-F à la différence entre les variétés de Imperata cylindrica (Imperata cylindrique) qui ne peuvent pas être distingués par la seule morphologie. Ces variétés sont la mauvaise herbe nuisible sur la liste fédérale, imperata cylindrique et étroitement liée, répandue variété ornementale, je. cylindrica Var. Koenigii (Herbe de sang japonais).
Résumé
Wild-type I. cylindrica (imperata cylindrique) est l'un des dix meilleurs pires plantes envahissantes dans le monde, un impact négatif sur les ressources agricoles et naturelles dans 73 pays différents à travers l'Afrique, en Asie, en Europe, en Nouvelle-Zélande, Océanie et les 1-2 Amériques. Imperata cylindrique se forme rapidement-étalement, stands monodominantes qui déplacent une grande variété d'espèces végétales indigènes et à leur tour menacer les animaux indigènes qui dépendent des espèces de plantes indigènes déplacées pour le fourrage et un abri. Pour ajouter au problème, une variété ornementale [I. cylindrica var. koenigii (Retzius)] est largement commercialisé sous les noms de 'Rubra' Imperata cylindrica, Red Baron, et de l'herbe de sang japonais (JBG). Cette variété est présumément stérile et non invasive et est considéré comme une plante ornementale souhaitable pour ses feuilles de couleur rouge. Toutefois, dans des conditions correctes, JBG peut produire des graines viables (Carol Holko, 2009 communication personnelle) et peut revenir à un vert iforme nvasive qui est souvent impossible de distinguer imperata cylindrique comme il faut sur les caractéristiques distinctives de la variété de type sauvage invasive 4 (figure 1). Cela rend l'identification à l'aide la morphologie d'une tâche difficile, même pour les taxonomistes de plantes bien formés. La réversion de la JBG à un phénotype agressif vert n'est pas non plus un phénomène rare. En utilisant des comparaisons de séquences de codage et de régions variables de l'ADN nucléaire et chloroplastique, nous avons confirmé que JBG est revenu à la invasif verte au sein des Etats du Maryland, la Caroline du Sud, et le Missouri. JBG a été vendu et planté dans un état à peu près tous sur le continent américain où il n'ya pas une infestation active imperata cylindrique. La mesure du problème reviendra dans pas bien compris parce que les plantes redevenus sont sans papiers et souvent détruits.
L'application de ce protocole fournit une méthode moléculaire pour identifier les JBG revient et peut aider à garder ces variétés de co-produisent unee peut s'hybrider. Imperata cylindrique est une allofécondation obligatoire et, lorsqu'on les croise avec un génotype différent, peut produire dispersées par le vent viable semences qui se propagent sur de grandes distances imperata cylindrique 5-7. JBG a un génotype légèrement différente de celle imperata cylindrique et peut être en mesure de former des hybrides viables avec imperata cylindrique. Pour ajouter au problème, JBG est plus froid et tolérante à l'ombre de imperata cylindrique 8-10, et le flux génétique entre ces deux variétés est susceptible de générer des hybrides qui sont plus agressifs, tolérante à l'ombre, et résistant au froid que de type sauvage imperata cylindrique. Bien que de type sauvage imperata cylindrique infeste actuellement plus de 490 millions d'hectares dans le monde, aux États-Unis du Sud-Est il infeste plus de 500.000 hectares et est capable d'occuper la plupart des Etats-Unis car il se propage rapidement vers le nord en raison de sa niche large et géographique 3,7,11 potentiel. Le potentiel d'un croisement génétique est une grave préoccupation pour le Programme de l'USDA-APHIS fédérale Semaine nocives. Actuellement, l'USDA-APHIS interdit JBG dans les Etats whavant il ya les grands infestations imperata cylindrique (par exemple, en Floride, Alabama, Mississippi). Toutefois, la prévention des deux variétés de la combinaison peut s'avérer plus difficile que imperata cylindrique et JBG élargir leurs distributions. En outre, la répartition de la JBG revenir est actuellement inconnu et sans la capacité d'identifier ces variétés par la morphologie, certains infestations imperata cylindrique peut être le résultat de JBG revient. Malheureusement, les méthodes actuelles d'identification moléculaire s'appuient généralement sur AFLP (amplified fragment length polymorphisms) et séquençage de l'ADN, qui sont tous deux longues et coûteuses. Ici, nous présentons le premier coût-efficace et fiable basée sur la PCR méthode génotypage moléculaire de distinguer avec précision entre les imperata cylindrique et JBG revenir.
Protocole
1. Prélèvement et conservation
Cette méthode a été développée et testée en utilisant frais, congelés, et les tissus foliaires récemment séché.
- Identifier imperata cylindrique et / ou JBG tissus à l'aide d'un taxonomiste qui se spécialise dans l'herbe l'identification des espèces. Avec ses feuilles rouges lumineuses, JBG ornementale est facile de distinguer visuellement de type sauvage et imperata cylindrique JBG revenir, mais imperata cylindrique et JBG revenir sont presque impossibles à distinguer les uns des autres. Imperata cylindrique et le phénotype JBG revenue avoir verts, les feuilles plus longues, les rhizomes nettement plus étendues et plus, et la région plus de feuilles que JBG 12 (Figure 1).
- Tissus de la feuille fraîche fournit l'ADN de la qualité la plus abondante et la plus élevée et peut être recueillie à partir du champ ou en serre I. plantes cylindrica. Si les tissus frais doit être utilisé, extraire de l'ADN dans les 3 heures suivant le prélèvement pour aider à prévenir la dégradation. Sinon, préparez les tissus pour le stockage, keeping le tissu frais et à la lumière du soleil directe.
- Pour stocker les tissus pour l'extraction d'ADN à une date ultérieure, la méthode la plus optimale consiste à congeler et conserver le tissu à -80 ° C immédiatement. Ne laissez pas le tissu congelé pour dégeler avant extraction de l'ADN. Transférer le tissu congelé dans l'azote liquide avant les étapes de broyage de l'extraction d'ADN pour empêcher la décongélation.
- Si un congélateur à -80 ° C n'est pas disponible, le tissu sèche immédiatement. Pour le stockage à sec, placez le tissu dans une enveloppe en papier et de stocker l'enveloppe dans du gel de silice déshydraté ou d'autres déshydratants actifs à la température ambiante. Une petite quantité de silice indicateur mélangé avec de la silice non-indiquant veillera à ce que la silice est entièrement déshydraté et approprié pour le séchage des tissus végétaux.
- Utilisez au moins 10 fois gel de plus de silice que les tissus de feuilles fraîches en poids. Les tissus végétaux doivent sécher dans les 24 heures. La qualité et la quantité d'ADN est réduite par le stockage à sec au cours du temps (comme dans le cas de spécimen d'herbiers).
2. Extraction de l'ADN
Pour extraire l'ADN d'un tissu végétal, suivre le DNeasy usine Mini Kit (Qiagen, Valencia, CA; Cat # 69104 ou 69106) les instructions du fabricant avec une modification mineure. Au lieu d'utiliser l'a suggéré à moins de 100 mg de tissu frais ou inférieure à 20 mg de tissu sec pour chaque colonne, meuler supérieure à 100 mg, puis de transférer 100 mg de tissu frais ou congelé (ou> 20 mg de tissu sec) dans des tubes appropriés pour l'extraction. L'ADN nucléaire et plaste est extrait simultanément.
- Avant de commencer ces procédures, vérifiez que l'éthanol a été ajouté à tampons AP3 / E et AW.
- Broyer> 100 mg de tissu de feuilles fraîches ou congelées (ou> 20 mg de tissus de la feuille sèche) en une fine poudre à l'aide de trois séries de l'azote liquide avec un broyage dans un mortier et un pilon réfrigérés. Perturbations insuffisante du matériau de départ ou de lyse insuffisante peut également conduire à une baisse des rendements de l'ADN. Soigneusement moudre le tissue et ne surchargez pas les colonnes avec trop de tissu.
- Transférer 100 mg, poudre à partir de tissus congelés frais ou congelé (ou 20 mg de poudre à partir de tissu sec) à un microtube de 1,5 ml contenant 400 ul de tampon AP1 et 4 pl de RNase A. Chaque tube peut être placé dans un rack sur une petite équilibrer pour surveiller le poids exact du tissu par tube.
- Échantillon Vortex ou un bougé de (s) pour mélanger et incuber pendant 10 min à 65 ° C, en inversant le tube (s) 2-3 fois pendant l'incubation.
- Ajouter 130 pi de tampon AP2 à chaque échantillon. Mélanger en inversant le tube (s) à plusieurs reprises, et incuber pendant 5 min sur la glace.
- Pipeter chaque lysat dans une colonne spin QIAshredder Mini séparée, et placer chaque colonne dans un tube de 2 ml de collecte (fourni avec le kit). Centrifugeuse colonne (s) pendant 2 min à 20.000 xg (~ 14000 rpm), et transférer chaque accréditives fraction dans un nouveau tube (non fourni avec le kit) sans perturber formé à granulés.
- Ajouter 1,5 volumes de tampon AP3 /E, et mélanger à la pipette.
- Transfert 650 ul du mélange dans une colonne spin Mini DNeasy dans un tube de 2 ml de collecte. Centrifugeuse colonne (s) pendant 1 min à 6000 xg (~ 8000 rpm), et jeter le accréditives. Répétez cette étape avec le reste du mélange pour chaque échantillon.
- Placer la colonne spin (s) dans un nouveau tube collecteur de 2 ml (s), et ajouter 500 ul de tampon AW vers le haut de chaque colonne. Centrifugeuse colonne (s) pendant 1 min à 6000 xg (~ 8000 rpm), et jetez-débit par le biais.
- Ajouter une autre de 500 ul de tampon AW vers le haut de chaque colonne. Centrifuger pendant 2 min à 20.000 xg (~ 14000 rpm). Cette étape va sécher la colonne, éliminant ainsi toute l'éthanol résiduelle contenue dans les tampons qui peuvent inhiber la PCR.
- Transférer chaque colonne spin à un nouveau tube de 1,5 ml d'échantillon microcentrifugeuse. Ajouter 100 pi de tampon AE vers le haut de chaque colonne pour l'élution, et incuber la colonne (s) pendant 5 min à température ambiante. Centrifugeuse colonne (s) pendant 1 min à 6000 xg (~ 8,000 rpm) pour recueillir l'ADN.
- Répétez ces étapes d'élution un moment donné, éluer l'ADN dans le même tube de 1,5 ml de microcentrifugation pour donner 200 pi d'échantillon. Des échantillons d'ADN doivent être stockées à -20 ° C jusqu'à son utilisation. Concentrations d'ADN dépendent du type de tissu et les conditions de stockage. Rendements optimaux sont obtenus lorsque l'ADN élution avec un total de 200 pi de tampon AE; cependant, les concentrations peuvent être augmentées si les volumes d'élution sont réduits à aussi peu que 50 pl.
3. Vérification de la qualité de l'ADN et la quantité
- Testez la qualité et la quantité d'ADN extrait avant l'installation PCR en utilisant un spectrophotomètre ou fluorimètre et électrophorèse sur gel. Cela aidera à assurer le succès des étapes ultérieures.
- L'utilisation d'un spectrophotomètre, la qualité et la quantité d'ADN d'essai. Rendements d'ADN bonnes devrait se situer entre 50 et 150 ng / ul avec 260/280 et 230/280 ratios proches de 2.0. A titre d'exemple, la figure 2 montre les résultats de bonne qualité en utilisant le spectrophotomètre NanoDrop (ThermoScientific, Wilmington, DE).
- Effectuer l'électrophorèse sur gel en utilisant un standard d'agarose 1%. Vérifiez la présence de bandes relativement importantes (> 10 Ko) sans stries à peu de la contamination d'ARN (Figure 3).
- Si la concentration en ADN est élevé, des échantillons d'ADN dilués à 70 ng / ul pour les étapes ultérieures.
4. Les amorces de PCR
Les amorces de PCR utilisées dans ce protocole sont basées sur des différences de séquences entre la région espaceur trnL-F plaste de génotypes imperata cylindrique et JBG. Ces différences se présentent sous forme de SNP (Single Nucleotide Polymorphism) et les indels (insertions et suppressions) qui ont permis le développement de la variété des amorces spécifiques en localisant les amorces sur les sites de séquences uniques (Figure 4).
- La qualité des amorces peuvent avoir un impact significatif sur les résultats de la PCR. Amorces de commande à partir d'une entreprise digne de confiance. Nous commandons nos amorces de Oblique Bio, Inc (:/ / Www.obliquebio.com/web/ "target =" _blank "> http://www.obliquebio.com/web/, Huntsville, AL), demandant que les procédures standard de dessalement.
- trnL-F amorces de contrôle positifs: Ce jeu d'amorces amplifie la région espaceur plaste trnL-F de la plupart des taxons herbe 11-13 et sert un bon contrôle positif (résultant dans une bande de 890 pb).
Nom Séquence trnF (GAA)-F 5'-ATTTGAACTGGTGACACGAG-3 ' trnL (exon 5 ')-C 5'-CGAAATCGGTAGACGCTACG-3 ' - De type sauvage amorces imperata cylindrique: Ce jeu d'amorces est spécifique à imperata cylindrique et ne pas amplifier la région trnL-F de JBG génotypes. Les résultats présentés dans un groupe qui est de 595 pb.
Nom Sequence trnLF-C-F1 5'-TCCACTTTTTTGAAAAAACAAGTGCAA-3 ' trnLF-C-R1 5'-GCCGATACTCTAATAAATAAAAAAAAAAAAGAAAT-3 ' - JBG JBG et Revert amorces: Ce jeu d'amorces est spécifique à JBG génotype et n'amplifie pas la région trnL-F de imperata cylindrique. Les résultats présentés dans un groupe qui est de 594 pb.
Nom Séquence trnLF-R-F2 5'-CCAAATCCACTTTTTTGAAAAAACAAGTGGTT-3 ' trnLF-R-R2 5'-CGAGATTCCTTGCCGATACTCTAATAAAA-3 ' - Remettre en suspension chaque amorce dans un volume suffisant de nucléase-free O ddH 2 pour obtenir un 100 mm Stock solutions qui peuvent être stockés à -20 ° C, à long terme.
- Diluer chaque stock d'amorce à 12 mm avant les étapes de configuration de PCR.
5. PCR Setup
Extraction d'ADN sont amplifiés en utilisant chacun des ensembles d'amorces susmentionnées dans des réactions de PCR. Inclure un contrôle positif afin de s'assurer que tous les réactifs de PCR sont bien fonctionner et peut générer une bande. Inclure un contrôle négatif pour s'assurer qu'aucun des réactifs sont contaminés avec de l'ADN non désirées. Le contrôle négatif ne contient pas de modèle-et devrait aboutir à aucune production de bande.
- Préparez toutes les réactions dans des tubes PCR à paroi mince pour permettre un meilleur transfert de chaleur entre le bloc thermocycleur et l'échantillon. Nous vous recommandons d'utiliser des aérosols disponibles dans le commerce sans embouts de pipette pour éviter la contamination.
- Pour chaque échantillon d'ADN isolé, mis en place 50 ul réactions de PCR utilisant chacun des ensembles d'amorces ci-dessus dans 0,2 ml tubes PCR à paroi mince en ajoutant les réactifs suivants sur la glace dans l'ordre indiqué ci-dessous. Si plusieurs échantillons sont en cours de préparation, faire un cocktail contenant tous les réactifs à l'exceptionde la matrice d'ADN pour établir des conditions uniformes entre toutes les réactions.
Réactif PCR Volume d'occasion Concetration final Nucléase-free ddH 2 O 40,5 pi 10X Advantage 2 PCR Buffer (Clonetech, CA) 5,0 pl 10% (v / v) Avantage UltraPure PCR dNTP Mix (10 mM chacun, Clonetech, CA) 1,0 pl 0,2 mM Primer 1 (12μM dans le trou DDH 2 O) 1,0 pl 0,24 uM Primer 2 (12μM dans le trou DDH 2 O) 1,0 pl 0,24 uM Mix Polymerase Avantage 2 (Clonetech, CA) 0,5 pl 1% (v / v) Extraction de l'ADN(70 ng / réaction; ajuster ddH volume 2 O en fonction des besoins) 1,0 pl 1,4 ng / ul Total: 50,0 pi - Afin de s'assurer que tous les réactifs de PCR sont bien fonctionner, configurer le contrôle positif à l'aide des amorces de contrôle positifs. Ce jeu d'amorces fonctionne aussi bien pour imperata cylindrique, JBG, JBG revenir et d'autres graminées et se traduira par un groupe qui est de 890 pb.
- Configurer le contrôle négatif en utilisant l'amorce même contrôle défini comme le contrôle positif, à l'aide ddH 2 O au lieu de l'extraction de l'ADN. Si tous les réactifs sont exempts de l'ADN contamine, cette réaction se traduira par aucune bande.
- Si la concentration en ADN est faible, plus l'ADN peuvent être ajoutés à chaque réaction, en ajustant la quantité de nucléase-free O ddH 2 d'occasion pour porter le volume total de réaction de 50 ul. Ne pas utiliser plus de 5 pi de l'ADN (10% des le volume total) par reaction, les impuretés que possible contenues dans les échantillons d'ADN peuvent inhiber les réactions PCR.
6. PCR à vélo
- Effectuer amplifications par PCR dans un thermocycleur équipé d'un couvercle chauffant à l'aide des paramètres suivants à vélo PCR. Nous utilisons le Mastercycle pro S thermocycleur (Eppendorf, Hauppauge, NY) réglé pour fonctionner avec des conditions standard de température de rampes. Toute thermocycleur qualité devraient bien performer.
Cycle Dénaturation Recuit Polymérisation 1 2 min à 95 ° C 2 30 sec à 95 ° C 30 sec à 61 ° C 90 sec à 68 ° C 35 cycles 3 5 min à 68 ° C Maintenir à 4 ° C jusqu'à ce que l'échantillon est retirée - Optimiser les conditions de PCR (y compris la température de recuit primaire, le temps de vulgarisation, et le nombre de cycles) selon les besoins en fonction de la qualité de l'ADN, les amorces, la Taq polymérase ou le type de thermocycleur utilisé. Nous vous recommandons d'utiliser un thermocycleur gradient capables lorsqu'il s'agit de déterminer l'optimal températures de recuit.
- Si le thermocycleur utilisé n'a pas de couvercle chauffant, ajouter une goutte d'huile minérale à la partie supérieure de chaque échantillon pour empêcher l'évaporation durant le cyclage de PCR.
7. Électrophorèse sur gel des produits de PCR
Pour visualiser les résultats de l'analyse, séparés des produits de PCR sur un gel d'agarose 1% en utilisant l'électrophorèse standard.
- Mélanger 2 ul d'un tampon de chargement standard de l'ADN (généralement une solution 5x ou 6x) avec 5 pi de chaque produit amplifié par PCR.
- Charger les échantillons sur un gel d'agarose 1% contenant EtBr (bromure d'éthidium pour coloration de l'ADN) fait avec esystèmes tampons ither TAE ou SB (borate de sodium) 16. Nous utilisons 1 pi d'une 10 mg / ml solution mère EtBr par 100 ml d'agarose 1% (0,1 pg / ml).
- Exécuter échantillons à ~ 120 jusqu'à ce que le front atteint ¾ de la longueur totale du gel.
- Sous une lumière ultraviolette (par exemple une boîte de courte longueur d'onde la lumière UV), d'inspecter les bandes résultant de voir si un fragment approprié a été amplifié.
- Documenter le gel et les bandes résultant en utilisant une disposition de photo-documentation du système ou l'appareil photo.
8. Les résultats représentatifs
Lors de la visualisation des produits de PCR, imperata cylindrique a un motif unique de bandes par rapport à celle de JBG ou rétablie JBG (figure 5). Pour chaque échantillon d'ADN, l'ensemble F-trnL positif amorce de contrôle devrait se traduire par un seul haut-intensité de la bande à ~ 890 pb. Cela permet de vérifier que tous les réactifs de PCR sont bien fonctionner. De même, le contrôle négatif (pas de modèle) la réaction ne doit pas contenir des bandes pour toute s amorceet utilisé. Cela permet de vérifier qu'aucun des réactifs ont été contaminés.
Si l'échantillon d'ADN est dérivé de type sauvage imperata cylindrique, une réaction de PCR en utilisant le jeu d'amorces imperata cylindrique spécifique se traduira par une seule bande à ~ 595 pb, tandis que les amorces spécifiques JBG se traduira par aucune bande. De même, si l'échantillon d'ADN est dérivée de JBG JBG ou rétabli, une réaction de PCR en utilisant le jeu d'amorces JBG-spécifique se traduira par une seule bande à ~ 594 pb, tandis que les amorces spécifiques imperata cylindrique se traduira par aucune bande. Parce que JBG JBG, et revint ont identiques séquence d'acide nucléique, ils ont donc des profils de bandes identiques. Si de nombreux échantillons doivent être comparés sur un gel dans le même temps, nous vous recommandons d'exécuter tous les échantillons provenant de chaque amorce situé à côté de l'autre, ce qui rend plus facile à balayer les échantillons pour obtenir des résultats positifs.
Les différences morphologiques entre les JBG JBG et revient sont assez évidents (par exemple la couleur rouge des feuilles et plus petite taille de JB G vs la coloration verte, plus grande stature et la croissance agressive de la JBG revenir), et bien que les résultats de PCR sera le même, les variétés JBG sont faciles à distinguer à l'aide morphologie de la plante.

Figure 1. Comparaison des culture en serre Imperata cylindrica var. Koenigii (herbe de sang japonais), robot I. cylindrica var. koenigii (JBG Revert) et I. cylindrica (Wild-type de imperata cylindrique).
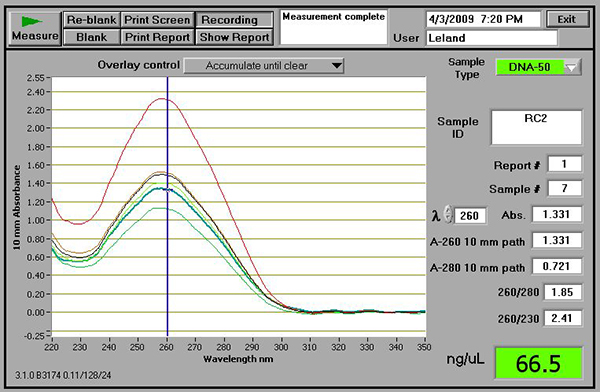
Figure 2. Un exemple d'échantillons d'ADN vérifiées à l'aide d'un spectrophotomètre NanoDrop. Noter que, indépendamment du spectrophotomètre utilisé, le rapport 260/280 doit être proche de 1,8 et le rapport 260/230 doit proche de 2,0.
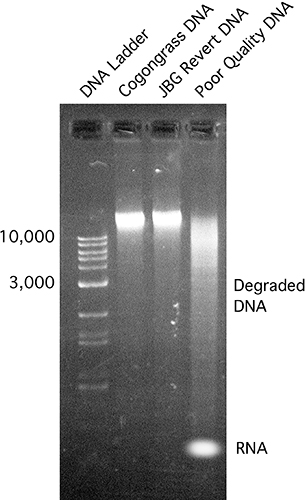
Des échantillons d'ADN Figure 3. Vérifiée par électrophorèse sur gel standard sur un gel d'agarose 1%. Un marqueur d'ADN commercial a été utilisé pour l'analyse granulométrique. Lane # 4 est un exemple de l'échantillon d'ADN de qualité médiocre, montrant bavures et une certaine contamination ARN.
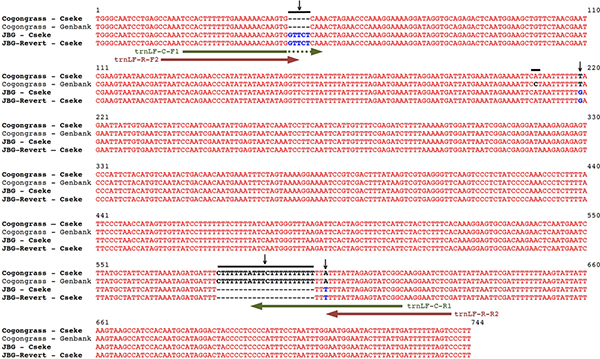
Alignements de séquences Figure 4. Des régions trnL-F de Imperata cylindrica var. Koenigii (herbe de sang japonais), robot I. cylindrica var. koenigii (JBG Revert) et I. cylindrica (Wild-type de imperata cylindrique). Vertical flèches noires indiquent des différences dans les séquences résultant de SNPs et indels. Horizontaux flèches vertes indiquent les positions des amorces imperata cylindrique de type sauvage utilisés pour imperata cylindrique-PCR spécifique. Horizontaux flèches rouges indiquent la positions de la JBG JBG et Revert amorces utilisées pour JBG-PCR spécifique.
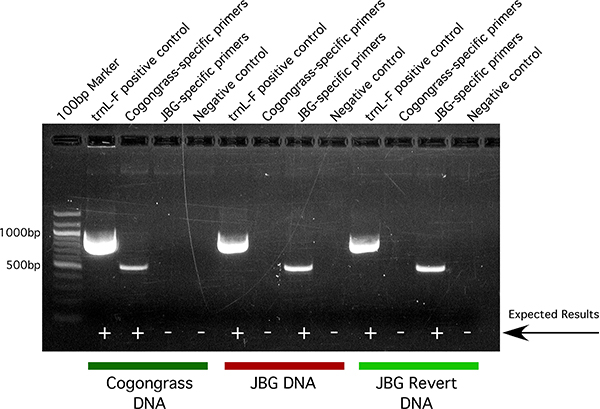
Figure 5. Résultat représentative de l'électrophorèse sur gel des produits de PCR dérivés de imperata cylindrique, et JBG JBG revenir échantillons d'ADN associés à des imperata cylindrique et JBG amorces spécifiques ainsi que l'trnL-F témoin positif et un contrôle de modèle pas négative.
Discussion
La pépinière des États-Unis et de l'aménagement paysager se développent sur la culture et la vente d'espèces de plantes exotiques et roman. Ceci, couplé avec la mondialisation croissante du commerce, augmente les chances que des espèces végétales envahissantes entreront, établir et diffuser aux Etats-Unis La capacité de réguler le gouvernement fédéral ces plantes repose sur l'information qui n'est souvent pas disponible, y compris le potentiel de devenir envahissantes , la taxonomie correcte, et la distinction génétique de taxons indigènes et naturalisées. Parce que notre connaissance des plantes envahissantes est souvent limitée, avec des plantes importées cachés caractéristiques envahissantes ont été introduites volontairement pour apprendre plus tard qu'ils envahissent nos ressources agricoles et naturelles. Ce protocole vise à résoudre ces problèmes liés à I. variétés cylindrica en fournissant la première méthode simplifiée moléculaire qui peut distinguer précisément de type sauvage imperata cylindrique et la forme est revenue de son homologue JBG ornementales.
jove_content "> Pour le développement de ce protocole, de type sauvage imperata cylindrique ont été recueillies auprès des populations naturalisées à l'unité de l'étang du ruisseau forestier à Santa Rosa County, près de Jay, Floride en Juin 2008. JBG a été obtenue à partir d'une pépinière commerciale (Bluebird Nursery, Inc ..) en Juin 2008 ainsi que de la collection d'un particulier de à Columbia, MO JBG revient ont été obtenus à partir de la cour de la Campbell Musée géologique à l'Université de Clemson, Caroline du Sud en Juin 2008; de l'Université Park à Riverdale, MA en Juin 2009, et de la cour avant d'un propriétaire à Columbia, MO en 2009 (identifié par Leland Cseke). Toutes les plantes ont été maintenus dans une serre située à l'Université d'Alabama à Huntsville (Huntsville, AL).Le séquençage génétique de l'ADN recueilli à partir de ces plantes incluses dans les comparaisons de la profondeur de 9 régions d'ADN indépendantes couramment utilisées pour 2 plantes de codes à barres. Dans tous les cas, les séquences de JBG étaient un bonus de 100% à ceux de la JBG revenir, contribuant ainsi àvérifier que JBG en effet revenir à un vert, forme invasive. Seule la SA nucléaire et les chloroplastes trnL-F régions présentent des différences qui peuvent être utilisés pour distinguer génétiquement imperata cylindrique et JBG. La région ITS a un total de 3 SNP (polymorphismes nucléotidiques simples) entre imperata cylindrique et JBG, tandis que la région trnL-F dispose de 2 SNP et 2 indels (insertions et des suppressions). Ces différences génétiques a permis diverses amorces spécifiques de PCR pour être mis au point qui permet de distinguer entre de type sauvage et imperata cylindrique JBG revient. Les résultats les plus fiables proviennent de amorces dérivées de la plaste trnL-F région. Ainsi, ce protocole est basé sur les différences de séquences entre les régions trnL-F du génome chloroplastique de imperata cylindrique, JBG JBG et revenues (figure 4).
Pour aider à générer des amorces qui sont plus spécifiques aux variétés en question et d'aider à éviter les faux positifs à partir d'espèces étroitement apparentées, all connus trnL-F séquences de I. cylindrica variétés ont été comparées avec trnL-F séquences provenant d'espèces de graminées connexes (43 séquences indépendantes de 29 espèces, par exemple, Cymbopogon citratus, Sorghastrum incompletum, les Coix lacryma-jobi, de Miscanthus sinensis, de Saccharum officinarum, Sorghum halepense). Bien, nous avons examiné les séquences des amorces spécifiques à travers divers-29 espèces d'herbe, la spécificité des amorces spécifiques variété n'a pas été complètement examinée pour sa capacité à amplifier l'ADN de la plupart des autres espèces de graminées. Par conséquent, le tissu utilisé pour l'extraction d'ADN doivent être soigneusement identifiés comme I. cylindrica avant de commencer ce protocole. Si l'herbe ne peut pas être identifié soit comme imperata cylindrique ou JBG, nous suggérons le séquençage du produit PCR afin de s'assurer que les séquences sont une correspondance exacte ou JBG imperata cylindrique. L'heure actuelle, la méthode la plus exacte pour vérifier l'identité d'un échantillon d'herbe donnée consiste à effectuer la PCR à la fois sur le trnl-F et de ses régions, suivie d'une vérification séquence de produits de PCR et la comparaison des séquences avec des séquences connues de taxons identifiés avec précision. L'ADN peut être amplifié en utilisant des amorces de contrôle décrites dans le présent protocole (pour la région trnL-F) ou des amorces d'autres disponibles dans d'autres publications 13-15. Le séquençage est beaucoup plus de travail et coûteux que d'utiliser notre procédure simplifiée.
La qualité des amorces utilisées pour la PCR est essentielle à la réussite de la procédure. Nous avons fait des amorces pour cette procédure disponible à partir Oblique Bio, Inc ( http://www.obliquebio.com/web/ , Huntsville, AL). L'avantage de commander les amorces de Bio Oblique, c'est qu'ils stocker un grand nombre de parties aliquotes de chaque amorce de la production en série d'échantillon identique à celle des amorces que nous avons utilisées pour l'optimisation dans notre laboratoire. Par conséquent, les amorces non seulement avoir la même séquence, mais they proviennent du même lot de production exacte qui a été utilisé dans ce protocole. En utilisant des amorces provenant du même lot, peut aider à éviter les variables parasites dans la procédure qui pourrait résulter de différences dans la qualité des amorces de PCR. De même, tandis que d'autres polymérases Taq devrait fonctionner correctement pour la PCR, la qualité de la Taq polymérase utilisée aura un impact sur la qualité des résultats de PCR. Afin de permettre une meilleure cohérence dans les réactifs de PCR, nous avons optimisé le protocole en utilisant des réactifs de Clontech. Le Avantage 2 polymérase (Clontech, Mountain View, CA, Cat # 639201 ou 639202) est un mélange d'un solide, démarrage à chaud Taq polymérase et une enzyme de relecture qui permet de fournir une haute spécificité et plus précises amplifications.
Parce que ce protocole repose sur l'ADN des chloroplastes, qui est hérité de la mère dans les herbes, les événements d'hybridation entre imperata cylindrique et les génotypes JBG ne peut pas être capturé avec notre procédure d'identification moléculaire. Dans les cas où hypridization est suspeDECT, nous recommandons d'utiliser les régions nucléaires qui sont héritées des deux parents. Le plus couramment utilisé non-plaste région variable à considérer dans le génotypage des plantes est le ribosomique nucléaire région ITS 13-15,17. Actuellement, nous faisons des progrès vers le multiplexage de l'amplification du chloroplaste trnL-F région avec celle de la centrale nucléaire de sa région dans le même tube PCR. Multiplexage plaste avec les régions d'ADN nucléaire serait potentiellement contourner les limites de l'utilisation soit seul, mais de telles méthodes nécessitent l'optimisation et l'évaluation supplémentaire pour déterminer la faisabilité au cas par cas. L'utilisation d'indicateurs quantitatifs PCR en temps réel (qPCR) et les nouvelles technologies, telles que les balises moléculaires (sondes fluorescentes d'amorces), sont également en cours d'évaluation à sécurité intrinsèque et précise les méthodes de génotypage des plantes.
Le protocole présenté ici fournit une approche rapide et fiable de distinguer le JBG revenir de celle de type sauvage imperata cylindrique. Nous encourageons l'utilisationrs de ce protocole à nous contacter pour signaler les résultats découlant de l'utilisation de ce protocole. Cette information partagée permettra de fournir des informations sur la distribution de JBG revient. Cela permettra également d'aider les régulateurs à l'USDA de prendre des décisions éclairées sur les mesures qui peuvent être nécessaires pour contourner la propagation et l'hybridation potentiel des variétés imperata cylindrique hautement invasifs.
Déclarations de divulgation
Nous n'avons rien à communiquer.
Remerciements
Nous tenons à remercier Alan Tasker (USDA-APHIS, Riverdale, MD), Stephen Compton (Clemson), Sherry Aultman (Clemson), Craig Ramsey (USDA-APHIS, Fort Collins, CO), et Betty Marose (UMD) de l'aide pour obtenir des échantillons . Nous remercions les étudiants Andrew Adrian (UA-Huntsville) et Derek Thacker (UA-Huntsville) pour leur aide dans l'essai de ce protocole, et Joseph Herdy pour son travail dans le tournage de la vidéo. Ce travail a été financé par National Fish and Wildlife Foundation.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| DNeasy usine Mini Kit | Qiagen, Valencia, CA | 69104 ou 69106 | Toute kit génomique réputé plante à ADN qui donne l'ADN de bonne qualité devrait fonctionner correctement pour ces procédures. |
| trnF (GAA)-F | Oblique Bio, Inc | 3-0578 | Amorce le contrôle positif avant |
| trnL (exon 5 ')-C | Oblique Bio, Inc | 3-0579 | Inverser amorce contrôle positif |
| trnLF-C-F1 | Oblique Bio, Inc | 3-0864 | Transférer de type sauvage amorce imperata cylindrique |
| trnLF-C-R1 | Oblique Bio, Inc | 3-0865 | Inverserde type sauvage amorce imperata cylindrique |
| trnLF-R-F2 | Oblique Bio, Inc | 3-0866 | Transférer JBG JBG et revenir amorce |
| trnLF-R-R2 | Oblique Bio, Inc | 3-0867 | Inverser JBG JBG et revenir amorce |
| Avantage UltraPure PCR dNTP Mix | Clontech, Mountain View, CA | 639125 | |
| Avantage 2 polymérase | Clontech, Mountain View, CA | 639201 639202 ou | Une bonne relecture, de démarrage à chaud Taq polymérase |
Références
- Holm, L. G., Pancho, J. V., Herberger, J. P., Plucknett, D. L. A Geographical Atlas of World Weeds. , Krieger Publishing Company. Malabar, Florida, U.S. (1979).
- CABI, Crop Protection Compendium. Commonwealth Agricultural Bureau International (CABI). , Wallingford, UK. (2007).
- MacDonald, G. E. Cogongrass (Imperata cylindrica) - Biology, ecology, and management. Critical Reviews in Plant Sciences. 23, 367-380 (2004).
- Talley, S. M., Cseke, L. J. Molecular diagnostic technologies for invasive plants. CPHST Fort Collins Laboratory 2009 Annual Report. Zink, R. , USDA-APHIS-PPQ-CPHST. Fort Collins, Colorado. 27-28 (2009).
- Dozier, H., Gaffney, J. F., McDonald, S. -K., Johnson, E. R. R. L., Shilling, D. G. Cogongrass in the United States: History, ecology, impacts, and management. Weed Technology. 12, 737-743 (1998).
- Chikoye, D., Ekeleme, F. Weed flora and soil seedbanks in fields dominated by Imperata cylindrica in the moist savannah of West Africa. Weed Research. 41, 475-490 (2001).
- Weber, E. Invasive Plant Species of the World: A Reference Guide to Environmental Weeds. , CABI Publishing. Wallingford, UK. (2003).
- Patterson, D. T. Shading effects on growth and partitioning of plant biomass in Cogongrass (Imperata cylindrica) from shaded and exposed habitats. Weed Science. 28, 735-740 (1980).
- Cole, J. T., Cole, J. C. Ornamental grass growth response to three shade intensities. Journal of Environmental Horticulture. 18, 18-22 (2000).
- Bryson, C. T., Koger, C. H., Byrd, J. D. Effects of temperature and exposure period to heat on cogongrass (Imperata cylindrica) viability. Weed Technology. 21, 141-144 (2007).
- Capo-chichi, L. J. A., Faircloth, W. H., Williamson, A. G., Patterson, M. G., Miller, J. H., van Santen, E. Invasion Dynamics and Genotypic Diversity of Cogongrass (Imperata cylindrica) at the Point of Introduction in the Southeastern United States. Invasive Plant Science and Management. 1 (2), 133-141 (2008).
- Talley, S. M., Ramsey, C. L. Experimentally assessing the invasive potential of plants. CPHST Laboratory 2009 Annual Report. Zink, R. , USDA-APHIS-PPQ-CPHST. Fort Collins, Colorado. 29-30 (2009).
- Hodkinson, T. R., Chase, M. W., Lledó, M. D., Salamin, N., Renvoize, S. A. Phylogenetics of Miscanthus, Saccharum and related genera (Saccharinae, Andropogoneae, Poaceae) based on DNA sequences from ITS nuclear ribosomal DNA and plastid trnLintron and trnL-F intergenic spacers. J. Plant Res. 115, 381-392 (2002).
- Kress, W. J., Wurdack, K. J., Zimmer, E. A., Weigt, L. A., Janzen, D. H. Use of DNA barcodes to identify flowering plants. Proc. Natl. Acad. Sci. U.S.A. 102 (23), 8369-8374 (2005).
- Roodt-Wilding, R., Spies, J. J. Phylogenetic relationships in southern African chloridoid grasses (Poaceae) based on nuclear and chloroplast sequence data. Systematics and Biodiversity. 4, 401-415 (2006).
- Brody, J. R., Kern, S. E. Sodium boric acid: a Tris-free, cooler conductive medium for DNA electrophoresis. BioTechniques. 36, 214-216 (2004).
- Chou, C. -H., Tsai, C. C. Genetic variation in the intergenic spacer of ribosomal DNA of Imperata cylindrica (L.) Beauv. var. major (Cogongrass) populations in Taiwan. Botanical Bulletin of Academia Sinica (Taipei). 40, 319-332 (1999).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon