Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Reinigung und Aggregation des Amyloid Precursor Protein intrazelluläre Domäne
In diesem Artikel
Zusammenfassung
Ein Verfahren für die großtechnische Reinigung des APP intrazelluläre Domäne (AICD) beschrieben. Wir beschreiben auch Methoden zu induzieren In vitro AICD Aggregation und Visualisierung mittels Rasterkraftmikroskopie. Die beschriebenen Methoden sind nützlich für biochemische / strukturelle Charakterisierung der AICD und die Auswirkungen der molekularen Chaperone auf die Aggregation.
Zusammenfassung
Amyloid-Vorläuferprotein (APP) ist ein Typ I Transmembran-Proteins mit der Pathogenese der Alzheimer-Krankheit (AD) zugeordnet ist. APP wird durch einen großen extrazellulären Domäne und einer kurzen zytosolische Domäne bezeichnet das APP intrazelluläre Domäne (AICD) aufweist. Während der Reifung durch den sekretorischen Weg, kann APP durch Proteasen genannt α, β und γ-Sekretase 1 gespalten werden. Sequentielle proteolytische Spaltung von APP mit β und γ-Sekretasen führt zu der Produktion eines kleinen Peptids proteolytische, genannt Aß, das amyloidogene ist und der Kernbestandteil von senilen Plaques. Die AICD ist auch befreit von der Membran nach Sekretase Verarbeitung und durch Interaktionen mit Fe65 und Tip60 kann in den Kern in Transkriptionsregulation mehrerer Zielgene 2,3 teilzunehmen translozieren. Protein-Protein-Wechselwirkungen zwischen den AICD beeinträchtigen Menschenhandel, Verarbeitung und zelluläre Funktionen von Holo-APP und seine C-terminal Fragmente. Wir haben kürzlich gezeigt, dass AICD wird Aggregat kann in vitro, und dieser Prozess durch den AD-verwickelt molekularen Chaperon Ubiquilin-1 4 inhibiert. Übereinstimmend mit diesen Ergebnissen, die AICD hat hydrophoben Domänen ausgesetzt und ist untrennbar in vitro 5,6 ungeordnet, aber es erhält stabile sekundäre Struktur, wenn sie Fe65 7 gebunden. Wir haben vorgeschlagen, dass Ubiquilin-1 unangemessen inter-und intramolekulare Wechselwirkungen von AICD verhindert die Aggregation in vitro und in intakten Zellen 4. Während die meisten Studien über die Rolle von APP in der Pathogenese von AD zu konzentrieren, ist die Rolle in diesem Prozess AICD nicht klar. Expression AICD hat sich gezeigt, Apoptose zu induzieren 8, zu modulieren Signalwege 9 und zum Calcium regulieren Signalisierung 10. Überexpression von AICD und Fe65 in einer transgenen Mausmodell induziert wie Alzheimer Pathologie 11, und vor kurzem hat AICD in BH nachgewiesenin Lysaten mittels Western Blot, wenn mit geeigneten Antigen-Retrieval-Techniken 12. Um strukturelle, biochemische und biophysikalische Untersuchungen des AICD erleichtern, haben wir ein Verfahren zur rekombinanten große Mengen an hochreinem AICD Protein produzieren. Wir beschreiben außerdem ein Verfahren zur Induktion der in vitro thermische Aggregation AICD und Analyse mittels Rasterkraftmikroskopie. Die beschriebenen Methoden sind für biochemische, biophysikalische und strukturelle Charakterisierung des AICD und die Auswirkungen der molekularen Chaperone auf AICD Aggregation nützlich.
Protokoll
Ein. Expression von rekombinantem APP intrazelluläre Domäne (AICD)
- Transform E. coli-Stamm BL21 mit menschlichen AICD (Reste 649-695 von APP, neuronale Isoform Nummerierung) in den Vektor pGEX-4T-1 (GE Healthcare) kloniert. Dieser Vektor wird als C-terminalen Teil eines Fusionsprotein von Glutathion-S-Transferase (GST) AICD exprimieren. Dieser Vektor kodiert für ein Thrombin auch Spaltsequenz um das Entfernen des GST-Einheit zu erleichtern. Details Klonierung in pGEX-4T-1 AICD finden Sie in unserem früheren Veröffentlichung 4 gefunden werden.
- Aus einer einzigen Kolonie, Animpfen von 10 ml LB-Medium mit Ampicillin (100 ug / ml) und bei 37 ° C über Nacht unter starkem Schütteln.
- Am nächsten Morgen impfen 400 ml LB / Ampicillin in einem Ein-Liter-Kolben mit 5 ml der Übernacht-Kultur.
- Bei 37 ° C unter kräftigem Schütteln bis zu einer optischen Dichte bei 600 nm erreicht hat 0,4 bis 0,6. Dies dauert in der Regel 2 bis 2,5 Stunden.
- Induzieren die Expression von GST-AICD durch Zugabe von 0,4 mM Isopropyl-beta-D-thiogalactopyranosid (IPTG). Bei 37 ° C unter kräftigem Schütteln für 5 Stunden.
- Zentrifugation der Zellen bei 6000 × g für 15 min bei 4 ° C. Das Zellpellet kann bei -80 ° C für mehrere Monate werden an diesem Punkt des Verfahrens abgelegt.
2. Reinigung von GST-AICD
- Planen 50 ml Lysepuffer wie folgt: 1 mM Dithiothreitol, 1 mM Ethylendiamintetraessigsäure (EDTA), 0,1% Triton-X100, 1 mM Phenylmethylsulfonylfluorid (PMSF), Complete Protease-Inhibitor-Cocktail (Roche Applied Science) und phosphatgepufferter Salzlösung. Chillen auf Eis.
- Auftauen Bakterienpellet (wenn vorher eingefroren) aufEis.
An diesem Punkt ist es wichtig, dass alle Schritte bei 4 ° C durchgeführt, um die Proteolyse zu begrenzen. Alle Rohre, Puffer und andere Reagenzien sollten vorgekühlt. Wenn Sie einen Französisch drücken oder Emulgator für die Lyse, sollten diese vorgekühlten als gut.
- Gründlich resuspendieren das Bakterienpellet in 20 ml Lyse-Puffer. Eine Kombination von Verwirbelung und Verreiben mit 10 ml Pipette auf Eis wird eine vollständige Resuspension des Pellets.
- Passieren die resuspendierten Bakterienzellen durch einen Emulgator (z. B. Avestin EmulsiFlex-C3) oder Französisch Druckzelle, vorgekühlt auf 4 ° C bei 30.000 psi. Führen Sie das Lysat durch die Emulgator ein zweites Mal. Sammeln Sie das Lysat in ein vorgekühltes 50 ml Tube.
Der Hauptvorteil dieser beiden Instrumente ist, dass sie Erwärmung der Probe zu minimieren. Die am häufigsten verwendete Technik der Ultraschallbehandlung erzeugt erhebliche Hitze an der Spitze der Sonde und der Ultraschallbehandlungkann Aggregation von rekombinanten Proteinen führen.
- Zentrifuge das Lysat bei 15000 × g für 30 min. Trennen Sie und halten Sie den Überstand in ein vorgekühltes 50 ml Tube.
- Add 500 pl einer 50% igen Aufschlämmung von Glutathion-Agarose (Sigma-Aldrich). Drehen für 30 min bei 4 ° C. Während die Probe rotiert, machen frische Elutionspuffer: 50 mM Tris 7,8, 0,1% Triton-X100, 0,5 mM Dithiothreitol, 10 mM reduziertes Glutathion.
Der einzige Schritt des Protokolls, das bei Raumtemperatur durchgeführt wird, ist der Elutionsschritt. Somit wird der Elutionspuffer bei Raumtemperatur gehalten. Elutionspuffer sollte frisch zubereitet werden.
- Gießen des Lysats und Schlamm in eine Einweg-5 "Polystyrol Chromatographiesäule mit grobem (90-130 um) Filtern (Evergreen Scientific). Die Säule wird 5 mal mit 3 ml Phosphat-gepufferter Kochsalzlösung.
- Verschließen Sie die unteren Ende der Säule und 500 ul der Raumtemperatur Elutionspuffer. Verschließen Sie die Spitze derSäule und rotieren für 5 min bei Raumtemperatur. Sammeln des Eluats in ein gekühltes 1,5 ml Mikrozentrifugenröhrchen. Wiederholen Sie die Elution zwei weitere Male (1,5 ml insgesamt Eluat).
- Dialysieren das Eluat in einem 10.000 Molekulargewichtsschnitt Dialysemembran (zB Thermo Scientific Slide-A-Lyzer Dialyse Kassetten) in 4 Liter phosphatgepufferter Kochsalzlösung über Nacht bei 4 ° C. Dialysieren gegen weitere 4 Liter Phosphat-gepufferter Kochsalzlösung bei 4 ° C für 1 Stunde am folgenden Tag.
- Entfernen des Proteins aus der Kassette und Dialyse durchführen Proteinquantifizierung Assay zur Ausbeute von GST-AICD bestimmen. Ein typisches Reinigungsverfahren aus 400 ml Kultur werden in Ausbeuten von> 20 mg gereinigtem Protein bei Konzentrationen von 10-20 mg / ml ergeben. Aliquot das Protein in 200 ul Aliquots und einfrieren bei -80 ° C.
3. Thrombin Spaltung von GST-AICD und Reinigung von AICD
- Thaw 200 ul des gereinigten GST-AICD und fügen Sie 20 ul 1 U / ul Thrombin. über Nacht bei 37 ° C.
Die Qualität und Reinheit von Thrombin variiert beträchtlich zwischen den Herstellern und können eine bedeutende Quelle von Verunreinigungen sein. Wir verwenden Thrombin von GE Healthcare (siehe Tabelle von Reagenzien). Nach Inkubation über Nacht, sollte> 95% des Fusionsproteins abgespalten (1B) ist.
- Nachdem über Nacht Spaltung mit Thrombin, ist es wahrscheinlich, dass eine Fraktion des freigesetzten AICD Aggregation unterzogen und die Lösung trüb erscheinen. Löschen des aggregiertes Material durch Zentrifugation bei 20.000 × g für 30 min bei 4 ° C. Entfernen Sie den Überstand in ein vorgekühltes Mikrozentrifugenröhrchen.
- Fügen Sie 50 ul 50% Glutathion-Agarose-Suspension und 50 ul 50% p-Aminobenzamidin-Agarose Aufschlämmung zu GST und Thrombin zu entfernen sind. Inkubieren mit einer Drehung 5 min bei Raumtemperatur.
- Zentrifuge kurz Sediment die Agarosekügelchen, und entfernen Sie den Überstand in ein frisches Röhrchen. Auszug ter GST vier weitere Male mit Glutathion-Agarose. Eine einzelne Extraktion mit p-Aminobenzamidin-Agarose ist ausreichend, um die 20 U Thrombin zu entfernen.
- Führen Sie ein Protein Quantifizierung Assay auf 5 ul des gereinigten AICD. Typische Ausbeuten aus einer 400 ml Kultur sind 50-100 ug bei einer Konzentration von 0,2 bis 0,5 mg / ml. Höhere Renditen sind möglich durch Spaltung größere Mengen an GST-AICD.
Gereinigtes AICD sollte vorbereitet biochemischen / biophysikalischen Charakterisierung frisch. Nicht verwendetes Material muss verworfen werden. Es ist auch möglich, die durch Lyophilisieren der AICD Probe und Resuspendieren in einem kleineren Volumen an Puffer 6 konzentrieren. Wenn Erfassen AICD erfordert Blotting (wie nach einer Filtereinheit oder Dot-Blot-Assay), Antigen Retrieval auf der Membran durchgeführt werden sollte, wie durch Pimplikar und Suryanarayana 12 beschrieben.
4. AICD Aggregation für Atomic Force Microscopy (AFM)
- Frisch p hergestellten verdünnen AICDhosphate Kochsalzlösung auf eine Konzentration von 0,1 mg / ml in einem Gesamtvolumen von 15 ul. An diesem Punkt können verschiedene Verbindungen und / oder Proteine hinzugefügt zu bestimmen, ob sie AICD Aggregation hemmen. Zum Beispiel haben wir gezeigt, dass äquimolare Konzentrationen von Ubiquilin-1 Aggregation AICD verhindern in diesem Assay 4.
Es ist auch möglich, größere Aggregat Reaktionsvolumina für biochemische Experimente wie Abfangfilter Assays und Lichtstreuung 4. Wir haben erfolgreich war Durchführen Abfangfilter Assays auf Reaktionsgemische so groß wie 400 ul mit AICD auf Konzentrationen so niedrig wie 1 uM verdünnt.
- Induzieren thermische Aggregation durch Inkubation der Proben bei 43 ° C unter Schütteln bei 800 rpm in einer Eppendorf-Thermomixer für 48 Stunden. Diese Temperatur wurde auf der Grundlage Chaperon Assays, die die thermische Aggregation des Modells Chaperon Substrat Citratsynthase 4,13 untersuchen gewählt.
Thermische Aggregation ist eine häufig verwendete Methode, um den strukturellen Übergang eines Proteins aus einem nativen veranlassen, eine nicht-native Struktur (was gewöhnlich Beta-Faltblatt-reich), was zur Bildung von intermolekularen Aggregate führt. Thermische Aggregation wird auch verwendet, um die Funktion von Chaperonen, die hydrophoben Segmente aus Bilden unangemessen intermolekulare Wechselwirkungen (und damit zu verzögern oder zu verhindern thermische Aggregation) abzuschirmen untersuchen.
- Verdünnen Sie die Probe 20-fach in deionisiertem Wasser und Spot 2 ul auf frisch gespaltenem Glimmer. Trocknen Sie die Probe in einem Exsikkator über Nacht. Es wird notwendig sein, um empirisch die optimale Verdünnung für die Bildgebung und somit mehrere Verdünnungen im Bereich von 10 - bis 100-fach wäre klug.
- Visualisieren Sie die aggregierten AICD mit dem AFM Betriebssystem im Tapping-Modus 14. Die Ausleger verwendet werden, sind Gold-Mikrotabletten scharfe Nitrid Hebel (MSNL, Bruker) mit einem nominalen Spitzenradius von 2 nm. Typische Klopfen Amplituden währendten Imaging sind 10-20 nm bei der Resonanzfrequenz der Ausleger (30-50 kHz).
Wir fanden, dass die Bildqualität und Kontrast verschiedener Proteinaggregate auf die aufgebrachte Kraft und die Dauer des Experiments abhing. Perturbation zu Probe durch die Spitze zu minimieren, behielten wir die angelegte Kraft relativ gering, indem sie einen Sollwert-Verhältnis oberhalb von 95% und die Begrenzung der Abtastung einer jeden Probe auf 5-10 min. Bildverarbeitung wurde mit WSxM Software (Nanotec) durchgeführt. Standard-Bildverarbeitungsfunktionen bestand aus dem Flugzeug Subtraktion und Abflachung. Unsere Laboratorien ein "Home-built" AFM 15 eine Schnittstelle zu einem kommerziellen Rastersondenmikroskop Steuerung (Nanotec).
5. Repräsentative Ergebnisse
Die Expression und relative Anreicherung von GST-AICD ist in 1A gezeigt. Nach Lyse ist die Mehrheit von dem Fusionsprotein in der löslichen Fraktion und damit Extraktion aus Inclusiauf Körper ist nicht erforderlich. Das Material, aus dem Glutathion-Agarose-Säule eluiert ist> 95% rein. Bei der Herstellung in 1 gezeigt, war die Konzentration des Proteins aus der Säule eluiert 14,5 mg / ml, und die Ausbeute betrug 22 mg (aus einer Startkultur von 400 ml). Spaltung von 200 ul dieses Präparats über Nacht mit 20 U Thrombin in Folge nahezu 100% Spaltung des Fusionsproteins (1B). Die Menge an Thrombin verwendet wird, ist niedrig genug, wie es nicht sein, nachweisbar durch Coomassie-Blau-Färbung (siehe "Cleaved" lane, Abbildung 1B). Entfernen des Thrombins und GST führte zu einem gewissen Verlust an Material, jedoch> 90% rein mit einer Ausbeute in dieser prep von 70 ug AICD in einer Konzentration von 0,35 mg / ml betrug. 1C zeigt das Flußdiagramm für die Aggregation von AICD und nachfolgende Analyse. AICD Aggregate durch AFM abgebildet sind typischerweise Sphäroids / amorph und in der Größe von 50 bis 100 nm (Abb. 1D, F). AggregierteMaterial ist nicht nachweisbar, wenn eine äquimolare Menge des Chaperon Ubiquilin-1 an die Aggregations-Reaktionsvorrichtung (1E) 4 zugesetzt.
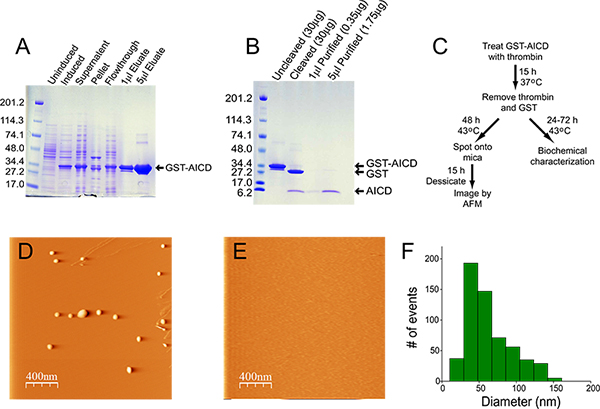
Abbildung 1. Aufreinigung und Aggregation von AICD. A) Coomassie-Blau-Färbung der Proben in jedem Schritt des Reinigungsverfahrens gesammelt. Für den nicht induzierten und induzierten Proben wurden 20 ul gesamten Bakterien auf der Gel laufen. Für die Überstandsfraktion wurden 5 ul Lysat (aus einem Gesamtvolumen von 20 ml) auf dem Gel laufen. Für die Pelletfraktion wurde das Pellet in 20 ml Lyse-Puffer resuspendiert und 5 ul dieses Materials wurden in einem Wasserbad Sonicator während 30 min bei 4 ° C beschallt vor dem Laden auf das Gel. Für die Durchströmung wurden 5 pl auf dem Gel laufen. Die Mengen des Eluats auf das Gel geladen sind angegeben. B) Coomassie-Blau gefärbten Gel ungespaltenen und gespaltenen GST-AICD (2 ul) und 1 und 5 & mu; l gereinigte AICD. C) Flussdiagramm der AICD Reinigung, Aggregation und Analyse. D, E) Repräsentative AFM-Bild von thermisch aggregiert AICD in Abwesenheit (D) und in Gegenwart (E) des molekularen Chaperons Ubiquilin-1. F) Histogramm der Größenverteilung der AICD Aggregate.
Diskussion
In diesem Protokoll haben wir ein Verfahren zur Gewinnung von hochreinem AICD für strukturelle, biophysikalischen und biochemischen Analysen skizziert. Dieses Verfahren erfordert keine ausgeklügelte Chromatographie Geräte und ist damit zugänglich für die meisten Laboratorien. Andere Gruppen haben AICD 5-7,16 gereinigt, einschließlich GST-AICD 17-19, für biochemische / Strukturanalysen. Nachteile zu früheren Protokollen gehören schlechte Löslichkeit von AICD 16, weniger als idea...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren bedanken sich bei Dr. Hui Zheng (Baylor College of Medicine) für die APP cDNA danken. Diese Arbeit wurde vom NIH Zuschüsse R21AG031948 (DB, JMB), F30AG030878 (ESS), R01DK073394 (AFO), die John Sealy Memorial Endowment Fund for Biomedical Research (AFO) und der Jean C. und William D. Willis Neuroscience Research Endowment finanziert (ESS). JMB ist ein Gelehrter in der Translational Research Scholar Program und ein Mitglied der University Of Texas Medical Branch Claude E. Pfeffer Ältere Amerikaner Independence Center (unterstützt durch NIH Grants UL1RR029876 und P30-AG-024.832, jeweils).
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| pGEX-4T-1 | GE Healthcare | 28-9545-49 | |
| Thrombin | GE Healthcare | 27-0846-01 | |
| Ampicillin | Fisher Scientific | BP1760 | |
| Bradford-Protein-Assay-Reagenz | Bio-Rad | 500-0002 | |
| Coomassie-Blau | Bio-Rad | 161-0786 | |
| IPTG (Isopropyl-beta-D-thiogalactopyranosid) | Sigma-Aldrich | I6758 | |
| Glutathion-Agarose | Sigma-Aldrich | G4510 | |
| p-Aminobenzamidin-Agarose | Sigma-Aldrich | A7155 | |
| Komplette Proteaseinhibitor-Cocktail | Roche | 11836170001 | |
| Slide-A-Lyzer Dialyse Kassetten | Thermo Scientific | 66380 | |
| Chromatographiesäulen | Evergreen Scientific | 208-3367-050 | |
| Emulgator | Avestin, Inc. | EmulsiFlex-C3 | Highly recommended |
| Eppendorf Thermomixer | Eppendorf | 022670107 | |
| Mica Disks | Ted Pella | 50-12 | |
| AFM Cantilever | Bruker | MSNL-10 | |
| WSxM Software | Nanotec | N / A | Free Download |
Referenzen
- De Strooper, B., Vassar, R., Golde, T. The secretases: enzymes with therapeutic potential in Alzheimer disease. Nature reviews. Neurology. 6, 99-107 (2010).
- Chang, K. A., Suh, Y. H. Possible roles of amyloid intracellular domain of amyloid precursor protein. BMB reports. 43, 656-663 (2010).
- McLoughlin, D. M., Miller, C. C. The FE65 proteins and Alzheimer's disease. J. Neurosci. Res. 86, 744-754 (2008).
- Stieren, E. S. Ubiquilin-1 is a molecular chaperone for the amyloid precursor protein. J. Biol. Chem. 286, 35689-35698 (2011).
- Ramelot, T. A., Nicholson, L. K. Phosphorylation-induced structural changes in the amyloid precursor protein cytoplasmic tail detected by NMR. J. Mol. Biol. 307, 871-884 (2001).
- Ramelot, T. A., Gentile, L. N. Transient structure of the amyloid precursor protein cytoplasmic tail indicates preordering of structure for binding to cytosolic factors. Biochem. 39, 2714-2725 (2000).
- Radzimanowski, J. Structure of the intracellular domain of the amyloid precursor protein in complex with Fe65-PTB2. EMBO Rep. 9, 1134-1140 (2008).
- Ohkawara, T., Nagase, H., Koh, C. S., Nakayama, K. The amyloid precursor protein intracellular domain alters gene expression and induces neuron-specific apoptosis. Gene. 475, 1-9 (2011).
- von Rotz, R. C. The APP intracellular domain forms nuclear multiprotein complexes and regulates the transcription of its own precursor. J. Cell Sci. 117, 4435-4448 (2004).
- Hamid, R. Amyloid precursor protein intracellular domain modulates cellular calcium homeostasis and ATP content. J. Neurochem. 102, 1264-1275 (2007).
- Ghosal, K., Stathopoulos, A., Pimplikar, S. W. APP intracellular domain impairs adult neurogenesis in transgenic mice by inducing neuroinflammation. PLoS ONE. 5, e11866 (2010).
- Pimplikar, S. W., Suryanarayana, A. Detection of APP intracellular domain in brain tissue. Met. Molecul. Biol. 670, 85-91 (2011).
- Buchner, J., Grallert, H., Jakob, U. Analysis of chaperone function using citrate synthase as nonnative substrate protein. Met. Enzymol. 290, 323-338 (1998).
- Hansma, H. G. Recent advances in atomic force microscopy of DNA. Scanning. 15, 296-299 (1993).
- Valbuena, A. Quasi-simultaneous imaging/pulling analysis of single polyprotein molecules by atomic force microscopy. Rev. Sci. Instrum. 78, 113707 (2007).
- Radzimanowski, J., Beyreuther, K., Sinning, I., Wild, K. Overproduction, purification, crystallization and preliminary X-ray analysis of human Fe65-PTB2 in complex with the amyloid precursor protein intracellular domain. Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun. 64, 409-412 (2008).
- Chen, T. Y., Liu, P. H., Ruan, C. T., Chiu, L., Kung, F. L. The intracellular domain of amyloid precursor protein interacts with flotillin-1, a lipid raft protein. Biochem. Biophys. Res. Commun. 342, 266-272 (2006).
- Kim, M. Y. Regulation of Notch1 signaling by the APP intracellular domain facilitates degradation of the Notch1 intracellular domain and RBP-Jk. J. Cell Sci. 124, 1831-1843 (2011).
- Lazarov, O. Axonal transport, amyloid precursor protein, kinesin-1, and the processing apparatus: revisited. J. Neurosci. 25, 2386-2395 (2005).
- Kakuda, N. Equimolar production of amyloid beta-protein and amyloid precursor protein intracellular domain from beta-carboxyl-terminal fragment by gamma-secretase. J Biol. Chem. 281, 14776-14786 (2006).
- Gosal, W. S., Myers, S. L., Radford, S. E., Thomson, N. H. Amyloid under the atomic force microscope. Protein Pept. Lett. 13, 261-270 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten