Method Article
A Guided Materialien Screening-Ansatz für die Entwicklung von Quantitative Sol-Gel-Derived Protein Microarrays
In diesem Artikel
Zusammenfassung
Eine geführte Material-Screening Ansatz zur Sol-Gel abgeleiteten Protein-Microarrays mit dotierten eine aufstrebende Pin-Druckverfahren der Fertigung entwickeln beschrieben. Diese Methodik wird durch die Entwicklung von Acetylcholinesterase-und Multi-Kinase-Microarrays, die für eine kostengünstige Screening kleiner Moleküle verwendet werden aufgezeigt.
Zusammenfassung
Microarrays haben den Einsatz in der Entwicklung von High-Throughput-Assays für neue Materialien und Entdeckung von niedermolekularen Leitstrukturen gefunden. Hier beschreiben wir eine geführte Material-Screening Ansatz zur Sol-Gel-basierten Materialien, die zur Herstellung von dreidimensionalen Protein-Microarrays zu identifizieren. Der Ansatz identifiziert zunächst Materialien, die als Microarrays gedruckt werden können, schränkt die Anzahl von Materialien durch diejenigen zu identifizieren, die mit einem bestimmten Enzym-Assay sind, und dann schärft in der optimalen Materialien basierend auf Beibehaltung der maximalen Enzymaktivität. Dieser Ansatz wird angewendet, um Microarrays geeignet für zwei verschiedene Enzym-Assays, eine mit Acetylcholinesterase und die andere mit einem Satz von vier zentralen Kinasen bei Krebs eine Rolle zu entwickeln. In jedem Fall war es möglich, die für die quantitative Mikroarrays niedermolekulare Screening-Assays und Produktion von Dosis-abhängigen Inhibitor-Wirkungs-Kurven verwendet werden könnten, zu erzeugen. Wichtig ist, dass die Fähigkeit zu screenen viele Kumpelstoffe produziert Informationen über die Arten von Materialien, die am besten geeignet sowohl Microarray Produktion und Speicherung von Enzymaktivität. Die Materialien Daten geben einen Einblick in Grundmaterial Anforderungen für optimale Anpassung, High-Density-Sol-Gel-abgeleiteten Microarrays.
Einleitung
Microarrays haben Popularität innerhalb der wissenschaftlichen Gemeinschaft als eine Methode zur Steigerung der Durchsatz Assay gewonnen. Da die Entwicklung der Microarray-Technologie, um die Genexpression 1 in der Mitte der 1990er Jahre zu bewerten, haben Microarrays Einsatz in der Entwicklung von High-Throughput-Assays gefunden, um Protein-Protein-und Protein-Interaktionen kleines Molekül zu identifizieren und neue Materialien mit einzigartigen Eigenschaften zu finden. 2-5 In jüngerer Zeit wurden Mikroarrays, wobei bestimmte Materialien verwendet werden, um funktionelle Proteine zu immobilisieren, wodurch ein dreidimensionales Mikroarrayelement in denen Proteine eingeschlossen entwickelt, das einfache Messung der enzymatischen Aktivität und Hemmung auf dem Microarray selbst mit einem geeigneten Fluoreszenz Assay, der auf das Substrat Umsatz gekoppelt ist. 5-10 Wichtig ist, dass solche Mikroarrays entwickelt, um alle notwendigen Komponenten, um Bildschirm Proben und Kontrollen gehören zusammen in einem sehr parallel Mode werden. 5,8
Frühe Beispiele von Protein-Microarrays wurden typischerweise unter Verwendung von Standard-Verfahren für die Immobilisierung von Proteinen an feste Träger, wie eine kovalente Bindung, erfassen 11 Affinität 3 und physikalische Adsorption. 12 Während diese Verfahren von Bio-Immobilisierung erhöht Probenkonzentration und beschleunigte Kinetik erforderlich für erlauben Assay Miniaturisierung, leiden sie jeweils Nachteile. Im Allgemeinen führen alle eine Reduktion der nativen Biomolekül Funktionalität durch chemische Modifikation der Oberfläche, erschwerter Zugang zum aktiven Zentren oder unsachgemäße Ausrichtung durch unspezifische Immobilisierung. So sind alle Methoden führen zu geringeren Empfindlichkeit des Assays trotz einer Steigerung der Abscheidung Biomolekül, eine Antwort, die wahrscheinlich entsteht durch die Notwendigkeit, künstlich binden Biomolekülen an einer Oberfläche.
Eine aufstrebende Ansatz für die Herstellung von funktionellen Biomolekülen Microarrays ist durch den Stift-Druck von Protein-dotiertenKieselsole auf festen Trägern, die in der Regel entweder funktionalisierte Glasplättchen oder einzelnen Vertiefungen von Mikrotiterplatten. Das Sol-Gel-Prozess selbst erfolgt in einer wässrigen Umgebung bei Raumtemperatur, und es ist die flüssige Vorstufe, die gedruckt und dann Gele zum Einschluss von Biomolekülen in der 3D-Matrix, was eine hohe Eiweissbelastung 13 sowie Einschluss von mehreren Komponenten in gleich Mikroarrayelement. 13,14 Tailoring des Sol-Gel-abgeleitete Material durch sorgfältige Auswahl der verschiedenen Silikavorstufen durchgeführt werden, sowie durch die Veränderung der wässrigen Komponente durch die Verwendung von verschiedenen Puffern (pH, Ionenstärke) und Aufnahme verschiedene Zusätze (Polymere, kleine Moleküle), um einen optimalen Materialfluss zu erreichen, hängt die spezifische Natur von denen auf dem Biomolekül, das eingeschlossen ist. 10
Eine mögliche Einschränkung mit den Entwicklungsländern Sol-Gel abgeleiteten Protein-Microarrays über Pin-Druck verbunden ist,die Notwendigkeit, die Sol-Gel-basierte Verbundwerkstoffe, die ohne Geliermittel in den Stift gedruckt werden können oder unerwünschte Eigenschaften zeigen (nicht reproduzierbar Messfelder, Rissbildung, schlechte Haftung, Inkompatibilität mit Assay-Komponenten, schlechte Protein-Aktivität) einmal auf eine Oberfläche gedruckt identifizieren. 5 Gleichzeitige Optimierung all dieser Parameter schließt einen Ansatz, bei de novo Materialien ausgelegt werden kann, oder untersucht langsam in serieller Weise. Auf der anderen Seite ist Zufalls-Screening von mehreren tausend oder zehntausend Materialien weder zeitlich noch kostengünstiger.
In diesem Artikel beschreiben wir ein direktes Screening Ansatz, der die schnelle Identifizierung von geeigneten Materialien können für die Herstellung von Protein-Arrays, ohne dass nach dem Zufallsprinzip Screening einer großen Anzahl von Materialien. Mit einer geführten Ansatz werden Materialien, die für Microarray Druck zunächst identifiziert, gefolgt von einer Reihe von kleinen Bildschirme zu identifizieren optimale Sol-Gel-abgeleitete Materialienal-Kombinationen, die reproduzierbar gedruckt werden, ohne zu reißen und sind kompatibel mit einer gegebenen Assay. Schließlich werden optimale Materialien basierend auf Beibehaltung der Enzymaktivität und Leistung in einem abschließenden kleinen Molekülen Screening-Assay identifiziert. Auf diese Weise kann eine optimale Materialien aus vielen tausend Kandidaten mit nur ein paar hundert Schritte Assay identifiziert werden. Wir zeigen, diesen Ansatz für die Herstellung von sowohl hoher Dichte Acetylcholinesterase und Multi-Kinase-Microarrays und die Verwendung solcher Microarrays für kleine Molekül-Screening.
Protokoll
1. Herstellung von Additiv-Lösungen
- Planen 25 und 50 mM Tris (hydroxymethyl) aminomethan (Tris-Base, M r 121,14 g / mol) und HEPES (M r 238,3 g / mol) bei pH 7,0, 7.2, 7.4, 7.6, 7.8, 8.0 und 8.2 in 50 ml Zentrifugenröhrchen Rohre. Den pH-Wert mit 1 N HCl und NaOH sind. Bei Raumtemperatur lagern und ersetzen nach 3 Monaten.
- Planen 24% (w / v) Poly (vinylalkohol) (PVA): wiegen 2,4 g PVA (M w 9.000 - 10.000, 80% hydrolysiert) und in den 10 ml entionisiertem destilliertem Wasser (ddH 2 O), lösen die Polymer-Pellets durch Vortexen. Über die 24% (w / v) Lösung in gleichen Volumina von 16% (w / v), 12% (w / v) und 8% (w / v) PVA-Lösungen herzustellen. Degas Lösungen durch Ultraschall vor dem Gebrauch. Lagerung bei 4 ° C für bis zu 1 Monat.
- Planen 24% (w / v) Polyethylenimin (PEI): in 4,8 ml PEI (M w 1.300, 50% (w / w) in H 2 O) auf 5,2 ml ddH 2 O. Verwenden Sie den 24% (w / v) Lösung zu gleichen Volumina vorbereitenvon 16% (w / v), 12% (w / v) und 8% (w / v) PEI-Lösungen. Degas Lösungen durch Ultraschall vor dem Gebrauch. Lagerung bei 4 ° C für bis zu 1 Monat.
- Planen 60% (w / v) Polyethylenglycol (PEG): wiegen 6 g PEG (Mw 600) und in den 10 ml ddH 2 O, das Polymer lösen Pellets durch Vortexen. Verwenden serieller Verdünnung auf ein gleiches Volumen an 30% (w / v) PEG Lösung herzustellen. Degas Lösungen durch Ultraschall vor dem Gebrauch. Lagerung bei 4 ° C für bis zu 1 Monat.
- Bereiten Sie 24% (v / v) N-(3-triethoxysilylpropyl) gluconamid (GLS): add 480 ul GLS (50% in Ethanol - stock GLS die Verwendung von einem Wasserbad zu erwärmen und auflösen Lösung, wenn kristalline verlangen kann), um 520 ul 95% (v / v) Ethanol und Mischen für 20 min mit Ultraschall. Über die 24% (v / v) Lösung zu einem gleichen Volumen von 16% (v / v) GLS Lösung herzustellen. Als frische Lösungen für den Einsatz pro Tag.
- Bereiten Sie 24% (v / v) Methyltrimethoxysilan (MTMS): Fügt 240 ul MTMS bis 760 ul bestehender angesäuert ddH 2 O (pH 2,0, 1 N HCl) und Mix für 20 min durch Sonifikation. Über die 24% (v / v) Lösung zu einem gleichen Volumen von 16% (v / v) MTMS Lösung herzustellen. Als frische Lösungen für den Einsatz pro Tag.
- Bereiten Sie 24% (v / v) bis [(3-methyldimethoxylsilyl) propyl] Polypropylenoxid (MDSPPO): Fügt 240 ul MDSPPO bis 760 ul bestehender angesäuert ddH 2 O (pH 2.0, 1 N HCl) und Mix für 20 min durch Sonifikation. Über die 24% (v / v) Lösung zu einem gleichen Volumen von 16% (v / v) MDSPPO Lösung herzustellen. Als frische Lösungen für den Einsatz pro Tag.
- Bereiten Sie 24% (v / v) 3 - (aminopropyl)-triethoxysilane (APTES): Fügt 240 ul APTES bis 760 ul bestehender angesäuert ddH 2 O (pH 2.0, 1 N HCl) und Mix für 20 min durch Sonifikation. Verwenden Sie den 24% (v / v) Lösung, um ein gleiches Volumen von 16% machen (v / v) APTES Lösung. Als frische Lösungen für den Einsatz pro Tag.
- Planen 24% (v / v) bis (triethoxysily) ethan (bis-TEOS): in 240 ul bis-TEOS zu 760 ul bestehenden angesäuerten ddH 2 O (pH 2,0, 1 N HCl) Und für 20 min durch Sonifikation mischen. Über die 24% (v / v) Lösung zu einem gleichen Volumen von 16% (v / v) Bis-TEOS-Lösung zu machen. Als frische Lösungen für den Einsatz pro Tag.
- Bereiten Sie 24% (v / v) carboxyethylsilanetriol (Si-COOH): 960 ul Si-COOH (25% in ddH 2 O) bis 40 ul der bestehenden angesäuert ddH 2 O (pH 2.0, 1 N HCl) und Mix für 20 min durch Sonifikation. Über die 24% (v / v) Lösung zu einem gleichen Volumen von 16% (v / v) Si-COOH Lösung herzustellen. Als frische Lösungen für den Einsatz pro Tag.
- Getrennt herzustellen 3 mM N ε - Acetyl-L-Lysin, D-Sorbit, α-α-Trehalose (M r 188.22 g / mol, 182.17 g / mol, und 378.33 g / mol) und 1,5 mM Triton X-100 (Mw 625 g / mol) in ddH 2 O. Volumes können je nachdem, wie viel benötigt wird, machen neue Lösungen für den Einsatz pro Tag.
- Bereiten Sie 60% (w / w) Glycerin: add 2,4 g wasserfreies Glycerin zu 1,6 g ddH 2 O, mischen durch Wirbel. Degas Lösungdurch Beschallung vor der Verwendung. Lagerung bei 4 ° C für bis zu 1 Monat.
- Bereiten Sie 30% (w / w) Glycerin: add 1,6 g wasserfreies Glycerin zu 2,4 g ddH 2 O, mischen durch Wirbel. Degas Lösung durch Ultraschall vor dem Gebrauch. Lagerung bei 4 ° C für bis zu 1 Monat.
2. Herstellung von Silica Sols
Die folgenden Schritte ausführen, die entsprechenden Solen, wenn es auf Eis gehalten, bis zu 1 h werden nach Zugabe von Wasser. Sols über 1 Stunde in Folge gesunken / inkonsistent Material Gelierzeiten verwendet.
- Herstellung einer Natrium-Silikat (SS) Sol auf der Basis
- Man wiegt 120 g des Ionenaustauscherharzes in einen 500 ml-Kunststoffbecher.
- In 150 ml 0,1 N HCl und Rühren für 1 h mit einem 2-Zoll-magnetischen Rührstab.
- Filtern Sie die Lösung unter Verwendung eines Büchner-Trichter mit Vakuum.
- Langsam ddH 2 O, um das gefilterte Harz waschen, bis das resultierende Filtrat klar ist. Dies dauert etwa 100ml ddH 2 O.
- Sammeln und Speichern der vorbereiteten Harz in einem Kunststoff-Behälter bei Raumtemperatur bis zu 1 Monat.
- Man wiegt 2,59 g Natriumsilicat (SS, 27% (w / w) SiO 2, 10% (w / w) NaOH) in einen 50 ml-Kunststoffbecher.
- 10 ml ddH2O der SS. Swirl vorsichtig von Hand, um die Lösung zu mischen.
- Abwiegen 5,60 g vorbereitete Harz in einem separaten 50 ml Kunststoffbecher. In der Natriumsilikat-Lösung und Mischung für 2 min mit einem Ein-Zoll-magnetischen Rührstab.
- Das Gemisch wird filtriert Lösung mit einem Büchner-Trichter, der mit einem Sauger an einem Wasserhahn.
- Verwenden Sie eine 10 ml Kunststoff-Spritze mit einer 0,2 um-Membranfilter, um die Sol-Lösung ausgestattet filtern. Daraus ergibt sich ein Sol mit 5,6% (w / w) Kieselsäure genannt SS im weiteren Text. Halten Sie das Sol auf Eis, wenn sie nicht in Gebrauch ist.
- ½ SS: 1 ml der vorbereiteten SS Sol zu 1 ml ddH2O, mischen durch Wirbel. Halten Sie das Sol auf Eis, wenn sie nicht inverwenden.
- Herstellung eines diglycerylsilane (DGS) basierte Sol
- Synthesize DGS als anderswo. 15,16 Shop kristallinen DGS in einem Exsikkator bei Raumtemperatur beschrieben für bis zu 6 Monaten.
- Abwiegen etwa 1 g kristallines DGS.
- Verwenden Sie einen Mörser und Stößel, die DGS zu einem feinen Pulver mahlen. Führen Sie diesen Schritt so schnell wie möglich an Feuchtigkeitsaufnahme aus der Luft zu verhindern.
- Sorgfältig übertragen Sie die fein gemahlenen DGS zu einem leeren 20-ml Szintillationsfläschchen, notieren Sie die Masse (bis zu einem tausendstel Gramm).
- In ddH 2 O, um eine 0,5 g / ml DGS ergeben.
- Beschallen die hydrolysiert DGS auf Eis für 20 min, mischen von Vortex für 5 Sekunden alle 5 min. Während feuchten Tagen kann eine vollständige Auflösung der DGS erfordern die Zugabe von 10 ul 1 N HCl. Dies sollte zu der Lösung vor der Beschallung zugegeben werden.
- Verwenden Sie ein 3-ml Kunststoff-Spritze mit einer 0,2-um-Membranfilter, um die Sol-Lösung filtern ausgestattet, Entfernen von feinen Partikeln. Daraus ergibt sich ein Sol mit 5,0% (w / w) Kieselsäure genannt DGS im weiteren Text. Halten Sie das Sol auf Eis, wenn sie nicht in Gebrauch ist.
- DGS ½: 1 ml der vorbereiteten DGS Sol zu 1 ml ddH2O, mischen durch Wirbel. Halten Sie das Sol auf Eis, wenn sie nicht in Gebrauch ist.
3. Pre-Screening, um die Ursache von bedruckbaren Materialien
Ein verallgemeinertes Schema zeigt die verschiedenen Phasen der geführten Faktor-Analyse verwendete bedruckbaren Materialien zu identifizieren, ist in 1 gezeigt. Ein Minimum gesamten Materials Volumen von 100 ul wird empfohlen. Verwendung von kleineren Volumina macht Visualisierung Material Gelierung schwierig.
- Stufe 1: Puffer und Silan
- In 50 ul Puffer (25 und 50 mM Tris-oder HEPES bei pH 7.0, 7.2, 7.4, 7.6, 7.8, 8.0, 8.2) in ein 2 ml Glasszintillationsröhrchen.
- In 50 ul des entsprechenden Sol (DGS, DGS ½, SS, SS ½) und Vortex Mischung in einem on-und off-Mode für 10 sec durchBewegen Sie das Fläschchen auf und neben dem Vortex-Mischer.
- Set Materialien bei Raumtemperatur ruhen, die Überwachung der Zeit von Material Gelierung. Die Gelierzeit ist die Grenze, bei der das Material frei fließen aufhört beim Drehen der Flasche in einen 45 °-Winkel definiert sind.
- Ausschließen Materialkombinationen mit Gelierzeiten weniger als 2,5 Stunden von den nachfolgenden Material Siebstufen.
- Stufe 2: Puffer, Silan und Polymer
- In einer 2-ml Glasszintillationsröhrchen hinzu 3,13 ul entweder 16% (w / v) oder 8% (w / v) PVA / PEI-Lösung oder 6,3 ul 30% (w / v) oder 60% (w / v) PEG-Lösung.
- In 50 mM HEPES (pH 8,0), um das kombinierte Volumen von 50 ul bringen, Mischen der Lösung durch Wirbel.
- In 50 ul des entsprechenden Sol (DGS, DGS ½, SS, SS ½), mischen in der Mode in Schritt 3.1.2 beschrieben.
- Ausschließen Materialkombinationen mit Gelierzeiten (wie in Schritt 3.1.3 definiert) von weniger als 2,5 Stunden von den nachfolgenden Material Siebstufen.
- Stufe 3: Puffer, Silan, Polymer und Organosilan
- In einer 2-ml Glasszintillationsröhrchen hinzu 3,13 ul entweder 16% (w / v) oder 8% (w / v) PVA-Lösung oder 6,3 ul 30% (w / v) oder 60% (w / v) PEG-Lösung.
- In 3.13 ul 16% (w / v) Organosilan (GLS, MTMS, MDSPPO, Bis-TEOS, Si-COOH oder Aminopropyltriethoxysilan).
- In 50 mM HEPES (pH 8,0), um das kombinierte Volumen von 50 ul bringen, Mischen der Lösung durch Wirbel.
- In 50 ul des entsprechenden Sol (DGS, DGS ½, SS, SS ½) und mischen in der Mode in Schritt 3.1.2 beschrieben. Ausschließen Materialkombinationen mit Gelierzeiten (wie in Schritt 3.1.3 definiert) von weniger als 2,5 Stunden von den nachfolgenden Material Siebstufen.
- Stufe 4: Puffer, Silan, Polymer, Organosilan und kleinen Molekülen additive
- In einer 2-ml Glasszintillationsröhrchen hinzu 2,08 ul entweder 24% (w / v) oder 12% (w / v) PVA-Lösung oder 6,3 ul 30% (w / v) oder 60% (w / v) PEG-Lösung.
- Hinzufügen2,08 ul 24% (w / v) Organosilan (GLS, Si-COOH).
- In 2,08 ul der 3 mM Lösung kleiner Moleküle (N ε - Acetyl-L-Lysin, D-Sorbit, α-α-Trehalose) oder 1,5 mM (Triton X-100).
- In 50 mM HEPES (pH 8,0), um das kombinierte Volumen von 50 ul bringen, Mischen der Lösung durch Wirbel.
- In 50 ul des entsprechenden Sol (DGS, DGS ½, SS, SS ½) und mischen in der Mode in Schritt 3.1.2 beschrieben.
- Ausschließen Materialkombinationen mit Gelierzeiten (wie in Schritt 3.1.3 definiert) von weniger als 2,5 Stunden von den nachfolgenden Material Druckstadien.
4. Herstellung von Druck-Protein-dotierten Materialien
Die Materialien am Ende von Schritt 3.4.6 erkannt werden vorwärts in Bedruckbarkeit Studien durchgeführt, mit dem entsprechenden Enzym nun im Sol enthalten. In allen Fällen unten ausgeführt, wässrigen Lösung, die sämtliche Komponenten mit Ausnahme des Silanwird in einem Mikrotiterplatten vorbereitet, gefolgt von der Zugabe des Sols nur vor dem Drucken. Um die Verwendung eines 384-Mikrotiterplatte aufzunehmen, ist eine Gesamtlösung von 50 ul anstelle von 100 ul verwendet.
- Acetylcholinesterase (AChE)-Microarray
- Bereiten Sie eine 2 kU / ml Lösung von AChE in 50 mM HEPES (pH 8,0) nach der Charge-spezifischen Enzymaktivität (Einheiten der Aktivität pro mg) zur Verfügung gestellten Informationen durch den Hersteller.
- Aliquot 2 kU / ml Enzym-Stammlösung in 5 ul Fraktionen unter Verwendung von 500-ul Reaktionsgefäße und bei -20 ° C 2 kU / ml AChE Lagerlösungen bleiben für bis zu 4 Monaten stabil bei -20 ° C gelagert
- Bereiten Basismaterialien durch die Kombination die Hälfte der Volumina von entsprechenden Polymeren, Organosilane und kleinen Molekülen durch Zusatzstoffe Schritt 3.4.6 in einer 384-well Mikrotiterplatten identifiziert.
- In 5 ul 2 kU / ml AChE in 50 mM HEPES (pH 8,0).
- Unter Verwendung von 50 mM HEPES (pH 8,0), bringen die kombinierteWell-Volumen um 25 ul-Mix durch Pipettieren der Lösung nach oben und unten mehrere Male.
- Multikinase Microarray
- Bereiten 100 ng / ul Kinase-Lösungen (p38a, MAPK2, EGFR, GSK-3β) in ddH2O nach der Aktivität (U / ml bzw. U / mg) und Menge Informationen durch den Hersteller zur Verfügung gestellt. Bewahren Kinase Lösungen in Aliquots von 2 ul bei -80 ° C.
- Bereiten Kinasesubstrate (MBP, p (E 4 Y), GSM) bei 5 mg / ml, 50 mg / ml oder 300 pM in ddH 2 O, nach der Menge Informationen durch den Hersteller zur Verfügung gestellt. Shop Substrate in Aliquots von 2,5 ul bei -80 ° C.
- Bereiten Basismaterialien durch die Kombination die Hälfte der Volumina von entsprechenden Polymeren, Organosilane und kleinen Molekülen durch Zusatzstoffe Schritt 3.4.6 in einer 384-well Mikrotiterplatten identifiziert.
- Zu jedem Well mit den Basismaterialien, fügen Sie 2,5 ul Kinase und 2 ul seiner jeweiligen Substrat (p38a und MBP, MBP und MAPK2, EGFR und p (E 4 Y), GSK-3β und GSM) wie in den Schritten 4.2.1 und 4.2.2 vorbereitet sind.
- Unter Verwendung von 50 mM HEPES (pH 7,4), bringen die kombinierte gut Lautstärke auf 25 ul. Durch Pipettieren gründlich mischen.
5. Microarray Formation
Dieser Abschnitt erklärt die genaue Vorgehensweise für das Drucken Materialien auf einem einzigen Gleitfläche. Um mehrere modifizierte Gleitflächen vorhanden (Amin-, Epoxy-, Aldehyd-und PMMA), ist dieses Verfahren 4 mal wiederholt.
- Formular Microarrays mit einem Kontaktstift-Druck Roboter mit einer XYZ-Bühne ausgestattet. Mit geschlitzten Mantel Stifte von 100 Mikrometer Durchmesser, um die Protein-dotierten Sole hinterlegen.
- Stellen Feuchtigkeit in der Druckkammer auf 80-90%. Luftfeuchtigkeit <80% kann in Probenverdampfung und Ungereimtheiten in Druck, aufgrund der geringen Volumina Abscheidung führen.
- Beschallen Stifte in ddH2O für 15 min vor dem Drucken trocken und unter einem Strom von Stickstoff. Verwenden Sie ein Rohr cleaner auf eine Restfeuchte aus dem Stifthalter entfernen und vorsichtig die Nadel in den Druckkopf. Nichtbeachtung Restfeuchte zu entfernen, kann der Stift sich frei zu bewegen mit dem Halter, was verpasst Flecken zu verhindern.
- Control-Array-Muster durch den Chip Writer Pro (CWP) Programm. Für jede Probe und in der 384-Well-Source-Platte werden 200 Punkte (maximale Anzahl von Punkten pro Aufnahme über die SMP3 Hüllstift) auf einem Oberflächen-modifizierten Glas-Objektträger gedruckt.
- Beginnen Sie den Druckvorgang durch die Software. Set Reise in der XY-Richtung bis 10 mm / s und Probe Anfluggeschwindigkeit (Z-Richtung) bis 2 mm / s mit einem 2,5 sec Probe Ladezeit.
- Hält das Lauf durch CWP. Senken Sie den Stift in die Probe und fügen Sie 25 ul des jeweiligen Sol (DGS, DGS ½, SS, SS ½) an die gut mit der Pipette. Mischen Sie die Lösung mit einem up-and-down Pipettieren Bewegung wiederholt 50x. Beim Mischen durch Pipettieren, verringert sich die Menge von Luft in die Lösung eingebracht. Luftblasen Präventiont kompletten Beladung Probe innerhalb des Stiftes.
- Beginnen Sie den Druckvorgang durch CWP, und drucken Sie die Probe auf einer Gleitfläche. Pause den Druckvorgang vor dem Laden Probe aus der nachfolgenden Source-Platte gut.
- Nehmen Sie den Druck Stift mit Hilfe eines Magneten und legen einen Pfeifenreiniger in den Druckkopf, um die Bildung von Feuchtigkeit in den Druckkopf zu verhindern.
- Spülen Sie den Druck mit Stift ddH 2 O und beschallen in saubere ddH 2 O für 30 Sekunden.
- Trocknen Sie den Druck Stift unter einem Strom von Stickstoff und entfernen Sie den Rohrreiniger, indem Sie den Stift wieder in den Druckkopf.
- Beginnen Druck durch folgende Schritte 5,6-5,10, für alle Proben noch innerhalb der Source-Platte. Bis zu 12.000 Stellen von 100 Mikrometer Durchmesser können auf einem einzigen Objektträger aufgebracht werden kann.
- Dieses Verfahren kann auch auf Bedruckstoffe auf den Boden der Wells einer Mikroplatte mit 96 Vertiefungen aufgebracht werden. Nach der Kalibrierung CWP-Datei, kalibrieren Sie den Stift, um den Druck zu gewährleisten Distanzence reiste zwischen Kassa-Abscheidung ist über die Höhe der Mikrotiterplatte. Dies ermöglicht der Stift konsequent ausdrucken von Brunnen zu Brunnen in einer linearen Weise ohne Beschädigung der Pin.
- Age das Array für mindestens 30 min und bis zu 24 Stunden in der Druckkammer bei 80-90% Luftfeuchtigkeit nach Abschluss der gesamten Druckversuch.
6. Acetylcholinesterase-Aktivität Assay
- Vorbereiten positive Kontrolle (PC) Proben
- Bereiten 1 mM Acetylthiocholinjodid (ATCH: M r 289.18 g / mol) in 4% Glycerin, 25 mM Tris (pH 7,0) in einem 1,5-ml-Röhrchen. ATCH Bedürfnisse bereit vor jedem Gebrauch frisch und mit einem Puffer bei pH 7.0 als höhere pH-Puffer schnelle Autohydrolyse verursachen, wodurch Fehlalarme. Das Endvolumen kann in Abhängigkeit von der Anzahl der Proben (25 ul pro Probe) werden.
- In 0,14 ul 5 mM bodipy-Fl-L-Cystin zu 25 ul 1 mM in die Vertiefung einer 384-Mikrotiterplatten pla ATCHte.
- In 24,86 ul 4% Glycerin, 25 mM Tris (pH 7,0); Mix durch Pipettieren sorgfältig, um eine Blasenbildung in der Lösung zu vermeiden. Potenzielle Enzym-Inhibitoren können in den PC-Lösung, bevor sie die Lautstärke auf 50 ul mit Puffer eingebaut werden.
- Vorbereiten negative Kontrolle (NC) Proben
- In 0,14 ul 5 mM Bodipy-FL-L-Cystin 49,86 ul 4% Glycerin, 25 mM Tris (pH 6,5) in die Vertiefung einer 384-Mikrotiterplatte. Durch Pipettieren gründlich mischen.
- Überdrucken PC und NC-Lösungen
- Überdrucken die Alters-Microarrays folgenden Schritte 5,1-5,10 (ignorieren Schritt 5.6) des Protokolls. Mit geschlitzten Mantel Stifte von 235 Mikrometer Durchmesser, um den PC und NC Überdrucken Lösungen hinterlegen. Dies gewährleistet die Lösung umfasst die zuvor abgeschiedenen Stelle vollständig.
- Alter Arrays bei 80-90% Luftfeuchtigkeit für 1 Stunde bei Raumtemperatur. Aufgrund der Autohydrolyse ATCH kann längere Inkubationszeiten in Fehlalarmen führen oder erhöht enEnzym-Aktivität.
7. Kinaseaktivitätsassay
- Bereiten 500 uM Adenosin-5'-Triphosphat (ATP)-Lösung unter Verwendung stock ATP (100 mM) bei -20 ° C gelagert Als frische Lösung für jedes Experiment und die Lautstärke anpassen, um experimentelle Notwendigkeit: Jeder Drucklösung erfordert 5 ul.
- Bereiten Sie eine 100 mM Magnesiumchlorid (MgCl 2: M r 95,21 g / mol) Stammlösung in ddH 2 O.
- Positive Kontrolle (PC)
- In 2,5 ul 100 mM MgCl 2 und 5 ul 500 uM ATP zur Vertiefung einer 384-Mikrotiterplatten.
- Bringen Sie die insgesamt gut ein Volumen von 50 ul mit 50 mM HEPES (pH 7,4); durch Pipettieren gründlich mischen. Potenzielle Enzym-Inhibitoren können in den PC-Lösung, bevor sie die Lautstärke auf 50 ul mit Puffer eingebaut werden.
- Negative Kontrolle (NC)
- In 2,5 ul 100 mM MgCl 2 und 5 ul ddH2O zur Vertiefung einer 384-microtiter Platte.
- Bringen Sie die insgesamt gut ein Volumen von 50 ul mit 50 mM HEPES (pH 7,4) und durch Pipettieren gründlich mischen.
- Überdrucken PC und NC-Assay Cofaktoren
- Führen Sie Schritt 6.3.1 des Protokolls.
- Alter Arrays bei 80-90% Feuchtigkeit für 2 Stunden bei Raumtemperatur.
- Slide Färbung
- Platz gedruckt Microarray gleitet einzeln in einer Petrischale mit genügend Farbstoff (aus dem Kit), um die gesamte Folie abdecken. Schütteln mäßig (~ 200 rpm) für 45 min mit einem Schüttler.
- Entfernen Sie die Folie mit einer Pinzette und in einen sauberen Petrischale mit Waschpuffer (aus dem Kit). Schütteln mäßig (~ 200 rpm) für 45 min mit einem Schüttler.
- Entfernen Sie die Folie mit einer Pinzette und schleudern mit einem herkömmlichen Desktop-Microarray Mikrozentrifuge mit einer Rutsche ausgestattet Inhaber.
8. Microarray Imaging and Analysis
- Imaging
Beachten Sie, dass das bildgebende Verfahren wird s seinpecific um den Typ von Array-Reader verfügen. Für diese Untersuchungen ein Alpha Innotech NovaRay Imager mit einer weißen Lichtquelle und CCD-Detektionssystem mit einem 478 ± 17 nm Anregungs-und 538 ± 21 nm Emission Filter für die AChE-Mikroarrays und 530 ± 40 nm Anregungs-und 614 ± 62 nm Emission ausgestattet Filter für Kinase-Microarrays verwendet wurde. Konfokalen Laser-Scanning-Systeme können auch für das Lesen der Arrays verwendet werden, obwohl die Einstellungen werden speziell für das Instrument.
- Legen Sie gleiten in den Dia-Halter mit den Punkten nach oben zeigt.
- Legen Sie die Anzahl der Vorschaubilder Abschnitte (ein Minimum von 2 wird empfohlen, eine an jedem Ende der Objektträger), Auflösung (mindestens 4 um wird empfohlen) und Belichtung (Auto empfohlen) innerhalb der Imaging-Software.
- Erwerben slide Bild und speichern als ". Tiff"-Datei.
- Analyse
- Öffnen erworben slide image in ImageJ64.
- Klicken Sie auf das Auswahl-Werkzeug oval und messen dieSignalintensität jeden Spot mit der Maßnahme Option unter der Registerkarte Analysieren. Bei der Auswahl der Fläche zu messen, wählen Sie eine Region geringfügig größer als die beobachtete Stelle und einheitliche Größe unter Flecken ImageJ Subjektivität zu reduzieren.
- Durchschnittliche die Intensität von 25 ähnlichen PC-Spots, die von einem durchschnittlichen Intensität von 25 ähnlichen NC Flecken geteilt, um PC / NC-Verhältnisse für die einzelnen Material-Zusammensetzungen zu erhalten.
Ergebnisse
Durch die Durchführung einer geführten Faktor für das Material-Bildschirm, konnten wir die Anzahl der getesteten Materialien von ~ 20.000 bis ein paar hundert, die Gelierzeiten geeignet für den Druck hatte minimieren. Durch die Anwendung eines strengen Richtlinien erfordern Material Gelbildungszeit von 2,5 Stunden oder mehr, wurden Materialien, die die Druckstiften verstopfen oder produzieren nicht reproduzierbaren Arrays nie gedruckt. Die bedruckbaren Materialien identifiziert, um eine ausreichende (> 2,5 h) Gelierzeiten haben wurden auf 4 verschiedenen funktionalisierten Glasträger Oberflächen gedruckt. Um als "druckbaren" zu werden, musste die maximale Anzahl von Punkten pro Aufnahme Volumen des Stiftes zu bedruckenden (SMP3 = 200) werden. Spots wurden auch für Spotmorphologie bewertet, um sicherzustellen, dass keine Rissbildung oder unerwünschte Phasentrennung eingetreten mit einfachen Hellfeld, wie in 2 gezeigt.
Von diesem Stadium identifiziert bedruckbaren Materialien wurden Microarrays mit AChE produziert undKinasen in die gepufferte wässrige Komponente eingearbeitet. Materialien, die mit dem Assay-Verfahren (einschließlich der möglichen Überdruck und Waschen oder Färben Stufen) waren, wurden durch Beobachtung Beibehaltung der Mikroarray Spots (keine Rissbildung, Verlust von Flecken oder ungewöhnliche Fluoreszenzmuster) und eine positive Kontrolle (PC) Negativkontrolle (NC identifiziert ) von größer als 1, da durch Bild beobachtet. Da es sich um etwa 50% der Materialien, eine größere PC / NC-Verhältnis von 3 wurde verwendet, um optimale Materialien unter Beibehaltung der Protein-Aktivität definieren. Durch diese Methode ist, zeigen 26 Sol-Gel-abgeleiteten Materialien, die AChE und 2 enthaltenden Materialien Kinasen zufrieden die> 3 PC / NC Kriterien. Abbildung 3 und Abbildung 4 eine grafische Aufschlüsselung der 5 geführte Material angezeigten Schritte zur Identifizierung der optimalen AChE und Kinase Mikroarrays sind.
Die Assays können auch durch die Erzeugung eines Z 'Ergebnis überprüft werden. 17 Dies geschah mit dem Material, das die höchste PC / NC Verhältnis hergestellt. Abbildung 5 zeigt die Z 'Grundstück durch den Vergleich des Signals von 200 Punkten, 100 PC und 100 NC nach Überdrucken die Indikator-Farbstoff und Substrat auf der AChE Array erzeugt wird, erhalten. Der Schmerz und die Kinase Arrays führte in den jeweiligen Z 'Noten von 0,60 und 0,67, was auf eine ausgezeichnete Test. Es sollte jedoch beachtet werden, dass vor dem Assay-Validierung, on-Array Enzym, Farbstoff, Substrat und Cofaktor Konzentrationen durch Überdrucken einen Bereich von Konzentrationen der einzelnen Komponenten und Auswählen der Konzentration, die die höchste Signalqualität produziert optimiert werden, wie ausführlich an anderer Stelle beschrieben werden musste . 5
Um die Tests zu validieren, wurden quantitative Hemmung erhaltenen Daten mit bekannten und unbekannten AChE-Inhibitoren, wobei die Ergebnisse in doppelte durchgeführt und verwendet, um doppelte Grundstücke (6A) und Hemmkurven (6B und 6C) zu erzeugen. Spots wurden zunächst mit Mischungen von bekannten biologisch aktiven niedermolekularen Inhibitoren dann mit Farbstoff und Substrat überdruckt und Kontrolle Mischungen, die entweder bekannt oder Inhibitoren keinen Inhibitor enthalten waren. Doppelte Parzellen wurden erzeugt, um die Enzymaktivität zu beurteilen, und beliebige Mischungen, die in weniger als 25% Enzymaktivität führte wurden als positiv für die Hemmung. Einzelne Verbindungen aus solchen Mischungen wurden dann in zweifacher Ausfertigung an die spezifische kleines Molekül (en), die für die Hemmung identifizieren getestet. Einmal identifiziert, diese kleinen Moleküle wurden verwendet, um quantitative Hemmkurven generieren IC 50-Werte und Hemmkonstanten bestimmen.
Ähnliche qualitative Ergebnisse wurden mit dem Multi-Kinase-Array mit einer gemeinsamen Kinase-Inhibitor, Staurosporin. 7A und 7B zeigen die Microarray-Bild und zeigen, dass die Signalintensitäten nach Überdrucken und Färben der multikinase(- ATP), Positivkontrolle (+ ATP) und bekannter Inhibitor (+ ATP + inh) Anordnung wie für eine negative Kontrolle erwartet. Um die Fähigkeit zur Hemmung quantitative Daten aus den Microarrays erhalten zu demonstrieren, wurde eine konzentrationsabhängige Hemmung Assay für eine Kinase durchgeführt. Wie in den 8A und 8B gezeigt, nimmt Signalintensität Inhibitorkonzentration zu, und die Reaktion folgt der erwarteten Konzentration Hemmung Kurve für die p38α/MBP Kinase / Substrat-System.
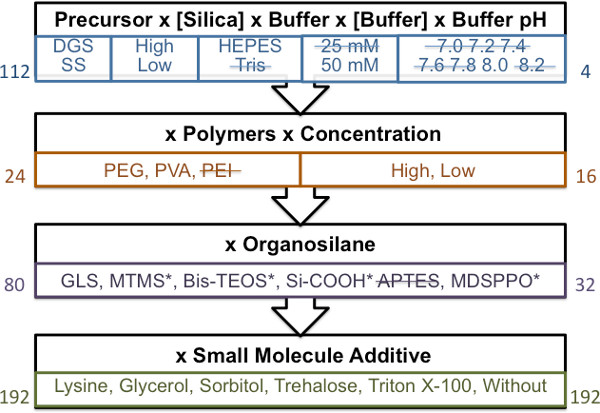
Abbildung 1. Allgemeines Schema für die geführte Materialien Screening-Ansatz. Jeder Block stellt einen Schritt auf dem Bildschirm in der angegebenen Reihenfolge. Die Zahlen auf der linken Seite stellen die Anzahl von Materialien für die Analyse vorbereitet. Mit einem Gelierdauer größer als 2,5 h (Materialien mit Gelierzeiten weniger als 2,5 h are, die durch die durchgestrichen), wird die Anzahl der Materialien, die jede Stufe weitergegeben und vorgetragen während der Material-Bildschirm durch die Zahl auf der rechten Seite angegeben. * Für Materialien mit weniger als optimale Phasentrennung.

Abbildung 2. Optische Bilder mit verschiedenen Störungsarten Materialien an der Bedruckbarkeit Schritt des Bildschirms. Ein Bild von einem "guten" Material (zweite Reihe, dritte Spalte) wird ebenfalls zum Vergleich gezeigt. Nachdruck mit freundlicher Genehmigung aus Lit. 8, Copyright 2013 American Chemical Society.
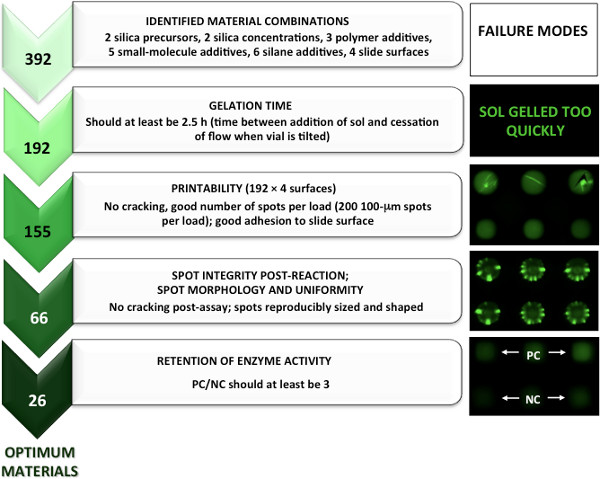
Abbildung 3. Ein gerichteter Material Screening-Ansatz für die Identifizierung der optimalen Materialien für die Herstellung von Sol-Gel-abgeleiteten AChE Microarrays. Dauerwelle mit Nachdruckission von Referenz 5, Copyright 2013 American Chemical Society.

Abbildung 4. Ein gerichteter Materialien Screening-Ansatz für die Identifizierung der optimalen Materialien für die Herstellung von Sol-Gel-abgeleiteten Kinase-Microarrays. Mit Genehmigung aus Lit. 8, Copyright 2013 American Chemical Society Nachdruck.
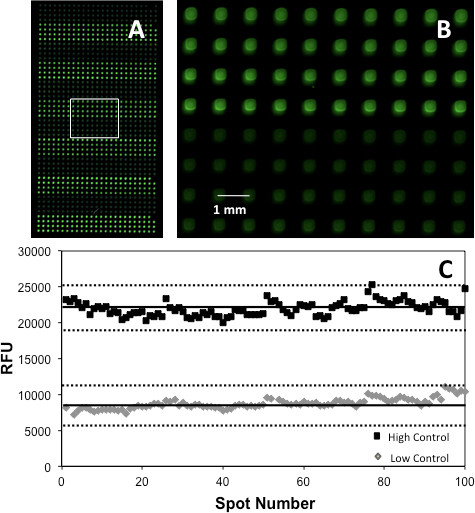
Abbildung 5. (A) Ein Teil der AChE Microarray zeigt HC (hellgrün) und LC (hellgrün) Spots (ein schwarz-grün-Palette wurde als Pseudocolor für die Klarheit der Präsentation angewendet), (B) eine vergrößerte Ansicht der boxed Bereich zu markieren Ort Morphologie und Ausrichtung;. und (c) eine Z 'plot durchgezogenen Linien zeigen den Mittelwert der repliziert, während gestrichelte Linien zu 3SD entsprechen. Nachdruck mit freundlicher Genehmigung aus Lit. 5, Copyright 2013 American Chemical Society.
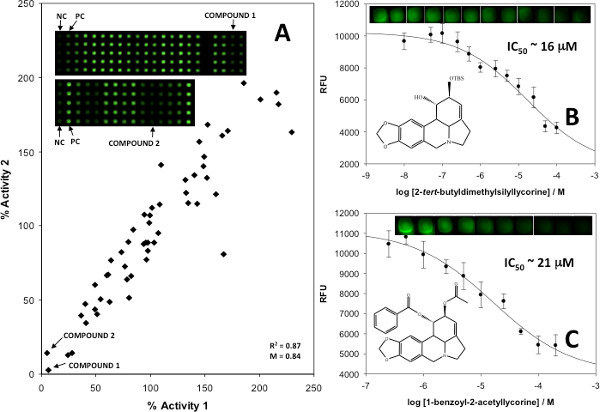
Abbildung 6. (A) Duplizieren Grundstück für on-Array-Screening von synthetischen Analoga von Amaryllidaceae Alkaloide; (B) IC 50 Stellplätze der identifizierten potentiellen Inhibitoren als Verbindungen 1 und (C) Verbindung 2, mit Fehlerbalken repräsentieren eine Standardabweichung des Mittelwertes von 25 markiert repliziert. Repräsentative Spots gezeigt werden, um Unterschiede in proportionales Signal Inhibitorkonzentrationen illustrieren. Nachdruck mit freundlicher Genehmigung aus Lit. 5, Copyright 2013 American Chemical Society. Klicke hier, um eine größere Abbildung anzuzeigen .
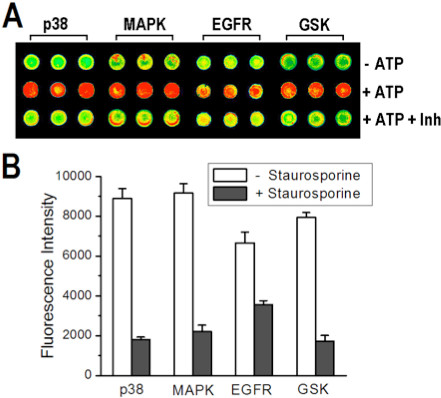
Abbildung 7. On-Array-Assay unter Verwendung von vier Kinasen 1.4SS/1.0PVA zum Mitreißen und gedruckt an ein Amin-derivatisierten Folie. (A) Ein Bild von einem Abschnitt der Mikroarray in denen Punkte mit Kinasen mit ihren jeweiligen Substraten zusammen mit Puffer eingeschlossen wurden überdruckt (NC, obere Reihe), oder Lösungen, die ATP (PC, mittlere Reihe) oder ATP + Staurosporin (untere Reihe). (B) Balkendiagramme Vergleich Signalintensitäten zwischen gesperrt und ungehemmt Reaktionen, nach Abzug der Hintergrund-Signale und Fehlerbalken repräsentieren eine Standardabweichung des Mittelwertes von 25 Wiederholungen. Nachdruck mit freundlicher Genehmigung aus Lit. 8, Copyright 2013 American Chemical Society.
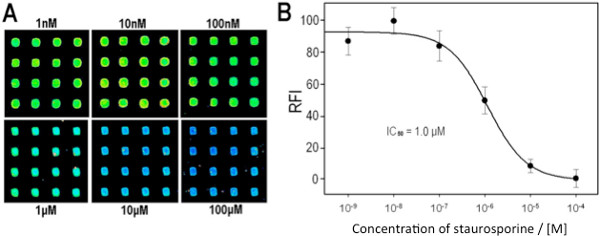
Abbildung 8. Inhibiti. auf Assay auf einem Mikroarray p38a/MBP (A) Sektionen von Mikroarrays, welche repräsentative Punkte overspotted mit variierenden Konzentrationen von Staurosporin, wie angegeben (die Bilder wurden von einer einzigen Abtastung der gleichen Folie erhalten wird; zusammengesetzte Bild wird zur Klarheit gezeigt). (B) 50 IC-Kurve aus den analysierten Array Bilder erzeugt. Die Intensität bei 100 mM erhalten wurde aus allen Bildern subtrahiert, alle anderen Intensitäten wurden durch Einstellen der Intensität bei 10 nM auf einen Wert von 100% der Aktivität erhalten normalisiert. Nachdruck mit freundlicher Genehmigung aus Lit. 8, Copyright 2013 American Chemical Society.
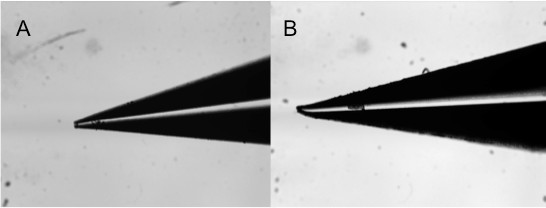
Abbildung 9. Bilder von mikroskopischen Stealth-Stift für Kontaktstift-Druck mit verschiedenen Unvollkommenheiten verwendet: (A) verstopft ist, (B) gebogen.
Diskussion
Die Methodik hier beschrieben wurde als die am besten geeignete für die Identifizierung druckbare Sol-Gel-Materialien abgeleitet mit einem Kontakt Drucker ausgewählt, wodurch ein zeit-und kostengünstige Verfahren zur schnellen Identifizierung optimalen Materialien, ohne eine große Zahl von Materialien zu screenen. Von den insgesamt 20.000 ~ potenziellen Materialien, war es möglich, ~ 200 Materialien, die für den Druck auf der Grundlage Gelierdauer allein waren zu identifizieren. Diese deutlich die Anzahl der benötigten Materialien, um für nachfolgende Druckversuche hergestellt werden reduziert. Diese bedruckbaren Materialien wurden dann auf 4 Gleitflächen für insgesamt 768 Material-Gleitpaarungen gedruckt. Im Durchschnitt können 50 Punkte / Replikate von einem Material in ~ 3 min, einschließlich Probenbeladung, Spot Abscheidung und Reinigung Stift gedruckt werden. Von diesen 155 Materialien oder etwa 20%, für den Druck der maximalen Anzahl von Punkten pro Lösungsaufnahme erlaubt und reproduzierbar hergestellt Punktgrößen. Es sollte angemerkt werden, dass der 4 slide Oberflächen getestet, gedruckte Materialien besser in der Reihenfolge: Amin> Epoxidharz> Aldehyd> PMMA, PMMA Folien nicht produzieren nützliche Arrays für alle Materialien. Dies war wahrscheinlich auf die Polarität der Oberflächenbeschichtung zugeschrieben. Vergleichen der genannten Gleitflächen wurden das polarere Amin und Epoxid besser für die wässrigen Sole im Vergleich zu den PMMA Folien geeignet. Außerdem der getesteten Oberflächen bieten die Amin-beschichtete Objektträger ein Potential positiv geladenen Oberfläche der abgeschiedenen anionischen Sol zu binden. Wir vermuten, dass die Nanopartikel Siliciumdioxid an der Grenzfläche zwischen dem Schieber und das Sol auf der Oberfläche interagieren. Sowohl die Epoxy-und die Aldehyd Gleitflächen fehlt die gleiche anfängliche Lade-Interaktion. Um eine optimale Ort Abscheidung zu gewährleisten ist es sehr empfehlenswert, um pre-beschichtete Objektträger von einem Anbieter wie Arrayit verwenden. In-house Beschichtung erzeugt inkonsistente Flächen, die zu schlechter Reproduzierbarkeit Spot 13 und führen, in einigen Fällen kann eine Quantifizierung prob führenbleme. 18. Von gleicher Bedeutung, Einfluss auf Temperatur und Luftfeuchtigkeit die "Bedruckbarkeit" der Materialien. Während keine detaillierten Studien zu den Auswirkungen im Zusammenhang mit Temperatur durchgeführt wurden, wurde das Drucken immer bei Raumtemperatur (23 ± 3 ° C). Feuchtigkeit (mehr als 80%) wurde auch in der Kammer Druck gesteuert wird, um unregelmäßige Form Ablagerung aufgrund der geringen Volumina Abscheidung (0,7-2,3 nl) und eine Verdampfung zu verhindern.
Während das Material Bildschirm wurde zur Identifizierung optimaler Sol-Gel-abgeleitete Materialien, die speziell zum Drucken von AChE und Kinasen, eine kleine Reihe von Materialien identifiziert, die für beide Proteine gearbeitet geführt. Tatsächlich wurden beide Materialien, die für die Kinase-Mikroarray Herstellung identifiziert wurden SS + PVA + Basis Glycerin, und beide Materialien wurden in den 26 Materialien für AChE Microarrays ausgewählt identifiziert. Diese "optimal" Materialien können eine generische Ausgangspunkt für die Entwicklung weiterer prote bietenin-dotierten Sol-Gel-basierten Microarrays und kleine Bildschirme um diesen Zusammensetzungen zentriert identifizieren können noch bessere Werkstoffe für Microarray-Herstellung. Ein zweiter Punkt ist die Bedeutung des Enzyms verwendet. Im Falle von AChE (a ziemlich robust Enzym), gehalten 26 (oder etwa 40%) der ursprünglichen 66 Materialien als Assay-kompatibel identifiziert die Aktivität des eingeschlossenen AChE. Doch für die zarteren Kinasen, waren nur 2 der 69 Test-kompatiblen Zusammensetzungen oder rund 3% der Materialien in der Lage, die Aktivität aller Kinasen zu behalten. Während eine ausreichende Zahl von verschiedenen Enzymen nicht studiert haben, um schlüssige Aussagen zu machen, scheint es, dass die Optimierung Arrayherstellung mit relativ instabil Enzyme können zur Identifizierung der Materialien, die eine breite Palette von Proteinen an mutliplexed Microarray Herstellung erlauben einzufangen führen kann.
Unabhängig von der gewählten Proteins wurde die große cut-off Faktor zur Identifizierung bedruckbaren Materialien die Notwendigkeit für eine langeMaterial Gelierzeiten (> 2,5 h). Bei der Entwicklung SS Sol-Gel-Materialien, ist es sehr wichtig, um sicherzustellen, dass nach Ionenaustausch und Filtration wurde das Sol bei etwa pH 4 ist. Sols mit einem geringeren anfänglichen pH-Wert kann in Materialien mit einer geringeren als den neutralen pH-Wert, die Enzymaktivität beeinflussen können. 19 Einstellen der Menge an Dowex (Ionenaustauscher) zu SS kann die endgültige pH des Sol verändern führen. Wenn eine neue Charge des Harzes wird das Verhältnis von Harz zu SS muss so eingestellt werden, dass Sole bei etwa pH 4 nach dem Verfahren in Abschnitt 2 des Protokolls zu produzieren.
Ebenso ist die Herstellung von kristallinen DGS oft eine Fehlerquelle mit Materialversagen verbunden, wenn mit DGS based Sole für das Biomolekül Einschluss. Obwohl hier nicht im Detail angegeben, muss große Sorgfalt, um während der Synthese des kristallinen DGS, insbesondere die Notwendigkeit, die Anwesenheit von Wasser während der Synthese zu vermeiden, die polyglycerated Siliciumdioxid produzieren kann entnommen werdentes statt monomere DGS. Auch aufgrund der hygroskopischen Natur der DGS, muss die kristalline Probe zu speichernden ausgetrocknet werden und innerhalb von 6 Monaten nach der Synthese. Kristalline DGS älter als 6 Monate nicht vollständig auflösen (wegen teilweise kondensierten Polyglyceryl Silikat-Material), auch mit Ultraschall in einer sauren Umgebung. Unvollständige DGS Auflösung produziert Solen mit unbekannten und unkontrollierbaren Silica-Anteil und damit weniger robusten Materialien.
Ein wichtiger Punkt bei Kontaktdruck beachten ist die Qualität der Stifte. Beschädigte oder misshandelt Stifte (Abbildung 9) wird nie reproduzierbar Arrays unabhängig von dem Material gedruckt wird. Es wird empfohlen, um den Stift mit einem Qualitäts-Check Dissektionsmikroskop um sicherzustellen, gebrochen oder verstopft Stifte nicht verwendet werden. Sorgfältige Behandlung garantiert eine lange Lebensdauer für die Stifte. Freie Bewegung des Stifts ist ebenfalls wichtig. In Fällen, in denen Feuchtigkeit in dem Druckkopf zwischen dem Kopf und der Stift der Stift eingeschlossen ist,er wird dann nicht vollständig und somit nicht guten Kontakt mit der Oberfläche, was zu einem Mangel der Abscheidung von Material.
Zusammenfassend haben wir einen detaillierten, mehrstufigen Screening-Ansatz für die Entwicklung von High-Density-Protein-dotierten pin gedruckten Microarrays zur Verfügung gestellt. Das Screening beinhaltet die Optimierung der Materialeigenschaften (Gelierdauer und Bedruckbarkeit), damit Druck von Materialien, um mehr konzentriert Screening gefolgt, um Materialien, die mit einem gegebenen Assay und in der Lage, um die Enzymaktivität zu behalten sind zu identifizieren. Diese geführte Material Screening-Ansatz können zusätzliche Microarray-Formate angewendet werden, um Zeit und Kosten mit der Herstellung von effizienten High-Density-Microarrays zu reduzieren.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Die Autoren danken Maria Monton, Julie Lebert, Jessamyn Kleine, Xin Ge und Laura Lautens für die Unterstützung bei der Entwicklung von Protein-Microarrays. Die Autoren danken dem Natural Sciences and Engineering Research Council of Canada (NSERC) für die Finanzierung dieser Arbeit. Die Autoren danken auch dem Canada Foundation for Innovation und dem Ontario Innovation Trust für die Unterstützung dieser Arbeit. JDB hält den Canada Research Chair in Bioanalytik und Biointerfaces.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Poly(vinyl alcohol) (PVA) | Sigma-Alderich | 360627 | 80% hydrolozyed, Mw 600 |
| Polyethylene glycol 600 (PEG) | Sigma-Alderich | 87333 | |
| Polyethylenimine (PEI) | Sigma-Alderich | 482595 | 50 wt% solution in water |
| Carboxyethylsilanetriol (Si-COOH) | Gelest, Inc. | SIC2263.0 | 25% in water |
| N-(3-triethoxysilylpropyl) gluconamide(GLS) | Gelest, Inc. | SIT8189.0 | 50% in ethanol |
| bis[(3-methyldimethoxysilyl)propyl]polypropylene oxide (MDSPPO) | Gelest, Inc. | SIB1660.0 | |
| Methyltrimethoxysilane (MTMS) | Gelest, Inc. | SIM6560.1 | |
| Bis(triethoxysiyly)ethane (Bis-TEOS) | Gelest, Inc. | SIB1817.0 | |
| 3-Aminopropyltriethoxysilane (APTES) | Gelest, Inc. | SIA0610.0 | |
| Glycerol | Sigma-Alderich | 49767 | |
| D-Sorbitol | Sigma-Alderich | 240850 | |
| D-(+)-Trehalose dihydrate | Sigma-Alderich | T9531 | |
| Triton X-100 | Sigma-Alderich | X-100 | |
| Nε-Acetyl-L-lysine | Sigma-Alderich | A4021 | |
| Tris(hydroxymethyl)aminomethane | Sigma-Alderich | 154563 | |
| HEPES | Sigma-Alderich | H3375 | |
| Sodium hydroxide, 1.0 N | LabChem Inc. | LC24350-2 | |
| Hydrochloric Acid, 1.0 N/0.1 N | LabChem Inc. | LC15300-2/LC152220-2 | |
| Magnesium chloride | Sigma-Alderich | M8266 | |
| Diglycerolsilane (DGS) | Prepared in laboratory | ||
| Sodium silicate solution | Fisher Scientific | SS338-1 | |
| Dowex 50WX8-100 ion exchance resin | Sigma-Alderich | 217492 | |
| Acetylthiocholine iodide | Sigma-Alderich | 1480 | |
| Acetylcholinesterase from Electrophorus electricus (electric eel) | Sigma-Alderich | C2888 | |
| BODIPY FL L-Cystine | Invitrogen | B-20340 | |
| Pro-Q Diamond Phosphoprotein/Phosphopeptide Microarray Stain Kit | Invitrogen | P33706 | |
| Adenosine 5'triphosphate disodium salt (ATP) solution | Sigma-Alderich | A6559 | |
| MAP Kinase 2 (MAPK2) | EMD Millipore | 454850 | |
| p38α/SAPK2a (T106M), active | EMD Millipore | 14-687M | |
| Epidermal growth factor (EGFR) | EMD Millipore | Donated by Millipore | |
| Glycogen synthase kinase 3β (GSK-3β) | EMD Millipore | 14-306 | |
| Myelin basic protein (MBP) | EMD Millipore | Substrate for MAPK2 and p38α, Donated by Millipore | |
| GSM | EMD Millipore | 12-533 | Substrate for GSK-3β |
| Poly-glu-tyr polypeptide p(E4Y) | EMD Millipore | 12-440 | Substrate for EGFR |
| Stealth pin | ArrayIt | SMP3 | |
| Stealth pin | ArrayIt | SMP7 | |
| Amine coated slides | ArrayIt | SMM2 | |
| Aldehyde coated slides | ArrayIt | SMA2 | |
| Exposy coated slides | ArrayIt | SME2 | |
| Poly(methylmethacrylate) (PMMA) coated slides | Exakt Technologies Inc. | 41500 | |
| 0.2-μm syringe filter | PALL Life Sciences | 4612 | |
| Equipment | |||
| Virtek Contact Printer | BioRad | ||
| Novaray Fluorescence Slide Imager | Alpha Innotech Corporation | ||
| Desktop microarray centrifuge | ArrayIt | MHC110V | |
| MilliQ Synthesis A10 | Millipore | Used to filter all water required for experiments |
Referenzen
- Schena, M. M., Shalon, D. D., Davis, R. W. R., Brown, P. O. P. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science (New York, N.Y.). 270 (5235), 467-470 (1995).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science (New York, N.Y.). 289 (5485), 1760-1763 (2000).
- Zhu, H. H., Bilgin, M. M., et al. Global analysis of protein activities using proteome chips. Science (New York, N.Y.). 293 (5537), 2101-2105 (2001).
- Hook, A. L., Chang, C. -. Y., et al. Combinatorial discovery of polymers resistant to bacterial attachment. Nature Biotechnology. , (2012).
- Monton, M. R. N., Lebert, J. M., Little, J. R. L., Nair, J. J., McNulty, J., Brennan, J. D. A Sol-Gel-Derived Acetylcholinesterase Microarray for Nanovolume Small-Molecule Screening. Analytical Chemistry. 82 (22), 9365-9373 (2010).
- Cho, E. J., Tao, Z., Tehan, E. C., Bright, F. V. Multianalyte pin-printed biosensor arrays based on protein-doped xerogels. Analytical Chemistry. 74 (24), 6177-6184 (2002).
- Cho, E. J., Tao, Z., et al. Tools to Rapidly Produce and Screen Biodegradable Polymer and Sol-Gel-Derived Xerogel Formulations. Applied Spectroscopy. Society for Applied Spectroscopy. 56 (11), 1385-1389 (2002).
- Ge, X., Lebert, J. M., Monton, M. R. N., Lautens, L. L., Brennan, J. D. Materials Screening for Sol-Gel-Derived High-Density Multi-Kinase Microarrays. Chemistry of Materials. 23 (16), 3685-3691 (2011).
- Rupcich, N., Green, J. R. A., Brennan, J. D. Nanovolume Kinase Inhibition Assay Using a Sol-Gel-Derived Multicomponent Microarray. Analytical Chemistry. 77 (24), 8013-8019 (2005).
- Rupcich, N. Coupled enzyme reaction microarrays based on pin-printing of sol-gel derived biomaterials. Analytica Chimica Acta. 500 (1-2), 3-12 (2003).
- MacBeath, G. G., Schreiber, S. L. S. Printing proteins as microarrays for high-throughput function determination. Science (New York, N.Y.). 289 (5485), 1760-1763 (2000).
- Stillman, B. A. B., Tonkinson, J. L. J. FAST slides: a novel surface for microarrays. BioTechniques. 29 (3), 630-635 (2000).
- Rupcich, N., Goldstein, A., Brennan, J. D. Optimization of sol-gel formulations and surface treatments for the development of pin-printed protein microarrays. Chemistry of Materials. 15 (9), 1803-1811 (2003).
- Gill, I., Ballesteros, A. Encapsulation of biologicals within silicate, siloxane, and hybrid sol-gel polymers: An efficient and generic approach. Journal of the American Chemical Society. 120 (34), 8587-8598 (1998).
- Brook, M. A., Chen, Y., et al. Proteins entrapped in silica monoliths prepared from glyceroxysilanes. Journal of Sol-gel Science and Technology. 31 (1), 343-348 (2004).
- Brook, M. A., Chen, Y., Guo, K., Zhang, Z., Brennan, J. D. Sugar-modified silanes: precursors for silica monoliths. Journal of Materials Chemistry. 14 (9), 1469 (2004).
- Zhang, J., Chung, T., Oldenburg, K. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- . Next generation of protein microarray support materials: Evaluation for protein and antibody microarray applications. Journal of Chromatography A. , 1-8 (2013).
- Bhatia, R. B., Brinker, C. J., Gupta, A. K., Singh, A. K. Aqueous Sol-Gel Process for Protein Encapsulation. Chemistry of Materials. 12 (8), 2434-2441 (2000).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten