Method Article
A Materials Guided Approach Triagem de Desenvolvimento de quantitativos Sol-gel derivados de microarrays de proteína
Neste Artigo
Resumo
Uma abordagem de triagem de material guiada para desenvolver sol-gel de proteína derivadas de microarrays dopados, utilizando um método de fabrico do pino-impressão emergente é descrito. Esta metodologia é demonstrada através do desenvolvimento de acetilcolinesterase e multiquinase microarrays, que são utilizados para rentável de pequenas moléculas de triagem.
Resumo
Microarrays encontraram utilização no desenvolvimento de ensaios de alto rendimento para a descoberta de novos materiais e de pequenas moléculas de droga leva. Descrevemos aqui uma abordagem de triagem de material guiada para identificar materiais à base de sol-gel, que são adequados para a produção de microarrays de proteína em três dimensões. A primeira abordagem identifica os materiais que podem ser impressas como microarrays, se reduz o número de materiais, identificando aqueles que são compatíveis com um determinado ensaio enzimático e afia em em materiais ideais com base na retenção de atividade máxima da enzima. Esta abordagem é aplicada para desenvolver microarrays adequados para dois ensaios diferentes de enzimas, utilizando uma enzima acetilcolinesterase e o outro utilizando um conjunto de quatro cinases-chave envolvidas no cancro. Em cada caso, foi possível produzir microarrays que poderiam ser usados para ensaios quantitativos de rastreio de pequenas moléculas e de produção de curvas de resposta de inibidores dependentes da dose. Mais importante, a capacidade de filtrar muitos companheirorials produzido informações sobre os tipos de materiais que melhor se adequam tanto a produção de microarray e retenção da atividade enzimática. Os dados materiais fornecer informações sobre os requisitos materiais básicos necessários para a adaptação ideal, de alta densidade sol-gel microarrays derivados.
Introdução
Microarrays ganharam popularidade no seio da comunidade científica como um método para aumentar o rendimento do ensaio. Desde o desenvolvimento da tecnologia de microarranjo para avaliar a expressão do gene 1 em meados da década de 1990, microarrays de ter encontrado utilização no desenvolvimento de ensaios de alto rendimento para identificar a proteína-proteína e proteína-interacções molécula pequena, e para encontrar novos materiais com propriedades únicas. 2-5 Mais recentemente, foram desenvolvidos os microarrays em que os materiais específicos são utilizados para imobilizar proteínas funcionais, produzir um elemento de microarray tridimensional dentro do qual as proteínas são aprisionadas, permitindo a fácil medição de actividade enzimática e a inibição no próprio usando um microarranjo de fluorescência adequada ensaio que é acoplada ao volume de substrato. 5-10 Importante, tais microarrays pode ser concebido para incluir todos os componentes necessários para as amostras de tela e controlos em conjunto de uma maneira altamente paralela. 5,8
Os primeiros exemplos de microarrays de proteína foram tipicamente preparados usando métodos padrão para a imobilização da proteína em suportes sólidos, tais como ligação covalente, 11 afinidade capturar 3 e adsorção física. 12 Embora estes métodos de bio-imobilização para permitir o aumento da concentração da amostra e da cinética de reacção acelerados necessário para miniaturização de ensaio, cada um deles sofre desvantagens. Em geral, todos causar uma redução na funcionalidade biomolécula nativa devido a modificação química da superfície, dificultando o acesso a sítios activos, ou na orientação incorrecta devido a imobilização não específica. Assim, todos os métodos resultam em sensibilidade do ensaio inferior apesar de um aumento na deposição de biomoléculas, uma resposta que surge provavelmente devido à necessidade de se ligar artificialmente biomoléculas a uma superfície.
Uma abordagem emergente para a produção de microarrays de biomoléculas funcionais é através do pino-impressão de proteína dopadosoles de sílica em suportes sólidos, os quais são normalmente lâminas de vidro ou funcionalizados ou poços individuais de placas de microcavidades. O processo sol-gel de si tem lugar num meio aquoso, à temperatura ambiente, e é o precursor líquido, que é impresso e, em seguida, os geles para biomoléculas aprisionam dentro da matriz 3D, permitindo a elevada carga de proteína 13, bem como o aprisionamento de múltiplos componentes dentro o mesmo elemento de microarray. 13,14 Adaptação do material derivado de sol-gel pode ser feito através de uma selecção cuidadosa de diferentes precursores de sílica, bem como alterando o componente aquoso através do uso de tampões diferentes (pH, força iónica) e inclusão de vários aditivos (polímeros, pequenas moléculas) para obter um material ideal, a natureza específica dos quais depende a biomolécula que está retido 10.
Uma limitação potencial associado com o desenvolvimento de sol-gel de microarrays de proteínas derivados via pin-impressão éa necessidade de identificar materiais compósitos baseados em sol-gel que podem ser impressas sem gelificação no pino ou mostrando as propriedades indesejáveis (tamanhos irreproduzíveis local rachaduras, falta de adesão, a incompatibilidade com componentes de ensaio, a má atividade da proteína), uma vez impressas em uma superfície. 5 simultânea otimização de todos estes parâmetros impede uma abordagem em que os materiais podem ser projetados de novo, ou examinou lentamente de uma forma serial. Por outro lado, a seleção aleatória de muitos milhares ou dezenas de milhares de materiais não é nem de tempo nem de baixo custo.
Neste artigo, é descrito um método de rastreio dirigido, que permite a rápida identificação de materiais apropriados para a produção de microarrays de proteína, sem a necessidade de rastrear aleatoriamente um grande número de materiais. Utilizando uma abordagem orientada, materiais adequados para a impressão de microarray são identificadas em primeiro lugar, seguido por uma série de telas de pequena escala para identificar óptima de sol-gel derivado material combinações que podem ser impressos de forma reprodutível, sem rachaduras e são compatíveis com um determinado ensaio. Finalmente, os materiais óptimos são identificados com base na manutenção da actividade da enzima e desempenho em um teste de rastreio em pequena molécula final. Desta forma, os materiais podem ser identificados óptimas de muitos milhares de candidatos usando apenas algumas centenas de passos de ensaio. Nós demonstramos esta abordagem para o fabrico de ambos acetilcolinesterase de alta densidade e microarranjos multiquinase e a utilização de tais microarrays para pequenas moléculas de triagem.
Protocolo
1. Preparação das soluções aditivas
- Preparar 25 e 50 mM de Tris (hidroximetil) aminometano (Tris-base, M r 121,14 g / mol) e HEPES (M r 238,3 g / mol) a pH 7.0, 7.2, 7.4, 7.6, 7.8, 8.0 e 8.2 em 50 ml de centrífuga tubos. Ajustar o pH usando HCI 1N e NaOH, respectivamente. Guarde-o em temperatura ambiente e substitua após 3 meses.
- Prepare a 24% (w / v) de poli (álcool vinílico) (PVA): pesa-se 2,4 g de PVA (M w 9.000 - 10.000, 80% hidrolisado) e adicionar 10 ml de água desionizada-destilada (ddH2O), dissolver as pastilhas de polímero por vórtex. Usar a solução 24% (w / v) para preparar volumes iguais de 16% (w / v), 12% (w / v) e 8% (w / v), soluções de PVA. Soluções desgaseifica por sonicação antes da utilização. Armazenar a 4 ° C durante até 1 mês.
- Prepare a 24% (w / v) de polietilenoimina (PEI): adicionar 4,8 ml de PEI (M w 1300, 50% (w / w) em H 2 O) a 5,2 ml de ddH 2 O. Usar a solução 24% (w / v) para preparar volumes iguaisde 16% (w / v), 12% (w / v) e 8% (w / v), soluções de PEI. Soluções desgaseifica por sonicação antes da utilização. Armazenar a 4 ° C durante até 1 mês.
- Prepare a 60% (w / v) de polietilenoglicol (PEG): pesa-se 6 g de PEG (M w 600) e adicionar 10 ml de ddH2O, dissolver as pastilhas de polímero em vórtice. Utilização de diluição em série para se preparar um volume igual de 30% (w / v) de solução de PEG. Soluções desgaseifica por sonicação antes da utilização. Armazenar a 4 ° C durante até 1 mês.
- Prepare a 24% (v / v) de N-(3-trietóxissililpropilo) gluconamide (GLS): adicionar 480 uL de GLS (50% em etanol - GLS imagens pode requerer o uso de um banho de água e aquecer até se dissolver solução cristalina) para 520 ul de 95% (v / v) de etanol e mistura-se durante 20 minutos por sonicação. Usar a solução de 24% (v / v) para fazer um volume igual de 16% (v / v) de solução de GLS. Faça novas soluções para o uso por dia.
- Prepare a 24% (v / v) metiltrimetoxissilano (MTMS): adicionar 240 uL de MTMS a 760 ul de existir acidificada ddH2O (pH 2,0, 1 N HCl) e mistura-se durante 20 minutos por sonificação. Usar a solução de 24% (v / v) para fazer um volume igual de 16% (v / v) de solução de MTMS. Faça novas soluções para o uso por dia.
- Prepare a 24% (v / v) de bis [(3-methyldimethoxylsilyl) propil] óxido de polipropileno (MDSPPO): adicionar 240 uL de MDSPPO a 760 ul de existir acidificada ddH2O (pH 2,0, HC1 IN) e mistura-se durante 20 min pela sonorização. Usar a solução de 24% (v / v) para fazer um volume igual de 16% (v / v) de solução MDSPPO. Faça novas soluções para o uso por dia.
- Prepare a 24% (v / v) 3 - (aminopropil) trietoxissilano (APTES): adicionar 240 uL de APTES e 760 ul de existir acidificada ddH2O (pH 2,0, HC1 IN) e mistura-se durante 20 minutos por sonificação. Usar a solução de 24% (v / v) para fazer um volume igual de 16% (v / v) de solução de APTES. Faça novas soluções para o uso por dia.
- Prepare a 24% (v / v) de bis (triethoxysily) etano (bis-TEOS): adicionar 240 uL de cloreto de bis-TEOS para 760 ul de existir acidificado ddH2O (pH 2,0, HC1 IN) E misture por 20 minutos de sonorização. Usar a solução de 24% (v / v) para fazer um volume igual de 16% (v / v) de solução de bis-TEOS. Faça novas soluções para o uso por dia.
- Prepare a 24% (v / v) carboxyethylsilanetriol (Si-COOH): adicionar 960 uL de Si-COOH (25% em ddH2O) a 40 ul de existir acidificada ddH2O (pH 2,0, HC1 IN) e mistura para 20 min por sonorização. Usar a solução de 24% (v / v) para fazer um volume igual de 16% (v / v) de solução Si-COOH. Faça novas soluções para o uso por dia.
- Separadamente preparar a 3 mM ε N - acetil-L-lisina, D-sorbitol, α-α-trealose (M r 188.22 g / mol, 182,17 g / mol, e 378,33 g / mol, respectivamente) e 1,5 mM de Triton X-100 (média M w 625 g / mol) em DDH 2 O. Os volumes podem variar, dependendo de quanto é necessário, tornar as soluções frescas para utilização por dia.
- Prepare a 60% (w / w) de glicerol: adicionar 2,4 g de glicerol anidro até 1,6 g de ddH2O, mistura por vórtice. Solução Degaspor ultra-sons antes da utilização. Armazenar a 4 ° C durante até 1 mês.
- Prepare a 30% (w / w) de glicerol: adicionar 1,6 g de glicerol anidro até 2,4 g de ddH2O, mistura por vórtice. Desgaseifica solução antes da utilização por sonicação. Armazenar a 4 ° C durante até 1 mês.
2. Preparação de soluções coloidais de sílica
Seguindo os procedimentos descritos abaixo, as respectivas soluções coloidais, quando mantidos em gelo, pode ser utilizado até 1 hora após a adição de água. Sóis utilizados para além de 1 hr resultado na diminuição tempos de gelificação material / inconsistente.
- Preparação de um silicato de sódio (SS) baseado sol
- Pesam-se 120 g de resina de permuta iónica em um copo de plástico de 500 ml.
- Adicionar 150 ml de HCl 0,1 N e agita-se durante 1 hora usando uma barra de agitação magnética de 2 polegadas.
- Filtrar a solução através de um funil de Buchner ligado a vácuo.
- Lentamente, adicionar ddH2O para lavar a resina filtrada até o filtrado resultante é clara. Isto demora cerca de 100ml de DDH 2 O.
- Recolha e armazenar a resina preparada num recipiente de plástico à temperatura ambiente durante 1 mês.
- Pesar 2,59 g de silicato de sódio (SS, 27% (w / w) de SiO2, 10% (w / w), NaOH) num copo de 50 ml de plástico.
- Adicionar 10 ml de DDH 2 O para a SS. Agitar suavemente com a mão para misturar a solução.
- Pesar 5,60 g de resina preparada em um copo de 50 ml de plástico separado. Adicionar a solução de silicato de sódio e mistura-se durante 2 minutos utilizando uma barra de agitação magnética de uma polegada.
- Filtra-se a solução de mistura com um funil de Buchner ligado a um aspirador ligado a uma torneira de água.
- Usar uma seringa de plástico de 10 ml equipado com um filtro de membrana de 0,2 um para filtrar a solução de sol. Isto produz um sol com 5,6% (w / w) de sílica denominada SS em texto adicional. Mantenha o sol no gelo quando não estiver em uso.
- ½ SS: adicionar 1 ml de preparado SS sol para 1 ml de DDH 2 O, mix de vórtice. Manter o solenóide no gelo quando não emusar.
- Preparação de um diglycerylsilane (DGS) sol baseado
- Sintetizar DGS conforme já descrito. 15,16 Loja DGS cristalinas num secador em temperatura ambiente por até 6 meses.
- Pesar cerca de 1 g da DGS cristalinas.
- Use um almofariz e pilão para moer os DGS a um pó fino. Conclua esta etapa tão rapidamente quanto possível para evitar a absorção de humidade a partir do ar.
- Transferir cuidadosamente os DGS finamente moído para um frasco de cintilação vazio de 20 ml, registrar a massa (a um milésimo de um grama).
- Adicionar ddH2O para dar uma solução a 0,5 g / ml DGS.
- Sonicate os DGS hidrolisadas no gelo por 20 minutos, misture por vórtex por 5 segundos a cada 5 min. Durante os dias húmidos, a dissolução completa do DGS podem exigir a adição de 10 ul de HC1 IN. Este deve ser adicionado à solução antes da sonicação.
- Usando uma seringa de plástico de 3 ml, equipado com um filtro para filtrar a solução de sol de membrana de 0,2 um, a remoção de partículas finas. Isto produz um sol com 5,0% (w / w) de sílica denominada DGS em texto adicional. Mantenha o sol no gelo quando não estiver em uso.
- ½ DGS: adicionar 1 ml de preparado DGS sol para 1 ml de DDH 2 O, mix de vórtice. Mantenha o sol no gelo quando não estiver em uso.
3. Pré-triagem para identificar materiais para impressão
Um esquema generalizado que mostra as diferentes fases da análise factorial guiada usado para identificar materiais de impressão está representada na Figura 1. Recomenda-se um volume mínimo de material total de 100 ul. A utilização de volumes mais pequenos torna visualizando gelificação material difícil.
- Fase 1: tampão e silano
- Adicionar 50 ul de tampão (Tris 25 mM e 50 ou HEPES a pH 7.0, 7.2, 7.4, 7.6, 7.8, 8.0, 8.2) a um frasco de cintilação de vidro ml 2.
- Adicionar 50 ml da sol apropriado (DGS, ½ DGS, SS, ½ SS) e mistura de vórtice de forma on-e-off por 10 segundos pormover o frasco dentro e fora do vortex.
- Conjunto de materiais repousar à temperatura ambiente, o controlo da época de gelificação do material. O tempo de gelificação é definido como o ponto no qual o material pára de fluir livremente rotativo sobre o frasco para um ângulo de 45 °.
- Excluir combinações de materiais com tempos de gelificação inferior a 2,5 hr de etapas de triagem de materiais subseqüentes.
- Fase 2: tampão, silano e polímero
- Em um frasco de 2 ml de cintilação de vidro, adicionar 3,13 ml de solução ou 16% (w / v), 8% (w / v) de PVA / PEI ou 6,3 ul de 30% (w / v) ou 60% (w / v) de solução de PEG.
- Adicionar 50 mM de HEPES (pH 8,0) para perfazer um volume combinado de 50 ul, misturar a solução por vórtice.
- Adicionar 50 ml da sol apropriado (DGS, ½ DGS, SS, ½ SS), mistura da forma descrita no passo 3.1.2.
- Excluir combinações de materiais com tempos de gelificação (conforme definido no passo 3.1.3) inferior a 2,5 h de etapas de triagem de materiais subseqüentes.
- Fase 3: tampão, de silano, polímero e organossilano
- Em um frasco de 2 ml de cintilação de vidro, adicionar 3,13 ml de solução de PVA ou 16% (w / v), 8% (w / v), ou 6,3 ml de 30% (w / v) ou 60% (w / v) solução de PEG.
- Adicionar 3,13 mL de 16% (w / v) de organosilano (GLS, MTMS, MDSPPO, bis-TEOS, Si-COOH ou APTES).
- Adicionar 50 mM de HEPES (pH 8,0) para perfazer um volume combinado de 50 ul, misturar a solução por vórtice.
- Adicionar 50 ml da sol apropriado (DGS, ½ DGS, SS, ½ SS) e misturar da forma descrita no passo 3.1.2. Excluir combinações de materiais com tempos de gelificação (conforme definido no passo 3.1.3) inferior a 2,5 h de etapas de triagem de materiais subseqüentes.
- Fase 4: buffer, aditivo de silano, polímero, organossilanos e pequenas moléculas
- Em um frasco de 2 ml de cintilação de vidro, adicionar 2,08 ml de solução de PVA ou 24% (w / v) ou 12% (w / v), ou 6,3 ml de 30% (w / v) ou 60% (w / v) solução de PEG.
- Adicionar2,08 mL de 24% (w / v) de organosilano (GLS, Si-COOH).
- Adicionar 2,08 mL da solução 3 mM de pequenas moléculas (N ε - acetil-L-lisina, D-sorbitol, α-α-trealose) ou 1,5 mM (Triton X-100).
- Adicionar 50 mM de HEPES (pH 8,0) para perfazer um volume combinado de 50 ul, misturar a solução por vórtice.
- Adicionar 50 ml da sol apropriado (DGS, ½ DGS, SS, ½ SS) e misturar da forma descrita no passo 3.1.2.
- Excluir combinações de materiais com tempos de gelificação (conforme definido no passo 3.1.3) inferior a 2,5 h de estágios de material de impressão subseqüentes.
4. Preparação de Materiais para impressão Proteína dopados
Os materiais identificados no final do passo 3.4.6 são transportados em estudos de capacidade de impressão, com a enzima apropriada agora a ser incluído no sol. Em todos os casos descritos a seguir, solução aquosa contendo todos os componentes, excepto para o silanoé preparado de uma microplaca, seguido da adição do sol imediatamente antes da impressão. Para acomodar a utilização de uma placa de microtitulação de 384, num volume total de 50 ul de solução é utilizada em vez de 100 ul.
- Acetilcolinesterase (AChE) microarray
- Prepara-se uma solução de 2 kU / mL de AChE em 50 mM de HEPES (pH 8,0), após a actividade enzimática específica do lote (unidades de actividade por mg), as informações fornecidas pelo fabricante.
- Alíquota de 2 solução estoque de enzima kU / ml em 5 frações ul utilizando microtubos de 500 ul e armazenar a -20 ° C. Duas soluções estoque AChE KU / ml permanecer estável por até 4 meses, quando armazenadas a -20 ° C.
- Preparar os materiais de base, combinando a metade dos volumes de polímeros correspondentes, organossilanos e aditivos de pequenas moléculas identificadas através do passo 3.4.6 numa placa de microtitulação de 384 poços.
- Adicionar 5 mL de 2 kU / mL de AChE em 50 mM de HEPES (pH 8,0).
- Usando HEPES 50 mM (pH 8,0) e levar o conjuntobem volume para 25 mL mix pipetando a solução cima e para baixo várias vezes.
- Multiquinase microarray
- Preparar soluções de 100 quinase ng / ul (p38a, MAPK2, EGFR, a GSK-3β) em ddH2O sequência da actividade (U / ml ou U / mg) e a informação da quantidade fornecida pelo fabricante. Armazenar as soluções quinase em alíquotas de 2 ul a -80 ° C.
- Prepara-quinase substratos (MBP, p (E4 Y), GSM) a 5 mg / ml, 50 mg / ml ou 300 uM em ddH2O, seguindo a informação de quantidade fornecida pelo fabricante. Loja substratos em alíquotas de 2,5 mL a -80 ° C.
- Preparar os materiais de base, combinando a metade dos volumes de polímeros correspondentes, organossilanos e aditivos de pequenas moléculas identificadas através do passo 3.4.6 numa placa de microtitulação de 384 poços.
- Para cada uma das cavidades contendo os materiais de base, adicionar 2,5 mL de quinase e 2 ul do respectivo substrato (p38a e PAM, PAM e MAPK2, EGFR e p (E4 Y), GSK-3β e GSM), como preparados nos passos 4.2.1 e 4.2.2, respectivamente.
- Usando HEPES 50 mM (pH 7,4), levar o volume bem combinada 25 ul. Misture por pipetagem.
5. Formação Microarray
Esta seção explica o procedimento detalhado para a impressão de materiais em uma superfície de um único diapositivo. Para imprimir em várias superfícies de deslizamento modificados (amina, epoxi, aldeído e PMMA), este procedimento é repetido 4 vezes.
- Formar microarrays usando um contato robô pin-impressão equipado com um palco XYZ. Utiliza pinos ranhurados bainha de 100 um de diâmetro para depositar os soles de proteína dopados.
- Conjunto de humidade no interior da câmara de impressão para 80-90%. Humidade <80% pode resultar na evaporação da amostra e inconsistências na impressão, devido aos volumes de deposição pequenas.
- Sonicar pinos em ddH2O durante 15 min antes da impressão e seco sob uma corrente de azoto. Use um tubo Cleaner para remover a umidade residual de dentro do suporte do pino e coloque cuidadosamente o pino na cabeça de impressão. Falha para remover a umidade residual pode dificultar o pino de mover-se livremente com o titular, o que resulta em manchas perdidas.
- Padrões de matriz de controle através do programa Pro Escritor Chip (CWP). Para cada poço de amostra no interior da placa de fonte de 384 poços, 200 pontos (número máximo de pontos por absorção utilizando o pino de bainha SMP3) são impressas sobre a superfície de uma lâmina de vidro modificado.
- Iniciar o processo de impressão por meio do software. Conjunto de viagens na direcção XY a 10 mm / s, velocidade de aproximação e de amostra (sentido Z) a 2 mm / s, com uma amostra de 2,5 seg O tempo de carregamento.
- Pausar a corrida através CWP. Abaixar o pino na amostra e adicionar 25 uL da respectiva solução coloidal (DGS, ½ DGS, SS, ½ SS) do poço com uma pipeta. Misture a solução utilizando um movimento para cima e para baixo pipetagem repetida 50x. Durante a mistura por pipetagem, minimizar a quantidade de ar incorporada na solução. Bolhas de ar prevençãot completa carregamento de amostra dentro do pino.
- Iniciar o processo de impressão através CWP, e imprimir a amostra sobre uma superfície slide. Pausar o processo de impressão antes de colocar amostra da placa de fonte posterior também.
- Retire o pino de impressão utilizando um ímã e coloque um limpador de cachimbo na cabeça de impressão para evitar o acúmulo de umidade na cabeça de impressão.
- Lavar o pino de impressão com DDH 2 O, e sonicate em limpo DDH 2 O por 30 segundos.
- Seca-se o pino de impressão sob uma corrente de azoto e remover o tubo limpo, colocando a parte traseira do pino na cabeça de impressão.
- Começar a impressão seguindo os passos 5,6-5,10, para todas as amostras restantes dentro da placa fonte. Até 12.000 pontos de 100 um de diâmetro podem ser depositadas sobre uma única lâmina.
- Este método também pode ser aplicado a materiais de impressão para o fundo dos poços no prazo de uma microplaca de 96 poços. Após o arquivo de calibração CWP, recalibrar o pino de impressão para garantir a distance viajou até entre deposição local está acima da altura da microplaca. Isto permite que o pino de imprimir de forma consistente de poço para poço de uma forma linear, sem danificar o pino.
- Idade da matriz por um período mínimo de 30 minutos ou superior a 24 horas na câmara de impressão a 80-90% de humidade após a conclusão de toda a experiência de impressão.
6. Acetilcolinesterase Atividade Assay
- Preparando-se amostras de controlo positivas (PC)
- Prepare 1 mM de iodeto de acetiltiocolina (Atch: M r 289,18 g / mol), em 4% de glicerol, 25 mM Tris (pH 7,0) num tubo de microcentrífuga de 1,5 ml. Atch precisa de ser preparada antes de cada utilização e, utilizando um tampão de pH 7,0 como tampão de pH mais elevadas causam autohydrolysis rápida, produzindo os falsos positivos. O volume final pode ser variada, dependendo do número de amostras (25 ul por amostra).
- Adicionar 0,14 mL de 5 mM de BODIPY-fl-L-cistina para 25 ul de 1 mM Atch no poço de microtitulação-384 plate.
- Adicionar 24,86 mL de 4% de glicerol, 25 mM Tris (pH 7,0) e misturar, pipetando cuidadosamente para evitar a formação de bolhas na solução. Potenciais inibidores de enzima podem ser incorporados na solução de PC antes de levar o volume a 50 ul com tampão.
- Preparação de amostras de controlo negativo (CN)
- Adicionar 0,14 mL de 5 mM de BODIPY-fl-L-cistina para 49,86 ml de 4% de glicerol, 25 mM Tris (pH 6,5) na cavidade de uma placa de microtitulação de 384. Misture por pipetagem.
- Overprinting PC e soluções de NC
- Sobrepor os microarrays idade seguintes passos 5,1-5,10 (ignorando o passo 5.6) do protocolo. Use pinos bainha fendas de diâmetro 235 mM para depositar o PC e soluções de impressão sobreposta NC. Isto assegura a solução abrange o local anteriormente depositado inteiramente.
- Matrizes A idade de 80-90% de humidade durante 1 hora à temperatura ambiente. Devido à autohydrolysis de ATCH, os tempos de incubação mais longos podem resultar em falsos positivos ou aumentada brZYME atividade.
7. Ensaio da Actividade de Quinase
- Preparar solução de 500 uM de adenosina 5'-trifosfato (ATP), utilizando estoque de ATP (100 mM), armazenada a -20 ° C. Faça solução nova para cada experimento e ajustar o volume com a necessidade experimental: cada solução de impressão requer 5 mL.
- Prepare a 100 mM de cloreto de magnésio (MgCl 2: r g / mol M 95,21) solução estoque em DDH 2 O.
- Controle positivo (PC)
- Adicionar 2,5 mL de MgCl 2 100 mM e 5 ul de 500 uM de ATP para o poço de uma placa de microtitulação de 384.
- Levar o volume total a 50 bem uL com 50 mM de HEPES (pH 7,4) e misturar por pipetagem. Potenciais inibidores de enzima podem ser incorporados na solução de PC antes de levar o volume a 50 ul com tampão.
- Controle negativo (NC)
- Adicionar 2,5 mL de 100 mM de MgCl 2 e 5 ul de ddH2O para o poço de uma de 384 mplaca icrotiter.
- Traga o volume bem total para 50 mL com 50 mM HEPES (pH 7,4) e misture por pipetagem.
- Overprinting PC e NC cofatores ensaio
- Siga o passo 6.3.1 do protocolo.
- Matrizes A idade de 80-90% de humidade durante 2 horas à temperatura ambiente.
- Coloração das lâminas
- Lugar microarray impresso desliza individualmente em uma placa de Petri com corante suficiente (do kit) para cobrir todo o slide. Agitação moderadamente (~ 200 rpm) durante 45 min usando um agitador de placas.
- Remover o slide usando uma pinça e coloque em um prato de Petri limpa com tampão de lavagem (de kit). Agitação moderadamente (~ 200 rpm) durante 45 min usando um agitador de placas.
- Remover o slide usando uma pinça e girar seco usando uma microcentrifuge microarray desktop convencional equipado com um suporte de slide.
8. Microarray Imaging and Analysis
- Imagem
Note-se que o método de imagem será sESPECÍFICAS para o tipo de leitor de matriz disponível. Para estes estudos, um Alpha Innotech NovaRay gerador de imagens com uma fonte de luz branca e o sistema de detecção CCD equipado com um 478 ± 17 nm de excitação e 538 ± 21 nm, filtro de emissão para os microarrays de AChE, e 530 ± 40 nm de excitação e de emissão de 614 ± 62 nm filtrar foi usado microarrays quinase. Sistemas de varredura confocal a laser também pode ser usada para a leitura das matrizes, embora as configurações irão ser específicos para o instrumento.
- Coloque o slide no compartimento de slides com os pontos para cima.
- Defina o número de seções pré-visualização (um mínimo de 2 é recomendado, uma em cada extremidade da lâmina de microscópio), resolução (mínimo de 4 mM é recomendado) e exposição (auto é recomendado) dentro do software de imagem.
- Adquirir imagem do slide e salvar como um arquivo. "Tiff".
- Análise
- Abra imagem do slide adquirido em ImageJ64.
- Clique na ferramenta de seleção oval e medir aintensidade de sinal de cada ponto usando a opção sob medida o guia analisar. Ao selecionar a área para medir, selecionar uma região um pouco maior do que o ponto observado e de tamanho consistente entre os pontos de reduzir ImageJ subjetividade.
- Média da intensidade de 25 pontos PC semelhantes, dividido pela intensidade média de 25 pontos semelhantes NC obter proporções PC / CN para as composições de material individuais.
Resultados
Através da realização de uma análise factorial guiada para a tela de material, fomos capazes de minimizar o número de materiais testados, a partir de ~ 20.000 a algumas centenas que tinham tempos de gelificação adequados para impressão. Através da aplicação de uma orientação estrita exige tempo de gelificação de material de 2,5 horas ou mais, os materiais susceptíveis de entupir os pinos de impressão ou produzir matrizes irreprodutíveis nunca foram impressas. Os materiais de impressão identificados tenham tempos (> 2,5 hr) de gelificação suficientes foram impressas em quatro diferentes superfícies de deslizamento de vidro funcionalizadas. De modo a ser considerado como "impressão", o número máximo de pontos por unidade de volume a absorção do pino teve de ser impresso (SMP3 = 200). Manchas foram também avaliadas para a morfologia local para assegurar nenhuma separação de fases de cracking ou indesejável tinha ocorrido utilizando microscopia de campo brilhante simples, como mostrado na Figura 2.
A partir desta fase de materiais de impressão identificados, microarrays foram produzidos com a AChE equinases incorporada no componente aquoso tamponado. Materiais que eram compatíveis com o procedimento de ensaio (incluindo potencial overprinting e máquina de lavar ou coloração etapas) foram identificados pela observação retenção de manchas microarray (sem rachaduras, perda de pontos ou padrões incomuns de fluorescência) e um controle positivo (CP) para o controle negativo (NC ) rácio superior a 1, como observado através da imagem. Como este foi cerca de 50% dos materiais, uma maior proporção PC / CN, de 3 foi utilizada para definir materiais óptimos com retenção da actividade da proteína. Através deste método, 26 sol-gel de materiais derivados contendo materiais doer e 2 contendo quinases satisfeitos os critérios> 3 PC / NC. Figuras 3 e 4 mostram uma avaria gráfica dos 5 passos da tela materiais guiadas para a identificação de ótima AChE e quinase microarrays, respectivamente.
Os ensaios também podem ser validado através da geração de uma pontuação Z »17. Isto foi feito utilizando o material que produziu a proporção PC / CN maior. Figura 5 mostra o plano Z 'obtida através da comparação do sinal gerado a partir de 200 pontos, 100 e 100 PC NC após a impressão sobreposta de corante indicador e do substrato sobre a matriz de AChE. A dor e as matrizes quinase resultou nos respectivos escores Z 'de 0,60 e 0,67, indicativo de um excelente ensaio. No entanto, deve notar-se que antes da validação do ensaio, enzima, corante, substrato e co-factor em concentrações de matriz teve de ser optimizadas por impressão sobreposta uma gama de concentrações de cada componente e seleccionando a concentração que produziu o sinal mais elevado, tal como descrito em pormenor noutro 5.
Para validar os ensaios, dados de inibição quantitativos foram obtidos utilizando conhecidos e desconhecidos, os inibidores da acetilcolinesterase, com resultados realizados em duplicado e utilizada para produzir os gráficos de duplicados (Figura 6A) e as curvas de inibição (Figuras 6B e 6C). <> Spots fortes foram sobreposta primeiro com misturas de inibidores de pequenas moléculas biologicamente activas que, em seguida, com o corante e o substrato, e as misturas de controlo contendo tanto conhecidos inibidores ou inibidores não foram incluídos. Lotes duplicados foram gerados para avaliar a actividade da enzima, e quaisquer misturas que resultaram em pelo menos 25% a actividade da enzima foi considerada positiva para a inibição. Os compostos individuais de tais misturas foram então testadas em duplicado, para identificar a molécula pequena específico (s) responsável pela inibição. Uma vez identificadas, estas pequenas moléculas foram usadas para gerar curvas de inibição quantitativos para determinar valores de IC50 e as constantes de inibição.
Resultados qualitativos semelhantes foram obtidos utilizando a matriz multiquinase com um inibidor da quinase comum, estaurosporina. Figura 7A e 7B mostram a imagem de microarray e indicam que as intensidades de sinal após a coloração e impressão sobreposta multiquinasematriz são tal como esperado para um controlo negativo (- ATP), controlo positivo (+ ATP) e inibidor conhecido (+ ATP + INH). Para demonstrar a capacidade de obter dados de inibição quantitativa dos microarrays, um ensaio de inibição dependente da concentração foi feita por uma única cinase. Tal como mostrado nas Figuras 8A e 8B, a intensidade do sinal diminui à medida que aumenta a concentração de inibidor, e a resposta segue a curva de inibição, dependente da concentração esperada para o sistema de quinase / substrato p38α/MBP.
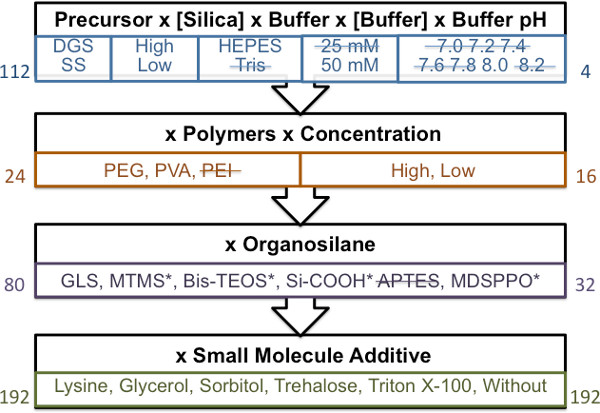
Figura 1. Esquema geral para a abordagem de triagem de materiais guiada. Cada bloco representa um passo da tela em ordem sequencial. Os números do lado esquerdo indicam o número total de materiais preparados para análise. Utilizando um tempo de gelificação maior do que 2,5 horas (materiais com tempos de gelificação inferior a 2,5 hr are indicado pelo riscado), o número de materiais que passaram cada etapa e transportado durante a tela de material são indicadas pelo número à direita. * Representa materiais com menos do que a separação de fases óptima.

Figura 2. Imagens ópticas mostram vários modos de materiais na etapa capacidade de impressão da tela fracasso. Uma imagem de um "bom" do material (segunda linha, terceira coluna) também é mostrado para comparação. Reproduzido com permissão da referência 8, copyright 2013 American Chemical Society.
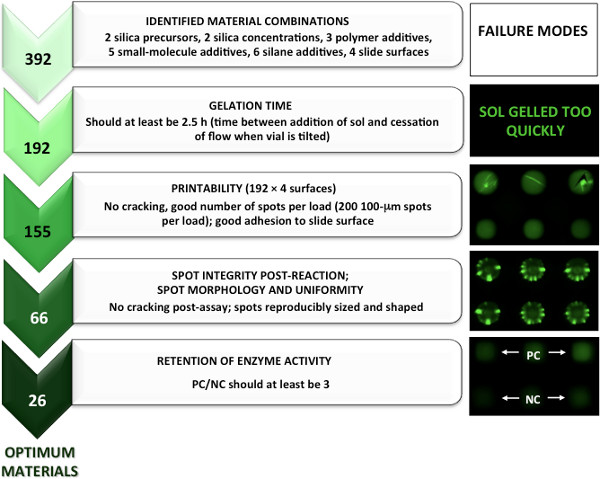
Figura 3. Uma abordagem de triagem de material direcionado para a identificação de materiais ideais para a fabricação de sol-gel derivados de microarrays doer. Reproduzido com permissão de referência 5, copyright 2013 American Chemical Society.

Figura 4. A triagem de materiais abordagem direcionada para a identificação de materiais ideais para a fabricação de microarrays quinase sol-gel derivados. Reproduzido com permissão da referência 8, copyright 2013 American Chemical Society.
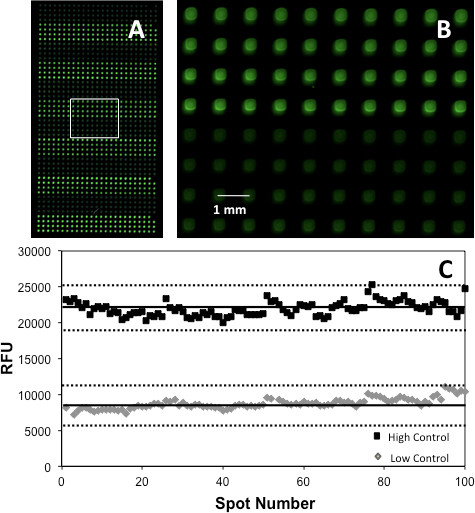
Figura 5. (A) A seção de microarray AChE mostrando HC (verde brilhante) e LC (luz verde) pontos (uma paleta de preto-verde foi aplicado como pseudo para maior clareza de apresentação), (B) uma visão ampliada da área de box para destacar local Morfologia e alinhamento;. e (C) um lote de Z 'linhas a cheio indicam a média da repetições, enquanto que as linhas tracejadas correspondem a 3DP. Reproduzido com permissão da referência 5, copyright 2013 American Chemical Society.
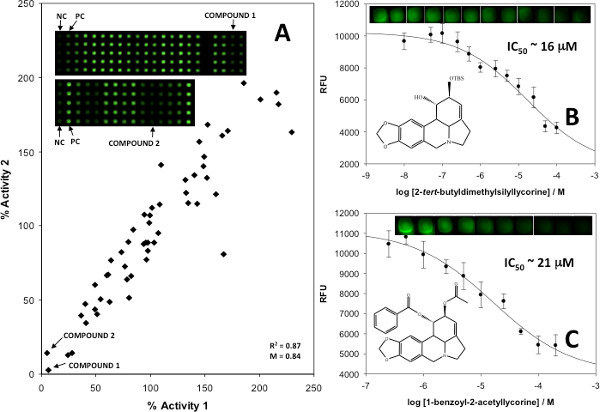
Figura 6. (A) Duplicado lote para o rastreio em série de análogos sintéticos dos alcalóides de Amaryllidaceae, (B) IC 50 parcelas de inibidores potenciais identificadas marcadas como compostos 1 e (C), composto 2, com barras de erro que representa um desvio padrão da média de 25 repetições. pontos representativos são apresentados para ilustrar as diferenças de sinal proporcional às concentrações de inibidor. Reproduzido com permissão da referência 5, copyright 2013 American Chemical Society. Clique aqui para ver a figura maior .
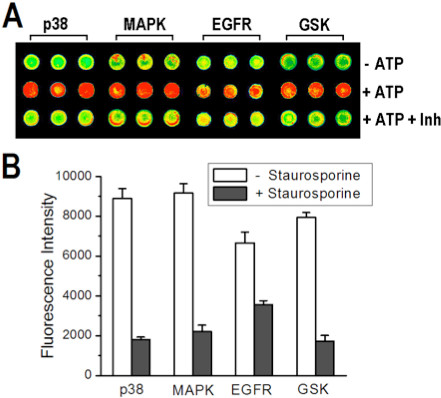
Figura 7. Ensaio On-matriz de quatro quinases usando 1.4SS/1.0PVA para o aprisionamento e impressos em um slide de amina derivados. (A) Uma imagem de uma seção de microarray na qual os pontos com quinases co-aprisionados com seus respectivos substratos foram sobreposta com tampão (NC, linha superior), ou soluções contendo ATP (PC, linha do meio) ou ATP + staurosporina (linha de fundo). (B) Os gráficos de barras comparando a intensidade do sinal entre as reacções inibidas e não inibidas, após a subtracção dos sinais de branco e barras de erro representam um desvio padrão da média de 25 repetições. Reproduzido com permissão da referência 8, copyright 2013 American Chemical Society.
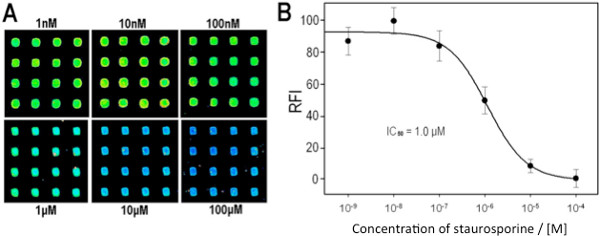
Figura 8. Inhibiti. em ensaio em uma p38a/MBP microarray (D) Secções de microarrays que mostram pontos representativos overspotted com concentrações variadas de estaurosporina, como indicado (as imagens foram obtidas através de um único exame da mesma lâmina; imagem compósita é mostrado para maior clareza). (B) curva de IC 50 gerada a partir das imagens de matriz analisados. A intensidade obtida a 100 mM foi subtraída de todas as imagens, todas as outras intensidades foram normalizados definindo a intensidade obtida a 10 nM para um valor de 100% de actividade. Reproduzido com permissão da referência 8, copyright 2013 American Chemical Society.
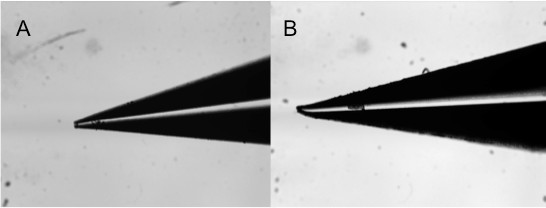
Figura 9. Imagens de pin discrição microscópico utilizado para contato pin-impressão mostrando várias imperfeições: (A) entupido, (B) dobradas.
Discussão
A metodologia aqui descrita foi escolhido como o mais adequado para identificação de impressão de materiais sol-gel derivados, com uma impressora de contacto, produzindo um tempo de processo e de custo eficaz para identificar rapidamente os materiais óptimos sem ter de analisar grandes quantidades de materiais. De um total de ~ 20000 materiais potenciais, foi possível identificar ~ 200 materiais que eram adequadas para impressão com base no tempo de gelificação sós. Isto reduz significativamente o número de materiais necessárias para estar preparado para os ensaios de impressão subsequente. Estes materiais de impressão foram então impressas em quatro superfícies de deslizamento para um total de 768 combinações de material de deslizamento. Em média, 50 pontos / repetições de um material pode ser impresso no ~ 3 min, incluindo o carregamento da amostra, local de deposição e limpeza pin. Destes, os materiais 155, ou cerca de 20%, permitido para imprimir o número máximo de pontos por absorção da solução e produzido local tamanhos reproduzíveis. Deve-se notar que o 4 slide superfícies testadas, materiais impressos melhor na ordem: amina> epóxi> aldeído> PMMA; lâminas PMMA não produzir matrizes úteis para todos os materiais. Isto foi provavelmente atribuído à polaridade do revestimento de superfície. Comparando-se as superfícies de deslizamento atrás mencionadas, a amina mais polar e epóxi eram mais adequados para os sóis aquosos, em comparação com as corrediças de PMMA. Além disso, as superfícies de teste, as lâminas revestidas com amina proporcionam uma superfície carregada positivamente para o potencial depositado aniónico sol de ligação. Suspeita-se, a sílica nanopartículas na interface entre a corrediça e o sol de interagir ao longo da superfície. Tanto o epoxi e as superfícies de deslizamento de aldeído não têm a mesma interacção inicial baseada em carga. Para garantir a deposição local óptimo é altamente recomendada a usar lâminas pré-revestidas de um fornecedor tal como Arrayit. O revestimento em casa produz superfícies inconsistentes que levam a uma má reprodutibilidade ponto 13 e, em alguns casos, pode conduzir a problemas de quantificaçãoblemas. 18 De igual importância, temperatura e umidade afetam a "qualidade de impressão" dos materiais. Enquanto não existem estudos detalhados sobre os efeitos relacionados com a temperatura foram realizadas, a impressão era sempre realizado à temperatura ambiente (23 ± 3 ° C). Humidade (superior a 80%) foi também controlada no interior da câmara de impressão para evitar a deposição de forma irregular, devido à deposição de pequenos volumes de 0,7-2,3 (nl) e a evaporação.
Enquanto a tela material foi orientada para identificar materiais derivados de sol-gel ótimas especificamente para impressão de AChE e quinases, um pequeno conjunto de materiais foi identificado que trabalhou para os dois tipos de proteínas. Com efeito, ambos os materiais que foram identificados para a fabricação de microarray quinase foram baseados em PVA SS + + glicerol, e ambos os materiais foram também identificados dentro dos 26 materiais seleccionados para microarranjos de AChE. Estes materiais "ótimas" pode oferecer um ponto de partida genérico para desenvolver ainda mais protein-dopados microarrays baseados em sol-gel, e telas pequenas centradas em torno destas composições podem identificar ainda melhores materiais para fabricação de microarray. Um segundo ponto a salientar é a importância da enzima utilizada. No caso da AChE (uma enzima bastante robusta), 26 (ou seja, aproximadamente 40%) das originais 66 materiais identificados como ensaio compatível retida a actividade da AChE aprisionada. No entanto, para as cinases mais delicado, apenas 2 das composições compatíveis com o ensaio 69, ou cerca de 3% do material, foram capazes de manter a actividade de todas as quinases. Quando um número suficiente de diferentes enzimas não têm sido estudados para fazer afirmações conclusivas, parece que a fabricação matriz otimização com enzimas relativamente instáveis podem levar à identificação de materiais que podem entrap uma ampla gama de proteínas para permitir mutliplexed microarray fabricação.
Independente da proteína escolhida, o principal factor de cut-off para a identificação de materiais de impressão era a necessidade de uma longatempos de gelificação de material (> 2,5 hr). Quando o desenvolvimento de materiais à base de sol-gel de SS, que é muito importante para garantir que, na sequência de troca iónica e filtração, o sol é de cerca de pH 4. Os sóis com um pH inicial inferior pode resultar em materiais com um pH mais baixo do que o neutro, o que pode afectar a actividade da enzima. 19 ajustando a quantidade de Dowex (resina de troca de iões) para SS pode alterar o pH final da solução coloidal. Quando um novo lote de resina é preparada a proporção de resina a SS tem de ser ajustada de modo a produzir soluções coloidais a cerca de pH 4 seguindo o processo no ponto 2 do protocolo.
Da mesma forma, a preparação da DGS cristalinas é frequentemente uma fonte de erro associado à insuficiência de material ao utilizar DGS sóis base para o aprisionamento biomolécula. Apesar de não ser relatado aqui em pormenor, um grande cuidado deve ser feita durante a síntese do DGS cristalinos, em particular a necessidade de evitar a presença de água durante a síntese, o que pode produzir sílica polyglyceratedtes, em vez de DGS monoméricos. Além disso, devido à natureza higroscópica do DGS, a amostra cristalina necessita de ser armazenado dessecado e utilizado dentro de 6 meses após a síntese. DGS cristalinos com mais de 6 meses, não pode dissolver completamente (devido ao material de silicato de poliglicerilo parcialmente condensada), mesmo com sonicação em um ambiente ácido. DGS incompleta dissolução produz sóis com desconhecido e incontrolável teor de sílica e, portanto, os materiais menos robustos.
Um ponto importante a notar com a impressão de contacto é a qualidade dos pinos. Dos pinos danificadas ou extraviadas (Figura 9) nunca irá produzir matrizes reprodutíveis independentes do material a ser impresso. Recomenda-se verificar a qualidade do pino usando um microscópio de dissecação para garantir pinos quebrados ou entupidos não são usados. Manuseio cuidadoso garante vida longa para os pinos. O movimento livre do pino também é importante. Nos casos em que a humidade é preso na cabeça de impressão entre a cabeça e o pino, o pinonão irá assentar correctamente e, portanto, não entra em contacto adequado com a superfície, o que resulta numa falta de deposição de material.
Em conclusão, nós fornecemos uma abordagem de triagem detalhada, várias etapas para o desenvolvimento de proteínas dopados microarrays pin-impressos de alta densidade. O rastreio envolve a optimização das propriedades dos materiais (o tempo de gelificação e capacidade de impressão) para permitir a impressão de materiais, seguido por triagem mais focado para identificar materiais que sejam compatíveis com um determinado ensaio e capaz de manter a actividade da enzima. Esta abordagem de triagem de material orientado pode ser aplicado a formatos microarray adicionais para reduzir o tempo e os custos associados com a produção eficiente microarranjos de alta densidade.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Os autores agradecem a Maria Monton, Julie Lebert, Jessamyn Little, Xin Ge e Laura Lautens de assistência em desenvolvimento de microarrays de proteína. Os autores também agradecem as Ciências Naturais eo Conselho de Pesquisa em Engenharia do Canadá (NSERC) para o financiamento deste trabalho. Os autores também agradecer à Fundação Canadense para Inovação e do Ontário Confiança Inovação para o apoio a este trabalho. JDB detém o Canada Research Chair in Chemistry bioanalíticos e Biointerfaces.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Poly(vinyl alcohol) (PVA) | Sigma-Alderich | 360627 | 80% hydrolozyed, Mw 600 |
| Polyethylene glycol 600 (PEG) | Sigma-Alderich | 87333 | |
| Polyethylenimine (PEI) | Sigma-Alderich | 482595 | 50 wt% solution in water |
| Carboxyethylsilanetriol (Si-COOH) | Gelest, Inc. | SIC2263.0 | 25% in water |
| N-(3-triethoxysilylpropyl) gluconamide(GLS) | Gelest, Inc. | SIT8189.0 | 50% in ethanol |
| bis[(3-methyldimethoxysilyl)propyl]polypropylene oxide (MDSPPO) | Gelest, Inc. | SIB1660.0 | |
| Methyltrimethoxysilane (MTMS) | Gelest, Inc. | SIM6560.1 | |
| Bis(triethoxysiyly)ethane (Bis-TEOS) | Gelest, Inc. | SIB1817.0 | |
| 3-Aminopropyltriethoxysilane (APTES) | Gelest, Inc. | SIA0610.0 | |
| Glycerol | Sigma-Alderich | 49767 | |
| D-Sorbitol | Sigma-Alderich | 240850 | |
| D-(+)-Trehalose dihydrate | Sigma-Alderich | T9531 | |
| Triton X-100 | Sigma-Alderich | X-100 | |
| Nε-Acetyl-L-lysine | Sigma-Alderich | A4021 | |
| Tris(hydroxymethyl)aminomethane | Sigma-Alderich | 154563 | |
| HEPES | Sigma-Alderich | H3375 | |
| Sodium hydroxide, 1.0 N | LabChem Inc. | LC24350-2 | |
| Hydrochloric Acid, 1.0 N/0.1 N | LabChem Inc. | LC15300-2/LC152220-2 | |
| Magnesium chloride | Sigma-Alderich | M8266 | |
| Diglycerolsilane (DGS) | Prepared in laboratory | ||
| Sodium silicate solution | Fisher Scientific | SS338-1 | |
| Dowex 50WX8-100 ion exchance resin | Sigma-Alderich | 217492 | |
| Acetylthiocholine iodide | Sigma-Alderich | 1480 | |
| Acetylcholinesterase from Electrophorus electricus (electric eel) | Sigma-Alderich | C2888 | |
| BODIPY FL L-Cystine | Invitrogen | B-20340 | |
| Pro-Q Diamond Phosphoprotein/Phosphopeptide Microarray Stain Kit | Invitrogen | P33706 | |
| Adenosine 5'triphosphate disodium salt (ATP) solution | Sigma-Alderich | A6559 | |
| MAP Kinase 2 (MAPK2) | EMD Millipore | 454850 | |
| p38α/SAPK2a (T106M), active | EMD Millipore | 14-687M | |
| Epidermal growth factor (EGFR) | EMD Millipore | Donated by Millipore | |
| Glycogen synthase kinase 3β (GSK-3β) | EMD Millipore | 14-306 | |
| Myelin basic protein (MBP) | EMD Millipore | Substrate for MAPK2 and p38α, Donated by Millipore | |
| GSM | EMD Millipore | 12-533 | Substrate for GSK-3β |
| Poly-glu-tyr polypeptide p(E4Y) | EMD Millipore | 12-440 | Substrate for EGFR |
| Stealth pin | ArrayIt | SMP3 | |
| Stealth pin | ArrayIt | SMP7 | |
| Amine coated slides | ArrayIt | SMM2 | |
| Aldehyde coated slides | ArrayIt | SMA2 | |
| Exposy coated slides | ArrayIt | SME2 | |
| Poly(methylmethacrylate) (PMMA) coated slides | Exakt Technologies Inc. | 41500 | |
| 0.2-μm syringe filter | PALL Life Sciences | 4612 | |
| Equipment | |||
| Virtek Contact Printer | BioRad | ||
| Novaray Fluorescence Slide Imager | Alpha Innotech Corporation | ||
| Desktop microarray centrifuge | ArrayIt | MHC110V | |
| MilliQ Synthesis A10 | Millipore | Used to filter all water required for experiments |
Referências
- Schena, M. M., Shalon, D. D., Davis, R. W. R., Brown, P. O. P. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science (New York, N.Y.). 270 (5235), 467-470 (1995).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science (New York, N.Y.). 289 (5485), 1760-1763 (2000).
- Zhu, H. H., Bilgin, M. M., et al. Global analysis of protein activities using proteome chips. Science (New York, N.Y.). 293 (5537), 2101-2105 (2001).
- Hook, A. L., Chang, C. -. Y., et al. Combinatorial discovery of polymers resistant to bacterial attachment. Nature Biotechnology. , (2012).
- Monton, M. R. N., Lebert, J. M., Little, J. R. L., Nair, J. J., McNulty, J., Brennan, J. D. A Sol-Gel-Derived Acetylcholinesterase Microarray for Nanovolume Small-Molecule Screening. Analytical Chemistry. 82 (22), 9365-9373 (2010).
- Cho, E. J., Tao, Z., Tehan, E. C., Bright, F. V. Multianalyte pin-printed biosensor arrays based on protein-doped xerogels. Analytical Chemistry. 74 (24), 6177-6184 (2002).
- Cho, E. J., Tao, Z., et al. Tools to Rapidly Produce and Screen Biodegradable Polymer and Sol-Gel-Derived Xerogel Formulations. Applied Spectroscopy. Society for Applied Spectroscopy. 56 (11), 1385-1389 (2002).
- Ge, X., Lebert, J. M., Monton, M. R. N., Lautens, L. L., Brennan, J. D. Materials Screening for Sol-Gel-Derived High-Density Multi-Kinase Microarrays. Chemistry of Materials. 23 (16), 3685-3691 (2011).
- Rupcich, N., Green, J. R. A., Brennan, J. D. Nanovolume Kinase Inhibition Assay Using a Sol-Gel-Derived Multicomponent Microarray. Analytical Chemistry. 77 (24), 8013-8019 (2005).
- Rupcich, N. Coupled enzyme reaction microarrays based on pin-printing of sol-gel derived biomaterials. Analytica Chimica Acta. 500 (1-2), 3-12 (2003).
- MacBeath, G. G., Schreiber, S. L. S. Printing proteins as microarrays for high-throughput function determination. Science (New York, N.Y.). 289 (5485), 1760-1763 (2000).
- Stillman, B. A. B., Tonkinson, J. L. J. FAST slides: a novel surface for microarrays. BioTechniques. 29 (3), 630-635 (2000).
- Rupcich, N., Goldstein, A., Brennan, J. D. Optimization of sol-gel formulations and surface treatments for the development of pin-printed protein microarrays. Chemistry of Materials. 15 (9), 1803-1811 (2003).
- Gill, I., Ballesteros, A. Encapsulation of biologicals within silicate, siloxane, and hybrid sol-gel polymers: An efficient and generic approach. Journal of the American Chemical Society. 120 (34), 8587-8598 (1998).
- Brook, M. A., Chen, Y., et al. Proteins entrapped in silica monoliths prepared from glyceroxysilanes. Journal of Sol-gel Science and Technology. 31 (1), 343-348 (2004).
- Brook, M. A., Chen, Y., Guo, K., Zhang, Z., Brennan, J. D. Sugar-modified silanes: precursors for silica monoliths. Journal of Materials Chemistry. 14 (9), 1469 (2004).
- Zhang, J., Chung, T., Oldenburg, K. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- . Next generation of protein microarray support materials: Evaluation for protein and antibody microarray applications. Journal of Chromatography A. , 1-8 (2013).
- Bhatia, R. B., Brinker, C. J., Gupta, A. K., Singh, A. K. Aqueous Sol-Gel Process for Protein Encapsulation. Chemistry of Materials. 12 (8), 2434-2441 (2000).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados