Method Article
Entwicklung und Validierung einer quantitativen PCR-Methode für Equiden Herpesvirus-2-Diagnose in Atem Flüssigkeiten
In diesem Artikel
Zusammenfassung
Here, we present a protocol for the development and validation of a quantitative PCR method used for the detection and quantification of EHV-2 DNA in equine respiratory fluids. The EHV-2 qRT-PCR validation protocol involves a three-part procedure: development, characterization of qRT-PCR assay alone, and characterization of the whole analytical method.
Zusammenfassung
The protocol describes a quantitative RT-PCR method for the detection and quantification of EHV-2 in equine respiratory fluids according to the NF U47-600 norm. After the development and first validation step, two distinct characterization steps were performed according to the AFNOR norm: (a) characterization of the qRT-PCR assay alone and (b) characterization of the whole analytical method. The validation of the whole analytical method included the portrayal of all steps between the extraction of nucleic acids and the final PCR analysis.
Validation of the whole method is very important for virus detection by qRT-PCR in order to get an accurate determination of the viral genome load. Since the extraction step is the primary source of loss of biological material, it may be considered the main source of error of quantification between one protocol and another. For this reason, the AFNOR norm NF-U-47-600 recommends including the range of plasmid dilution before the extraction step. In addition, the limits of quantification depend on the source from which the virus is extracted. Viral genome load results, which are expressed in international units (IU), are easier to use in order to compare results between different laboratories.
This new method of characterization of qRT-PCR should facilitate the harmonization of data presentation and interpretation between laboratories.
Einleitung
Equid Herpes - Virus-2 (EHV-2) in einem Respiratorisches Syndrom, mit potentiellen klinischen Manifestationen wie Nasenausfluss, Pharyngitis und geschwollene Lymphknoten 1-3 beteiligt. Dieses Virus wird vermutet , auch mit den Armen-Leistung von Pferden in Verbindung gebracht werden, die zwei in einer signifikanten und negativen wirtschaftlichen Auswirkungen für die Pferdebranche führen kann.
Bisher war der Goldstandard für die Gamma-EHV (γ-EHV) Detektion der Zellkulturverfahren. Der erste Nachteil dieses Verfahrens war das Fehlen der Diskriminierung zwischen EHV-2 und γ-EHV ist (zB EHV-5). Der zweite Nachteil war die langsame Entwicklung des cytopathischen Verfahren, die vom 12. bis 28 Tage dauert 4,5 zu manifestieren.
Entwicklung eines validierten und normalisierte quantitative Echtzeit-Polymerase-Kettenreaktion (qRT-PCR) -Methode wird dazu beitragen, schnell das Virus zu erkennen, zwischen EHV-2 und EHV- zu diskriminieren5 und die Beziehung zwischen dem viralen Genom Last und die Krankheit durch die Quantifizierung Aspekt zu studieren.
Die Polymerase - Kettenreaktion (PCR) wurde 1986 von Mullis 6 zum ersten Mal beschrieben und ist über die neue Goldstandard zu werden in die meisten Felder der biologischen Diagnose (Mensch, Umwelt und Tier). Dieses Verfahren, das auf die Amplifikation eines Teils des Genoms der Pathogene basiert, stellt viele Vorteile: Spezifität, Empfindlichkeit und Schnelligkeit. Darüber hinaus zurückgetreten das Risiko von Amplikonkontamination seit dem Aufkommen der qRT-PCR und Qualitätssicherung 7. Dennoch erforderte die Anerkennung der PCR als neuen Goldstandard-Methode mehr als nur Spieldaten verbessert, sondern auch die Demonstration der Kontrolle der Entwicklung und Validierung Schritte des gesamten Verfahrens ohne Verschlechterung der Leistung im Laufe der Zeit.
Die erste molekulare Werkzeuge für die Detektion verwendet EHV-2 wurden Zeit consuming und beteiligten unspezifische Amplifikation mit nested PCR 8 durch Sequenzierung gefolgt. Die gezielte Gene für Herpes - Viren wurden Desoxyribonukleinsäure (DNA) Polymerase und DNA Verpackung 9. Jedoch nested PCR stellt ein hohes Risiko der Kontamination durch Amplikons. Seitdem haben sich herkömmliche PCR - Tests entwickelt worden , um die Interleukin-10-like - Gen oder Glycoprotein B - Gens zu amplifizieren, überprüft 2009 2. In jüngerer Zeit, Echtzeit - PCR Eigenschaften für die Quantifizierung von EHV-2 beschrieben sind, wurden 10 jedoch keine Daten zur Verfügung standen in Bezug auf die Validierung des gesamten Verfahrens einschließlich des Extraktionsprozesses.
In diesem Protokoll, Entwicklungs- und Validierungsverfahren sind für eine quantitative PCR - Verfahren zum Nachweis und zur Quantifizierung von EHV-2 - DNA in der Pferdeatmungsflüssigkeiten nach der Association Française de normalisation (AFNOR) Norm NF U47-600 3,11,12 beschrieben, das ist der Französisch Vertreter indie internationale Normalisierungskommission. Diese Norm beschreibt die "Anforderungen und Empfehlungen für die Umsetzung, Entwicklung und Validierung von Tier PCR in der Tiergesundheit Analyseverfahren" 11,12, nach dem NF EN ISO / CEI 17025, 2005 13 und OIE (Weltorganisation für Tiergesundheit) . Empfehlungen, 2010 14 der EHV-2 qRT-PCR - Validierungsprotokoll beinhaltet ein dreiteiliges Verfahren: (a) Entwicklung der qRT-PCR - Test, (b) Charakterisierung der qRT-PCR - Test allein und (c) Charakterisierung des gesamten Analyseverfahren (von der Extraktion von Nukleinsäuren aus der biologischen Probe zur PCR-Analyse).
Die Charakterisierung der qRT-PCR-Assays und des gesamten Analysemethode umfassen die Definition von zwei Grenzen: Die Nachweisgrenze (LOD) und der Bestimmungsgrenze (LOQ). Die LOD 95% PCR stellt die niedrigste Anzahl von Nukleinsäure Kopien pro Volumeneinheit , die in 95% aller cas nachgewiesen werden kannes. Die Bestimmungsgrenze von 95% PCR stellt die niedrigste Menge an Nukleinsäure Kopien, die die Unsicherheiten unter Berücksichtigung bestimmt werden kann.
Diese qRT-PCR-Methode ermöglicht die präzise Erfassung und schnelle Quantifizierung von EHV-2 in der Atem Flüssigkeiten. Darüber hinaus könnte das Verfahren in anderen Laboratorien angewendet werden, um ein standardisiertes Verfahren, um sicherzustellen, und als allgemeine Vorlage für die Entwicklung weiterer neuer qRT-PCR-Assays.
Protokoll
Hinweis: Siehe all die verschiedenen Schritte , die in 1 dargestellt sind.
1. Extraktion von Nukleinsäuren
Hinweis: Führen Sie Extraktion unter einer Abzugshaube Atemwegs Kontamination mit Nukleinsäuren zu begrenzen. Umfassen eine Extraktions negative Kontrolle mit DEPC-behandeltem Wasser, um sicherzustellen, dass keines der Reagenzien mit unerwünschten DNA verunreinigt sind.
- Extrahieren von Nukleinsäuren aus der biologischen Probe nach einem zuvor beschriebenen Protokoll 15 und Protokoll des Herstellers.
- Hinzufügen 140 ul biologischen Probe auf 560 & mgr; l Lyselösung (AVL-Puffer) und Inkubation für 10 min bei Raumtemperatur. Hinzufügen 560 ul Ethanol. Gelten die ersten 630 ul dieser Lösung (Probe + Lyselösung + Ethanol) auf einer Silica-Säule und zentrifugieren.
- Anwenden, um die verbleibenden 630 & mgr; l dieser Lösung wurden zu gleichen Silicasäule und zentrifugiert. Dann waschen Sie die Säule mit 500 ulvon 2 verschiedenen Waschpuffer (AW1 und AW2).
- Eluieren Nukleinsäure mit 50 & mgr; l Elutionspuffer (AVE-Puffer) und auf Raumtemperatur äquilibrieren. Schließen Sie die Kappe und Inkubation für 1 min bei Raumtemperatur. Zentrifuge bei 6.000 g für 1 min.
2. Amplifikationsprozedur
- Bereiten 22,5 ul Reaktionsmischung für jede Reaktion. Hinzufügen, 12,5 & mgr; l PCR-Mastermix, x & mgr; l von 20 & mgr; M Vorwärtsprimer, x & mgr; l von 20 & mgr; M Rückwärts-Primer, y & mgr; l von 10 & mgr; M Sonde und z & mgr; l ultrareinem Wasser als 22,5 & mgr; l zu erreichen erforderlich (x, y und z Volumina erhalten nach der Titration, siehe die Abschnitte 3.2.3 und 3.2.6).
- Aliquot von 22,5 & mgr; l der geeigneten Reaktionsgemisch gut zu jeder Reaktion in einer 96-Well-Platte.
Hinweis: Fügen negative Kontrollen für die Extraktion und für die PCR, um sicherzustellen, dass keines der Reagenzien mit unerwünschten DNA verunreinigt sind. - Hinzufügen, 2,5 & mgr; l der Probe wurden 2,5 & mgr; l Extraktions negative-Steuerung, 2,5 ul der PCR-negative Kontrolle und 2,5 ul der positiven Probe (Referenzstamm oder Plasmid) in die entsprechende Reaktion gut. Nach der Verteilung, decken Sie die Platte mit einer Klebeplatte Dichtung. Zentrifugieren Sie die Platte für 10 Sekunden bei 6.000 g.
- Die Platte wird in einer Real-Time PCR-System. Wählen Sie die Vorlage für den Test-Layout und den Lauf starten. Verwenden Sie die PCR Programmeinstellung: 10 min bei 95 ° C , gefolgt von 45 Zyklen von 15 sec bei 95 ° C und 1 min bei 60 ° C (Tabelle 1).
- Übertragen Sie die Rohdaten von der Real-Time PCR-System in eine Tabelle. Stellen Sie die Schwelle in den Verstärkungsstücke oberhalb der Basislinie und in der exponentiellen Wachstumsregion des Schwellenzyklus für jede Probe zu erhalten. Zeichnen Sie jeden Punkt Standard als Standardkurve gesetzt Linearität zu erhalten. Berechnen der Kopienzahl für die unterschiedlichen Proben auf der Standardkurve berechnet.
3. Entwicklung der quantitativen RT-PCR
Hinweis: Die Entwicklung eines qRT-PCR-Test erfordert Referenzstämme, eine spezifische titriert Plasmid und verschiedene Kontrollen und beinhaltet die Titration der Primer und Sonde.
- Vorversuch
- Design - Primer und Sonden mit spezieller Software nach den bisherigen Empfehlungen 16.
- Auszug Referenzstämme, wie in Kapitel 1 beschrieben.
- Amplify, wie in Abschnitt 2 mit 900 nM für die endgültige Konzentration von Primern und 250 nM für die endgültige Konzentration der Sonde beschrieben. Analysieren Sie das Signal als in Abschnitt 2.5 beschrieben.
- Zur gleichen Zeit, führen Amplifikation von DNA-Referenzstämme ohne Sonden (wie in Abschnitt 3.1.3 beschrieben). Sequenz , die die Amplifikate durch die Sanger - Methode 17,18 erhalten. Analysieren Sie die Sequenzen , die durch eine Nucleotid - BLAST 19 ausgeführt wird .
- Titration von Primern und Sonde
- Bereiten Sie drei verschiedene Mischungen mit 250 nM Endkonzentration von Sonde und mit unterschiedlichen endgültigen concentrtionen der Vorwärts- und Rückwärts-Primer (50 nM / 50 nM, 300 nM / 300 nM, 900 nM / 900 nM) für die 3 Replikate der positiven Probe und eine negative Kontrolle. Für jede Mischung, die gleiche entsprechende Volumen von 20 & mgr; M Vorwärtsprimer hinzufügen und 20 & mgr; M Reverse-Primer (0,25 & mgr; l der 50 nM Endkonzentration zu erhalten, 1,5 ul der 300 nM Endkonzentration oder 4,5 & mgr; l zu erhalten, die 900 nM Endkonzentration zu erhalten) , 50 & mgr; l PCR Master Mix, 2,5 ul 10 uM Sonde und Reinstwasser nach Bedarf 90 & mgr; l zu erreichen.
- Führen Sie die Amplifikationsverfahren, wie in Abschnitt 2 beschrieben.
- Wählen Sie die beste Voraussetzung für die höchste Stufe der Verstärkung, früheste Zyklus Schwellenwert (Ct) und beste Wiederholbarkeit zwischen den drei Bedingungen zu erhalten. Bestimme die beste Konzentration und das entsprechende Volumen x ul der Vorwärts- und Rückwärts-Primern.
- Bereiten Sie 5 verschiedene Mischungen für die 4 Proben (3 Replikate der positiven Probe und eine Negativkontrolle)mit 5 verschiedenen Endkonzentrationen von Sonde (50 nM, 100 nM, 150 nM, 200 nM, 250 nM). Für jede Mischung, fügen x ul 20 uM Vorwärtsprimer und x ul 20 uM Reverse-Primer (vorher bestimmt, in Abschnitt 3.2.3), 50 & mgr; l PCR-Master-Mix, entsprechende Volumen von 10 & mgr; M-Sonde (0,5 ul der zu erhalten 50 nM Konzentration, 1 & mgr; l der 100 nM Konzentration zu erhalten, 1,5 ul der 150 nM Konzentration zu erhalten, 2 & mgr; l der 200 nM Konzentration oder 2,5 ul zu erhalten, die 250 nM Konzentration) und Reinstwasser zu erhalten, wie es erforderlich ist, 90 & mgr; l zu erreichen.
- Führen Sie die Amplifikationsverfahren, wie in Abschnitt 2 beschrieben.
- Wählen Sie die beste Voraussetzung für die höchste Stufe der Verstärkung, früheste Zyklus Schwellenwert (Ct) und beste Wiederholbarkeit zwischen den fünf Bedingungen zu erhalten. Ermitteln Sie die beste Konzentration und das entsprechende Volumen y ul für die Sonde.
4. Charakterisierung von Quantitative Real-time PCR (qRT-PCR)
Hinweis: Nach der Entwicklungsschritt und die Bestimmung der besten Bedingungen zu verwenden, wird die Charakterisierung Schritt der PCR die Spezifität umfasst, die Nachweisgrenze, die Linearitätsbereich und die Grenze der Quantifizierung der qRT-PCR.
- Herstellung und Titration von Plasmid
- Bestellen kommerziellen Plasmid , das das relevante Fragment der DNA - Zielgens für die PCR ausgewählt , die (in diesem Protokoll, Nukleotide 2081 bis 2381 von EHV-2 - Glykoprotein B - Gen, siehe Tabelle 1).
Hinweis: Um das Risiko von Atemwegs Kontamination mit synthetischen DNA begrenzen, führen Reihenverdünnungen von Plasmiden unter einer Abzugshaube in einem separaten Raum und arbeiten mit verdünnter DNA während aller Schritte der Entwicklung und Validierungsverfahren. - Re-suspend das Plasmid mit Reinstwasser, um eine Stammlösung bei 50 ng / ml herzustellen. Vortex und Zentrifuge kurz die Plasmid-Stammlösung.
Hinweis: Bestimmen Sie die wirkliche Konzentration des Plasmid stock Lösung nach Resuspension, um die Kopienzahl des Plasmids zu berechnen. - 1 & mgr; l Plasmid zu dem Spektrophotometer. Lesen Sie die optische Dichte (OD) bei 230 nm, 260 nm und 280 nm. Lesen Sie die DNA-Konzentration auf dem Spektralphotometer Software (OD 260 nm) aus.
- Berechnen Sie die Kopienzahl des Plasmids unter Verwendung von Avogadro-Zahl (N A) und die Formel:
Die Kopienzahl / ul = (N A [Plasmid in ng / ul]) / (Plasmid Länge x 10 9 x durchschnittliche Gewicht eines Basenpaar) = (6,022 x 10 23 x [Plasmid]) / (Plasmid Länge x 10 9 x 660)
- Bestellen kommerziellen Plasmid , das das relevante Fragment der DNA - Zielgens für die PCR ausgewählt , die (in diesem Protokoll, Nukleotide 2081 bis 2381 von EHV-2 - Glykoprotein B - Gen, siehe Tabelle 1).
- Testen der Spezifität (Inklusivität und Exklusivität) von qRT-PCR
- Testen Sie die Inklusivität des PCR-System. Wählen Sie EHV-2 positive DNA-Proben, die zuvor durch Sequenzierung charakterisiert (positive Status festgestellt).
- Führen Sie die Amplifikationsverfahren, wie in Abschnitt 2 beschrieben.
- analysieren Sie diePCR-Daten für jede Probe, wie in Abschnitt 2.5 beschrieben. Prüfen Sie das Vorhandensein einer exponentiellen Kurve für alle in Abschnitt ausgewählten Proben 4.2.1 und bestätigen Inklusivität von PCR.
- Testen der Exklusivität des PCR-System unter Verwendung von DNA-Extrakten aus Pathogenen mit genetischen Ähnlichkeiten zu dem Ziel (in diesem Fall andere equid Herpesviren, wie EHV-1, EHV-4, EHV-3, EHV-5 und asinine herpesvirus-5 beschrieben in Tabelle 2) und andere in respiratorischen Erkrankungen des Wirts beteiligt Pathogene (in diesem Fall equine Arteritis - Virus, Pferde - Influenza - Virus, Coxiella burnetii, Rhodococcus equi, Streptococcus equi equi, Streptococcus equi zooepidemicus, Chlamydophila abortus und Klebsiella pneumoniae, siehe Tabelle 2).
- Führen Sie die Amplifikationsverfahren, wie in Abschnitt 2 beschrieben.
- Analysieren Sie die PCR-Daten für jede Probe, wie in Abschnitt 2.5 beschrieben. Überprüfen Sie, ob das Fehlen einer exponentiellen Kurve für alle Proben gewählt inAbschnitt 4.2.4 die Exklusivität der PCR zu bestätigen.
- Nachweisgrenze von qRT-PCR
- Dispense 90 & mgr; l von Reinstwasser in 6 Röhrchen.
- Führen 6 zehnfache serielle Verdünnungen des Plasmids die Minderungszone (Verlust von Ct detection) zu zielen. Transfer 10 ul aus dem Plasmid in das Röhrchen mit 90 & mgr; l Reinstwasser Arbeitsverdünnung. Vortex und Zentrifuge kurz das Rohr. Wiederholen Sie Schritt 4.3.2 bis zur letzten Rohr in der Verdünnungsreihe wurde das Plasmid erhalten.
- Führen Sie die Verstärkung der 6 zehnfache serielle Verdünnungen des Plasmids, wie in Kapitel 2 beschrieben.
- Bestimmen Sie die Minderungszone: die Zone zwischen der letzten Verdünnung von Plasmid , das ein positives Signal und die erste Verdünnung ohne Nachweis präsentiert (siehe Abbildung 2).
- Um die 6 zweifachen Reihenverdünnungen starten, wählen Sie die letzte Verdünnung von Plasmid, das ein positives Signal gibt (siehe Abschnitt 4.3.4).
Hinweis: Führen Sie 3 unabhängige trials die Nachweisgrenze von qRT-PCR (LOD 95% PCR) zu bestimmen , - Dispense 25 ul Reinstwasser in 6 Röhrchen.
- Führen Sie 6 zweifache serielle Verdünnungen des Plasmids. Transfer 25 ul aus dem Plasmid Arbeitsverdünnung, wie 4.3.5, in das Röhrchen mit 25 ul Reinstwasser in Abschnitt bestimmt. Vortex und Zentrifuge kurz das Rohr. Wiederholen Sie Schritt 4.3.7 bis zur letzten Rohr in der Verdünnungsreihe wurde das Plasmid erhalten.
- Führen die Verstärkung der 6 zweifachen seriellen Verdünnungen des Plasmids, wie in Kapitel 2. Wiederholen 4.3.7 Schritte zweimal 4.3.8, zu erhalten, 3 Versuche mit 8 Replikaten (24 Replikaten) von jedem der 6 Zehnfache Reihenverdünnungen des Plasmids.
Anmerkung: Die Nachweisgrenze der qRT-PCR (LOD 95% PCR) als die niedrigste DNA Kopien Anzahl von Volumeneinheit definiert ist, die in 95% der Fälle festgestellt wird. - Berechnen Sie die Anzahl der positiven Replikate von 24 Wiederholungen für jede Ebene von Plasmid Konzention.
- Bestimmen Sie die LOD 95% PCR. Die LOD 95% PCR ist das Niveau , das aus 24 Replikaten in der Detektion von 23 Replikaten positive Ergebnisse.
- Linearitätsbereich und Bestimmungsgrenze von qRT-PCR
Anmerkung: Führen 4 unabhängige Versuche mit 6 zehnfache serielle Verdünnungen des Plasmids , um sicherzustellen , dass die niedrigste Konzentration in dem Bereich verwendet wird, entspricht dem LOD 95% PCR zuvor in 4.3.10 bestimmt.- Dispense 45 & mgr; l von Reinstwasser in 6 Röhrchen.
- Starten Sie die 6 zehnfache serielle Verdünnungen mit der Konzentration von Plasmid - Arbeitsverdünnung bis 10 7 LOD 95% PCR entspricht.
- Führen 6 zehnfache serielle Verdünnungen des Plasmids. Jeweils 5 & mgr; l aus dem Plasmid Arbeitsverdünnung (bestimmt in Abschnitt 4.4.2) in das Röhrchen mit 45 & mgr; l von Reinstwasser. Vortex und Zentrifuge kurz das Rohr. Wiederholen Sie Schritt 4.4.3 bis zur letzten Rohr in der seriellen Verdünnung p erhaltenlasmid.
- Amplify die 6 zehnfache serielle Verdünnungen des Plasmids, wie in Kapitel 2 beschrieben.
- Verfolgen Sie die lineare Regression y = ax + b mit einem für die Steigung und b für den Schnittpunkt (Abbildung 3).
- Berechnen Sie die Verstärkungseffizienz (E) aus der Steigung eine der Standardkurve (siehe 4.4.5) unter Verwendung der Gleichung:
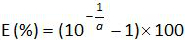 .
.
Wiederholen Sie die Schritte 4.4.1 bis 4.4.6 dreimal.
Hinweis: Amplifikationseffizienz stellt die Menge an PCR-Produkt Erhöhung nach jedem Zyklus. Eine ideale Reaktion erreicht Wirkungsgrad von nahezu 100%. In der Praxis E (%) wurde zwischen 75% und 125%. Höhere E kann Amplifikation von unspezifischen Produkten oder Pipettieren Fehler in der seriellen Verdünnung an. Lower E kann in der Verdünnungsreihe, schlechte Primer-Design oder nicht optimalen Reaktionsbedingungen auch eine Pipettieren Fehler an. - Berechnen Sie die Vorspannung (Tabelle3). Überprüfen Sie für jedes Plasmid - Ebene , die die absolute Bias - Wert ist kleiner als der kritische Bias - Wert (0,25 log 10). Bestimmen Sie die mittlere Vorspannung und die Linearität Unsicherheiten (U Lini) für jedes Plasmid - Ebene (Tabelle 3) , um die Leistung der linearen Regression für EHV-2 qPCR zu bewerten (Abbildung 4). U Lini ist die Linearität Unsicherheit für jeden bestimmt i Ebene Plasmid berechnet von Standardabweichung (SD'i) und mittlere Bias. Bestimmen Sie die kombinierte Linearität Unsicherheit (U LIN) von EHV-2 qPCR durch die Formel:

Hinweis: Die Annahme der Vorspannung wird durch das Labor festgelegt (in der Regel absolute Bias 0,25 log 10) und entspricht der Differenz zwischen dem niedrigsten Wert und dem höchsten Wert von 0,5 log 10 (Menge in 10 Kopienzahl log gemessen). U LIN Wert könnte dazu beitragen, aus verschiedenen Laboratorien Leistung von qPCR zu vergleichen. - Bestimmen Sie die Grenze der Quantifizierung der qRT-PCR (LOQ PCR): LOQ PCR die niedrigste Konzentration mit einer Vorspannung von 0,25 log 10 für den Linearitätsbereich (Tabelle 3) verwendet wird.
5. Charakterisierung des gesamten Analysemethode (von DNA-Extraktion in der qRT-PCR-Ergebnis)
Anmerkung: Die Charakterisierung des gesamten Verfahrens ist die Validierung aller notwendigen Schritte qRT-PCR - Daten zu erhalten (dh aus der Gewinnung von DNA aus den Atemprobe (siehe Abschnitt 1) zur Amplifikation und Quantifizierung des Ziels (siehe Abschnitt 2 )).
- Stellen Sie Korrespondenz zwischen der Kopien Anzahl in der PCR-Reaktion und die Kopien Nummer in der biologischen Probe mit der Formel:
 .
.
Das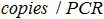 Ist die Anzahl der Plasmid-Kopie in der PCR-Reaktion,
Ist die Anzahl der Plasmid-Kopie in der PCR-Reaktion, 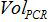 das Volumen der Probe wird auf PCR zur Amplifizierung Mischung hinzugefügt,
das Volumen der Probe wird auf PCR zur Amplifizierung Mischung hinzugefügt, 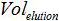 das Volumen von Puffer AVE verwendet Nukleinsäuren zu eluieren und
das Volumen von Puffer AVE verwendet Nukleinsäuren zu eluieren und 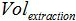 ist das Volumen der entnommenen Probe.
ist das Volumen der entnommenen Probe. - Testen Sie die Sensitivität und Spezifität des gesamten Analysemethode
Hinweis: Nur bekannte EHV-2 positiv (oder negativ) Proben werden in diesem Abschnitt verwendet.- Testen Sie die "Empfindlichkeit" der qRT-PCR-Methode von positiven Proben für das Ziel (EHV-2) zu analysieren.
- Wählen Sie EHV-2 positive DNA-Proben, die zuvor charakterisiert (positive Status).
- Extrahieren Sie die Nukleinsäure, wie in Kapitel 1 beschrieben, ein Verstärkungs Verfahren durchführen, wie in Kapitel 2 beschrieben.
- Bestimmen Sie die Anzahl der realen Positive (positive Probendie positiv arbeitend sind mit dieser RT-PCR) und die Anzahl der falschen Negativen (bekannt positive Proben, die mit dieser RT-PCR-negativ sind).
- Testen Sie die "Spezifität" der qRT-PCR-Methode von negativen Proben zu analysieren
- Wählen EHV-2 DNA negativen Proben (negative Zustand).
- Extrahieren Sie die Nukleinsäure, wie in Kapitel 1 beschrieben, ein Verstärkungs Verfahren durchführen, wie in Kapitel 2 beschrieben.
- Bestimmen der Anzahl von echten Negativen (negative Proben, die mit dieser RT-PCR-negativ bekannt sind) und die Anzahl von Fehlalarmen (bekannten negativen Proben, die mit dieser RT-PCR positiv sind).
- Berechnen Sie die "diagnostische Sensitivität" (Se) und "diagnostische Spezifität" (Sp) des gesamten Verfahrens (Tabelle 4) wie folgt: Se = Anzahl der realen Positive / (Anzahl der realen Positive + falsch negative Ergebnisse ) und Sp = Anzahl der realen Negative / (Anzahl der realen negativens + False Positives) (siehe Tabelle 4).
- Berechnen Sie die 95% Konfidenzintervall für Sensitivität und Spezifität des gesamten Verfahrens mit der Formel von Greiner und Gardner Beziehung 12,20:
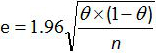
wobei e der geschätzte Fehler ist, θ der Se (oder Sp) und n die Anzahl der untersuchten Proben.
Hinweis: Für eine kleine Anzahl von Proben, verwenden Sie die Schwartz Tabelle nach AFNOR - Norm 12 , um das 95% Konfidenzintervall für Sensitivität und Spezifität des gesamten Verfahrens berechnen.
- Testen Sie die "Empfindlichkeit" der qRT-PCR-Methode von positiven Proben für das Ziel (EHV-2) zu analysieren.
- Herstellung der negativen Quellenmaterial zur Charakterisierung des gesamten Analysemethode
ACHTUNG: Um die Grenzen der Nachweis und die Quantifizierung des gesamten Verfahrens (LOD - Methode und die LOQ - Methode) zu bestimmen, fügen bekannten Konzentrationen von Plasmid zu den biologischen Proben , die frei sind dafür bekannt,des Targets (EHV-2 in diesem Fall). Diese Proben, zusammen mit dem quantifizierten Plasmid, bilden positive Standards , mit denen die LOD - Methode und die Methode LOQ zu bestimmen.- Bestätigen Sie die Abwesenheit von Ziel (EHV-2) in den biologischen Proben verwendet, um positive Standards konstruieren.
- Wählen Sie verschiedene bekannte negative biologische Proben.
- Extrahieren von biologischen Proben, wie in Kapitel 1 beschrieben, die Verstärkung Verfahren durchführen, wie in Kapitel 2 beschrieben.
- Bestätigen Sie die Abwesenheit von Ziel durch das Fehlen von PCR-Signal in diesen Proben.
Hinweis: Verwenden Sie die gleiche negative Ressource Material für alle Stufen zwischen 5,4 und 5,5.
- Pool, die verschiedenen negativen Proben zu 15 ml negativen Ressourcenmaterial zu erhalten (ein Volumen, das notwendig für die vollständige Validierung). Dispense 135 ul dieser negativen Ressource Material in 100 Röhrchen. Halten Röhrchen bei 4 ° C oder bei -80 ° C für lange Lagerung.
- Bestätigen Sie die Abwesenheit von Ziel (EHV-2) in den biologischen Proben verwendet, um positive Standards konstruieren.
- Limit Nachweis des gesamten Analysemethode
- Bestimmen Sie die Minderungszone des gesamten Verfahrens.
- Dispense 45 & mgr; l von Reinstwasser in 6 Röhrchen.
- Führen 6 zehnfache serielle Verdünnungen des Plasmids. Jeweils 5 & mgr; l aus dem Plasmid Arbeitsverdünnung bis 10 7 LOD 95% PCR entspricht mit 45 & mgr; l von Reinstwasser in das Röhrchen (wie in Abschnitt 4.3.10 festgelegt). Vortex und Zentrifuge kurz das Rohr. Wiederholen Sie Schritt 5.4.1.2 bis zum letzten Rohr in der seriellen Verdünnung erhielt Plasmid.
- Jeweils 5 & mgr; l von jeder Verdünnung von Plasmid 2 Röhrchen mit 135 & mgr; l der negativen Quellenmaterial 2 zu erhalten repliziert für die 6 positive Standards. Vortex und kurz Zentrifuge.
- Führen Sie die Extraktion der zwei Replikate für die 6 positive Standards, wie in Kapitel 1 beschrieben, die Verstärkung Verfahren durchführen, wie in Kapitel 2 beschrieben.
- Bestimmen Sie die Minderungszone: die Zone zwischen dem letzten konzentration von Plasmid, das ein positives Signal und die erste Konzentration zu geben, die kein Signal zeigt.
- Nachweisgrenze der Methode (LOD - Methode) von 2 unabhängigen Trials
Anmerkung: Führen 2 unabhängigen Versuchen der Nachweisgrenze des gesamten Verfahrens (LOD - Verfahren) zu bestimmen.- Starten Sie die 6 zweifache serielle Verdünnungen mit dem Plasmid Arbeitsverdünnung, die 4-mal stärker konzentriert, dass die letzte Konzentration des Plasmid ist, das ein positives Signal gegeben (siehe Abschnitt 5.4.1.5).
- Dispense 25 ul Reinstwasser in 6 Röhrchen.
- Führen 5 zweifache serielle Verdünnungen des Plasmids. Transfer 25 ul aus dem Plasmid Arbeitsverdünnung in das Röhrchen mit 25 ul Reinstwasser (wie 5.4.2.1 in Abschnitt bestimmt). Vortex und Zentrifuge kurz das Rohr. Wiederholen Sie Schritt 5.4.2.3 bis zum letzten Rohr in der seriellen Verdünnung erhielt Plasmid.
- Jeweils 5 & mgr; l von jeder Verdünnung von plasmid bis 4 Röhrchen mit 135 ul des negativen Ressourcenmaterials 4 Replikate der 5 positive Standards zu erhalten. Vortex und Zentrifuge kurz auf die Rohre.
- Führen Sie die Extraktion der vier Replikate für die 5 positive Standards, wie in Kapitel 1 beschrieben, die Verstärkung Verfahren durchführen, wie in Kapitel 2 beschrieben.
- Wiederholen Sie die Schritte 5.4.2.1 einmal 5.4.2.5 8 Wiederholungen für jede Ebene von Plasmid-Konzentration zu erhalten.
- Zählen Sie die Anzahl der positiven Wiederholungen von 8 Wiederholungen für jede Ebene von Plasmid-Konzentration.
- Bestimmen Sie die LOD - Methode. Die LOD - Methode ist die letzte Ebene , auf der 8 Wiederholungen von 8 Replikate positiv sind (wie in Abschnitt 5.4.2.7 beschrieben).
- Bestimmen Sie die Minderungszone des gesamten Verfahrens.
- Linearitätsbereich und Quantifizierungsgrenze des gesamten Analysemethode
Hinweis: Um die Grenze der Quantifizierung des gesamten Verfahrens (LOQ - Methode) zu bestimmen, fügen bekannte Konzentrationen des Plasmid biologischenProben, die frei von dem Ziel (EHV-2 in diesem Fall) zu sein, sind bekannt. Diese Proben bilden positive Standards der LOQ Methode zu bestimmen.- Dispense 45 & mgr; l von Reinstwasser in 6 Röhrchen.
- Führen 6 zehnfache serielle Verdünnungen des Plasmids. Jeweils 5 & mgr; l aus dem Plasmid Arbeitsverdünnung bis 10 7 LOD - Methode entspricht mit 45 & mgr; l von Reinstwasser in das Röhrchen (wie in Abschnitt 5.4.2.8 bestimmt). Vortex und Zentrifuge kurz das Rohr. Wiederholen Sie Schritt 5.5.2 bis zur letzten Rohr in der Verdünnungsreihe wurde das Plasmid erhalten.
- Jeweils 5 & mgr; l von jeder Verdünnung des Plasmid 2 Röhrchen mit 135 & mgr; l negative biologische Ressource Material 2 Replikate der 6 positive Standards zu erhalten. Vortex und kurz Zentrifuge.
- Führen Sie die Extraktion der zwei Replikate für die 6 positive Standards, wie in Kapitel 1 beschrieben, die Verstärkung Verfahren durchführen, wie in Kapitel 2. Wiederholen Sie die Schritte 5.5.1bis 5.5.4 dreimal.
- Definieren Sie eine Genauigkeit Profil zu bewerten, um und die quantitative Durchführung des Verfahrens zu bestätigen.
- Definieren Sie die Akzeptanzgrenzen des gesamten Verfahrens für das Labor. In diesem Protokoll werden die Akzeptanzgrenzen definiert als ± 0,75 Log 10 durch die labeo Frank Duncombe.
- Für eine Studie, Spuren eine erste lineare Regression y = ax + b (a ist die Steigung und b der Schnittpunkt ) mit einer Wiederholung von Ct - Werte für jeden Schätzwert der Standardkurve entspricht. Mit dieser ersten linearen Regression, die Berechnung der experimentelle Wert der Kopienzahl für die Replikate von Ct-Werte für diese erste Standardkurve verwendet. Wiederholen Sie Schritt 5.5.5.2 mit den zweiten Replikate von Ct-Werte, um eine zweite lineare Regression erhalten wird, und dem experimentellen Wert der Kopienzahl für die zweite Replikate von Ct-Werte für die zweite Standardkurve berechnen.
- Wiederholen Sie Schritt 5.5.5.2 mit dem zweiten und dritten Studien.
- Berechnen Sie die Genauigkeit, Richtigkeit und Genauigkeitsgrenzen für jedes Plasmid Ebene nach NF U47-600-2 12.
- Erstellen Sie eine Tabelle, die Informationen für alle Standard-Punkte zu kompilieren (5.5.5.4) und erstellen Sie eine Genauigkeit Profil mit den zuvor definierten Akzeptanzgrenzen in 5.5.5.1 und die Richtigkeit der Daten zusammen mit den unteren und den oberen Genauigkeitsgrenzen wie in 5.5 berechnet .5.4 (Abbildung 5).
- Bestimmen Sie die LOQ Methode: das mit Richtigkeits 0,75 log 10 auf die niedrigste Konzentration einer Standardkurve entspricht , wie für den Linearitätsbereich berechnet in 5.5.5.5 verwendet.
- Bewertung der Wiederholbarkeit und Reproduzierbarkeit
Hinweis: Überprüfen Sie die Wiederholbarkeit und Reproduzierbarkeit, indem das Verhältnis zwischen der Standardabweichung unter Verwendung von und den Mittelwert von Wiederholungsmessungen (Variationskoeffizient oder CV = Standardabweichung /bedeuten).- Bewerten Sie die Wiederholbarkeit der ganze Methode von 1 Analyst:
- Wählen Sie 3 biologischen Proben mit 3 verschiedenen viralen Genoms Lasten (zuvor in PCR zum Beispiel getestet).
- Auszug 8 Wiederholungen der drei Proben, wie in Kapitel 1 beschrieben, die Verstärkung Verfahren durchführen, wie in Kapitel 2 beschrieben.
- Berechnen Sie den Mittelwert und Standardabweichung des Ct - Wert für jede Probe gesammelt.
- Berechnen Sie die Intra-Assay-CV mit der Formel CV = Standardabweichung / Mittelwert.
- Bewerten Sie die Reproduzierbarkeit der Methode insgesamt um 3 Analysten:
- Wählen Sie 3 biologischen Proben mit 3 verschiedenen viralen Genoms Lasten (zuvor in PCR zum Beispiel getestet)
- Auszug 2 Wiederholungen der 3 Proben gewählt in 5.6.2.1 (wie in Abschnitt 1 beschrieben). Führen Sie die Verstärkung Verfahren wie in Abschnitt 2. Wiederholen Sie die Schritte beschrieben 5.6.2.2 von 2 unabhängigen Analysten.
- Berechnen Sie den Mittelwert und Standardabweichungdes Ct - Wert für jede Probe gesammelt.
- Berechnen Sie die Inter-Assay-CV mit der Formel CV = Standardabweichung / Mittelwert.
- Bewerten Sie die Wiederholbarkeit der ganze Methode von 1 Analyst:
Ergebnisse
Die quantitative RT-PCR - Methode, wie oben beschrieben, durchgeführt wurde , zu detektieren und equid herpesvirus-2 in Atmungsflüssigkeiten quantifizieren. 1 stellt einen schematischen Arbeitsablauf - Diagramm für die Entwicklung und Validierung eines quantitativen RT-PCR - Verfahren nach der AFNOR - Norm NF U47-600. Spezifität der Primer und Sonden wurden während der Schritt-für-Schritt Entwicklung der PCR validiert. Nur EHV-2-Stämme wurden in diesem System amplifiziert. Anschließend war die Leistung der qRT-PCR charakterisiert.
Erstens, die LOD PCR abzuschätzen, a 6 zehnfachen Reihenverdünnung durchgeführt wurde , das Minderungszone (Figur 2) zu schaffen. In diesem Beispiel 6 zehnfache serielle Verdünnungen wurden zwischen 10 -5 und 10 bis 10 (zwischen 26.000 und 0,26 Kopien / 2,5 & mgr; l - Probe) zu schätzen , die LOD PCR hergestellt. Die Minderungszone lies zwischen Verdünnungen von 10 -9 und 10 -10 (zwischen 2,6 und 0,26 Kopien / 2,5 ul Probe). Um die LOD PCR - Wert in diesem Fall 6 zweifache serielle Verdünnungen des Plasmids bestimmen wurden in dieser Minderungszone zwischen 5,2 und 0,16 Kopien / 2,5 ul Probe gemacht. Die LOD PCR - Wert lag bei 2,6 Kopien / 2,5 ul Probe.
Um den Linearitätsbereich und LOQ PCR bestimmen, wurde die LOD PCR - Wert verwendet , um den Bereich von 6 zehnfache Reihenverdünnungen zu starten, zwischen 2,6 (LOD PCR) und 260.000 Kopien / 2,5 & mgr; l Probe. 3 veranschaulicht eine lineare Regression für die EHV2 qRT-PCR von einer Studie. Die Leistungen der linearen Regression (Abbildung 4) sind in vierfacher Ausfertigung validiert unter Verwendung der beschriebenen Berechnungen in Tabelle 3. Die Berechnungen durchgeführt werden , nach den Linearitätsbereich zu definieren , um die Kriterien absolute Bias i value ≤ 0,25 log 10, unabhängig von der Ebene i von Plasmid - Last. In diesem Fall Bereich lag die Linearität zwischen 2,6 und 260.000 Kopien / 2,5 ul Probe. LOQ PCR ist die niedrigste Konzentration in der Linearitätsbereich (dh 2,6 Kopien / 2,5 & mgr; l Probe in diesem Fall). U LIN wurde 2.6-260,000 Kopien / 2,5 ul DNA zu 0,12 log 10 im Bereich bestimmt.
Nach der Entwicklung (Abbildung 1, blau) und Charakterisierung der qRT-PCR (Abbildung 1, gelb), empfiehlt die AFNOR NF U47-600 norm Charakterisierung des gesamten Analyseverfahrens von DNA Extraktion qRT-PCR (Abbildung 1, orange). Die diagnostische Sensitivität und Spezifität wurden in Tabelle 4. Die quantitativen Leistungen der qRT-PCR ganze analytische Verfahren , wie es berechnet wurde mit einer Genauigkeit Profil ausgewertet und validiert ( Abbildung 5).
Dieses Protokoll, das state-of-the-Art molekularen Technologie nutzt, erlaubt uns die EHV-2-Virusgenom Last in 172 Nasentupferproben von Pferden mit Atemwegserkrankungen und / oder klinischer Verdacht auf Infektion erhalten nachzuweisen und zu quantifizieren. Die Inzidenz von EHV-2 aus dem Feld (biologischen) Proben betrug 50% (86/172) in dieser Population. Die quantitativen Analysen zeigten , dass virale Genom Lasten von EHV-2 bei jungen Pferden waren signifikant höher und die Neuverteilung der viralen Genoms Lasten verringert mit dem Alter (Abbildung 6). In der vorliegenden Studie wurde die höchste EHV-2 Genom virale Belastung (1,9 x 10 11 Kopien / ml) wurde in Fohlen (Abbildung 6) detektiert.
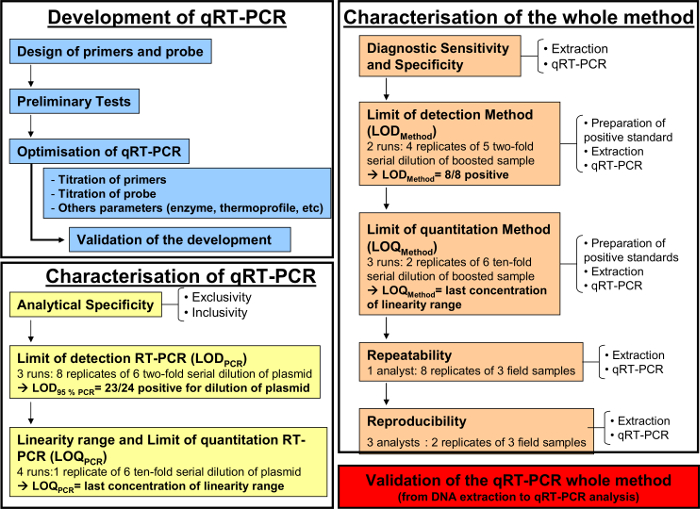
Abbildung 1: Workflow - Diagramm für die Entwicklung (blau), die Charakterisierung der quantitativen RT-PCR(gelb) und die Charakterisierung des gesamten Analyseverfahrens von DNA Extraktion qRT-PCR (orange) gemäß der AFNOR - Norm NF U47-600-2. Der Arbeitsablauf - Diagramm nimmt die verschiedenen Schritte für die Entwicklung, die Charakterisierung der quantitative RT -PCR und die Charakterisierung des gesamten Analyseverfahrens von DNA Extraktion qRT-PCR. Für jeden Schritt, die Anzahl der erforderlichen Durchläufe zeigt das Workflow - Diagramm, führen zu Verdünnungen und die Anzahl der benötigten Analysten. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
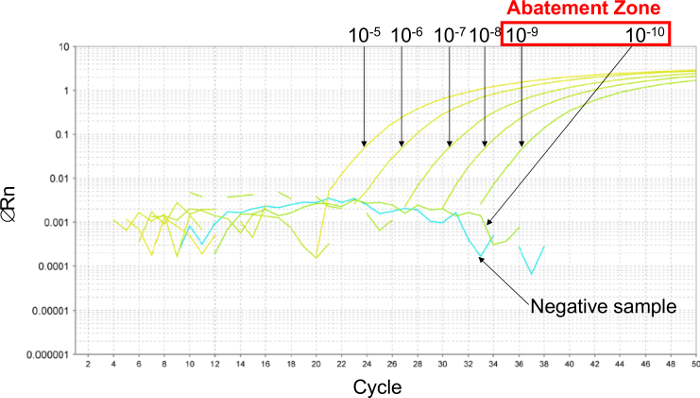
Abbildung 2: Bestimmung der Bekämpfungszone mit repräsentativen Ergebnissen von real-time PCR - Kurven mit 6 zehnfache serielle Verdünnungen von Plasmid erhalten. Um die Minderungszone schätzen, 6 zehnfache SerienVerdünnungen werden zwischen 10 -5 (26.000 Kopien / 2,5 ul Probe) und 10 bis 10 (0,26 Kopien / 2,5 ul Probe) hergestellt. Die Vermeidungszone liegt zwischen Verdünnungen von 10 -9 (2.6 Kopien / 2,5 ul Probe) und 10 bis 10 (0,26 Kopien / 2,5 ul Probe). In diesem Fall 6 zweifache serielle Verdünnungen von Plasmid wurden in dieser Minderungszone machte die LOD 95% PCR, zwischen 5,2 und 0,16 Kopien / 2,5 ul Probe. , Um zu bestimmen Bitte klicken hier , um eine größere Version dieser Figur.
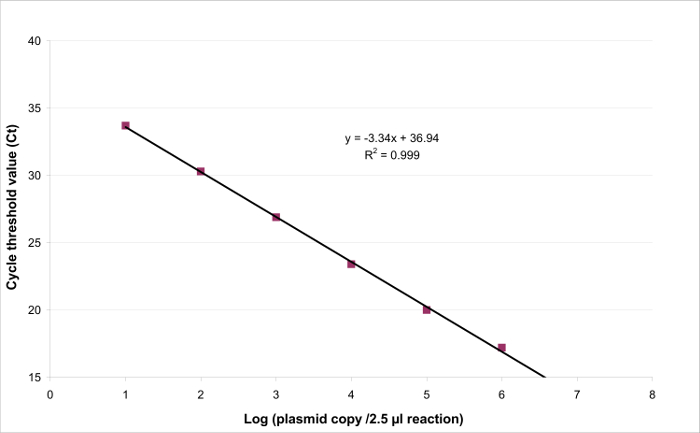
Fig . 3: Die lineare Regression für EHV2 qRT-PCR Die Linearität der quantitativen Tests ist die Fähigkeit , Ergebnisse zu erzeugen , die in einem bestimmten Bereich auf die Konzentration des Ziel proportional sind. Dies kann dadurch modelliert werden,lineare Regression (y = ax + b) zwischen der Instrumentenantwort (Zyklus Schwelle oder Ct) und dem Logarithmus der Menge des Ziels (Anzahl der Ziel Kopien / 2,5 ul Probe). Bitte hier klicken um eine größere Version dieser Figur zu sehen .
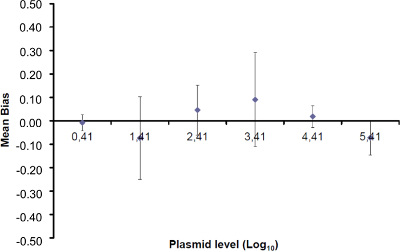
Abb . 4: Durchführung der linearen Regression von EHV-2 qPCR mittlere Vorspannung stellen die mittlere Differenz zwischen der gemessenen Menge Plasmid ( 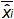 ) Und die theoretische Plasmid Menge (x 'i) für jedes Plasmid - Ebene. Vertikale Balken repräsentieren die Linearität Unsicherheit (U Lini) durch die Formel
) Und die theoretische Plasmid Menge (x 'i) für jedes Plasmid - Ebene. Vertikale Balken repräsentieren die Linearität Unsicherheit (U Lini) durch die Formel 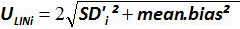
wo SD'i ist die Standardabweichung o f gemessen Plasmid Menge. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
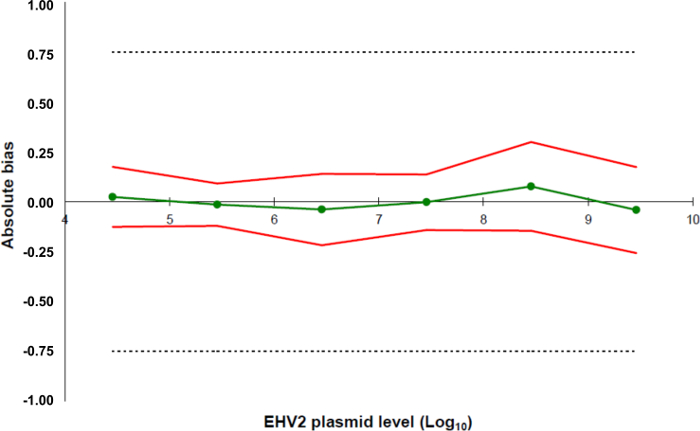
Abbildung 5:. Genauigkeit Profile auf der Basis der Validierungsergebnisse des EHV-2 qRT-PCR - Methode Die grüne Linie (Kreise) stellt die Richtigkeit der Daten (systematischer Fehler oder Bias). Die Akzeptanzgrenzen sind bei ± 0,75 log 10 durch das Labor (gestrichelte Linien) definiert. Die untere und die obere Genauigkeitsgrenzen für jedes Plasmid Lastniveau aus dem zweimal bedeuten Bias bestimmt ± Standardabweichung der Zuverlässigkeitsdaten (rote Linien). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
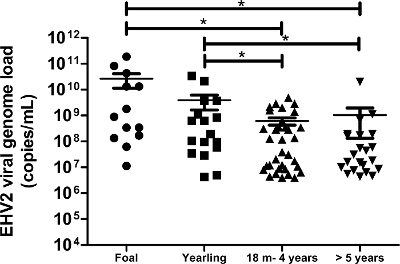
Abbildung 6:. Die Quantifizierung der viralen Genoms Lasten von EHV-2 nach Alter Das virale Genom Lastverteilung von EHV-2 in Nasentupferproben nachgewiesen wird für die verschiedenen Altersgruppen vertreten. Die horizontalen Linien stellen die Medianwerte innerhalb der Standardabweichung (m = Monate). * Signifikant verschieden von ANOVA mit Newman-Keuls post-hoc - Test (p <0,05). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| Zielgen | Grundierungen, Sonde und Plasmid - Sequenzen (5'-3 ') | Nukleotidposition | Produktgröße (Nukleotide) | Thermozyklusbedingungen | Referenzen | |||
| EHV2 gB (HQ247755.1) | Forward: GTGGCCAGCGGGGTGTTC | 2113-2130 | 78 | 95 ° C 5 min | 11 | |||
| Reverse: CCCCCAAAGGGATTYTTGAA | 2189-2170 | 95 ° C 15 sec | 45 Zyklen | |||||
| Sonde: FAM-CCCTCTTTGGGAGCATAGTCTCGGGG-MGB | 2132-2157 | 60 ° C 1 min | ||||||
| Plasmid: ACCTGGGCACCATAGGCAAGGTGGTGGTCA ATGTGGCCAGCGGGGTGTTCTCCCTCTTTG GGAGCATAGTCTCGGGGGTGATAAGCTTTTT CAAAAATCCCTTTGGGGGCATGCTGCTCATA GTCCTCATCATAGCCGGGGTAGTGGTGGTG TACCTGTTTATGACCAGGTCCAGGAGCATAT ACTCTGCCCCCATTAGAATGCTCTACCCCGG GGTGGAGAGGGCGGCCCAGGAGCCGGGCG CGCACCCGGTGTCAGAAGACCAAATCAGGA ACATCCTGATGGGAATGCACCAATTTCAG | 2081-2381 | |||||||
Tabelle 1:. Die Sequenzen der Primer, Sonden und positive synthetische DNA Kontrollen in diesem Protokoll verwendet Die Sequenz von Plasmid (positive synthetische DNA) entspricht Positionen 2081 bis 2381 der Sequenz EHV2gB bis Nukleotid (HQ247755.1). Das Design von Primern und Sonden in diesem Protokoll verwendet wurde, durch Verwendung spezieller Software erhalten.
| PATHOGENS | Referenz (Ursprung) | Anzahl der Stämme | ERGEBNISSE |
| EHV-2 | |||
| EHV-2 | VR701 (ATCC) | 20 | Positiv |
| 20 Proben (FDL-Sammlung) | |||
| EHV-5 | KD05 (GERC) | 20 | Negativ |
| 20 Proben (FDL-Sammlung) | |||
| EHV-3 | VR352 (ATCC) | 2 | Negativ |
| T934 WSV (GERC) | |||
| EHV-1 | Kentucky Stamm Ky A (ATCC) | 3 | Negativ |
| 2 Proben (FDL-Sammlung) | |||
| EHV-4 | VR2230 (ATCC) | 1 | Negativ |
| Asinine herpesvirus AHV5 | FDL-Sammlung | 1 | Negativ |
| PferdeInfluenza-Virus | A / Pferde / Jouars / 4/2006 (H3N8) | 1 | Negativ |
| (Zugangsnummer JX091752) | |||
| Equine Arteritis Virus | VR796 (ATCC) | 2 | Negativ |
| Rhodococcus equi | FDL-Sammlung | 1 | Negativ |
| Streptococcus equi subsp. zooepidemicus | FDL-Sammlung | 1 | Negativ |
| Streptococcus equi subsp. equi | FDL-Sammlung | 1 | Negativ |
| Coxiella burnetii | ADI-142-100 (Adiagene) | 1 | Negativ |
| Chlamydophila abortus | ADI-211-50 (Adiagene) | 1 | Negativ |
| Klebsiella pneumoniae | FDL-Sammlung | 1 | Negativ |
Tabelle 2: Analytische Spezifität der qRT-PCR für EHV-2.
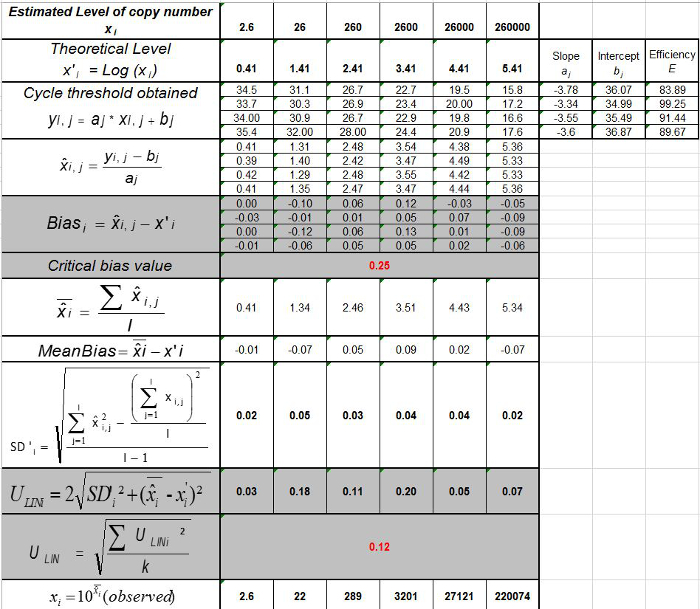
Tabelle 3: Berechnung der Vorspannung und Linearität Unsicherheit (angepasst von NF U47-600-2 12). Für jeden Versuch, die Leistungen der linearen Regression (y = ax + b) werden unter Verwendung der Tabelle validiert , wobei y der Zyklus Schwellenwert erreicht ist, eine die Neigung erhalten wird;. X ist das Plasmid Ebene und b ist der Schnittpunkt i ist das Plasmid Pegel (i variiert von 1 bis k Stufen); k die Anzahl der Plasmid - Ebenen verwendet wird (beispielsweise k = 6 in tseinen Tisch); j der Studie (j von 1 bis I - Studien variiert); I ist die Anzahl der Versuche, zwischen 3 und 6 Studien (zB I = 4 in dieser Tabelle) x i die geschätzte Plasmid Menge für jeden ist. i Plasmid - Ebene. x i 'i die theoretische Gleichung x erhaltene Plasmid Menge ist' = 10 log (x i) für jedes i Ebene Plasmid. Während jeder j - Studie erhalten die Zyklus Schwellenwert für jedes i Plasmid-Ebene wird mit der linearen Regression berechnet, y i, j = a j x i, j + b j. 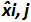 ist die gemessene Menge Plasmid während des Prozesses j. Bias i sub> ist der Unterschied zwischen der gemessenen Plasmid Menge und der theoretischen Plasmid Menge für jeden Versuch und jedes Plasmid Ebene beobachtet.
ist die gemessene Menge Plasmid während des Prozesses j. Bias i sub> ist der Unterschied zwischen der gemessenen Plasmid Menge und der theoretischen Plasmid Menge für jeden Versuch und jedes Plasmid Ebene beobachtet. 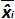 ist der Mittelwert von
ist der Mittelwert von 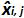 von jeder i - Ebene Plasmid; SD 'i die Standardabweichung der Messgröße ist
von jeder i - Ebene Plasmid; SD 'i die Standardabweichung der Messgröße ist 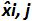 für jedes i Plasmid - Ebene; Mittlere Vorspannung ist der Mittelwert von Bias i; U Lini die Linearität Unsicherheit ist für jedes i - Ebene aus SD'i berechnet Plasmid bestimmt und die mittlere Bias. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
für jedes i Plasmid - Ebene; Mittlere Vorspannung ist der Mittelwert von Bias i; U Lini die Linearität Unsicherheit ist für jedes i - Ebene aus SD'i berechnet Plasmid bestimmt und die mittlere Bias. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Tabelle 4:. Berechnung der diagnostischen Sensitivität (Se) und Spezifität (Sp) des gesamten Verfahrens A Schwartz Tabelle verwendet , um die confid zu berechnenrenzIntervall bei 95% der Empfindlichkeit und Spezifität des gesamten Verfahrens wie in NF U47-600-2 beschrieben.
Diskussion
Seit den 2000er Jahren, Echtzeit-PCR wurde ersetzt Goldstandardtechniken (Zellkultur und Bakterienkultur Methoden) in einer wachsenden Zahl von Laboratorien. Die Umsetzung der Technik ist relativ einfach. Jedoch Validierung von Laborverfahren ist wesentlich für die molekulare Erkennung und Quantifizierung von Pathogenen genaue, wiederholbare und zuverlässige Daten zu gewährleisten.
Da der Extraktionsschritt die Hauptquelle für den Verlust der biologischen Material ist, kann es die Hauptfehlerquelle der Quantifizierung zwischen einem Protokoll und einem anderen betrachtet. Als solche kann die Schaffung einer Standardkurve von DNA Plasmids während qRT-PCR, vor allem in der Literatur berichtet wurde, zeigt die Viruslast Genom aber nicht berücksichtigt nicht der Extraktionsschritt.
Beschreibung eines De - novo - Strategie für eine ganze Methode Validierungsprozess in der AFNOR - Norm NF U47-600-2 stellt einen bedeutenden Fortschritt auf diesem Gebiet. Wie veranschaulicht indieses Papier für EHV-2 bei Pferden oder von anderen in Bienen 21 erfordert diese klare Unterscheidung zwischen dem Entwicklungsschritt und der Validierungsschritt mit Charakterisierung der PCR und Charakterisierung des gesamten Verfahrens. Eine Einschränkung in diesem interessanten Ansatz ist, dass jede Änderung des Protokolls in der Verpflichtung führen werden, den gesamten Prozess erneut zu validieren, die sehr teuer sein könnte. Diese Begrenzung wurde auch durch die Tatsache hervorgehoben , dass die Grenzen der Quantifizierung auf der Quelle abhängen , aus dem das Virus extrahiert wird (beispielsweise Atmungsflüssigkeiten, Organen, Blut oder Urin). In der Tat stellt jede Matrix unterschiedliche Spezifitäten in ihren physikalisch-chemischen Eigenschaften und es ist wichtig, unabhängig jede unterschiedliche Matrix für virale Detektion und Quantifizierung durch qRT-PCR verwendet zu definieren. Somit kann das virale Genom Last jeder biologischen Probe genauer aus der Extraktion werden quantifiziert. Die Charakterisierung berücksichtigt auch die Thermocycler model und wenn die Verwendung eines zuvor gut charakterisierten Verfahren (beispielsweise das EHV-2 qPCR Verfahren in diesem Papier beschrieben) , um eine neue Art von Maschine in Laboratorium Mutter erfordert oder einem anderen Labor, muß man die Leistung des Instruments zu bestätigen. Die Bestätigung der Durchführung eines Assays qPCR ist Voraussetzung für alle Tests in einem Labor zu bringen. Dies wird normalerweise durch die Analyse einer Bezugsprobe mit bekannten Eigenschaften erreicht. Eine solche Prüfung ist Voraussetzung und zwingend angesehen, wie durch die NF 47-600-1 AFNOR-Norm gefordert, um die Leistung der qPCR (LOD, LOQ Effizienz) und die Robustheit des gesamten Verfahrens (LOD, LOQ) zu validieren. Nicht nur während der Entwicklung und Charakterisierung Schritte sondern auch, wenn in der Forschung oder für diagnostische Zwecke verwendet werden, können die Risikofaktoren identifiziert werden und gut kontrollierte Standardisierung des Protokolls zu gewährleisten. Von besonderer Bedeutung ist eine angemessene Ausbildung des Personals, hoch qualifiziertes Personal, Qualitätskontrolle der Verbrauchsmaterialiengebrauchte und ihre Lagerung, Kontrolle über die unmittelbaren Umgebungsbedingungen und das Bewusstsein für messtechnische Bedingungen, die in dem Test beteiligt, die Leistung der wissenschaftlichen Instrumente beeinflussen können. Verwendung von Referenzproben für die Interlabor-Vergleiche könnten auch helfen, die Unsicherheiten zu steuern. Auf diese Weise Vergleich von Daten zwischen Laboratorien kann erleichtert werden. Tatsächlich sind Interlabor-Eignungsprüfungen unerlässlich, um die Reproduzierbarkeit des Verfahrens zu bewerten und zu bestätigen.
Viralen Genoms Last Ergebnisse, die in internationalen Einheiten (IE) der analysierten biologischen Matrix ausgedrückt werden (IU: Kopien / ml für Flüssigkeiten oder Kopien / g Gewebe) sind einfacher zu verwenden, um Ergebnisse zwischen verschiedenen Labors zu vergleichen. Alle Ergebnisse über dem LOQ als Kopien ausgedrückt / ml und ein Ergebnis zwischen dem LOD und LOQ genommen wird als nicht quantifizierbare positive Ergebnis. quantificational Daten des Genoms Presenting entspricht mehr auf diese Weise genau auf die Vorarbs von Analysen (Amplifikation des Genoms). In der Tat, in Zellkulturexperimenten, die Expression der Viruslast durch TCID 50 (mittlere Gewebekulturinfektionsdosis) von der Art der Zellen und Virusstämme abhängt. Jeder Stamm Linie besitzt eine einzigartige Infektion Kinetik und einige Viren wie EHV-2 kann mehrere Tage dauern, bis die ersten zytopathogener Effekt ist offensichtlich.
Abschließend diese neue Methode zur Charakterisierung von qRT-PCR sollte die Harmonisierung der Datendarstellung und Interpretation zwischen den Labors erleichtern. Dies wird für potenzielle neue Anwendungen der qRT-PCR in Zukunft wie die Einrichtung eines Cut-off-Wert für die Erklärung der Krankheitsstatus sehr nützlich sein, anstatt nur das Vorhandensein oder Fehlen des Erregers.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen haben.
Danksagungen
The authors would like to thank Sophie Castagnet and Nadia Doubli-Bounoua for their technical support. This work received financial support from the General Council of Calvados and the agreement of Region Basse-Normandie and French Government (CPER 2007-2013; project R25 p3). The authors would like to thank the experts of the AFNOR group and particularly Jean-Philippe Buffereau and Eric Dubois.
Materialien
| Name | Company | Catalog Number | Comments |
| AB-1900 natural color ABgene 96 well plate | Dutsher | 16924 | |
| Adhesive film qPCR Absolute | Dutsher | 16629 | Adhesive film used for sealing the plate prior to the qRT-PCR run |
| 0.5 ml microtubes, skirted, caps | Dutsher | 039258 | |
| Ethanol 98% | Sodipro | SAF322941000 | |
| Primers | Eurofins | Custom order | |
| Probe | Life Technologies | Custom order | |
| Plasmid | Eurofins | Custom order | |
| QIAamp RNA viral Mini Kit (containing: QIAamp Mini column, AVL buffer, AW1 buffer, AW2 buffer, AVE buffer, collection tubes) | Qiagen | 52906 | AVL buffer: pre-warm 5 min at 72 °C |
| Sequencing by Sanger method | Eurofins | Custom order | |
| Taqman Universal PCR Master Mix | Life Technologies | 4364340 | |
| Tris-EDTA buffer solution | Santa Cruz | sc-296653A | |
| NanoDrop 2000c Spectrophotometer | Thermoscientific | ND-2000C | |
| StepOnePlus Real-Time PCR systems | Life Technologies | 4376600 | pre-warm 15 min |
Referenzen
- Brault, S. A., et al. The immune response of foals to natural infection with equid herpesvirus-2 and its association with febrile illness. Vet.Immunol.Immunopathol. 137 (1-2), 136-141 (2010).
- Fortier, G., Van Erck, E., Pronost, S., Lekeux, P., Thiry, E. Equine gammaherpesviruses: pathogenesis, epidemiology and diagnosis. Vet.J. 186 (2), 148-156 (2010).
- Hue, E. S., et al. Detection and quantitation of equid gammaherpesviruses (EHV-2, EHV-5) in nasal swabs using an accredited standardised quantitative PCR method. J Virol.Methods. 198 (1), 18-25 (2014).
- Diallo, I. S., et al. Multiplex real-time PCR for the detection and differentiation of equid herpesvirus 1 (EHV-1) and equid herpesvirus 4 (EHV-4). Vet.Microbiol. 4 (1-3), 93-103 (2007).
- Williams, K. J., et al. Equine multinodular pulmonary fibrosis: a newly recognized herpesvirus-associated fibrotic lung disease. Vet.Pathol. 44 (6), 849-862 (2007).
- Mullis, K., et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb.Symp.Quant.Biol. 51 (1), 263-273 (1986).
- EPA Office of Water (4607). EPA-815-B-04-001. Quality Assurance/Quality Control Guidance for Laboratories Performing PCR Analyses on Environmental Samples. , (2004).
- Telford, E. A., et al. Equine herpesviruses 2 and 5 are gamma-herpesviruses. Virology. 195 (2), 492-499 (1993).
- Fortier, G., et al. Identification of equid herpesvirus-5 in respiratory liquids: A retrospective study of 785 samples taken in 2006-2007. Vet.J. 182 (2), 346-348 (2009).
- Brault, S. A., Bird, B. H., Balasuriya, U. B., MacLachlan, N. J. Genetic heterogeneity and variation in viral load during equid herpesvirus-2 infection of foals. Vet.Microbiol. 147 (3-4), 253-261 (2011).
- Association Francaise de Normalisation. NFU 47-600-1. Animal health analysis methods-PCR-Part 1: Requirements and recommandations for the implementation of veterinary PCR. , (2015).
- Association Francaise de Normalisation. NFU 47-600-2. Animal health analysis methods-PCR-Part 2: Requirements and recommendations for the development and the validation of veterinary PCR. , (2015).
- ISO. EN ISO-CEI 17025. General requirements for the competence of testing and calibration laboratories. , (2005).
- Manual of Diagnostic Tests and Vaccines for Terrestrial Animals 2010. Chapter1.1.1.4/ 5. Principles and Methods of Validation of Diagnostic Assay for Infectious Diseases. , This thoroughly revised chapter replaces Chapter 1.1.4 Principles of validation of diagnostic assays for infectious diseases and Chapter 1.1.5 Validation and quality control of polymerase chain reaction methods used for the diagnosis of infectious diseases from the sixth edition of the OIE Terrestrial Manual (2009).
- Apaza, S., et al. Detection and genogrouping of noroviruses from children's stools by Taqman One-step RT-PCR. J Vis.Exp. (65), e3232(2012).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis.Exp. (63), e3998(2012).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol.Biol. 94 (3), 441-448 (1975).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc.Natl.Acad.Sci.U.S.A. 74 (12), 5463-5467 (1977).
- NCBI. BLAST Homepage and Selected Search Pages. Introducing the BLAST homepage and form elements/functions of selected search pages. , (2015).
- Greiner, M., Gardner, I. A. Application of diagnostic tests in veterinary epidemiologic studies. Prev.Vet Med. 45 (1-2), 43-59 (2000).
- Blanchard, P., Regnault, J., Schurr, F., Dubois, E., Ribiere, M. Intra-laboratory validation of chronic bee paralysis virus quantitation using an accredited standardised real-time quantitative RT-PCR method. J Virol.Methods. 180 (1-2), 26-31 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten