Method Article
Développement et validation d'une méthode de PCR quantitative pour équidé herpèsvirus-2 Diagnostics dans respiratoires Fluides
Dans cet article
Résumé
Here, we present a protocol for the development and validation of a quantitative PCR method used for the detection and quantification of EHV-2 DNA in equine respiratory fluids. The EHV-2 qRT-PCR validation protocol involves a three-part procedure: development, characterization of qRT-PCR assay alone, and characterization of the whole analytical method.
Résumé
The protocol describes a quantitative RT-PCR method for the detection and quantification of EHV-2 in equine respiratory fluids according to the NF U47-600 norm. After the development and first validation step, two distinct characterization steps were performed according to the AFNOR norm: (a) characterization of the qRT-PCR assay alone and (b) characterization of the whole analytical method. The validation of the whole analytical method included the portrayal of all steps between the extraction of nucleic acids and the final PCR analysis.
Validation of the whole method is very important for virus detection by qRT-PCR in order to get an accurate determination of the viral genome load. Since the extraction step is the primary source of loss of biological material, it may be considered the main source of error of quantification between one protocol and another. For this reason, the AFNOR norm NF-U-47-600 recommends including the range of plasmid dilution before the extraction step. In addition, the limits of quantification depend on the source from which the virus is extracted. Viral genome load results, which are expressed in international units (IU), are easier to use in order to compare results between different laboratories.
This new method of characterization of qRT-PCR should facilitate the harmonization of data presentation and interpretation between laboratories.
Introduction
Equid herpesvirus-2 (EHV-2) est impliqué dans un syndrome respiratoire, avec des manifestations cliniques potentielles telles que l' écoulement nasal, la pharyngite et des ganglions lymphatiques enflés 1-3. Ce virus est également soupçonné d'être associé à la mauvaise performance des chevaux, ce qui peut entraîner un impact économique significatif et négatif pour l'industrie du cheval 2.
Jusqu'à présent, l'étalon-or pour le gamma-EHV (γ-EHV) la détection a été la méthode de culture cellulaire. Le premier inconvénient de cette procédure était l'absence de discrimination entre EHV-2 et d' autres γ-EHV (par exemple EHV-5). Le second inconvénient est la lenteur du développement du processus cytopathologique, qui prend de 12 à 28 jours pour manifester 4,5.
Élaboration d'un temps réel de réaction en chaîne par polymérase quantitative validée et normalisée (qRT-PCR) permettra de détecter rapidement le virus, afin de discriminer entre EHV-2 et EHV5, et d'étudier la relation entre la charge du génome viral et la maladie grâce à l'aspect de quantification.
Réaction en chaîne par polymérase (PCR) a été décrite pour la première fois en 1986 par Mullis 6 et est sur le point de devenir la nouvelle norme d'or dans la plupart des domaines du diagnostic biologique (humaine, l' environnement et vétérinaire). Cette méthode, qui est basée sur l'amplification d'une partie du génome d'agents pathogènes, présente de nombreux avantages: la spécificité, la sensibilité et la rapidité. En outre, le risque de contamination de l' amplicon a reculé depuis l'avènement de qRT-PCR et l' assurance de la qualité 7. Néanmoins, la reconnaissance de la PCR comme une nouvelle méthode de l'étalon-or a nécessité plus qu'une simple amélioration des données de performance, mais aussi la démonstration du contrôle du développement et de validation des étapes du procédé entier sans dégradation des performances au fil du temps.
Les premiers outils moléculaires utilisés pour la détection de EHV-2 ont été temps camplification non spécifique onsuming et impliqué dans la PCR nichée suivie d' un séquençage 8. Les gènes ciblés pour les virus de l' herpès sont l' acide désoxyribonucléique (ADN) polymérase et l' ADN emballage 9. Cependant, la PCR nichée présente un risque élevé de contamination par des amplicons. Depuis lors, les tests PCR conventionnels ont été conçus pour amplifier l'interleukine gène 10-like ou gène de la glycoprotéine B, révisé en 2009 2. Plus récemment, les caractéristiques de PCR en temps réel ont été décrits pour la quantification de EHV-2 10 mais aucune donnée étaient disponibles relatives à la validation de la méthode incluant l'ensemble du processus d'extraction.
Dans ce protocole, les procédures de développement et de validation sont décrits pour une méthode PCR quantitative pour la détection et la quantification de EHV-2 ADN dans les fluides respiratoires équins selon l'Association française de normalisation (AFNOR) norme NF U47-600 3,11,12, qui est le représentant françaisle comité international de normalisation. Cette norme détaille les «Exigences et recommandations pour la mise en œuvre, le développement et la validation de PCR vétérinaire animale méthode d'analyse de la santé» 11,12, selon la norme NF EN ISO / CEI 17025 2005 13 et à l' OIE (Organisation mondiale de la santé animale) . recommandations 2010 14 le qRT-PCR protocole de validation EHV-2 implique une procédure en trois parties: (a) le développement du dosage qRT-PCR, (b) la caractérisation de l' essai qRT-PCR seul et (c) la caractérisation de l'ensemble méthode analytique (provenant de l'extraction des acides nucléiques de l'échantillon biologique pour l'analyse PCR).
La caractérisation de l'essai qRT-PCR et de toute méthode d'analyse comprend la définition de deux limites: la limite de détection (LOD) et la limite de quantification (LQ). La limite de détection de 95% PCR représente le plus faible nombre de copies d'acide nucléique par unité de volume qui peuvent être détectés dans 95% de tous les NCAes. La LQ 95% PCR représente la plus faible quantité de copies d'acides nucléiques qui peuvent être déterminés en tenant compte des incertitudes.
Cette méthode qRT-PCR permet la détection précise et la quantification rapide de EHV-2 dans les fluides respiratoires. En outre, le procédé peut être appliqué dans d'autres laboratoires afin d'assurer une procédure normalisée et un modèle général pour que le développement d'autres nouveaux tests qRT-PCR.
Protocole
Remarque: Reportez - vous à toutes les différentes étapes qui sont illustrés dans la Figure 1.
1. Extraction des acides nucléiques
Remarque: Effectuez l'extraction sous une hotte pour limiter la contamination des voies respiratoires avec des acides nucléiques. Inclure un contrôle négatif d'extraction avec de l'eau traitée par DEPC pour veiller à ce qu'aucun des réactifs sont contaminés avec de l'ADN non désirées.
- Extraire les acides nucléiques de l'échantillon biologique selon un protocole protocole 15 du fabricant et décrits précédemment.
- Ajouter 140 ul d'échantillon biologique à 560 ul de solution de lyse (tampon AVL) et incuber pendant 10 min à température ambiante. Ajouter 560 ul d'éthanol. Appliquer les premiers 630 pi de cette solution (échantillon + solution de lyse + éthanol) à une colonne de silice et on centrifuge.
- Appliquer les 630 ul restants de cette solution dans la colonne de silice et même centrifugeuse. Laver ensuite la colonne avec 500 pide 2 tampons de lavage différentes (AW1 et AW2).
- Éluer l'acide nucléique avec 50 pi de tampon d'élution (tampon AVE) et équilibrer à la température ambiante. Fermez le bouchon et incuber à température ambiante pendant 1 min. Centrifuger à 6000 g pendant 1 min.
2. Procédure Amplification
- Préparer 22,5 pi de mélange réactionnel pour chaque réaction. Ajouter 12,5 ul de PCR Master Mix, x pi de 20 pM amorce avant, x pi de 20 pM amorce inverse, y ul de sonde de 10 uM et z ul eau ultrapure comme nécessaire pour atteindre 22,5 pi (x, on obtient les volumes y et z après titrage, voir les sections 3.2.3 et 3.2.6).
- Aliquoter 22,5 ul du mélange de réaction appropriée à chaque puits de réaction dans une plaque de 96 puits.
Note: Inclure les contrôles négatifs pour l'extraction et pour la PCR pour veiller à ce qu'aucun des réactifs sont contaminés avec de l'ADN non désirées. - Ajouter 2,5 pi de l'échantillon, 2,5 pi d'extraction negativcontrôle e, 2,5 pi de PCR témoin négatif et 2,5 pi d'échantillon positif (souche de référence ou plasmide) à la réaction correspondante bien. Après la distribution, couvrir la plaque avec un joint de plaque adhésive. Centrifuger la plaque pendant 10 secondes à 6000 g.
- Placer la plaque dans un système PCR en temps réel. Sélectionnez le modèle pour la mise en page de test et commencer la course. Utiliser le paramètre de programme de PCR: 10 min à 95 ° C , puis 45 cycles de 15 s à 95 ° C et 1 min à 60 ° C (tableau 1).
- Transférer les données brutes du système de PCR en temps réel à une feuille de calcul. Régler le seuil dans les graphes d'amplification au-dessus du niveau de référence et dans la région de croissance exponentielle pour obtenir le cycle seuil pour chaque échantillon. Tracer chaque norme de point de consigne comme une courbe standard pour obtenir la linéarité. Calculer le nombre de copie pour les différents échantillons en fonction de la courbe étalon.
3. Développement de RT-PCR quantitative
Remarque: Le développement d'un test qRT-PCR nécessite des souches de référence, un plasmide spécifique, et titré différents contrôles et entraîne le titrage des amorces et la sonde.
- Essai préliminaire
- Amorces de conception et sondes avec des logiciels spécifiques selon les recommandations précédentes 16.
- Extrait des souches de référence comme décrit dans la section 1.
- Amplifier comme décrit dans la section 2 avec 900 nM pour la concentration finale d'amorces et 250 nM pour la concentration finale de la sonde. Analyser le signal tel que décrit dans la section 2.5.
- Dans le même temps, effectuer l'amplification de l'ADN des souches de référence (tel que décrit dans la section 3.1.3) sans sondes. Séquencer les amplicons obtenus par la méthode de Sanger 17,18. Analyser les séquences en exécutant un BLAST nucléotidique 19.
- Titration de Amorces et sonde
- Préparer 3 mélanges différents avec 250 nM concentration finale de la sonde et concentr finale différenteations de la marche avant et arrière amorces (50 nM / 50 nM, 300 nM / 300 nM, 900 nM / 900 nM) pour les 3 répétitions de l'échantillon positif et un contrôle négatif. Pour chaque mélange, ajouter le même volume approprié de 20 uM de l'amorce sens et 20 uM d'amorce inverse (0,25 ul pour obtenir la concentration finale 50 nM, 1,5 pl pour obtenir les 300nm concentration finale ou 4,5 ul pour obtenir la concentration finale 900 nM) , 50 pl de PCR master mix, 2,5 pi de sonde de 10 uM et de l'eau ultrapure que nécessaire pour atteindre 90 pi.
- Effectuez la procédure d'amplification tel que décrit dans la section 2.
- Choisissez la meilleure condition pour obtenir le plus haut niveau d'amplification, seuil plus tôt du cycle (Ct) et la meilleure répétabilité entre les 3 conditions. Déterminer la meilleure concentration et le volume correspondant x pi du amorces avant et inverse.
- Préparer 5 mélanges différents pour les 4 échantillons (3 répétitions de l'échantillon positif et un témoin négatif)avec 5 concentrations finales différentes de la sonde (50 nM, 100 nM, 150 nM, 200 nM, 250 nM). Pour chaque mélange, ajouter x pi de 20 pM amorce avant et x pi de 20 pM amorce inverse (précédemment déterminé, à la section 3.2.3), 50 pl de PCR master mix, volume approprié de 10 uM sonde (0,5 pi pour obtenir le 50 concentration en nM, 1 pi pour obtenir la concentration de 100 nM, 1,5 pl pour obtenir la concentration nM 150, 2 ul pour obtenir la concentration de 200 nM ou 2,5 ul pour obtenir la 250 concentration en nM) et de l'eau ultra-pure comme nécessaire pour atteindre 90 ul.
- Effectuez la procédure d'amplification tel que décrit dans la section 2.
- Choisissez la meilleure condition pour obtenir le plus haut niveau d'amplification, seuil plus tôt du cycle (Ct) et la meilleure répétabilité entre les 5 conditions. Déterminer la meilleure concentration et le pl volume y correspondant pour la sonde.
4. Caractérisation du quantitative Real-PCR en temps (qRT-PCR)
Remarque: Après l'étape de développement et de la détermination des meilleures conditions d'utilisation, l'étape de caractérisation de la PCR comprend la spécificité, la limite de détection, la plage de linéarité et la limite de quantification de qRT-PCR.
- Préparation et Titration du plasmide
- Commander un plasmide commercial contenant le fragment correspondant du gène cible d'ADN choisi pour la PCR (dans ce protocole, les nucléotides 2081-2381 de EHV-2 glycoprotéine gène B, voir le tableau 1).
Remarque: Pour limiter le risque de contamination des voies respiratoires avec de l'ADN synthétique, effectuer des dilutions en série de plasmides sous une hotte dans une pièce séparée et de travailler avec l'ADN dilué au cours de toutes les étapes du développement et de la procédure de validation. - Re-suspendre le plasmide avec de l'eau ultrapure pour préparer une solution mère à 50 ng / ml. Vortex et centrifuger brièvement la solution plasmidique de stock.
Remarque: Déterminer la concentration réelle de la sto plasmidiquesolution ck après remise en suspension afin de calculer le nombre de copies du plasmide. - Ajouter 1 pi de plasmide au spectrophotomètre. Lire la densité optique (DO) à 230 nm, 260 nm et 280 nm. Lire la concentration d'ADN sur le logiciel spectrophotomètre (OD, 260 nm).
- Calculer le nombre de copies du plasmide en utilisant le nombre d'Avogadro (N A) et la formule:
Le nombre de copies / pl = (N A [plasmide en ng / pl]) / (longueur de plasmide x 10 9 x poids moyen d'une paire de base) = (6.022 x 10 23 x [plasmide]) / (longueur de plasmide x 10 9 x 660)
- Commander un plasmide commercial contenant le fragment correspondant du gène cible d'ADN choisi pour la PCR (dans ce protocole, les nucléotides 2081-2381 de EHV-2 glycoprotéine gène B, voir le tableau 1).
- Test de la Spécificité (inclusivité et exclusivité) de qRT-PCR
- Testez le inclusivité du système PCR. Choisir des échantillons d'ADN positifs 2 EHV, précédemment caractérisé par séquençage (établi le statut positif).
- Effectuez la procédure d'amplification tel que décrit dans la section 2.
- Analyser leles données PCR pour chaque échantillon comme décrit dans la section 2.5. Vérifiez la présence d'une courbe exponentielle pour tous les échantillons choisis dans la section 4.2.1 et confirmer l'inclusivité de la PCR.
- Testez l'exclusivité du système PCR en utilisant des extraits d'ADN provenant de pathogènes présentant des similitudes génétiques à la cible (dans ce cas, d'autres herpèsvirus équidés tels que EHV-1, EHV-4, EHV-3, EHV-5 et asinine herpesvirus-5 comme décrit dans le tableau 2) et d' autres agents pathogènes impliqués dans les maladies respiratoires de l'hôte (dans ce cas, le virus de l' artérite équine, le virus de la grippe équine, Coxiella burnetii, Rhodococcus equi, Streptococcus equi equi, Streptococcus equi zooepidemicus, Chlamydophila abortus et Klebsiella pneumoniae, voir le tableau 2).
- Effectuez la procédure d'amplification tel que décrit dans la section 2.
- Analyser les données de PCR pour chaque échantillon comme décrit dans la section 2.5. Vérifiez l'absence d'une courbe exponentielle pour tous les échantillons choisissection 4.2.4 pour confirmer l'exclusivité de la PCR.
- Limite de détection de qRT-PCR
- Distribuer 90 pl d'eau ultrapure dans 6 tubes.
- Effectuer 6 dilutions en série de dix fois du plasmide pour cibler la zone de réduction (de perte de détection Ct). Transfert de 10 pl à partir du plasmide dilution de travail dans le tube avec 90 ul d'eau ultrapure. Vortex et centrifuger brièvement le tube. Répétez l'étape 4.3.2 jusqu'à ce que le dernier tube de la dilution en série a reçu le plasmide.
- Effectuer l'amplification des 6 dilutions en série de dix fois du plasmide tel que décrit dans la section 2.
- Déterminer la zone de réduction: la zone située entre la dernière dilution du plasmide présentant un signal positif et la première dilution sans détection (voir la figure 2).
- Pour démarrer les 6 dilutions en série deux fois, choisissez la dernière dilution du plasmide qui donne un signal positif (voir section 4.3.4).
Remarque: Effectuez 3 tri indépendantals pour déterminer la limite de détection de qRT-PCR (LOD 95% PCR) - Distribuer 25 ul d'eau ultrapure dans 6 tubes.
- Effectuer 6 dilutions en série de deux en deux du plasmide. Transfert 25 ul de la dilution de travail plasmidique, tel que déterminé dans la section 4.3.5, le tube avec 25 pi d'eau ultrapure. Vortex et centrifuger brièvement le tube. Répétez l'étape 4.3.7 jusqu'à ce que le dernier tube de la dilution en série a reçu le plasmide.
- Effectuer l'amplification des 6 dilutions en série de deux en deux du plasmide tel que décrit dans la section 2. Répétez les étapes 4.3.7 à 4.3.8 deux fois, pour obtenir 3 essais avec 8 répétitions (24 répétitions) de chacun des 6 dix fois des dilutions en série du plasmide.
Remarque: La limite de détection de la qRT-PCR (PCR à 95% LOD) est défini comme étant le plus petit nombre de copies d'ADN par unité de volume qui est détecté dans 95% des cas. - Calculer le nombre de répétitions positives sur 24 répétitions pour chaque niveau du plasmide concentration.
- Déterminer le LOD 95% PCR. La limite de détection de 95% de PCR est le niveau qui se traduit par la détection de 23 réplicats positifs sur 24 réplicats.
- Linéarité Range et limite de quantification de qRT-PCR
Remarque: Effectuez 4 essais indépendants avec 6 dilutions en série de dix fois du plasmide pour assurer que la plus faible concentration utilisée dans la gamme correspond à la LOD 95% PCR déterminé précédemment dans 4.3.10.- Distribuer 45 pl d'eau ultrapure dans 6 tubes.
- Démarrez les 6 dilutions en série de dix fois avec la concentration du plasmide dilution de travail correspondant à 10 7 LOD 95% PCR.
- Effectuer 6 dilutions en série de dix fois du plasmide. Transfert 5 pi de la dilution de plasmide de travail (déterminé à l'article 4.4.2) au tube avec 45 pi d'eau ultrapure. Vortex et centrifuger brièvement le tube. Répétez l'étape 4.4.3 jusqu'à ce que le dernier tube de la dilution en série a reçu plasmid.
- Amplifier les 6 dilutions en série de dix fois du plasmide tel que décrit dans la section 2.
- Tracer la régression linéaire y = ax + b avec la pente et b pour l'interception (Figure 3).
- Calculer l'efficacité d'amplification (E) à partir de la pente de la courbe d' un standard (voir 4.4.5) en utilisant l'équation:
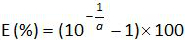 .
.
Répétez les étapes 4.4.1 à 4.4.6 trois fois.
Note: Le rendement d'amplification représente la quantité de produit PCR augmentation après chaque cycle. Une réaction idéale atteint l'efficacité proche de 100%. En pratique, E (%) se situe entre 75% et 125%. Supérieur E peut indiquer l'amplification de produits non spécifiques ou une erreur de pipetage dans la dilution en série. Basse-E peut également indiquer une erreur de pipetage dans la dilution en série, la conception d'amorce pauvres ou des conditions de réaction non optimales. - Calculer le biais (tableau3). Vérifiez pour chaque niveau de plasmide que la valeur de polarisation absolue est inférieure à la valeur de polarisation critique (0,25 log 10). Déterminer le biais moyenne et les incertitudes de linéarité (U Lini) pour chaque niveau de plasmide (tableau 3) pour évaluer la performance de la régression linéaire pour EHV-2 qPCR (Figure 4). U Lini est l'incertitude de linéarité déterminée pour chaque i plasmide de niveau calculé à partir de l' écart - type (SD'i) et biais moyen. Déterminer la linéarité de l' incertitude associée (U LIN) de EHV-2 qPCR donnée par la formule:

Remarque: L' acceptation de partialité est spécifié par le laboratoire (en biais absolu générale 0,25 log 10) et correspondent à la différence entre la valeur la plus faible et la valeur la plus élevée de 0,5 log 10 (quantité mesurée en log 10 nombre de copies). U LIN valeur pourrait aider à comparer les performances de qPCR de différents laboratoires. - Déterminer la limite de quantification de la qRT-PCR (LQ PCR): LQ PCR est la plus faible concentration avec un biais de 0,25 log 10 utilisé pour la gamme de linéarité (tableau 3).
5. Caractérisation de la méthode d'analyse entier (de l'extraction de l'ADN à la Résultat qRT-PCR)
Note: La caractérisation de la méthode globale est la validation de toutes les mesures nécessaires pour obtenir des données qRT-PCR (ie, de l'extraction de l' ADN de l'échantillon respiratoire (voir section 1) à l'amplification et la quantification de la cible (voir la section 2 )).
- Établir une correspondance entre le nombre d'exemplaires de la réaction de PCR ainsi que le nombre de copies d'présents dans l'échantillon biologique à la formule:
 .
.
le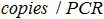 Est le nombre de copies du plasmide dans la réaction PCR,
Est le nombre de copies du plasmide dans la réaction PCR, 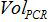 est le volume de l'échantillon ajouté au mélange PCR pour l'amplification,
est le volume de l'échantillon ajouté au mélange PCR pour l'amplification, 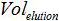 est le volume de tampon AVE utilisé pour éluer les acides nucléiques et
est le volume de tampon AVE utilisé pour éluer les acides nucléiques et 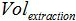 est le volume de l'échantillon extrait.
est le volume de l'échantillon extrait. - Test de la sensibilité et la spécificité de la méthode analytique Whole
Note: EHV-2 échantillons seulement connus positifs (ou négatifs) sont utilisés dans cette section.- Tester la "sensibilité" du procédé qRT-PCR en analysant des échantillons positifs pour la cible (EHV-2).
- Choisir des échantillons d'ADN positifs 2 EHV, caractérisé précédemment (statut positif).
- Extraire l'acide nucléique tel que décrit dans la section 1. Effectuer une procédure d'amplification tel que décrit dans la section 2.
- Déterminer le nombre de vrais positifs (échantillons positifsqui sont positifs à la présente par RT-PCR) et le nombre de faux négatifs (appelés échantillons positifs qui sont négatifs avec cette RT-PCR).
- Testez la «spécificité» de la méthode qRT-PCR en analysant des échantillons négatifs
- Choisir des échantillons d'ADN négatifs 2 EHV-(statut négatif).
- Extraire l'acide nucléique tel que décrit dans la section 1. Effectuer une procédure d'amplification tel que décrit dans la section 2.
- Déterminer le nombre de vrais négatifs (échantillons négatifs qui sont connus pour être négatif avec cette RT-PCR) et le nombre de faux positifs (échantillons négatifs connus qui sont positifs à la présente par RT-PCR).
- Calculer la "sensibilité diagnostique" (Se) et la «spécificité de diagnostic" (Sp) de la méthode globale (tableau 4) comme suit: Se = nombre de vrais positifs / (nombre de vrais positifs + faux négatifs) et Sp = nombre de biens négatifs / (nombre de réel négatifs + faux positifs) (voir le tableau 4).
- Calculer l'intervalle de confiance de 95% pour la sensibilité et la spécificité de l'ensemble du procédé avec la formule de Greiner et Gardner 12,20 relation:
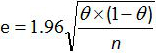
où e est l'erreur estimée, θ est le Se (ou Sp) et n le nombre d'échantillons analysés.
Remarque: Pour un petit nombre d'échantillons, utilisez le tableau Schwartz selon la norme AFNOR 12 pour calculer l'intervalle de confiance de 95% pour la sensibilité et la spécificité de la méthode ensemble.
- Tester la "sensibilité" du procédé qRT-PCR en analysant des échantillons positifs pour la cible (EHV-2).
- Préparation du négatif Matériel de ressource pour la caractérisation de la méthode analytique Whole
ATTENTION: Pour déterminer les limites de détection et de quantification de l'ensemble du procédé (LOD et la méthode LDD), ajouter des concentrations connues du plasmide dans les échantillons biologiques qui sont connus pour être librede la cible (EHV-2 dans ce cas). Ces échantillons, ainsi que le plasmide quantifiée, constituent des normes positives avec lesquelles pour déterminer la méthode LOD et la méthode LQ.- Confirmez l'absence de cible (EHV-2) dans les échantillons biologiques utilisés pour construire des normes positives.
- Choisissez différents échantillons biologiques négatifs connus.
- Extrait des échantillons biologiques tels que décrits dans la section 1. Effectuer la procédure d'amplification tel que décrit dans la section 2.
- Confirmer l'absence de cible en l'absence d'un signal de PCR dans ces échantillons.
Remarque: Utilisez le même matériel de ressource négative pour toutes les étapes entre 5,4 et 5,5.
- Regrouper les différents échantillons négatifs pour obtenir 15 ml de matériau de ressource négative (un volume nécessaire à la validation complète). Distribuer 135 ul de cette matière de ressources négatif en 100 tubes. Garder les tubes à +4 ° C ou à -80 ° C pour un stockage.
- Confirmez l'absence de cible (EHV-2) dans les échantillons biologiques utilisés pour construire des normes positives.
- Limit de détection de la méthode analytique Whole
- Déterminer la zone de réduction de la méthode ensemble.
- Distribuer 45 pl d'eau ultrapure dans 6 tubes.
- Effectuer 6 dilutions en série de dix fois du plasmide. Transfert 5 pi de la dilution de travail plasmidique correspondant à 10 7 LOD 95% PCR (tel que déterminé dans la section 4.3.10) au tube avec 45 pi d'eau ultrapure. Vortex et centrifuger brièvement le tube. Répétez l'étape 5.4.1.2 jusqu'à ce que le dernier tube de la dilution en série a reçu le plasmide.
- Transfert 5 ul de chaque dilution de plasmide dans 2 tubes avec 135 ul de la matière de ressource négative pour obtenir deux répétitions pour les 6 étalons positifs. Vortex et centrifuger brièvement.
- Effectuer l'extraction des 2 répétitions pour les 6 normes positives comme décrit dans la section 1. Effectuer la procédure d'amplification tel que décrit dans la section 2.
- Déterminer la zone de réduction: la zone entre le dernier concntration du plasmide donnant un signal positif et la première concentration qui montre l'absence de signal.
- Limite de détection de la méthode (LOD Méthode) Déterminé par 2 essais indépendants
Remarque: Effectuez 2 essais indépendants pour déterminer la limite de détection de l'ensemble de la méthode (LOD Method).- Démarrer les 6 dilutions en série deux fois avec de la dilution de plasmide qui est 4 fois plus concentrée que la dernière concentration du plasmide qui a donné un signal positif (voir section 5.4.1.5).
- Distribuer 25 pl d'eau ultrapure dans 6 tubes.
- Effectuer 5 dilutions en série de deux en deux du plasmide. Transfert 25 pi du plasmide de travail dilution (tel que déterminé dans la section 5.4.2.1) sur le tube avec 25 pi d'eau ultrapure. Vortex et centrifuger brièvement le tube. Répétez l'étape 5.4.2.3 jusqu'à ce que le dernier tube de la dilution en série a reçu le plasmide.
- Transfert 5 pi de chaque dilution de plasmid à 4 tubes avec 135 ul de la matière de ressource négative pour obtenir 4 réplicats de 5 standards positifs. Vortex et centrifuger brièvement les tubes.
- Effectuer l'extraction des 4 répétitions pour les 5 normes positives comme décrit dans la section 1. Effectuer la procédure d'amplification tel que décrit dans la section 2.
- Répétez les étapes 5.4.2.1 à 5.4.2.5 une fois pour obtenir 8 répétitions pour chaque niveau de concentration de plasmide.
- Comptez le nombre de répétitions positives sur 8 répétitions pour chaque niveau de concentration de plasmide.
- Déterminer la méthode LOD. La méthode LOD est le dernier niveau auquel 8 répétitions de 8 répétitions sont positifs (comme décrit dans la section 5.4.2.7).
- Déterminer la zone de réduction de la méthode ensemble.
- Linéarité Range et limite de quantification de la méthode analytique Whole
Remarque: Pour déterminer la limite de quantification de l'ensemble de la méthode (LQ Méthode), ajouter des concentrations connues du plasmide au biologiqueéchantillons qui sont connus pour être libres de la cible (EHV-2 dans ce cas). Ces échantillons constituent des normes positives pour déterminer la méthode LQ.- Distribuer 45 pl d'eau ultrapure dans 6 tubes.
- Effectuer 6 dilutions en série de dix fois du plasmide. Transfert 5 pi de la dilution de travail plasmidique correspondant à 10 7 LOD Méthode (tel que déterminé dans la section 5.4.2.8) au tube avec 45 pi d'eau ultrapure. Vortex et centrifuger brièvement le tube. Répétez l'étape 5.5.2 jusqu'à ce que le dernier tube de la dilution en série a reçu le plasmide.
- Transférer 5 ul de chaque dilution du plasmide à 2 tubes avec 135 pi de matière négative des ressources biologiques pour obtenir 2 répétitions des 6 normes positives. Vortex et centrifuger brièvement.
- Effectuer l'extraction des 2 répétitions pour les 6 normes positives comme décrit dans la section 1. Effectuer la procédure d'amplification tel que décrit dans la section 2. Répétez les étapes 5.5.15.5.4 à trois fois.
- Définir un profil de précision afin d'évaluer et de valider la performance quantitative de la méthode.
- Définir les limites d'acceptabilité de toute la méthode pour le laboratoire. Dans ce protocole, les limites d'acceptabilité sont définis comme ± 0,75 Log 10 par le labeo Frank Duncombe.
- Pour un essai, tracer une première régression linéaire y = ax + b (a est la pente et b est l'interception) avec une répétition de valeurs Ct correspondant à chaque valeur estimée de la courbe standard. Avec cette première régression linéaire, le calcul de la valeur expérimentale du nombre de copies pour les essais répétés des valeurs Ct utilisées pour cette première courbe standard. Répétez l'étape 5.5.5.2 avec les deuxièmes répétitions de valeurs Ct pour obtenir une deuxième régression linéaire et calculer la valeur expérimentale du nombre de copies pour les deuxièmes répétitions de valeurs Ct utilisées pour la deuxième courbe standard.
- Répétez l'étape 5.5.5.2 avec les deuxième et troisième essais.
- Calculer les limites de précision, la justesse et de précision pour chaque niveau plasmidique selon NF U47-600-2 12.
- Créer une feuille de calcul pour compiler les informations pour tous les points standards (5.5.5.4) et de créer un profil de précision avec les limites d'acceptabilité définies précédemment dans 5.5.5.1 et la véracité des données, ainsi que la partie inférieure et les limites de précision supérieures tel que calculé à 5.5 .5.4 (figure 5).
- Déterminer la méthode LQ: cela correspond à la plus faible concentration d'une courbe standard avec justesse 0,75 log 10 que celui utilisé pour la gamme de linéarité calculée 5.5.5.5.
- Évaluation de répétabilité et de reproductibilité
Remarque: Vérifiez la répétabilité et la reproductibilité en utilisant le rapport entre l'écart type et la moyenne des mesures répétées (de coefficient de variation, ou CV = écart type /signifier).- Évaluer la répétabilité de la méthode Whole par 1 analyste:
- Choisir 3 échantillons biologiques avec 3 charges de génome viral distinctes (préalablement testés dans une PCR par exemple).
- Extrait 8 répétitions des 3 échantillons comme décrit dans la section 1. Effectuer la procédure d'amplification tel que décrit dans la section 2.
- Calculer la moyenne et l' écart type de la valeur Ct recueillies pour chaque échantillon.
- Calculer le CV intra-essai en utilisant la formule CV = déviation standard / moyenne.
- Évaluer la reproductibilité de la méthode Whole par 3 analystes:
- Choisissez 3 échantillons biologiques avec 3 charges du génome viral distinctes (préalablement testés dans la PCR, par exemple)
- Extrait 2 répétitions des 3 échantillons choisis en 5.6.2.1 (tel que décrit dans la section 1). Effectuez la procédure d'amplification tel que décrit dans la section 2. Répétez les étapes 5.6.2.2 par 2 analystes indépendants.
- Calculer la moyenne et l'écart typede la valeur Ct recueillies pour chaque échantillon.
- Calculer le CV inter-essai en utilisant la formule CV = déviation standard / moyenne.
- Évaluer la répétabilité de la méthode Whole par 1 analyste:
Résultats
La méthode RT-PCR quantitative, comme décrit ci - dessus, a été mis en œuvre pour détecter et quantifier équidé herpesvirus-2 dans les fluides respiratoires. La figure 1 illustre un diagramme de flux de travail schématique pour le développement et la validation d'une méthode RT-PCR quantitative selon la norme AFNOR NF U47-600. Spécificité des amorces et des sondes ont été validées lors de l'élaboration étape par étape de la PCR. Seulement EHV-2 souches ont été amplifiées dans ce système. Par la suite, les performances de la qRT-PCR doit être caractérisée.
Tout d' abord, pour estimer la LOD PCR, une dilution en série 6 dix fois a été effectuée pour établir la zone de réduction (Figure 2). Dans cet exemple, 6 dilutions en série de dix fois ont été effectuées entre 10 -5 et 10 -10 (entre 26.000 et 0.26 copies / 2,5 ul d' échantillon) pour estimer la PCR LOD. La zone de réduction ls entre des dilutions de 10 -9 et 10 -10 (entre 2.6 et 0.26 copies / 2,5 pi d' échantillon). Pour déterminer la valeur LOD PCR dans ce cas, 6 dilutions en série de deux en deux du plasmide ont été faites dans cette zone de réduction entre 5.2 et 0.16 copies / 2,5 échantillon ul. La valeur LOD PCR était de 2,6 copies / 2,5 échantillon ul.
Pour déterminer la plage de linéarité et de LQ PCR, la valeur LOD PCR a été utilisée pour démarrer la gamme de 6 dilutions en série de dix fois, entre 2,6 (LOD PCR) et 260.000 copies / 2,5 pi d' échantillon. La figure 3 illustre une régression linéaire pour le EHV2 qRT-PCR à partir d'un essai. Les performances de régression linéaire (figure 4) sont validés en quatre en utilisant les calculs décrits dans le tableau 3. Les calculs sont effectués pour définir la plage de linéarité selon les critères Bias absolue i vallog 10 ue ≤ 0,25, quel que soit le niveau i de la charge de plasmide. Dans ce cas, la linéarité gamme était entre 2,6 et 260.000 copies / 2,5 pi d'échantillon. La PCR LQ est la plus faible concentration dans la gamme de linéarité (ie, 2,6 copies / 2,5 échantillons ul dans ce cas). U LIN a été établie à 0,12 log 10 dans la gamme 2.6-260,000 copies / 2,5 pi d'ADN.
Après le développement (Figure 1, bleu) et la caractérisation de la qRT-PCR (Figure 1, jaune), la norme AFNOR NF U47-600 norme recommande la caractérisation de l'ensemble méthode d' analyse de l' extraction d'ADN à qRT-PCR (Figure 1, orange). La sensibilité et la spécificité ont été calculées comme décrit dans le tableau 4. Les rendements quantitatifs de la méthode analytique ensemble qRT-PCR ont été évaluées et validées avec un profil de précision ( Figure 5).
Ce protocole, qui utilise la technologie moléculaire state-of-the-art, nous a permis de détecter et de quantifier l'EHV-2 charge virale du génome dans 172 échantillons d'écouvillons nasaux obtenus à partir des chevaux souffrant de troubles respiratoires et / ou suspicion clinique d'infection. L'incidence de EHV-2 de terrain échantillons (biologiques) était de 50% (86/172) dans cette population. Les analyses quantitatives ont montré que les charges du génome viral de EHV-2 étaient significativement plus élevés chez les jeunes chevaux et la répartition des charges du génome viral diminue avec l' âge (figure 6). Dans la présente étude, le EHV-2 charge de génome viral le plus élevé (1,9 x 10 11 copies / ml) a été détectée chez les poulains (figure 6).
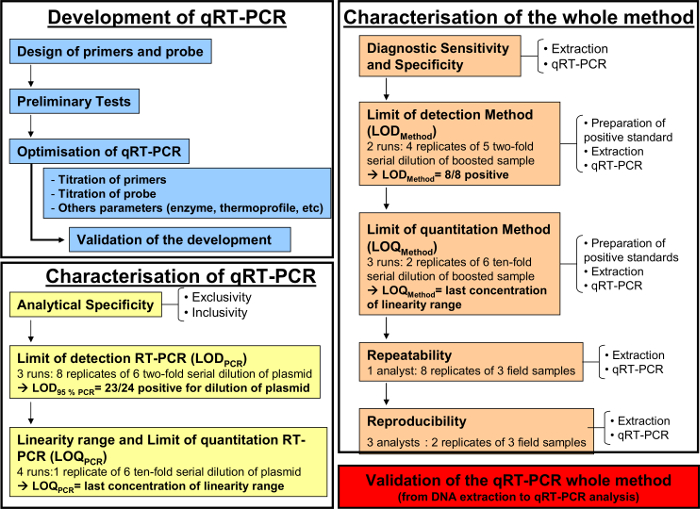
Figure 1: Diagramme de workflow pour le développement (bleu), la caractérisation de la RT-PCR quantitative(jaune) et la caractérisation de l'ensemble méthode d' analyse de l' extraction d'ADN à qRT-PCR (orange) selon la norme AFNOR NF U47-600-2. Le tableau de flux de travail reprend les différentes étapes de l'élaboration, la caractérisation de la RT quantitative -PCR et la caractérisation de l'ensemble méthode d'analyse de l'extraction d'ADN à qRT-PCR. Pour chaque étape, le tableau de flux de travail indique le nombre de pistes nécessaires, des dilutions à effectuer et le nombre d'analystes requis. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
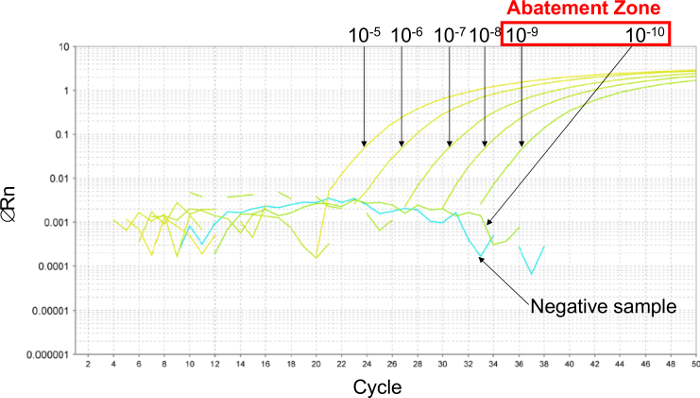
Figure 2: Détermination de la zone de réduction avec des résultats représentatifs de courbes de PCR en temps réel obtenues avec 6 dilutions en série de dix fois de plasmide. Pour estimer la zone de réduction, série 6 dix foisdilutions sont faites entre 10 -5 (26.000 copies / 2,5 pi d' échantillon) et 10 -10 (0,26 copies / 2,5 pi d' échantillon). La zone de réduction se situe entre des dilutions de 10 -9 (2,6 copies / 2,5 pi d' échantillon) et 10 -10 (0,26 copies / 2,5 pi d' échantillon). Dans ce cas, 6 dilutions en série de deux en deux du plasmide ont été faites dans cette zone de réduction pour déterminer la LOD 95% PCR, entre 5.2 et 0.16 copies / 2,5 pi d' échantillon. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
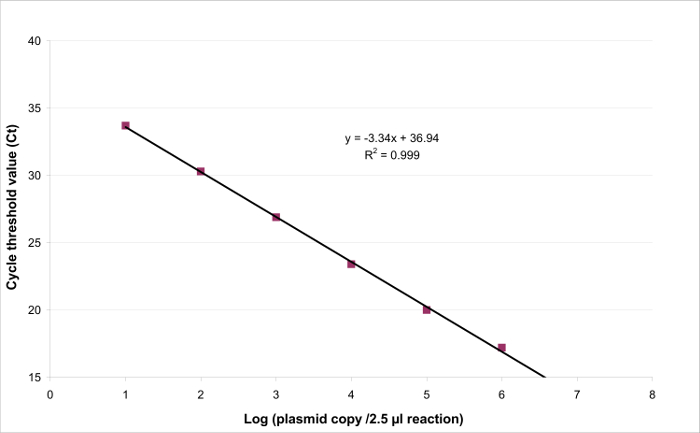
Figure 3:. Une régression linéaire pour EHV2 qRT-PCR La linéarité de l' essai quantitatif est la capacité à produire des résultats qui sont proportionnels à la concentration de la cible présente dans une plage spécifique. Ceci peut être modélisé parrégression linéaire (y = ax + b) entre le (seuil de cycle ou Ct) réponse instrumentale et le logarithme de la quantité de la cible (nombre de cibles copies / 2,5 pi d' échantillon). S'il vous plaît cliquer ici pour voir une version plus grande de cette figure .
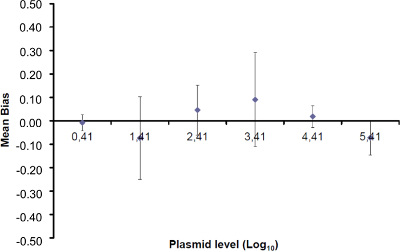
Figure 4:. La performance d' une régression linéaire de EHV-2 qPCR moyen de polarisation représentent la différence moyenne entre la quantité de plasmide mesurée ( 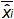 ) Et la quantité de plasmide théorique (x 'i) pour chaque niveau de plasmide. Les barres verticales représentent la linéarité incertitude (U Lini) donnée par la formule
) Et la quantité de plasmide théorique (x 'i) pour chaque niveau de plasmide. Les barres verticales représentent la linéarité incertitude (U Lini) donnée par la formule 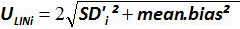
où SD'i est la déviation standard o f quantité de plasmide mesurée. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
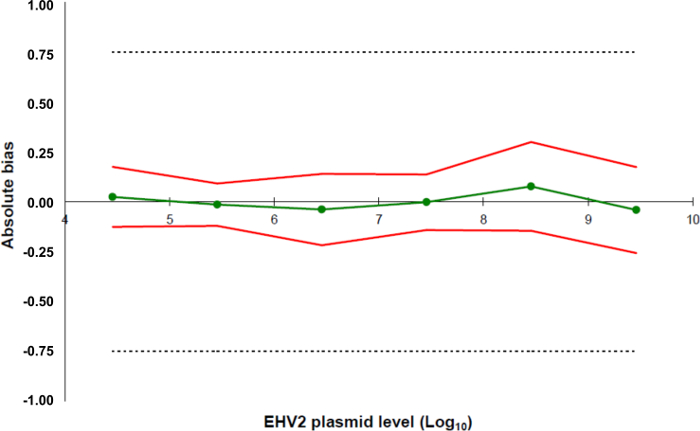
Figure 5:. Profils de précision sur la base des résultats de la validation de la méthode qRT-PCR EHV-2 La ligne verte (cercles) représente la justesse des données (erreur systématique ou biais). Les limites d'acceptabilité sont définis à ± 0,75 Log 10 par le laboratoire (lignes en pointillés). Les limites de précision inférieure et la partie supérieure ont été déterminées pour chaque niveau de la polarisation moyenne ± deux fois l'écart type des données de fiabilité (lignes rouges) de charge plasmidique. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
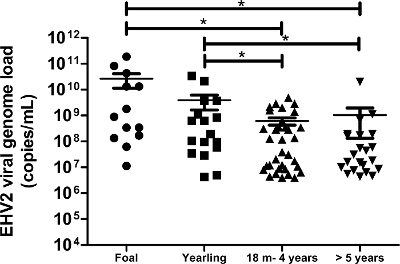
Figure 6:. Quantification des charges du génome viral de EHV-2 selon l'âge La répartition de la charge virale du génome de EHV-2 détectée dans les échantillons d'écouvillons nasaux est représenté pour les différents groupes d'âge. Les lignes horizontales représentent les valeurs moyennes à l'intérieur de l'écart-type (m = mois). * Différence significative par ANOVA avec un test de Newman-Keuls post-hoc (p <0,05). S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| gène cible | Amorces, des sondes et des séquences PLASMIDE (5'-3 ') | Position nucléotidique | Taille du produit (nucléotides) | conditions de cyclage thermique | Les références | |||
| EHV2 gB (HQ247755.1) | Forward: GTGGCCAGCGGGGTGTTC | 2113-2130 | 78 | 95 ° C 5 min | 11 | |||
| Revers: CCCCCAAAGGGATTYTTGAA | 2189-2170 | 95 ° C 15 sec | 45 cycles | |||||
| Sonde: FAM-CCCTCTTTGGGAGCATAGTCTCGGGG-MGB | 2132-2157 | 60 ° C 1 min | ||||||
| Plasmide: ACCTGGGCACCATAGGCAAGGTGGTGGTCA ATGTGGCCAGCGGGGTGTTCTCCCTCTTTG GGAGCATAGTCTCGGGGGTGATAAGCTTTTT CAAAAATCCCTTTGGGGGCATGCTGCTCATA GTCCTCATCATAGCCGGGGTAGTGGTGGTG TACCTGTTTATGACCAGGTCCAGGAGCATAT ACTCTGCCCCCATTAGAATGCTCTACCCCGG GGTGGAGAGGGCGGCCCAGGAGCCGGGCG CGCACCCGGTGTCAGAAGACCAAATCAGGA ACATCCTGATGGGAATGCACCAATTTCAG | 2081-2381 | |||||||
Tableau 1:. Sequences d'amorces, des sondes et des contrôles d'ADN synthétiques positifs utilisés dans ce protocole La séquence du plasmide (ADN synthétique positif) correspond au nucléotide positions 2081-2381 de la séquence EHV2gB (de HQ247755.1). La conception des amorces et des sondes utilisées dans ce protocole a été obtenu à l'aide d'un logiciel spécifique.
| PATHOGÈNES | Référence (origine) | Nombre de souches | RÉSULTATS |
| EHV-2 | |||
| EHV-2 | VR701 (ATCC) | 20 | Positif |
| 20 échantillons (collection FDL) | |||
| EHV-5 | KD05 (GERC) | 20 | Négatif |
| 20 échantillons (collection FDL) | |||
| EHV-3 | VR352 (ATCC) | 2 | Négatif |
| T934 WSV (GERC) | |||
| EHV-1 | souche Kentucky Ky A (ATCC) | 3 | Négatif |
| 2 échantillons (collection FDL) | |||
| EHV-4 | VR2230 (ATCC) | 1 | Négatif |
| Asinine herpesvirus AHV5 | FDL Collection | 1 | Négatif |
| chevalinVirus de la grippe | A / equine / Jouars / 4/2006 (H3N8) | 1 | Négatif |
| (Numéro d'accession JX091752) | |||
| Equine Virus de l' artérite | VR796 (ATCC) | 2 | Négatif |
| Rhodococcus equi | FDL Collection | 1 | Négatif |
| Streptococcus equi ssp. zooepidemicus | FDL Collection | 1 | Négatif |
| Streptococcus equi ssp. équi | FDL Collection | 1 | Négatif |
| Coxiella burnetii | ADI-142-100 (Adiagène) | 1 | Négatif |
| Chlamydophila abortus | ADI-211-50 (Adiagène) | 1 | Négatif |
| Klebsiella pneupneumoniae | FDL Collection | 1 | Négatif |
Tableau 2: Spécificité analytique qRT-PCR pour EHV-2.
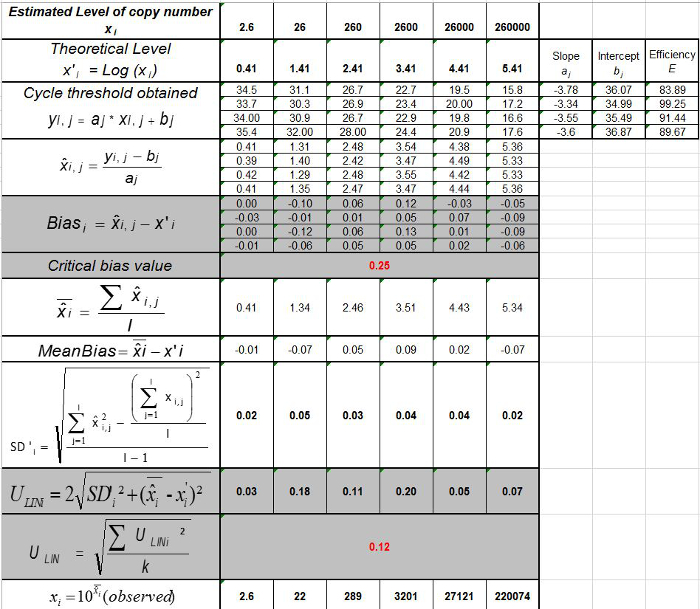
Tableau 3: Calcul de la polarisation et la linéarité incertitude (adapté de NF U47-600-2 12). Pour chaque essai, les performances de régression linéaire (y = ax + b) sont validés à l' aide de la table où y est le seuil de cycle obtenu; a est la pente obtenue;. X est le niveau de plasmide et b est l'interception i est le plasmide niveau (i varie de 1 à niveaux k); k est le nombre de niveaux de plasmide utilisé (par exemple, k = 6 en tsa table); j est le procès (j varie de 1 à I des essais); I est le nombre d'essais, comprise entre 3 et 6 essais (par exemple I = 4 dans ce tableau) x i est la quantité de plasmide estimée pour chacun. i plasmide de niveau. x 'i est la quantité théorique de plasmide obtenu avec l' équation x' i = log 10 (x i) pour chaque i plasmide de niveau. Au cours de chaque essai j, le seuil de cycle obtenu pour chaque i le niveau de plasmide est calculé par régression linéaire y i, j = a j x i, j + b j. 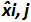 est la quantité de plasmide mesurée pendant l'essai j. Bias i sub> est la différence observée entre la quantité de plasmide mesurée et la quantité de plasmide théorique pour chaque essai et chaque niveau de plasmide.
est la quantité de plasmide mesurée pendant l'essai j. Bias i sub> est la différence observée entre la quantité de plasmide mesurée et la quantité de plasmide théorique pour chaque essai et chaque niveau de plasmide. 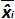 est la valeur moyenne de
est la valeur moyenne de 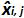 par chaque i plasmide de niveau; SD 'i est l'écart type de la quantité mesurée
par chaque i plasmide de niveau; SD 'i est l'écart type de la quantité mesurée 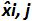 pour chaque i niveau plasmidique; polarisation moyenne est la moyenne de Bias i; U Lini est l'incertitude de linéarité déterminée pour chaque i plasmide de niveau calculé de SD'i et de partialité veux dire. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
pour chaque i niveau plasmidique; polarisation moyenne est la moyenne de Bias i; U Lini est l'incertitude de linéarité déterminée pour chaque i plasmide de niveau calculé de SD'i et de partialité veux dire. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Tableau 4:. Calcul de la sensibilité diagnostique (Se) et la spécificité (Sp) du procédé une table entière Schwartz a été utilisé pour calculer la CONFIDintervalle de réfé- à 95% de la sensibilité et de la spécificité du procédé entier tel que décrit dans U47-600-2 NF.
Discussion
Depuis les années 2000, la PCR en temps réel a été de remplacer les techniques d'or standard (culture cellulaire et des méthodes de culture de bactéries) dans un nombre croissant de laboratoires. La mise en oeuvre de cette technique est relativement facile. Cependant la validation des méthodes de laboratoire est essentiel pour la détection moléculaire et la quantification des agents pathogènes pour garantir des données précises, reproductibles et fiables.
Etant donné que l'étape d'extraction est la principale source de perte de matière biologique, elle peut être considérée comme la principale source d'erreur de quantification entre un protocole et un autre. A ce titre, la création d'une courbe standard de l'ADN du plasmide au cours de la qRT-PCR, principalement dans la littérature, indique que la charge du génome viral, mais ne tient pas compte de l'étape d'extraction.
Description d'une stratégie de novo pour tout un processus de validation de la méthode dans la norme AFNOR NF U47-600-2 représente un progrès significatif dans ce domaine. Comme cela est illustré dansce document pour EHV-2 chez les chevaux, ou par d' autres dans les abeilles 21, cela nécessite une différenciation claire entre l'étape de développement et de l'étape de validation avec la caractérisation de la PCR et la caractérisation de toute la méthode. Une limitation de cette approche intéressante est que tout changement dans le protocole se traduira par l'obligation de revalider le processus complet qui pourrait être très coûteux. Cette limitation a été également mis en évidence par le fait que les limites de quantification dépendent de la source à partir de laquelle le virus est extrait (par exemple, des fluides respiratoires, les organes, le sang ou l' urine). En effet, chaque matrice présente des spécificités différentes, dans leurs caractéristiques physico-chimiques et il est important de définir, indépendamment dans chaque matrice différente utilisée pour la détection et la quantification virale par qRT-PCR. Ainsi, la charge du génome viral de chaque échantillon biologique peut être quantifiée plus précisément à partir de l'extraction. La caractérisation prend également en compte le mod thermocycleurel et quand l'utilisation d'une méthode précédemment bien caractérisé (par exemple, la méthode EHV-2 qPCR décrite dans cet article) nécessite un nouveau type de machine dans le laboratoire de parent ou d' un autre laboratoire, il faut confirmer l'efficacité de cet instrument. La confirmation de l'exécution d'une analyse qPCR est une condition sine qua non pour tous les tests mettent en laboratoire. Ceci est normalement obtenu par l'analyse d'un échantillon de référence possédant des propriétés connues. Un tel contrôle est une condition préalable et considéré obligatoire tel que demandé par la norme NF 47-600-1 AFNOR afin de valider la performance du (efficacité LOD, LOQ) qPCR et la robustesse de toute la méthode (LOD, LOQ). Non seulement pendant les étapes de développement et de caractérisation, mais également lorsqu'ils sont utilisés dans la recherche ou à des fins de diagnostic, les facteurs de risque peuvent être identifiés et bien contrôlées pour assurer la normalisation du protocole. Il est particulièrement préoccupant formation adéquate du personnel, du personnel hautement qualifié, le contrôle de la qualité des consommablesutilisés et leur stockage, le contrôle des conditions environnementales immédiates et de sensibilisation des conditions métrologiques qui peuvent affecter la performance des instruments scientifiques impliqués dans le dosage. Utilisation d'échantillons de référence pour les comparaisons inter-laboratoires pourrait aussi aider à contrôler les incertitudes. De cette manière, la comparaison des données entre les laboratoires peut être facilitée. En effet, des essais inter-laboratoires sont indispensables pour évaluer et confirmer la reproductibilité de la méthode.
Résultats de la charge du génome viral qui sont exprimées en unités internationales (UI) de la matrice biologique analysé (UI: copies / ml pour des fluides ou des copies / g pour les tissus) sont plus faciles à utiliser afin de comparer les résultats entre différents laboratoires. Tous les résultats ci-dessus la LQ sont exprimés en copies / ml et un résultat entre le LOD et LOQ est considéré comme un résultat positif non quantifiable. La présentation des données quantificationnels du génome de cette manière est plus conforme précisément aux procès-s d'analyses (amplification du génome). En fait, dans des expériences de culture cellulaire, expression de la charge virale par TCID 50 (tissu médiane culture infectieuse dose) est fonction de la nature des cellules et des souches de virus. Chaque ligne de souche possède sa cinétique d'infection uniques et certains virus comme EHV-2 peuvent prendre plusieurs jours avant le premier effet cytopathogène est apparente.
En conclusion, cette nouvelle méthode de caractérisation des qRT-PCR devrait faciliter l'harmonisation de la présentation des données et l'interprétation entre les laboratoires. Ce sera très utile pour de nouvelles applications potentielles de qRT-PCR à l'avenir, comme la mise en place d'une valeur de coupure pour la déclaration de l'état de la maladie au lieu de simplement la présence ou l'absence de l'agent pathogène.
Déclarations de divulgation
Les auteurs déclarent qu'ils ont aucun intérêt financier concurrents.
Remerciements
The authors would like to thank Sophie Castagnet and Nadia Doubli-Bounoua for their technical support. This work received financial support from the General Council of Calvados and the agreement of Region Basse-Normandie and French Government (CPER 2007-2013; project R25 p3). The authors would like to thank the experts of the AFNOR group and particularly Jean-Philippe Buffereau and Eric Dubois.
matériels
| Name | Company | Catalog Number | Comments |
| AB-1900 natural color ABgene 96 well plate | Dutsher | 16924 | |
| Adhesive film qPCR Absolute | Dutsher | 16629 | Adhesive film used for sealing the plate prior to the qRT-PCR run |
| 0.5 ml microtubes, skirted, caps | Dutsher | 039258 | |
| Ethanol 98% | Sodipro | SAF322941000 | |
| Primers | Eurofins | Custom order | |
| Probe | Life Technologies | Custom order | |
| Plasmid | Eurofins | Custom order | |
| QIAamp RNA viral Mini Kit (containing: QIAamp Mini column, AVL buffer, AW1 buffer, AW2 buffer, AVE buffer, collection tubes) | Qiagen | 52906 | AVL buffer: pre-warm 5 min at 72 °C |
| Sequencing by Sanger method | Eurofins | Custom order | |
| Taqman Universal PCR Master Mix | Life Technologies | 4364340 | |
| Tris-EDTA buffer solution | Santa Cruz | sc-296653A | |
| NanoDrop 2000c Spectrophotometer | Thermoscientific | ND-2000C | |
| StepOnePlus Real-Time PCR systems | Life Technologies | 4376600 | pre-warm 15 min |
Références
- Brault, S. A., et al. The immune response of foals to natural infection with equid herpesvirus-2 and its association with febrile illness. Vet.Immunol.Immunopathol. 137 (1-2), 136-141 (2010).
- Fortier, G., Van Erck, E., Pronost, S., Lekeux, P., Thiry, E. Equine gammaherpesviruses: pathogenesis, epidemiology and diagnosis. Vet.J. 186 (2), 148-156 (2010).
- Hue, E. S., et al. Detection and quantitation of equid gammaherpesviruses (EHV-2, EHV-5) in nasal swabs using an accredited standardised quantitative PCR method. J Virol.Methods. 198 (1), 18-25 (2014).
- Diallo, I. S., et al. Multiplex real-time PCR for the detection and differentiation of equid herpesvirus 1 (EHV-1) and equid herpesvirus 4 (EHV-4). Vet.Microbiol. 4 (1-3), 93-103 (2007).
- Williams, K. J., et al. Equine multinodular pulmonary fibrosis: a newly recognized herpesvirus-associated fibrotic lung disease. Vet.Pathol. 44 (6), 849-862 (2007).
- Mullis, K., et al. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb.Symp.Quant.Biol. 51 (1), 263-273 (1986).
- EPA Office of Water (4607). EPA-815-B-04-001. Quality Assurance/Quality Control Guidance for Laboratories Performing PCR Analyses on Environmental Samples. , (2004).
- Telford, E. A., et al. Equine herpesviruses 2 and 5 are gamma-herpesviruses. Virology. 195 (2), 492-499 (1993).
- Fortier, G., et al. Identification of equid herpesvirus-5 in respiratory liquids: A retrospective study of 785 samples taken in 2006-2007. Vet.J. 182 (2), 346-348 (2009).
- Brault, S. A., Bird, B. H., Balasuriya, U. B., MacLachlan, N. J. Genetic heterogeneity and variation in viral load during equid herpesvirus-2 infection of foals. Vet.Microbiol. 147 (3-4), 253-261 (2011).
- Association Francaise de Normalisation. NFU 47-600-1. Animal health analysis methods-PCR-Part 1: Requirements and recommandations for the implementation of veterinary PCR. , (2015).
- Association Francaise de Normalisation. NFU 47-600-2. Animal health analysis methods-PCR-Part 2: Requirements and recommendations for the development and the validation of veterinary PCR. , (2015).
- ISO. EN ISO-CEI 17025. General requirements for the competence of testing and calibration laboratories. , (2005).
- Manual of Diagnostic Tests and Vaccines for Terrestrial Animals 2010. Chapter1.1.1.4/ 5. Principles and Methods of Validation of Diagnostic Assay for Infectious Diseases. , This thoroughly revised chapter replaces Chapter 1.1.4 Principles of validation of diagnostic assays for infectious diseases and Chapter 1.1.5 Validation and quality control of polymerase chain reaction methods used for the diagnosis of infectious diseases from the sixth edition of the OIE Terrestrial Manual (2009).
- Apaza, S., et al. Detection and genogrouping of noroviruses from children's stools by Taqman One-step RT-PCR. J Vis.Exp. (65), e3232(2012).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis.Exp. (63), e3998(2012).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol.Biol. 94 (3), 441-448 (1975).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc.Natl.Acad.Sci.U.S.A. 74 (12), 5463-5467 (1977).
- NCBI. BLAST Homepage and Selected Search Pages. Introducing the BLAST homepage and form elements/functions of selected search pages. , (2015).
- Greiner, M., Gardner, I. A. Application of diagnostic tests in veterinary epidemiologic studies. Prev.Vet Med. 45 (1-2), 43-59 (2000).
- Blanchard, P., Regnault, J., Schurr, F., Dubois, E., Ribiere, M. Intra-laboratory validation of chronic bee paralysis virus quantitation using an accredited standardised real-time quantitative RT-PCR method. J Virol.Methods. 180 (1-2), 26-31 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon