Method Article
ODELAY: Eine groß angelegte Methode für die Mehrparameter-Quantifizierung des Hefewachstums
In diesem Artikel
Zusammenfassung
Wir stellen eine Methode zur Quantifizierung von Wachstumsphänotypen einzelner Hefezellen dar, da sie in festen Medien unter Verwendung der Zeitraffer-Mikroskopie in Kolonien aufwachsen, die als eine zellige Verdopplungsbewertung von lebenden Arrays von Hefe (ODELAY) bezeichnet werden. Die Populations-Heterogenität von genetisch identischen Zellen, die zu Kolonien wachsen, kann direkt beobachtet und quantifiziert werden.
Zusammenfassung
Wachstums-Phänotypen von Mikroorganismen sind ein starker Indikator für ihre zugrunde liegende genetische Fitness und können in 3 Wachstumsregimes getrennt werden: Lag-Phase, Log-Phase und stationäre Phase. Jede Wachstumsphase kann verschiedene Aspekte der Fitness zeigen, die mit verschiedenen ökologischen und genetischen Bedingungen zusammenhängen. Hochauflösende und quantitative Messungen aller 3 Phasen des Wachstums sind in der Regel schwer zu erreichen. Hier präsentieren wir eine detaillierte Methode zur Charakterisierung aller 3 Wachstumsphasen auf festen Medien mit einem Assay namens One-Cell Doubling Evaluation von lebenden Arrays von Hefe (ODELAY). ODELAY quantifiziert die Wachstumsphänotypen einzelner Zellen, die unter Verwendung von Zeitraffer-Mikroskopie zu Kolonien auf festen Medien wachsen. Diese Methode kann die Heterogenität der Population mit jedem Wachstumsparameter in genetisch identischen Zellen, die zu Kolonien wachsen, direkt beobachten. Diese Heterogenität der Bevölkerung bietet eine einzigartige Perspektive für das Verständnis der genetischen und epigenetischen Regulierung und der Reaktionen aufGenetische und umweltbedingte Störungen. Während die ODELAY-Methode unter Verwendung von Hefe demonstriert wird, kann sie auf jedem koloniebildenden Mikroorganismus verwendet werden, der durch eine helle Feldmikroskopie sichtbar ist.
Einleitung
Wachstums-Phänotypen von Mikroorganismen sind ein starker Indikator für ihre zugrunde liegende genetische Eignung zu einem gegebenen Umgebungszustand. Das Wachstum wird klassisch in 3 verschiedene Wachstumsregimes getrennt: Lag-Phase, Log-Phase und stationäres Phasenwachstum 1 . Jede Wachstumsphase kann verschiedene Aspekte der Fitness zeigen, die von verschiedenen ökologischen und genetischen Bedingungen abhängig sind. Zum Beispiel kann die Verzögerungszeit oder die Zeitspanne, in der ein Organismus vor dem Beginn des exponentiellen Wachstums in der Verzögerungsphase verbringt, auf die Fähigkeit eines Organismus hinweisen, auf veränderte Umgebungsbedingungen zu reagieren 2 . Die Verdopplungszeit während des Log-Phase-Wachstums, die häufigste Metrik der zellulären Fitness, zeigt die Gesamteffizienz der Fähigkeit eines Organismus, sich durch Metabolisierung und Nutzung von Umweltmaterialien für die Replikation zu teilen. Stationäre Phase, wo Wachstum nach Log-Phase schnell reduziert wird, ist ein weiterer Indikator für Fitness, die regelmäßig istAls Wachstumsendpunkt in punktbasierten Hefewachstums-Assays verwendet.
Mehrere Hefewachstumstests sind derzeit verfügbar und gelten als Standardmethoden zur Bewertung von Wachstumsphänotypen in Hefe 3 , 4 , 5 . Diese Assays basieren in erster Linie auf Methoden zum Anbau von Hefe entweder auf festen oder in flüssigen Medien. Auf festen Medien übertragen Kolonie-Pinning-Assays eine kleine Anzahl von Zellen auf einen festen Agar mit einem Pin und Hefe-Zellen können für eine definierte Zeitspanne wachsen. Kolonien werden dann abgebildet und ihre Größen werden an einem Endpunkt 6 verglichen. Diese Kolonie-Pinning-Assays haben sich als robust und skalierbar erwiesen, um genomweite Bildschirme zu erzeugen. In jüngster Zeit wurde eine periodische Bildgebung mit Flachbett-Scannern und Einzelobjektivreflex (SLR) -Kameras in diese Assays integriert, um das Koloniewachstum über die Zeit 7 , 8 aufzuzeichnen, 9 Die Auflösung dieser Vorrichtungen verhindert jedoch, dass sie einzelne Zellen detektieren und somit diese Kolonie-Pinning-Assays nicht direkt die Verzögerungszeit beobachten und die Variation zwischen den einzelnen Zellen, die in Kolonien wachsen, nicht beobachten können.
Flüssigbasierte Wachstumstests wurden auch zur Durchführung von genomweiten Schirmen eingesetzt 3 . Die Kopplung eines Liquidwachstums-Assays mit der Zeitraffer-Mikroskopie zeigte die Heterogenität der Population in der Verdopplungszeit genetisch identischer Einzelzellen, die eine wichtige Perspektive für das Verständnis der genetischen Regulation und der Umweltanpassung bietet. Dieser Assay misst jedoch nicht andere Aspekte des Wachstums wie Verzögerungszeit und Tragfähigkeit 10 . Hier stellen wir eine Methode vor, um alle drei Wachstumsphasen von koloniebildenden Mikroorganismen auf festen Medien unter Verwendung eines Assays zu charakterisieren, den wir ODELAY 11 bezeichnen . ODELAY besteht aus utiliZing hohe Durchlauf-Zeitraffer-Mikroskopie zur Aufzeichnung von Bildern von einzelnen Zellen, die in Kolonien auf festen Medien wachsen. Diese Population einzelner Zellen, die in Kolonien wachsen, zeigt die zugrundeliegende Heterogenität der Bevölkerung, die von anderen weniger empfindlichen Messungen, wie z. B. terminaler Endpunkt-Scoring, nicht erkannt wird. Wir zeigen die Methode auf Hefe, aber ODELAY kann auf jeden Organismus angewendet werden, der Kontrast in der Hellfeldmikroskopie zeigt.
Protokoll
1. Vorbereitung der Agarose Gel Stock
- 2 g hochreine Agarose abwiegen.
- Füge die Agarose zu einer 500 ml Flasche hinzu und notiere ihre kombinierte Masse.
- Berechnen Sie die Zielmasse der Flasche plus 2 g Agarose mit 150 g ultrareinem Wasser und fügen Sie dann 150 g ultrareines Wasser innerhalb von 0,1 g dieser Zielmasse hinzu.
- Beachten Sie die Masse der Flasche plus die Agarose und Wasser.
- Legen Sie die Flasche in eine Mikrowelle und achten Sie darauf, die Flasche mit einer losen Kappe auf die Oberseite zu passen, um die Wasserverdampfung beim Erwärmen der Agarose zu minimieren.
- Mikrowelle die Flasche in 15 - 20 s Bursts gefolgt von kurz wirbeln die Flasche, um die Agarose und Wasser zu mischen. Wiederholen Sie diesen Vorgang, bis die Lösung kocht und die Mischung homogen ist.
ACHTUNG: Achten Sie darauf, dass die Haut mit Dampf aus der Flasche entweicht. Auch wird die Flasche heiß an die Berührung und ein geeigneter Schutz, wie ein Autoklavenhandschuh, ist erforderlich, um Verletzungen zu vermeiden. - Nachdem die geschmolzene Agarose homogen ist, wiegt sie die Flasche wieder und fügt innerhalb von 0,1 g der ursprünglichen Masse hochreines Wasser hinzu. Dadurch wird das durch Verdunstung verlorene Wasser ersetzt.
- Bevor die geschmolzene Agarose erstarrt, wirbeln, um das Zusatzwasser zu mischen, um eine Homogenität zu gewährleisten.
- Aliquot 15,2 g Agarose in 9 - 50 ml Plastikröhrchen.
- Die Röhrchen bis zum Anbau kühlen.
2. Vorbereitung von ODELAY Agarose-Medien
- Man erhitze 400 ml entionisiertes Wasser, um in einem überdachten Becher zu kochen, auf einer Kochplatte unter leichtem Rühren mit einem Magnetrührstab.
- Füge 2 ml 10X-Medium ( z . B. Hefeextrakt Pepton (YEP) oder Komplement Ergänzungsmischung (CSM) zu einem 15,2 g Agarosealiquot in einem 50 ml konischen Röhrchen aus Schritt 1.10 hinzu.
HINWEIS: CSM-Medium neigt dazu, an den Glasrutschen zu haften. Bei Verwendung von CSM-Medienformulierungen werden 5 μl 50% iges Polyethylenglykol (PEG) in steriles Wasser der Mediumformulierung zugesetzt. Das PEG verhindert das aGar beim Ankleben an die Glasrutschen während der Formtrennung und hemmt das Hefewachstum nicht. - Fügen Sie zusätzliche 100X Nährstoffergänzungen hinzu, falls erforderlich, und verwenden Sie Wasser, um das gesamte hinzugefügte Volumen von diesem Schritt auf 1 ml zu bringen.
- Wiegen Sie das Agarose-Aliquot mit Zusatzmedien und Ergänzungen, bevor Sie das 50 ml konische Röhrchen aus Schritt 2.1 in kochendes Wasser geben.
- Nach 16 min das 50 ml Röhrchen herausnehmen und die Mischung mit einem Wirbel homogenisieren. Halten Sie die Kappe fest auf dem 50 mL konischen Rohr befestigt.
HINWEIS: Ein Deckel auf dem Becher hilft, das ganze Rohr gleichmäßig zu erwärmen, sonst wird ein Film aus fester Agarose bilden und möglicherweise nicht schmelzen. Auch während dieser Zeit ist es zweckmäßig, die Agaroseform zusammenzubauen ( Abbildung 1 ). - Homogenisieren der Mischung unter Verwendung eines Wirbels.
- Kochen für weitere 2 min, um sicherzustellen, dass alle Agar gemischt und geschmolzen ist.
- Wiegen Sie Rohr und ersetzen Sie jede Masse verloren mit ultra-reinem sterilen Wasser.
- Füge 2 ml 10X kohlensäure hinzuCe-Reagenz ( z . B. 20% w / v Glucose) und Vortex, um Agarose-Medium-Lösung zu erhalten.
HINWEIS: Trennung von Glasschienen: Beim Zusammenbau der Form ist darauf zu achten, dass die langen Distanzstücke mit der richtigen Ausrichtung in die Form gebracht werden. Dies liegt daran, da der Laser das Acryl schneidet, neigt es dazu, als ein Kegel zu schneiden, der das Teil mit leicht abgewinkelten Seiten anstelle von genau 90 ° Seiten verlässt. Dann, wenn die Form auf die Tischplatte gelegt wird, um das Glas aus dem Agar freizugeben, sollte die Kante des Abstandshalters in einem spitzen Winkel mit dem Agar sein. Der spitze Winkel hilft, die Kante des Agars von dem oberen Glasstück weg zu komprimieren, wenn die Außenkante des Formabstandshalters um die untere Kante geschwenkt wird (siehe Abbildung 1 ). Das Ergebnis ist eine konsistentere Trennung des Agars vom oberen Stück Glas.
HINWEIS: Formtrennmittel, die auf das Glas aufgebracht werden, wie Öle, Silikon-Sprays oder sogar kommerzielle Fensterbehandlungen sollten nicht verwendet werden, da sie die s verunreinigen Ursprung des Agars und möglicherweise hemmen das Wachstum. Diese Methoden nehmen einige Praxis, um konsistent zu sein. - Reinigen Sie vier 2 in x 3 in x 1 mm dicken Glasrutschen mit 70% Ethanol und trocknen Sie mit Zwangsluft.
- Saubere Acrylformen mit 70% Ethanol, trocknen und zusammenbauen, wie in Abbildung 1 gezeigt . Nehmen Sie die drei unteren Stücke, wie gezeigt, und montieren Sie sie, so dass die beiden identischen Stücke sandwich das dritte Stück ( Abbildung 1B ). Klemmen Sie die Basisstücke auf jeder Seite mit zwei kleinen Binder-Clips ( Abbildung 1C ). Legen Sie die Ständer in die Form, um sicherzustellen, dass der Laser kerf korrekt positioniert ist ( Abbildung 1E ).
- Legen Sie den gereinigten und getrockneten Glasschieber auf die Form und halten Sie ihn fest, während Sie einen zweiten Glasschieber auf die andere Seite legen. Klemmen Sie die Baugruppe mit einem größeren Binderclip ( Abbildung 1D ). Klemmen Sie beide Folien mit größeren Binder-Clips (Lass = "xfig"> Abbildung 1D). Vergewissern Sie sich, dass der obere Binderclip mit der Glasscheibe übereinstimmt, die mit dem Acryl überlappt.
- Hinzufügen von verbleibenden Binder-Clips. Wieder stellen Sie sicher, dass die Klemmen den Schieber berühren, wo er das Acryl überlappt ( Abbildung 1D ).
HINWEIS: Bevor Sie die zusammengesetzte Form mit geschmolzenem Agar füllen, stellen Sie sicher, dass die Kanten durch Pipettieren von ca. 70 μl geschmolzenem Agarmedium entlang der Innenseite der Form versiegelt werden. Dieser Agar wird schnell verfestigen und verhindert Lecks. - Füllen Sie die Form mit geschmolzenem Agar, um sicherzustellen, dass Sie Luftblasen in der Form einfangen.
HINWEIS: Dies kann durch langsames Pipettieren des geschmolzenen Agars am Rand der Form erreicht werden. Nachdem es gefüllt ist, lassen Sie die Form für 40 min bis 1 h bei etwa 23 ° C Umgebungstemperatur abkühlen. Wenn man sich zu lange abkühlen lässt, kann der Agar in der Form brechen.
HINWEIS: Formtrennung: Eine ordnungsgemäße Entfernung der Form aus dem gegossenen Agar ist wichtig, um einen gleichmäßigen Feststoff zu gewährleistenAgar-Pad für Hefe-Wachstum. Das Versagen, eine effiziente Trennung der Form bei diesem Schritt zu gewährleisten, führt zu Wachstumsdifferenzen, die auf Unvollkommenheiten in dem festen Agar-Pad zurückzuführen sind. Das Üben dieses Schrittes wird ein paar Mal empfohlen. - Beginnen Sie mit dem Entfernen der unteren Binder-Clip. Entfernen Sie den unteren Teil der Form, die aus den drei Sandwich-Stücke besteht. Halten Sie die Folien durch Komprimieren und entfernen Sie dann die Binder-Clips. Achten Sie darauf, dass Sie die Formscheiben nicht stoßen, während Sie die Binder-Clips entfernen. Dadurch werden die Medien verformt.
- Legen Sie die Form auf den Rand der Tischplatte, so dass die Seite der Form mit dem blauen Punkt nach oben und in der unteren linken Ecke ( Abbildung 1E ). Legen Sie Daumen unter den Acryl und den ersten Finger auf die Oberseite in Richtung der inneren Kante der Form.
- Langsam und vorsichtig mit Daumen gegen die Form nach oben drücken, als ob der Formabstandhalter um seine Unterkante schwenkbar ist ( Abbildung 1F ). Konstant anwendenAber langsam zunehmender Druck Die Benutzer sehen eine Pause und Luftblase erscheinen entlang der Linie, wo das obere Glas bedeckt die Form spacer.
- Nachdem du die anfängliche Bruchlinie gesehen hast, gehst du mit Daumen hoch und drückst einen konstanten Druck an. Der Agar sollte an diesem Punkt vom Glas abbrechen ( Abbildung 1F ). Die Form weiter nach oben drehen. Das hebt das Glas an, ohne es zu schieben. Eine Linie, in der sich der Agar vom Glas abzieht, sollte sich von der anfänglichen Bruchlinie entfernen.
HINWEIS: An diesem Punkt klebt der Agar sowohl an der Unterseite als auch an der Oberseite des Glases. Dies geschieht gelegentlich mit dem am weitesten links liegenden Rand. Solange die Fläche deformiert ist nicht in der Gegend, wo Hefe entdeckt wird, sollte dies gut sein. - Wenn das Glas ganz frei kommt, packen Sie es und entfernen Sie es vollständig aus der Form. Dann entfernen Sie das andere Formteil mit einer ähnlichen Bewegung, um das Stück frei zu heben, ohne den Agar zu bewegen. Legen Sie die Folien in eineSterile Spitzenbox mit etwas sterilem hochreinem Wasser im Boden. Schließen Sie die Schachtel und lag bei 4 ° CO / N für den nächsten Tag. Bei längerer Lagerung werden die Wachstumsraten inkonsistent.
3. ODELAY Kulturvorbereitung
- Legen Sie Stämme in eine 96-Well-Platte für O / N-Wachstum.
- Am nächsten Tag verdünnen Sie 20 μl Overnight-Kultur in 200 μL Medium für 220 μl Gesamtvolumen in einer neuen Platte.
- Messen Sie die optische Dichte bei 600 nm (OD600) jeder Kultur in einem Plattenleser. Mit einem Plattenleser und einem Flüssigkeitshandhabungsroboter folgen Sie dem automatischen Verdünnungsprotokoll der Schritte 3.3.1 - 3.3.6. Manuell verdünnen die Kulturen in der Platte auf ca. 0,1 OD600 (optional).
- Auf dem Plattenleser traf "Experiment" unter "neu erstellen". Wähle ODELAYDilution.exp und lauf das Experiment. Geben Sie den Namen des Experiments als ODELAY "Datum" "Zeit" "Experimentname" Iteration ein.
- Klicken Sie auf die sTatistics Tab und klicken Sie dann auf das Excel-Symbol. Sichern Sie die Daten auf einen Daumen-Laufwerk und übertragen Sie es auf einen Flüssigkeits-Handling-Roboter-Computer.
- Öffnen Sie den Layout- und Methoden-Editor des Roboters. Öffnen Sie die Methodendatei "ODELAYDilution_v1.med". Führen Sie die ausführbare Datei "Convert_SynergyFiles.exe" aus. Geben Sie 0,09 für Ziel-OD600 ein.
- Klicken Sie auf "Glu Correction" und "Gal Correction" auf 0,05. Messen Sie leere Medien in einer identischen Platte. Klicken Sie auf "Datei generieren". Wählen Sie die Datei im Methodeneditor aus. Klicken Sie auf das Ampel-Symbol, um die Methode zu starten, um das Laufzeit-Verdünnungsprogramm zu öffnen.
- Vergewissern Sie sich, dass die Rohre nicht verschlossen sind und die Platten mit Deckel versehen sind und die Abdeckungen von den Spitzen entfernt sind. Legen Sie Platten, Rohre und Spitzen auf das Flaschenhandling Roboter Deck, wie durch das Deck-Layout angezeigt.
- Klicken Sie auf die Schaltfläche "Play", um das Verdünnungsprogramm auszuführen. Wählen Sie die richtige Anzahl von 50 μl Spitzen. Wählen Sie die richtige Anzahl von 300 μl Spitzen. Warte mal12 min für den laufenden lauf.
- Nehmen Sie die Verdünnungsplatte vom Roboter, bedecken Sie die Spitzen und rekapitulieren Sie die 15 ml Medienröhrchen. Kultur die Platte für 5 - 6 h bei 30 ° C.
- Etwa 1 - 2 h vor Beginn der zweiten Verdünnung stellen Sie sicher, dass die Inkubationskammern für das Mikroskop eingeschaltet werden. Lassen Sie sie bei ° C äquilibrieren oder die für den Versuch erforderliche Temperatur.
- Wiederholen Sie die Verdünnungsschritte 3.3 - 3.4, aber verdünnen Sie die Kulturen auf eine OD600 von 0,01 - 0,02, um die Kulturen auf Agaroseschlitten aufzuspüren.
- Die Verdünnungsplatte mit einer Metallgefrierdichtung abdecken.
- Sonde die Verdünnungsplatte in einem Eisbad für 30 s mit der Platte schweben in Eiswasser mit einer Zentrifuge Metall Eimer-Passung, um die Platte zu halten ( Abbildung 2A ). Übertragen Sie die beschallten Kulturen von der Verdünnungsplatte zu einer flachen Bodenplatte. Etiketten 4 x 96-well Flachbodenplatten 1, 2, 3 und 4 ( Abbildung 2B ).
- Transfer 150 &Nr. 181, L der Verdünnungsplatte aus den Vertiefungen A01 - D06 in die Wells C04 - F09 der mit 1 bezeichneten Platte. Dann werden 150 μl der Verdünnungsplatte aus den Vertiefungen A07 - D12 in die Wells C04 - F09 der mit 2 bezeichneten Platte überführt. Dann 150 μl übergeben Der Verdünnungsplatte aus den Vertiefungen E01 - H06 in die Vertiefungen C04 - F09 der mit der Beschriftung 3 versehenen Platte. 3. Umfassen Sie 150 μl der Verdünnungsplatte aus den Vertiefungen E07 - H12 in die Wells C04 - F09 der mit 4 bezeichneten Platte.
- Gehen Sie zu Schritt 4, Spotting auf Agar.
4. Spotting auf Agar mit einem automatisierten Liquid Spotting Roboter
- Entfernen Sie die bei 4 ° CO / N (ab Schritt 2.19) gespeicherten Agarose-Objektträger aus ihren befeuchteten sterilen Pipettenboxen.
- Wenn nötig, schneiden Sie die Ecken des Agarose-Mediums mit einer sauberen Rasierklinge ab, um sicherzustellen, dass sie auf die Schieberkammer passen.
- Entfernen Sie die Schiebeklemme von der Halterung. Setzen Sie die Agarplatte vorsichtig in den vertieften Bereich der Bühnenkammerhalterung.
HINWEIS: Stellen Sie sicher, dass die oderDie Entstehung der Folie ist vom Experiment bis zum Experiment konsistent. - Überprüfen Sie, ob die Klemme bündig mit der Unterseite der Kammer ist. Wenn es krumm ist, dann kann der Schieber nicht vollständig im vertieften Bereich sitzen. Setzen Sie den Nivellier-Abstandshalter in die mittlere Plattenposition des Spotting-Roboters. Dieser Abstandshalter berührt die Nivellierschrauben, so dass die Schieberkammer mit den Spitzen geebnet werden kann.
- Legen Sie die Teller auf den Tisch in der Reihenfolge ihres Quadranten. Die Platten 1, 2, 3 und 4 wurden von links nach rechts geordnet ( Abbildung 2B ).
- Legen Sie die Spitzen so aus, dass die inneren 24 Wells C04 - F09 mit Spitzen in 4 leere Tipboxen besetzt sind. Eine fünfte Box muss eine Spitze in der C10-Position haben, sowie 4 Spitzen, deren Enden in den Positionen A01, A12, H01 und H12 abgeschnitten sind. Diese 4 abgeschnittenen Spitzen sorgen für Stabilität für die Spitzenplattenklemme ( Abbildung 2C und 2D ).
- Entfernen Sie die obere Abdeckung von der slIde Kammerhalterung
- Legen Sie den ersten Tip-Punch in die Start-Site des Roboter Control Spotting-Programm. Dies sollte ein Loch in die Agarose, die später verwendet werden, um die Ausrichtung der Ursprungs-Koordinate verwendet werden. Legen Sie die Platte 1 auf den Flüssigkeitshandhabungsroboter, um den ersten Quadranten zu lokalisieren. Vergewissern Sie sich, dass der Deckel entfernt ist und fahren Sie mit dem Spotting-Programm fort.
- Überprüfen Sie, ob alle Stellen vorhanden sind. Warten Sie auch 30 Minuten, bis sie trocknen. Wenn die Flecken etwa 1 mm im Durchmesser sind, kann das Programm fortfahren. Entleeren Sie die gebrauchten Spitzen in den Biohazard-Behälter und legen Sie frische Tipps auf den Roboter. Tauschen Sie die Platte 1 für die 2. Quadrantenplatte aus.
- Wiederholen Sie für den dritten Quadranten. Und wiederholen Sie sich wieder für den 4. Quadranten.
- Wenn die Flecken trocken sind, ersetzen Sie die Schieberkammerabdeckung und schlagen Sie das Gerät um.
- Installieren Sie die Schlauchverbindungen mit einem Luftfilter.
- Legen Sie einen Glasschieber auf die Oberseite der Kammer.
HINWEIS: Diese Folie ist entscheidend für reduciNg den Wärmestrom zum Agar und minimiert die Bildung von Kondenswasser auf die Objektivabdeckungsseite der Kammer. Vergiss diesen Deckzettel nicht. Abhängig von der Mikroskopkonfiguration verhindert dieser Deckel, dass das Erwärmen von der Beleuchtungsseite zu einer Kondensation auf der Objektivseite führt. - Positionieren Sie einen Ventilator, um heiße Luft auf die Objektivseite der Kammer zu blasen. Dieser Ventilator verhindert das Ausbilden von Kondenswasser auf dem Deckel. Stellen Sie den Luftstrom durch den Bubbler auf 10 mL / min ein.
5. ODELAY auf dem Mikroskop ausführen
- Führen Sie das Skript "ODELAY_Microscope_Control.m" aus.
- Klicken Sie auf den "Shutter", um den Durchblendlicht zu öffnen und dann die "Focus" -Taste, um die hohe Kamera-Rate einzuleiten ( Abbildung 3A , Rote Pfeile).
- Klicken Sie auf "Go Origin" und verschieben Sie die Bühne, um die Ursprungsmarke zu finden, die im Agar gestanzt wurde.
- Dann ziehe dich leicht nach rechtsFokus auf Hefezellen im Bereich E07.
- Gehe zurück zum Ursprung-Punsch und zentriere ihn im Gesichtsfeld.
- Stellen Sie den Ursprung auf diesen Wert ein, indem Sie die Taste "set" drücken ( Abbildung 3A , blaue Pfeile). Gehen Sie nun zur Position H18 und fokussieren Sie mit der Sechskantschraube, die dieser Stelle am nächsten liegt. Dann in Position L07 bewegen und mit der Sechskantschraube fokussieren, die dieser Stelle am nächsten liegt. Dann in Position E07 verschieben und mit der Sechskantschraube fokussieren, die dieser Stelle am nächsten liegt.
- Wiederholen Sie die Schritte 5.5 - 5.7, wenn diese Positionen im Fokus bleiben.
- Überprüfen Sie den Fokus in der Mitte und an den Kanten an den Stellen E12, H12, L12. Stellen Sie den Autofokusbereich ein, wenn die Fokus-Z-Werte, wie durch den Z-Wert angezeigt, größer als ± der Autofokusbereich sind ( z . B. stellen Sie den Autofokusbereich auf 60 μm den Z-Wert für einen Mittelpunkt ein, der größer als ± ist 40 μm).
- Drücken Sie die Reset-Taste, da dies die Tasteneigenschaften aktiviert und drücken Sie dannODELAY ( Abbildung 3A , grüne Pfeile). Wählen Sie das Verzeichnis, um die Daten zu speichern. Stellen Sie sicher, dass genügend Festplattenspeicher vorhanden ist.
HINWEIS: Das Mikroskop sammelt Daten für 48 Stunden oder bis das Programm geschlossen ist.
6. Verarbeitung von ODELAY-Daten
- Öffnen Sie die ODELAY_IPT.exe oder verwenden Sie das Skript ODELAY_Image_Processing_Tool_v7.m Skript ( Abbildung 3C ).
- Vorbereiten einer * ODELAYExpDisc.xlsx Excel-Tabelle.
- Benennen Sie das Experiment in Zelle B1.
- Wählen Sie das Bildverzeichnis aus, in dem die Bilddateien E07 - H18 gespeichert sind.
- Wählen Sie das Datenverzeichnis aus, in dem Daten geschrieben werden sollen.
- Schreiben Sie das Datum, an dem das Experiment in die Zelle B04 gestartet wurde. Verwenden Sie das Format MM / TT / JJJJ.
- Schreiben Sie die Verdünnungszeit in Zelle C04. Diese Zeit dauert etwa fünf Minuten, bevor das erste Bild gesammelt wird. Verwenden Sie das Format HH: MMpm, wo HH für h und MM ist für min.
- Fügen Sie in die Stammnamen nach tO die Quellplatte (Schritt 3.1). Wegen der Art, wie Stämme entdeckt werden, werden sie auf der ODELAY-Agarose-Folie umgeordnet. Zellen in der Kalkulationstabelle B31-B126 sind die Quellplatte, während die Zellen C31 - C126 die Spotorte in der ODELAY Agarplatte sind.
- Dann betätigen Sie die Schaltfläche "Prozessdaten" und wählen Sie die * ODELAYExpDisc.xlsx, die gerade vorbereitet wurde.
- Warten Sie 16-24 h, abhängig vom Computersystem, das verwendet wird, um Daten zu verarbeiten.
- Wenn die Bilder verarbeitet werden, erscheint ein Verzeichnis mit dem Namen "ODELAY Well Data" und eine Datei "* _Index_ODELAYData.mat". Drücken Sie die Taste "Daten laden" und wählen Sie die gerade erzeugte Datei "* _Index_ODELAYData.mat" aus. Damit wird der gerade verarbeitete Datensatz geladen. Das "*" im Dateinamen wird nach dem in der Excel-Tabelle eingegebenen Experimentnamen aufgelistet.
- Nach dem Laden der Daten, überprüfen Sie es mit der Zeitleiste, die auf der Unterseite gefunden wird, klicken Sie auf ein Bild Quadrat, um Wachstumskurven zu sehen fOder diesen Ort, oder finden Sie einen Brunnen von Interesse in der Liste auf der linken Seite und laden Sie dann die Bilder für diese Liste.
- Erzeugen Sie Histogramme von Populationswachstumsparametern, indem Sie die Taste "Violine Plot" drücken. Dies erzeugt eine Darstellung, die in Fig. 4 oder Fig. 6 gezeigt ist .
Ergebnisse
Beispielbilder von Hefe, die in der Zeitraffer-Mikroskopie wachsen, sind in Fig. 3B gezeigt . Nach der Verarbeitung der Zeitrafferbilder ist ein repräsentativer Datensatz, der die Hefestämme BY4741 & BY4742 vergleicht, in Fig. 4 gezeigt . In diesem Beispiel-Datensatz gibt es sehr wenig Abweichung in der Verdopplungszeit zwischen verschiedenen Positionen auf der Platte. Wenn das Agarosemedium schlecht vorbereitet wird, dann wäre eine offensichtliche Abweichung sowohl bei der Verdopplungszeit als auch bei der Verzögerungszeit in den Fleckpositionen offensichtlich, die mit dem deformierten Bereich des Agarosegels übereinstimmen. Während die Verdopplungszeiten relativ gleichmäßig erscheinen, zeigt dieses Beispiel Variationen der Verzögerungszeitmessungen. Ein konsistenterer Datensatz ist in Abbildung 6 dargestellt . In diesem Datensatz sind sowohl Verzögerungszeit als auch Verdopplungszeit einheitlich.
Dateien / ftp_upload / 55879 / 55879fig1.jpg "/>
Abbildung 1: Agar Mould Assembly.
Die Komponenten der Agarform sind in ( A ) dargestellt. Montieren Sie den Sockel wie in ( B ) gezeigt und klemmen Sie den Sockel mit kleinen Binderclips. Legen Sie die längeren Ständer in den Hohlraum der Basis ( C ) und legen Sie dann die Schieber wie in ( D ) gezeigt in die Form. Eine Seitenansicht, die den Winkel der Form und die Ausrichtung des Formkörpers zeigt, der für eine gleichmäßige Trennung des Agars von dem Glasschieber ( E ) benötigt wird. Beachten Sie die Position des Daumens und der Zeigefinger sowie die Gerade des Agars, die sich von der Folie ( F ) trennt, und notieren Sie den horizontalen Pfeil. Die Trennlinie sollte sich gleichmäßig in Richtung der vertikalen Pfeile bewegen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
= "Jove_content" fo: keep-together.within-page = "1"> 
Abbildung 2: Sonication and Spotting Methode.
Sonden Sie die Platte in Eiswasser und verwenden Sie einen Zentrifugen-Eimerhalter, um die Platte ( A ) zu unterstützen. Legen Sie die Platten aus den Schritten 3.8.1 für das Aufsprühen auf die Agaroseplatte ( B ). Auch die Spitzen so anordnen, dass die linke meisten Kasten eine Spitze an Position C10 hat und dann vier andere Spitzen mit ihren Enden abgeschnitten, also stoßen sie nicht den Plattenhalter ( C ) an. Legen Sie die restlichen Spitzen in vier Boxen, so dass die inneren 24 Spitzenpositionen besetzt sind ( D ). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
55879fig3.jpg "/>
Abbildung 3: ODELAY Grafische Benutzeroberfläche.
Ein Screenshot der grafischen Benutzeroberfläche für "ODELAY_Microscopecontrol.m" ( A ). Diese Schnittstelle ermöglicht die Überwachung der Kamera und die Einstellung der Mikroskop-Beleuchtungseinstellungen für Epifluoreszenz- und Hellfeldmodi. Rote Pfeile zeigen auf die Focus- und Transmit-Tasten, die die Kamera aktivieren, um Bilder schnell zu erfassen und den Durchlicht-Shutter zu öffnen. Blaue Pfeile werden verwendet, um die Bühne für den Ursprung zu bewegen und dann den Ursprung mit der Taste "Go Origin" und "Set" zu setzen. Grüne Pfeile zeigen auf "Reset" und "ODELAY !!!" Tasten, die ODELAY-Bildmodi auf die aktuellen Bedingungen zurücksetzen und die ODELAY-Bildsammlung initiiert. Zeitraffer Bilder von Hefe, die auf festem Medium bei 0, 3, 6 und 9 h nach dem Spotting ( B ) wachsen. Ein Screenshot der grafischen Benutzeroberfläche für "ODELAY_IPT.m" oder th E ODELAY Bildverarbeitungswerkzeug ( C ). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 4: Beispiel ODELAY Ausgang.
Dieser Datensatz vergleicht die Stämme BY4741 und BY4742 auf YPD-Medien. Diese Figur ist ein Beispiel für eine gut vorbereitete Agarose-Folie; Autofokuseinstellungen sind jedoch nicht optimal. Die Daten, die in jeder Spalte von links nach rechts dargestellt werden, sind: die Tragfähigkeit in Log 2 des Kolonienbereichs; Verdopplungszeit, gegeben in min; Und Verzögerungszeit, gegeben in min. In diesem Beispiel verdoppeln sich die Verdopplungszeiten aller Flecken auf der Agar-Folie gut mit einer kleinen Menge an erhöhter Verdopplungszeit in Richtung der Säule. Allerdings variieren die Lagzeiten in diesem Datensatz erheblich._upload / 55879 / 55879fig4large.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
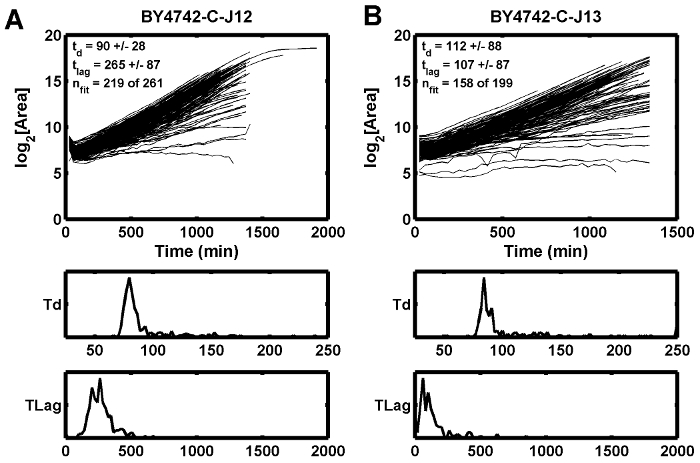
Abbildung 5: Growth Curve Beispiele.
Dieses Beispiel zeigt, wie ein schlechter Anfangsfokus die geschätzte Verzögerungszeit (t lag ) erhöhen kann ( A ), während die angrenzende Position eine kürzere Verzögerungszeit ( B ) aufweist. T d ist die Verdopplungszeit in min und t lag die Verzögerungszeit in min. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
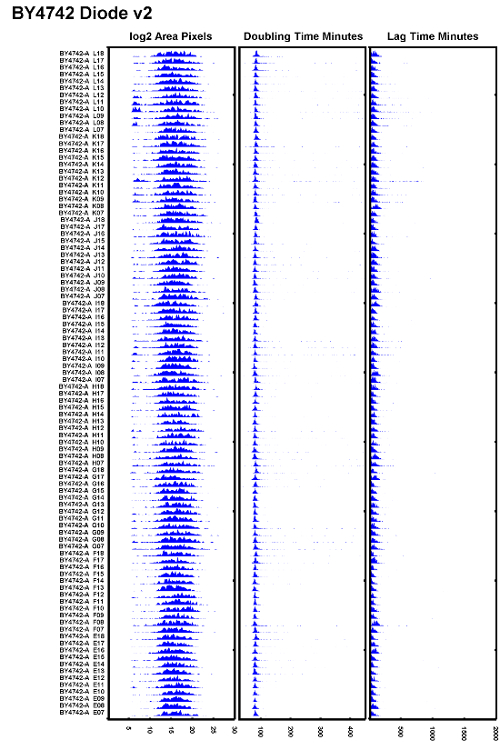
Abbildung 6: Beispiel für gut durchgeführte Testversuche.
Ein eBeispiel des BY4742-Stammes, der nach dem Austausch einer Wolfram-Halogenbirne mit einem Dioden-Illuminator getestet wurde und sichergestellt wurde, dass der Autofokus korrekt eingestellt ist. Alle verdoppelnden Zeiten scheinen sich gut zu überlappen, und die Verzögerungszeiten scheinen konsistent zu sein. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
Der ODELAY-Test hat mehrere kritische Punkte, um reproduzierbare und zuverlässige phänotypische Messungen zu gewährleisten. Der erste kritische Punkt ist die konsequente Vorbereitung der Hefekulturen. Es muss darauf geachtet werden, die Hefezellen aus logarithmischem Wachstum zu ernten. Wenn die Kulturen gesättigt sind, dann wird ihre Heterogenität der Bevölkerung erhöht, die Heterogenität durch genetische oder umweltbedingte ( z. B. Kohlenstoffquellen) Faktoren beeinträchtigen kann 11 . Der zweite kritische Punkt ist die konsequente Vorbereitung der Medien. Im Allgemeinen sollte ein großes Volumen von 10X-Stammmedien-Lösung generiert und dann im Laufe der Zeit verwendet werden, um Batch-Effekte zu minimieren. Die Formulierung von Medien nach Gewicht, wann immer möglich, hilft, die Konsistenz des Mediums im Laufe der Zeit zu verbessern, indem die Dichte des Agars sichergestellt wird und der Gesamtwassergehalt der Agarose genau überwacht werden kann. Der dritte kritische Punkt beinhaltet die Minimierung oder Beseitigung jeder mechanischen Verformung der Agarose michDia Eine mechanische Verformung des Mediums erfolgt am häufigsten bei der Trennung der Agarose aus den Glasscheiben. Wie bei vielen Labortechniken ist die Praxis erforderlich, diesen Schritt zu beherrschen.
Die Variation der Verzögerungszeit, wie in Fig. 4 dargestellt , ist oft mit einem der drei Faktoren verbunden: mechanische Verformung des Agarosemediums, Veränderung der geformten Agardicke oder einer instabilen Lichtquelle. Wenn sich das Agarose-Medium in der Z-Höhe über das gefleckte Array ändert, kann die Höhenvariation den Bereich der Autofokus-Routine überwältigen, wodurch die Anfangsbilder leicht unscharf werden. Aus diesem Grund überprüfen Sie die Fokushöhe an mehreren Stellen in der Mitte und entlang der Kanten des gefleckten Arrays, um sicherzustellen, dass die Autofokusroutine genügend Z-Bereich hat, um Fokus zu finden. Wenn nötig, verwenden Sie das Autofocus-Bedienfeld, um den Fokusbereich zu erhöhen und die Anzahl der Fokussierungsschritte zu erhöhen.
Ein dritter möglicher conditIonen, die zu einem schlechten Fokus führen können, ist eine instabile oder flackernde Lichtquelle, die die berechnete Fokusbewertung für eine bestimmte Z-Höhe stören kann. Wolfram Halogenbirnen neigen dazu, gut zu flackern, bevor die Birnen ausbrennen. Die Wirkung eines schlechten Fokus wird in einem Beispiel beobachtet, bei dem die Wachstumskurven zwischen den ersten und zweiten Zeitpunkten eintauchen ( Abbildung 5 A ), während der benachbarte Punkt nicht das gleiche Dip hat ( Abbildung 5 B ). In diesem Fall wurde die schlechte Fokusbedingung durch Ersetzen der Wolfram-Halogen-Lichtquelle gemildert.
In der Praxis haben die Autoren festgestellt, dass, um das Flackern von 100W-Wolfram-Halogenlampen zu reduzieren, die Glühbirnen alle 500 h oder etwa alle 2 Monate ausgetauscht werden müssen, wenn die Mikroskope stark untergebracht sind. Um eine schlechte Fokusausgabe von einer flackernden Glühlampe zu vermeiden, ersetzen Sie die Wolfram-Halogen-Lichtquelle oft oder ersetzen Sie die Halogenlampe mit einer Diodenlichtquelle. EinBeispiel eines Datensatzes, der eine geringe Variation der Verdopplungszeiten sowie einheitlichere Verzögerungszeiten zeigt, ist in Fig. 6 gezeigt . Dieser Datensatz wurde mit einem Dioden-Illuminator aufgenommen, der eine stabilere Ausleuchtung über die Zeit bietet, während er den Autofokus durchführt.
Während viele der hier erwähnten Punkte zur Optimierung der Medienvorbereitung offensichtlich erscheinen, zeigen sich in der Literatur die meisten Großbildschirme nicht gut miteinander, 8 , 11 . Daher haben wir sorgfältig die Vorbereitung von Kulturen und Agarose-Medien beschrieben, so dass mehr reproduzierbare phänotypische Bildschirme erzeugt werden können.
Der ODELAY-Assay ist derzeit im Durchsatz im Vergleich zu Pinning-basierten Assays wie synthetischen genetischen Arrays oder dem Scan-O-Matic-Assay begrenzt. Während diese Methoden die Anzahl der gemessenen Stämme erhöhen, fehlt ihnen die Fähigkeit, einzelne Zellen zu lösenDaher kann man die Heterogenität der Bevölkerung nicht messen, die wir in klonalen Hefestämmen beobachten. Der Ursprung dieser Heterogenität wird derzeit nicht verstanden, aber die hier beschriebene Verschmelzung von Technik und Berechnungen bietet eine Möglichkeit zur objektiven Adressierung der zugrunde liegenden zellulären Mechanismen 12 .
Die Autoren möchten beachten, dass ODELAY derzeit nur für eine bestimmte Mikroskopmarke und Körpertyp optimiert ist. Das Ändern von ODELAY für andere Mikroskopsysteme ist einfach, erfordert jedoch Kenntnisse der Open Source API 13 . Allerdings werden sowohl die API als auch die ODELAY Scripts geschrieben, um leicht an verschiedene Systeme und experimentelle Assays angepasst zu werden.
Während ODELAY ursprünglich für Hefe entwickelt wurde, konnten wir es ohne Veränderung nutzen, um das Wachstum von Mycobacterium smegmatis zu beobachten. Die Beobachtung der anderen Kolonie bildenden Mikroorganismen istMöglich mit Änderungen am Quellcode 11 . Im Allgemeinen ist ODELAY ein leistungsfähiges und flexibles Werkzeug für den Vergleich von Mikroorganismen, die unter verschiedenen Umgebungsbedingungen und genetischen Störungen gewachsen sind.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Autoren bestätigen die Unterstützung für diese Arbeit durch Zuschüsse U54 RR022220 und P50 GM076547 an JDA von den US National Institutes of Health. FDM ist ein Postdoktorand mit den kanadischen Instituten für Gesundheitsforschung. Wir danken dem luxemburgischen Zentrum für Systembiomedizin und der Universität Luxemburg für die Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose UltraPure | ThermoFisher | 16500500 | Gel Temp 36C, Gel Strength (1%) 1.2 g/sq cm |
| Yeast Extract Peptone (YEP) | Fisher Scientific | BP1422-2 | |
| Complete Suplement Mixture (CSM) | Fisher Scientific | MP114560222 | |
| Polyethylene Glycol 3350 (av. mol. wt. 3000-3700) | SigmaAldrich | P2906 | |
| Yeast Strain BY4741 | ThermoFisher | 95400.BY4741 | |
| Yeast Strain BY4742 | ThermoFisher | 95400.BY4742 | |
| 50 mL Falcon tubes | Corning | 430291 | 1 case |
| 15 mL Falcon tubes | Corning | 352096 | |
| 2 x 3 inch 1.0 mm thick slides 1/2 gross | VWR | 48382-179 | |
| 96-well plate flat bottom | Corning | 353072 | |
| Hydra liquid handleing robot | Thermo | 1096-DT-100 | |
| Hamilton Microlab Star Liquid Handleing Robot | Hamilton | ||
| hydra 100 mL tips Extended Length DARTS | Thermo | 5527 | |
| Synergy H4 Plate Reader | Biotek | H4MLFAD | |
| Leica DMI6000 B Microscope | Leica | ||
| Leica 10X/0.3NA objective | Leica | 11506289 | |
| Hamamatsu ORCA Flash 4.0 Camera | Hamamatsu | C11440-22CU | |
| MATLAB with image processing tool box | Mathworks | ||
| MicroManager | Open Imaging | https://micro-manager.org/ | |
| ODELAY Microscope Control (MATLAB scripts and GUI) | www.aitchisonlab.com\ODELAY for Matlab scripts and software | ||
| ODELAY Microscope Chamber | www.aitchisonlab.com\ODELAY for Mechanincal Drawings | ||
| ODELAY Agar Molds | www.aitchisonlab.com\ODELAY for mold drawings |
Referenzen
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van't Riet, K. Modeling of the bacterial growth curve. Appl Environ Microbiol. 56 (6), 1875-1881 (1990).
- Sellick, C. A., Campbell, R. N., Reece, R. J. Galactose metabolism in yeast-structure and regulation of the leloir pathway enzymes and the genes encoding them. Int Rev Cell Mol Biol. 269, 111-150 (2008).
- Yoshikawa, K., et al. Comprehensive phenotypic analysis for identification of genes affecting growth under ethanol stress in Saccharomyces cerevisiae. FEMS Yeast Res. 9 (1), 32-44 (2009).
- Bryan, A. K., Goranov, A., Amon, A., Manalis, S. R. Measurement of mass, density, and volume during the cell cycle of yeast. Proc Natl Acad Sci U S A. 107 (3), 999-1004 (2010).
- Baryshnikova, A., et al. Quantitative analysis of fitness and genetic interactions in yeast on a genome scale. Nat Methods. 7 (12), 1017-1024 (2010).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327 (5964), 425-431 (2010).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446 (7137), 806-810 (2007).
- Zackrisson, M., et al. Scan-o-matic: High-Resolution Microbial Phenomics at a Massive Scale. G3 (Bethesda). 6 (9), 3003-3014 (2016).
- Bean, G. J., Jaeger, P. A., Bahr, S., Ideker, T. Development of ultra-high-density screening tools for microbial 'omics'. PloS One. 9 (1), e85177 (2014).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS Biol. 10 (5), e1001325 (2012).
- Herricks, T., et al. One-Cell Doubling Evaluation by Living Arrays of Yeast. ODELAY! G3 (Bethesda). 7 (1), 279-288 (2017).
- Mast, F. D., Ratushny, A. V., Aitchison, J. D. Systems cell biology. J Cell Biol. 206 (6), 695-706 (2014).
- Edelstein, A. D., et al. Advanced methods of microscope control using µManager software. J Biol Methods. 1 (2), e10 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten