Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erhebung und Verarbeitung von Lymphknoten von großen Tieren für RNA-Analyse: Vorbereitung auf Lymphknoten Transkriptomischen Studien der großen Tierarten
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet einen Überblick über Verfahren zur Isolierung von RNA für die transkriptomischen Profilierung der Lymphknoten Gewebe von großen Tieren, einschließlich Schritte bei der Identifizierung und Entfernung der Lymphknoten von Vieh und Wildtiere, Probenahme Ansätze Konsistenz über mehrere Tiere, und Überlegungen sowie repräsentative Ergebnisse für die Post-Sammlung, Erhaltung und Verarbeitung für RNA-Analyse zu schaffen.
Zusammenfassung
Große Tiere (Vieh und Wildtiere) dienen als wichtiges Reservoir von Zoonoseerregern, einschließlich Brucella, Mycobacterium Bovis, Salmonellenund Escherichia Coli, und eignen sich für die Untersuchung der Pathogenese und/oder Ausbreitung der Bakterien in natürlichen Wirte. Mit der wichtigen Funktion der Lymphknoten in der Host-Immunantwort dienen Lymphknoten Gewebe als eine potentielle Quelle von RNA für nachgeschaltete transkriptomischen Analysen, um die zeitlichen Veränderungen in der Genexpression in Zellen im Verlauf einer Infektion zu bewerten. Dieser Artikel bietet eine Übersicht über den Prozess der Lymphknoten Sammlung, Gewebe-Sampling und nachgelagerten RNA Verarbeitung in Vieh, weitere Beispiele zur Verfügung gestellt von der amerikanischen Bison (Bison Bison Rind (Bos Taurus) als ein Modell mit ). Das Protokoll enthält Informationen über die Lage, Ermittlung und Entfernung der Lymphknoten aus mehreren wichtigen Standorten im Körper. Darüber hinaus wird eine Biopsie Sampling-Methode vorgestellt, die für eine Konsistenz der Probenahme über mehrere Tiere ermöglicht. Einige Überlegungen zur Probe Erhaltung werden diskutiert, einschließlich der Erzeugung von RNA geeignet für nachgeschaltete Methoden wie RNA-Sequenzierung und RT-PCR. Aufgrund der langen Verzögerungen große Tier vs. Maus Zeitstudien Kurs innewohnt sind repräsentative Ergebnisse von Bisons und Rinder Lymphknoten Gewebe präsentiert um zu beschreiben, den zeitlichen Verlauf der Abbau in diesem Gewebetyp, im Rahmen einer Überprüfung der methodische Vorarbeiten auf RNA-Abbau in anderen Geweben. Insgesamt wird dieses Protokoll sinnvoll, beide tierärztliche Forscher Anfang Transkriptom Projekte auf große Tiere Proben und Molekularbiologen Lerntechniken für in Vivo Gewebe-Probeentnahme und in-vitro- Verarbeitung interessiert sein.
Einleitung
RNA-Sequenzierung Analyse des transkriptoms Lymphknoten bietet die Möglichkeit, die Immunantwort von Tieren zu einer Vielzahl von Krankheitserregern zu charakterisieren. Während diese Methode bei Mäusen ausgiebig genutzt wurden, haben vor kurzem Analysen in größeren Säugetiere1,2ausgebaut. Vieh/großes Tier Lymphknoten kann verwendet werden, um Host Reaktion auf eine Infektion, nicht nur für den Gebrauch im Impfstoff oder genetische Anfälligkeit Studien und für die Identifizierung von Zielen für die Entwicklung von Medikamenten, sondern auch als Modellsysteme für Studien am Menschen zu charakterisieren über Zoonosen. Zum Beispiel im Falle von Brucellose (eine bakterielle Zoonose, dass Auswirkungen eine halbe Million auf der ganzen Welt jedes Jahr Menschen), trotz deutlich erhöhte Kosten, Studien bei Schafen oder Ziegen sind relevanter für die Infektion beim Menschen und menschliche Impfstoff Entwicklung als Labor Tiermodelle. Maus Infektionsmodelle rekapitulieren die retikuloendothelialen System Infektion aber nicht die charakteristischen klinischen Symptome3.
Im großen Tierversuch im Vergleich zu tierischen Laborstudien umfasst den Prozess des Gewebes Ernte unbedingt eine längere Verzögerung zwischen der Euthanasie und die Gewebe-Kollektion, die eine mögliche für die Erhaltung der Herausforderung qualitativ hochwertige RNA. Intakte RNA ist essentiell für die Generation der biologisch relevanten transkriptomischen Daten. Die Erzeugung von qualitativ hochwertigen RNA aus Gewebeproben erfolgt im Containment Einrichtungen für große Tiere Pathogen Studien besonders kritisch. Solche Studien sind von Natur aus schwieriger durchzuführen, da sie nicht nur genehmigte Anlagen und hoch qualifiziertes Personal erfordern aber auch erhebliche finanzielle Kosten, die abhängig von der Arbeit von Dutzende bis Hunderte von Tausenden von Dollar reichen können. Diese Arten von Studien beinhalten auch eine interdisziplinäre Zusammenarbeit und fachübergreifende Kenntnisse für ihren Abschluss, ihre Komplexität hinzufügen. Daher bietet Ausbildung auf, Entwicklung und Einhaltung ein optimiertes System für die Entnahme von Proben und Erhaltung erhebliche Vorteile für nachgeschaltete molekulare Untersuchungen von Geweben von infizierten Tieren.
Die Sammlung von größeren Lymphknoten präsentiert zusätzliche Herausforderungen für die Gewebe-Sammlung im Vergleich zu ähnlichen Probenahme von murinen Lymphknoten. Die Vorbereitung auf die Probe-Exzision erfordert ein grundlegendes Verständnis der Anatomie des Lymphknotens, einschließlich der relevanten internen Strukturen. Die Struktur eines Lymphknotens besteht aus lymphoiden Läppchen umgeben von Nebenhöhlen mit Lymphe gefüllt. Diese Strukturen sind in eine zähe, fibröse Kapsel eingeschlossen. 4 eine lymphoide Lobule ist die "grundlegende anatomische und funktionelle Einheit des Lymphknotens" und besteht aus Follikel, eine tief kortikale Einheit und medulläre Schnüre und Nebenhöhlen4 (Abbildung 1A). B- und T-Lymphozyten sind Heimat der Follikel und tiefen kortikalen Einheiten. Diese Strukturen bieten eine 3D Gerüst und der Interaktion zwischen Lymphozyten und Antigen oder antigenpräsentierende Zellen.
Grob, Follikel und tiefen kortikalen Einheiten identifiziert werden können auf Schnittfläche als sie eine dichtere netzartige Geflecht enthalten und dunkler als die Nebenhöhlen, die bestehen aus einem zarteren netzartige Geflecht und heller erscheinen (Abbildung 1 b). Gemäß der Konvention beziehen sich Pathologen auf die Regionen der Lymphknoten als oberflächliche Kortex (Follikel), Parakortex (tief kortikalen Einheiten) und der Medulla (medulläre Schnüre und Nebenhöhlen). Eine ordnungsgemäße Untersuchung aller drei Regionen wurde als beste Praxis in Routine pathologischen Untersuchung Leitlinien für Lymphknoten5angesehen. Beachten Sie, dass es erhebliche Unterschiede in der Konsistenz, Größe und Farbe der Lymphknoten, auch innerhalb eines einzelnen Tieres. Zunehmendem Alter der Tiere ihre Lymphknoten wird tendenziell kleiner und werden fester als bei jüngeren Tieren, in der Regel auf eine Erhöhung ihrer Bindegewebe und eine Verminderung der normalen lymphatischen Struktur6,7.

Abbildung 1. Anatomie des Lymphknotens. (A) Diese Karikatur zeigt die Anatomie des Lymphknotens, Schlüsselstrukturen darstellt. (B) dieses noch Bild zeigt einen bovinen Lymphknoten schneiden im Querschnitt. Die relevanten Strukturen/Layer, die mit dem bloßen Auge sichtbar sind, werden hervorgehoben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Abhängig von der experimentellen Frage wird verschiedenen Lymphknoten für die Erhebung und Analyse von Interesse sein. Peripheren Lymphknoten sind diejenigen befindet sich tief im subkutanen Gewebe. Bei Rindern, periphere oder oberflächlichen Lymphknoten oft verwendet in der klinischen und experimentellen Praxis umfassen Parotis, mandibulären, retropharyngeal, prescapular, Thymus (precrural) und oberflächlichen inguinalen (Supramammary bei Frauen, bei Männern scrotal) () ( Abbildung 2). In Tabelle 1sind die Eigenschaften der wichtigsten oberflächlichen Lymphknoten, wie beschrieben in die Rinder System8beschrieben. Im folgenden sind einige mögliche Lymphknoten Sammlung Pläne für bakterielle Infektionskrankheiten von Rindern als Ausgangspunkt für die Untersuchung vorgestellt.
Brucella Abortus/Brucella Melitensis: Standard necropsies für B. Abortus-infizierten Rindern und B. Melitensis-infizierten Ziegen am National Animal Disease Center zu erholen, Supramammary, Prescapular und Parotis Lymphknoten Gewebe , sowohl für das Schleifen für die bakterielle Enumeration und für die RNA-Vorbereitung für den Host RNA Ausdruck profilieren. B. Abortus zurückgewonnen werden regelmäßig in jedem der diese Lymphknoten in experimentell infizierten Rindern9. Das Vorhandensein von Bakterien in jedem solcher Lymphknoten nachweisbar in B. Melitensis-infiziert Ziegen bis zu mindestens neun Monate nach der Infektion mit der RNA-basierten Methoden aus unseren Studien (Boggiatto Et Al., unveröffentlicht). Salmonellen SP.: Prescapular, subiliac (Thymus), mesenterialen Lymphknoten nützlich erwiesen haben, während die Profilierung der Rinder Kadaver Salmonellen Prävalenz10,11,12 und wäre von Interesse für transkriptomischen Studien. E. Coli O157: H7: mesenterialen Lymphknoten (in den mittleren Dünndarm und distalen Dünndarm Standorten) sind die Niederlassungen einer gelegentlichen Erholung der Bakterien im infizierten Kälbern (aber nicht bei infizierten Erwachsenen Rindern)13. Leptospirose (Leptospira SP.): eine chronische Persistenz der Bakterien in den Lymphknoten Entwässerung der Brustdrüse14beobachtet worden. Mycobacterium bovis : Bei Rindern wurden die Bakterien wieder Post-experimentelle Infektion von mediastinalen und tracheobronchialer Lymphknoten Kälber15. Darüber hinaus ist Lymphknoten RNA verwendet worden, um große Tier Host Reaktionen auf Viren, wie die Schweine reproduktive und respiratorische Syndrom Virus2zu untersuchen. Abbildung 2 zeigt den Speicherort der eine Teilmenge von diesen großen Lymphknoten im Körper Rinder.
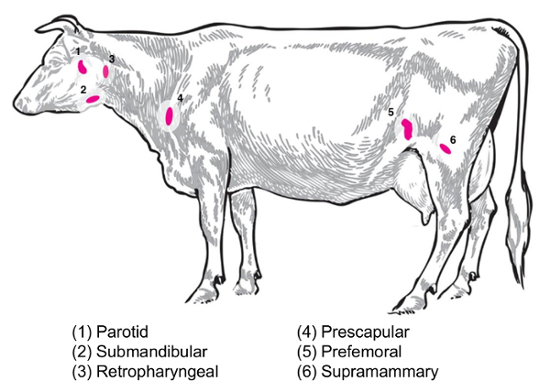
Abbildung 2: Cartoon Darstellung ausgewählter Lymphknoten Standorte in Bos taurus . Die nummerierten Lymphknoten sind kommentiert. Bitte klicken Sie hier für eine größere Version dieser Figur.
In diesem Papier und das zugehörige Video präsentieren wir ein Protokoll für die Isolierung von großen Tieren Lymphknoten für RNA-Studien, soll informativ für Molekularbiologen transkriptomischen Studien von großen Tieren Infektionen beteiligt sein. Erstens bieten wir einen Überblick über die Isolierung-Verfahren für die Lymphknoten mit Probenahme von Rindern und Bisons Gewebe als Beispiele. Gepaart mit dieser Demo, wie im Video dargestellt, ist ein Workflow für eine reproduzierbare Gewebe Probenahme zur RNA-Isolierung. Anschließend beschreiben wir wichtige Überlegungen für die Verarbeitung eines infizierten Lymphknotens mit Fokus auf Sicherheit, Beständigkeit und RNS-Qualität.
Die Vorbereitung der RNA aus dem Gewebe mit einem versauerten Phenol-Guanidin erfolgt Reagenz basiert auf der Originalmethode von Chomczynski und Sacchi16,17, mit einer Reinigung über Silica-basierten Spin Spalten in Anwesenheit von chaotropen Agenten basierend auf der ursprünglichen Arbeit von Vogelstein und Gillespie18. Wir untersuchen auch das Potenzial für die Verwertung von RNA für Transkriptom von Rindern Lymphknoten durch alternative Methoden haltbar gemacht. Schließlich beschäftigen wir die Auswirkungen der Zeitvariablen RNS-Qualität in großen Tier Autopsien, einschließlich eine repräsentativere Experiment Darstellung der Wirkung einer Erhöhung in der Zeit zwischen der Euthanasie und die Probenahme auf dem wiederhergestellten RNA-Profil von Bison und Bovine Lymphknoten. Dieser Artikel wird nicht nur Molekulare Biologen, sondern auch zu tierärztliche Forscher beginnend transkriptomischen Studien nützlich.
Protokoll
Die hier abgebildeten Tier Autopsie-Verfahren fallen unter zugelassenen IACUC Protokolle an die National Animal Disease Center, Ames, IA. Alle Experimente wurden gemäß den genehmigten Richtlinien für Tierhaltung und Tierschutz.
1. Vorplanung vor der Autopsie
- Bereiten Sie vor der Autopsie die folgenden Materialien für den Transport nach der Autopsie-Suite:
- 1,5 oder 2 mL Mikrozentrifugenröhrchen gefüllt mit ~ 1,0 mL RNA-Erhaltung-Lösung in einem Rack oder Transport-Container. Achten Sie darauf, die Anweisungen des Herstellers für das Verhältnis des Volumens der Bewahrung Lösung auf das Volumen des Gewebes zu folgen.
- Einweg-Skalpelle und saubere, autoklaviert Zange: einen Satz für sezieren die Haut und einen anderen Satz für das Sammeln der Lymphknoten (eine für jede Lymphknoten, pro Tier), und verhindert so eine Cross-Kontamination der Haut und der Umwelt Flora mit den Lymphknoten Gewebe.
- 3 Stanzen mm Biopsie Werkzeuge, Autopsie Messer, Sharps Container für gebrauchte Skalpelle und Biopsie Werkzeuge und Schneidbretter oder Einweg-Schalen für den Einsatz in den Lymphknoten-Dissektion.
- Verwenden Sie persönliche schützende Ausrüstung, einschließlich der EVP für die Arbeit auf der entsprechenden BSL-Ebene für das System erforderlich.
- Autoklaven wieder verwendbare Metallwerkzeuge, einschließlich der Zange in Metall Töpfe vor der Autopsie, die Utensilien/trockene Ware einstellen.
2. Identifikation und Auswahl von Lymphknoten bei Rindern und Bisons
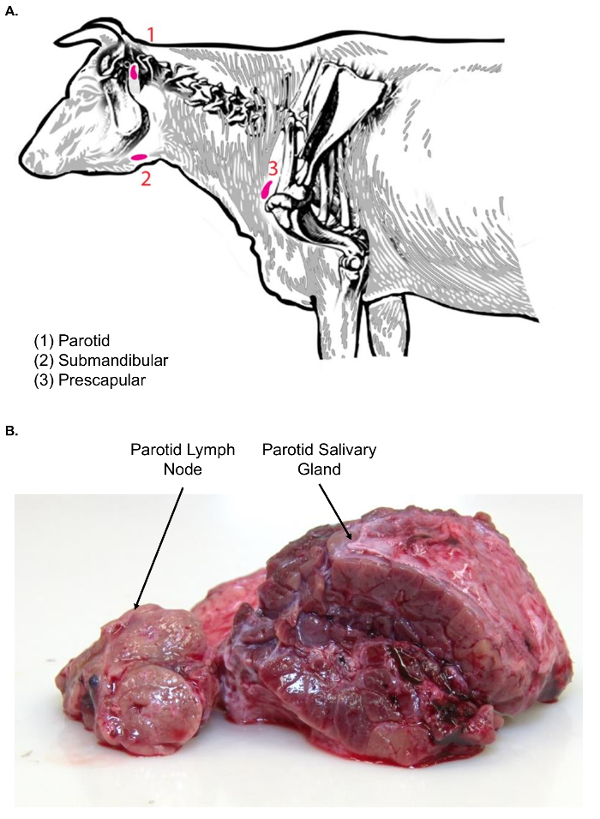
Abbildung 3 . Lymphknoten des bovinen Kopf- und halskrebs. (A) Diese Karikatur zeigt ausgewählte Lymphknoten von Kopf und Hals von Bos Taurus. (B) zeigt dieses Bild die Parotis Lymphknoten im Querschnitt (links) im Vergleich zu der Parotis Speicheldrüse im Querschnitt (rechts). Beachten Sie den Unterschied zwischen den beiden Gewebetypen in Texturen. Bitte klicken Sie hier für eine größere Version dieser Figur.
-
Isolierung der prescapular Lymphknoten bei Rindern und Bisons
- Einschläfern des Tieres (Rind oder Bison jeden Alters, je nach Bedarf für das Experiment) basierend auf dem lokalen IACUC-Protokoll. Bestätigen Sie den Tod durch Methoden, die speziell in der institutionellen IACUC Protokoll, einschließlich den Mangel an einem Herzschlag, das Fehlen der Atmung und das Fehlen der Hornhaut Reflex beschrieben. Bewegen Sie das Tier auf den Boden der Autopsie.
Hinweis: Es gibt verschiedene Methoden der humane Euthanasie in der Laborpraxis von großen Tieren verwendet. Die intravenöse Gabe von Natrium Pentobarbital und Phenytoin-Natrium-Lösung ist jedoch die am häufigsten genehmigt und Form genutzt. Beachten Sie die AVMA Richtlinien für die Euthanasie von Tieren. 19
Achtung: Befolgen Sie alle relevanten Biosafety-Protokolle für eine Autopsie an der Gasthochschule, einschließlich der Richtlinien für die sichere Entsorgung von Kleie in einem pannensicheren Container und die Dekontamination von wiederverwendbaren Metall Messer und Zangen. Dieses Protokoll generiert Sharps Abfälle aus der Verwendung von Einweg-Skalpelle. Tragen Sie persönlichen Schutzausrüstung, wie durch die institutionellen Richtlinien, einschließlich Overalls, Stiefel, Handschuhe und Augenschutz vorgeschrieben. - 3 Teilnehmer an der tierischen Geweben arbeiten zu identifizieren.
Hinweis: Hier Sezierer 1 wird sein verantwortlich für die groß angelegte Schneiden der gewünschten tierischen Gewebe, Sezierer 2 wird der Lymphknoten von den tierischen Sektionen zu erholen und Sezierer 3 funktioniert an einem separaten Tisch, Abschnitte von jeder Lymphknoten zu isolieren und zu Röhren mit einem Konservierungsmittel Lösung übertragen. - Beginnen Sie, indem man das Tier im seitlichen liegen.
- Erweitern Sie die Schulter durch Verschieben der vorderen Gliedmaßen zurück auf die Ebene des Knies (Carpus); Diese Bewegung wird die Schulter zurück zu bringen und ermöglichen ein leichter Abtasten der Lymphknoten. Das Gefühl für das Vorhandensein des Lymphknotens "Klumpen".
Hinweis: Bei Erkrankungen, Lymphadenopathie möglicherweise vorhanden, was erleichtert die Identifizierung der Lymphknoten, aber dieser Prozess kann auch Auswirkungen auf die Konsistenz, die Zusammensetzung und die Architektur des Lymphknotens. - Verwenden Sie sobald diese gefunden ist, und mit der Schulter noch erweitert, ein Skalpell oder Autopsie Messer um ein Hautschnitt entlang der Kontur der Schulter.
Hinweis: Beschränken Sie die Größe des Schnittes, nicht, auch wenn die Lymphknoten liegt über Abtasten wurde. Ein größeren Schnitt ermöglicht leichteren Zugriff auf tiefer gelegene Gewebe und erleichtert die Sezierer der Exzision des Lymphknotens. - Mit einer Zange, ziehen Sie die Haut um den Punkt der Schulter und Nacken Muskulatur.
Hinweis: Die prescapular Lymphknoten findet oft im subkutanen Fett, vor allem bei overconditioned Tieren. - Wieder einmal ertasten Sie die Position des Lymphknotens (Abb. 3A).
Hinweis: Anders als das subkutane Fett hält die Lymphknoten seine Form beim getastet. Darüber hinaus sind Lymphknoten nicht zusammenhängend mit einer umgebenden Gewebe, wie sie von einer dünnen fibrösen Kapsel eingeschlossen sind. - Nach dem Auffinden der prescapular Lymphknoten, sorgfältig das subkutane Fett, mit einem Skalpell und Pinzette, sezieren Sie und isolieren Sie den Lymphknoten.
- Suchen Sie nach das Vorhandensein von einer dünnen fibrösen Kapsel, wie in Abbildung 1 b, um die Lymphknoten und Fettgewebe unterscheiden; Lymphknoten sind nicht zusammenhängend mit einer umgebenden Gewebe.
- Übertragen Sie die Lymphknoten an den Lymphknoten Dissektion Tisch, mit einem Plastikbehälter für die Gewebe-Übertragung für weiter Schnitt.
Hinweis: Die Darstellung dieser Lymphknoten im Bison ist ähnlich. Das zugehörige Video zeigt das Erscheinungsbild des prescapular Lymphknotens als seziert aus der rechten Schulter von einem amerikanischen Bison.
- Einschläfern des Tieres (Rind oder Bison jeden Alters, je nach Bedarf für das Experiment) basierend auf dem lokalen IACUC-Protokoll. Bestätigen Sie den Tod durch Methoden, die speziell in der institutionellen IACUC Protokoll, einschließlich den Mangel an einem Herzschlag, das Fehlen der Atmung und das Fehlen der Hornhaut Reflex beschrieben. Bewegen Sie das Tier auf den Boden der Autopsie.
-
Isolierung der Parotis Lymphknoten bei Rindern und Bisons
- Beginnen Sie, indem man das Tier im seitlichen liegen auf dem Boden der Autopsie. Suchen Sie das Kiefergelenk (TMJ).
- Wenn dies schwer zu lokalisieren ist, bewegen Sie den Unterkiefer langsam und bestimmen Sie den Speicherort, an dem der Unterkiefer mit dem Schädel verbunden; Dies ist das TMJ.
- Machen Sie einen Einschnitt in die Haut mit einem Skalpell oder Autopsie Messer sorgfältig werden. Starten des Schnittes auf das Gelenk dorsal und ventral/vertikal entlang der Jawline zu verlängern.
- Sezieren Sie die Haut entfernt, um das subkutane Gewebe sichtbar zu machen. Der Parotis Speicheldrüse ist erstens wegen seiner großen Größe, gelappt Oberflächenaussehen und rote Färbung sichtbar.
- Pinzette und Skalpell, bewegen Sie mit Hilfe der Parotis Speicheldrüse beiseite, der Parotis Lymphknoten sitzen nur kranial und medial der Speicheldrüse (Abbildung 3A) zu finden. Prüfen Sie die Parotis Lymphknoten Gewebe für Textur vor der Weiterverarbeitung.
Hinweis: Lymphknoten des Gesichts (Parotis und mandibulären) sind mit Speicheldrüsen, die die Lymphknoten Isolation verwirren können. Im Vergleich zu den Lymphknoten Speicheldrüsen sind wesentlich größer und haben eine lappenden Oberflächenaussehen. Darüber hinaus auf Schnittfläche, Speicheldrüsen sind homogen in der Natur und weisen nicht auf die klassische kortikalen und medulläre architektonische Muster der Lymphknoten (Abb. 3 b). - Verbrauchssteuern der Drüse mit einem Skalpell. Übertragen sie die Lymphknoten Dissektion Tabelle, mit einem Plastikbehälter für die Gewebe-Übertragung für weiter schneiden.
- Beginnen Sie, indem man das Tier im seitlichen liegen auf dem Boden der Autopsie. Suchen Sie das Kiefergelenk (TMJ).
-
Isolierung der Supramammary Lymphknoten bei weiblichen Rindern und Bisons
- Legen Sie das Tier im seitlichen liegen.
- Das Hinterbein zu entlarven die inguinalen Region und suchen das Euter zu erheben.
Hinweis: Bei einem gesunden, nicht-stillende Tier kann Abtasten der diese Lymphknoten nicht möglich. In einer laktierenden Tieren möglicherweise diese Lymphknoten spürbar an der Caudale Grenze der Basis des Euters. - Mit einem Skalpell, einen Einschnitt machen Sie nur dorsal zum Euter, entlang der Oberschenkel, die Supramammary Drüsen verfügbar zu machen.
- Sezieren Sie das subkutane Gewebe zu und finden Sie die Supramammary Lymphknoten, eingebettet in einer dünnen Schicht des subkutanen Fettgewebes. Wenn nicht visuell unterscheidbar, ertasten Sie das exponierte Gewebe auf das Vorhandensein einer festen Struktur innerhalb des subkutanen Fettgewebes.
- Mit Pinzette und Skalpell, sorgfältig aus den Lymphknoten aus dem Fett zu sezieren. Übertragen sie die Lymphknoten Dissektion Tabelle, mit einem Plastikbehälter für die Gewebe-Übertragung für weiter schneiden.
3. Schneiden und Lagerung von Lymphknoten
- Übergeben Sie die isolierte Lymphknoten aus Sezierer 2 bis 3 auf einem Seziertisch angrenzend an den wichtigsten Autopsie Raum dissector. Legen Sie jeder Lymphknoten auf einem frischen ein Schneidebrett. Pre-label Schneidebretter in Abschnitten von Lymphknoten, um den Empfang von mehreren Lymphknoten in Folge zu erleichtern. Verwenden Sie für infektiöse Proben Einweg-Schalen.
- Mit Pinzette und einem Einweg Skalpell oder Autopsie Messer, einen Abschnitt des Lymphknotens entfernen. Mit einem scharfen Messer, machen Sie einen sagittalen Schnitt zu öffnen, den Lymphknoten.
- Untersuchen Sie das Innere des Lymphknotens, vor allem in Proben von infizierten Tieren, auf der Suche nach Asymmetrie, Läsionen, Farbunterschiede usw. Notieren der Beobachtungen.
-
(Option 1) Kleinen Gewebeschnitte
- Verwenden Sie Einweg-Skalpelle, um Stücke von jedem Lymphknoten Verbrauchsteuern, die weniger als 5 mm in der Dicke und jedes Stück zu einem Schlauch mit einer RNA-Erhaltung-Lösung mit einer sauberen Pinzette zu übertragen.
-
(Option 2) Lymphknoten-Biopsie oder schneiden-Methoden
Hinweis: Biopsien oder speziellen Methoden, angewandt auf parallele Lymphknoten Abschnitte über verschiedene Knoten und Tiere, bieten Konsistenz der Stichprobe Gewebe für eine Massenanalyse RNA.-
Keil-Schnitt Methode
- Nach der Prüfung die sagittalen Abschnitts erfassen Zellen über das Profil des Lymphknotens, Verbrauchsteuern einen Kuchen-förmigen Keil aus den Lymphknoten aus der Mitte der äußeren Kapsel. Für einen Knoten 2 cm in der Breite (standard-Größe, siehe Tabelle 1), schneiden Sie ein Keil in die Mitte, die 4 mm in der äußeren Bogenlänge und 5 mm Dicke haben ein nasses Gewicht von ~ 100 mg.
- Mit dem Skalpell, den Keil klein geschnitten Sie, nicht dicker als 5 mm und ca. 50-100 mg jeden feuchten Tuch Gewicht.
Hinweis: aus Gründen der Kohärenz verarbeiten Sie alle Keil Stücke aus einer einzigen Lymphknoten entweder in einem Batch oder in separaten Röhren von RNA bündeln gefolgt um eine repräsentative RNA-Profil aus den Lymphknoten von Interesse bieten. - Legen Sie die Gewebe-Stücke mit der Pinzette in Röhren mit mindestens 10 Bänden der RNA Bewahrung Lösung im Vergleich zu der Menge des Gewebes.
-
Punch-Biopsie-Methode
- Wählen Sie eine Biopsie Stanzwerkzeug, wenn das Ziel ist es, bestimmte Bereiche, wie z. B. spezifische Follikel aus dem Knoten sammeln. Wählen Sie ein Werkzeug konsistent mit der Größe der Stichprobe für die Sammlung gewünscht. Biopsie Stanzwerkzeuge sind in einer Vielzahl von Größen, von 1 bis 8 mm Durchmesser erhältlich.
- Suchen Sie visuell der Regionen von Interesse zur Probe in den Lymphknoten.
- Verwenden Sie das Stanzwerkzeug Biopsie um zu Verbrauchsteuern die Abschnitte von Interesse, drücken und Drehen des Werkzeuges in das Gewebe um den Kern zu erstellen. Übertragen Sie das Stück Gewebe mit der Pinzette auf ein Rohr mit mindestens 10 Bänden der RNA Bewahrung Lösung. Wenn dicker als 5 mm, verwenden Sie Pinzette und Skalpell, das Gewebe weiter in kleinere Stücke teilen.
-
Keil-Schnitt Methode
- Sobald die Proben zur Bewahrung Lösung übertragen wurden (zB., RNALater), transportieren sie zurück ins Labor bei Raumtemperatur.
- Speichern Sie die Proben bei 4 ° C über Nacht, um Gewebe eindringen zu ermöglichen.
- Am nächsten Tag Gießen Sie die überschüssige Konservierungsmittel RNA-Lösung und speichern Sie die Gewebeproben bei-80 ° C. Entfernen Sie so viel Konservierungsmittel wie möglich vor dem Einfrieren der Proben. Verwenden Sie einen P1000 und/oder P200 Mikropipette Tipp für ein effizientes RNA Konservierungsmittel Lösung entfernen.
Achtung: Entsorgen Sie dekantiert Erhaltung-Lösung entsprechend, basierend auf den infektiösen Eigenschaften des Erregers sowie die chemischen und umweltbedingte Gefahren der Konservierungsmittel Lösung wie angegeben auf dem Sicherheitsdatenblatt genutzt.
Hinweis: Die hier bereitgestellten Richtlinien sind für die RNA-Erhaltung-Lösung in der Tabelle der Materialienbeschrieben bereitgestellt. Wenden Sie alternative Konservierung Lösungen mit den Richtlinien des Herstellers.
4. Verarbeitung von RNA Lymphknoten
Achtung: Tragen Sie einen Laborkittel, Handschuhe und richtige Augenschutz für die Bearbeitungsschritte.
Hinweis: Das Phenol basierende Reagenz verwendet hier die Tabelle der Materialien beschrieben ist (und das Protokoll basiert auf den Richtlinien des Herstellers)20. Die Verwendung von alternativen Phenol basierende Reagenzien erfordern eine Änderung des Verfahrens, basierend auf den Empfehlungen des Herstellers für das jeweilige Produkt erworben.
- Vor dem Zugriff auf die Proben, Pre-aliquoten gekühlt (4 ° C) Phenol basierende Reagenz zur Homogenisierung Röhren (1,5 mL/Rohr), mit Hilfe einer Pipette.
Achtung: Phenol ist giftig, ätzend, und eine Gefahr für die Gesundheit. Chloroform ist giftig und gesundheitsschädlich. Füllen Sie Verarbeitungsschritte 4.1-4.9 in einem chemischen Abzug, und tragen Sie Handschuhe, um den Kontakt mit den Chemikalien zu vermeiden. In den Vereinigten Staaten ist Chloroform als gefährlicher Abfall; Röhrchen mit gefährlicher Abfälle nach lokalen und institutionellen Richtlinien entsorgen. Prozess-Gewebeproben von mit Krankheitserregern unter den entsprechenden BSL-2, BSL-3 oder BSL-4 Leitlinien infizierten Tieren. - Sobald eine Probe mit einer sauberen Pinzette aus der Mikrozentrifugenröhrchen gelöst werden kann, sofort etwa 50-100 mg des erhaltenen Gewebes zum Übertragen der Homogenisierung Röhrchen mit Phenol-basierte Reagenz
- Das Auftauen vor der Zugabe zu Phenol basierende Reagenz zu begrenzen. Verwenden Sie Speicher auf Trockeneis, wenn mehrere Proben aus dem Gefrierfach zur Verarbeitung entfernt werden.
- Fest Röhrchen Sie der, legen Sie es auf den Homogenisator und homogenisieren Sie die Probe mit einem Rotor-Stator Gewebe Homogenisator in das Phenol basierende Reagenz zu. Füllen Sie die Verarbeitung bei Raumtemperatur. Wenn abgeschlossen haben, überprüfen Sie für ein trübes Aussehen um sicherzustellen, dass die Probe erfolgreich homogenisiert wurde; das Gewebe sollte in eine trübe Suspension verteilt werden.
Hinweis: Eine 2 min RNA-Extraktion-Einstellung wird verwendet (die voreingestellte RNA_02_1-Einstellung für den Homogenisator beschrieben in der Tabelle der Materialien; für gefrorenes Gewebe entwickelt). Der Rotor-Stator in diesem Protokoll angegeben (siehe Tabelle der Materialien) ist große gezahnt und angepasst für die Homogenisierung der unterschiedlichsten Gewebearten, einschließlich Fasermaterial. Die RNA_02_01-Einstellung richtet sich speziell an (im Gegensatz zu frisch) eingefroren Gewebe. Wie Lymphknoten Bindegewebe Komponenten haben, empfiehlt sich eine ähnlich optimierten Rotor-Stator-Homogenisator, eine effektive Dissoziation zu erreichen. Das National Institute of Environmental Health Sciences21 Weitere Informationen zur Homogenisierung Empfehlungen zu sehen.- Wenn große Teile des Gewebes nach der Homogenisierung bleiben, wiederholen Sie die Homogenisierung. Um effizient die RNA wiederherzustellen, muss das Gewebe gut getrennt werden.
- Verwenden Sie für mehr faserig Lymphknoten Stücke Pinzetten und Scheren Lymphknoten Stück in mehrere kleinere Stücke vor der Übertragung an die Homogenisierung Röhre Pre hacken. Wie in der Einleitung beschrieben, möglicherweise Lymphknoten Gewebe von älteren Tieren mehr faserig.
Hinweis: Wenn eine dual Analyse der Wirt und Erreger RNA gewünscht wird, kann zusätzliche Behandlung (z. B. Wulst Homogenisierung) notwendig sein für die Erholung der bakteriellen RNA besonders wenn Gram-positive Erreger im Gewebe vorhanden sind.
- Sofort entfernen der homogenisierten Probe aus den Rohren und überträgt es auf RNase-freie Mikrozentrifugenröhrchen (2,0 mL Größe) mit einer Mikropipette P1000.
- Zentrifugieren Sie die Probe für 5 min bei 12.000 x g bei 4 ° C, jede Probe Ablagerungen zu entfernen. Mit einer P1000 Mikropipette Spitze, übertragen des Überstands zu einem frischen Microcentrifuge Schlauch, das Pellet hinterlässt.
- Inkubieren Sie den überstand für 5 min bei Raumtemperatur. Jede Probe zwischen zwei 1.5 Mikrozentrifugenröhrchen aufgeteilt.
- Fügen Sie 0,2 mL Chloroform pro 1 mL des Phenol-basierte Reagenz (d.h. 0,3 mL für eine 1,5 mL Probe); invertiere es eigenhändig für 1-2 min, mischen Sie die Phasen, aber tun nicht Wirbel der Probenmaterials. 2 min bei Raumtemperatur inkubieren.
- Zentrifugieren Sie es für 15 min bei 12.000 x g bei 4 ° C.
- Entfernen Sie vorsichtig die obere, klaren wässrige Phase mit einer Mikropipette Spitze (P1000 oder P200) bilden die obere Schicht wird ohne Unterbrechung der Interphase oder die rote Phenol-Chloroform-Schicht. Auf einen neuen Microcentrifuge Schlauch übertragen.
- Mischen Sie es mit einem gleichen Volumen von 100 % Ethanol; Mischen Sie gründlich, durch das Rohr 4 - 5 mal umdrehen. Die Röhre erscheint oft bewölkt.
- Übertragen Sie mit einer Mikropipette Spitze die Flüssigkeit auf eine Silica-basierten kommerziellen Spin Spalte weiter zu reinigen und eluieren RNA, nach dem Hersteller Anweisungen22. Eluieren generierte aus RNA ~ 50 mg des Gewebes in 50-100 µl RNase-freies Wasser.
Hinweis: Während die Gewinnung von Phenol-basierte Reagenz DNA in die organische Phase für die nachgeschaltete Verwendung von RNA in qRT-PCR und/oder RNA-Sequenzierung Anwendungen entfernt empfiehlt sich eine zusätzliche Behandlung der RNA-Proben mit DNase. - Aliquot der eluierten RNA, Schläuche für den Einsatz in eine Quantifizierung und Qualität Bewertung zu jedem Einfrieren Auftauen der wichtigsten RNA-Probe zu trennen. Übertragen Sie die Rohre bis-80 ° C für die Lagerung.
- Bestätigen Sie bei Lymphknoten Proben mit Zoonoseerregern, vor allem bei der BSL-3 Aufhaltestufe infizierten Tieren abgeleitet resultierende RNS-Probe für die Abwesenheit von Krankheitserregern, wenn die Probe zu einem niedrigeren Biosafety Level Containment Bereich verschoben werden für die Qualitätsanalyse, RT-PCR und/oder RNA-Sequenzierung Bibliothek Vorbereitung.
Hinweis: Es ist entscheidend für die örtliche Vorschriften zu prüfen und im Falle der CDC (Centers for Disease Control and Prevention) Inaktivierung Leitlinien für die Arbeit mit ausgewählten Agenten, es ist notwendig, alle Inaktivierung Verfahren am lokalen Standort des Forschers zu validieren.- 1/10th Volume jeder eluierten RNA-Probe aus Schritt 4.12 (d.h. 5 µl von 50 µl der Gesamt-RNS) entfernen. Mit einer sterilen Technik, mischen Sie die Probe mit 100 µl Bakterienwachstum Brühe in einen sterilen 1,5 mL Microcentrifuge Schlauch. Die RNA-Brühe-Gemisch verteilt mit einem sterilen Kunststoff Spreader sind die Oberfläche einer nährbodenplatte.
Hinweis: Wählen Sie eine Brühe Art und Agar Platte konsistent mit dem Wachstum des Erregers mit denen die Tiere in Frage gestellt wurden. - Inkubieren Sie die Agarplatte unter Bedingungen, die spezifisch für das Wachstum des Erregers mit denen die Tiere in Frage gestellt wurden. Überprüfen Sie für das Fehlen einer wiederhergestellten Bakterien vor dem Entfernen der Proben von BSL-3 Containment.
Hinweis: Die resultierende eluierten RNA ist nach Quantifizierung und Tests von Integrität, geeignet für downstream-Anwendungen, einschließlich einer RNA-Sequenzierung und RT-PCR-Analyse.
- 1/10th Volume jeder eluierten RNA-Probe aus Schritt 4.12 (d.h. 5 µl von 50 µl der Gesamt-RNS) entfernen. Mit einer sterilen Technik, mischen Sie die Probe mit 100 µl Bakterienwachstum Brühe in einen sterilen 1,5 mL Microcentrifuge Schlauch. Die RNA-Brühe-Gemisch verteilt mit einem sterilen Kunststoff Spreader sind die Oberfläche einer nährbodenplatte.
- Beurteilen Sie die RNA-Erholung und Reinheit mit einem Spektralphotometer. Zur Beurteilung der RNA-Qualität laden Sie 1-2 µl der eluierten RNA-Probe zu Spektralphotometer für eine Analyse. Nehmen Sie ein Spektrum der Probe im ultravioletten (UV) Bereich.
- Aus dem UV-Spektrum aufzunehmen, A260/a280 Verhältnis, A260/a230 Verhältnis und den A-260 -Wert für die Probe (A = Absorption).
- Da einsträngige RNA in einer Konzentration von 40 µg/mL eine Extinktion von 1,0 bei 260 hat nm, die RNA-Konzentration (µg/mL) multipliziert die A260 40 für die gereinigten Proben zu berechnen.
- Als schnelle Methode zur Beurteilung der Widerstandsfähigkeit gegen RNA degradierten Proben von der weiteren Analyse ausgeschlossen, mischen Sie 2 µl jeder RNA-Probe mit 4 µl 1,5 X formamide Belastung Farbstoff [95 % entionisiertem Formamid, 0,025 % (w/V) Bromophenol blue, 0,025 % (w/V) Xylol Cyanol, 5 mM EDTA (pH 8.0), 0,025 % (w/V) SDS] in 1,5 mL Röhrchen Microcentrifuge23,24.
- Erhitzen Sie die Rohre bei 70 ° C für 1 min und übertragen Sie dann die Röhren für 1 min Eis.
- Laden Sie die Proben zu einem 1 % Agarose-Gel vorbereitet in 1 X TBE (für eine vollständige Beschreibung der eine native Agarose-Gelelektrophorese für RNA, siehe Rio, Ares Jr., Hannon und Nilsen)24. Trennen der Proben durch standard-Gel-Elektrophorese Methoden und bestätigen Sie die Anwesenheit von 28 s und 18 s ribosomalen RNA-Bands.
- Follow-up der Gelelektrophorese mit Quantitative Analysemethoden für die RNA-Integrität-Bestimmung [z.B., einem Bioanalyzer und RIN (RNA-Integrität-Nummer) Analyse] wie beschrieben in die Ergebnisse repräsentativ und Müller, O. Et Al. 25 und Schröder, A. Et Al. 26.
5. alternative Extraktionsmethode von Formalin fixiert, Paraffin-eingebetteten (FFPE) Gewebe
Hinweis: Obwohl FFPE Gewebe Erhaltung nicht die stabilste Methode der Nukleinsäure-Konservierung darstellen, das Protokoll dargestellt ist eine Möglichkeit, einige transcriptional Änderungen zu studieren, wenn andere erhaltene Gewebe nicht verfügbar sind.
- Bereiten Sie Formalin Reagenzien für den Erhalt von Gewebe durch den Verzicht auf Formalin in Kunststoffbehältern. Gewebe sollte bzw. in Formalin mit einem Verhältnis von 20:1 Formalin: Gewebe Volumen/Gewicht, beibehalten werden.
Achtung: Formalin ist ein bekanntes menschliches karzinogen und obwohl nicht akut toxisch, chronischer Exposition (in der Regel durch eingeatmete Dämpfe) kann eine erhebliche Gesundheitsgefahr darstellen. Ausreichende Belüftung und PPE (d.h. die Verwendung von Nitril-Handschuhe, innerhalb von 15 Minuten nach der ersten Exposition geändert werden) sind erforderlich, um die gesundheitlichen Risiken zu minimieren. Beziehen sich auf die OSHA Formaldehyd Standard (29 CFR 1910.1048) für weitere Informationen zur Belichtung Überwachung und Risikobewertung.
Hinweis: Mit zu wenig Formalin kann die Fähigkeit des Gewebes, vollständig erhalten werden, wenn in Konservierungsmittel untergetaucht auswirken. - Nach der Isolierung des Gewebes von Interesse sorgfältig Trimmen des Gewebes auf weniger als 5 mm Dicke.
- Tauchen Sie mit Pinzette, sorgfältig getrimmte Gewebe in Formalin, Spritzer zu minimieren.
Hinweis: Das Gewebe sollte vollständig in Formalin, um eine komplette Fixierung ermöglichen getaucht werden. Es ist wichtig, dass alle Flächen des Gewebes mit Formalin vollständig bedeckt sind.
- Tauchen Sie mit Pinzette, sorgfältig getrimmte Gewebe in Formalin, Spritzer zu minimieren.
- Sobald das Gewebe in Formalin getaucht wurde, ermöglichen Sie die Gewebe Fixierung für mindestens 24 Stunden bei Raumtemperatur erfolgen.
Hinweis: Formalin Gewebe dringt langsam, mit einer Rate von 1 mm pro Stunde von allen Oberflächen27,28. Daher halten die Dicke der Gewebeschnitte auf weniger als 5 mm Ergebnisse in eine schnelle Fixierung des gesamten Gewebes. - Übertragen Sie nach der Fixierung der Gewebe auf eine entsprechende Reagenz für die Einbettung von Paraffin.
Hinweis: zum Beispiel waren das Gewebe untersucht in dieser Handschrift zu 70 % Ethanol vor der Einbettung übertragen. - Des Gewebes in Paraffin einbetten. 29
- Abschnitt des Gewebes bis 10-80 µm Dicke (Einsatz 10-20 µm MiRNA ist auf Wunsch extrahiert werden)29.
- Mit Skalpell und Pinzette, entfernen Sie eine 40 mg Gewebe Stück von jeder Probe verarbeitet werden und legen Sie sie in ein steriles, Nuklease-free Microfuge Rohr.
- Nutzen Sie eine kommerzielle Set für 4.wenn und Nukleinsäure-Recovery von Proteinkinase Gewebeproben RNA aus den erhaltenen Proben zu extrahieren.
Hinweis: Das Kit verwiesen in der Tabelle der Materialien nutzt Xylol und Ethanol wäscht entfernen Paraffin, einem Behandlungsschritt Protease und Glasfaser Filter, RNA zu reinigen.
Ergebnisse
Die Nutzung der in diesem Artikel (Schritte 1 bis 4 des Protokolls) vorgestellten Überlegungen helfen bei der Beitreibung von RNA aus großen Tier Proben, die für eine nachgeschaltete Analyse in Host genexpressionsstudien geeignet ist. Die RNA-Qualität für downstream-Anwendungen wird durch mehrere standard-Maßnahmen beurteilt. Spektrophotometrie A260/a280 Verhältnis liefert ein Maß für die Protein-Verschmutzung und A260/a230 Verhältnis stellt ein weiteres Mittel zur Re...
Diskussion
Die Mehrheit der transkriptomischen Studien und die dazugehörigen Protokolle auf Maus, Ratte oder Post-Mortem Humanproben konzentrieren. Jedoch bieten Untersuchungen in Vieh und Wildtiere eine breite Palette von Möglichkeiten für die Charakterisierung der Immunantwort gegen Krankheiten, sowohl als für Veterinärmedizin und in Bezug auf zoonotische Krankheiten, für die menschliche Gesundheit. Dieses Protokoll zur Verfügung gestellt, einer Übersicht der wichtigsten Überlegungen für hoher Integrität RNA-Extraktion...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenlegen. Alle Forschung wird mit intramurale Mittel aus dem US Department of Agriculture, Agricultural Research Service finanziert. Alle Verweise auf bestimmte Produkte werden für die Zwecke der experimentellen Reproduzierbarkeit zur Verfügung gestellt und stellen keine Billigung dieser Produkte durch die Bundesregierung.
Danksagungen
Die Autoren möchten James Fosse für seine hervorragende Arbeit auf allen Videografie und Videoverarbeitung danken; Michael Marti für seine hervorragende Arbeit bei der Erzeugung von digitalisierten Rinder Bilder; Lilia Walther für ihre Hilfe mit RNA-Extraktion und Bioanalyzer läuft; Mitch Palmer und Carly Kanipe für ihre hilfreiche Bewertung und Feedback auf Lymphknoten Bilder; und die Tierpflege und Tierärzten bei der National Animal Disease Center für all ihre harte Arbeit und Unterstützung bei der Tierhaltung und der Vorbereitung auf Autopsien.
Materialien
| Name | Company | Catalog Number | Comments |
| RNA preservation solution (we used RNALater for all experiments) | ThermoFisher | AM7020 | |
| 1.5 ml or 2 ml polypropylene microcentrifuge tubes | Fisher Scientific | 05-408-129 | |
| Disposable scalpels | Daigger Scientific | EF7281 | |
| Tissue forceps, rat tooth | Fisher Scientific | 12-460-117 | Other tissue forceps available including curved tip, tapered edge, etc. , depends on user preference |
| 3 mm punch biopsy needles | Fisher Scientific | NC9949469 | |
| Sharps container (small and transportable for necropsy) | Stericycle | 8900SA | 1 qt. size shown here |
| Cutting boards or disposable trays | Fisher Scientific | 09-002-24A | Available in a variety of sizes, depends on user preference |
| Personal protective equipment | Varies with pathogen (gloves, respirator masks, goggles, etc.) | ||
| Phenol-based RNA extraction reagent (we used TRIzol Reagent for all experiments) | ThermoFisher | 15596026 | |
| Silica column-based RNA extraction kit (we used the PureLink RNA Mini kit for all experiments) | ThermoFisher | 12183018A | Designed for up to 100 mg tissue |
| 100% Ethanol (200 proof for molecular biology) | Sigma-Aldrich | E7023 | |
| Tissue homogenizer with enclosed homogenization tubes (we used the gentleMACS dissociator for all experiments) | Miltenyi Biotec | 130-093-235 | |
| Agarose (General, for gel electrophoresis) | Sigma-Aldrich | A9539 | |
| 1X TBE | Fisher Scientific | BP24301 | Can also make from scratch in the laboratory |
| Deionized formamide | EMD Millipore | S4117 | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Bromophenol blue | Sigma-Aldrich | 114391 | |
| Xylene cyanol | Sigma-Aldrich | X4126 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | EDS | |
| UV-Vis Spectrophotometer (we used the NanoDrop Spectrophotometer) | ThermoFisher | ND-2000 | |
| Device for quantitative RNA assessment (we used the Bioanalyzer, with associated components and protocols) | Agilent | G2939BA | |
| FFPE RNA extraction kit (we used the RecoverAll Total Nucleic Acid Isolation Kit for Formalin Fixed, Paraffin Embedded Tissue) | ThermoFisher | AM1975 | |
| Plastic spreader (L-shaped spreader) | Fisher Scientific | 14-665-231 | Only needed for sterility testing for samples from infected animals |
| Necropsy knives | Livestock Concepts | WI-0009209 |
Referenzen
- Tizioto, P. C., et al. Immunological response to single pathogen challenge with agents of the bovine respiratory disease complex: an RNA-sequence analysis of the bronchial lymph node transcriptome. PLoS One. 10 (6), e0131459 (2015).
- Miller, L. C., et al. Analysis of the swine tracheobronchial lymph node transcriptomic response to infection with a Chinese highly pathogenic strain of porcine reproductive and respiratory syndrome virus. BMC Veterinary Research. 8, 208 (2012).
- Silva, T. M. A., Costa, E. A., Paixao, T. A., Tsolis, R. M., Santos, R. L. Laboratory animal models for brucellosis research. Journal of Biomedicine and Biotechnology. 2011, 518323 (2011).
- Willard-Mack, C. L. Normal structure, function, and histology of the lymph nodes. Toxicologic Pathology. 34, 409-424 (2006).
- Elmore, S. A. Histopathology of the lymph nodes. Toxicologic Pathology. 34 (5), 425-454 (2006).
- Luscieti, P., Hubschmid, T. h., Cottier, H., Hess, M. W., Sobin, L. H. Human lymph node morphology as a function of age and site. Journal of Clinical Pathology. 33 (5), 454-461 (1980).
- Hadamitsky, C., et al. Age-dependent histoarchitectural changes in human lymph nodes: an underestimated process with clinical relevance?. Journal of Anatomy. 216 (5), 556-562 (2010).
- Sisson, S., Grossman, J. D., Getty, R. . Sisson and Grossman’s The Anatomy of Domestic Animals, Volume 1. , (1975).
- Olsen, S. C., Johnson, C. Comparison of abortion and infection after experimental challenge of pregnant bison and cattle with Brucella abortus strain 2308. Clinical and Vaccine Immunology. 18 (12), 2075-2078 (2011).
- Brichta-Harhay, D. M., et al. Microbiological analysis of bovine lymph nodes for the detection of Salmonella enterica. Journal of Food Protection. 75 (5), 854-858 (2012).
- Samuel, J. L., O’Boyle, D. A., Mathers, W. J., Frost, A. J. Isolation of Salmonella from mesenteric lymph nodes of healthy cattle at slaughter. Research in Veterinary Science. 28 (2), 238-241 (1980).
- Arthur, T. M., et al. Prevalence and characterization of Salmonella in bovine lymph nodes potentially destined for use in ground beef. Journal of Food Protection. 71 (8), 1685-1688 (2008).
- Cray, W. C., Moon, H. W. Experimental infection of calves and adult cattle with Escherichia coli O157:H7. Applied and Environmental Microbiology. 61 (4), 1586-1590 (1995).
- Thiermann, A. B. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. American Journal of Veterinary Research. 43 (5), 780-784 (1982).
- Palmer, M. V., Waters, W. R., Whipple, D. L. Investigation of the transmission of Mycobacterium bovis from deer to cattle through indirect contact. American Journal of Veterinary Research. 65 (11), 1483-1489 (2004).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA, and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-537 (1993).
- Chomczynski, P., Sacchi, N. Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Vogelstein, B., Gillespie, D. Preparative and analytical purification of DNA from agarose. Proceedings of the National Academy of Sciences of the United States of America. 76 (2), 615-619 (1979).
- . . AVMA Guidelines for the Euthanasia of Animals. , (2013).
- . . TRIzol Reagent Manual. , (2016).
- . . Disruption and Homogenization of Tissue for the Extraction of RNA. , (2014).
- . . PureLink RNA Mini Kit Protocol . , (2012).
- . . RNA Gel-loading Buffer. , (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing Agarose Gel Electrophoresis of RNA. Cold Spring Harbor Protocols. , (2010).
- Mueller, O., et al. A microfluidic system for high-speed reproducible DNA sizing and quantitation. Electrophoresis. 21 (1), 128-134 (2000).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (3), (2006).
- Suvarna, K. S., Layton, C., Bancroft, J. D. . Bancroft’s Theory and Practice of Histological Techniques. , (2012).
- Medawar, P. B. The rate of penetration of fixatives. Journal of Microscopy. 61 (1-2), 46-57 (1941).
- Canene-Adams, K. Preparation of formalin-fixed paraffin-embedded tissue for immunohistochemistry. Methods in Enzymology. 533, 225-233 (2013).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27, 126-139 (2006).
- Fajardy, I., et al. Time course analysis of RNA stability in human placenta. BMC Molecular Biology. 10 (21), (2009).
- Bahar, B., et al. Long-term stability of RNA in post-mortem bovine skeletal muscle, liver and subcutaneous adipose tissues. BMC Molecular Biology. 8, 108 (2007).
- Yamagishi, A., et al. Gene profiling and bioinformatics analyses reveal time course differential gene expression in surgically resected colorectal tissues. Oncology Reports. 31 (4), 1532-1538 (2014).
- Choi, S., Ray, H. E., Lai, S. H., Alwood, J. S., Globus, R. K. Preservation of multiple mammalian tissues to maximize science return from ground based and spaceflight experiments. PLoS One. 11 (12), e0167391 (2016).
- Almeida, A., Thiery, J. P., Magdelenat, H., Radvanyi, F. Gene expression analysis by real-time reverse transcription polymerase chain reaction: influence of tissue handling. Analytical Biochemistry. 328 (2), 101-108 (2004).
- Lee, S. M. L., Schelcher, C., Thasler, R., Schiergens, T. S., Thasler, W. E. Pre-analytical determination of the effect of extended warm or cold ischaemia on RNA stability in the human ileum mucosa. PLoS One. 10 (9), e0138214 (2015).
- Kap, M., et al. The influence of tissue procurement procedures on RNA integrity, gene expression, and morphology in porcine and human liver tissue. Biopreservation and Biobanking. 13 (3), 200-206 (2015).
- Hong, S. H., et al. Effects of delay in the snap freezing of colorectal cancer tissues on the quality of DNA and RNA. Journal of the Korean Society of Coloproctology. 26 (5), 316-325 (2010).
- Lee, S. M., et al. RNA stability in human liver: comparison of different processing times, temperatures and methods. Molecular Biotechnology. 53 (1), 1-8 (2013).
- Morrison, P. K., et al. Post-mortem stability of RNA in skeletal muscle and adipose tissue and the tissue-specific expression of myostatin, perilipin and associated factors in the horse. PLoS One. 9 (6), e100810 (2014).
- Seear, P. J., Sweeney, G. E. Stability of RNA isolated from post-mortem tissues of Atlantic salmon (Salmo salar L.). Fish Physiology and Biochemistry. 34 (1), 19-24 (2008).
- Marchuk, L., Sciore, P., Reno, C., Frank, C. B., Hart, D. A. Postmortem stability of total RNA isolated from rabbit ligament, tendon, and cartilage. Biochimica et Biophysica Acta. 1379 (2), 171-177 (1998).
- Inoue, H., Kimura, A., Tuji, T. Degradation profile of mRNA in a dead rat body: basic semi-quantification study. Forensic Science International. 130 (2-3), 127-132 (2002).
- Micke, P., et al. Biobanking of fresh frozen tissue: RNA is stable in nonfixed surgical specimens. Laboratory Investigation. 86 (2), 202-211 (2006).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- Ahmed, A. M., et al. Variation in the ovine abomasal lymph node transcriptome between breeds known to differ in resistance to the gastrointestinal nematode. PLoS One. 10 (5), e0124823 (2015).
- Russell, G. C., et al. Host gene expression changes in cattle infected with Alcelaphine herpesvirus 1. Virus Research. 169 (1), 246-254 (2012).
- Avraham, R., et al. A highly multiplexed and sensitive RNA-seq protocol for simultaneous analysis of host and pathogen transcriptomes. Nature Protocols. 11, 1477-1491 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten