Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Titration ELISA als eine Methode zur Bestimmung der Dissoziationskonstante Rezeptor Liganden Interaktion
In diesem Artikel
Zusammenfassung
Ein detailliertes Protokoll, eine Titrierung ELISA durchzuführen wird beschrieben. Darüber hinaus wird ein neuartiger Algorithmus, Titration ELISAs zu bewerten und zu einer Dissoziationskonstante Bindung eines lösliche Liganden an einen Mikrotiter Platte immobilisiert Rezeptor präsentiert.
Zusammenfassung
Die Dissoziationskonstante beschreibt die Wechselwirkung zwischen zwei Partnern in das verbindliche Gleichgewicht und ist ein Maß für die Affinität. Es ist ein entscheidender Parameter verschiedener Liganden, z.B., wettbewerbsfähige Inhibitoren, Protein-Isoformen und Mutanten, für ihre Bindungsstärke an einen Bindungspartner zu vergleichen. Dissoziation-Konstanten werden durch Konzentrationen von gebundenen gegenüber freien Liganden als verbindliche Kurven Plotten bestimmt. Im Gegensatz dazu sind Titrierung Kurven, in denen ein Signal, das proportional zur Konzentration des gebundenen Liganden ist gegen die Gesamtkonzentration an zusätzlichen Liganden geplottet wird leichter zu erfassen. Das Signal kann spektroskopisch und durch Enzym-linked Immunosorbentprobe Assay (ELISA) erkannt werden. Dies wird in einem Protokoll für eine Titration ELISA illustriert, die die Bindung von der Schlange Venom-abgeleitete Rhodocetin in seiner immobilisierten Zieldomäne Integrin α2β1 misst. Titration ELISAs sind vielfältig und weit verbreitet. Jedes Paar von interagierenden Proteine können als immobilisierte Rezeptor und lösliche Liganden verwendet werden, vorausgesetzt, dass beide Proteine rein sind, und deren Konzentrationen sind bekannt. Die Schwierigkeit war bisher die Dissoziationskonstante aus einer Titration-Kurve zu bestimmen. In dieser Studie wird eine mathematische Funktion, die zugrunde liegenden Titrierung Kurven eingeführt. Ohne keine fehleranfällige grafische Schätzung einer Sättigung Rendite ermöglicht dieser Algorithmus Verarbeitung der Rohdaten (Signalintensitäten bei verschiedenen Konzentrationen von zusätzlichen Liganden) direkt durch mathematische Auswertung über eine nichtlineare Regression. So mehrere Titrierung Kurven können gleichzeitig aufgenommen und umgewandelt in eine Reihe von charakteristischen Parameter, darunter die Dissoziationskonstante und die Konzentration der Bindung-aktiv-Rezeptor, und sie können statistisch ausgewertet werden. In Kombination mit diesem Algorithmus Titration ELISAs profitieren die Dissoziationskonstante direkt zu präsentieren. Daher können sie in Zukunft effizienter genutzt werden.
Einleitung
Die Dissoziationskonstante K ist ein wichtiger Parameter, die Affinität eines Rezeptors (R) für seine Liganden (L) zu beschreiben. Basierend auf dem Gesetz der Masse, wird K für das Gleichgewicht definiert in denen die Rezeptor-Ligand-komplexen RL in den R-Rezeptor und Ligand L: distanziert sich
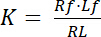 Gleichung 1
Gleichung 1
mit den Indizes f , die frei/ungebundenen Zustand von Rezeptor und Ligand. Die Konzentration der Rezeptor-Ligand-Komplex, RL, ist identisch mit der Konzentration der Rezeptor-gebundenen Liganden Lb. Da die Gesamtkonzentration an Rezeptor Rt die Summe der freien Rezeptor Rf und Liganden gebundenes Rezeptor Rb ist = Lb, die Dissoziationskonstante kann auch als geschrieben werden:
 Gleichung 2
Gleichung 2
Daher die Sättigung Ertrag Y, definiert als der Anteil der gebundenen Liganden Lb im Verhältnis der Konzentration des Rezeptors Rt,
 Gleichung 3
Gleichung 3
hängt von der Konzentration der freien Liganden Lf:
 Gleichung 4
Gleichung 4
Diese hyperbolische Beziehung beschreibt die verbindliche Kurve ein Rezeptor-Ligand-Wechselwirkungen und die Handlung zeigt die Konzentration der gebundenen Liganden Lb als eine Funktion der Konzentration der freien Liganden Lf. Die verbindliche Kurve kann die Dissoziationskonstante K als die Konzentration des freien Liganden mit Hälfte-maximale Sättigung Rendite ableiten. Darüber hinaus wurden verschiedene Algorithmen zur Bindung Kurven zu linearisieren, wie die doppelt-reziproke Handlung von Klotz1,2, oder Transformationen nach Scatchard oder Hanes (rezensiert von Bisswanger3) eingerichtet. Alle Algorithmen leiden jedoch das Problem, das der maximale Wert der Sättigung-Ausbeute, die asymptotisch bei hohen Konzentrationen an freien Liganden in der Bindung Kurve nähert, hat in eine grafische Vorauswertung geschätzt werden und ist daher fehleranfällig.
Darüber hinaus erfordert die Bestimmung einer verbindlichen Kurve die Quantifizierung der freien und gebundenen Liganden während der Bindung Gleichgewicht. Zu diesem Zweck hat die freie Liganden von Rezeptor-gebundenen Liganden getrennt und quantifiziert werden. Daher müssen die Ligand und Rezeptor unterscheiden sich in ihren Eigenschaften, wie ein nicht-Protein-Ligand im Gegensatz zu einem Protein-Rezeptor. Wenn beide Bindungspartner Proteine sind, haben sie in ihrer Größe, Gebühren oder andere molekulare Eigenschaften unterscheiden. Dennoch ist die Quantifizierung der Liganden-Konzentrationen im kleinen verbindliche Ansätze eine schwierige Aufgabe. Radioaktive Markierung des Liganden ist oft notwendig sein, die geringe Konzentration von gebundenen Liganden zu erkennen gewesen, vor allem dann, wenn erhebliche Mengen an Rezeptoren nicht verfügbar oder erschwinglich waren. Darüber hinaus kann die Rezeptor-gebundenen Liganden während und nach der Isolierung in gewissem Sinne nicht zu vernachlässigenden distanzieren. Daher müssen komplexe Methoden, z. B. Gleichgewicht Gel Filtration4Kapillarelektrophorese5und Puls Proteolyse6, Rezeptor-gebundenen Liganden zu quantifizieren und trennen Sie ihn vom freien Liganden.
Im Gegensatz zu diesen verbindlichen Assays erfordern Titration Experimente nicht die quantitative Trennung von freien und gebundenen Liganden. Zu diesem Zweck ist ein Rezeptor auf eine konstante Konzentration mit verschiedenen Konzentrationen von zusätzlichen Liganden titriert. Durch die Bindung an den Rezeptor hat der gebundene Liganden eine biophysikalische Eigenschaft unterscheidet sie von den kostenlosen Liganden und ist messbar, z.B., Photometrie, Fluorometry oder Antikörpernachweis. So ein Signal S, die proportional zur Sättigung ist Ausbeute Y und folglich auch auf die Konzentration der Rezeptor-gebundenen Liganden (Lb) wird erkannt als Funktion der Gesamtkonzentration an zusätzlichen Liganden (Lt). Beide Parameter, das Signal S und die Gesamtkonzentration an zusätzlichen Liganden sind in gewissem Sinne direkter und einfacher als die Konzentrationen der freien und gebundenen Liganden quantifiziert. Vor allem, die Erkennung von Rezeptor-gebundenen Liganden durch Enzym-linked Immunosorbentprobe Assay (ELISA) erlaubt die Reduktion von unter 100 µL Probenvolumen sowie parallele Messungen von mehreren Liganden-Konzentrationen in Multi-gut Mikrotiter-Platten. In eine Titrierung ELISA ist ein Rezeptor physisch zu einer Mikrotiterplatte in der gleichen Konzentration adsorbiert und titriert mit lösliche Liganden. Der Rezeptor wird im Wesentlichen durch hydrophobe Adsorption auf der Kunststoffoberfläche immobilisiert. Die Oberflächenkonzentration der immobilisierten Rezeptor korreliert mit der Beschichtung Konzentration des Rezeptors in eine nicht-lineare Beziehung, wahrscheinlich nach Langmuir´s Adsorption Isotherm7. Neben der Gesamtzahl der adsorbierten Rezeptormoleküle ist ihren Aktivitätszustand ein weiterer wichtiger Parameter für die Titration Assays. Nur immobilisiert Rezeptoren, die Liganden Bindung Aktivität bewahrt haben, sind relevant für die Titration-Assay und schließlich dazu beitragen, die Gesamtkonzentration der aktiven Rezeptoren Rt die Titration-Assays, die nicht direkt ermittelt werden kann.
Websites auf der Kunststoffoberfläche, die nicht durch die immobilisierten Rezeptor fallen sind anfällig für andere Proteine, wie z. B. die Liganden adsorbieren. Physikalische Adsorption des Liganden zu solchen Kunststoff Oberfläche Seiten würde in ein ähnliches Signal wie der Rezeptor-gebundenen Liganden und dennoch in einer unspezifischen Weise führen. Dieses unspezifische Signal Kunststoff Oberfläche Stätten der Mikrotiter-Platten zu reduzieren, die haben nicht mit Protein beschichtet wurden noch mit Rinderserumalbumin (BSA) blockiert werden. Jedoch können für einige Rezeptor-Ligand-Titration-Assays, unspezifischen Hintergrund Signale beobachtet werden. Dann, andere Blockern, wie eine Lösung von 0,2 % Gelatine oder von 0,04 % Tween 20, werden empfohlen.
Nach der Bindung an den Rezeptor, wird die freie Liganden durch zwei Waschschritte entfernt. Gebundenen Liganden bleibt mit dem Rezeptor, die auf die Kunststoffoberfläche des Brunnens Mikrotiter immobilisiert, und optional durch chemische Fixierung verstärkt. Für die anschließende kovalente Vernetzung von gebundenen Liganden und immobilisierten Rezeptor mit Glutaraldehyd ist die Puffer Substanz TRIS HEPES, ohne Änderung der Liganden Bindung ersetzt. HEPES, im Gegensatz zu TRIS, nicht Glutaraldehyd inaktivieren. Die kovalente Vernetzung mit Glutaraldehyd behebt die gebundenen Liganden mit seinen Rezeptor und verhindert seine Dissoziation bei Wasch- und Inkubation Folgeschritte. So die Rezeptor-Ligand-Wechselwirkungen ist chemisch fixiert und gewährleistet eine Titration-Kurve, die von nachfolgenden Schritte des Waschens und Inkubation nicht betroffen ist. Glutaraldehyd Fixierung kann jedoch chemisch ändern, der Ligand und Rezeptor derart, dass ihre Interaktion reduziert oder abgeschafft ist. Modifikation von Epitopen innerhalb der Liganden kann darüber hinaus der Bindungsaffinität der Nachweis von Antikörper ändern, vor allem, wenn ein monoklonaler Antikörper verwendet wird, um gebundene Liganden zu quantifizieren. Obwohl keiner von diesen Nebenwirkungen von Glutaraldehyd Fixierung in dieser Titration ELISA auftritt, muss die Empfindlichkeit des Tests in Richtung Glutaraldehyd bei jeder Rezeptor-Ligand Interaktion vor der Titration Experiment getestet werden. Nach der Fixierung wird überschüssige Glutaraldehyd in drei Waschschritten mit TRIS-haltigen Puffer entfernt. TRIS inaktiviert verbleibenden Aldehyd-Gruppen, die nonspecifically mit Nachweis von Antikörpern in der Folgeschritt reagieren könnte.
Die Menge des gebundenen Liganden ist mit Enzym-linked Antikörper, quantifiziert die bieten eine photometrische Signal ELISA S. Dies ist im Vergleich zu den gesamten Liganden Konzentration Lt hinzugefügt geplottet in jede Vertiefung. Trotz seiner einfacher Erwerb ist die Titration Kurve keine hyperbolische Funktion im Gegensatz zu der Bindung-Kurve. Darüber hinaus war es unklar wie die Dissoziationskonstante K aus einer Titration Kurve zu berechnen. Obwohl Algorithmen zu linearisieren spektroskopisch erworbenen Titrierung Kurven unabhängig von Stockell8 und Heyn und Weischet9gemeldet wurden, fielen sie wegen ihrer Unsicherheit der Schätzung der maximalen Signals kurz schätzen, dass die Sättigung Ertrag nähert sich bei hohen Konzentrationen an zusätzlichen Liganden.
Hier sind eine Titrierung ELISA und eine nicht-lineare Regression-Algorithmus beschrieben, die Dissoziationskonstante K für eine Rezeptor-Ligand Interaktion aus einer Titration Kurve abzuleiten. Dieses Protokoll ist für das Zusammenspiel von Kollagen-Bindung A-Domäne der Integrin α2β1 mit einer Schlange Venom abgeleitet Inhibitor beispielhaft dargestellt. Integrine sind Zelle Adhäsionsmoleküle, die die Verankerung der Zellen der umgebenden extrazellulären Matrix oder die zugrunde liegenden Basalmembran10,11zu vermitteln. Darüber hinaus vermitteln Integrine wichtige Signale zwischen Zellen und der extrazellulären Matrix durch die Rekrutierung von zusätzlichen Signalmoleküle und bilden neue Zellorganellen, Adhesomes, bei der Zellmatrix Interaktion12,13, 14. Kollagen, dem Liganden α2β1 Integrin, ist das am häufigsten vorkommende Protein des menschlichen Körpers und ist ein entscheidender Gerüstbau Bestandteil des Bindegewebe15. Die Interaktion zwischen α2β1 Integrin und Kollagen wird durch A-Domäne der Integrin α2 Untereinheit vermittelt. Die Integrin α2A-Domäne enthält einen zweiwertigen kationen, die für Kollagen Bindung erforderlich ist und seine Struktur stabilisiert. Der Wildtyp Form sowie Mutanten des Geschäftsfeldes α2A, wie dem, in dem die Oberfläche ausgesetzt Rückstände Y216 für ein Glycin ersetzt worden können leicht in eine bakterielle Expressionssystems rekombinant hergestellt und über ihre Oligo-His-Tags mit einem NiNTA isoliert Superflow Säule mit einer anschließenden Dialyse gegen TRIS gepufferte Kochsalzlösung (TBS; 50 mM TRIS/HCl, pH 7,4, 150 mM NaCl) mit 2 mM MgCl216. Deren Konzentrationen wurden mit dem Bicinchoninic Säure Assay (BCA) ermittelt und ihre Reinheiten von herkömmlichen SDS-PAGE getestet und mit Coomassie Brilliant Blue R250 befleckt sind.
Die Interaktion zwischen α2β1 Integrin und Kollagen wird durch die Bindung der Schlange Venom Komponente, Rhodocetin, von den malayischen Grubenotter (Calloselasma Rhodostoma)16,17blockiert. Verwendet als lösliche Liganden in dieser Titration ELISA, Rhodocetin aus dem Rohöl gereinigt wurde Venom bereits16beschrieben. Es wird in HEPES-gepufferte Kochsalzlösung (HBS; 10 mM HEPES/NaOH, pH 7,4, 150 mM NaCl) gelöst und bei-20 ° c eingefroren werden gespeichert Seine Konzentration wurde von BCA und seiner Reinheit wurde nachgewiesen durch SDS-PAGE. Als Antagonist, Rhodocetin blockiert nicht nur Kollagen, die Bindung an das Integrin α2β1 A-Domäne, aber auch die inaktiven Konformation der Integrin und verhindert somit Signalisierung von Kollagen in den Zellen oder Thrombozyten18stabilisiert. Es ist von großer biomedizinischen Bedeutung zu bestimmen, die Dissoziationskonstante des Rhodocetin mit seinen Rezeptor-Ziel und somit zu entwirren, seiner molekularen Mechanismus und pharmazeutischen Potenzial z.B.als Vermittler antithrombotische. Zu diesem Zweck eine Titrierung ELISA beschrieb ist einschließlich ihrer Bewertung gilt das für fast jeder Rezeptor-Ligand Interaktion mit einer Stöchiometrie von 1:1 Interaktion.
Access restricted. Please log in or start a trial to view this content.
Protokoll
(1) auf lagerlösungen
- Um 100 mL 10 X TBS pH 7.4 Lösung vorzubereiten, 6,06 g TRIS und 8,77 g NaCl in 90 mL entionisiertem Wasser auflösen, anpassen des pH-Werts 7,4 mit 37 % HCl-Lösung, die Lautstärke auf 100 mL mit entionisiertem Wasser füllen und Filtern Sie die Lösung.
- 100 mL 1 vorbereiten M HEPES/NaOH, pH 7.4 Lösung 23,83 g HEPES in 90 mL entionisiertem Wasser auflösen, anpassen den pH-Wert 7,4 mit 1,5 M NaOH, füllen Sie die Lautstärke auf 100 mL mit entionisiertem Wasser und Filtern Sie die Lösung.
- Lösen Sie um 100 mL 5 M NaCl-Lösung vorzubereiten, 29,2 g NaCl in 90 mL entionisiertem Wasser. Füllen Sie die Lautstärke auf 100 mL mit entionisiertem Wasser und Filtern Sie die Lösung.
- Um 100 mL 1 M MgCl2 Lösung vorzubereiten, lösen Sie 20,33 g MgCl2 · 6H2O in 90 mL entionisiertem Wasser. Füllen Sie die Lautstärke auf 100 mL mit entionisiertem Wasser und Filtern Sie die Lösung.
- Um 5 % Hitze-inaktivierten BSA im Wasser vorzubereiten, wiegen Sie 2,5 g der BSA (Teil V, pH 7.0) in einem 50 mL-Tube und in 45 mL entionisiertem Wasser auflösen. Füllen Sie die Lösung für 50 mL mit entionisiertem Wasser und erhitzen Sie die Lösung in einem Wasserbad bei 68 ° C für 45 min. im Eisbad abkühlen und bei-20 ° c lagern
- 25 % Glutaraldehyd Wasserlösung vorzubereiten.
Achtung: Glutaraldehyd ist schädlich, wenn geschluckt, giftig beim Einatmen und verursacht Verätzungen. Tragen Sie schützende Kleidung, Handschuhe, und Auge/Gesicht Schutz. - Erhöhen Sie die Kaninchen Antiserum als zuvor beschriebenen19. Der Titer von Antiserum wurde nach Standardprotokollen20bestimmt.
- Bereiten Sie Anti-Kaninchen-Immunglobulin-Antikörpern aus Ziege konjugiert mit alkalischer Phosphatase.
- Um 100 mL 0,1 M Glycin Lösung vorzubereiten, lösen Sie 0,75 g Glycin in 90 mL entionisiertem Wasser auf. Einstellen Sie des pH-Werts auf 10.4 mit 1,5 M NaOH-Lösung, füllen Sie die Lautstärke auf 100 mL mit entionisiertem Wasser, und Filtern Sie die Lösung.
- 100 mL 0,5 M Zn (II) vorbereiten-Acetat-Lösung, lösen Sie 10,98 g Zn (II)-Acetat · 2H2O in 90 mL entionisiertem Wasser. Füllen Sie die Lautstärke auf 100 mL mit entionisiertem Wasser und Filtern Sie die Lösung.
- Um 100 mL 1,5 M NaOH Lösung vorzubereiten, lösen Sie auf, 6,0 g NaOH in 90 mL entionisiertem Wasser. Füllen Sie die Lautstärke auf 100 mL mit entionisiertem Wasser und Filtern Sie die Lösung.
2. bereiten Sie Puffer und funktionierende Lösungen
- 5 mL 10 X TBS pH 7.4, mit 45 mL entionisiertem Wasser verdünnen und 100 µL einer 1 M MgCl2 Vorratslösung hinzufügen. Bei Zimmertemperatur aufbewahren. Die TBS ist pH 7.4, 2 mM MgCl2 Lösung für Immobilisierung des Rezeptors und das Waschen der Mikrotiterplatte.
- 10 mL 5 % der Hitze-inaktivierten BSA im Wasser und 5 mL 10 X TBS pH 7.4 mit entionisiertem Wasser auf 50 mL verdünnt. Fügen Sie 100 µL der Stammlösung 1 M MgCl2 hinzu und mischen Sie die Lösung gut. Halten Sie es auf dem Eis und bewahren Sie sie für weitere Versuche bei-20 ° C. Hinweis, dass 2,5 mL 1 % BSA in TBS, pH 7,4, 2 mM MgCl2 sind erforderlich für jede Titration-Kurve.
- Verdünnen Sie 2,5 mL 1 M HEPES/NaOH, pH 7,4 und 1,5 mL 5 M NaCl-Lösung, 50 mL mit entionisiertem Wasser, 100 µL 1 M MgCl2 Lösung hinzufügen und mischen Sie die Lösung gründlich um 50 mL HBS, pH 7.4: 50 mM HEPES/NaOH vorzubereiten , 150 mM NaCl.
- Fügen Sie 5 µL der Stammlösung 1M MgCl2 und 2 µL 0,5 M Zn (II)-Acetat-Stammlösung zu 5 mL 0,1 M Glycin Lösung, pH 10.4 vorzubereitende alkalische Phosphatase (AP) Puffer (Glycin-Lösung 0,1 M, pH 10.4, 1 mM MgCl2, 0, 2 mM Zn(II)-acetate).
(3) Immobilisierung des Rezeptors (Integrin α2A-Domain) auf einer Mikrotiterplatte
- Die Stammlösung Integrin α2A-Domäne in TBS, pH 7,4, 2 mM MgCl2 für eine Endkonzentration von 5 µg/mL zu verdünnen.
Hinweis: Die Lautstärke der Beschichtungslösung ist 650 µL für eine Titration Kurve bestehend aus 12 Brunnen-Folge von einer halben Bereich-Mikrotiterplatte. - Füllen Sie jeden Brunnen einer Zeile auf einer halben Bereich-Mikrotiterplatte mit 50 µL/Well von 5 µg/mL-Beschichtungslösung Integrin α2A-Domäne (Wildtyp oder Mutanten). Führen Sie jede Zeile der Titration zumindest in Duplikate (in diesem Beispiel als Vierlinge; siehe Layout der Mikrotiterplatte in Abbildung 1).
- Die Dichtplatte mit Folie oder einem Deckel schließen. Lassen Sie die Platte über Nacht bei 4 ° C.
4. Waschen Sie beschichtet Vertiefungen der Platte zweimal mit TBS, pH 7,4, 2 mM MgCl2
- Löslichen Rezeptor entfernen nun Moleküle, die haben nicht auf die Kunststoffoberfläche durch physikalische Adsorption immobilisiert wurde, entfernen Sie die Beschichtungslösung und füllen jeweils mit 50 µL TBS, pH 7,4, 2 mM MgCl2.
- Entfernen Sie die Waschlösung. Stellen Sie sicher, dass der Brunnen nicht zu trocken werden. Tippen Sie daher nicht die Mikrotiterplatte auf ein Gewebe Tuch, Restflüssigkeit zu entfernen. Verwenden einer mehrstufigen Pipette oder eine Multi-Kanal-Pipette, um die Brunnen schnell zu füllen.
- Wiederholen Sie waschen einmal.
5. unspezifische Bindungsstellen blockieren
- In TBS, pH 7,4, 2 mM MgCl2 in jede Vertiefung 50 µL 1 % BSA Lösung hinzufügen.
- Der Brunnen mit Folie verschließen oder mit einem Deckel schließen.
- Inkubieren Sie der Brunnen für 1 h bei Raumtemperatur.
Hinweis: Die Inkubation Schritte dieses Protokolls kann auf einer rockig oder schütteln Plattform durchgeführt werden. Aber dies ist nicht erforderlich und ändert nicht das Ergebnis des Experiments.
6. Vorbereitung der serielle Verdünnung Folge des Liganden, Rhodocetin
- Variieren Sie die Start-Konzentration der Rhodocetin und der Verdünnungsfaktor der serielle Verdünnung, um eine angemessene Bandbreite an Liganden Konzentrationen zu erhalten und eine vollständige Titration Kurve mit einem minimalen und maximalen Signal aufzuzeichnen. Beschäftigen Sie in diesem Experiment eine Start-Konzentration von 243 nM Rhodocetin und ein Verdünnungsfaktor von 2,3. Verdünnen Sie Rhodocetin-Stammlösung, die höchste Konzentration des Liganden der Zeile serielle Verdünnung. Für jede Titration Kurve mit einem Verdünnungsfaktor von 2,3, bereiten 115 µL 243 nM (d.h. 15,2 µg/mL) Rhodocetin Lösung in 1 % BSA/TBS, pH 7,4, 2 mM MgCl2 im Reagenzglas #1.
- Füllen Sie 65 µL 1 % BSA Lösung in TBS, pH 7,4, 2 mM MgCl2 10 Reagenzgläser, #2-#11 beschriftet.
- Übertragen von 50 µL der Rhodocetin Verdünnung aus dem Reagenzglas #1, Reagenzglas #2, mischen Sie beide Lösungen (Gesamtvolumen: 115 µL; Verdünnungsfaktor: 1:2. 3) durch Verreibung und dann Transfer 50 µL aus dieser Mischung Reagenzglas #3 usw.
- Diese serielle Verdünnung bis Reagenzglas #11 weiter.
Hinweis: Die Bände in Schritten 6.1-6.4 gegeben sind ausreichend für eine Titration Kurve. Multiplizieren Sie diese Volumes durch die Anzahl der Wiederholungen. In diesem Fall führen Sie acht Titrierung Kurven (Vierlinge von zwei α2A-Domäne Formen) und bereiten die folgenden Bände: 920 µL Rhodocetin-Lösung in der höchsten Konzentration im Reagenzglas #1; 520 µL 1 % BSA in TBS, pH 7,4, 2 mM MgCl2 füllen Sie jeweils die Reagenzgläser #2 und #11; und 400 µL des Transfervolumen von einem Rohr auf die nächste.
(7) die Bindung des Liganden (Rhodocetin) in verschiedenen Konzentrationen zu immobilisiert Rezeptor (Integrin α2A-Domain)
- Entfernen Sie die blockierende Lösung aus den Mikrotiter-Platte-Brunnen von einer Vakuumleitung.
- Fügen Sie sofort 50 µL der Rhodocetin Lösung in 1 % BSA/TBS, pH-Wert 7,4/MgCl2 Lösung von Reagenzglas #1 in Vertiefungen von Spalte 1, Lösung von Reagenzglas #2 in Spalte 2,etc. hinzufügen 50 µL 1 % BSA in TBS, pH 7.4 Vertiefungen , 2 mM MgCl2 (Sperrung und Verdünnung Puffer) als Liganden-freie Kontrolle in Vertiefungen Spalte 12 (siehe Layout der Mikrotiterplatte, Abbildung 1).
- Der Brunnen mit Folie verschließen oder mit einem Deckel schließen.
- Inkubieren Sie die Brunnen für 1,5 h bei Raumtemperatur (ca. 20-22 ° C).
8. Waschen Vertiefungen der Platte zweimal mit HBS, pH 7,4, 2 mM MgCl2
- Um nicht gebundenen Liganden Moleküle zu entfernen, entfernen Sie die verbindliche Lösung und füllen jeweils gut mit 50 µL von HBS, pH 7,4, 2 mM MgCl2. Dann entfernen Sie die Waschlösung.
- Achten Sie darauf, dass der Brunnen nicht zu trocken werden. Tippen Sie daher nicht die Mikrotiterplatte auf ein Gewebe Tuch, Restflüssigkeit zu entfernen. Verwenden einer mehrstufigen Pipette oder eine Multi-Kanal-Pipette, um die Brunnen schnell zu füllen.
- Wiederholen Sie waschen einmal.
9. Befestigen Sie Rezeptor gebundene Liganden mit 2,5 % Glutaraldehyd in HBS, pH 7,4, 2 mM MgCl2
- Bereiten Sie eine frische 2,5 % Glutaraldehyd Lösung durch Mischen von 1 Teil 25 % Glutaraldehyd Lösung und 9 Teile von HBS, pH 7,4, 2 mM MgCl2.
- Füllen Sie jeder Brunnen der Mikrotiterplatte mit 50 µL der Lösung 2,5 % Glutaraldehyd in HBS, pH 7,4, 2 mM MgCl2. Inkubieren Sie die Mikrotiterplatte für 10 min bei Raumtemperatur.
10. Waschen Vertiefungen der Platte dreimal mit 50 µL/Well von TBS, pH 7,4, 2 mM MgCl2
- Um zu entfernen und deaktivieren überschüssige Glutaraldehyd, die Fixierung Lösung entfernen und füllen jeweils gut mit 50 µL TBS, pH 7,4, 2 mM MgCl2. Entfernen Sie dann die Waschlösung.
Hinweis: Gießen Sie die Glutaraldehyd-haltige Fixierung Lösung aus der Mikrotiterplatte in eine Schale und verwerfen Sie die Fixierung Lösung zu, nachdem es durch ein gleiches Volumen von TBS, pH 7.4 inaktiviert wurden. - Achten Sie darauf, dass der Brunnen nicht zu trocken werden. Tippen Sie daher nicht die Mikrotiterplatte auf ein Gewebe Tuch, Restflüssigkeit zu entfernen. Verwenden einer mehrstufigen Pipette oder eine Multi-Kanal-Pipette, um die Brunnen schnell zu füllen.
- Wiederholen Sie waschen zweimal.
11. die Quantifizierung der Rezeptor-gebundenen Liganden von ELISA
- Fügen Sie 50 µL/Well der primären Antikörper-Lösung in 1 % BSA in TBS, pH 7,4, 2 mM MgCl2. Die primären Antikörper-Lösung ist ein Kaninchen Antiserum gegen Rhodocetin19, 1:2,000 in 1 % BSA in TBS, pH 7,4, 2 mM MgCl2verdünnt.
- Inkubieren Sie die Platte für 75-90 min bei Raumtemperatur. Waschen Sie alle Vertiefungen der Platte dreimal mit 50 µL/Well von TBS, pH 7,4, 2 mM MgCl2. Während der drei Waschschritte, Abhören von der Mikrotiterplatte auf ein Tuch ist Tuch nicht erforderlich.
- Fügen Sie 50 µL/Well der Sekundärantikörper Lösung in 1 % BSA in TBS, pH 7,4, 2 mM MgCl2. Zu diesem Zweck Verdünnen der Sekundärantikörper, Kaninchen Immunglobulin-targeting Ziege Antikörper konjugiert mit alkalischer Phosphatase, 1:2,000 in 1 % BSA in TBS, pH 7,4, 2 mM MgCl2. Inkubieren Sie die Platte für 75-90 min bei Raumtemperatur.
- Ansetzen Sie AP-Erkennung Lösung, indem man eine 5 mg Tablette mit 4-Nitrophenyl Phosphat Binatrium Salz Hexahydrat (Phosphatase Substrat) in 5 mL AP Puffer (Glycin-Lösung 0,1 M, pH 10.4, mit 1 mM MgCl2 und 0,2 mM Zn(II)-acetate).
- Waschen Sie alle Vertiefungen der Platte dreimal mit 50 µL/Well von TBS, pH 7,4, 2 mM MgCl2, unmittelbar vor der nächsten Schritt. Tippen Sie auf der Mikrotiterplatte auf ein Gewebe Tuch nach dem letzten Waschschritt, alle Spuren von Flüssigkeit zu entfernen.
- Die Vertiefungen der Mikrotiterplatte 50 µL/Well der AP-Erkennung Lösung hinzufügen. Fügen Sie hinzu, AP-Erkennung Lösung prompt alle Brunnen, die enzymatische Umwandlung so gleichzeitig starten. Verwenden Sie daher eine Multi-Kanal-Pipette.
- Inkubieren Sie die Platte bei Raumtemperatur, bis die Lösung in den Brunnen mit der höchsten Konzentration des Liganden vergilben.
Hinweis: Die Inkubationszeit variieren zwischen 5 min und 1 h je nach der Intensität des Signals. - Beenden Sie Konvertierung des Substrats Phosphatase durch Zugabe von 50 µL/Well 1,5 M NaOH Lösung. Lassen Sie die Platte stehen für einige Minuten um streifenfreie Mischen der beiden Lösungen zu gewährleisten. Um die gleiche Inkubationszeit in alle Wells zu rechtfertigen, verwenden Sie eine Multi-Kanal-Pipette und fügen Sie 1,5 M NaOH in der gleichen Reihenfolge in die Vertiefungen wie die des Substrats im Schritt 11,8 AP-Erkennung hinzugefügt.
- Messen Sie die optische Dichte (OD) bei 405 nm von jedem Bohrloch durch einen ELISA-Reader.
12. Bewertung der Titration Signale
- Öffnen Sie die Tabelle der Rohdaten, die OD405nm Werte mit Excel. Wie diese Signalwerte der Titrierung Kurven in Zeilen gelesen werden, übertragen Sie die Werte einer Spalte und Label mit der Konzentrationen von zusätzlichen Liganden in einer anderen Spalte.
- Öffnen Sie den Graphpad Prism 5 (Version 5.0). Öffnen Sie eine neue Projektdatei im Hauptmenü. Wählen Sie das XY -Format unter neue Daten & Grafik. Wählen Sie die Option Enter und Handlung eine zentrale Anlaufstelle für jeden Wert für die Y-Achse.
- Kopieren Sie die beiden Spalten, Konzentration von zusätzlichen Liganden und signalisieren Werte (OD405nm ) aus der Excel-Datei und fügen Sie sie in das Datenblatt von GraphPad Prism als X- und Y-Werte, beziehungsweise.
- Öffnen Sie die Sub-Analyseprogramm von GraphPad Prism 5 und wählen Sie die Option nicht-lineare Regression unter XY-Analyse. Wählen Sie benutzerdefinierte Gleichung , und drücken Sie die Schaltfläche "neu" um eine neue Gleichung zu erstellen.
- Geben Sie die Titration Kurve Gleichung in der Form: Y-=(Smax-Smin)*((X+R+K)-sqrt((X+R+K)^2-4*R*X)) /(2*R) + Smin + B * X in der neu eröffneten Vorlagenblatt mit Y wird der Signalwert S, wobei die Konzentration von zusätzlichen Liganden L X , Wird die Konzentration von R immobilisiert Rezeptor, wird die Dissoziationskonstante K und B, der Hintergrund Steigung. Definieren Sie die entsprechenden Einschränkungen, z. B. K > 0 und R > 0.
Hinweis: Diese Gleichung ist die gleiche Gleichung der Gleichung 9 in einer anderen Form. - Analysieren Sie die Werte des Datenblatts, durch die Wahl der benutzerdefinierte Gleichung, die neu erstellt wurde. Öffnen Sie die Tabelle mit den Werten der berechneten Näherung (K, Rt, S-max, Sminund B) die im Abschnitt " Ergebnis " auf der linken Seite des Bildschirms Software dargestellt sind.
Hinweis: Die Software bestimmt die 5 Parameter durch iterative Anpassung der nichtlinearen Regression nur dann, wenn die Titration Kurve aus mindestens 5 Datenpunkte besteht. - Werten Sie der Parameter K, Rt, S-max, Sminund B für jede Gruppe von Titrierung Kurven statistisch aus und Korrelieren Sie die Parameter mit der Besonderheit der Gruppe (mutierte oder chemisch modifiziert Liganden oder Rezeptor).
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Nach der ELISA entwickelt wurde, die gelbe Farbe des Substrats konvertierten alkalische Phosphatase, Para- Nitrophenolate, weist darauf hin, dass die Menge des gebundenen Rhodocetin Liganden sinkt mit abnehmender Konzentration von zusätzlichen Rhodocetin aus Spalten 1 bis 11 (Abbildung 1). Die farblosen Vertiefungen in der Rhodocetin-freien Brunnen in Spalte 12 zeigen eine niedrige Hintergrundsignal.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Titration ELISA ist ein vielseitiger Testsystem für die Dissoziation von einem Rezeptor-Ligand Interaktion bestimmen. Als die Titration ELISA umgeht die Notwendigkeit, freie und gebundene Liganden effektiv zu trennen und ihre Konzentration quantitativ analysieren wesentlich mehr Studien und Publikationen haben Titration ELISAs beschäftigt, anstatt Bindung Kurven . Darüber hinaus sind Titration ELISAs einfach durchzuführen und erfordert relativ geringe Mengen an Rezeptor und Ligand. Für die genaue Analyse der Dis...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Der Autor hat nichts preisgeben.
Danksagungen
Das Protokoll und den Algorithmus wurden in einem Projekt, finanziert von der Deutschen Forschungsgemeinschaft (DFG-Stipendium SFB1009 A09 und EB177/13-1) entwickelt. Der Autor dankt Barbara Schedding und Felix Schmalbein für technischen Support und Dr. Niland für kritisch liest das Manuskript.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| TRIS | neoFrox | 1125KG001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| NaCl | Applichem | 1,316,591,214 | |
| MgCl2 | Merck | 172571 | |
| integrin a2A, wild-type and mutant, recombinant | isolated in author's lab | ||
| NiNTA superflow column | Qiagen, Germany | 30821 | |
| Coomassie-Brilliant Blue R250 | Serva | 35050 | |
| bicinchoninic acid assay (BCA), protein concentration determination kit | Fisher Scientific | 23225 | |
| bovine serum albumine (BSA), fraction V | Applichem | A1391 | |
| 25 % solution of glutaraldehyde | Merck | 354400 | |
| anti-rabbit immunglobulin-antibodies from goat, conjugated with alkaline phosphatase | Sigma-Aldrich | A9919 | |
| Glycine | Applichem | A1377 | |
| Zn(II)-acetate | Applichem | A4324 | |
| NaOH | Applichem | A1551 | |
| Alkaline phosphatase substrate tablet (5 mg) | Sigma-Aldrich | S0942 | |
| Costar half-area microtiter plate | Thermo Scientific | Corning 3690 | |
| micro reaction tubes | Eppendorf | 30120086 | |
| Microplate ELISA reader | BioTek | Synergy HT |
Referenzen
- Klotz, I. M. The application of the law of mass action to binding by proteins; interactions with calcium. Arch Biochem. 9, 109-117 (1946).
- Klotz, I. M. Ligand-receptor complexes: origin and development of the concept. J Biol Chem. 279 (1), 1-12 (2004).
- Bisswanger, H. Ch. 1: Multiple Equilibria, Principles and Derivations. Enzyme Kinetics: Principles and Methods. , Wiley VCH Verlag GmbH. 1-26 (2017).
- Shimura, K., Kasai, K. Affinity gel titration: quantitative analysis of the binding equilibrium between immobilized protein and free ligand by a continuous titration procedure. Anal Biochem. 149 (2), 369-378 (1985).
- Gong, M., Nikcevic, I., Wehmeyer, K. R., Limbach, P. A., Heineman, W. R. Protein-aptamer binding studies using microchip affinity capillary electrophoresis. Electrophoresis. 29 (7), 1415-1422 (2008).
- Hanes, M. S., Ratcliff, K., Marqusee, S., Handel, T. M. Protein-protein binding affinities by pulse proteolysis: application to TEM-1/BLIP protein complexes. Protein Sci. 19 (10), 1996-2000 (2010).
- Latour, R. A. The Langmuir isotherm: a commonly applied but misleading approach for the analysis of protein adsorption behavior. J Biomed Mater Res A. 103 (3), 949-958 (2015).
- Stockell, A. The binding of diphosphopyridine nucleotide by yeast glyceraldehyde-3-phosphate dehydrogenase. J Biol Chem. 234 (5), 1286-1292 (1959).
- Heyn, M. P., Weischet, W. O. Circular dichroism and fluorescence studies on the binding of ligands to the α subunit of tryptophan synthase. Biochem. 14 (13), 2962-2968 (1975).
- Campbell, I. D., Humphries, M. J. Integrin structure, activation, and interactions. Cold Spring Harb Perspect Biol. 3 (3), (2011).
- Zeltz, C., Gullberg, D. The integrin-collagen connection--a glue for tissue repair? J Cell Sci. 129 (4), 653-664 (2016).
- Kanchanawong, P., et al. Nanoscale architecture of integrin-based cell adhesions. Nature. 468 (7323), 580-584 (2010).
- Luo, B. H., Carman, C. V., Springer, T. A. Structural basis of integrin regulation and signaling. Annu Rev Immunol. 25, 619-647 (2007).
- Luo, B. H., Springer, T. A. Integrin structures and conformational signaling. Curr Opin Cell Biol. 18 (5), 579-586 (2006).
- Ricard-Blum, S. The collagen family. Cold Spring Harb Perspect Biol. 3 (1), a004978(2011).
- Eble, J. A., Tuckwell, D. S. The α2β1 integrin inhibitor rhodocetin binds to the A-domain of the integrin α2 subunit proximal to the collagen-binding site. Biochem J. 376 (Pt 1), 77-85 (2003).
- Eble, J. A., et al. The α2β1 integrin-specific antagonist rhodocetin is a cruciform, heterotetrameric molecule. FASEB J. 23 (9), 2917-2927 (2009).
- Eble, J. A., et al. Dramatic and concerted conformational changes enable rhodocetin to block α2β1 integrin selectively. PLoS Biol. 15 (7), e2001492(2017).
- Bracht, T., Figueiredo de Rezende, F., Stetefeld, J., Sorokin, L. M., Eble, J. A. Monoclonal antibodies reveal the alteration of the rhodocetin structure upon α2β1 integrin binding. Biochem J. 440 (1), 1-11 (2011).
- Harlow, E., Lane, D. Chapter 5: Immunizations. Antibodies, a laboratory manual. 5, Cold Spring Harbor Laboratory. 53-138 (1988).
- Lu, D. Analyzing interactions between SSB and proteins by the use of fluorescence anisotropy. Methods Mol Biol. 922, 155-159 (2012).
- Fielding, L. NMR methods for the determination of protein-ligand dissociation constants. Curr Top Med Chem. 3 (1), 39-53 (2003).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr Opin Struct Biol. 11 (5), 560-566 (2001).
- McDonnell, J. M. Surface plasmon resonance: towards an understanding of the mechanisms of biological molecular recognition. Curr Opin Chem Biol. 5 (5), 572-577 (2001).
- Rich, R. L., Myszka, D. G. Advances in surface plasmon resonance biosensor analysis. Curr Opin Biotechnol. 11 (1), 54-61 (2000).
- Pesquero, N. C., et al. Real-time monitoring and kinetic parameter estimation of the affinity interaction of jArtinM and rArtinM with peroxidase glycoprotein by the electrogravimetric technique. Biosens Bioelectron. 26 (1), 36-42 (2010).
- Scheuermann, T. H., Padrick, S. B., Gardner, K. H., Brautigam, C. A. On the acquisition and analysis of microscale thermophoresis data. Anal Biochem. 496, 79-93 (2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten