É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Titulação ELISA como um método para determinar a constante de dissociação de interação do ligante do Receptor
Neste Artigo
Resumo
Um protocolo detalhado para executar uma titulação ELISA é descrito. Além disso, um algoritmo de romance é apresentado para avaliar a titulação ELISAs e obter uma constante de dissociação de ligação de um ligante solúvel a um receptor de imobilizado-placa de microtitulação.
Resumo
A constante de dissociação descreve a interação entre os dois parceiros em equilíbrio a ligação e é uma medida de sua afinidade. É um parâmetro fundamental para comparar diferentes ligantes, por exemplo, inibidores do competidor, isoformas de proteínas e mutantes, para sua força de ligação a um parceiro de vinculação. Constantes de dissociação são determinados pela plotagem concentrações de limite contra o ligante livre como curvas de ligação. Em contraste, curvas de titulação, em que um sinal que é proporcional à concentração de ligante ligado é plotado contra a concentração total de ligante adicionado, são mais fáceis de gravar. O sinal pode ser detectado espectroscopicamente e por ensaio imunoenzimático (ELISA). Isto é exemplificado em um protocolo para uma titulação ELISA que mede a ligação do rhodocetin derivado de veneno de cobra para seu domínio alvo imobilizado da integrina α2β1. Titulação ELISAs são versáteis e amplamente utilizado. Qualquer par de interação de proteínas pode ser usado como receptor de imobilizado e solúvel ligante, desde que ambas as proteínas são puras, e as respectivas concentrações são conhecidas. Até agora, a dificuldade tem sido determinar a constante de dissociação de uma curva de titulação. Neste estudo, é introduzida uma função matemática subjacente de curvas de titulação. Sem qualquer estimativa propenso gráfica de um rendimento de saturação, este algoritmo permite o processamento dos dados brutos (intensidades de sinal em diferentes concentrações de ligante adicionado) diretamente pela avaliação matemática através de regressão não-linear. Assim, várias curvas de titulação podem ser gravadas simultaneamente e transformadas em um conjunto de parâmetros característicos, entre elas a constante de dissociação e a concentração de vinculação-ativo do receptor, e elas podem ser avaliadas estatisticamente. Quando combinado com este algoritmo, titulação ELISAs ganhar a vantagem de apresentar diretamente a constante de dissociação. Portanto, eles podem ser usados mais eficientemente no futuro.
Introdução
A constante de dissociação K é um parâmetro chave para descrever a afinidade de um receptor (R) para seu ligante (L). Com base na lei de ação das massas, K é definido para o equilíbrio, no qual a RL complexo ligante-receptor dissocia-se para o receptor R e o ligante l:
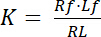 Equação 1
Equação 1
com os índices f indicando o estado livre/desacoplado do receptor e ligante. A concentração do complexo receptor-ligante, RL, é idêntica à concentração do ligante do receptor-limite Lb. Como a concentração total de receptores Rt é a soma do receptor livre Rf e ligados a ligante receptor Rb = Lb, a constante de dissociação também pode ser escrita como:
 Equação 2
Equação 2
Portanto, a saturação rendimento Y, definido como a fração do limite ligante Lb em relação a concentração total de receptores Rt,
 Equação 3
Equação 3
depende da concentração do ligante livre Lf:
 Equação 4
Equação 4
Esta relação hiperbólica descreve a curva de ligação de uma interação receptor-ligante e seu enredo mostra a concentração de ligante limite Lb em função da concentração de ligante livre Lf. A partir da curva de ligação, a constante de dissociação K pode ser derivada, como a concentração de ligante livre no escoamento de metade-máximo de saturação. Além disso, diferentes algoritmos para linearizar curvas de ligação foram estabelecidos, tais como o enredo de duplo recíproco por Klotz1,2, ou transformações de acordo com Scatchard ou Hanes (revisto por Bisswanger3). No entanto, todos os algoritmos sofrem com o problema que o valor máximo do rendimento de saturação, que é assintoticamente aproximou-se em concentrações elevadas de ligante livre na curva de ligação, tem de ser estimada em uma gráfica pré-avaliação e, portanto, é sujeito a erros.
Além disso, a determinação de uma curva de ligação requer a quantificação de ligante livre e ligado durante o equilíbrio da ligação. Para este fim, o ligante livre deve ser separada do ligante-receptor-limite e quantificado. Portanto, o ligante e receptor tem que diferem em suas propriedades, tais como um ligante não proteico em oposição a um receptor de proteína. Se ambos os parceiros de ligação são proteínas, eles precisam ser distinguíveis em seus tamanhos, encargos ou outras características moleculares. No entanto, a quantificação das concentrações de ligante em abordagens de pequena escala de vinculação é uma tarefa difícil. Etiquetar radioativo do ligante tem sido muitas vezes necessário para detectar a baixa concentração de ligante acoplado, especialmente se quantidades substanciais de receptores não estavam disponíveis ou acessíveis. Além disso, o ligante-receptor-limite pode dissociar durante e após o isolamento de forma não negligenciável. Daí, métodos complexos, tais como equilíbrio gel filtração4, eletroforese capilar5e pulso proteólise6, são necessários para quantificar o ligante do receptor ligado e separá-lo de ligante livre.
Em contraste com estes ensaios obrigatórios, experimentos de titulação não requerem a separação quantitativa de ligante ligado e livre. Para este fim, um receptor em uma concentração constante é titulado com diferentes concentrações de ligante adicionado. Pela ligação ao receptor, o ligante ligado tem uma propriedade biofísica que diferencia o ligante livre e é mensurável por, por exemplo, fotometria, fluorometry ou de anticorpos. Assim, um sinal S, que é proporcional a saturação Y de rendimento e, consequentemente, também para a concentração de ligante do receptor ligado (Lb), é detectado como uma função da concentração total de ligante adicionado (Lt). Ambos os parâmetros, o sinal de S e a concentração total de ligante adicionado são quantificados de forma direta e mais fácil do que as concentrações de ligante ligado e livre. Especialmente, a detecção de ligante do receptor ligado por ensaio imunoenzimático (ELISA) permitiu a redução dos volumes de amostra para abaixo de 100 µ l, bem como medidas paralelas de várias concentrações de ligante em placas de microtitulação multi bem. Em uma titulação, ELISA, um receptor é fisicamente adsorvido a uma placa de microtitulação na mesma concentração e titulado com ligante solúvel. O receptor está imobilizado na superfície de plástico essencialmente por adsorção hidrofóbica. A concentração superficial de imobilizado receptor correlaciona-se com a concentração de revestimento do receptor em uma relação não-linear, provável, de acordo com Langmuir´s de isoterma de adsorção7. Além do número total de moléculas do receptor adsorvida, seu estado de atividade é outro parâmetro importante para os ensaios de titulação. Apenas imobilizado receptores que tem mantido a actividade de ligação do ligante, são relevantes para o ensaio de titulação e eventualmente contribuam para a concentração total de receptores ativa Rt do ensaio de titulação, o que não pode ser determinado diretamente.
Sites sobre a superfície de plástico, não são abrangidas pelo receptor de imobilizado são propensas a adsorver outras proteínas, tais como o ligante. Adsorção física do ligante a tais sites superfície plásticas resultaria em um sinal similar como o ligante do receptor ligado, ainda de forma não específica. Para reduzir este sinal inespecífica, os sites de superfície plásticos das placas de microtitulação que não têm sido revestidos com proteína ainda serão bloqueados com albumina de soro bovino (BSA). No entanto, para alguns ensaios de titulação do receptor-ligante, sinais de fundo inespecíficos podem ser observados. Em seguida, outro agentes de bloqueio, tais como uma solução de 0,2% de gelatina ou de 0,04% Tween 20, são recomendados.
Após a ligação ao receptor, o ligante livre é removido por duas etapas de lavagem. Ligante ligado permanece com o receptor, que é imobilizado à superfície plástica do poço da microplaca e, opcionalmente, reforçado por fixação química. Para a subsequente covalent ligação transversal de limite ligante e receptor imobilizado com glutaraldeído, a substância tampão TRIS é substituída para HEPES, sem qualquer mudança na ligação do ligante. HEPES, em contraste com TRIS, não inativar glutaraldeído. A ligação covalente transversal em glutaraldeído corrige o ligante acoplado com seu receptor e impede sua dissociação durante as etapas subsequentes de lavagem e incubação. Assim, a interação receptor-ligante é quimicamente congelada e garante uma curva de titulação, que é não afetada por etapas subsequentes de lavagem e incubação. No entanto, fixação de glutaraldeído quimicamente pode modificar o ligante e receptor de tal forma que sua interação é reduzida ou abolida. Além disso, modificação de epítopos dentro o ligante pode mudar a afinidade de ligação do anticorpo de detecção, especialmente se um anticorpo monoclonal é usado para quantificar o ligante acoplado. Embora nenhum destes efeitos adversos da fixação de glutaraldeído ocorre nesta titulação ELISA, a sensibilidade do teste para glutaraldeído deve ser testada para cada interação receptor-ligante antes do experimento de titulação. Após a fixação, glutaraldeído em excesso é removido em três etapas de lavagem com tampão TRIS-contendo. TRIS inactivates restantes grupos aldeído, que nonspecifically podem reagir com a detecção de anticorpos na etapa subsequente.
A quantidade de ligante ligado é quantificada com anticorpos enzima-lig, que fornecem um sinal fotométrico de ELISA S. Isto é plotado versus o ligante total concentração Lt adicionado a cada poço. Apesar de sua aquisição mais fácil, a curva de titulação não é uma função de hiperbólicas em contraste com a curva de ligação. Além disso, tem sido pouco claras como calcular a constante de dissociação K de uma curva de titulação. Embora algoritmos para linearizar curvas de titulação espectroscopicamente adquiridos independentemente foram relatados por Stockell8 e Heyn e Weischet9, ela falhou devido a sua incerteza de estimar o sinal máximo valor que o rendimento de saturação aproxima-se em concentrações elevadas de ligante adicionado.
Aqui, uma titulação ELISA e um algoritmo de regressão não-linear são descritos para derivar a constante de dissociação K para uma interação do ligante do receptor de uma curva de titulação. Este protocolo é exemplificado para a interação da-domínio de ligação de colágeno da integrina α2β1 com um inibidor de derivada de veneno de cobra. Integrinas são moléculas de adesão celular, que mediam a ancoragem das células para a matriz extracelular circundante ou membrana basal subjacente10,11. Além disso, integrinas transmitem sinais importantes entre as células e a matriz extracelular através do recrutamento de moléculas de sinalização adicional e formando novas organelas celulares, adhesomes, em cima da célula-matriz interação12,13, 14. colágeno, o ligante de integrina α2β1, é a proteína mais abundante do corpo humano e é um componente crucial do andaime do tecido conjuntivo15. A interação entre a integrina α2β1 e colágeno é mediada pela-domínio de subunidade α2 integrina. O integrina α2A-domínio contém um cátion divalente, que é necessária para a ligação de colágeno e estabiliza sua estrutura. O selvagem-tipo formulário, bem como os mutantes do domínio α2A, como aquele em que o resíduo da superfície exposta Y216 tinha sido substituído por uma glicina, podem ser facilmente recombinantes produzidos em um sistema de expressão bacteriana e isolados através de seus oligo-His-tags com um NiNTA coluna de Superflow com uma diálise subsequente contra salino TRIS (TBS; 50 mM TRIS/HCl, pH 7,4, 150 mM NaCl) contendo 2 mM MgCl216. As concentrações foram determinadas com o ensaio de ácido bicinchoninic (BCA) e suas purezas são testadas por SDS-PAGE convencional e coradas com Coomassie Brilliant Blue R250.
A interação entre a integrina α2β1 e colágeno é bloqueada pela vinculação do componente de veneno de cobra, rhodocetin, a partir do Malayan pit viper (Calloselasma rhodostoma)16,17. Usado como um ligante solúvel nesta titulação ELISA, rhodocetin foi purificado a partir do crude veneno conforme descrito anteriormente,16. Ele é dissolvido em solução salina tampão HEPES (HBS; 10 mM HEPES/NaOH, pH 7,4, 150 mM NaCl) e é armazenado congelado a-20 ° C. Sua concentração foi determinada pelo BCA e sua pureza foi comprovada por SDS-PAGE. Como um antagonista, rhodocetin não só bloqueia o colágeno vinculativos para a integrina α2β1 A-domínio, mas também estabiliza a conformação inativa a integrina, evitando qualquer sinalização de colágeno em células ou plaquetas18. É de grande importância biomédica para determinar a constante de dissociação de rhodocetin com seu alvo do receptor e, portanto, desvendar seu mecanismo molecular e farmacêutica potencial por exemplo, como um agente antitrombótico. Para este fim, uma titulação ELISA é descrita, incluindo sua avaliação, que é aplicável a praticamente qualquer interação receptor-ligante com uma estequiometria 1:1 interação.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. as soluções
- Para preparar 100 mL de 10 x TBS solução de pH 7,4, dissolver 6,06 g de TRIS e 8,77 g de NaCl em 90 mL de água desionizada, ajustar o pH para 7,4 com solução de HCl 37%, preencher o volume a 100 mL com água desionizada e filtrar a solução.
- Para preparar 100 mL de 1 M HEPES/NaOH, solução de pH 7,4, dissolver 23,83 g de HEPES em 90 mL de água desionizada, ajustar o pH para 7,4 com 1,5 M de NaOH, preencher o volume a 100 mL com água desionizada e filtrar a solução.
- Para preparar 100 mL de solução de NaCl 5m, dissolva 29,2 g de NaCl em 90 mL de água desionizada. Preencher o volume a 100 mL com água desionizada e filtrar a solução.
- Para preparar 100 mL de uma solução 1 M MgCl2 , dissolver 20,33 g de MgCl2 · 6H2O em 90 mL de água desionizada. Preencher o volume a 100 mL com água desionizada e filtrar a solução.
- Para preparar 5% inactivadas pelo calor BSA em água, pesar 2,5 g de BSA (fração V, pH 7,0) em um tubo de 50 mL e dissolvê-lo em 45 mL de água desionizada. Encha a solução de 50 mL com água desionizada e aqueça a solução em um banho de água a 68 ° C por 45 min. resfriá-lo em um banho de gelo e armazená-lo a-20 ° C.
- Prepare a solução aquosa de glutaraldeído 25%.
Atenção: O glutaraldeído é prejudicial se engolido, tóxico por inalação e provoca queimaduras. Usar vestuário de protecção, luvas e olhos/face. - Levante o soro de coelho, como descrito anteriormente,19. O título do anti-soro foi determinado de acordo com protocolos padrão20.
- Prepare a imunoglobulina de coelho antianticorpos de cabra conjugada com fosfatase alcalina.
- Para preparar 100 mL de solução 0,1 M de glicina, dissolva 0,75 g de glicina em 90 mL de água desionizada. Ajustar o pH a 10.4 com solução de NaOH de 1,5 M, preencher o volume a 100 mL com água desionizada e filtrar a solução.
- Para preparar 100 mL de 0,5 M de Zn (II)-solução de acetato, dissolver 10,98 g de Zn (II)-acetato · 2H2O em 90 mL de água desionizada. Preencher o volume a 100 mL com água desionizada e filtrar a solução.
- Para preparar 100 mL de solução de NaOH de 1,5 M, dissolva 6,0 g de NaOH em 90 mL de água desionizada. Preencher o volume a 100 mL com água desionizada e filtrar a solução.
2. prepare a Buffers e trabalhar soluções
- Diluir 5 mL de 10 x TBS pH 7,4, com 45 mL de água desionizada e adicionar 100 µ l de uma solução stock de 1m MgCl2 . Mantê-lo em temperatura ambiente. A TBS, pH 7,4, 2 mM MgCl2 solução é para a imobilização do receptor e a placa de microtitulação de lavagem.
- Dilua 10 mL de 5% de BSA inactivadas pelo calor na água e 5 mL de 10 x TBS, pH 7,4 com água destilada a 50 mL. Adicionar 100 µ l de solução-mãe 1 M MgCl2 e homogeneizar a solução. Mantê-lo no gelo e armazená-lo para novas experiências a-20 ° C. Nota que 2,5 mL de 1% de BSA na TBS, pH 7,4, 2 mM MgCl2 são necessários para cada curva de titulação.
- Diluir 2,5 mL de 1 M HEPES/NaOH, solução de NaCl de 5 M pH 7,4 e 1,5 mL a 50 mL com água desionizada, adicionar 100 µ l MgCl2 uma solução 1 M e misturar a solução completamente para preparar 50 mL de HBS, pH 7.4: 50 mM HEPES/NaOH , 150 mM de NaCl.
- Adicionar 5 µ l de solução-mãe 1M MgCl2 e 2 µ l 0,5 M Zn (II)-solução de acetato de solução de glicina 5 mL 0,1 M, pH 10.4 para preparar o tampão de fosfatase alcalina (AP) (solução de glicina 0,1 M, pH 10.4, 1 mM MgCl2, Zn(II)-acetate) de 0.2 mM.
3. imobilização do Receptor (integrina α2A-domínio) para uma placa de microtitulação
- Dilua a solução-mãe de integrina α2A-domínio na TBS, pH 7,4, 2 mM MgCl2 para uma concentração final de 5 µ g/mL.
Nota: O volume da solução de revestimento é 650 µ l para uma curva de titulação, que compreende um poço 12-linha de uma placa de metade da área-microplaca. - Encha cada poço de uma linha em uma placa de microtitulação de área metade com 50 µ l/poço da solução de revestimento 5 µ g/mL de integrina α2A-domínio (selvagem-tipo ou mutante). Executar todas as linhas de titulação pelo menos em duplicatas (neste exemplo como quadrigêmeos; consulte o layout da placa de microtitulação na Figura 1).
- Selar o prato com papel alumínio ou fechá-la com uma tampa. Deixe o prato a 4 ° C durante a noite.
4. lavagem de Poços revestidos da placa duas vezes com TBS, pH 7,4, 2 mM MgCl2
- Para remover o receptor solúvel moléculas, que não têm sido imobilizadas na superfície plástica por adsorção física, remover a solução de revestimento e encha cada um bem com 50 µ l de TBS, pH 7,4, 2 mM MgCl2.
- Remova a solução de lavagem. Certifique-se de que os poços não tornar-se seco. Portanto, não toque a placa de microtitulação sobre um pano de tecido para remover o líquido residual. Use uma pipeta de várias etapas ou uma pipeta multicanal para encher os poços rapidamente.
- Repita essa etapa de lavagem uma vez.
5. bloquear Sites de ligação inespecífica
- Adicione 50 µ l de solução a 1% BSA na TBS, pH 7,4, 2 mM MgCl2 a cada poço.
- Reseal poços com papel alumínio ou fechar com uma tampa.
- Incube os poços por 1h à temperatura ambiente.
Nota: Qualquer uma das etapas de incubação do presente protocolo pode ser executada em uma plataforma de balanço ou agitação. No entanto, isto não é necessário e não altera o resultado do experimento.
6. preparação de uma linha de diluição Serial do ligante, Rhodocetin
- Varie a concentração inicial de rhodocetin e o fator de diluição de diluição serial, para obter um conjunto adequado de concentrações de ligante e gravar uma curva de titulação completa com um sinal mínimo e máximo. Neste experimento, empregam uma concentração inicial de 243 nM rhodocetin e um factor de diluição de 2.3. Dilua a solução-mãe de rhodocetin para a maior concentração de ligante da linha serial diluição. Para cada curva de titulação com um factor de diluição de 2.3, preparar 115 µ l de solução de rhodocetin 243 nM (ou seja, 15,2 µ g/mL) em 1% BSA/TBS, pH 7,4, 2 mM MgCl2 em tubo de ensaio #1.
- Preencha 65 µ l de solução 1% de BSA na TBS, pH 7,4, 2 mM MgCl2 em 10 tubos de ensaio, rotulado #2 a #11.
- Transferir 50 µ l da diluição do rhodocetin do tubo de ensaio #1 para o tubo de ensaio #2, misturar as duas soluções (total volume: 115 µ l; factor de diluição: 1:2. 3) por trituração e, em seguida, transferência 50 µ l de mistura para tubo de ensaio #3, etc.
- Continue esta diluição serial até o tubo de ensaio #11.
Nota: Os volumes dados em etapas 6.1-6.4 são suficientes para uma curva de titulação. Multiplica esses volumes pelo número de repetições. Neste caso, realizar oito curvas de titulação (quadrigêmeos de duas formas de α2A-domínio) e preparar os seguintes volumes: 920 µ l de solução de rhodocetin em maior concentração no tubo de ensaio #1; 520 µ l de 1% de BSA na TBS, pH 7,4, 2 mM MgCl2 para preencher cada um dos tubos de teste #2 a #11; e 400 µ l de volume de transferência de um tubo para o próximo.
7. ligação de ligante (Rhodocetin) em diferentes concentrações a imobilizado do Receptor (integrina α2A-domínio)
- Remova a solução de bloqueio dos poços da microplaca placa por uma linha de vácuo.
- Imediatamente adicione 50 µ l da solução de rhodocetin em 1% BSA/TBS, solução de pH 7,4/MgCl2 do tubo de ensaio #1 nos poços da coluna 1, solução de tubo de ensaio #2 poços da coluna 2,etc. adicionar 50 µ l de 1% de BSA na TBS, pH 7,4 , 2 mM MgCl2 (tampão de bloqueio e diluição) como um ligante livre do controle de poços da coluna 12 (consulte layout de placa de microtitulação, Figura 1).
- Reseal poços com papel alumínio ou fechar com uma tampa.
- Incube os poços por 1,5 h em temperatura ambiente (cerca de 20-22 ° C).
8. lavagem de poços da placa duas vezes com HBS, pH 7,4, 2 mM MgCl2
- Para remover moléculas de ligante vinculados, remover a solução de ligação e encha cada um com 50 µ l de HBS, pH 7,4, 2 mM MgCl2. Em seguida, remova a solução de lavagem.
- Cuide-se que os poços não tornar-se seco. Portanto, não toque a placa de microtitulação sobre um pano de tecido para remover o líquido residual. Use uma pipeta de várias etapas ou uma pipeta multicanal para encher os poços rapidamente.
- Repita essa etapa de lavagem uma vez.
9. fixar o ligante-Receptor ligado com 2,5% glutaraldeído em HBS, pH 7,4, 2 mM MgCl2
- Prepare uma solução de glutaraldeído 2,5% fresco misturando 1 parte de solução de glutaraldeído 25% e 9 partes de HBS, pH 7,4, 2 mM MgCl2.
- Encha cada poço da placa de microtitulação com 50 µ l da solução de glutaraldeído 2,5% em HBS, pH 7,4, 2 mM MgCl2. Incube a placa de microtitulação durante 10 minutos à temperatura ambiente.
10. Lave poços da placa três vezes com 50 µ l/poço de TBS, pH 7,4, 2 mM MgCl2
- Para remover e inativar glutaraldeído em excesso, remover a solução de fixação e encha cada um com 50 µ l de TBS, pH 7,4, 2 mM MgCl2. Em seguida, remova a solução de lavagem.
Nota: Despeje a glutaraldeído contendo solução de fixação da placa de microtitulação em um prato e deite fora a solução de fixação após ele tem sido inativado por um volume igual de TBS, pH 7,4. - Cuide-se que os poços não tornar-se seco. Portanto, não toque a placa de microtitulação sobre um pano de tecido para remover o líquido residual. Use uma pipeta de várias etapas ou uma pipeta multicanal para encher os poços rapidamente.
- Repita este passo de lavar duas vezes.
11. quantificação de ligante do Receptor ligado por ELISA
- Adicione 50 µ l/poço da solução de anticorpo primário em 1% de BSA na TBS, pH 7,4, 2 mM MgCl2. A solução de anticorpo primário é um anti-soro coelho levantadas contra rhodocetin19, dilui-se 1:2,000 em 1% de BSA na TBS, pH 7,4, 2 mM MgCl2.
- Incube a placa por 75-90 min à temperatura ambiente. Lave todas as cavidades da placa três vezes com 50 µ l/poço de TBS, pH 7,4, 2 mM MgCl2. Durante as três etapas de lavagem, rosqueamento da placa de microtitulação sobre um tecido de pano não é necessário.
- Adicione 50 µ l/poço da solução anticorpo secundário em 1% de BSA na TBS, pH 7,4, 2 mM MgCl2. Para este fim, dilua o anticorpo secundário, coelho imunoglobulina-direcionamento cabra anticorpos conjugados com fosfatase alcalina, para 1:2,000 em 1% de BSA na TBS, pH 7,4, 2 mM MgCl2. Incube a placa por 75-90 min à temperatura ambiente.
- Preparar a solução de detecção de AP pela dissolução de um comprimido de 5 mg contendo 4-nitrofenil fosfato dissódico sal hexaidratado (substrato de fosfatase) em 5 mL de tampão de AP (solução de glicina 0,1 M, pH 10.4, contendo 1 mM MgCl2 e 0,2 mM Zn(II)-acetate).
- Lave todas as cavidades da placa três vezes com 50 µ l/poço de TBS, pH 7,4, 2 mM MgCl2, imediatamente antes de executar o próximo passo. Bata a placa do microtiter sobre um pano de tecido após a última etapa de lavagem para remover todos os vestígios de líquido.
- Adicione 50 µ l/poço da AP-detectando solução aos poços da placa de microtitulação. Adicione solução de AP-detectando prontamente a todas as cavidades para iniciar a conversão enzimática tão simultaneamente quanto possível. Portanto, use uma pipeta multicanal.
- Incube a placa à temperatura ambiente até que a solução dos poços com a maior concentração de ligante amarelar.
Nota: O tempo de incubação pode variar entre 5 min e 1 h, dependendo da intensidade do sinal. - Pare a conversão do substrato fosfatase pela adição de 50 µ l/poço de solução de NaOH de 1,5 M. Deixe a placa de pé durante vários minutos assegurar-se de raia livre mistura de ambas as soluções. Para justificar o período de incubação a mesma em todos os poços, usar uma pipeta multicanal e adicionar a 1,5 M NaOH na mesma ordem adicionada aos poços do substrato AP-detectando na etapa 11,8.
- Medir a densidade óptica (OD) em 405 nm de cada poço por um leitor de ELISA.
12. avaliação dos sinais de titulação
- Abra a tabela de dados brutos, os valores de405nm OD, com o Excel. Como esses valores de sinal das curvas de titulação são lidos em linhas, transpor os valores de uma coluna e a etiqueta com as concentrações de ligante adicionado em outra coluna.
- Abra o Graphpad Prism 5 (versão 5.0). Abra um novo arquivo de projeto no menu principal. Escolha o formato XY sob gráfico & novos dados. Escolha a opção Enter e trama um único ponto para cada valor do eixo y.
- Copie as duas colunas, a concentração de ligante adicionado e sinal de valores (valores de OD405nm ) a partir do excel arquivo e colá-los para a folha de dados do GraphPad Prism como X e Y valores, respectivamente.
- Abra o programa de análise sub GraphPad Prism 5 e escolha a opção de regressão não-linear em XY-análise. Escolha equação definida pelo usuário e pressione o botão novo para criar uma nova equação.
- Digite a equação da curva de titulação na forma: Y =(Smax-Smin)*((X+R+K)-sqrt((X+R+K)^2-4*R*X)) /(2*R) + Smin + B * X em folha modelo recém inaugurado, com Y sendo o valor de sinal S, X sendo a concentração de ligante adicionado L , R, sendo a concentração de imobilizado do receptor, K é a constante de dissociação, e B, sendo o plano de fundo da inclinação. Definir as restrições adequadas, tais como K > 0 e R > 0.
Nota: Esta equação é a mesma equação da equação 9 , de uma forma diferente. - Analise os valores da folha de dados escolhendo a equação definida pelo usuário, que foi recentemente criada. Abra a tabela com os valores calculados de aproximação (K, R,t, Smáximo, Smine B) que são mostradas na seção resultado no lado esquerdo da tela software.
Nota: O software determina os 5 parâmetros por encaixe iterativo da regressão não-linear, somente se a curva de titulação consiste de pontos de dados de pelo menos 5. - Avaliar os parâmetros K, R,t, Smáx, Smine B para cada grupo de curvas de titulação, estatisticamente e correlacionar os parâmetros com a característica específica do grupo (ligante mutado ou quimicamente modificados ou receptor).
Access restricted. Please log in or start a trial to view this content.
Resultados
Após o ELISA foi desenvolvido, a cor amarela do substrato convertidos da fosfatase alcalina, pará- nitrophenolate, indica que a quantidade de ligante rhodocetin limite diminui com diminuindo as concentrações de rhodocetin adicionado de colunas de 1 a 11 (Figura 1). Os poços incolores nos poços rhodocetin-livre na coluna 12 mostram um sinal de baixo fundo.
Fotométrica quantificação ...
Access restricted. Please log in or start a trial to view this content.
Discussão
A titulação de ELISA é um sistema versátil de teste para determinar a dissociação de uma interação receptor-ligante. Como a titulação ELISA contorna a necessidade de separar ligands livre e limite efetivamente e analisar as suas concentrações quantitativamente, substancialmente mais estudos e publicações têm empregado titulação ELISAs em vez de gravação curvas de ligação . Além disso, titulação ELISAs são fáceis de realizar e requerem quantidades razoavelmente baixas do receptor e ligante. Para ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
O autor não tem nada para divulgar.
Agradecimentos
O protocolo e o algoritmo foram desenvolvidos dentro de um projeto financiado pela Deutsche Forschungsgemeinschaft (DFG grant A09 SFB1009 e EB177/13-1). O autor agradece Barbara Schedding e Felix Schmalbein apoio técnico e Dr. Niland para ler criticamente o manuscrito.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| TRIS | neoFrox | 1125KG001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| NaCl | Applichem | 1,316,591,214 | |
| MgCl2 | Merck | 172571 | |
| integrin a2A, wild-type and mutant, recombinant | isolated in author's lab | ||
| NiNTA superflow column | Qiagen, Germany | 30821 | |
| Coomassie-Brilliant Blue R250 | Serva | 35050 | |
| bicinchoninic acid assay (BCA), protein concentration determination kit | Fisher Scientific | 23225 | |
| bovine serum albumine (BSA), fraction V | Applichem | A1391 | |
| 25 % solution of glutaraldehyde | Merck | 354400 | |
| anti-rabbit immunglobulin-antibodies from goat, conjugated with alkaline phosphatase | Sigma-Aldrich | A9919 | |
| Glycine | Applichem | A1377 | |
| Zn(II)-acetate | Applichem | A4324 | |
| NaOH | Applichem | A1551 | |
| Alkaline phosphatase substrate tablet (5 mg) | Sigma-Aldrich | S0942 | |
| Costar half-area microtiter plate | Thermo Scientific | Corning 3690 | |
| micro reaction tubes | Eppendorf | 30120086 | |
| Microplate ELISA reader | BioTek | Synergy HT |
Referências
- Klotz, I. M. The application of the law of mass action to binding by proteins; interactions with calcium. Arch Biochem. 9, 109-117 (1946).
- Klotz, I. M. Ligand-receptor complexes: origin and development of the concept. J Biol Chem. 279 (1), 1-12 (2004).
- Bisswanger, H. Ch. 1: Multiple Equilibria, Principles and Derivations. Enzyme Kinetics: Principles and Methods. , Wiley VCH Verlag GmbH. 1-26 (2017).
- Shimura, K., Kasai, K. Affinity gel titration: quantitative analysis of the binding equilibrium between immobilized protein and free ligand by a continuous titration procedure. Anal Biochem. 149 (2), 369-378 (1985).
- Gong, M., Nikcevic, I., Wehmeyer, K. R., Limbach, P. A., Heineman, W. R. Protein-aptamer binding studies using microchip affinity capillary electrophoresis. Electrophoresis. 29 (7), 1415-1422 (2008).
- Hanes, M. S., Ratcliff, K., Marqusee, S., Handel, T. M. Protein-protein binding affinities by pulse proteolysis: application to TEM-1/BLIP protein complexes. Protein Sci. 19 (10), 1996-2000 (2010).
- Latour, R. A. The Langmuir isotherm: a commonly applied but misleading approach for the analysis of protein adsorption behavior. J Biomed Mater Res A. 103 (3), 949-958 (2015).
- Stockell, A. The binding of diphosphopyridine nucleotide by yeast glyceraldehyde-3-phosphate dehydrogenase. J Biol Chem. 234 (5), 1286-1292 (1959).
- Heyn, M. P., Weischet, W. O. Circular dichroism and fluorescence studies on the binding of ligands to the α subunit of tryptophan synthase. Biochem. 14 (13), 2962-2968 (1975).
- Campbell, I. D., Humphries, M. J. Integrin structure, activation, and interactions. Cold Spring Harb Perspect Biol. 3 (3), (2011).
- Zeltz, C., Gullberg, D. The integrin-collagen connection--a glue for tissue repair? J Cell Sci. 129 (4), 653-664 (2016).
- Kanchanawong, P., et al. Nanoscale architecture of integrin-based cell adhesions. Nature. 468 (7323), 580-584 (2010).
- Luo, B. H., Carman, C. V., Springer, T. A. Structural basis of integrin regulation and signaling. Annu Rev Immunol. 25, 619-647 (2007).
- Luo, B. H., Springer, T. A. Integrin structures and conformational signaling. Curr Opin Cell Biol. 18 (5), 579-586 (2006).
- Ricard-Blum, S. The collagen family. Cold Spring Harb Perspect Biol. 3 (1), a004978(2011).
- Eble, J. A., Tuckwell, D. S. The α2β1 integrin inhibitor rhodocetin binds to the A-domain of the integrin α2 subunit proximal to the collagen-binding site. Biochem J. 376 (Pt 1), 77-85 (2003).
- Eble, J. A., et al. The α2β1 integrin-specific antagonist rhodocetin is a cruciform, heterotetrameric molecule. FASEB J. 23 (9), 2917-2927 (2009).
- Eble, J. A., et al. Dramatic and concerted conformational changes enable rhodocetin to block α2β1 integrin selectively. PLoS Biol. 15 (7), e2001492(2017).
- Bracht, T., Figueiredo de Rezende, F., Stetefeld, J., Sorokin, L. M., Eble, J. A. Monoclonal antibodies reveal the alteration of the rhodocetin structure upon α2β1 integrin binding. Biochem J. 440 (1), 1-11 (2011).
- Harlow, E., Lane, D. Chapter 5: Immunizations. Antibodies, a laboratory manual. 5, Cold Spring Harbor Laboratory. 53-138 (1988).
- Lu, D. Analyzing interactions between SSB and proteins by the use of fluorescence anisotropy. Methods Mol Biol. 922, 155-159 (2012).
- Fielding, L. NMR methods for the determination of protein-ligand dissociation constants. Curr Top Med Chem. 3 (1), 39-53 (2003).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr Opin Struct Biol. 11 (5), 560-566 (2001).
- McDonnell, J. M. Surface plasmon resonance: towards an understanding of the mechanisms of biological molecular recognition. Curr Opin Chem Biol. 5 (5), 572-577 (2001).
- Rich, R. L., Myszka, D. G. Advances in surface plasmon resonance biosensor analysis. Curr Opin Biotechnol. 11 (1), 54-61 (2000).
- Pesquero, N. C., et al. Real-time monitoring and kinetic parameter estimation of the affinity interaction of jArtinM and rArtinM with peroxidase glycoprotein by the electrogravimetric technique. Biosens Bioelectron. 26 (1), 36-42 (2010).
- Scheuermann, T. H., Padrick, S. B., Gardner, K. H., Brautigam, C. A. On the acquisition and analysis of microscale thermophoresis data. Anal Biochem. 496, 79-93 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados