Method Article
Visualisierung von genetischen Varianten, kurze Ziele und Punktmutationen in Zusammenhang mit morphologischen Gewebe mit einem RNA In Situ Hybridisierung Assay
In diesem Artikel
Zusammenfassung
Hier beschreiben wir einen in Situ Hybridisierung Test sensitiver und spezifischer Nachweis von Sequenzen so kurz wie 50 Nukleotide mit Einzel-Nukleotid-Auflösung auf die einzelne Zelle Ebene ermöglicht. Assays, die manuell oder automatisch ausgeführt werden kann, kann Visualisierung von Splice-Varianten, kurze Sequenzen und Mutationen im Zusammenhang mit Gewebe ermöglichen.
Zusammenfassung
Da präzisionsmedizin stark abhängig von der genauen Nachweis von Biomarkern ist, gibt es ein zunehmender Bedarf an standardisierten und robuste Technologien, die RNA Biomarker in Situ in klinischen Proben zu messen. Während die Grind-Bind-Assays wie RNAseq und quantitative RT-PCR ermöglichen hochsensiblen Gen Ausdruck Messungen, sie erfordern auch RNA-Extraktion und verhindern somit wertvolle Expressionsanalyse morphologische Gewebe im Rahmen. Die in Situ Hybridisierung (ISH)-Assay hier beschriebenen erkennt RNA Zielsequenzen 50 Nukleotide auf Einzel-Nukleotid-Auflösung und die einzelligen Ebene möglichst kurz. Dieser Test ist eine Ergänzung zu den bisher entwickelten kommerziellen Assay und ermöglicht die Sensitivität und Spezifität in Situ Detektion der Splice-Varianten, kurze Ziele und Punktmutationen im Gewebe. In diesem Protokoll wurden Sonden entwickelt, um einzigartige Exon Kreuzungen für zwei klinisch relevanten Splice-Varianten, EGFRvIII und METΔ14 Ziel. Die Erkennung von kurzen Zielsequenzen zeigte sich durch den spezifischen Nachweis des CDR3 Sequenzen von T-Zell-Rezeptoren-α und β in der Jurkat T-Zell-Linie. Auch ist das Dienstprogramm des ISH Assays für die Unterscheidung von RNA-Zielsequenzen bei Einzel-Nukleotid-Auflösung (Punktmutationen) durch die Visualisierung der EGFR L858R und KRAS G12A Einzel-Nukleotid-Variationen in Zelllinien mit automatisierten Färbung Plattformen. Zusammenfassend zeigt das Protokoll einen spezialisierten RNA ISH-Assay, der es die Erkennung von Splice-Varianten, kurze Sequenzen und Mutationen in Situ für manuelle und automatische freigesprochen ermöglicht.
Einleitung
Transkriptomischen von Hochdurchsatz-Technologien wie Microarrays und Next-Gen-RNA-Sequenzierung (RNAseq) haben die Entdeckung der RNA-Biomarker mit diagnostischen, prognostischen und prädiktiven klinischen Wert für verschiedene Krankheiten einschließlich exponentiell verbessert. Krebs1,2. Um den Einsatz dieser Biomarker in der präzisionsmedizin zu fördern, gibt es ein hohen Bedarf an standardisierten und robuste Technologien, die RNA Biomarker die Gewebe im Rahmen von klinischen Proben messen kann. Während allgemein etabliert, Grind und Bind-Assays wie RNAseq und quantitative RT-PCR ermöglichen hochsensiblen Gen Ausdruck Messungen, die erforderlichen Gewebe Homogenisierung und RNA Isolierung bedeuten den Verlust der in Vivo Zelltyp Spezifität und morphologische Informationen3. Konventionelle in Situ RNA Erkennung Methoden fehlen die Sensitivität und Spezifität erforderlich, um seltene oder niedrigen exprimierenden RNA Biomarker innerhalb der Gewebe Kontext4zuverlässig messen.
Eine kommerzielle in Situ -Hybridisierung (ISH)-Assay (zB., die RNAscope-Assay) ist eine Technologie, die diese in Angriff genommen hat Herausforderungen und ermöglicht auch die hoch empfindliche und spezifische Visualisierung der einzelnen RNA-Moleküle, die größer als 300 Nukleotide im Gewebe morphologischen Kontext. Diese Art von Test verwendet eine einzigartige Oligonukleotid Sonde Design von ca. 6 – 20 Doppel-Z-Sonde Paaren kombiniert mit einer erweiterten Hybridisierung-basierte Signal Verstärkung5.
Diese Studie beschreibt einen speziellen RNA-ISH Assay, BaseScope, ergänzend zu der zuvor entworfenen kommerzielle Technologie, die RNA-Zielsequenzen so kurz wie 50 Nukleotide bei Einzel-Nukleotid-Auflösung erkennen kann. Dieser Test befasst sich mit der komplizierten Komplexität des transkriptoms und gilt für die präzise Erkennung von Exon Kreuzungen, kurze Zielsequenzen und Punktmutationen im Gewebe Kontext (Tabelle 1) mit so wenig wie ein Doppel-Z-Sonde. Dieser Bericht zeigt das komplette Test-Protokoll und seine Verwendung bei der Erkennung von Splice-Varianten, CDR3-Sequenzen für T-Zell-Rezeptor-Klone, und Einzel-Nukleotid-Mutationen im FFPE Zell-Linien und Tumorgewebe.
Protokoll
In dieser Studie verwendeten menschlichen tumorproben wurden deidentifiziert und erwarb aus kommerziellen Quellen entsprechend den lokalen ethischen Richtlinien für Forschung am Menschen.
1. Probe, Ausrüstung und Reagenz Vorbereitung
-
Proteinkinase Probenvorbereitung
- Gewebe
- Unmittelbar nach der Dissektion, das Gewebe (in Blöcken von 3 – 4 mm Dicke geschnitten) in 10 % Neutral gepufferte Formalin (NBF) für 16-32 h bei Raumtemperatur (RT) befestigen.
Hinweis: Fixierzeit variiert je nach Gewebetyp und Größe. - Waschen Sie die Probe mit 1 x Phosphat-gepufferte Kochsalzlösung (PBS) und entwässern mit Hilfe einer standard Ethanol (EtOH) Reihe (70 % EtOH für 30 – 60 min, 80 % EtOH für 30 – 60 min, 90 % EtOH für 30 – 60 min, 95 % EtOH für 30 – 60 min, 3 x 100 % EtOH 30 – 60 min) gefolgt von Xylol.
- Betten Sie die Probe in Paraffin mit Standardverfahren6 ein und trimmen Sie der Paraffin-Blöcke je nach Bedarf entfernen überschüssiges Paraffin.
Hinweis: Blockgröße ist abhängig von des Gewebe-Stichprobenumfangs, aber eine typische Größe ist 0,75 x 0,75 Zoll2 oder kleiner.
- Unmittelbar nach der Dissektion, das Gewebe (in Blöcken von 3 – 4 mm Dicke geschnitten) in 10 % Neutral gepufferte Formalin (NBF) für 16-32 h bei Raumtemperatur (RT) befestigen.
- Zell-Linien
- Sammeln und pellet-Zellen nach den empfohlenen Methoden für die spezifische Zell-Linie.
- Zellen in 10 % Formaldehyd bei RT für 24 h auf ein Rotator zu beheben.
- Bereiten Sie Zellen als ein Pellet in vorgewärmte Verarbeitung Gel (zB., Histogel). Ermöglichen Sie das Pellet, indem das Gel-Zell-Pellet auf ein Stück Parafilm auf Eis zu festigen, und lassen Sie es für 2 – 3 min. Submerge Gel-Zell-Pellet mit 1 X PBS-Puffer.
- Entwässern Sie und Betten Sie der Zelle Pellets wie in Schritt 1.1.1 beschrieben ein.
- Abschnitt Vorbereitung
- Die eingebetteten Gewebe/Zellen in 5 ± 1 μm Abschnitte mit einem Mikrotom geschnitten, montieren Sie die Abschnitte an elektrostatisch anhaftenden Objektträger und an der Luft trocknen sie über Nacht bei RT
Hinweis: Die Folien können bei RT unter Austrocknung bis zu 3 Monate lang gespeichert werden. - Legen Sie die Folien in einem Dia-Rack und Backen Sie montiert Gewebe Folien in einem zirkulierenden Luft Ofen bei 60 ° C für 1 h vor der Durchführung des Tests.
Hinweis: Verwenden Sie die Folien sofort oder speichern sie bei RT mit Trockenmittel für bis zu 1 Woche. Längerer Lagerung kann RNA-Abbau führen.
- Die eingebetteten Gewebe/Zellen in 5 ± 1 μm Abschnitte mit einem Mikrotom geschnitten, montieren Sie die Abschnitte an elektrostatisch anhaftenden Objektträger und an der Luft trocknen sie über Nacht bei RT
- Gewebe
-
Vorbereitung der Ausrüstung
- Legen Sie die Hybridisierung Ofen bis 40 ° C. Gründlich nass befeuchtenden Papier entfernen Rückstände dH2O. Legen Sie das Papier in das Fach ein Feuchtigkeit Kontrolle und setzen Sie die Schale in den Ofen Hybridisierung, für mindestens 30 min vor Gebrauch prewarm.
-
Reagenz-Vorbereitung
- Füllen Sie zwei clearing Agent Gerichte mit 200 mL Xylol und zwei Färbung Tellern mit 200 mL 100 % EtOH, die für 4.wenn der Abschnitte verwendet werden.
- Bereiten Sie 200 mL handelsübliche 1 X Ziel abrufen Reagenz durch Hinzufügen von 180 mL dH2O bis 20 mL 10 X Ziel abrufen Reagenz vor. Legen Sie zwei Objekthalter in einem Dampfer. Füllen Sie einen Diahalter mit 200 mL 1 x Ziel abrufen Reagenz und füllen Sie die anderen Diahalter mit 200 mL dH2O. Hitze beide Lösungen zum Kochen mit dem Steamer.
- Warmen 50 X Waschpuffer auf 40 ° C für 10 – 20 min. Vorbereitung 3 L 1 X Waschpuffer durch Verdünnung 60 mL vorgewärmt 50 X Waschpuffer mit 2,94 L dH2O.
- Bereiten Sie in einer Dampfhaube 50 % Gill Hämatoxylin gegenfärbung Lösung durch Zugabe von 100 mL Gills Hämatoxylin, 100 mL von dH2O. Bereiten Sie in der Dunstabzugshaube Bläuen Reagenz (0,02 % (w/V) Ammoniak Wasser) durch Zugabe von 1,43 mL 28 – 30 % Ammonium Hydroxid, 250 mL von dH2O.
- Prewarm Ziel-Sonden bei 40 ° C für 10 min vor der Sonde Hybridisierung und bringen die verstärkenden Reagenzien (AMP 0 – 6), RT
(2) RNA In Situ Hybridisierung Assay
-
4.wenn und Austrocknung
- Nach dem Backen wie im Schritt 1.1.3.2 beschrieben (optional Haltepunkt 1), deparaffinize die Abschnitte in Xylol für 5 min. unter Rühren. Deparaffinize wieder in frischem Xylol für 5 min. dehydrieren in 100 % EtOH für 2 min mit Agitation und Wiederholung wieder in frische 100 % EtOH 2 min. lang.
- Luft trocknen die Folien für 5 min bei 60 ° C in einem zirkulierenden Luft Ofen oder bei RT Trocknen bis sie vollständig sind (optional Haltepunkt 2).
-
Probe Vorbehandlungen
- Inkubieren Sie die Abschnitte mit ~ 4 Tropfen von Ready-to-Use Wasserstoffperoxid für 10 min bei RT die endogenen Peroxidase-Aktivität zu stillen. Dekantieren Sie die Lösung von den Folien zu und spülen Sie sie zweimal mit dH2O.
- Inkubieren Sie die Abschnitte mit 200 mL das Ziel abrufen Reagenz für 15-30 min bei 100 ° C in einem Dampfer.
Hinweis: Inkubationszeit variieren je nach Gewebetyp. In diesem Protokoll wurde Ziel abrufen für 15 min für tumorproben und Zelle Pellets durchgeführt. - Die Lösung von den Folien zu Dekantieren, spülen Sie zweimal mit dH2O, Tauchen Sie ein in 100 % EtOH für 3 min und trocknen die Folien bei 60 ° C in einem zirkulierenden Luft Ofen oder bei RT bis Sie vollständig trocknen.
- Zeichnen Sie eine hydrophobe Barriere rund um den Bereich mit einem hydrophoben Stift, ca. 0,75 x 0,75 Zoll2.
Hinweis: Es wird nicht empfohlen, ein kleineres Hindernis zu ziehen. Für größere Teile müssen eine größere Barriere gezogen werden. - Lassen Sie die Barriere für 1 min vollständig trocknen, oder lassen Sie über Nacht bei RT (optional Haltepunkt 3).
- Legen Sie die Folien in einen Diahalter und legen Sie den Diahalter in der Feuchtigkeit Kontrolle Fach. ~ 4 Tropfen Protease III zu jeder Folie und inkubieren sie 15-30 min bei 40 ° C im Backofen Hybridisierung Proteinverdauung.
Hinweis: Inkubationszeit variieren je nach Gewebe. In diesem Protokoll wurde Protease Verdauung für 30 min für tumorproben und 15 min für Zelle Pellets durchgeführt. - Dekantieren Sie die Lösung von den Folien zu und spülen Sie sie zweimal mit dH2O.
-
Sonde Hybridisierung
- ~ 4 Tropfen der entsprechenden Ready-to-Use-Sonde Lösung um den gesamten Bereich abzudecken. Wenn größere Teile zu verwenden, fügen Sie ca. 5 – 6 Tropfen.
- Hybridisieren Sie die Sonden für 2 h bei 40 ° C im Ofen Hybridisierung. Dekantieren Sie die Lösung von den Folien zu und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie in diesem Schritt des Waschvorgangs.
-
Signalverstärkung
- Inkubieren Sie die Abschnitte mit ~ 4 Tropfen des AMP 0 pro Folie bei 40 ° C im Ofen für 30 min. Dekantieren die Lösung Hybridisierung und waschen Sie die Folien in 200 mL 1 X Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie in diesem Schritt des Waschvorgangs.
- Inkubieren Sie die Abschnitte mit ~ 4 Tropfen des AMP 1 pro Folie bei 40 ° C im Ofen für 15 min. Dekantieren die Lösung Hybridisierung und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie den Waschvorgang.
- Inkubieren Sie die Abschnitte mit ~ 4 Tropfen des AMP 2 pro Folie bei 40 ° C im Ofen für 30 min. Dekantieren die Lösung Hybridisierung und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie den Waschvorgang.
- Inkubieren Sie die Abschnitte mit ~ 4 Tropfen des AMP 3 pro Folie bei 40 ° C im Ofen für 30 min. Dekantieren die Lösung Hybridisierung und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie den Waschvorgang.
- Inkubieren Sie die Abschnitte mit ~ 4 Tropfen des AMP 4 pro Folie bei 40 ° C im Ofen für 15 min. Dekantieren die Lösung Hybridisierung und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie den Waschvorgang.
- Brüten Sie die Abschnitte mit ~ 4 Tropfen des AMP 5 pro Folie bei RT für 30 min. Dekantieren die Lösung und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie den Waschvorgang.
- Brüten Sie die Abschnitte mit ~ 4 Tropfen des AMP 6 pro Folie bei RT für 15 min. Dekantieren die Lösung und waschen Sie die Folien in 200 mL 1 x Waschpuffer für 2 min bei RT mit gelegentlichen Agitation. Wiederholen Sie den Waschvorgang.
-
Signalerkennung
- Vorbereiten des schnell rote Farbstoffs Lösung arbeiten. Eine Folie mit einer 0,75 x 0,75 Zoll2 Barriere hinzufügen 2 μL des Farbstoffes schnell rot-B 120 μL schnell rot-A in eine Röhre und gut mischen.
Hinweis: Abhängig von der Größe der hydrophobe Barriere und die Anzahl der Folien variiert Datenmengen schnell rot funktionierende Lösung. - Die überschüssige Flüssigkeit aus den Folien zu dekantieren und Hinzufügen des schnell rote Farbstoffs Lösung zu den Folien arbeiten. Inkubieren Sie die Folien für 10 min bei RT in das Fach mit einer Abdeckung, um Lichteinfall zu vermeiden.
Hinweis: Verwenden Sie die Fast Red arbeiten Lösung innerhalb von 5 min Vorbereitung, und setzen Sie es nicht direktem Sonnenlicht oder UV-Licht. - Dekantieren Sie Fast Red-Lösung und spülen Sie die Folien zweimal mit Leitungswasser. Legen Sie die Folien wieder in einem Dia-Rack.
- Vorbereiten des schnell rote Farbstoffs Lösung arbeiten. Eine Folie mit einer 0,75 x 0,75 Zoll2 Barriere hinzufügen 2 μL des Farbstoffes schnell rot-B 120 μL schnell rot-A in eine Röhre und gut mischen.
-
Gegenfärbung
- Counterstain Gewebeschnitte mit 50 % Gill Hämatoxylin Lösung für 2 min bei RT. Wash Folien mit Leitungswasser und dies mehrmals wiederholen, bis die Folien liegen auf der Hand, während die Abschnitte lila bleiben. Tauchen Sie die Folien in 0,02 % Ammoniak Wasser zum Bläuen (dip 2 – 3 mal). Ersetzen Sie das Ammoniak-Wasser mit Leitungswasser und waschen Sie der Objektträger 3 – 5 Mal.
-
Schieben Sie Montage
- Trocken trocknen Sie die Folien in einem zirkulierenden Luft Ofen bei 60 ° C für 15 min oder bei RT bis Sie vollständig. Platzieren Sie 1 – 2 Tropfen Reagenz Montage auf jeder Folie und Ort Deckgläsern, über jeden Abschnitt. Überfüllung von Luftblasen zu vermeiden. Die Folien für mindestens 5 min der Luft trocknen lassen.
Hinweis: Das Substrat schnell rot ist Alkohol-und Kleinschreibung. Folien in Alkohol nicht austrocknen.
- Trocken trocknen Sie die Folien in einem zirkulierenden Luft Ofen bei 60 ° C für 15 min oder bei RT bis Sie vollständig. Platzieren Sie 1 – 2 Tropfen Reagenz Montage auf jeder Folie und Ort Deckgläsern, über jeden Abschnitt. Überfüllung von Luftblasen zu vermeiden. Die Folien für mindestens 5 min der Luft trocknen lassen.
-
Visualisierung
- Die Folien unter dem standard Hellfeld-Mikroskop zu beobachten.
Ergebnisse
Die in Situ Hybridisierung Assay Workflow:
Der Workflow ist in Abbildung 1 dargestellt und besteht aus vier Teilen: Permeabilisierung der Zellen oder Gewebe mit Ziel abrufen und Protease Lösungen, Hybridisierung der Sonden, die Ziel-RNA, signal-Verstärkung und Visualisierung des Signals. Das Signal kann auch mit Softwaresystemen für digitale Bildverarbeitung quantifiziert werden oder in einer semi-quantitative Weise anhand der Anzahl der Punkte pro Zelle. Manuelle Verfahren Sie wie in Abbildung 2 wurde auch in kommerziellen Auto Färbung Systemen vollständig automatisiert.
Repräsentative Färbung für Exon-Junction-Erkennung (EGFRvIII spleißvariante): EGFRvIII ist eine Variante des epidermalen Wachstumsfaktor-Rezeptors, das durch eine in-Frame genomische Löschung der Exons 2 bis 7 entsteht, führt zu konstitutiv aktiven Onkogenen Signalisierung7 . Der Test wurde verwendet, um EGFRvIII Status im tumorproben FFPE Glioblastom (GBM) zu identifizieren. Einzel-Doppel-Z-Probes wurden entwickelt, um der Exon-Kreuzungen erstrecken, um entweder WT zu erkennen Mutant oder beide Protokolle (Abb. 3A). WT EGFR-Sonden umfassen die Kreuzungen von Exons 1 und 2 (E1/E2) oder Exons 7 und 8 (E7/E8), während der EGFRvIII-spezifischen Sonden die Kreuzung von Exons 1 und 8 (E1/E8) erstrecken. Eine gemeinsame Sonde, die Kreuzung der Exons 8 und 9 (E8/E9) erstreckt wurde auch verwendet, um insgesamt EGFR (WT und EGFRvIII Transkripte) zu erkennen. Die Sonden wurden dann zur EGFR-Status in FFPE GBM Proben zu bestimmen. Die zwei Repräsentative Beispiele in Abb. 3 b stammen aus einer größeren Studie. EGFR-Status wurde durch eine unabhängige Methode, RT-PCR bestätigt. Beide Sonden WT erkannt Signal in beiden Proben, darauf hinweist, dass die beiden Proben WT EGFR (Abb. 3 b) zum Ausdruck bringen. Die mutierte Sonde ergab jedoch nur Signalerkennung im EGFRvIII + Beispiel bestätigt, dass dieses Beispiel zwar positiv für die EGFRvIII-Variante (Abb. 3 b, linken Panels). Im Gegensatz dazu hat die mutierte Sonde Signal in der EGFRvIII-Probe (Abb. 3 b, richtigen Platten) nicht erkannt. Zusammengenommen zeigen diese Ergebnisse, dass die Exon-Junction-Assay EGFRvIII Status im GBM FFPE tumorproben identifizieren kann.
Vertreter für kurze Ziele Färbung:
Die CDR3 oder ergänzende bestimmenden Region 3, ist eine sehr Variable Domäne in T-Zell-Rezeptoren. Die CDR3-Sequenz sind in der Regel recht kurz; Beispielsweise sind die CDR3 α und β-Sequenzen aus der Jurkat T-Zell-Linie 51 und 48 Nukleotide in der Länge, bzw. (Abb. 4A). Spezifischen CDR-Sequenzen in Jurkat Zellen, antisense Sonden für CDR3 ausgedrückt zu identifizieren α und β, die in der Jurkat T-Zell-Linie ausgedrückt sind entstanden, neben Sinn Sonden für CDR3 α und β, dienen als negative Kontrolle Sonden. Die Sonden wurden dann in FFPE vorbereitet Jurkat-Zellen mit dem Assay getestet. Robuste Färbung beobachtet wurde mit Anti-Sense-Sonden für beide CDR3 α und β in den Jurkat-Zellen, während Sinn-Sonden entdeckt wenig bis gar kein Signal (Abbildung 4 b). Diese Ergebnisse zeigen die Fähigkeit des kurzen Ziel-Assays, zwischen höchst Variable aber kurze CDR-Sequenzen für T-Zell-Rezeptor-Klone zu unterscheiden.
Vertreter für Punktmutation L858R EGFR Färbung:
Punktmutation Sonden wurden entwickelt, um Einzel-Nukleotid-Variationen und kleine Einfügungen oder Löschungen (INDELs) im Zusammenhang mit Tumor zu erkennen. Abbildung 5A zeigt die Fähigkeit zur in Situ Detektion von Punktmutation L858R EGFR (2573T > G). Zwei Sonden wurden entwickelt: die Feststellung, die L858R mutierten EGFR-Sequenz, und eine andere, die EGFR L858 WT-Sequenz zu erkennen. Beide Sonden wurden in zwei FFPE vorbereitet Zelllinien getestet: H2229, das nur EGFR L858 WT ausdrückt; und H1975, die heterozygot für die Mutation L858R EGFR ist. Die L858R mutierte Sonde erkannt Signal nur in der H1975-Zell-Linie, aber nicht in die H2229-Zell-Linie. Jedoch entdeckt die WT-Sonde Signal in beiden Zelllinien. In ähnlicher Weise in Situ Detektion von Punktmutation KRAS G12A visualisiert Abbildung 5 b (35 G > C). Zwei Sonden wurden entwickelt, um die KRAS G12A MT und KRAS G12 WT Sequenzen zu erkennen und dann auf die HuT78-Zell-Linie (die nur KRAS G12 WT drückt) und die SW116-Zell-Linie (die heterozygot für die Mutation des KRAS G12A) getestet. Während die KRAS G12 WT-Sonde in beiden Zelllinien Signal erkannt, erkannt KRAS G12A Sonde nur Signal in die SW116-Zell-Linie. Zusammengenommen zeigen diese Daten die technische Fähigkeiten des Assays Punktmutation bei der Aufdeckung von Einzel-Nukleotid-Polymorphismen im Zusammenhang mit Zellen und Gewebe.
Vertreter für die automatisierte in Situ -Assay Färbung:
Automatisierte Tests ermöglichen eine größere Anzahl von Proben mehr zuverlässig ausgeführt werden Inter Benutzer Variabilität und Hands-on-Zeit zu minimieren und erzeugen konstant reproduzierbare Ergebnisse. Daher wurde eine automatisierte Version des Assays entwickelt. Um automatisierte Färbung mit dieser Assay zu demonstrieren, wurde Erkennung von spleißvariante METΔ14 untersucht. Diese Variante ist das Ergebnis von Exon 14 im MET-gen während der Pre-mRNA Spleißen, führt zu konstitutiven Aktivierung und Onkogenen Transformation der MET Rezeptor8,9übersprungen. Um speziell die METΔ14 Variante zu erkennen, wurden zwei Exon-Junction-Sonden entwickelt: eine, die über der Kreuzung von Exons 13 und 15 (E13/E15), die METΔ14 Variante Abschrift zu erkennen, und eine andere, die Kreuzung der Exons 14 und 15 (E14/E15) zur Erkennung der WT MET erstreckt Transkript (Abb. 6A). Beide Sonden wurden dann in 2 FFPE vorbereitet Zelllinien getestet: H596, die die METΔ14-Variante zum Ausdruck bringt, und A549, die das WT traf gen zum Ausdruck bringt. Beide Sonden zeigten sich gegenseitig ausschließende Expressionsmuster E13/E15-Sonde nur Erkennung Signal in den H596 Zellen und die E14/E15-Sonde nur Erkennung Signal in den A549 Zellen (Zahlen 6 b und 6 C). Zu guter Letzt zeigte die Sonde für DapB kein Signal, kein Hintergrundsignal (Zahlen 6 b und 6 C) angibt. Insgesamt zeigt diese Daten spezifische Erkennung des MET Variante METΔ14 in Situ unter Verwendung der automatisierten BaseScope Assay.
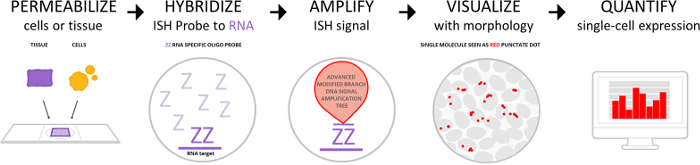
Abbildung 1 : Der Assay-Workflow. Der Workflow besteht aus 4 großen Schritten: Vorbehandlung, Zellen oder Gewebe, Sonde Hybridisierung, Ziel-RNA, permeabilize signal-Verstärkung und signal-Erkennung durch Visualisierung unter Hellfeld oder fluoreszierende Mikroskop. Einzelne Punkte können mit Hilfe eines digitalen Bildes Analyseplattform quantifiziert werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
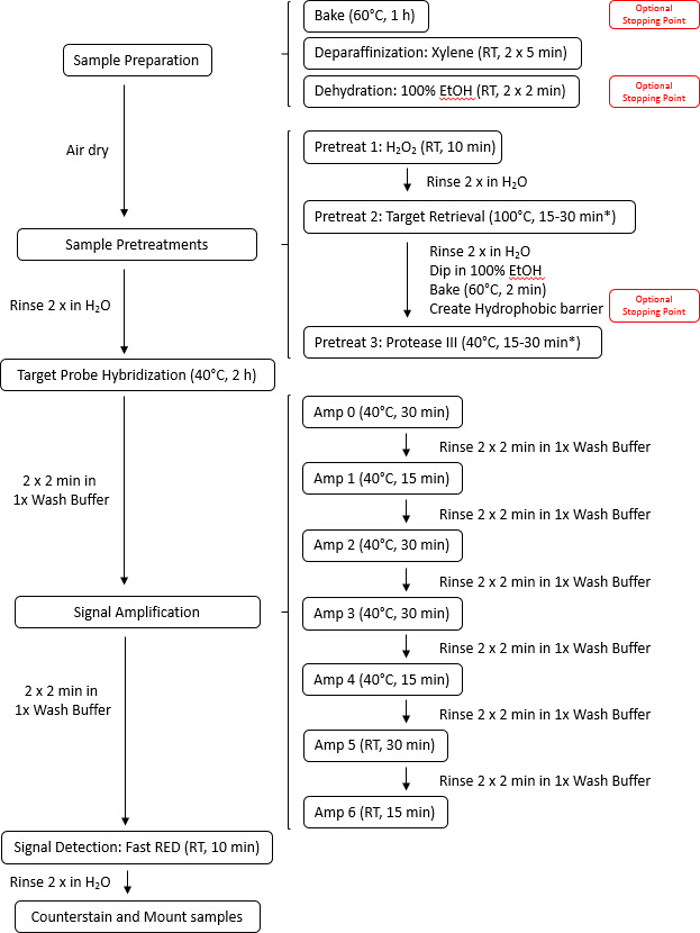
Abbildung 2 : Abbildung des Protokolls manuelle Assay. Der gesamte Test kann in 9 h. Vorbehandlung erfolgen, die Zeiten je nach Gewebetyp, variieren so ist es ratsam, Anhang A in der Bedienungsanleitung für Gewebe Vorbehandlung Empfehlungen bezüglich Inkubationszeit zu konsultieren. Bitte klicken Sie hier für eine größere Version dieser Figur.
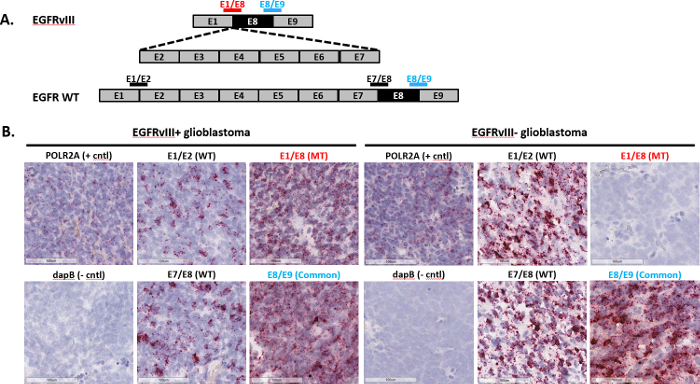
Abbildung 3 : Repräsentative Bilder der Exon-Junction-Erkennung. (A) siehe hier sind die Exon-Organisation für EGFR WT und EGFRvIII Transkripte und eine schematische Darstellung der Doppel-Z Exon Kreuzung Sonden, die die Kreuzung um EGFR WT oder EGFRvIII erkennen zu überspannen. (B) das Exon-Junction-Assay wurde auf zwei FFPE Glioblastom Proben mit den Sonden aufgeführt in (A), sowie eine positive Kontrolle Sonde, POLR2A und Negativkontrolle Sonde, DapB durchgeführt. Bitte klicken Sie hier für eine größere Version dieser Figur.
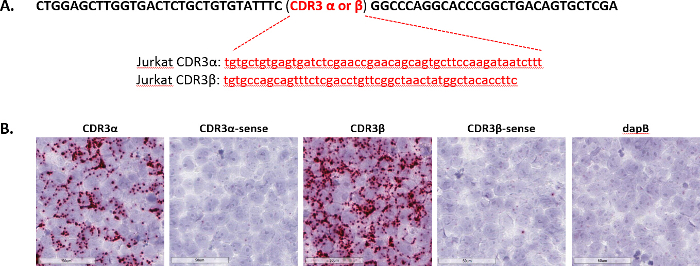
Abbildung 4 : Repräsentative Bilder von kurzen Ziel-Sequenz Erkennung. (A) siehe hier sind CDR3-Sequenzen in Jurkat Zellen. Die Sequenz in schwarz ist eine begleitende gemeinsame Sequenz und die Sequenz in rot ist einzigartig, entweder CDR3α oder CDR3β. Doppel-Z kurze Einzelziel Sequenz Sonden wurden gegen diese Sequenzen entwickelt. (B) die kurzen Ziel-Assay wurde auf Jurkat Zellen als eine Proteinkinase Zelle Pellet mit Anti-Sinn oder Sonden für die Sequenzen in (A) vorbereitet, und DapB diente als Negativkontrolle. Bitte klicken Sie hier für eine größere Version dieser Figur.
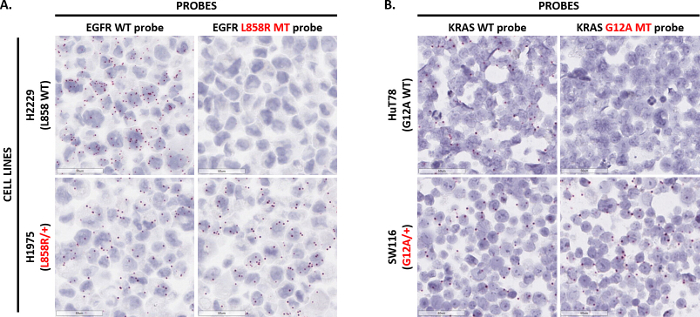
Abbildung 5 : Repräsentative Bilder Punktmutation Nachweisgrenze. (A) der Test erfolgte am H2229 Zellen (homozygot für EGFR L858) und H1975 (heterozygot für die Mutation L858R EGFR) als eine Proteinkinase Zelle Pellet mit einzelnen Doppel-Z-Punktmutation Sonden Ausrichtung der EGFR L858 WT Sequenz oder EGFR L858R vorbereitet mutierten Sequenz. (B) die Punktmutation-Assay wurde am HuT78 (homozygot für KRAS G12A) und SW116 Zellen (heterozygot für die Mutation des KRAS G12A) als eine Proteinkinase Zelle Pellet mit einzelnen Doppel-Z-Punktmutation Sonden Ausrichtung der KRAS G12 WT-Sequenz durchgeführt oder KRAS G12A mutierten Sequenz. Bitte klicken Sie hier für eine größere Version dieser Figur.
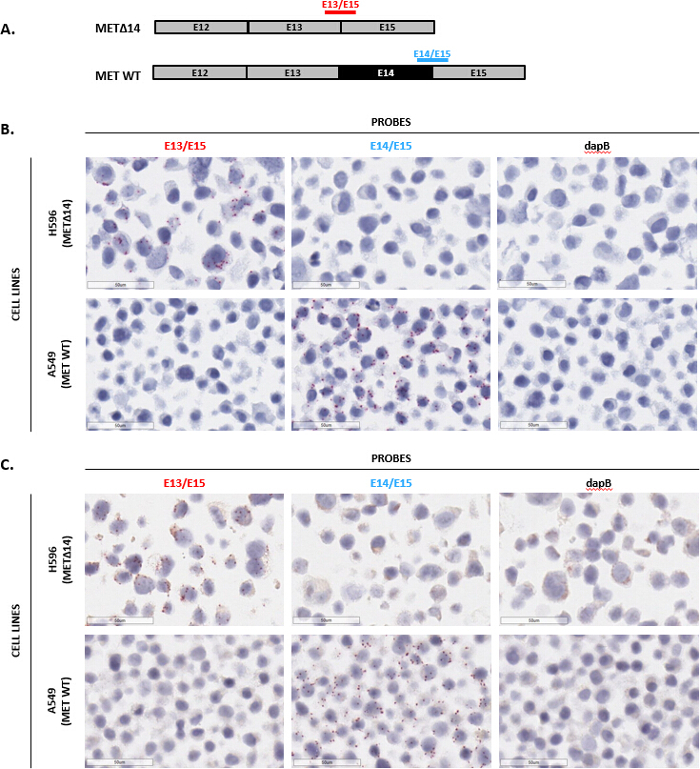
Abbildung 6 : Repräsentative Bilder der automatisierten Färbung mit dem Exon-Junction-Assay. (A) siehe hier ist die Exon-Organisation für traf WT und METΔ14 Zeugnisse und eine schematische Darstellung der einzelnen Doppel-Z-Exon Kreuzung Sonden, die überspannen die Kreuzung um traf WT oder METΔ14 zu erkennen. (B) und (C) automatisierte Exon-Junction-Assay wurde am H596 (Ausdruck von METΔ14) und A549 Zellen (Ausdruck erfüllt WT) als eine Proteinkinase Zelle Pellet mit den Sonden in (A) aufgeführten vorbereitet sowie Negativkontrolle Sonde DapB durchgeführt. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Exon-Junction | Kurze Sequenz | Punktmutation |
| Splice-Variante/isoform | Sequenzen zwischen 50 und 300 nt | Punktmutation |
| Kreisförmige RNA (CircRNA) | Hochgradig homologe Sequenzen | Kurze indel |
| Gen fusion | CDR3-Sequenz für TCR Klone | Gen, die Bearbeitung |
| Gen Knockout (KO) | Pre-miRNA | |
| Kleinen Nukleolus RNA (SnoRNA) | ||
| Gen, die Bearbeitung |
Tabelle 1: Anwendungen von der in situ Assay. Es gibt 3 Hauptkategorien für Anwendungen des Assays: Exon-Junction, kurze Zielsequenz und Punktmutation. In jeder Spalte aufgeführt sind einige spezifische Anwendungsbeispiele für jede Kategorie.
Diskussion
In diesem Bericht wurden die neuartigen ISH Assay-Protokoll und seine Anwendungen im Detail besprochen. Der Test ermöglicht die direkte Visualisierung der Exon-Kreuzungen, kurz-Ziel und hochgradig homologe Sequenzen und Punktmutationen im Zusammenhang mit Gewebe. Der Test basiert auf RNAscope Technologie5 und ist daher in der Lage der Einzelmolekül-Erkennung. Jedoch kann durch eine erweiterte Verstärkersystem, das Signal mit Sonden mit so wenig wie ein Doppel-Z-Paar oder mit einer Ziel-Vorlage-Länge von nur 50 Nukleotide erkannt werden. Da die Sonden so kurz wie ein einziges Doppel-Z Länge sein können, können zum Nachweis von Exon Kreuzungen, kurz-Ziel und hochgradig homologe Sequenzen und Punktmutationen (Tabelle 1).
Für eine erfolgreiche Leistung des Assays gibt es mehrere technische Empfehlungen. Zuerst Gewebe in frischen 10 % Neutral gepufferte Formalin (NBF) bei Raumtemperatur für 16-32 h10festzusetzen. Underfixation (< 16 h) oder Overfixation (> 32 h) wird die Leistung des Assays beeinträchtigen und möglicherweise zusätzliche Optimierung. Zweitens sollte dafür optimale Steuerung von Temperatur und Luftfeuchtigkeit, die für robuste Sonde Hybridisierung und Signal-Verstärkung erforderlich sind, die Folie Verarbeitung System und Hybridisierung Ofen für Protokoll Schritte 2,3 bis 5 (Protease Vorbehandlung, Sonde verwendet werden Hybridisierung, Signalverstärkung und Signalerkennung). Drittens: überschüssige Rest Puffer sollte vor jedem Schritt im gesamten Protokoll richtig dekantiert (aber nicht so sehr, dass die Gewebeschnitte Austrocknen). Wenn die Folien austrocknen, werden erhebliches unspezifisches Signal entwickeln. Viertens kann je nach Gewebetyp, Vorbehandlung Optimierung erforderlich. Verwendung der falschen Protease oder eine suboptimale lang durchführen kann in unter - oder over - digestion und wirken sich negativ auf das Signal. Zu guter Letzt ist es wichtig, immer positive und negative Kontrollen mit den Prüfspitzen laufen. Negativkontrolle Sonden sicherzustellen, dass keine Hintergrundsignal vorhanden ist, und Positivkontrolle Sonden stellen Sie sicher, dass der Test korrekt durchgeführt wurde und die RNA-Qualität in der Probe optimal ist für die Interpretation der Testergebnisse Sonde. Wenn es kein Signal mit der Positivkontrolle Sonde, dann die RNA-Qualität in der Stichprobe dürfte suboptimal und ein Signal nicht mit der Prüfspitze gesehen werden kann.
Neben der manuellen Test wurde die Leistungsfähigkeit des Tests auf automatisierte freigesprochen auch gezeigt (Abbildung 6). Dieser automatisierte ISH-Assay ergibt sich ein hohes Signal-Rausch-Verhältnis und eignet sich für die gleichen Anwendungen wie in Tabelle 1dargestellt; jedoch Vorteile einer automatisierten Tests gehören Standardisierung der Assay-Bedingungen, Minimierung von Inter Benutzer Variabilität und Hands-on-Zeit und Zulage für Hochdurchsatz-Screening von Gewebeproben auf zuverlässige Weise.
Während Immunhistochemie (IHC) und qRT-PCR zur Detektion von Splice-Varianten (besonders EGFRvIII), in klinischen Proben FFPE erlauben, diese Techniken können nicht über die erforderliche Spezifität und bieten keinen Einblick in die räumliche Auflösung des spleißes Variant Ausdruck, bzw.11,12. Ein entscheidender Vorteil des Assays in diesem Protokoll ist seine sehr empfindlich und spezifisch Visualisierung der Spleiß Kreuzungen unter Beibehaltung der morphologischen Gewebe-Kontext. Hier war der Assay Fähigkeit, einzigartige Exon Kreuzungen für mehrere Splice-Varianten, einschließlich EGFRvIII und METΔ14, präzise Ziel gezeigt (Abbildungen 3 und 6). Darüber hinaus hat der Test gezeigt, spleißvariante AR-V7 in Prostata-Krebs, mehrere Isoformen von ErbB4 im Gehirn und Bestätigung der Ko von kreisförmigen RNA-Cdr1as in der Maus Gehirn3,13,14erkennen.
Kurze Sequenz Zielerfassung von ISH Assays kann zur Visualisierung der RNA-Sequenzen so kurz wie 50 Nukleotide in der Länge, wie gezeigt durch den Nachweis des CDR3 Sequenzen Jurkat Zellen (Abbildung 3) abgeleitet. Der Test kann auch erkennen, Gensequenzen, die höchst Homolog zu anderen Familienmitgliedern oder Arten, dargestellt durch Revêchon Et Al., wer den kurzen Ziel-Assay verwendet, um menschliche Progerin ausgedrückt in Maus subkutane weißen Fettgewebe15erkennen. Darüber hinaus können kleine Nukleolus RNA (SnoRNA), CRISPR-vermittelten gen bearbeiten und Vorläufer-RNA erkannten in Situ mit der kurzen Ziel-Test sein. In jüngerer Zeit, kombiniert Fu Et Al. ISH Assays mit IHC, präzise Zellen in der Netzhaut, die mit dem Ausdruck der Pre-MiRNA mir125b16zu identifizieren.
Mutation, die Profilerstellung in Tumoren ist von entscheidender Bedeutung für das Studium der Progression von Tumoren und für gezielte Therapien zu entwickeln. Während Mutation Profilierung durch Hochdurchsatz-Sequenzierung erreicht werden kann, kann nicht diese Technologie intratumorale Heterogenität oder Link genetische Veränderungen mit zellulären Morphologie17,18vollständig behandelt. Der Nachweis von Punktmutationen mit dieser ISH-Assay kann zur Unterscheidung von RNA-Ziel-Sequenzen mit einer Single-Base-Auflösung als validiert durch den Nachweis des EGFR L858R und KRAS G12A Einzel-Nukleotid-Variationen in Zelllinien (Abbildung 5). Darüber hinaus verwendet Baker Et Al. die Punktmutation Assay mehrere Mutationen im BRAF, KRAS und PIK3CA Onkogene in colorectal Krebs18. Identifizieren und räumlich Karte selten mutierten Subclones von Tumorzellen, letztlich zeigen, wie sie zur Intra-Tumor Heterogenität beitragen konnten.
Zusammenfassend lässt sich sagen wurde eine spezielle RNA-ISH-Assay entwickelt. Diese Methodik ermöglicht die Erkennung von Splice-Varianten, kurze Sequenzen und Mutationen in Situ. Es ist sensibel, konkrete, quantifizierbare und anpassungsfähig Leistung durch manuellen Methoden und auf automatisierten freigesprochen.
Offenlegungen
Alle Autoren werden von Advanced Cell Diagnostics, Inc. beschäftigt.
Materialien
| Name | Company | Catalog Number | Comments |
| HybEZ Oven (110 or 220 VAC) or HybEZ II Oven (110 or 220V) | ACD | 310010 or 310013 (HybEZ™), 321710 or 321720 (HybEZ™ II) | |
| HybEZ Humidity Control Tray (with lid) | ACD | 310012 | |
| ACD EZ-Batch Slide Rack (20 slide capacity) 1 rack 310017 | ACD | 310017 | |
| HybEZ Humidifying Paper | ACD | 310015 | |
| ImmEdge Hydrophobic Barrier Pen (required) | Vector Laboratory | H-4000 | |
| SuperFrost Plus Slides (required) | Fisher Scientific | 12-550-15 | |
| 10% neutral-buffered formalin (NBF) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paraffin wax | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microtome | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Gill’s Hematoxylin I | American Master Tech Scientific/MLS | HXGHE1LT | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Xylene | Fisher Scientific/MLS | X3P-1GAL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Vertical 24 Slide Rack | American Master Tech Scientific/MLS | LWSRA24 | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Staining Dishes | American Master Tech Scientific/MLS | LWT4457EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Clearing Agent Dishes, xylene resistant | American Master Tech Scientific/MLS | LWT4456EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| 100% alcohol (EtOH) | American Master Tech Scientific/MLS | ALREACS | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| VectaMount Permanent Mounting Medium (required) | Vector Labs | H-5000 | |
| Cover Glass, 24 mm x 50 mm | Fisher Scientific/MLS | 12-545-F | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Ammonium hydroxide, 28–30% | Sigma-Aldrich/MLS | 320145-500mL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Carboy (>3L) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Oster Steamer Model 5712, Black and Decker Steamer HS3000, or the Braun Multiquick FS 20 Steamer | / | / | |
| Digital thermometer | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Water bath or incubator, capable of holding temperature at 40 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Pipettors and tips, 1–1,000 μL | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Distilled water | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tubes (various sizes) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Fume hood | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Graduated cylinder | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Parafilm | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paper towel or absorbent paper | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microcentrifuge | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microscope and accessories | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Drying oven, capable of holding temperature at 60 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Formaldehyde | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Histogel | Fisher Scientific/MLS | 22-110-678 | |
| BaseScope Reagent Kit - RED | Advanced Cell Diagnostics | 322900 | |
| BaseScope Hs-EGFR-E1E2 | Advanced Cell Diagnostics | 701701 | |
| BaseScope Hs-EGFR-E1E8 | Advanced Cell Diagnostics | 701711 | |
| BaseScope Hs-EGFR-E7E8 | Advanced Cell Diagnostics | 701721 | |
| BaseScope Hs-EGFR-E8E9 | Advanced Cell Diagnostics | 701731 | |
| BaseScope Hs-MET-E14E15 | Advanced Cell Diagnostics | 701811 | |
| BaseScope Hs-MET-E13E15 | Advanced Cell Diagnostics | 701801 | |
| BaseScope Hs-KRAS-G12A | Advanced Cell Diagnostics | 705491 | |
| BaseScope Hs-KRAS-G12-nt35WT | Advanced Cell Diagnostics | 705531 | |
| BaseScope Hs-EGFR-L858R | Advanced Cell Diagnostics | 705451 | |
| BaseScope Hs-EGFR-L858WT | Advanced Cell Diagnostics | 705461 | |
| BaseScope Control Probe Pack Human | Advanced Cell Diagnostics | 322975 |
Referenzen
- Bolha, L., Ravnik-Glavač, M., Glavač, D. Circular RNAs: Biogenesis, Function, and a Role as Possible Cancer Biomarkers. International Journal of Genomics. 2017, 6218353 (2017).
- Yamada, A., Yu, P., Lin, W., Okugawa, Y., Boland, C. R., Goel, A. A RNA-Sequencing approach for the identification of novel long non-coding RNA biomarkers in colorectal cancer. Scientific Reports. 8 (1), 575 (2018).
- Erben, L., He, M. X., Laeremans, A., Park, E., Buonanno, A. A Novel Ultrasensitive In Situ Hybridization Approach to Detect Short Sequences and Splice Variants with Cellular Resolution. Molecular Neurobiology. , (2017).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods in Molecular Biology. 461, 675-686 (2008).
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Paraffin processing of tissue. Protocols Online Available from: https://www.protocolsonline.com/histology/sample-preparation/paraffin-processing-of-tissue/ (2016)
- An, Z., Aksoy, O., Zheng, T., Fan, Q. W., Weiss, W. A. Epidermal growth factor receptor and EGFRvIII in glioblastoma: signaling pathways and targeted therapies. Oncogene. , (2018).
- Frampton, G. M., et al. Activation of MET via diverse exon 14 splicing alterations occurs in multiple tumor types and confers clinical sensitivity to MET inhibitors. Cancer Discovery. 5 (8), 850-859 (2015).
- Awad, M. M., et al. MET Exon 14 Mutations in Non-Small-Cell Lung Cancer Are Associated With Advanced Age and Stage-Dependent MET Genomic Amplification and c-Met Overexpression. Journal of Clinical Oncology. 34 (7), 721-730 (2016).
- Hammond, M. E., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for immunohistochemical testing of estrogen and progesterone receptors in breast cancer. Archives of Pathology and Laboratory Medicine. 134 (6), 907-922 (2010).
- Gan, H. K., Cvrljevic, A. N., Johns, T. G. The epidermal growth factor receptor variant III (EGFRvIII): where wild things are altered. Federation of European Biochemical Societies Journal. 280 (21), 5350-5370 (2013).
- Wheeler, S. E., Egloff, A. M., Wang, L., James, C. D., Hammerman, P. S., Grandis, J. R. Challenges in EGFRvIII detection in head and neck squamous cell carcinoma. Public Library of Science One. 10 (2), e0117781 (2015).
- Zhu, Y., et al. Novel Junction-specific and Quantifiable In Situ Detection of AR-V7 and its Clinical Correlates in Metastatic Castration-resistant Prostate Cancer. European Urology. , (2017).
- Piwecka, M., et al. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function. Science. , (2017).
- Revêchon, G., et al. Rare progerin-expressing preadipocytes and adipocytes contribute to tissue depletion over time. Scientific Reports. 7 (1), 4405 (2017).
- Fu, Y., et al. Functional ectopic neuritogenesis by retinal rod bipolar cells is regulated by miR-125b-5p during retinal remodeling in RCS rats. Scientific Reports. 7 (1), 1011 (2017).
- Yates, L. R., et al. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nature Medicine. 21 (7), 751-759 (2015).
- Baker, A. M., et al. Robust RNA-based in situ mutation detection delineates colorectal cancer subclonal evolution. Nature Communications. 8 (1), 1998 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten