Method Article
Visualização de variantes genéticas, metas de curtas e mutações pontuais no contexto morfológico do tecido com um ensaio de RNA In Situ da hibridação
Neste Artigo
Resumo
Aqui, descrevemos um em situ ensaio de hibridização que permite detecção sensível e específica de sequências tão curtas quanto 50 nucleótidos com single-nucleotide resolução a nível de célula única. O ensaio, que pode ser executado manualmente ou automaticamente, pode habilitar a visualização de variantes da tala, sequências curtas e mutações dentro do contexto de tecido.
Resumo
Porque a medicina de precisão é altamente dependente da detecção precisa de biomarcadores, há uma necessidade crescente de tecnologias padronizadas e robustas que medir RNA biomarcadores em situ em amostras clínicas. Enquanto os ensaios de rotina e vincular como RNAseq e RT-PCR quantitativo permitem medições de expressão do gene altamente sensíveis, eles também exigem a extração do RNA e assim impedem análise valiosa expressão dentro do contexto de tecido morfológica. Em situ da hibridação (ISH) ensaio descrito aqui pode detectar sequências de RNA alvo tão curtos quanto 50 nucleótidos em single-nucleotide resolução e a nível de célula única. Este ensaio complementar o teste comercial anteriormente desenvolvido e permite sensível e específico em situ detecção de variantes da tala, metas de curtas e mutações de ponto dentro do tecido. Neste protocolo, sondas foram projetadas para junções exon exclusivo para duas variantes da tala clinicamente importante, EGFRvIII e METΔ14-alvo. A detecção de sequências de destino curto foi demonstrada pela detecção específica de CDR3 sequências de células T receptores α e β na linha de células Jurkat T. Também mostrado é a utilidade deste teste ISH para a distinção de sequências de RNA alvo em single-nucleotide resolução (mutações pontuais), através da visualização de variações de EGFR L858R e KRAS G12A single-nucleotide em linhas celulares usando coloração automatizada plataformas. Em resumo, o protocolo mostra um ensaio de RNA ISH especializado que permite a detecção de variantes da tala, sequências curtas e mutações em situ para desempenho manual e na mácula automatizada.
Introdução
Tecnologias de transcriptomic de alto rendimento como microarrays e sequenciamento de RNA de próxima geração (RNAseq) melhoraram exponencialmente a descoberta de biomarcadores de RNA com valor clínico diagnóstico, prognóstico e preditivo para várias doenças, incluindo câncer,1,2. Para promover o uso desses biomarcadores em medicina de precisão, há uma alta necessidade de tecnologias padronizadas e robustas que pode medir biomarcadores de RNA dentro do contexto de tecido de amostras clínicas. Embora amplamente estabelecido moagem e vincular ensaios como RNAseq e RT-PCR quantitativo permitem medições de expressão do gene altamente sensível, a homogeneização do tecido necessário e isolamento de RNA implicam a perda de na vivo especificidade do tipo de célula e informação morfológica3. Metodologias de deteção convencional em situ RNA faltam a sensibilidade e especificidade necessários para mensurar fiavelmente raras ou baixa-expressando biomarcadores de RNA dentro de contexto o tecido4.
Um ensaio de hibridização (ISH) comercial em situ (ex., o ensaio de RNAscope) é uma tecnologia que abordou estes desafios e também permite a visualização altamente sensível e específica de moléculas de RNA única maiores que 300 nucleotídeos dentro do contexto morfológico do tecido. Este tipo de ensaio usa um projeto de sonda do oligonucleotide exclusivo de aproximadamente 6 – 20 pares de sonda dupla-Z combinada com uma amplificação de sinal avançado baseado em hibridação5.
Este estudo descreve um ensaio de RNA ISH especializado, BaseScope, complementar à tecnologia comercial previamente concebida que pode detectar sequências de RNA alvo tão curtos quanto 50 nucleótidos em resolução de nucleotídeo único. Este ensaio aborda as intrincadas complexidades da transcriptoma e é aplicável para a detecção precisa de junções exon, sequências de destino curto e mutações de ponto no contexto de tecido (tabela 1), usando como um par de sonda dupla-Z. Este relatório demonstra o protocolo de ensaio completo e sua utilização na detecção de variantes da tala, CDR3 sequências para clones do receptor de células T, e single-nucleotide mutações em FFPE célula linhas e tecidos de tumor.
Protocolo
As amostras de tumor humano utilizadas neste estudo foram deidentified e adquiridas de fontes comerciais em conformidade com as diretrizes éticas locais para pesquisa humana.
1. amostra, equipamento e preparação dos reagentes
-
Preparação da amostra FFPE
- Tecidos
- Imediatamente após a dissecação, corrigir o tecido (cortado em blocos de 3 – 4 mm de espessura) em tampão neutro formol a 10% (NBF) para 16 – 32 h à temperatura ambiente (RT).
Nota: O tempo de fixação pode variar dependendo do tamanho e tipo de tecido. - Lavar a amostra com 1 x tampão fosfato salino (PBS) e desidrata-se usando uma série de padrão etanol (EtOH) (70% EtOH por 30 a 60 min, 80% EtOH por 30 a 60 min, 90% EtOH por 30 a 60 min, 95% EtOH por 30 a 60 min, 3 x 100% para EtOH 30 a 60 min) seguido de xileno.
- Incorporar a amostra em parafina usando procedimentos padrão6 e aparar os blocos de parafina, conforme necessário para remover a parafina em excesso.
Nota: Bloco tamanho irá variar dependendo do tamanho de amostra de tecido, mas um tamanho típico é de 0,75 x 0,75 polegadas2 ou menor.
- Imediatamente após a dissecação, corrigir o tecido (cortado em blocos de 3 – 4 mm de espessura) em tampão neutro formol a 10% (NBF) para 16 – 32 h à temperatura ambiente (RT).
- Linha celular
- Coletar e células de acordo com os métodos recomendados para a linha de célula específica de Pelotas.
- Consertar as células em formol 10% em RT para 24h em rotacao.
- Preparar as células como um sedimento no processamento escaldadas gel (EG., Histogel). Permitir a pelota solidificar, colocando a pelota de gel de célula em um pedaço de parafilm no gelo e deixe descansar por 2 – 3 min submergir a pelota de gel de célula em 1X PBS.
- Desidratar e incorporar os pellets de célula conforme descrito na etapa 1.1.1.
- Preparação de seção
- Corte os tecidos/células incorporados em 5 ± 1 μm seções usando um micrótomo, montar as seções em lâminas de vidro eletrostaticamente adesivo e secá-los durante a noite no RT
Nota: Os slides podem ser armazenados no RT sob dessecação por até 3 meses. - Coloque os slides em um porta-lâminas e asse os slides de tecido montado em um forno de ar circulante a 60 ° C, durante 1 h antes de executar o ensaio.
Nota: Use os slides imediatamente ou armazená-los em RT com dessecantes para até 1 semana. Armazenamento prolongado pode resultar em degradação do RNA.
- Corte os tecidos/células incorporados em 5 ± 1 μm seções usando um micrótomo, montar as seções em lâminas de vidro eletrostaticamente adesivo e secá-los durante a noite no RT
- Tecidos
-
Preparação do material
- Programar o forno de hibridização para 40 ° C. Completamente o humidificação papel molhado e remover qualquer residual de dH2O. Insert o papel na bandeja de controle de umidade e insira a bandeja do forno de hibridização para escaldar pelo menos 30 min antes de usar.
-
Preparação do reagente
- Preencher dois limpar pratos de agente com 200 mL de xileno e encher dois pratos de coloração com 200ml de 100% EtOH, que será usado para deparaffinization de seções.
- Prepare 200 mL de reagente de recuperação de destino disponíveis comercialmente 1x adicionando 180 mL de dH2O a 20 mL de reagente de recuperação 10 x alvo. Coloque dois suportes de lâmina num barco a vapor. Encha um titular de slide com 200 mL de reagente de recuperação de destino de 1x e preencher o outro titular do slide com 200 mL de dH2Heat O. ambas as soluções para ferver usando o vapor.
- Pré-aquecido de tampão de lavagem quente x 50 a 40 ° C para 10-20 min. Prepare-se 3 L de tampão de lavagem 1X diluindo 60 mL de tampão de lavagem x 50 com 2,94 L de dH2O.
- Em uma coifa, preparar a hematoxilina de Gill 50% counterstaining solução adicionando-se 100 mL de hematoxilina de Gill para 100 mL de dH2O. Na coifa, preparar o reagente de Anil (0,02% (p/v) amônia água) adicionando 1,43 mL de hidróxido de amónio 28 – 30% a 250 mL de dH2O.
- Escaldar sondas de alvo a 40 ° C por 10 min antes da hibridização da sonda e trazer os reagentes amplificação (AMP 0-6) para RT
2. o RNA In Situ da hibridação ensaio
-
Deparaffinization e desidratação
- Após o cozimento, conforme descrito na etapa 1.1.3.2 (ponto de parada opcional 1), deparaffinize as seções em xilol por 5 min com agitação. Deparaffinize novamente em xilol fresco 5 min. desidratando em 100% EtOH por 2 min com agitação e repetir novamente no fresco 100% EtOH por 2 min.
- Ar seco os slides por 5 min a 60 ° C em um forno de ar circulante ou no RT até que estejam completamente secos (ponto de parada opcional 2).
-
Pré-tratamentos de amostra
- Incube as secções com ~ 4 gotas de peróxido de hidrogênio de ready-to-use para 10 min a RT para saciar da peroxidase endógena. Decantar a solução de slides e enxaguá-los duas vezes com dH2O.
- Incube as secções com 200 mL de reagente de recuperação o alvo por 15 a 30 min a 100 ° C, num barco a vapor.
Nota: O tempo de incubação pode variar dependendo do tipo de tecido. Neste protocolo, recuperação de destino foi realizada por 15 min para amostras de tumor e pelotas de célula. - Decantar a solução de slides, lave duas vezes com dH2O, mergulhe na 100% EtOH por 3 min e seco os slides a 60 ° C em um forno de ar circulante ou no RT até completamente seco.
- Desenhe uma barreira hidrofóbica ao redor da seção usando uma caneta hidrofóbica, aproximadamente 0,75 x 0,75 polegadas2.
Nota: Não é recomendável desenhar uma barreira menor. Para maiores seções, uma barreira maior precisará ser desenhado. - Deixar a barreira secar completamente por 1 min, durante a noite no RT (ponto de parada opcional 3).
- Coloque os slides em um suporte de corrediça e coloque o suporte do slide na barra de controle de umidade. Adicionar ~ 4 gotas de Protease III para cada slide e incube-os por 15 a 30 min a 40 ° C no forno hibridização para digestão de proteínas.
Nota: O tempo de incubação pode variar dependendo do tipo de tecido. Neste protocolo, digestão de protease foi realizada por 30 min. para amostras de tumor e 15 min para pelotas de célula. - Decantar a solução de slides e enxaguá-los duas vezes com dH2O.
-
Sonda de hibridização
- Adicione ~ 4 gotas da solução de sonda apropriado de ready-to-use para cobrir toda a seção. Se usando seções maiores, adicionar ~ 5-6 gotas.
- Cruzar as sondas para 2 h a 40 ° C, no forno da hibridação. Decantar a solução de slides e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem nesta etapa.
-
Amplificação de sinal
- Incubar as secções com ~ 4 gotas do AMP 0 por slide a 40 ° C no forno por 30 min. decantar a solução de hibridização e lavar as lâminas em 200 mL de tampão de lavagem 1 x por 2 min em RT com agitação ocasional. Repita o procedimento de lavagem nesta etapa.
- Incubar as secções com ~ 4 gotas de 1 AMP por slide a 40 ° C no forno por 15 min. decantar a solução de hibridização e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem.
- Incubar as secções com ~ 4 gotas de 2 AMP por slide a 40 ° C no forno por 30 min. decantar a solução de hibridização e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem.
- Incubar as secções com ~ 4 gotas do AMP 3 por slide a 40 ° C no forno por 30 min. decantar a solução de hibridização e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem.
- Incubar as secções com ~ 4 gotas de 4 AMP / diapositivo a 40 ° C no forno por 15 min. decantar a solução de hibridização e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem.
- Incubar as secções com ~ 4 gotas de 5 AMP por slide no RT por 30 min. decantar a solução e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem.
- Incubar as secções com ~ 4 gotas de 6 AMP por slide no RT durante 15 min. decantar a solução e lavar as lâminas em 200 mL de tampão de lavagem por 2 min em RT com agitação ocasional de 1x. Repita o procedimento de lavagem.
-
Detecção do sinal
- Prepare o corante Fast Red solução de trabalho. Para um slide com uma barreira de2 polegadas de 0,75 x 0,75, adicionar 2 μL da tintura rápido vermelho-B para 120 μL de Fast Red-A em um tubo e misture bem.
Nota: Dependendo do tamanho da barreira hidrofóbica e número de slides, volumes da solução de trabalho de Fast Red pode variar. - Decantar o líquido em excesso de slides e adicione o corante Fast Red, solução para os slides de trabalho. A incubar durante 10 minutos a RT na bandeja com uma tampa para evitar a exposição à luz.
Nota: Utilize o Fast Red solução dentro de 5 min de preparação de trabalho e não o exponha à luz directa do sol ou luz UV. - Decantar a solução Fast Red e enxágue as lâminas duas vezes com água da torneira. Coloque os slides em um rack de slide.
- Prepare o corante Fast Red solução de trabalho. Para um slide com uma barreira de2 polegadas de 0,75 x 0,75, adicionar 2 μL da tintura rápido vermelho-B para 120 μL de Fast Red-A em um tubo e misture bem.
-
Counterstaining
- Counterstain as seções de tecido com solução de hematoxilina de Gill 50% por 2 min em RT. Wash os slides com água da torneira e repita isso várias vezes até que as lâminas são claras, enquanto as seções permanecem roxas. Mergulhe os slides na água de amônia de 0,02% para Anil (mergulhe 2 – 3 vezes). Substituir a água de amônia com água da torneira e lave os slides 3 a 5 vezes.
-
Deslize a montagem
- Seca os slides em um forno de ar circulante a 60 ° C por 15 min ou em RT até completamente seco. Coloque 1 a 2 gotas de reagente de montagem em cada slide e lugar de lamelas sobre cada seção. Evite qualquer interceptação de bolhas de ar. Ar seco os slides pelo menos 5 min.
Nota: O substrato de Fast Red é sensíveis ao álcool. Sem desidratar os slides em álcool.
- Seca os slides em um forno de ar circulante a 60 ° C por 15 min ou em RT até completamente seco. Coloque 1 a 2 gotas de reagente de montagem em cada slide e lugar de lamelas sobre cada seção. Evite qualquer interceptação de bolhas de ar. Ar seco os slides pelo menos 5 min.
-
Visualização
- Observe os slides sob um microscópio brightfield padrão.
Resultados
Em situ da hibridação do ensaio fluxo de trabalho:
O fluxo de trabalho está representado na Figura 1 e consiste de quatro partes: permeabilização de células ou tecidos com recuperação de alvo e soluções de protease, hibridização de sondas para o destino do RNA, sinal de amplificação e visualização do sinal. O sinal também pode ser quantificado, usando sistemas de software de imagem digital ou de forma semi-quantitativa, baseado no número de pontos por celular. O procedimento manual descrito na Figura 2 também foi totalmente automatizado em sistemas comerciais de automancha.
Coloração representativo para a deteção de junção exon (variante de tala EGFRvIII): EGFRvIII é uma variante do receptor de fator de crescimento epidérmico que surge de uma exclusão de genômica em-frame de exões 2 a 7, levando a sinalização oncogênica constitutivamente ativo7 . O ensaio foi usado para identificar o status do EGFRvIII em amostras de tumor FFPE glioblastoma (GBM). Single Duplo-Z sondas foram projetadas para abranger as junções exon a fim de detectar qualquer WT, mutante, ou ambas as transcrições (Figura 3A). As sondas de EGFR WT abrangem as junções de exões 1 e 2 (E1/E2) ou exões 7 e 8 (E7/E8), enquanto as sondas EGFRvIII específicos abrangem a junção de exões 1 e 8 (E1/E8). Uma sonda comum que atravessa a junção de exões 8 e 9 (E8/E9) também foi usada para detectar EGFR total (ambos WT e EGFRvIII transcrições). Todas as sondas foram então usadas para determinar o status EGFR em amostras FFPE GBM. Os dois exemplos representativos, mostrados na Figura 3B foram tirados de um estudo maior. EGFR status foi confirmado por um método independente, RT-PCR. Ambas as sondas WT detectou sinal em ambas as amostras, indicando que as duas amostras expressam EGFR WT (Figura 3B). No entanto, a sonda mutante só mostrou a detecção do sinal na EGFRvIII + amostra, confirmando que esta amostra é realmente positiva para a variante EGFRvIII (Figura 3B, painéis à esquerda). Por outro lado, a sonda mutante não detectou sinal na EGFRvIII-amostra (Figura 3B, painéis certo). Tomados em conjunto, estes resultados demonstram que o ensaio de junção exon pode identificar o status do EGFRvIII em amostras de tumor GBM FFPE.
Representante de coloração para alvos curtos:
O CDR3, ou complementar determinante região 3, é um domínio altamente variável em receptores de células T. Normalmente, a sequência CDR3 são bastante curtos; por exemplo, as sequências α e β CDR3 da linha de células Jurkat T são 48 e 51 nucleotídeos de comprimento, respectivamente (Figura 4A). Para identificar a sequências específicas de CDR, expressado nas células Jurkat, antisentidas sondas para CDR3 α e β que são expressas na linha de células Jurkat T foram gerados, além de sondas de sentido para CDR3 α e β para servir como controle negativo sondas. Então, todas as sondas foram testadas em células Jurkat FFPE-preparado com o ensaio. Coloração robusto foi observada com antisentido sondas para ambos CDR3 α e β nas células Jurkat, Considerando que o sentido sondas não detectado pouco ou nenhum sinal (Figura 4B). Estes resultados demonstram a capacidade do ensaio curto alvo de discernir entre sequências de CDR altamente variáveis mas curtas para clones do receptor de células T.
Representante de coloração para o ponto de mutação de EGFR L858R:
Ponto de mutação sondas foram desenvolvidas para detectar variações de nucleotídeo único e pequenas inserções ou exclusões (puntuais) no contexto de tumor. Figura 5A demonstra a capacidade para a deteção em situ da mutação ponto L858R EGFR (2573T > G). Duas sondas foram criadas: um para detectar o L858R uma mutação de EGFR sequência e outro para detectar a sequência de EGFR L858 WT. As duas sondas foram testadas em duas linhas de célula FFPE-preparado: H2229, que só expressa EGFR L858 WT; e H1975, que é heterozigoto para a mutação de EGFR L858R. A sonda mutante L858R detectado sinal apenas na linha de célula de H1975 mas não em linhagem celular H2229. No entanto, a sonda WT detectou sinal em ambas as linhas de célula. Da mesma forma, Figura 5B visualiza em situ deteção do ponto de mutação KRAS G12A (35 G > C). Duas sondas foram projetadas para detectar as sequências KRAS G12A MT e KRAS G12 WT e então testadas sobre a linhagem de células de HuT78 (que apenas expressa KRAS G12 WT) e a linha de celular de SW116 (que é heterozigota para a mutação KRAS G12A). Enquanto a sonda KRAS G12 WT detectou sinal em ambas as linhas de célula, a sonda G12A KRAS só detectou sinal na linha de celular SW116. Tomados em conjunto, estes dados demonstram a capacidade técnica de ensaio de mutação pontual na detecção de polimorfismos de nucleotídeo único no contexto de célula e tecido.
Representante de coloração para o ensaio automatizado em situ :
Ensaios automatizados permitem um maior número de amostras para ser executado de forma mais confiável, minimizando a variabilidade inter usuário e tempo de prático e gerando resultados consistentemente reprodutíveis. Portanto, uma versão automatizada do ensaio foi desenvolvida. Para demonstrar a coloração automatizada com este ensaio, deteção da variante da tala METΔ14 foi examinada. Esta variante é o resultado de exon 14 no gene MET sendo ignorado durante a pre-mRNA emendar, que leva à ativação constitutiva e transformação oncogênica do MET receptor8,9. Para detectar especificamente a variante METΔ14, destinavam-se duas sondas de junção exon: um que atravessa a junção de exões 13 e 15 (E13/E15) para detectar a transcrição variante METΔ14 e outro que atravessa a junção de exões 14 e 15 (E14/E15) para detectar o WT conheceu transcrição (figura 6A). Ambas as sondas foram então testadas em 2 linhas de célula FFPE-preparado: H596, que expressa a variante METΔ14, e A549, que expressa o gene WT conheceu. Ambas as sondas mostraram padrões de expressão mutuamente exclusivos, com a sonda E13/E15 somente detecção de sinal nas células H596 e a sonda E14/E15 somente detecção de sinal nas células A549 (figuras 6B e 6C). Por último, a sonda para dapB não mostrou nenhum sinal, não indicando nenhum sinal de fundo (figuras 6B e 6C). Em geral, esses dados demonstram específica deteção do MET variante METΔ14 em situ utilizando o ensaio de BaseScope automatizado.
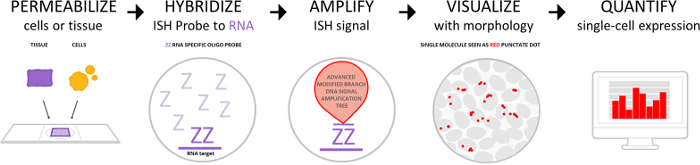
Figura 1 : O fluxo de trabalho ensaio. O fluxo de trabalho consiste em 4 etapas principais: pré-tratamento para permeabilize células ou tecidos, hibridização da sonda para alvo do RNA, amplificação de sinal e deteção de sinal por visualização sob microscópio brightfield ou fluorescente. Pontos individuais possam ser quantificados utilizando uma plataforma de análise de imagem digital. Clique aqui para ver uma versão maior desta figura.
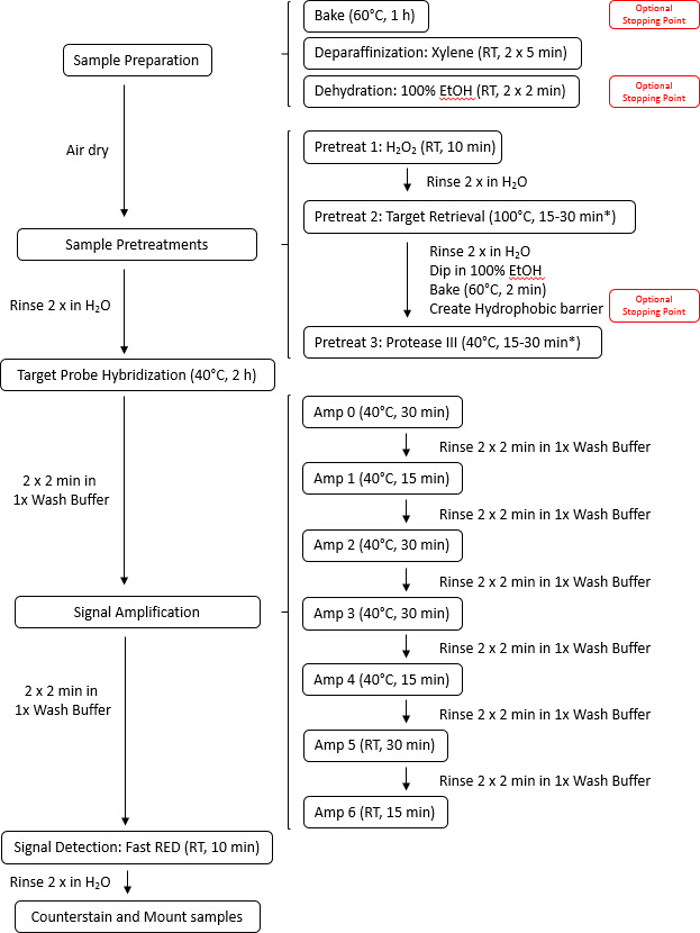
Figura 2 : Ilustração do protocolo do ensaio manual. O ensaio inteiro pode ser concluído em 9 h. pré-tratamento vezes podem variar dependendo do tipo de tecido, então é aconselhável consultar o apêndice A no manual do usuário para recomendações de pré-tratamento de tecido sobre o tempo de incubação. Clique aqui para ver uma versão maior desta figura.
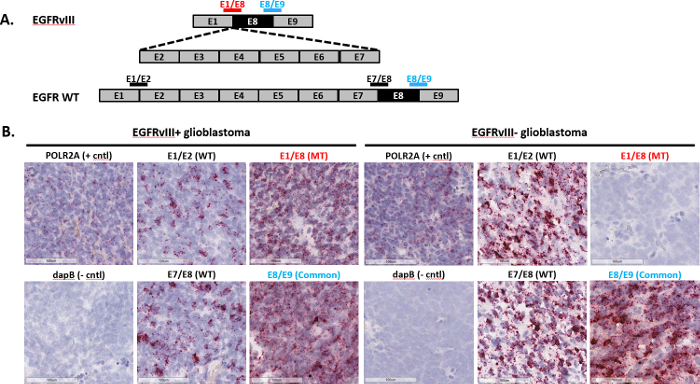
Figura 3 : Imagens representativas da deteção de junção exon. (A) mostrado aqui são a organização de exon para transcrições de EGFR WT e EGFRvIII e um esquema representando sondas de junção exon duplo-Z que escarrancham a junção para detectar EGFR WT ou EGFRvIII. Ensaio de junção (B) o exon foi executado em duas amostras de glioblastoma FFPE usando as sondas enumeradas na alínea A, bem como um dapB sonda de controle, POLR2A e sonda de controlo negativo, positivo. Clique aqui para ver uma versão maior desta figura.
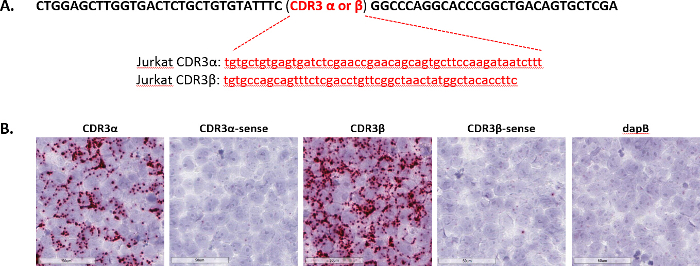
Figura 4 : Deteção de sequências de imagens representativas do destino curto. (A) mostrado aqui são sequências CDR3 nas células Jurkat. A sequência de preto é uma sequência comum de acompanhamento e a sequência em vermelho é exclusiva para CDR3α ou CDR3β. Sondas de sequência único duplo-Z curta alvo foram projetadas contra essas sequências. (B) o ensaio de curto alvo foi realizada em células Jurkat preparadas como uma pelota de célula FFPE usando sondas antisenso ou sentido como alvo as sequências no (A) e dapB foi usado como um controle negativo. Clique aqui para ver uma versão maior desta figura.
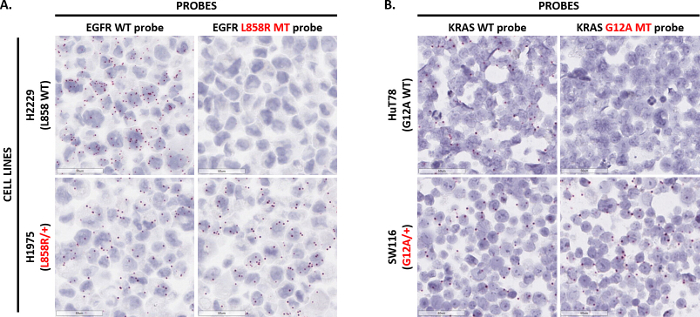
Figura 5 : Imagens representativas da deteção do ponto de mutação. (A), o ensaio foi realizada em células de H2229 (homozigotos para EGFR L858) e células de H1975 preparadas como uma pelota de célula FFPE usando sondas de mutação de ponto único duplo-Z visando a sequência do EGFR L858 WT ou L858R EGFR (heterozigotas para a mutação de EGFR L858R) sequência de mutante. (B) o ensaio de mutação pontual realizou-se em células HuT78 (homozigotos para KRAS G12A) e células de SW116 preparadas como uma pelota de célula FFPE usando sondas de mutação de ponto único duplo-Z visando a sequência de KRAS G12 WT (heterozigotas para a mutação KRAS G12A) ou G12A KRAS mutado sequência. Clique aqui para ver uma versão maior desta figura.
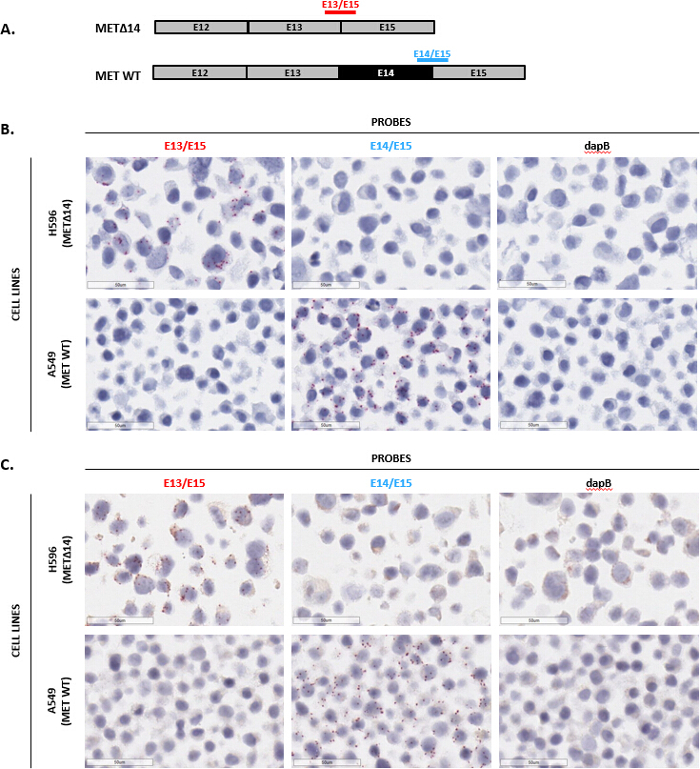
Figura 6 : Imagens representativas de coloração automatizada com o ensaio de junção exon. (A) mostrado aqui é a organização do exon para transcrições conheceu WT e METΔ14 e um esquema representando sondas de junção única dupla-Z exão que escarrancham a junção para detectar conheceu WT ou METΔ14. (B) e (C) o ensaio de junção exon automatizado foi realizada em células de H596 (expressando METΔ14) e células de A549 (expressando conheceu WT) preparadas como uma pelota de célula FFPE usando as sondas enumeradas na alínea A, bem como controlo negativo sonda dapB. Clique aqui para ver uma versão maior desta figura.
| Exon Junction | Sequência de curta | Ponto de mutação |
| Splice variante/isoform | Sequências de entre 50 e 300 nt | Ponto de mutação |
| RNA circular (circRNA) | Sequências homólogas altamente | Indel curto |
| Fusão de gene | CDR3 sequência para clones TCR | Gene de edição |
| Nocaute de gene (KO) | Pré-miRNA | |
| RNA nucleolar pequeno (snoRNA) | ||
| Gene de edição |
Tabela 1: aplicações da em situ ensaio. Há 3 principais categorias para aplicações deste teste: junção exon, sequência curta destino e ponto de mutação. Listados em cada coluna são alguns exemplos de aplicação específica para cada categoria.
Discussão
Neste relatório, o protocolo de ensaio novo ISH e suas aplicações foram discutidas em detalhe. O ensaio permite a visualização direta das junções exon, sequências curtas-alvo e altamente homólogas e mutações de ponto no contexto do tecido. O ensaio é baseado na tecnologia de RNAscope5 e, portanto, é capaz de deteção da molécula. No entanto, devido a um sistema avançado de amplificação, o sinal pode ser detectado com sondas contendo tão pouco como um par de duplo-Z ou com um comprimento de modelo alvo de apenas 50 nucleótidos. Porque as sondas podem ser tão curtas quanto um único double-Z de comprimento, isto permite a detecção de mutações pontuais (tabela 1), sequências curtas-alvo e altamente homólogas e junções exon.
Para o desempenho bem sucedido do ensaio, existem várias recomendações técnicas. Primeiro, tecidos devem ser fixados em fresco 10% tampão neutro formalina (NBF) à temperatura de 16 – 32 h10. Underfixation (< 16 h) ou overfixation (> 32 h) irá prejudicar o desempenho do ensaio e pode exigir a otimização adicional. Em segundo lugar, para garantir um controlo ideal de temperatura e umidade, que são necessários para amplificação de hibridização e sinal de sonda robusto, o slide forno de sistema e a hibridação de processamento deve ser usado para protocolo as etapas 2.3 para 5 (protease pré-tratamento, ponta de prova hibridização, sinal de amplificação e detecção do sinal). Em terceiros lugar, excesso buffers residuais devem ser adequadamente decantado antes de cada passo em todo o protocolo (mas não tanto que as secções de tecido secarem). Se os slides secarem, irá desenvolver o significativo sinal específico. Em quarto lugar, dependendo do tipo de tecido, otimização de pré-tratamento pode ser necessária. Usando a protease errada ou executar por um tempo qualidade inferior pode resultar em sob - ou over - digestion e afetará negativamente o sinal. Por último, é importante sempre executar controles positivos e negativos com as pontas de teste. Sondas de controlo negativo certifique-se que não há nenhum sinal de fundo, e sondas de controle positivo certifique-se que o ensaio foi feito corretamente e que a qualidade do RNA na amostra é ideal para interpretar os resultados da sonda. Se não há nenhum sinal com a sonda de controle positivo, então a qualidade do RNA na amostra é provável suboptimal e um sinal não pode ser visto com a sonda de teste.
Além do ensaio manual, a capacidade de realizar o ensaio na mácula automatizada também foi demonstrado (Figura 6). Este ensaio ISH automatizado produz uma alta relação sinal-ruído e é aplicável para as mesmas aplicações, conforme mostrado na tabela 1; no entanto, benefícios de um ensaio automatizado incluem a normalização das condições de ensaio, minimização da variabilidade inter usuário e tempo de hands-on e subsídio para seleção da elevado-produção de amostras de tecido de maneira confiável.
Enquanto a imuno-histoquímica (IHC) e qRT-PCR permitam detecção de variantes da tala (particularmente EGFRvIII), em amostras clínicas FFPE, estas técnicas podem falta a necessária especificidade e não oferecem insights sobre a resolução espacial da tala expressão variante, respectivamente de11,12. A principal vantagem do ensaio neste protocolo é sua visualização altamente sensível e específica dos cruzamentos da tala, preservando o contexto morfológico do tecido. Aqui, a capacidade do ensaio de segmentar com precisão as junções exon exclusivo para diversas variantes de tala, incluindo EGFRvIII e METΔ14, foi demonstrada (figuras 3 e 6). Além disso, o ensaio foi mostrado para detectar a variante da tala AR-V7 no cancro da próstata, múltiplas isoformas de ErbB4 no cérebro e confirmação de nocaute de Cdr1as de RNA circular no rato cérebro3,13,14.
Detecção de sequência curta alvos por este ensaio ISH permite a visualização de sequências de ARN tão curtas quanto 50 nucleotídeos de comprimento, como demonstrado pela detecção de sequências CDR3 derivadas de células Jurkat (Figura 3). O ensaio também pode detectar sequências de genes que são altamente homólogas de outros membros da família ou espécie, como mostrado por Revêchon et al., que usado o ensaio de curto-alvo para detectar progerin humana expressado no tecido adiposo branco subcutâneo de rato15. Além disso, a RNA nucleolar pequeno (snoRNA), edição de genes mediada por CRISPR e microRNA precursor podem ser detectado em situ com o ensaio de curto alvo. Mais recentemente, et al . Fu combinado este ensaio ISH com IHC para identificar células precisas na retina expressando o pre-miRNA mir125b16.
Mutação que perfila em tumores é fundamental para estudar a progressão dos tumores e para o desenvolvimento de terapias alvo. Enquanto a mutação de criação de perfil pode ser alcançada através do sequenciamento de alta produtividade, esta tecnologia não pode resolver completamente intratumoral heterogeneidade ou link alterações genéticas com morfologia celular17,18. A detecção de mutações pontuais usando este ensaio ISH permite a distinção de sequências de RNA alvo em uma resolução de base única, como validado pela detecção de variações de nucleotídeo único EGFR L858R e G12A KRAS em linhas celulares (Figura 5). Além disso, Baker et al usado o ensaio de mutação pontual para várias mutações nas oncogenes BRAF, KRAS e PIK3CA em câncer colorretal18-alvo. Eles foram capazes de identificar e mapear espacialmente raros subclones mutantes de células tumorais, em última análise, mostrando como eles contribuem para a heterogeneidade intra-tumor.
Em resumo, foi desenvolvido um ensaio de RNA ISH especializado. Esta metodologia permite a detecção de variantes da tala, sequências curtas e mutações em situ. É sensível, específico, quantificáveis e adaptável ao desempenho tanto por métodos manuais e automatizadas mácula.
Divulgações
Todos os autores são empregados por Advanced Cell Diagnostics, Inc.
Materiais
| Name | Company | Catalog Number | Comments |
| HybEZ Oven (110 or 220 VAC) or HybEZ II Oven (110 or 220V) | ACD | 310010 or 310013 (HybEZ™), 321710 or 321720 (HybEZ™ II) | |
| HybEZ Humidity Control Tray (with lid) | ACD | 310012 | |
| ACD EZ-Batch Slide Rack (20 slide capacity) 1 rack 310017 | ACD | 310017 | |
| HybEZ Humidifying Paper | ACD | 310015 | |
| ImmEdge Hydrophobic Barrier Pen (required) | Vector Laboratory | H-4000 | |
| SuperFrost Plus Slides (required) | Fisher Scientific | 12-550-15 | |
| 10% neutral-buffered formalin (NBF) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paraffin wax | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microtome | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Gill’s Hematoxylin I | American Master Tech Scientific/MLS | HXGHE1LT | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Xylene | Fisher Scientific/MLS | X3P-1GAL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Vertical 24 Slide Rack | American Master Tech Scientific/MLS | LWSRA24 | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Staining Dishes | American Master Tech Scientific/MLS | LWT4457EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Clearing Agent Dishes, xylene resistant | American Master Tech Scientific/MLS | LWT4456EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| 100% alcohol (EtOH) | American Master Tech Scientific/MLS | ALREACS | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| VectaMount Permanent Mounting Medium (required) | Vector Labs | H-5000 | |
| Cover Glass, 24 mm x 50 mm | Fisher Scientific/MLS | 12-545-F | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Ammonium hydroxide, 28–30% | Sigma-Aldrich/MLS | 320145-500mL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Carboy (>3L) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Oster Steamer Model 5712, Black and Decker Steamer HS3000, or the Braun Multiquick FS 20 Steamer | / | / | |
| Digital thermometer | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Water bath or incubator, capable of holding temperature at 40 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Pipettors and tips, 1–1,000 μL | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Distilled water | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tubes (various sizes) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Fume hood | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Graduated cylinder | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Parafilm | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paper towel or absorbent paper | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microcentrifuge | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microscope and accessories | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Drying oven, capable of holding temperature at 60 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Formaldehyde | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Histogel | Fisher Scientific/MLS | 22-110-678 | |
| BaseScope Reagent Kit - RED | Advanced Cell Diagnostics | 322900 | |
| BaseScope Hs-EGFR-E1E2 | Advanced Cell Diagnostics | 701701 | |
| BaseScope Hs-EGFR-E1E8 | Advanced Cell Diagnostics | 701711 | |
| BaseScope Hs-EGFR-E7E8 | Advanced Cell Diagnostics | 701721 | |
| BaseScope Hs-EGFR-E8E9 | Advanced Cell Diagnostics | 701731 | |
| BaseScope Hs-MET-E14E15 | Advanced Cell Diagnostics | 701811 | |
| BaseScope Hs-MET-E13E15 | Advanced Cell Diagnostics | 701801 | |
| BaseScope Hs-KRAS-G12A | Advanced Cell Diagnostics | 705491 | |
| BaseScope Hs-KRAS-G12-nt35WT | Advanced Cell Diagnostics | 705531 | |
| BaseScope Hs-EGFR-L858R | Advanced Cell Diagnostics | 705451 | |
| BaseScope Hs-EGFR-L858WT | Advanced Cell Diagnostics | 705461 | |
| BaseScope Control Probe Pack Human | Advanced Cell Diagnostics | 322975 |
Referências
- Bolha, L., Ravnik-Glavač, M., Glavač, D. Circular RNAs: Biogenesis, Function, and a Role as Possible Cancer Biomarkers. International Journal of Genomics. 2017, 6218353 (2017).
- Yamada, A., Yu, P., Lin, W., Okugawa, Y., Boland, C. R., Goel, A. A RNA-Sequencing approach for the identification of novel long non-coding RNA biomarkers in colorectal cancer. Scientific Reports. 8 (1), 575 (2018).
- Erben, L., He, M. X., Laeremans, A., Park, E., Buonanno, A. A Novel Ultrasensitive In Situ Hybridization Approach to Detect Short Sequences and Splice Variants with Cellular Resolution. Molecular Neurobiology. , (2017).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods in Molecular Biology. 461, 675-686 (2008).
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Paraffin processing of tissue. Protocols Online Available from: https://www.protocolsonline.com/histology/sample-preparation/paraffin-processing-of-tissue/ (2016)
- An, Z., Aksoy, O., Zheng, T., Fan, Q. W., Weiss, W. A. Epidermal growth factor receptor and EGFRvIII in glioblastoma: signaling pathways and targeted therapies. Oncogene. , (2018).
- Frampton, G. M., et al. Activation of MET via diverse exon 14 splicing alterations occurs in multiple tumor types and confers clinical sensitivity to MET inhibitors. Cancer Discovery. 5 (8), 850-859 (2015).
- Awad, M. M., et al. MET Exon 14 Mutations in Non-Small-Cell Lung Cancer Are Associated With Advanced Age and Stage-Dependent MET Genomic Amplification and c-Met Overexpression. Journal of Clinical Oncology. 34 (7), 721-730 (2016).
- Hammond, M. E., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for immunohistochemical testing of estrogen and progesterone receptors in breast cancer. Archives of Pathology and Laboratory Medicine. 134 (6), 907-922 (2010).
- Gan, H. K., Cvrljevic, A. N., Johns, T. G. The epidermal growth factor receptor variant III (EGFRvIII): where wild things are altered. Federation of European Biochemical Societies Journal. 280 (21), 5350-5370 (2013).
- Wheeler, S. E., Egloff, A. M., Wang, L., James, C. D., Hammerman, P. S., Grandis, J. R. Challenges in EGFRvIII detection in head and neck squamous cell carcinoma. Public Library of Science One. 10 (2), e0117781 (2015).
- Zhu, Y., et al. Novel Junction-specific and Quantifiable In Situ Detection of AR-V7 and its Clinical Correlates in Metastatic Castration-resistant Prostate Cancer. European Urology. , (2017).
- Piwecka, M., et al. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function. Science. , (2017).
- Revêchon, G., et al. Rare progerin-expressing preadipocytes and adipocytes contribute to tissue depletion over time. Scientific Reports. 7 (1), 4405 (2017).
- Fu, Y., et al. Functional ectopic neuritogenesis by retinal rod bipolar cells is regulated by miR-125b-5p during retinal remodeling in RCS rats. Scientific Reports. 7 (1), 1011 (2017).
- Yates, L. R., et al. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nature Medicine. 21 (7), 751-759 (2015).
- Baker, A. M., et al. Robust RNA-based in situ mutation detection delineates colorectal cancer subclonal evolution. Nature Communications. 8 (1), 1998 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados