Method Article
Visualizzazione di varianti genetiche, gli obiettivi di brevi e mutazioni puntiformi nel contesto morfologico del tessuto con una determinazione del RNA In Situ di ibridazione
In questo articolo
Riepilogo
Qui, descriviamo un in situ di ibridazione test che consente la rivelazione sensibile e specifica delle sequenze più breve 50 nucleotidi con risoluzione del singolo-nucleotide a livello di singola cellula. L'analisi, che possono essere eseguita manualmente o automaticamente, può abilitare la visualizzazione delle varianti di splicing, brevi sequenze e mutazioni all'interno del contesto del tessuto.
Abstract
Perché la medicina precisione dipende fortemente la rilevazione accurata di biomarcatori, c'è un crescente bisogno di tecnologie standardizzate e robusti che misurano RNA biomarcatori in situ in campioni clinici. Mentre i saggi di grind-e-bind come RNAseq e RT-PCR quantitativa, consentono misurazioni di espressione genica altamente sensibili, richiedono anche estrazione del RNA e impedire così l'analisi di espressione prezioso all'interno del contesto morfologico del tessuto. L'analisi in situ di ibridazione (ISH) descritto qui può rilevare sequenze di RNA bersaglio più breve 50 nucleotidi alla risoluzione del singolo-nucleotide e a livello di singola cellula. Questo test è complementare l'analisi commerciale precedentemente sviluppato e consente un sensibile e specifico in situ rilevamento delle varianti di splicing, obiettivi di brevi e mutazioni puntiformi all'interno del tessuto. In questo protocollo, le sonde sono stati progettati per giunzioni esone unico per due varianti di splicing clinicamente importante, EGFRvIII e METΔ14 di destinazione. La rilevazione di sequenze target di breve è stata dimostrata tramite la rilevazione specifica di sequenze CDR3 della T-cellula dei recettori α e β nella linea Jurkat T-cellulare. Viene inoltre illustrata l'utilità di questo test ISH per la distinzione di sequenze bersaglio RNA a singolo nucleotide risoluzione (mutazioni puntiformi) attraverso la visualizzazione delle variazioni di singolo-nucleotide L858R EGFR e KRAS G12A in linee cellulari usando la colorazione automatica piattaforme. In sintesi, il protocollo indica un'analisi di RNA ISH specializzata che consente il rilevamento di varianti di splicing, brevi sequenze e mutazioni in situ per prestazioni manuale e con coloratori automatici.
Introduzione
Tecnologie di alto-rendimento Transcrittomica come microarrays e sequenziamento di RNA di nuova generazione (RNAseq) hanno migliorato esponenzialmente la scoperta di biomarcatori di RNA con valore clinico diagnostico, prognostico e predittivo per varie malattie tra cui cancro1,2. Per spostare in avanti l'uso di questi biomarcatori in medicina di precisione, c'è un'alta necessità di tecnologie standardizzate e robusti che può misurare i biomarcatori di RNA all'interno del tessuto di campioni clinici. Mentre ampiamente stabilito grind-e-bind saggi come RNAseq e RT-PCR quantitativa, consentono misurazioni di espressione genica altamente sensibile, l'omogeneizzazione del tessuto richiesto e isolamento del RNA implicano la perdita di specificità in vivo delle cellule-tipo e informazioni morfologiche3. Convenzionale in situ RNA metodologie di rilevamento mancano la sensibilità e specificità necessarie per misurare in modo affidabile rare o basso-esprimendo biomarcatori di RNA all'interno del contesto del tessuto4.
Un'analisi di ibridazione (ISH) commerciale in situ (ad es., il dosaggio di RNAscope) è una tecnologia che ha affrontato queste sfide e consente inoltre la visualizzazione altamente sensibile e specifica di singole molecole di RNA superiore a 300 nucleotidi all'interno del contesto morfologico del tessuto. Questo tipo di analisi utilizza un design di sonda unica del oligonucleotide di circa 6 – 20 coppie di doppio-Z sonda combinata con un' amplificazione di segnale avanzato basato su ibridazione5.
Questo studio descrive un'analisi specializzata di RNA ISH, BaseScope, complementare alla tecnologia commerciale precedentemente progettata in grado di rilevare sequenze di RNA bersaglio più breve 50 nucleotidi a risoluzione di singolo nucleotide. Questo test consente di risolvere l'intricata complessità del trascrittoma ed è applicabile per la rilevazione accurata di giunzioni esone, sequenze target di breve e mutazioni puntiformi nel contesto del tessuto (tabella 1) utilizzando una coppia di sonde di doppio-Z. Questo rapporto dimostra il protocollo di analisi completa e il suo uso nella rilevazione delle varianti di splicing, CDR3 sequenze per cloni a cellula T del ricevitore, e mutazioni di singolo nucleotide in FFPE cellulare linee e tessuti del tumore.
Protocollo
I campioni di tumore umano utilizzati in questo studio erano compararli e acquisiti da fonti commerciali conformemente agli orientamenti etici locali per ricerca umana.
1. campione, attrezzatura e preparazione dei reagenti
-
Preparazione dei campioni FFPE
- Tessuti
- Immediatamente dopo la dissezione, fissare il tessuto (tagliato in blocchi di 3 – 4 mm di spessore) in formalina 10% neutra tamponata (NBF) per 16 – 32 h a temperatura ambiente (TA).
Nota: Tempo di fissazione varia a seconda delle dimensioni e del tipo di tessuto. - Lavare il campione con 1x tampone fosfato salino (PBS) e disidratare utilizzando una serie di standard etanolo (EtOH) (70% EtOH per 30 – 60 min, 80% EtOH per 30 – 60 min, 90% EtOH per 30 – 60 min, 95% EtOH per 30 – 60 min, 3 x 100% per EtOH 30 – 60 minuti) seguita da xilene.
- Incorporare il campione in paraffina utilizzando le procedure standard6 e tagliare i blocchi di paraffina come necessario per rimuovere la paraffina in eccesso.
Nota: Dimensione blocco variano a seconda della dimensione del campione di tessuto, ma una dimensione tipica è 0.75 x 0.75 pollici2 o più piccolo.
- Immediatamente dopo la dissezione, fissare il tessuto (tagliato in blocchi di 3 – 4 mm di spessore) in formalina 10% neutra tamponata (NBF) per 16 – 32 h a temperatura ambiente (TA).
- Linee cellulari
- Raccogliere e appallottolare celle secondo i metodi consigliati per la linea cellulare specifico.
- Difficoltà cellule in formaldeide al 10% a temperatura ambiente per 24 h in un rotatore.
- Preparare le cellule come una pallina in gel di trattamento preriscaldata (ad es., Histogel). Lasciar solidificare inserendo il pellet di gel-cellulare su un pezzo di parafilm sul ghiaccio il pellet e lasciate riposare per 2 – 3 min. Immergere il pellet di gel-cellulare in PBS 1X.
- Disidratare e incorporare il pellet cellulare come descritto al punto 1.1.1.
- Preparazione di sezione
- Tagliare le cellule del tessuto/incorporate in 5 sezioni di 1 μm ± utilizzando un microtomo, montare le sezioni su vetrini elettrostaticamente adesivo e li asciugare durante la notte a TA.
Nota: Le diapositive possono essere conservate a RT sotto essiccazione fino a 3 mesi. - Porre i vetrini in un portavetrini e cuocere i vetrini di tessuto montato in un forno ad aria circolanti a 60 ° C per 1 h prima di eseguire il test.
Nota: Utilizzare le diapositive immediatamente o conservarli a RT con materiale essicante per fino a 1 settimana. Conservazione prolungata può provocare degradazione del RNA.
- Tagliare le cellule del tessuto/incorporate in 5 sezioni di 1 μm ± utilizzando un microtomo, montare le sezioni su vetrini elettrostaticamente adesivo e li asciugare durante la notte a TA.
- Tessuti
-
Preparazione del materiale
- Impostare il forno di ibridazione a 40 ° C. Accuratamente bagnato l'umidificazione carta e rimuovere qualsiasi residuo di dH2O. Inserire la carta nel vassoio di controllo umidità e inserire il vassoio del forno di ibridazione per preriscaldare per almeno 30 min prima dell'uso.
-
Preparazione dei reagenti
- Riempire due piatti di agente con 200 mL di xilene di compensazione e riempire due piatti di colorazione con 200 mL di 100% EtOH, che verrà utilizzato per Sparaffinatura delle sezioni.
- Preparare 200 mL di reagente di reperimento del target commercialmente disponibile 1 x aggiungendo 180 mL di dH2O 20 mL di reagente di recupero target x 10. Posizionare due portavetrini in un piroscafo. Riempire un portavetrini con 200 mL di reagente di reperimento del target: 1x e riempire altri portavetrini con 200 mL di dH2Heat O. entrambe le soluzioni a bollore utilizzando il vapore.
- Preriscaldato caldo 50 x tampone di lavaggio a 40 ° C per 10 – 20 min preparare 3 L di tampone di lavaggio 1X diluendo 60 mL di tampone di lavaggio x 50 con 2,94 L del dH2O.
- In una cappa, preparare ematossilina di Gill 50% controcolorazione soluzione aggiungendo 100 mL di ematossilina di Gill a 100 mL di dH2O. In cappa, preparazione del reagente azzurrante (0,02% (p/v) acqua ammoniacale) aggiungendo 1,43 mL di idrossido di ammonio 28 – 30% a 250 mL di dH2O.
- Preriscaldare la destinazione sonde a 40 ° C per 10 min prima dell'ibridazione della sonda e portare i reagenti di amplificazione (AMP 0 – 6) a RT.
2. RNA In Situ ibridazione Assay
-
Sparaffinatura e disidratazione
- Dopo la cottura come descritto nel passaggio 1.1.3.2 (punto di arresto opzionale 1), deparaffinizzare le sezioni in xilene per 5 minuti con agitazione. Deparaffinizzare nuovamente in xilene fresco per 5 min, disidratare in 100% EtOH per 2 min con agitazione e ripetere nuovamente in fresco 100% EtOH per 2 min.
- Aria secca le diapositive per 5 min a 60 ° C in un forno ad aria circolanti o a RT fino a quando non sono completamente a secco (punto di arresto opzionale 2).
-
Pretrattamenti del campione
- Incubare le sezioni con ~ 4 gocce di perossido di idrogeno di ready-to-use per 10 min a RT per placare l'attività perossidasica endogena. Decantare la soluzione dalle diapositive e sciacquarli due volte con dH2O.
- Incubare le sezioni con 200 mL del reagente di recupero di destinazione per 15-30 min a 100 ° C in un piroscafo.
Nota: Il tempo di incubazione può variare a seconda del tipo di tessuto. In questo protocollo, recupero di destinazione è stato effettuato per 15 min per campioni tumorali e palline delle cellule. - Decantare la soluzione dalle diapositive, lavare due volte con dH2O, tuffo in 100% EtOH per 3 min e a secco le diapositive a 60 ° C in un forno ad aria circolanti o a RT fino a quando non è completamente asciutto.
- Disegnare una barriera idrofoba intorno alla sezione utilizzando una penna idrofobica, circa 0.75 x 0.75 pollici2.
Nota: Non è consigliabile disegnare una barriera più piccola. Per grandi sezioni, una più grande barriera dovrà essere disegnata. - Lasciate che la barriera asciugare completamente per 1 min, o lasciare durante la notte la RT (punto di arresto opzionale 3).
- Porre i vetrini in un portavetrini e posizionare il supporto per diapositive nella barra di controllo di umidità. Aggiungere ~ 4 gocce di proteasi III a ogni diapositiva e li Incubare per 15 – 30 min a 40 ° C in forno ibridazione per digestione delle proteine.
Nota: Il tempo di incubazione può variare a seconda del tipo di tessuto. In questo protocollo, digestione della proteasi è stata effettuata per 30 min per campioni tumorali e 15 min per palline delle cellule. - Decantare la soluzione dalle diapositive e sciacquarli due volte con dH2O.
-
L'ibridazione della sonda
- Aggiungi ~ 4 gocce della soluzione appropriata sonda di ready-to-use per coprire l'intera sezione. Se si utilizza più grandi sezioni, aggiungere ~ 5-6 gocce.
- Ibridare le sonde per 2 ore a 40 ° C in forno l'ibridazione. Decantare la soluzione dalle diapositive e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere la procedura di lavaggio in questo passaggio.
-
Amplificazione del segnale
- Incubare le sezioni con ~ 4 gocce di AMP 0 per ogni diapositiva a 40 ° C in forno per 30 min. decantare la soluzione di ibridazione e lavare i vetrini in 200 mL di tampone di lavaggio 1x 1 per 2 min a RT con agitazione occasionale. Ripetere la procedura di lavaggio in questo passaggio.
- Incubare le sezioni con ~ 4 gocce di 1 AMP per ogni diapositiva a 40 ° C in forno per 15 min. decantare la soluzione di ibridazione e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere il lavaggio.
- Incubare le sezioni con ~ 4 gocce di AMP 2 per ogni diapositiva a 40 ° C in forno per 30 min. decantare la soluzione di ibridazione e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere il lavaggio.
- Incubare le sezioni con ~ 4 gocce di 3 AMP per ogni diapositiva a 40 ° C in forno per 30 min. decantare la soluzione di ibridazione e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere il lavaggio.
- Incubare le sezioni con ~ 4 gocce di 4 AMP per ogni diapositiva a 40 ° C in forno per 15 min. decantare la soluzione di ibridazione e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere il lavaggio.
- Incubare le sezioni con ~ 4 gocce di 5 AMP per vetrino a temperatura ambiente per 30 min. decantare la soluzione e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere il lavaggio.
- Incubare le sezioni con ~ 4 gocce di 6 AMP per vetrino a temperatura ambiente per 15 min. decantare la soluzione e lavare i vetrini in 200 mL di tampone di lavaggio per 2 min a RT con agitazione occasionale: 1x. Ripetere il lavaggio.
-
Rilevamento del segnale
- Preparare il colorante Fast Red soluzione di lavoro. Per una diapositiva con una barriera di 0.75 x 0.75 pollici2 , aggiungere 2 μL di colorante Fast Red-B 120 μL di Fast Red-A in una provetta e mescolare bene.
Nota: A seconda delle dimensioni della barriera idrofoba e il numero di diapositive, volumi della soluzione di lavoro Fast Red variano. - Decantare il liquido in eccesso dalle diapositive e aggiungere il colorante Fast Red soluzione alle diapositive di lavoro. Incubare i vetrini per 10 min a RT nel vassoio con una copertura per evitare l'esposizione alla luce.
Nota: Utilizzare la soluzione entro 5 min di preparazione di lavoro Fast Red e non esporlo alla luce diretta del sole o luce UV. - Decantare la soluzione Fast Red e sciacquare i vetrini due volte con acqua di rubinetto. Porre i vetrini in un portavetrini.
- Preparare il colorante Fast Red soluzione di lavoro. Per una diapositiva con una barriera di 0.75 x 0.75 pollici2 , aggiungere 2 μL di colorante Fast Red-B 120 μL di Fast Red-A in una provetta e mescolare bene.
-
Colorazione di contrasto
- Colorante di contrasto le sezioni di tessuto con soluzione di ematossilina di Gill del 50% per 2 min a RT. Wash le diapositive con acqua di rubinetto e ripetere questa operazione più volte fino a quando le diapositive sono chiare, mentre le sezioni rimangono viola. Immergere i vetrini in acqua ammoniaca 0,02% per brunitura (dip 2 – 3 volte). Sostituire l'acqua di ammoniaca con l'acqua del rubinetto e lavare i vetrini 3 – 5 volte.
-
Montaggio a slitta
- A secco le diapositive in un forno ad aria circolanti a 60 ° C per 15 minuti o a temperatura ambiente fino a quando non è completamente asciutto. Posto 1-2 gocce di reagente di montaggio su ogni diapositiva e luogo coprioggetto sopra ogni sezione. Evitare qualsiasi intrappolamento di bolle d'aria. Asciugare i vetrini per almeno 5 min.
Nota: Il substrato Fast Red è alcol-sensibili. Non disidratare i vetrini in alcool.
- A secco le diapositive in un forno ad aria circolanti a 60 ° C per 15 minuti o a temperatura ambiente fino a quando non è completamente asciutto. Posto 1-2 gocce di reagente di montaggio su ogni diapositiva e luogo coprioggetto sopra ogni sezione. Evitare qualsiasi intrappolamento di bolle d'aria. Asciugare i vetrini per almeno 5 min.
-
Visualizzazione
- Osservare i vetrini con un microscopio a campo chiaro standard.
Risultati
In situ di ibridazione analisi flusso di lavoro:
Il flusso di lavoro è descritto in Figura 1 e consiste di quattro parti: permeabilization delle cellule o tessuti con soluzioni di proteasi, ibridazione delle sonde a RNA, dell'obiettivo e il recupero di destinazione del segnale amplificazione e visualizzazione del segnale. Il segnale può anche essere quantificato utilizzando sistemi di software di imaging digitale o in modo semi-quantitativo basato sul numero di punti per cella. La procedura manuale descritta in Figura 2 è stato anche completamente automatizzata in sistemi di auto-colorazione commerciale.
Macchiatura rappresentativo per il rilevamento di giunzione esone (variante di splicing EGFRvIII): EGFRvIII è una variante del recettore del fattore di crescita epidermico che nasce da un'omissione genomic in-struttura degli esoni 2 e 7, che porta a segnalazione oncogenica costitutivamente attiva7 . L'analisi è stata usata per identificare lo stato di EGFRvIII in campioni tumorali di glioblastoma (GBM) FFPE. Singoli doppio-Z sonde sono stati progettati per coprire le giunzioni esone al fine di rilevare entrambi WT, mutante, o entrambe le trascrizioni (Figura 3A). Le sonde di WT EGFR si estendono le giunzioni di esoni 1 e 2 (E1/E2) o esoni 7 e 8 (E7/E8), mentre le sonde specifiche EGFRvIII estendono la giunzione degli esoni 1 e 8 (E1/E8). Una sonda comune su cui si estende la giunzione degli esoni 8 e 9 (E8/E9) è stato anche utilizzata per rilevare totale EGFR (trascrizioni sia WT ed EGFRvIII). Tutte le sonde sono stati poi utilizzate per determinare lo stato EGFR in campioni FFPE GBM. I due esempi rappresentativi, mostrati nella Figura 3B sono state prese da un più grande studio. Stato EGFR è stato confermato da un metodo indipendente, RT-PCR. Entrambe le sonde WT rilevato segnale in entrambi i campioni, che indica che i due campioni esprimono EGFR WT (Figura 3B). Tuttavia, la sonda mutante ha mostrato solo rilevamento del segnale nel EGFRvIII + campione, confermando che questo esempio è davvero positivo per la variante di EGFRvIII (Figura 3B, pannelli a sinistra). Al contrario, la sonda mutante non ha rilevato il segnale nel EGFRvIII-campione (Figura 3B, pannelli di destra). Presi insieme, questi risultati dimostrano che il dosaggio di giunzione esone può identificare lo stato di EGFRvIII in campioni tumorali GBM FFPE.
Rappresentante di colorazione per le destinazioni di corti:
Il CDR3, o complementari regione determinante 3, è un dominio altamente variabile in ricevitori a cellula T. In genere, la sequenza di CDR3 sono piuttosto brevi; ad esempio, le sequenze α e β CDR3 dalla linea a cellula T di Jurkat sono 51 e 48 nucleotidi di lunghezza, rispettivamente (Figura 4A). Per identificare le sequenze CDR specifiche espresse in cellule Jurkat, sonde antisenso per CDR3 α e β che si esprimono nella linea Jurkat T-cellulare sono stati generati, oltre alle sonde di senso per CDR3 α e β per servire come controllo negativo sonde. Tutte le sonde poi sono stata esaminate in cellule Jurkat FFPE-preparato con il dosaggio. Macchiatura robusta è stata osservata con le sonde anti-senso per entrambi CDR3 α e β nelle cellule Jurkat, considerando che senso sonde non rilevato poco o nessun segnale (Figura 4B). Questi risultati dimostrano la capacità del saggio breve destinazione di discernere tra altamente variabile ma brevi sequenze di CDR per cloni a cellula T del ricevitore.
Rappresentante di macchiatura per punto mutazione EGFR L858R:
Mutazione puntiforme sonde sono state sviluppate per rilevare variazioni di singolo nucleotide e piccole inserzioni o delezioni (INDELs) nel contesto del tumore. Figura 5A dimostra la capacità per il rilevamento in situ della punto mutazione EGFR L858R (2573T > G). Due sonde sono stati progettati: uno per rilevare la L858R mutato sequenza EGFR e un altro per rilevare la sequenza di EGFR L858 WT. Entrambe le sonde sono state testate in due linee cellulari FFPE-preparato: H2229, che esprime solo EGFR L858 WT; e H1975, che è eterozigote per la mutazione di EGFR L858R. La sonda mutante L858R rilevato segnale solo nella linea cellulare H1975, ma non nella linea cellulare H2229. Tuttavia, la sonda WT rilevato segnale in entrambe le linee cellulari. Allo stesso modo, figura 5B Visualizza in situ rilevazione di mutazione di punto G12A KRAS (35 G > C). Due sonde sono stati progettati per rilevare le sequenze di KRAS G12A MT e KRAS G12 WT e quindi testati sulla linea cellulare HuT78 (che esprime solo KRAS G12 WT) e la linea cellulare SW116 (che è eterozigote per la mutazione di KRAS G12A). Mentre la sonda KRAS G12 WT rilevato segnale in entrambe le linee cellulari, la sonda di KRAS G12A rilevato solo segnale nella linea cellulare SW116. Presi insieme, questi dati dimostrano la capacità tecnica dell'analisi di mutazione di punto nella rilevazione di polimorfismi a singolo nucleotide nel contesto delle cellule e dei tessuti.
Rappresentante di macchiatura per il test automatizzato in situ :
Analisi automatizzate consentono un maggior numero di campioni deve essere eseguito in modo più affidabile, riducendo al minimo la variabilità inter-utente e hands-on tempo e generando risultati riproducibili in modo coerente. Di conseguenza, è stata sviluppata una versione automatizzata del test. Per dimostrare la colorazione automatica con questa analisi, rilevamento della variante della giuntura METΔ14 è stato esaminato. Questa variante è il risultato dell'esone 14 del gene MET essere saltato durante il pre-mRNA che impiomba, che conduce all'attivazione costitutiva e trasformazione oncogena del recettore MET8,9. Per rilevare in modo specifico la variante di METΔ14, sono stati progettati due sonde di giunzione esone: uno su cui si estende la giunzione degli esoni 13 e 15 (E13/E15) per rilevare la trascrizione variante METΔ14 e l'altro che attraversa la giunzione degli esoni 14 e 15 (E14/E15) per rilevare il WT MET trascrizione (Figura 6A). Entrambe le sonde sono state poi testate in 2 linee cellulari di FFPE-preparato: H596, che esprime la variante di METΔ14, e A549, che esprime il gene WT MET. Entrambe le sonde hanno mostrato i modelli di espressione reciprocamente esclusiva, con la sonda E13/E15 solo rilevamento segnale nelle cellule H596 e la sonda E14/E15 solo rilevamento segnale nelle cellule A549 (figure 6B e 6C). Infine, la sonda per dapB ha mostrato nessun segnale, che indica nessun segnale di fondo (figure 6B e 6C). Nel complesso, questi dati dimostrano la rilevazione specifica del MET variante METΔ14 in situ utilizzando il test automatizzato BaseScope.
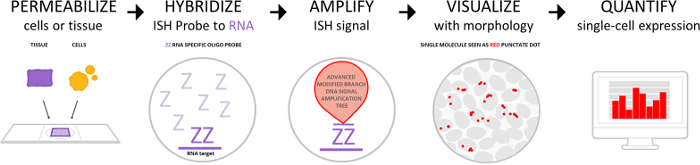
Figura 1 : Il flusso di lavoro test. Il flusso di lavoro è costituito da 4 fasi principali: pretrattamento per permeabilize cellule o tessuti, sonda di ibridazione a target RNA, amplificazione del segnale e rilevamento del segnale di visualizzazione al microscopio in campo chiaro o fluorescenti. Singoli punti possono essere quantificate utilizzando una piattaforma di analisi di immagine digitale. Clicca qui per visualizzare una versione più grande di questa figura.
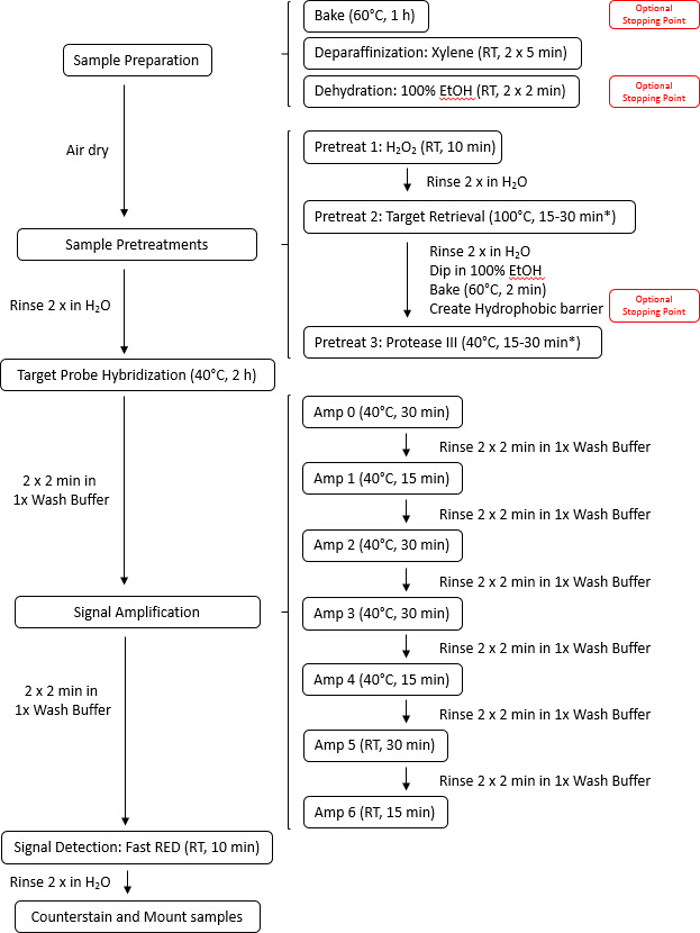
Figura 2 : Illustrazione del protocollo saggio manuale. Il dosaggio intero può essere completato in 9 h. pretrattamento volte possono variare a seconda del tipo di tessuto, quindi si consiglia di consultare l'Appendice A del manuale per consigli di pretrattamento del tessuto per quanto riguarda il tempo di incubazione. Clicca qui per visualizzare una versione più grande di questa figura.
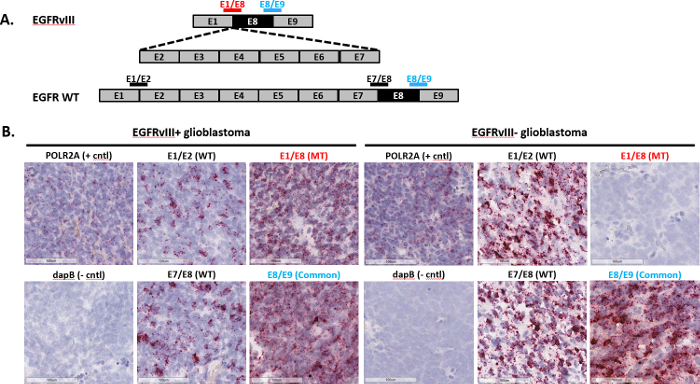
Figura 3 : Immagini rappresentative di rilevamento giunzione esone. (A) mostrato qui sono l'organizzazione di essone per EGFR WT ed EGFRvIII trascrizioni e un disegno schematico raffigurante sonde di giunzione esone double-Z che congiungono il bivio per rilevare EGFR WT o EGFRvIII. (B) dell'esone giunzione analisi è stata effettuata su due campioni di glioblastoma FFPE utilizzando le sonde elencate in (A), così come un dapB sonda di controllo, POLR2A e sonda di controllo negativo, positivo. Clicca qui per visualizzare una versione più grande di questa figura.
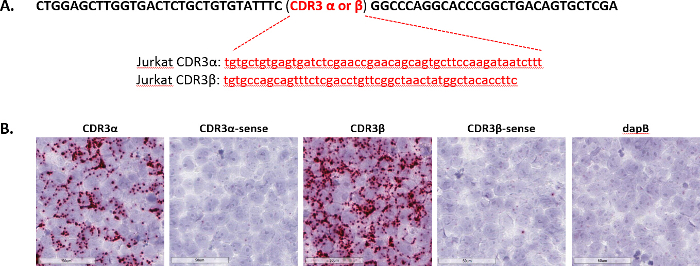
Figura 4 : Immagini rappresentative di destinazione breve sequenza rilevamento. (A) mostrato qui sono sequenze CDR3 in cellule Jurkat. La sequenza in nero è una sequenza comune di accompagnamento e la sequenza in rosso è unica a CDR3α o CDR3β. Sonde di sequenza bersaglio breve singolo doppio-Z sono stati progettati contro queste sequenze. (B), il saggio breve destinazione è stata eseguita su cellule Jurkat preparate come un pellet cellulare FFPE utilizzando sonde anti-senso o senso targeting per le sequenze in (A), e dapB è stato usato come controllo negativo. Clicca qui per visualizzare una versione più grande di questa figura.
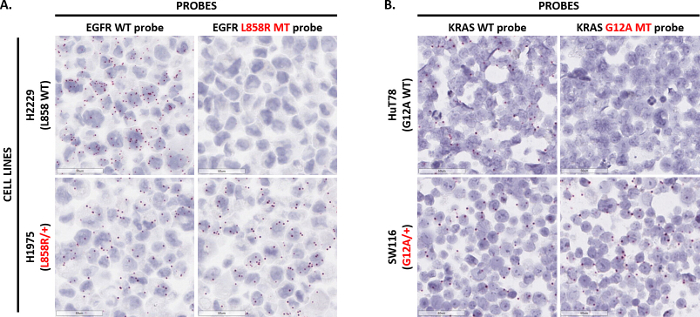
Figura 5 : Immagini rappresentative di rilevazione di mutazione di punto. (A) l'analisi è stata effettuata su H2229 (omozigoti per EGFR L858) e cellule H1975 (eterozigotiche per la mutazione di EGFR L858R) preparate come un pellet cellulare FFPE utilizzando sonde singola mutazione di punto doppio-Z targeting per la sequenza di EGFR L858 WT o EGFR L858R sequenza mutata. (B) l'analisi di mutazione di punto è stata eseguita su cellule HuT78 (omozigoti per KRAS G12A) e SW116 cellule (eterozigotiche per la mutazione di KRAS G12A) preparate come un pellet cellulare FFPE utilizzando sonde singola mutazione di punto doppio-Z targeting per la sequenza di KRAS G12 WT o Sequenza di G12A KRAS mutato. Clicca qui per visualizzare una versione più grande di questa figura.
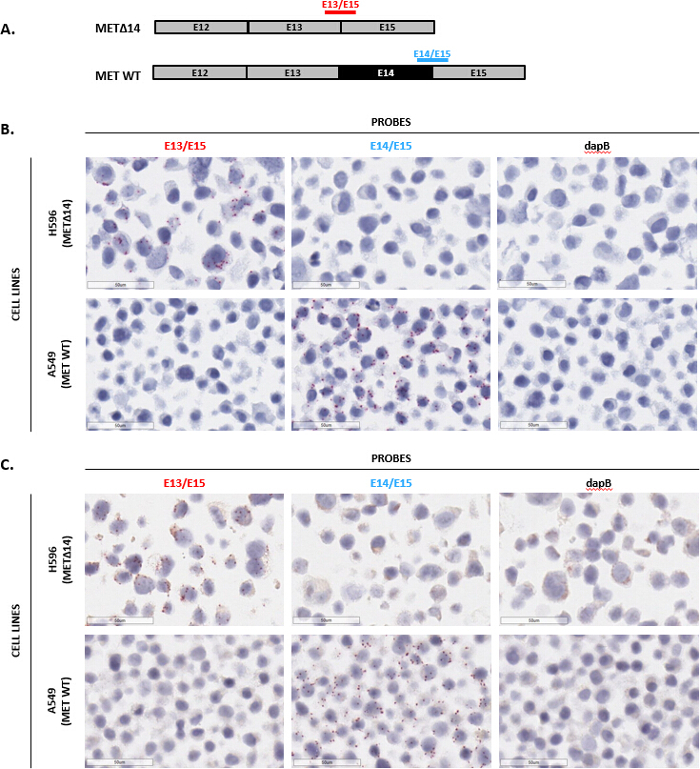
Figura 6 : Immagini rappresentative di colorazione automatica con il dosaggio di giunzione esone. (A) mostrato qui è l'organizzazione di essone per trascrizioni incontrato WT e METΔ14 e un disegno schematico raffigurante sonde di giunzione singola doppio-Z esone che congiungono il bivio per rilevare MET WT o METΔ14. (B) e (C), il dosaggio di giunzione esone automatizzata è stata eseguita su cellule H596 (che esprimono METΔ14) e cellule A549 (esprimendo incontrato WT) preparate come un pellet cellulare FFPE utilizzando le sonde elencate in (A), così come controllo negativo sonda dapB. Clicca qui per visualizzare una versione più grande di questa figura.
| Giunzione esone | Breve sequenza | Mutazione puntiforme |
| Splice variante/isoforma | Sequenze tra 50 e 300 nt | Mutazione puntiforme |
| RNA circolare (circRNA) | Sequenze altamente omologhe | Breve indel |
| Fusione genica | Sequenza di CDR3 per cloni TCR | Gene di editing |
| Knockout del gene (KO) | Pre-miRNA | |
| Small nucleolar RNA (snoRNA) | ||
| Gene di editing |
Tabella 1: applicazioni della in situ test. Ci sono 3 categorie principali per le applicazioni di questo test: giunzione esone, destinazione breve sequenza e mutazione di punto. Alcuni esempi di applicazione specifica per ogni categoria sono elencati in ogni colonna.
Discussione
In questo rapporto, il protocollo di dosaggio ISH romanzo e le sue applicazioni sono state discusse in dettaglio. Il test consente la visualizzazione diretta delle giunzioni esone, sequenze altamente omologhe e breve-destinazione e mutazioni puntiformi nel contesto del tessuto. Il dosaggio è basato su tecnologia RNAscope5 ed è quindi in grado di rivelazione della singola molecola. Tuttavia, a causa di un sistema di amplificazione avanzate, il segnale può essere rilevato con le sonde che contiene appena una coppia di doppio-Z o con una lunghezza di modello di destinazione di soli 50 nucleotidi. Poiché le sonde possono essere breve come un singolo doppio-Z di lunghezza, questo consente il rilevamento di giunzioni esone, sequenze altamente omologhe e breve-destinazione e mutazioni puntiformi (tabella 1).
Per la buona riuscita del dosaggio, sono disponibili numerosi consigli tecnici. In primo luogo, tessuti dovrebbero essere risolto in fresco formalina neutra tamponata al 10% (NBF) a temperatura ambiente per 16 – 32 h10. Underfixation (< h 16) o overfixation (> 32 h) potrebbe compromettere le prestazioni del dosaggio e può richiedere ulteriore ottimizzazione. In secondo luogo, per garantire un controllo ottimale della temperatura e l'umidità, che sono necessari per l'amplificazione del segnale e l'ibridazione sonda robusta, la diapositiva forno l'ibridazione e sistema di elaborazione deve essere utilizzata per la procedura di protocollo 2.3 a 5 (sonda di pretrattamento, di proteasi ibridazione, amplificazione del segnale e rilevamento del segnale). In terzo luogo, l'eccesso residuo buffer dovrebbe essere correttamente decantato prima di ogni passaggio in tutto il protocollo (ma non tanto che le sezioni di tessuto a secco). Se le diapositive si asciugano, si svilupperà significativo segnale aspecifico. In quarto luogo, a seconda del tipo di tessuto, pretrattamento ottimizzazione può essere necessario. Utilizzando la proteasi sbagliata o l'esecuzione per un tempo non ottimale può comportare in sotto - o sovra - digestion e influenzerà negativamente il segnale. Infine, è importante eseguire sempre i controlli positivi e negativi con i puntali. Sonde di controllo negativo verificare che non ci è segnale di sfondo, e sonde di controllo positivo assicurano che il test è stato fatto correttamente e che la qualità di RNA nel campione è ottima per l'interpretazione dei risultati di test sonda. Se non ci è segnale con la sonda di controllo positivo, allora è probabile che la qualità di RNA nel campione non ottimale e un segnale non può essere visto con la sonda di prova.
Oltre l'analisi manuale, la possibilità di eseguire il test con coloratori automatici fu dimostrata (Figura 6). Questo saggio ISH automatizzato produce un elevato rapporto segnale-rumore ed è applicabile per le stesse applicazioni come illustrato nella tabella 1; Tuttavia, i vantaggi di un test automatizzato includere standardizzazione delle metodiche, minimizzazione della variabilità inter-utente e hands-on tempo e indennità per high throughput screening dei campioni di tessuto in modo affidabile.
Mentre qRT-PCR e immunohistochemistry (IHC) consentire il rilevamento delle varianti di splicing (in particolare EGFRvIII), nei campioni clinici FFPE, queste tecniche possono mancare la necessaria specificità e non offrono la risoluzione spaziale della giunzione: spaccato espressione Variant, rispettivamente11,12. Un vantaggio fondamentale del dosaggio in questo protocollo è sua visualizzazione altamente sensibile e specifica delle giunzioni della giuntura, preservando il contesto morfologico del tessuto. Qui, capacità del dosaggio mira giunzioni esone unica per diverse varianti di splicing, tra cui EGFRvIII e METΔ14, è stata dimostrata (figure 3 e 6). Inoltre, il dosaggio è stato indicato per rilevare la variante di splicing AR-V7 nel carcinoma della prostata, molteplici isoforme di ErbB4 nel cervello e la conferma di knockout di circolare RNA Cdr1as nel topo cervello3,13,14.
Rilevamento della sequenza bersaglio breve di questo saggio ISH consente per la visualizzazione di sequenze di RNA più breve 50 nucleotidi di lunghezza, come dimostrato dalla rilevazione dei CDR3 sequenze derivate da cellule Jurkat (Figura 3). L'analisi può anche rilevare sequenze di geni che sono altamente omologhe ad altri membri della famiglia o specie, come illustrato da Revêchon et al., che utilizzato il saggio breve destinazione per rilevare progerin umana espressa in mouse bianco del tessuto adiposo sottocutaneo15. Inoltre, small nucleolar RNA (snoRNA), CRISPR-mediata del gene editing e precursore-microRNA può essere rilevato in situ con il saggio breve destinazione. Più recentemente, Fu et al combinato questo test ISH con IHC per identificare le celle precise nella retina che esprimono i pre-miRNA mir125b16.
Mutazione di profilatura in tumori è fondamentale per studiare la progressione dei tumori e per lo sviluppo di terapie mirate. Mentre l'analisi di mutazione può essere raggiunto tramite sequenziamento ad alte prestazioni, questa tecnologia non può risolvere completamente intratumoral eterogeneità o link di alterazioni genetiche con morfologia cellulare17,18. La rilevazione di mutazioni puntiformi che usando questa analisi ISH consente per distinzione di sequenze di RNA bersaglio a una risoluzione di singolo-base, come convalidato tramite la rilevazione delle variazioni di singolo nucleotide L858R di EGFR e KRAS G12A in linee cellulari (Figura 5). Inoltre, Baker et al. usato l'analisi di mutazione di punto di destinazione più mutazioni in oncogeni BRAF, KRAS e PIK3CA nel cancro colorettale18. Erano in grado di identificare e mappare spazialmente subcloni rari mutante delle cellule del tumore, in ultima analisi, mostrando come essi contribuiscono all'eterogeneità intra-tumorale.
In sintesi, è stata sviluppata un'analisi di RNA ISH specializzata. Questa metodologia consente il rilevamento di varianti di splicing, brevi sequenze e mutazioni in situ. È sensibile, specifico, quantificabili e adattabile per prestazioni sia di metodi manuali che con coloratori automatici.
Divulgazioni
Tutti gli autori sono impiegati da Advanced Cell Diagnostics, Inc.
Materiali
| Name | Company | Catalog Number | Comments |
| HybEZ Oven (110 or 220 VAC) or HybEZ II Oven (110 or 220V) | ACD | 310010 or 310013 (HybEZ™), 321710 or 321720 (HybEZ™ II) | |
| HybEZ Humidity Control Tray (with lid) | ACD | 310012 | |
| ACD EZ-Batch Slide Rack (20 slide capacity) 1 rack 310017 | ACD | 310017 | |
| HybEZ Humidifying Paper | ACD | 310015 | |
| ImmEdge Hydrophobic Barrier Pen (required) | Vector Laboratory | H-4000 | |
| SuperFrost Plus Slides (required) | Fisher Scientific | 12-550-15 | |
| 10% neutral-buffered formalin (NBF) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paraffin wax | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microtome | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Gill’s Hematoxylin I | American Master Tech Scientific/MLS | HXGHE1LT | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Xylene | Fisher Scientific/MLS | X3P-1GAL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Vertical 24 Slide Rack | American Master Tech Scientific/MLS | LWSRA24 | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Staining Dishes | American Master Tech Scientific/MLS | LWT4457EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tissue-Tek Clearing Agent Dishes, xylene resistant | American Master Tech Scientific/MLS | LWT4456EA | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| 100% alcohol (EtOH) | American Master Tech Scientific/MLS | ALREACS | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| VectaMount Permanent Mounting Medium (required) | Vector Labs | H-5000 | |
| Cover Glass, 24 mm x 50 mm | Fisher Scientific/MLS | 12-545-F | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Ammonium hydroxide, 28–30% | Sigma-Aldrich/MLS | 320145-500mL | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Carboy (>3L) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Oster Steamer Model 5712, Black and Decker Steamer HS3000, or the Braun Multiquick FS 20 Steamer | / | / | |
| Digital thermometer | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Water bath or incubator, capable of holding temperature at 40 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Pipettors and tips, 1–1,000 μL | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Distilled water | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Tubes (various sizes) | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Fume hood | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Graduated cylinder | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Parafilm | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Paper towel or absorbent paper | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microcentrifuge | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Microscope and accessories | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Drying oven, capable of holding temperature at 60 +/– 1 °C | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Formaldehyde | MLS | / | MLS Major Laboratory Supplier in North America. For other regions, please check Catalog Numbers with your local lab supplier. |
| Histogel | Fisher Scientific/MLS | 22-110-678 | |
| BaseScope Reagent Kit - RED | Advanced Cell Diagnostics | 322900 | |
| BaseScope Hs-EGFR-E1E2 | Advanced Cell Diagnostics | 701701 | |
| BaseScope Hs-EGFR-E1E8 | Advanced Cell Diagnostics | 701711 | |
| BaseScope Hs-EGFR-E7E8 | Advanced Cell Diagnostics | 701721 | |
| BaseScope Hs-EGFR-E8E9 | Advanced Cell Diagnostics | 701731 | |
| BaseScope Hs-MET-E14E15 | Advanced Cell Diagnostics | 701811 | |
| BaseScope Hs-MET-E13E15 | Advanced Cell Diagnostics | 701801 | |
| BaseScope Hs-KRAS-G12A | Advanced Cell Diagnostics | 705491 | |
| BaseScope Hs-KRAS-G12-nt35WT | Advanced Cell Diagnostics | 705531 | |
| BaseScope Hs-EGFR-L858R | Advanced Cell Diagnostics | 705451 | |
| BaseScope Hs-EGFR-L858WT | Advanced Cell Diagnostics | 705461 | |
| BaseScope Control Probe Pack Human | Advanced Cell Diagnostics | 322975 |
Riferimenti
- Bolha, L., Ravnik-Glavač, M., Glavač, D. Circular RNAs: Biogenesis, Function, and a Role as Possible Cancer Biomarkers. International Journal of Genomics. 2017, 6218353 (2017).
- Yamada, A., Yu, P., Lin, W., Okugawa, Y., Boland, C. R., Goel, A. A RNA-Sequencing approach for the identification of novel long non-coding RNA biomarkers in colorectal cancer. Scientific Reports. 8 (1), 575 (2018).
- Erben, L., He, M. X., Laeremans, A., Park, E., Buonanno, A. A Novel Ultrasensitive In Situ Hybridization Approach to Detect Short Sequences and Splice Variants with Cellular Resolution. Molecular Neurobiology. , (2017).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods in Molecular Biology. 461, 675-686 (2008).
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Paraffin processing of tissue. Protocols Online Available from: https://www.protocolsonline.com/histology/sample-preparation/paraffin-processing-of-tissue/ (2016)
- An, Z., Aksoy, O., Zheng, T., Fan, Q. W., Weiss, W. A. Epidermal growth factor receptor and EGFRvIII in glioblastoma: signaling pathways and targeted therapies. Oncogene. , (2018).
- Frampton, G. M., et al. Activation of MET via diverse exon 14 splicing alterations occurs in multiple tumor types and confers clinical sensitivity to MET inhibitors. Cancer Discovery. 5 (8), 850-859 (2015).
- Awad, M. M., et al. MET Exon 14 Mutations in Non-Small-Cell Lung Cancer Are Associated With Advanced Age and Stage-Dependent MET Genomic Amplification and c-Met Overexpression. Journal of Clinical Oncology. 34 (7), 721-730 (2016).
- Hammond, M. E., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for immunohistochemical testing of estrogen and progesterone receptors in breast cancer. Archives of Pathology and Laboratory Medicine. 134 (6), 907-922 (2010).
- Gan, H. K., Cvrljevic, A. N., Johns, T. G. The epidermal growth factor receptor variant III (EGFRvIII): where wild things are altered. Federation of European Biochemical Societies Journal. 280 (21), 5350-5370 (2013).
- Wheeler, S. E., Egloff, A. M., Wang, L., James, C. D., Hammerman, P. S., Grandis, J. R. Challenges in EGFRvIII detection in head and neck squamous cell carcinoma. Public Library of Science One. 10 (2), e0117781 (2015).
- Zhu, Y., et al. Novel Junction-specific and Quantifiable In Situ Detection of AR-V7 and its Clinical Correlates in Metastatic Castration-resistant Prostate Cancer. European Urology. , (2017).
- Piwecka, M., et al. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function. Science. , (2017).
- Revêchon, G., et al. Rare progerin-expressing preadipocytes and adipocytes contribute to tissue depletion over time. Scientific Reports. 7 (1), 4405 (2017).
- Fu, Y., et al. Functional ectopic neuritogenesis by retinal rod bipolar cells is regulated by miR-125b-5p during retinal remodeling in RCS rats. Scientific Reports. 7 (1), 1011 (2017).
- Yates, L. R., et al. Subclonal diversification of primary breast cancer revealed by multiregion sequencing. Nature Medicine. 21 (7), 751-759 (2015).
- Baker, A. M., et al. Robust RNA-based in situ mutation detection delineates colorectal cancer subclonal evolution. Nature Communications. 8 (1), 1998 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon