Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Messung der Spin-Lattice Relaxation Magnetfeldabhängigkeit von hyperpolarisiertem [1-13C]pyruvat
In diesem Artikel
Zusammenfassung
Wir präsentieren ein Protokoll zur Messung der Magnetfeldabhängigkeit der Spin-Gitter-Entspannungszeit von 13C-angereicherten Verbindungen, die mittels dynamischer kernatischer Polarisation mit schneller feldzyklusweiseer Entspannungsometrie hyperpolarisiert werden. Insbesondere haben wir dies mit [1-13C]pyruvat nachgewiesen, aber das Protokoll könnte auf andere hyperpolarisierte Substrate ausgedehnt werden.
Zusammenfassung
Die grundlegende Grenze für in vivo-bildgebende Anwendungen von hyperpolarisierten 13C-angereicherten Verbindungen sind ihre endlichen Spin-Gitter-Entspannungszeiten. Verschiedene Faktoren beeinflussen die Entspannungsraten, wie Pufferzusammensetzung, Lösungs-pH-Wert, Temperatur und Magnetfeld. In letzter Zeit kann die Spin-Gitter-Entspannungszeit an klinischen Feldstärken gemessen werden, aber in unteren Feldern, wo diese Verbindungen vom Polarisator abgegeben und zum MRT transportiert werden, ist die Entspannung noch schneller und schwer zu messen. Um ein besseres Verständnis der Magnetisierungsmenge zu haben, die während des Transports verloren ging, verwendeten wir eine schnelle feld-zyklus-entspannende Entspannungsometrie mit einer Magnetresonanzdetektion von 13C-Kernen bei 0,75 T, um die Kernspinresonanzdispersion der Spin-Gitter-Entspannungszeit von hyperpolarisiertem [1-13C]pyruvat. Die auflösungsdynamische kernatikale Polarisation wurde verwendet, um hyperpolarisierte Pyruvatproben in einer Konzentration von 80 mmol/L und physiologischen pH-Wert (7,8) zu produzieren. Diese Lösungen wurden schnell auf ein schnelles Feld-Zyklus-Relaxometer übertragen, so dass die Entspannung der Probenmagnetisierung als Funktion der Zeit mit einem kalibrierten kleinen Drehwinkel (3°-5°) gemessen werden konnte. Um die T1-Dispersion des C-1 von Pyruvat abzubilden, haben wir Daten für verschiedene Entspannungsfelder zwischen 0,237 mT und 0,705 T aufgezeichnet. Mit diesen Informationen haben wir eine empirische Gleichung ermittelt, um die Spin-Gitter-Entspannung des hyperpolarisierten Substrats innerhalb des genannten Bereichs von Magnetfeldern zu schätzen. Diese Ergebnisse können verwendet werden, um den Während des Transports verlorenen Magnetisierungsbetrag vorherzusagen und experimentelle Konstruktionen zu verbessern, um Signalverluste zu minimieren.
Einleitung
Magnetresonanzspektroskopische Bildgebung (MRSI) kann räumliche Karten von Metaboliten erzeugen, die durch spektroskopische Bildgebung erkannt werden, aber ihre praktische Anwendung wird oft durch ihre relativ geringe Empfindlichkeit eingeschränkt. Diese geringe Empfindlichkeit der in vivo Magnetresonanztomographie und Spektroskopie-Methoden ergibt sich aus dem geringen Grad der Kernmagnetisierung, der bei Körpertemperaturen und vernünftigen Magnetfeldstärken erreichbar ist. Diese Einschränkung kann jedoch durch den Einsatz der dynamischen kernatonischen Polarisation (DNP) überwunden werden, um die In-vitro-Magnetisierung flüssiger Substrate erheblich zu verbessern, die anschließend mit MRSI1,2 in den In-vivo-Stoffwechsel injiziert werden. , 3 , 4. DNP ist in der Lage, die Magnetisierung der meisten Kerne mit Nicht-Null-Kernspin zu verbessern und wurde verwendet, um in vivo MRSI-Empfindlichkeit von 13C-angereicherten Verbindungen wie Pyruvat5,6, Bicarbonat zu erhöhen 7,8, fumarate9, laktat10, glutamin11 ,und andere um mehr als vier Größenordnungen12. Seine Anwendungen umfassen Bildgebung der Gefäßerkrankung13,14,15, Organperfusion13,16,17,18, Krebs Nachweis1,19,20,21,22, Tumor-Staging23,24, und Quantifizierung des therapeutischen Ansprechens2 , 6 , 23 , 24 , 25 , 26.
Langsame Spin-Gitter-Entspannung ist wichtig für die In-vivo-Erkennung mit MRSI. Spin-Gitter-Entspannungszeiten(T1s) in der Größenordnung von zehn Sekunden sind für Kerne mit niedrigen gyromagnetischen Verhältnissen innerhalb kleiner Moleküle in Lösung möglich. Mehrere physikalische Faktoren beeinflussen den Energietransfer zwischen einem Kernspinübergang und seiner Umgebung (Gitter), was zu Entspannung führt, einschließlich der magnetischen Feldstärke, Temperatur und molekularen Konformation27. Die dipolare Entspannung wird in Molekülen für Kohlenstoffpositionen reduziert, ohne dass Direktprotonen angebracht sind, und die Deuteration von Auflösungsmedien kann die intermolekulare dipolare Entspannung weiter reduzieren. Leider haben deuterated Lösungsmittel begrenzte Fähigkeiten, um in vivo Entspannung zu erweitern. Erhöhte Entspannung von Carbonylen oder Carbonsäuren (wie Pyruvat) kann bei hohen Magnetfeldstärken durch chemische Verschiebungsanisotropie auftreten. Das Vorhandensein paramagnetischer Verunreinigungen aus dem Flüssigkeitsweg während der Auflösung nach der Polarisation kann zu einer schnellen Entspannung führen und muss mit Chelatoren vermieden oder beseitigt werden.
Für die Entspannung von 13C-haltigen Verbindungen auf niedrigen Feldern, bei denen die Spingitterentspannung deutlich schneller sein könnte, liegen nur sehr wenige Daten vor. Es ist jedoch wichtig, T1 bei niedrigen Feldern zu messen, um die Entspannung während der Vorbereitung des für die In-vivo-Bildgebung verwendeten Mittels zu verstehen, da die hyperpolarisierten Kontrastmittel in der Regel aus dem DNP-Gerät in der Nähe oder an der Erdoberfläche abgegeben werden. feld. Zusätzliche physikalische Faktoren wie 13C-angereicherte Substratkonzentration, Lösungs-pH-Wert, Puffer und Temperatur beeinflussen ebenfalls die Entspannung und wirken sich somit auf die Formulierung des Mittels aus. All diese Faktoren sind wesentlich für die Bestimmung der Schlüsselparameter bei der Optimierung des DNP-Auflösungsprozesses und die Berechnung der Größe des Signalverlustes, die beim Transport der Probe vom DNP-Gerät zum Bildmagneten auftritt.
Kernspinresonanzdispersionsmessungen (NMRD), d.h. T1-Messungen, als Funktion des Magnetfeldes werden typischerweise mit einem NMR-Spektrometer erfasst. Um diese Messungen zu erfassen, könnte eine Abschaltmethode verwendet werden, bei der die Probe zuerst aus dem Spektrometer geshuttlet wird, um sich an einem Feld zu entspannen, das durch seine Position im Randfeld des Magneten bestimmt wird28,29,30 und dann schnell wieder in den NMR-Magneten übertragen, um seine verbleibende Magnetisierung zu messen. Durch die Wiederholung dieses Prozesses an der gleichen Stelle im Magnetfeld, aber mit zunehmenden Entspannungsperioden kann eine Entspannungskurve erreicht werden, die dann analysiert werden kann, um T1zu schätzen.
Wir verwenden eine alternative Technik, die als schnelle Feldrad-Relaxometrie31,32,33 bekannt ist, um unsere NMRD-Daten zu erfassen. Wir haben ein kommerzielles Feld-Zyklus-Relaxometer (siehe Tabelle der Materialien) für T1 Messungen von Lösungen modifiziert, die hyperpolarisierte 13C-Kerne enthalten. Im Vergleich zur Shuttle-Methode ermöglicht das Field-Cycling diesem Relaxometer die systematische Erfassung von NMRD-Daten über einen kleineren Bereich von Magnetfeldern (0,25 mT bis 1 T). Dies wird durch eine schnelle Änderung des Magnetfeldes selbst erreicht, nicht durch die Probenposition im Magnetfeld. Daher kann eine Probe mit einer hohen Feldstärke magnetisiert, bei einer niedrigeren Feldstärke "entspannt" und dann durch Erfassung eines Freiinduktionszerfalls an einem festen Feld (und Larmor-Frequenz) gemessen werden, um das Signal zu maximieren. Das bedeutet, dass die Probentemperatur während der Messung gesteuert werden kann und die NMR-Sonde nicht an jedem Entspannungsfeld abgestimmt werden muss, was eine automatische Erfassung über den gesamten Magnetfeldbereich fördert.
Mit der Konzentration unserer Bemühungen auf die Auswirkungen der Abgabe und des Transports der hyperpolarisierten Lösungen bei niedrigen Magnetfeldern präsentiert diese Arbeit eine detaillierte Methodik zur Messung der Spin-Gitter-Entspannungszeit von hyperpolarisiertem 13C-Pyruvat mit Feld-Rad-Relaxometrie für Magnetfelder im Bereich von 0,237 mT bis 0,705 T. Die wichtigsten Ergebnisse dieser Methode wurden zuvor für [1-13C]pyruvat34 und 13C-angereichertes Natrium und Cäsiumbicarbonat35 vorgestellt, wo andere Faktoren wie die radikale Konzentration und der Auflösungs-pH-Wert auch untersucht.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Probenvorbereitung
ANMERKUNG: Die Schritte 1.1-1.8 werden nur einmal ausgeführt
- Bereiten Sie 1 ml Vorrat 13C-angereicherte Pyruvicsäurelösung, weit verbreitet für in vivo Forschung1,2,5,6, bestehend aus 15-mmol/L Triarylmethylradikal in [1- 13 C]pyruvicsäure (siehe Materialtabelle). Aliquots aus dieser Stammlösung werden für die Proben verwendet, die individuell polarisiert werden und anschließend bei verschiedenen Magnetfeldern einer Relaxometrie unterzogen werden. Eine Darstellung des [1-13C]pyruvicsäuremoleküls ist in Abbildung 1dargestellt.
- Klicken Sie auf der dynamischen Kernpolarisator-Software-Schnittstelle (siehe Tabelle der Materialien)auf die Cooldown-Taste, um die Temperatur des variablen Temperatureinsatzes (VTI) auf 1,4 K zu senken.
- Sobald der DNP die gewünschte Temperatur erreicht hat, laden Sie 10 l der Stammlösung in einen Probenbecher, öffnen Sie die Türöffnungen und stecken Sie den Becher mit einem speziell für diese Aufgabe entwickelten Einsteckstab in den VTI ein.
- Danach, schnell extrahieren Sie den Zauberstab und stellen Sie sicher, dass die Tasse freigegeben wird. Schließen Sie dann die Türder und fahren Sie mit den folgenden Schritten fort, während die Temperatur des VTI auf 1,4 K zurückgeht.
- Bereiten Sie den DNP auf einen Mikrowellen-Sweep vor, um die optimale HF-Frequenz für die Hyperpolarisation der Lagerlösung zu finden.
- Auf dem Computer, der das Spektrometer steuert (Teil des DNP-Systems), stellen Sie die Kommunikation zwischen dem Spektrometer und der DNP-Steuerungssoftware durch Doppelklick auf das HyperTerminal-Symbol her, das zuvor mit der entsprechenden seriellen Kommunikation konfiguriert war. rahmen.
- Sobald die Kommunikation hergestellt ist, starten Sie die RINMR-Software, geben Sie in ihre Befehlszeile ein. HYPERSENSENMR, und drücken Sie dann die Eingabetaste.
- Danach wird ein neues Fenster auf dem Bildschirm angezeigt und darauf die Nummer 1 (1) in das Feld Konfigurationsnummer eingegeben. Klicken Sie dann auf die Schaltfläche Konfiguration auswählen.
- Klicken Sie auf die Schaltfläche Mikrowelle sweep. Es wird ein kleines Fenster mit einem absteigenden Zähler von Sekunden gestartet, das anzeigt, dass das Spektrometer bereit ist und es auf periodische Triggersignale wartet, die von der DNP-Steuerungssoftware kommen, um die Polarisation zu testen.
- Wählen Sie in der DNP-Steuerungssoftware die Registerkarte Kalibrieren aus und klicken Sie auf die Schaltfläche Generieren.
- Geben Sie über das Kalibrierungs-Setup-Fenster die folgenden Informationen ein: Startfrequenz = 94,117 GHz, Endfrequenz = 94,137 GHz, Schrittgröße = 1 MHz, Schrittdauer = 300 s, Leistung = 50 mW, Flüssigheliumpegel = 65 % und Temperatur = 1,4 K.
- Klicken Sie auf die Schaltfläche Generieren, die das Setup-Fenster schließt und zur Registerkarte Kalibrieren zurückkehrt, auf der die Anzahl der Schritte und die Zeit angezeigt wird, die zum Ausführen des gewünschten Mikrowellen-Sweeps erforderlich ist.
- Sobald die gewünschte VTI-Temperatur erreicht ist, klicken Sie auf die Schaltfläche Aktivieren und starten Sie dann, um den Mikrowellen-Sweep-Prozess zu initialisieren.
- Am Ende des Mikrowellen-Sweeps, erholen Sie die Probe und notieren Sie die optimale Frequenz, wo die maximale Polarisation erreicht wird. Diese optimale Frequenz ist definiert als die Polarisationsfrequenz, die die maximale Polarisation liefert, wie in Abbildung 2dargestellt. Diese Frequenz wird zur Hyperpolarisierung aller Aliquots verwendet, die aus dieser spezifischen Stammlösung von Pyruvicsäure gewonnen werden.
- Bereiten Sie 250 ml Stammauflösungsmedium mit einer Lösung von 40-mmol/L Tris Basis, 50 mmol/L Natriumchlorid und 80-mlmol/L Natriumhydroxid in deionisiertem Wasser vor. Ethylendiamintetraessigsäure (EDTA) in einer Konzentration von 100 mg/L hinzufügen, um eine Metallionenkontamination zu beseitigen. Ähnlich wie bei der Pyruvicsäure-Stammlösung wird dieses Auflösungsmedium für alle verschiedenen Proben verwendet, die polarisiert werden. Genauere Informationen zu den verwendeten Chemikalien finden Sie in der Materialtabelle.
- Bereiten Sie auch 500 ml Stoffreinigungslösung vor, die aus 100 mg/L EDTA besteht, die in entionisiertem Wasser gelöst sind. Etwa 10 ml dieser Reinigungslösung werden nach jeder Polarisation verwendet, um den Auflösungsweg des DNP zu reinigen.
HINWEIS: Die Schritte 1.9-1.27 werden für jede einzelne Probe ausgeführt. - Kühlen Sie das DNP-Gerät auf 1,4 K zur Vorbereitung der Hyperpolarisierung einer [1-13C]pyruvicsäureprobe, indem Sie die Cooldown-Taste im DNP-Hauptfenster drücken.
- Wenn die für das Spektrometer verwendete Software bereits aktiv ist und Konfiguration 1 ausgewählt ist, fahren Sie mit den folgenden Schritten fort. Führen Sie andernfalls die Schritte 1.5.1 bis 1.5.3 aus, und fahren Sie dann mit den folgenden Schritten fort.
- Nachdem Sie überprüft haben, ob Konfiguration 1 im Fenster ausgewählt ist, in dem das DNP-Spektrometer gesteuert wird, klicken Sie auf die Schaltfläche Solid Build Up.
- Geben Sie den Dateinamen SSBuilupXXX ein, wobei "XXX" eine Zahl in der Reihenfolge der Dateien ist, die mit Aufbaudaten gespeichert sind. Diese Zahl wird von der Software automatisch erhöht. Klicken Sie dann auf OK. Ähnlich wie beim Mikrowellen-Sweep-Gehäuse wird ein kleines Fenster mit einem absteigenden Zähler von Sekunden gestartet, das anzeigt, dass das Spektrometer bereit ist und es auf periodische Triggersignale wartet, die von der DNP-Steuerungssoftware kommen, um die Polarisation zu testen. .
- Mit der pyruvicsäure - OX063 Stofflösung, die in Schritt 1.1 zubereitet wird, wiegen Sie 30 mg in einem Probenbecher ab.
- Wenn die gewünschte VTI-Temperatur erreicht ist (1,4 K) klicken Sie auf Sample einfügen, dann wählen Sie Normal Sample und dann auf Weiter. Setzen Sie den Becher gemäß den auf dem Bildschirm angezeigten Sicherheitsvorkehrungen in das kalte DNP-Gerät ein, indem Sie einen langen Zauberstab verwenden, der speziell für diese Aufgabe entwickelt wurde.
- Sobald der Becher eingefügt, der Zauberstab entfernt und die DNP-Türen geschlossen wurden, klicken Sie auf Weiter und dann auf Fertigstellen. An diesem Punkt senkt das Hyperpolarisatorsystem den Probenbecher auf die teilweise gefüllte Bestrahlungskammer (65%) mit flüssigem Helium.
- Warten Sie, bis die Temperatur auf 1.4K zurückgekehrt ist, und klicken Sie dann auf die Schaltfläche Polarisieren.
- Legen Sie im neuen Popup-Fenster den Frequenzwert in Schritt 1.6 auf den Frequenzwert fest, der aus dem Mikrowellen-Sweep erhalten wird. Legen Sie im selben Fenster auch die Leistung auf 50 mW und die Abtastzeit auf 300 s fest. Klicken Sie auf Weiter, aktivieren Sie das Kontrollkästchen Build-up-Überwachung aktivieren, und klicken Sie dann auf Fertig stellen.
HINWEIS: Sobald die Polarisation gestartet ist, erzeugt die DNP-Steuerungssoftware alle 300 s Triggersignale, um das Spektrometer anzuweisen, die Polarisation mit einem kleinen Spitzenwinkel zu beproben. Auf diese Weise fügt die Spektrometersoftware einen Beispielpunkt zu einer Solid-State-Magnetisierungskurve hinzu, die nun sowohl in der Spektrometersoftware als auch in der DNP-Steuerungssoftware unter der Registerkarte Polarisationsaufbauangezeigt wird. Nach der 4. Probe und jeder Probe danach passt die Spektrometersoftware die Kurve an eine exponentielle Wachstumsfunktion der Form an:
S = A *exp(-t/Tp) + y0
wobei A die Polarisationsamplitude ist, in beliebigen Einheiten, t die Abtastzeit, Tp die Polarisationszeitkonstante (beide in Sekunden) und y0 ein Offset. Basierend auf den angepassten Parametern berechnet die Software auch die bis zu diesem Zeitpunkt erreichte prozentuale Polarisation, die auch auf der Registerkarte Polarisationsstatus des DNP angezeigt wird. - Polarisieren, bis der Aufbau der Festkörpermagnetisierung mindestens 95% des Maximums (ca. eine Stunde) erreicht.
- Während die Probe polarisiert, bereiten Sie das Fast-Field-Cycling Relaxometer vor, wie in Abschnitt 2 unten erläutert.
- Wenn die gewünschte Polarisation erreicht ist, klicken Sie auf Auflösung ausführen und wählen Sie unter Methode Pyruvic Acid Test. Klicken Sie dann auf Weiter.
- Öffnen Sie nach den Anweisungen auf dem Bildschirm die DNP-Turmtüren und laden Sie die Heiz- und Druckkammer an der Oberseite des Geräts mit 4,55 ml des in Abschnitt 1.5 vorbereiteten Auflösungsmediums, um eine Konzentration von 80-mmol/L-Pyruvat auf bei einem pH-Wert von 7,75 °
- Positionieren Sie den erholenden Zauberstab in der richtigen Position, schließen Sie die Türer, und klicken Sie am Computer auf Weiter und dann auf Fertig. An diesem Punkt werden die Auflösungsmedien aufgeheizt, bis der Druck 10 bar erreicht.
- Sobald der 10 bar Druck erreicht ist, wird das gefrorene und hyperpolarisierte Pyruvat automatisch aus dem flüssigen Heliumbad gehoben, schnell vermischt und mit dem überhitzten Auflösungsmedium aufgetaut und durch einen Kapillarschlauch in einen birnenförmigen Kolben ausgestoßen. Während das hyperpolarisierte Pyruvat/Auflösungsmediengemisch ausgestoßen wird, wirbeln Sie den Kolben ständig, um eine homogene Mischung zu gewährleisten.
- Wenn die gesamte Mischung ausgeworfen ist, ziehen Sie schnell 1,1 ml der Flüssigkeit in eine Spritze, in ein vorgewärmtes (37 °C) NMR-Rohr mit 10 mm Durchmesser übertragen und schnell zum Feldrad-Relaxometer transportieren (siehe Schritt 2.2.12).
- Geben Sie das verbleibende Aliquot jeder Pyruvatauflösung in ein 0,55-T-Benchtop-NMR-Spektrometer (siehe Materialtabelle) aus, um mögliche systematische experimentelle Effekte zu überprüfen.
- Reinigen Sie den DNP-Flüssigkeitspfad sofort mit sauberem Auflösungsmedium, gefolgt von Ethanol. Blasen Sie Heliumgas durch den Flüssigkeitsweg, um verbleibende Reinigungsflüssigkeiten zu entfernen und den Sauerstoffweg zu reinigen. Reinigen Sie alle Glaswaren.
- Zeichnen Sie nach jeder Messung den pH-Wert der Proben sowohl des Tischspektrometers als auch des Feldrad-Relaxometers auf.
HINWEIS: JedeT1-Messung ist eine separate hyperpolarisierte Auflösung vom DNP-Gerät, daher ist Vorsicht geboten, um die Mess-zu-Messung-Reproduzierbarkeit der Probenzusammensetzung sicherzustellen. Dies wird durch das Wiegen aller Wirkstoffe und Lösungsmittel mit einer Genauigkeit von 0,1 mg erreicht, um eine genaue und reproduzierbare Vorbereitung der endgültigen hyperpolarisierten Lösungen zu gewährleisten.
2. Relaxometrie
HINWEIS Bitte beachten Sie Tabelle 1, um die Auswahl und Verwendung der verschiedenen Parameter, die in den folgenden Schritten beschrieben werden, besser zu verstehen. Vor der Auflösung muss der Relaxometer-Flip-Winkel berechnet und das Relaxometer muss eingerichtet und bereit für die Messung der hyperpolarisierten Lösung sein (siehe unten).
- Flip-Winkel-Kalibrierung
- Bereiten Sie 1 ml ordentliche [1-13C]Pyruvicsäure in einem NMR-Rohr vor und fügen Sie ein Gadolinium-Kontrastmittel hinzu, um die T1 der 13C-Kerne auf einen Wert von weniger als 200 ms, aber mehr als 50 ms zu reduzieren.
- Versiegeln Sie das NMR-Rohr, damit es mehrmals als Kalibrierstandard verwendet werden kann.
- Mit dem Tiefenmesser des Relaxometers, stellen Sie die Tiefe des Einführens des NMR-Rohrs auf die richtige Höhe, um sicherzustellen, dass die Probe in der Mitte der Relaxometer RF-Spule befindet.
- Markieren Sie die Einfügetiefe des 13C Pyruvat Kalibrierungsstandards mit Klebeband, um die Wiederholbarkeit zu gewährleisten.
- Legen Sie den Tiefenstopper am NMR-Rohr an die vom Band angegebene Position und legen Sie diesen Kalibrierstandard in die Bohrung des Feldrad-Relaxometers ein. Verwenden Sie ein Gewicht, um das NMR-Rohr in Position zu halten.
- Öffnen Sie das Instrumentenluftventil und stellen Sie von der Relaxometer-Frontplatte den Temperaturregler auf 37 °C ein. Dadurch wird die Temperatur der Probe während des Experiments bei 37 °C (bei 0,5 °C) mit erwärmter Luft beibehalten.
- Richten Sie die Feld-Zyklus-Relaxometer-Hardware ein, um 13C-Kernsignale zu erfassen. Dazu gehört die Installation und Energetik der externen Shim-Spule (siehe Materialtabelle),die Abstimmung und Anpassung der HF-Spule auf 8 MHz (bei 13C-Kernen) und die Verwendung des entsprechenden Kabels mit einem Kabel von 4 MHz.
- Führen Sie in der Instrumentensoftware die folgenden Schritte aus:
- Wählen Sie die Haupt-Par-Registerkarte
- Klicken Sie auf die Zelle neben der Beschriftung Experiment und scrollen Sie im Pop-up-Fenster nach unten, um die Pulssequenz "13CANGLE" auszuwählen. FFC ".
- Legen Sie die folgenden Erfassungsparameter fest: RFA = 5; SWT = 0,005, RD = 0,5, BPOL = 30 MHz, TPOL = 0,5.
- Wählen Sie die Registerkarte Acq. par aus, und wählen Sie dann die Unterregisterkarte Basic aus.
- Klicken Sie auf die Zelle neben der Bezeichnung Nucleus und scrollen Sie im Popup-Fenster nach unten, um 13Causzuwählen.
- Legen Sie dann die folgenden Parameter fest: SF = 8 MHz, SW = 1000000, BS = 652, FLTR = 100000, MS = 32.
- Wählen Sie die Unterregisterkarte Conf aus.
- Legen Sie die folgenden Parameter fest: RINH = 25, ACQD = 25.
- Wählen Sie die untertabuisierte Registerkarte nDim
- NBLK = 32, BINI = 2, BEND = 62 einstellen.
- Wählen Sie die Registerkarte Auswertung und dann die Unterregisterkarte Parameter aus.
- Legen Sie die folgenden Parameter fest: EWIP = 10, EWEP = 128, EWIB = 1, EWEB = 32.
- Klicken Sie dann auf das Symbol "Erfassung starten", um die Pulssequenz auszuführen.
- Sobald die Erfassung abgeschlossen ist, speichern Sie die Daten, wählen Sie das Dialogsymbol Auswertung aus und wählen Sie im Analysemenü WAM-Fenster: Absolute Magnitude. Wählen Sie dann Berichtsblatt, Graphen und Exportdatei aus und klicken Sie schließlich auf Ausführen.
- Suchen Sie im Berichtsfenster die HF-Pulsbreite, die die maximale Amplitude bereitstellt, und stimmen Sie den Wert mit Hilfe des Cursors im angezeigten Diagramm fein abzustimmen, was den Diagrammen in der unteren Zeile von Abbildung 3ähnelt. Diese Pulsbreite wird für den Parameter PW90 der folgenden Experimente verwendet.
- Klicken Sie auf das F1-Symbol, um die Frequenzverschiebung des Relaxometers anzupassen.
ANMERKUNG: WAM-Fenster: Absolute Magnitude ist eine Prozedur, um die Größe einer einzelnen oder einer Sequenz von Free-Induction-Zerfalls-Akquise (FIDs) vom von EWIP definierten Punkt bis zu dem von EWEP angegebenen Punkt und vom Block zu integrieren. von EWIB auf den von EWEBangegebenen Block definiert.
- T1-Messungen
- Stellen Sie sicher, dass die externe Shim-Spule installiert und mit Strom versorgt ist.
- Führen Sie in der Gerätesoftware die folgenden Schritte aus:
- Wählen Sie die Haupt-Par-Registerkarte
- Klicken Sie auf die Zelle neben der Beschriftung Experiment und scrollen Sie im Pop-up-Fenster nach unten, um die Pulssequenz HPUB/Sauszuwählen, die in Abbildung 4dargestellt ist.
- Legen Sie die folgenden Erfassungsparameter fest: RFA = 25, T1MX = Werte zwischen 3 und 5; SWT = 0,2, RD = 0, BRLX = Gewünschtes Entspannungsfeld in MHz (Protonen-Larmor-Frequenz).
- Wählen Sie die Registerkarte Acq. par aus, und wählen Sie dann die Unterregisterkarte Basic aus.
- Klicken Sie auf die Zelle neben der Bezeichnung Nucleus und scrollen Sie im Popup-Fenster nach unten, um 13Causzuwählen.
- Legen Sie dann die folgenden Parameter fest: SF = 8 MHz, SW = 1000000, BS = 652, FLTR = 50000.
- Wählen Sie die Unterregisterkarte Conf aus.
- Legen Sie die folgenden Parameter fest: PW90 entspricht dem Wert in Schritt 2.1.10, RINH = 25, ACQD = 25.
- Wählen Sie die Unterlader Puls aus und setzen Sie PW = 5.
- Wählen Sie die Unterlasche nDim aus und legen Sie NBLK = 100 fest.
- Warten Sie und machen Sie sich bereit, um die hyperpolarisierte Lösung zu erhalten, um die Datenerfassung zu initiieren.
- Unmittelbar vor dem Einsetzen der Probe in das Relaxometer starten Sie die Pulssequenz manuell von der Konsole aus, um zu vermeiden, dass die Probe in ein Null-Magnetfeld eingefügt wird. Aus diesem Grund ist es wichtig, den ersten Free Induction Decay (FID) während der Datenanalyse zu ignorieren.
- Nachdem die Erfassung abgeschlossen ist, speichern Sie die Daten, indem Sie auf die Schaltfläche Speichern klicken.
- Integrieren Sie mithilfe der Analysesoftware die Größe jedes FID-Signals, um eine Datenreihe zu erstellen, die aus der Probenmagnetisierung als Funktion der Zeit besteht.
- Extrahieren Sie die Spin-Gitter-Entspannungszeit aus einem Drei-Parameter-Exponentialmodell unter Verwendung eines standardmäßigen nichtlinearen Methodedes für die Anpassung am kleinsten Quadrat, der in einer kommerziellen Analysesoftware implementiert ist (siehe Tabelle der Materialien), vorausgesetzt, dass eine gleichmäßige Gewichtung für alle Daten:
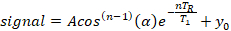
wobei A die Anfangssignalamplitude (y-intercept), T1 die Spin-Gitter-Entspannungszeit ist, TR die Wiederholungszeit ist, die ein bekannter Wert ist, y0 ist das Signal offset, und cos(n-1)(b) ist eine Korrektur für den Verlust der Längsmagnetisierung bei der nth Messung für einen Flip-Winkel, .
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Abbildung 2 zeigt ein Beispiel für einen hochauflösenden Vollbereichs-Mikrowellen-Sweep für Pyruvicsäure. Für den dargestellten Fall entspricht diese optimale Mikrowellenfrequenz 94,128 GHz, hervorgehoben in der Abbildung einfügen. Unser DNP-System kann normalerweise im Bereich von 93.750 GHz bis 94.241 GHz mit einer Schrittgröße von 1 MHz, einer Polarisationszeit von bis zu 600 s und einer Leistung von bis zu 100 mW arbeiten. Ein vollständiger Frequenzbereich wird nur auf neuartige...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Verwendung von DNP zur Verbesserung der Signalerfassung ist eine technische Lösung für unzureichende Magnetresonanzsignale, die von 13C-Kernen in begrenzten Konzentrationen zur Verfügung stehen, wie sie in Tierinjektionen verwendet werden, stellt aber andere experimentelle Herausforderungen dar. Jede in Abbildung 7 dargestellte Entspannungsmessung stellt eine Messung einer einzigartig vorbereiteten Probe dar, da sie nach auflösungsbedingt erwidert werden kann. Dies führt ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Angaben.
Danksagungen
Die Autoren danken dem Ontario Institute for Cancer Research, Imaging Translation Program und dem Natural Sciences and Engineering Research Council of Canada für die Finanzierung dieser Forschung. Wir möchten auch nützliche Gespräche mit Albert Chen, GE Healthcare, Toronto, Kanada, Gianni Ferrante, Stelar s.r.l., Italien, und William Mander, Oxford Instruments, UK, anerkennen.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| [1-13C]Pyruvic Acid | Sigma-Aldrich, St. Louis, MO, USA | 677175 | |
| 10mm NMR Tube | Norell, Inc., Morganton NC, USA | 1001-8 | |
| De-ionized water | |||
| Ethylenediaminetetraacetic acid disodium salt dihydrate (EDTA) | Sigma-Aldrich, St. Louis, MO, USA | E5134 | |
| HyperSense Dynamic Nuclear Polarizer | Oxford Instruments, Abingdon, UK | Includes the following: "DNP-NMR Polarizer" software used to control and monitor the whole DNP polarizer; "RINMR" used to monitor the solid state polarization levels; "HyperTerminal" used to communicate the DNP software with the RINMR software that monitors the solid state polarization level. Also includes the MQC bench top spectrometer to monitor the liquid state polarization in conjunction with it own RINMR software | |
| MATLAB R2017b | MathWorks, Natick, MA | Include scripts for non-linear fitting of magnetization decay over time and T1 NMRD analysis of hyperpolarized pyruvic acid. | |
| OX063 Triarylmethyl radical | Oxford Instruments, Abingdon, UK | ||
| pH meter - SympHony | VWR International, Mississauga, ON., Canada | SB70P | |
| ProHance | Bracco Diagnostics Inc. | Gadoteridol, Gd-HP-DO3A | |
| Pure Ethanol (100% pure) | Commercial Alcohols, Toronto, ON, Canada | P016EAAN | |
| Shim Coil | Developed in-house | ||
| Sodium Chloride | Sigma-Aldrich, St. Louis, MO, USA | S7653 | |
| Sodium Hydroxide | Sigma-Aldrich, St. Louis, MO, USA | S8045 | |
| SpinMaster FFC2000 1T C/DC | Stelar s.r.l., Mede (PV) Italy | Includes the software "AcqNMR" that is used to set experimental parameters, monitor the tuning and matching of the RF coil, loading different pulse sequences, calibrate flip angle, data acquisition and curve fitting, among other functions. Also includes a depth gauge, some weights and a depth stopper. | |
| Trizma Pre-Set Crystals (pH 7.6) | Sigma-Aldrich, St. Louis, MO, USA | T7943 |
Referenzen
- Golman, K., Zandt, R. I., Lerche, M., Pehrson, R., Ardenkjaer-Larsen, J. H. Metabolic imaging by hyperpolarized 13C magnetic resonance imaging for in vivo tumor diagnosis. Cancer Research. 66 (22), 10855-10860 (2006).
- Witney, T. H., Brindle, K. M. Imaging tumour cell metabolism using hyperpolarized 13C magnetic resonance spectroscopy. Biochemical Society Transactions. 38 (5), 1220-1224 (2010).
- Kurhanewicz, J., et al. Analysis of cancer metabolism by imaging hyperpolarized nuclei: prospects for translation to clinical research. Neoplasia. 13 (2), 81-97 (2011).
- Golman, K., et al. Cardiac metabolism measured noninvasively by hyperpolarized 13C MRI. Magnetic Resonance in Medicine. 59 (5), 1005-1013 (2008).
- Golman, K., in 't Zandt, R., Thaning, M. Real-time metabolic imaging. Proceedings of the National Academy of Science of the United States of America. 103 (30), 11270-11275 (2006).
- Day, S. E., et al. Detecting response of rat C6 glioma tumors to radiotherapy using hyperpolarized [1- 13C]pyruvate and 13C magnetic resonance spectroscopic imaging. Magnetic Resonance in Medicine. 65 (2), 557-563 (2011).
- Gallagher, F. A., et al. Magnetic resonance imaging of pH in vivo using hyperpolarized 13C-labelled bicarbonate. Nature. 453 (7197), 940-943 (2008).
- Wilson, D. M., et al. Multi-compound polarization by DNP allows simultaneous assessment of multiple enzymatic activities in vivo. Journal of Magnetic Resonance. 205 (1), 141-147 (2010).
- Gallagher, F. A., et al. Production of hyperpolarized [1,4-13C2]malate from [1,4-13C2]fumarate is a marker of cell necrosis and treatment response in tumors. Proceedings of the National Academy of Science of the United States of America. 106 (47), 19801-19806 (2009).
- Chen, A. P., et al. Feasibility of using hyperpolarized [1-13C]lactate as a substrate for in vivo metabolic 13C MRSI studies. Magnetic Resonance Imaging. 26 (6), 721-726 (2008).
- Gallagher, F. A., Kettunen, M. I., Day, S. E., Lerche, M., Brindle, K. M. 13C MR spectroscopy measurements of glutaminase activity in human hepatocellular carcinoma cells using hyperpolarized 13C-labeled glutamine. Magnetic Resonance in Medicine. 60 (2), 253-257 (2008).
- Ardenkjaer-Larsen, J. H., et al. Increase in signal-to-noise ratio of > 10,000 times in liquid-state NMR. Proceedings of the National Academy of Sciences of the United States of America. 100 (18), 10158-10163 (2003).
- Ishii, M., et al. Hyperpolarized 13C MRI of the pulmonary vasculature and parenchyma. Magnetic Resonance in Medicine. 57 (3), 459-463 (2007).
- Lau, A. Z., Chen, A. P., Cunningham, C. H. Integrated Bloch-Siegert B(1) mapping and multislice imaging of hyperpolarized (1)(3)C pyruvate and bicarbonate in the heart. Magnetic Resonance in Medicine. 67 (1), 62-71 (2012).
- Lau, A. Z., et al. Rapid multislice imaging of hyperpolarized 13C pyruvate and bicarbonate in the heart. Magnetic Resonance in Medicine. 64 (5), 1323-1331 (2010).
- Golman, K., Ardenkjaer-Larsen, J. H., Petersson, J. S., Mansson, S., Leunbach, I. Molecular imaging with endogenous substances. Proceedings of the National Academy of Sciences of the United States of America. 100 (18), 10435-10439 (2003).
- Johansson, E., et al. Cerebral perfusion assessment by bolus tracking using hyperpolarized 13C. Magnetic Resonance in Medicine. 51 (3), 464-472 (2004).
- Johansson, E., et al. Perfusion assessment with bolus differentiation: a technique applicable to hyperpolarized tracers. Magnetic Resonance in Medicine. 52 (5), 1043-1051 (2004).
- Albers, M. J., et al. Hyperpolarized 13C lactate, pyruvate, and alanine: noninvasive biomarkers for prostate cancer detection and grading. Cancer Research. 68 (20), 8607-8615 (2008).
- Chen, A. P., et al. Hyperpolarized C-13 spectroscopic imaging of the TRAMP mouse at 3T-initial experience. Magnetic Resonance in Medicine. 58 (6), 1099-1106 (2007).
- Lupo, J. M., et al. Analysis of hyperpolarized dynamic 13C lactate imaging in a transgenic mouse model of prostate cancer. Magnetic Resonance Imaging. 28 (2), 153-162 (2010).
- von Morze, C., et al. Imaging of blood flow using hyperpolarized [(13)C]urea in preclinical cancer models. Journal of Magnetic Resonance Imaging. 33 (3), 692-697 (2011).
- Brindle, K. M., Bohndiek, S. E., Gallagher, F. A., Kettunen, M. I. Tumor imaging using hyperpolarized 13C magnetic resonance spectroscopy. Magnetic Resonance in Medicine. 66 (2), 505-519 (2011).
- Park, I., et al. Detection of early response to temozolomide treatment in brain tumors using hyperpolarized 13C MR metabolic imaging. Journal of Magnetic Resonance Imaging. 33 (6), 1284-1290 (2011).
- Bohndiek, S. E., et al. Detection of tumor response to a vascular disrupting agent by hyperpolarized 13C magnetic resonance spectroscopy. Molecular Cancer Therapeutics. 9 (12), 3278-3288 (2010).
- Witney, T. H., et al. Detecting treatment response in a model of human breast adenocarcinoma using hyperpolarised [1-13C]pyruvate and [1,4-13C2]fumarate. British Journal of Cancer. 103 (9), 1400-1406 (2010).
- Levitt, M. H. Spin dynamics: basics of nuclear magnetic resonance. , John Wiley & Sons. (2001).
- Mieville, P., Jannin, S., Bodenhausen, G. Relaxometry of insensitive nuclei: optimizing dissolution dynamic nuclear polarization. Journal of Magnetic Resonance. 210 (1), 137-140 (2011).
- Redfield, A. G. Shuttling device for high-resolution measurements of relaxation and related phenomena in solution at low field, using a shared commercial 500 MHz NMR instrument. Magnetic Resonance in Chemistry. 41 (10), 753-768 (2003).
- Grosse, S., Gubaydullin, F., Scheelken, H., Vieth, H. -M., Yurkovskaya, A. V. Field cycling by fast NMR probe transfer: Design and application in field-dependent CIDNP experiments. Applied Magnetic Resonance. 17 (2), 211-225 (1999).
- Kimmich, R., Anoardo, E. Field-cycling NMR relaxometry. Progress in Nuclear Magnetic Resonance Spectroscopy. 44 (3-4), 257-320 (2004).
- Guðjónsdóttir, M., Belton, P., Webb, G. Magnetic Resonance in Food Science: Challenges in a Changing World. , The Royal Society of Chemistry. 65-72 (2009).
- Anoardo, E., Galli, G., Ferrante, G. Fast-field-cycling NMR: Applications and instrumentation. Applied Magnetic Resonance. 20 (3), 365-404 (2001).
- Chattergoon, N., Martinez-Santiesteban, F., Handler, W. B., Ardenkjaer-Larsen, J. H., Scholl, T. J. Field dependence of T1 for hyperpolarized [1-13C]pyruvate. Contrast Media & Molecular Imaging. 8 (1), 57-62 (2013).
- Martínez-Santiesteban, F. M., Dang, T. P., Lim, H., Chen, A. P., Scholl, T. J. T1 nuclear magnetic relaxation dispersion of hyperpolarized sodium and cesium hydrogencarbonate-13C. NMR in Biomedicine. 30 (9), 3749(2017).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten