Method Article
DNAzyme-abhängige Analyse der rRNA 2'-O-Methylierung
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur DNAzyme-abhängigen Spaltung der RNA vor. Dies ermöglicht eine schnelle und standortabhängige Analyse der RNA 2'-O-Methylierung. Dieser Ansatz kann für die vorläufige oder größere Bewertung der SnoRNA-Aktivität verwendet werden.
Zusammenfassung
Leitkasten C/D kleine Nukleolar-RNAs (snoRNAs) katalysieren 2'-O-Methylierung von ribosomaler und kleiner kernatischer RNA. Jedoch, eine große Anzahl von SnoRNA in höheren Eukaryoten können promiskuly erkennen andere RNA-Arten und 2'-O-Methylat mehrere Ziele. Hier bieten wir Schritt-für-Schritt-Anleitung für die schnelle und nicht-teure Analyse der standortspezifischen 2'-O-Methylierung mit einer etablierten Methode, die kurze DNA-Oligonukleotide namens DNAzymes verwendet. Diese DNA-Fragmente enthalten katalytische Sequenzen, die RNA an bestimmten Konsenspositionen spalten, sowie variable Homologiearme, die DNAzyme auf ihre RNA-Ziele lenken. Die DNAzym-Aktivität wird durch 2-'O-Methylierung des Nukleotids neben der Spaltungsstelle in der RNA gehemmt. Somit sind DNAzyme, die nur durch den Konsens der Spaltensequenz begrenzt sind, perfekte Werkzeuge für die schnelle Analyse der snoRNA-vermittelten RNA 2'-O-Methylierung. Wir analysierten snoRNA snR13- und snR47-geführte 2'-O-Methylierung von 25S ribosomaler RNA in Saccharomyces cerevisiae, um die Einfachheit der Technik zu demonstrieren und ein detailliertes Protokoll für den DNAzyme-abhängigen Assay bereitzustellen.
Einleitung
RNA-Modifikationen spielen eine wichtige Rolle bei der Regulierung der Genexpression. RNA 2'-O-Methylierung und Pseudouridylierung, die durch Kasten C/D und Box H/ACA kleine Nukleolar-RNAs (SnoRNAs) geführt werden, schützen die RNA vor Abbau und stabilisieren ihre höherrangigen Strukturen1,2,3 . SnoRNA-Ziele wurden hauptsächlich in ribosomalen RNAs (rRNA) und kleinen nuklearen RNAs (snRNAs) identifiziert. Jedoch, in höheren Eukaryoten, es gibt potenziell Hunderte von snoRNA ohne zugewiesene Funktionen und einige von ihnen können mehrere RNAs1,4,5,6,7erkennen. Daher sind Methoden, die die Identifizierung und Analyse von snoRNA-geführten Modifikationen ermöglichen, wichtige Werkzeuge zur Aufdeckung von Mechanismen, die zelluläre Prozesse regeln.
Eine Box C/D snoRNA-geführte putative 2'-O-Methylierungsstelle kann bioinforkokant identifiziert und experimentell durch viele Techniken bestätigt werden, einschließlich RNase H-gerichteter Spaltung oder ortsspezifische und genomweite Methoden, die Reverse-Transkription verwenden in niedrigen Nukleotiden (dNTPs) KonzentrationAnsatz8,9,10,11. Diese Techniken sind sehr empfindlich, aber auch mühsam und teuer, daher kann nicht für die anfängliche n- oder schnelles Testen geeignet sein. Eine der einfachsten und kostengünstigsten Methoden zur Identifizierung von 2'-O-Methylierungsstellen ist DNAzyme-abhängige RNA-Spaltung12. DNAzyme sind kurze, einsträngige und katalytisch aktive DNA-Moleküle, die in der Lage sind, die rna an bestimmten Positionen endonukleolytisch zu spalten. Sie bestehen aus einer konservierten und katalytisch aktiven Kernsequenz und 5' und 3' Bindungsarmen, die aus variablen Sequenzen bestehen, die von Watson-Crick Base-Pairing mit dem RNA-Ziel hybridisiert werden sollen (Abbildung 1). So liefern die 5' und 3' Arme die katalytische Sequenz an die spezifische RNA-Site. DNAzyme-abhängige Spaltung wird durch 2'-O-Methylierung des direkt vor der Spaltungsstelle positionierten Nukleotids12,13gehemmt. Dies macht DNAzymes sehr praktische Werkzeuge für die Analyse von vermeintlichen oder bekannten RNA 2'-O-Methylierungsstellen.
Für RNA-Modifikationsanalysen werden zwei Arten von DNAzymes verwendet12. Die aktive Sequenz von 10-23 DNAzyme (Abbildung 1A) besteht aus 15 Nukleotiden (5'RGGCTAGCTACAACGA3'), die eine Schleife um das zielgerichtete RNA-Purin-Pyrimid-Dinukleotid bilden und die Spaltung zwischen diesen beiden Nukleotiden katalysieren. Das RNA-Purin (R) ist nicht mit dem DNAzym base-gepaart und die 2'-O-Methylierung strotzt vor dNAzym die Spaltung. Die Bindungsarme von 10-23 DNAzymes sind in der Regel 10-15 Nukleotide lang. Die zweite DNAzyme-Klasse, 8-17 DNAzymes (Abbildung 1B) enthalten eine 14-Nukleotid-Katalytiksequenz (5'TCCGAGCCGGACGA3'). NukleotideC2, C3 und G4 paaren sich mit C9 G10 und G11 und bilden eine kurze Stammschleifenstruktur. 8-17 DNAzymes spalten RNA vor gelagert von jedem Guanin, das unvollkommen mit dem ersten Thymin aus der dNAzyme-Aktivsequenz gepaart ist. Das RNA-Nukleotid vor dem Guanin ist nicht mit DNAzyme base-gepaart und seine 2'-O-Methylierung beeinträchtigt die Spaltung. 8-17 DNAzyme benötigen längere Homologiearme von etwa 20 Nukleotiden, um DNAzyme auf seine spezifische Sequenz zu lenken.
Hier stellen wir ein Schritt-für-Schritt-Protokoll für die Analyse der 2'-O-Methylierung von rRNA in Saccharomyces cerevisiae mit 10-23 und 8-17 DNAzyme-abhängigen Ansätzen12,13 (Abbildung 1C) zur Verfügung. Dieses Protokoll kann leicht für andere Organismen und RNA-Arten angepasst und für die schnelle, vorläufige oder größere Analyse der standortspezifischen RNA 2'-O-Methylierung eingesetzt werden.
Protokoll
1. Sorten-, Medien- und Pufferrezepte
- Bereiten Hefe (S. cerevisiae) Medien wie hier detailliert: YP (1% w/v Hefeextrakt, 2% w / v bakteriologische pepton), und Glukose und Galaktose-Bestände bei 20% w/v.
- Bereiten Sie Natriumacetat (NaAc)-EDTA (AE) Puffer wie hier detailliert: 50 mM NaAc pH 5.3 und 10 mM EDTA.
- Bereiten Sie 10-23 DNAzyme 4x Inkubationspuffer wie hier detailliert: 24 mM Tris pH 8.0, 60 mM NaCl und 10-23 DNAzyme 4x Reaktionspuffer: 200 mM Tris pH 8.0 und 600 mMN NaCl.
- Bereiten Sie 8-17 DNAzyme 2x Reaktionspuffer wie hier detailliert: 200 mM KCl, 800 mM NaCl, 100 mM HEPES pH 7.0, 15 mM MgCl2und 15 mMM MnCl2.
- Bereiten Sie 10x MOPS Puffer wie hier detailliert: 200 mM MOPS, 50 mM NaAc, 1 mMEDTA; pH 7.0 und 1.5x Probendeaturing Buffer: 50% v/v Formamid, 20% v/v Formaldehyd, 1,5x MOPS Puffer.
- Erhalten Sie S. cerevisiae-Stämme, BY4741 (MATa his3-1 leu2-0 met15-0 ura3'0); GAL1::SNR13 (als BY4741 aber GAL1::SNR13:KANmX); GAL1::SNR47 (als BY4741 aber GAL1::SNR47:HIS3mX). Für diese Analyse kann jeder andere Hefestamm verwendet werden.
2. DNAzyme Design
- Finden Sie RNA-Sequenz von Interesse oder vermeintliche Methylierung Website mit einer geeigneten Datenbank. Verwenden Sie für S. cerevisiae snoRNA-Ziele die Hefe-SnoRNA-Datenbank: http://people.biochem.umass.edu/fournierlab/snornadb/mastertable.php14
- Um die von Interesse sindde Methylierungsstelle, z.B. snR13-abhängige Seite, zu finden, wählen Sie "snR13" und notieren Sie sich die Position des modifizierten Nukleotids (z.B. snR13-geführte A2281 in 25S rRNA).
- Suchen Sie Sequenzen vor und nach dem modifizierten Nukleotid mithilfe der entsprechenden Datenbank. Verwenden Sie für S. cerevisiae die Saccharomyces Genome Database: https://www.yeastgenome.org/
- Suchen Sie nach dem Zielgennamen, z.B. RDN25 (kodiert 25S rRNA).
- Wählen Sie auf der Registerkarte "Sequenz" 10-15 Nukleotide vor (5' Arm) und nachgelagert (3 'Arm) der Methylierungsstelle aus, wenn sie einen 10-23 DNAzyme-Assay und 20 Nukleotide vor (5' Arm) und nachgelagert (3'arm) der Methylierungsstelle für ein 8-17 DNAzyme verwenden.
- Erstellen Sie komplementäre Sequenzen von 5' und 3' Armen.
- Flank10-23 oder 8-17 DNAzyme katalytische Sequenz mit komplementären Sequenzen von 5' und 3' Armen.
- Bestellen Sie DNAzyme als normales DNA-Oligonukleotid beim Lieferanten.
3. S. cerevisiae Wachstumsbedingungen
HINWEIS: Es wurden S. cerevisiae BY4741 Stammderivate verwendet, bei denen die Expression von SNR13 oder SNR47 snoRNA vom induzierbaren GAL1-Promotor angetrieben wird. Um ihre Synthese zu induzieren oder zu hemmen, wachsen Zellen auf einem Medium, das Galaktose (GAL1-abhängige Transkription auf) oder Glukose (GAL1-abhängige Transkription aus) enthält. Als Kontrolle, verwenden Sie die Wild-Typ-Stamm (BY4741) entweder auf Galaktose oder Glukose angebaut.
- Wachsen Sie Hefestämme in einem geeigneten Medium und Bedingungen. Um GAL1::SNR13 und GAL1::SNR47-Stämme sowie den isogenen Wildtyp-Stamm zu analysieren, wachsen Zellen in 50 ml YP-Medien mit entweder 2% Glukose (YPD) oder Galaktose (YPGal) bei 30 °C bis zur mittleren Exponentialphase.
- Zentrifugenzellen bei 1.000 x g, für 3 min bei 4 °C.
- Entsorgen Sie den Überstand und halten Sie die Pellets.
- Zellpellets in flüssigem Stickstoff einfrieren und bei -80 °C lagern.
ACHTUNG: Flüssiger Stickstoff kann schwere kryogene Verbrennungen verursachen. Tragen Sie immer Schutzkleidung und üben Sie Sicherheitsvorkehrungen.
HINWEIS: Zellpellets können bei -80 °C bis zu 1 Monat gelagert werden. Das Protokoll kann hier bei Bedarf angehalten werden.
4. RNA-Isolation15
HINWEIS: Verwenden Sie die am besten geeignete Methode, um RNA zu isolieren. Für Hefe S. cerevisiaekann Die Hot-Phenol-RNA-Extraktion verwendet werden.
- 1 ml eiskaltes Wasser hinzufügen, die Pellets wieder aufhängen und resuspendierte Zellen in 1,5 ml Mikroröhren übertragen.
- Zentrifuge bei 20.000 x g für 10 s bei 4 °C und entfernen Sie den Überstand.
- Fügen Sie 400 L AE-Puffer hinzu, und setzen Sie die Zellen erneut aus.
HINWEIS: Die Schritte 4.4-4.15 werden bei Raumtemperatur durchgeführt, sofern nicht anders angegeben. - Fügen Sie 40 l 10 % SDS und 400 l saures Phenol (pH 4,5) hinzu.
ACHTUNG: Phenol ist giftig und sollte unter einer Dunstabzugshaube behandelt werden. Tragen Sie bei der Arbeit mit Phenol immer einen Labormantel, Schutzhandschuhe und eine Brille. Entsorgen Sie den Abfall gemäß den institutionellen Vorschriften. - Mischen Sie gut durch Wirbel für 20 s.
- Bei 65 °C für 10 min inkubieren. Alle 2 min, sanft öffnen und schließen Sie das Rohr, um den Druck zu lösen und drehen Sie das Rohr 2-3 mal, um die Phasen zu mischen.
- Die Rohre auf -80 °C übertragen und 10 min inkubieren.
- Auftauen der Rohre auf der Bank und Zentrifuge bei 20.000 x g, für 5 min bei Raumtemperatur.
- Übertragen Sie die obere Phase auf ein neues Rohr mit 400 l Säurephenol:Chloroform:Isoamylalkohol (25:24:1). Unterbrechen Sie die Interphase nicht.
ACHTUNG: Chloroform ist giftig und sollte unter einer Dunstabzugshaube behandelt werden. Tragen Sie bei der Arbeit mit Chloroform immer einen Labormantel, Schutzhandschuhe und eine Brille. Entsorgen Sie den Abfall gemäß den institutionellen Vorschriften. - Gut mischen, indem man 30 s und Zentrifuge bei 20.000 x g 10 min bei Raumtemperatur voranst.
- Übertragen Sie die obere Phase (ca. 400 l) in ein neues Rohr, das 400 l Chloroform enthält.
- Gut mischen, indem man 30 s und Zentrifuge bei 20.000 x g für 5 min bei Raumtemperatur verrühren.
- Übertragen Sie die obere Phase (ca. 300-350 l) in ein neues Rohr mit 1 ml EtOH und 40 l 7,5 M Ammoniumacetat (NH4AC). Mischen Sie, indem Sie das Rohr ein paar Mal umdrehen.
- Bei -80 °C für 2 h oder über Nacht bei -20 °C inkubieren.
HINWEIS: Das Verfahren kann hier angehalten werden. - Zentrifuge bei 20.000 x g, für 10 min bei 4 °C. Ein kleines, weißes RNA-Pellet wird auf der Unterseite des Rohres sichtbar.
- Entfernen Sie EtOH durch Pipettieren, um eine Störung des Pellets zu vermeiden.
- 1 ml 70% EtOH und Zentrifuge bei 20.000 x g für 5 min bei Raumtemperatur hinzufügen.
- Entfernen Sie 70% EtOH durch Pipettieren.
- Zentrifuge bei 20.000 x g für 15 s und entfernen Sie die restliche EtOH mit 2-20 l Pipette.
- Lassen Sie das Rohr 5 min auf der Bank offen, um das RNA-Pellet zu trocknen.
HINWEIS: RNA-Pellet ändert seine Farbe von weiß zu transparent, wenn trocken. - Setzen Sie das RNA-Pellet in 30 l RNase/DNase-freiH2O aus, übertragen Sie die Röhre sofort auf das Eis und messen Sie die RNA-Konzentration auf dem Mikrospektrophotometer.
- Einfrieren der Proben bei -20 °C.
HINWEIS: RNA kann bei -20 °C bis zu 1 Monat und bei -80 °C bis zu 1 Jahr gespeichert werden. Die Prozedur kann hier angehalten oder direkt mit dem nächsten Schritt fortgesetzt werden.
5. DNAzyme Verdauung
-
10-23 DNAzyme Verdauung
- In 1,5 ml bereiten Die Röhrchen einen Inkubationsmix her, indem sie 5 g RNA, 200 pmol von 10-23 DNAzyme (2 l von 100 mM Stammlösung) und 2,5 l 4x 10-23 Inkubationspuffer in einem Gesamtvolumen von 10 l kombinieren.
- Die Rohre auf einen trockenen Wärmeblock auf 95 °C übertragen und 3 min inkubieren.
- Die Rohre sofort auf Eis übertragen und 5 min inkubieren.
- Drehen Sie kurz nach unten und legen Sie die Rohre wieder auf das Eis.
- Fügen Sie 20 U RNase-Inhibitor (z. B. 0,5 l RiboLock RNase-Inhibitor) hinzu.
- Die Rohre in einen trockenen Wärmeblock für 25 °C legen und 10 min inkubieren.
- In der Zwischenzeit bereiten Sie ein Reaktionsgemisch in einem 1,5-L-Rohr vor, indem Sie 5 l 4x 10-23 Reaktionspuffer mit 4 l 300 mM MgCl2 und 1 lH2O kombinieren. Legen Sie das Rohr in einen trockenen Block, der auf 37 °C eingestellt ist.
- Übertragen Sie die Inkubationsmischung auf einen trockenen Wärmeblocksatz für 37 °C und fügen Sie 10 l vorgewärmter Reaktionsmix hinzu.
- Inkubieren Sie die Reaktion bei 37 °C für 1 h.
- Übertragen Sie die Rohre auf Eis und fahren Sie mit Schritt 5.3.1 fort.
-
8-17 DNAzyme Verdauung
- Bereiten Sie ein 1,5 ml Mikroröhren mit 5 g RNA in einem Gesamtvolumen von 6 l vor. Halten Sie die Röhre auf Eis.
- Bereiten Sie ein 1,5 ml Mikrorohr mit 400 pmol 8-17 DNAzyme (4 l aus 100 mM Lager) vor. Halten Sie die Röhre auf Eis.
- Die Rohre auf einen trockenen Wärmeblock satzfürh 95 °C übertragen und 2 min inkubieren.
- Bewegen Sie die RNA-Probe auf Eis.
- Drehen Sie das Rohr mit DNAzyme für 5 s und inkubieren bei 25 °C für 10 min.
- Gleichzeitig ein 1,5 ml-Rohr mit 10 l 2x 8-17 Reaktionspuffer vorbereiten und bei 25 °C inkubieren.
- Bereiten Sie ein Reaktionsgemisch vor, indem Sie dem Rohr mit DNAzyme 10 l vorgewärmten 2x Reaktionspuffer hinzufügen.
- Übertragen Sie 14 l des Reaktionsmixes mit RNA in die Röhre und fügen Sie 20 U RNase-Inhibitor hinzu.
- Inkubieren Sie die Reaktion bei 25 °C für 2 h.
- Übertragen Sie die Röhre auf Eis und fahren Sie zur RNA-Reinigung (Schritt 5.3.1).
-
RNA-Reinigung
- Fügen Sie dem Reaktionsröhrchen 350 l Wasser und 400 l Chloroform hinzu, mischen Sie sie gut durch Wirbeln für 30 s und Zentrifugen bei 20.000 x g für 5 min bei Raumtemperatur.
- Übertragen Sie die obere Phase (ca. 300-350 l) in ein neues Rohr mit 1 ml EtOH, 40 l mit 7,5 m NH4AC und 1 l Glykogen (10 g/l). Mischen Sie, indem Sie das Rohr ein paar Mal umdrehen.
- Bei -80 °C für 2 h oder über Nacht bei -20 °C inkubieren.
HINWEIS: Das Verfahren kann hier angehalten werden. - Wiederholen Sie die Schritte von 4.15 bis 4.21.
- Das RNA-Pellet in 10 l RNase/DNase-freiH2O wieder aufsetzen und die Rohre sofort auf Eis übertragen.
- Einfrieren der Proben bei -20 °C.
HINWEIS: RNA kann bei -20 °C bis zu einem Monat und bei -80 °C bis zu 1 Jahr gespeichert werden. Das Verfahren kann hier angehalten werden oder zur RNA-Elektrophorese gehen.
6. RNA-Elektrophorese
- Die Elektrophorese-Ausrüstung (Tank, Tablett, Kamm) mit 1% SDS besprühen, 15 min lassen und mit viel ddH2O abspülen.
- 1,5 g Agarose in 127,5 ml ddH2O durch Erhitzen in der Mikrowelle auflösen.
- 15 ml 10x MOPS und 7,5 ml 37% Formaldehyd in die Agaroselösung geben (Gesamtvolumen beträgt 150 ml).
ACHTUNG: Formaldehyd ist giftig und sollte unter einer Dunstabzugshaube behandelt werden. Tragen Sie bei der Arbeit mit Formaldehyd immer einen Labormantel, Schutzhandschuhe und eine Brille. Entsorgen Sie den Abfall gemäß den institutionellen Vorschriften. - Fügen Sie der Agarorose-Lösung eine angemessene Menge eines Gelflecks nach Wahl hinzu (z. B. 15 L SYBR Safe DNA Gel-Färbung). Gut mischen und die Agarose auf das Tablett gießen.
- Legen Sie sofort einen Kamm in das Gel ein.
- Lassen Sie es für 45 min unter der Dunstabzugshaube. Bedecken Sie das Fach mit Aluminiumfolie, wenn Sie einen lichtempfindlichen Gelfleck verwenden.
- Bereiten Sie 600 ml 1x MOPS Puffer vor.
-
RNA-Probenvorbereitung
- In einem 1,5 ml-Rohr 10 l der verdauten und gereinigten RNA-Probe, 5 l Probendeaturing Buffer und 0,5 l 6x Loading Dye kombinieren.
ACHTUNG: Formamid ist giftig und sollte unter einer Dunstabzugshaube behandelt werden. Tragen Sie bei der Arbeit mit Formamid immer einen Labormantel, Schutzhandschuhe und eine Brille. Entsorgen Sie den Abfall gemäß den institutionellen Vorschriften. - RNA-Proben bei 70 °C für 5 min inkubieren. Übertragen Sie die Proben auf Eis. Inkubieren Sie für 5 min.
- Drehen Sie kurz vor dem Laden auf das Gel.
- In einem 1,5 ml-Rohr 10 l der verdauten und gereinigten RNA-Probe, 5 l Probendeaturing Buffer und 0,5 l 6x Loading Dye kombinieren.
- Das Gel in den Elektrophoresetank geben und mit 1x MOPS Puffer füllen. Laden Sie das gesamte Volumen jeder Probe (15 l) auf das Gel. Laufen Sie bei 80 V, bis Bromphenolblau 2/3 der Gellänge erreicht.
- Stellen Sie das Gel mit einem Imager ab, der geeignet ist, den gewählten Gelfleck (z. B. UV-Transilluminator) zu erkennen.
Ergebnisse
Der Nutzen der DNAzyme-abhängigen Spaltung bei der Analyse von rRNA-Modifikationen wurde kürzlich im Kontext der snoRNAs Reifung13gezeigt. Der DNAzyme-abhängige Test wurde verwendet, um zu zeigen, dass der Mangel an 5'-End-Pre-SnoRNA-Verarbeitung 2'-O-Methylierungsniveaus von 25S und 18S rRNA in S. cerevisiae13betrifft.
Hier haben wir ein induzierbares SnoRNA-Transkriptionssystem verwendet, um die Wirksamkeit und Einfachheit der Technik zu demonstrieren. Box C/D snR13 leitet die Methylierung an zwei Positionen in 25S rRNA, einschließlich Adenin 2281 (Abbildung 2A). Auf dieses Nukleotid folgt Uracil, das das Konsensdinukleotid (RY) bildet, das durch ein 10-23 DNAzyme cleavable ist. Box C/D snR47 leitet auch die Methylierung von zwei Nukleotiden in 25S rRNA (Abbildung 2B). Auf das Adenin in Position 2220 folgt ein Guaninrückstand und dieses Dinukleotid kann durch ein 8-17 DNAzym gespaltet werden. Um die Synthese von snR13 oder snR47 snoRNA zu induzieren oder zu hemmen, haben wir den induzierbaren GAL1-Promotor vor SNR13- oder SNR47-Genen und kultivierten Zellen in mittelhaltige Galaktose (GAL1-abhängige Transkription auf) oder Glukose (GAL1-abhängige Transkription aus). Als nächstes wurde RNA, die aus GAL1::SNR13-Zellen isoliert wurde, mit 10-23 DNAzyme inkubiert, um 25S rRNA an snR13-abhängiger Stelle zwischen den Nukleotiden 2281 und 2282 zu spalten (Abbildung 2C). RNA aus GAL1::SNR47 Stamm wurde mit 8-17 DNAzyme Ziel snR47-abhängigen Standort zwischen Nukleotiden 2220 und 2221 behandelt (Abbildung 2D). Zur Kontrolle wurde DIE RNA des Wildtyps BY4741, der entweder auf Galaktose oder Glukose wächst, mit beiden DNAzymes inkubiert. Die Elektrophorese der mit DNAzyme behandelten RNA ergab, dass 25S rRNA aus GAL1::SNR13 und GAL1::SNR47-Stämmen, die auf Galaktose (GAL) wachsen, intakt blieben (Abbildung 3A,B; Fahrspuren 3). Im Gegensatz dazu wurde RNA, die aus GAL1::SNR13 und GAL1::SNR47-Zellen, die auf Glukose (GLC) anwachsen, von den jeweiligen DNAzymes verdaut(Abbildung 3A,B; Bahnen 4). In beiden Fällen verringerte sich das 25S rRNA-Band und 5' und 3' Trennmittel (A und B) wurden beobachtet. Dies deutet darauf hin, dass in GAL1::SNR13 und GAL1::SNR47-Stämmen 25S rRNA an snR13- oder snR47-geführten Stellen 2'-O-methyliert wurde, wenn Galaktose als Kohlenstoffquelle verwendet wurde und diese snoRNA exprimiert wurden. Das Fehlen von 25S rRNA-Methylierung, wenn snR13 oder snR47 Expression auf Glukose abgeschaltet wurde erlaubt für DNAzyme-abhängige Spaltung. Bei Wildproben(Abbildung 3A,B; Bahnen 1 und 2) wurde keine RNA-Verdauung beobachtet, da die Expression von snR13 und snR47 in diesem Stamm galaktos/glucose-unabhängig ist. Daher war rRNA normalerweise methyliert und so resistent gegen DNAzymes-Aktivität.
Insgesamt zeigt unser Experiment, dass die Spaltungsaktivität von 10-23 (Abbildung 3A) und 8-17 (Abbildung 3B) DNAzymes mit dem Fehlen von Feld C/D snR13 oder snR47 korreliert, was deutlich darauf hindeutet, dass diese snoRNA für 25S rRNA verantwortlich sind. 2'-O-Methylierung an bestimmten Standorten.
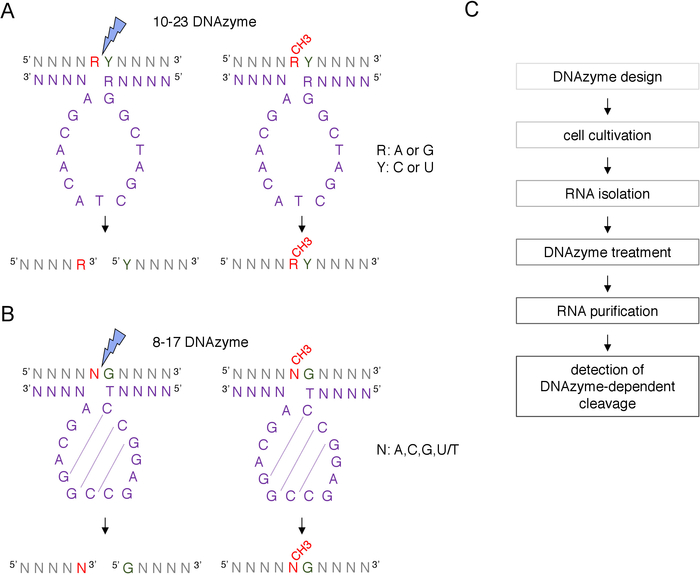
Abbildung 1: DNAzyme und ihre RNA-Substrate. (A) 10-23 DNAzymes spalten ein Purin-Pyrimidin (RY) RNA-Dinukleotid. R in der RNA ist nicht mit DNAzyme gekoppelt, während Y die R-Basis im DNAzyme ergänzt. Die Methylierung des Purins (R) in der RNA unterdrückt die DNAzyme-abhängige Spaltung. (B) 8-17 DNAzymes spalten RNA vor guanin, die unvollkommen mit dem ersten Thymin in der DNAzyme-Katalytiksequenz gepaart ist. Das Nukleotid vor Guanin ist nicht gepaart und seine Methylierung schützt vor DNAzyme-abhängiger Spaltung. RNA wird in Grau (abgesehen von der Methylierungsstelle) gezeigt, DNAzyme in Violett. N = nukleotid, R = Purin: Adenin oder Guanin, Y = Pyrimidin: Cytosin oder Uracil; CH3- bezeichnet die RNA-Methylierung. Die Basispaarung innerhalb der aktiven DNAzyme-Sequenzen wird durch gepunktete Linien gekennzeichnet. Ein blauer Blitz markiert die Dekolleté-Stelle. (C) Ein Flussdiagramm, das die Schritte einer DNAzyme-abhängigen Analyse zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: DNAzyme, die auf snR13- und snR47-abhängige Methylierungsstellen in 25S rRNA abzielen. (A, B) Browser-Screenshots, die snR13-abhängige (A) und snR47-abhängige (B) Methylierungsstellen in 25S rRNA (C) 25S rRNA-Sequenz um snR13-abhängige Methylierungsstelle (A2281) und 10-23 DNAzyme (in Violett) zeigen ENTWICKELT, um RNA zwischen A2281 und U2282zu spalten. Ein2281 ist nicht mit dem DNAzyme gekoppelt, während U2282 ein Paar mit dem ersten Nukleotid aus der aktiven DNAzyme-Sequenz (gekennzeichnet durch eine blaue Linie) bildet. Ein blauer Blitz markiert die Spaltung. (D) 25S rRNA-Sequenz um snR47-abhängige Methylierungsstelle (A2220) und 8-17 DNAzyme (in violett dargestellt), die entwickelt wurde, um RNA zwischen A2220 und G2221zu spalten. Ein2220 ist nicht mit dem DNAzyme hybridisiert, während G2221 unvollkommen mit Thymin gepaart ist (bezeichnet mit einer gestrichelten Linie). Ein blauer Blitz markiert die Dekolleté-Stelle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Analyse der standortspezifischen 2'-O-Methylierung von 25S rRNA mit 10-23 und 8-17 DNAzyme-abhängigem Assay. (A) Analyse der snR13-abhängigen 25S rRNA-Methylierung mit 10-23 DNAzym. (B) Analyse der snR47-abhängigen 25S rRNA-Methylierung mit 8-17 DNAzyme. RNA wurde visualisierte Färbung in einem denaturierenden Agarose-Gel. Die Klecleavage-Produkte A und B sind durch rote Pfeile gekennzeichnet. WT = Wild-Typ-Stamm; GAL = Galaktose, GLC = Glukose. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die DNAzyme-abhängige Verdauung kann als einfache und schnelle Methode zur Analyse der standortspezifischen RNA 2'-O-Methylierung12,13verwendet werden. DNAzymes spalten RNA, wenn das Nukleotid vor der Spaltungsstelle nicht methyliert ist. Im Gegensatz zu anderen Ansätzen, einschließlich RNase H-gerichteter Verdauung, alkalischer Abbau oder umgekehrter Transkription bei niedriger Nukleotidenkonzentration, gefolgt von quantitativer PCR oder Sequenzierung8,10,11 ,16, DNAzyme Ansatz erfordert ein einfaches DNA-Oligonukleotid und grundlegende Reagenzien, die in jedem molekularbiologischen Labor vorhanden sind. Darüber hinaus können DNAzyme in ähnlicher Weise verwendet werden, um die RNA-Pseudouridylierung zu analysieren, die durch die Box H/ACA snoRNA12vermittelt wird, was sie zu vielseitigen Werkzeugen bei der Untersuchung von SnoRNA-Zielen macht.
DNAzyme-abhängige Ansätze sind nur durch Spaltungsstellen-Konsenssequenzen17begrenzt. 10-23 DNAzyme können nur an Position R des RY-Dinukleotids zur Analyse der 2'-O-Methylierung verwendet werden, während 8-17 DNAzyme die Modifikation des vor gelagerten Nukleotids von Guanin erkennen. Daher können Modifikationen wie die 2'-O-Methylierung des ersten Nukleotids in den Dinukleotiden Guanin-Adenin (GA), Adenin-Adenin (AA), Pyrimidin-Adenin (YA) und Pyrimidin-Pyrimidin (YY) nicht analysiert werden. Darüber hinaus sollte der geringe Wirkungsgrad der DNAzyme-abhängigen Spaltung12 berücksichtigt werden. Obwohl einige DNAzymes RNA fast vollständig spalten (Abbildung 3B), verdauen viele DNAzymes ihre Ziele nur teilweise (Abbildung 3B). Die Effizienz kann von der Abfolge der Spaltungsstelle abhängen. Beispielsweise können RNA-Regionen mit Dehnungen desselben Nukleotids die korrekte Positionierung der aktiven DNAzyme-Sequenz beeinflussen. Darüber hinaus können RNA-Regionen, die eine starke sekundäre Struktur bilden, Die DNAzyme-Bindung an die Zielsequenz rehybridisieren und unterdrücken. Um diese Probleme zu überwinden, können Zyklen der Erwärmung und Kühlung des 10-23 DNAzyme und seines RNA-Substrats angewendet werden18.
Wir verwendeten den DNAzyme-Ansatz, um die 2'-O-Methylierung von rRNA zu untersuchen. Man kann diese Technik auch verwenden, um andere RNA-Modifikationen zu analysieren, wieZ. N6-Methyladenosin19. Ribosomale RNA kann aufgrund ihrer Fülle durch Elektrophorese analysiert und die Spaltprodukte unter dem UV-Licht visualisiert werden. Dies gilt jedoch nicht für weniger reichlich efizierte RNAs wie RNA Polymerase II-generierte coding RNAs (mRNA) und nicht-coding RNAs (ncRNA). Diese RNAs können in der Regel nicht direkt durch RNA-Färbung in Agarose- oder Polyacrylamid-Gelen nachgewiesen werden. In solchen Fällen kann die DNAzyme-abhängige Spaltung durch Nördliches Blotting visualisiert, indirekt durch PCR/quantitative PCR detektiert oder durch quantitative PCR mit Polymerasen (z.B. KlenTaq DNA Polymerase) analysiert werden, die in der Lage sind, 2-O-methylierte RNA aus unmethylierte RNA20,21.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Maya Wilson und Aneika Leney für die kritische Lektüre des Manuskripts. Diese Arbeit wurde von einem Sir Henry Dale Fellowship unterstützt, das gemeinsam vom Wellcome Trust und der Royal Society (200473/Z/16/Z) finanziert wurde.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Acid phenol | SIGMA | P4682 | |
| Agarose | VWR | A2114 | |
| Ammonium acetate | SIGMA | A1542 | |
| Chlorophorm | Fisher scientific | 10293850 | |
| DNase/RNase free water | Fischer Scientific | 10526945 | |
| DNAzyme | Integrated DNA Technology | Custom oligo DNA | |
| EDTA | SIGMA | E9884 | |
| Ethanol Absolute | Fisher scientific | 10437341 | |
| Formaldehyde | Sigma | F8775 | |
| Formamide | sigma | F9037 | |
| Galactose | SIGMA | G0750 | |
| Gel Loading Dye | Thermo Fisher Scientific | R0611 | |
| Glucose | SIGMA | G7021 | |
| Glycogen | Thermo Fisher Scientific | R0561 | |
| HEPES | SIGMA | H3375 | |
| Isoamyl | SIGMA | W205702 | |
| KCl | SIGMA | P9333 | |
| MgCl2 | SIGMA | M8266 | |
| MnCl2 | SIGMA | 244589 | |
| MOPS | SIGMA | M1254 | |
| NaCl | SIGMA | S7653 | |
| Oxoid Peptone Bacteriological | Thermo Fisher Scientific | LP0037 | |
| Oxoid Yeast Extract Powder | Thermo Fisher Scientific | LP0021 | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher Scientific | EO0382 | |
| SDS | SIGMA | 74255 | |
| Sodium acetate trihydrate | SIGMA | S8625 | |
| SYBR Safe DNA Gel Stain | Thermo Fisher Scientific | S33102 | |
| Tris base | SIGMA | TRIS-RO | |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| 1.5 mL microtubes | Sarstedt | ||
| 152VR5C01M -80°C freezer | Thermo Fisher Scientific | ||
| 250 mL Erlenmeyer flasks | Cole-Parmer | ||
| 50 mL conical tubes | Sarstedt | ||
| Combicup VX200 vortex | Appleton Woods | ||
| DS-11 microspectrophotometer | Denovix | ||
| Electrophoresis chamber (20 cm tray) | SIGMA | ||
| FiveEasy F20 pH meter | Appleton Woods | ||
| Gel documentation system | Syngene | ||
| Heraeus Fresco 21 micro centrifuge | Fisher Scientific | ||
| Megafuge 8R centrifuge with rotator suitable for 50 mL conical tubes | Fisher Scientific | ||
| Mini Fuge Plus mini centrifuge | Starlab | ||
| Mixer HC thermal block | Starlab | ||
| OLS26 Shaking Water Bath | Grant | ||
| PowerPac power supplier | BioRad |
Referenzen
- Dieci, G., Preti, M., Montanini, B. Eukaryotic snoRNAs: a paradigm for gene expression flexibility. Genomics. 94 (2), 83-88 (2009).
- Watkins, N. J., Bohnsack, M. T. The box C/D and H/ACA snoRNPs: key players in the modification, processing and the dynamic folding of ribosomal RNA. Wiley Interdisciplinary Review RNA. 3 (3), 397-414 (2012).
- Kufel, J., Grzechnik, P. Small Nucleolar RNAs Tell a Different Tale. Trends in Genetics. , (2018).
- Li, T., Zhou, X., Wang, X., Zhu, D., Zhang, Y. Identification and characterization of human snoRNA core promoters. Genomics. 96 (1), 50-56 (2010).
- Jorjani, H., et al. An updated human snoRNAome. Nucleic Acids Research. 44 (11), 5068-5082 (2016).
- Hubbard, T. J., et al. Ensembl 2009. Nucleic Acids Research. 37 (Database issue), D690-D697 (2009).
- Makarova, J. A., Kramerov, D. A. SNOntology: Myriads of novel snoRNAs or just a mirage?. BMC Genomics. 12, 543 (2011).
- Yu, Y. T., Shu, M. D., Steitz, J. A. A new method for detecting sites of 2'-O-methylation in RNA molecules. RNA. 3 (3), 324-331 (1997).
- Decatur, W. A., Liang, X. H., Piekna-Przybylska, D., Fournier, M. J. Identifying effects of snoRNA-guided modifications on the synthesis and function of the yeast ribosome. Methods in Enzymology. 425, 283-316 (2007).
- Dong, Z. W., et al. RTL-P: a sensitive approach for detecting sites of 2'-O-methylation in RNA molecules. Nucleic Acids Research. 40 (20), e157 (2012).
- Birkedal, U., et al. Profiling of ribose methylations in RNA by high-throughput sequencing. Angewandte Chemie International Edition, England. 54 (2), 451-455 (2015).
- Buchhaupt, M., Peifer, C., Entian, K. D. Analysis of 2'-O-methylated nucleosides and pseudouridines in ribosomal RNAs using DNAzymes. Analytical Biochemistry. 361 (1), 102-108 (2007).
- Grzechnik, P., et al. Nuclear fate of yeast snoRNA is determined by co-transcriptional Rnt1 cleavage. Nature Communication. 9 (1), 1783 (2018).
- Piekna-Przybylska, D., Decatur, W. A., Fournier, M. J. New bioinformatic tools for analysis of nucleotide modifications in eukaryotic rRNA. RNA. 13 (3), 305-312 (2007).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18 (10), 3091-3092 (1990).
- Maden, B. E. Mapping 2'-O-methyl groups in ribosomal RNA. Methods. 25 (3), 374-382 (2001).
- Santoro, S. W., Joyce, G. F. A general purpose RNA-cleaving DNA enzyme. Proceedings of the National Academy of Science, U. S. A. 94 (9), 4262-4266 (1997).
- Hengesbach, M., Meusburger, M., Lyko, F., Helm, M. Use of DNAzymes for site-specific analysis of ribonucleotide modifications. RNA. 14 (1), 180-187 (2008).
- Sednev, M. V., et al. N(6) -Methyladenosine-Sensitive RNA-Cleaving Deoxyribozymes. Angewandte Chemie International Edition, England. 57 (6), 15117-15121 (2018).
- Aschenbrenner, J., Marx, A. Direct and site-specific quantification of RNA 2'-O-methylation by PCR with an engineered DNA polymerase. Nucleic Acids Research. 44 (8), 3495-3502 (2016).
- Lee, K. W., Bogenhagen, D. F. Assignment of 2'-O-methyltransferases to modification sites on the mammalian mitochondrial large subunit 16 S ribosomal RNA (rRNA). Journal of Biological Chemistry. 289 (36), 24936-24942 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten