Method Article
Bildgebung der In Situ Interferon Gamma Produktion in der Maus Milz nach Listeria monocytogenes Infektion
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine einfache konfokale Bildgebungsmethode, um die In-situ-Lokalisierung von Zellen zu visualisieren, die das Zytokin Interferon-Gamma in murinen sekundären Lymphorganen absondern. Dieses Protokoll kann für die Visualisierung anderer Zytokine in verschiedenen Geweben erweitert werden.
Zusammenfassung
Zytokine sind kleine Proteine, die von Zellen abgesondert werden und die Zell-Zell-Kommunikation vermitteln, die für effektive Immunantworten entscheidend sind. Ein Merkmal von Zytokinen ist ihr Pleiotropismus, da sie von einer Vielzahl von Zelltypen produziert werden und beeinflussen können. Daher ist es wichtig, nicht nur zu verstehen, welche Zellen Zytokine produzieren, sondern auch in welcher Umgebung sie dies tun, um spezifischere Therapeutika zu definieren. Hier beschreiben wir eine Methode zur Visualisierung der Zytokinproduktion vor Ort nach einer bakteriellen Infektion. Diese Technik beruht auf bildgebenden Zytokin produzierenden Zellen in ihrer nativen Umgebung durch konfokale Mikroskopie. Dazu werden Gewebeabschnitte für Marker mehrerer Zelltypen zusammen mit einem Zytokinfleck gebeizt. Schlüssel zu dieser Methode, Zytokin-Sekretion wird direkt in vivo vor der Ernte des Gewebes von Interesse blockiert, so dass das Zytokin, das in den produzierenden Zellen angesammelt. Die Vorteile dieser Methode sind vielfältig. Erstens bleibt die Mikroumgebung erhalten, in der Zytokine hergestellt werden, was letztlich über die Signale informieren könnte, die für die Zytokinproduktion erforderlich sind, und über die zellen, die von diesen Zytokinen betroffen sind. Darüber hinaus gibt dieses Verfahren einen Hinweis auf den Standort der Zytokinproduktion in vivo, da es nicht auf einer künstlichen In-vitro-Restimulation der produzierenden Zellen angewiesen ist. Es ist jedoch nicht möglich, zytokin-nachgeschaltete Signalisierung in Zellen zu analysieren, die das Zytokin empfangen. In ähnlicher Weise entsprechen die beobachteten Zytokinsignale nur dem Zeitfenster, in dem die Zytokinsekretion blockiert wurde. Während wir die Visualisierung des Zytokin-Interferon(IFN)-Gammas in der Milz nach einer Mausinfektion durch die intrazellulären Bakterien Listeria monocytogenes beschreiben, könnte diese Methode potenziell an die Visualisierung jedes Zytokins in die meisten Organe.
Einleitung
Die Orchestrierung einer effizienten Immunantwort gegen einen Erreger erfordert eine komplexe Integration von Signalen, die von einer Vielzahl von Immunzellen angezeigt werden, die oft auf den Organismus verteilt sind. Um zu kommunizieren, produzieren diese Zellen kleine lösliche Proteine mit mehreren biologischen Funktionen, die als Immunmodulatoren namens Zytokine fungieren. Zytokine kontrollieren Zellrekrutierung, Aktivierung und Proliferation und sind daher bekannt, Schlüsselakteure bei der Förderung von Immunantworten zu sein1. Effektive Immunantworten erfordern, dass Zytokine in einem sehr organisierten raumzeitlichen Muster freigesetzt werden, das bestimmte Zellen verbindet, um bestimmte Signale zu induzieren. Daher ist es von entscheidender Bedeutung, die Zytokinproduktion und ihre Signalisierung vor Ort zu untersuchen, wobei die Mikroumgebung zu berücksichtigen ist, in der Zytokine hergestellt werden.
Listeria monocytogenes (L. monocytogenes) ist ein grampositives intrazelluläres Bakterium, das als Parademodell zur Untersuchung von Immunreaktionen auf intrazelluläre Krankheitserreger bei Mäusen verwendet wird. Ein Zytokin, IFN-Gamma (IFN) wird schnell produziert, innerhalb von 24 h nach L. monocytogenes Infektion. Es ist notwendig für die Erregung von Krankheitserregern, da Mäuse, die für IFN ausgeschlagen wurden, sehr anfällig für eine L. monocytogenes-Infektion sind2. IFN ist pleiotrop und wird von mehreren Zellen nach der Infektion3produziert. Während IFN, das von natürlichen Killerzellen (NK) produziert wird, für die direkte antibakterielle Aktivität4erforderlich ist, hat IFN aus anderen Quellen nachweislich andere Funktionen. Tatsächlich haben wir und andere vor kurzem festgestellt, dass DIE von CD8+ T-Zellen hergestellte IFN-Zelle eine spezifische Funktion bei der direkten Regulierung der T-Zelldifferenzierung5,6,7hat. Daher ist es entscheidend, zu verstehen, welche Zellen IFN (und in welcher Mikroumgebung) produzieren, um seine Funktion zu sezieren.
Die häufigste Technik zur Untersuchung der Zytokinproduktion beruht auf intrazellulärer Zytokinfärbung, die durch Durchflusszytometrie analysiert wird. Diese Methode ermöglicht die gleichzeitige Detektion mehrerer Zytokine in Kombination mit Zelloberflächenmarkern innerhalb einer einzigen Probe und stellt ein äußerst nützliches Werkzeug zur Untersuchung der Zytokinproduktion bereit. Die Verwendung der oben genannten Technik impliziert jedoch den Verlust jeglicher räumlicher Informationen. Darüber hinaus beruht der Zytokinnachweis häufig auf In-vitro-Restimulation, um die Zytokindetektion zu ermöglichen. Als solche wird die Fähigkeit einer bestimmten Zelle, ein Zytokin zu produzieren, analysiert, und es korreliert nicht notwendigerweise mit der tatsächlichen Zytokinsekretion in situ. Andere Methoden verwenden Reportermäuse, für die fluoreszierende Proteinexpression mit Zytokin-Transkription korreliert und eine Visualisierung auf einer einzelzelligen Ebene8ermöglicht. Obwohl diese Methode Zytokin-Transkription in situ verfolgen kann, gibt es eine begrenzte Anzahl von Zytokin-Reporter-Mäusen zur Verfügung. Darüber hinaus können Transkription, Übersetzung und Sekretion manchmal nicht verknüpft werden, und fluoreszierende Proteine haben eine andere Halbwertszeit als das Zytokin, das sie berichten, was diese Methode manchmal nicht für die Visualisierung von In-situ-Zytokinen geeignet macht.
Hier beschreiben wir eine Methode zur Visualisierung der In-situ-Zytokinproduktion durch konfokale Mikroskopie bei Einzelzellauflösung. Diese Technik ermöglicht die Visualisierung der zellulären Quelle und der umgebenden Nische im Gewebe. Dieses Protokoll beschreibt speziell die Visualisierung der IFN-Produktion in der Milz von L. monocytogenes infizierten Mäusen und konzentriert sich hier auf die IFN-Produktion durch NK-Zellen und antigenspezifische CD8+ T-Zellen. Es kann jedoch erweitert und an die Charakterisierung jeder Zytokinproduktion im Kontext anderer Situationen angepasst werden, in denen Zytokine wie Infektionen, Entzündungen oder Autoimmunerkrankungen produziert werden, solange das angestrebte Zytokin in Zellen zurückgehalten werden kann. durch intrazellulären Proteintransporthemmer.
Protokoll
Alle Versuche mit Mäusen waren mit dem UK Scientific Procedures Act von 1986 einverstanden.
1. Adoptivtransfer von antigenspezifischen CD8+ T-Zellen bei Mäusen
- Isolat ovalbumin (OVA)-spezifische CD8+ T-Zellen (OTI) mit grünem fluoreszierendem Protein (OTI-GFP) oder rotem fluoreszierendem Protein (OTI-RFP) aus Lymphknotensuspension von Transgenen Mäusen des T-Zellrezeptors9,10 mit einer Maus CD8+ T Zellisolationkit gemäß Herstellungsanleitung. Bereiten Sie die Zellsuspension vor, indem Sie Lymphknoten mit einem Spritzenkolben zerschlagen, wie zuvor beschrieben11.
- OTI-GFP- oder OTI-RFP-Zellen (3 x 106 Zellen) in C57BL/6 Wildtyp-Mäuseempfänger durch intravenöse Injektion übertragen, wie von Cahalan, et al.12beschrieben. Verwenden Sie Mäuse, die in der Regel 6-12 Wochen alt sind.
HINWEIS: Dieser Schritt ist optional und nur zum Nachverfolgen antigenspezifischer CD8+ T-Zellen erforderlich.
2. Listeria monocytogenes Infektion
- Erweitern Sie L. monocytogenes genetisch modifiziert, um OVA (LM-OVA)13 zu einer exponentiellen Wachstumsphase der Brüheherzinfusion bei 37 °C unter sanfter Erregung auszudrücken, bis die OD600 0,08–0,1 erreicht, wie zuvor in Bezug beschrieben 14.
- Injizieren Sie 100 l (maximales Volumen = 200 l) von 0,1–0,5 LD50 LM-OVA, die in Phosphatgepufferter Saline (PBS) durch intravenöse Injektion mit einer 29 G Insulinspritze verdünnt werden, in C57BL/6 Wildtyp-Mäuse, die OTI-GFP- oder OTI-RFP-Zellen tragen, wenn angegeben.
HINWEIS: In unseren Händen entspricht 0.1 x LD50 LM-OVA 2 x 104 Koloniebildenden Einheiten (CFU). L. Monocytogene, die genetisch verändert wurden, um OVA auszudrücken, werden verwendet, um die zuvor übertragenen OTI CD8+ T-Zellen zu aktivieren, aber es können andere Stämme von L. monocytogenes verwendet werden.
3. Behandlung mit Brefeldin A (BFA) zur Blockierung der Zytokinsekretion
- Injizieren Sie 250 g BFA in 200 l PBS intraperitoneal 6 h vor dem Mausopfer mit einer 29 G Insulinspritze.
HINWEIS: Lyophilisierte BFA wird zuerst in Dimethylsulfoxid (DMSO) resuspendiert, um einen Vorrat von 25 mg/ml Konzentration zu zuzubereiten. Der BFA wird dann in PBS bei Raumtemperatur (RT) verdünnt, um eine Kristallisation vor der Injektion zu vermeiden. Die Hemmung der Zytokinsekretion induziert eine Ansammlung von IFN in Zellen. Dies ist entscheidend für die Zytokin-Erkennung.
4. Ernte der Milz
- Euthanisieren Sie die Mäuse mit steigender Konzentration von CO 2, gefolgt von zervikalen Dislokation.
HINWEIS: Befolgen Sie die Richtlinien der lokalen Institutionen für die humane Sterbehilfe von Mäusen. - Reinigen Sie den Bauch mit 70% Ethanol, machen Sie einen Schnitt mit der Schere, um einen 1-2 cm schnitt durch die Haut auf der linken Flanke der Maus, wo die Milz befindet. Machen Sie vorsichtig einen Schnitt in das Peritoneum, um die Milz zu entblößen und nehmen Sie es mit einer Pinzette heraus. Ernten Sie die Milz, achten Sie darauf, sie nicht mit Zangen zu drücken oder zu schneiden, um die Milzarchitektur nicht zu stören.
5. Fixierung der Milz mit Paraformaldehyd (PFA)
- Bereiten Sie die fixative Lösung durch Mischen von 3,75 ml PBS und 3,75 ml 0,2 M L-Lysin vor. 21 mg Natriummperiodat hinzufügen und gut vermischen. Dann fügen Sie 2,5 ml von 4% PFA und 20 l von 12 N NaOH.
HINWEIS: Verwenden Sie die Fixierlösung am selben Tag und verwerfen Sie den Überschuss. Bewahren Sie es nicht auf. Dieser Fixierungsschritt ist wichtig, wenn die Probe fluoreszierende Proteine wie GFP enthält. Verwenden Sie kein PFA, das Spuren von Methanol enthält, da es fluoreszierende Proteine denaturiert.
ACHTUNG: PFA ist giftig und muss mit Vorsicht behandelt werden. - Untertauchen Sie die Milz in das Fixativ und fixieren Sie für mindestens 4 h, in der Regel 16–20 h bei 4°C unter sanfter Erregung.
- Entsorgen Sie die fixative Lösung und fügen Sie 5 ml PBS für 5 min bei RT unter sanfter Erregung hinzu.
- Ersetzen Sie die PBS durch 5 ml frische PBS-Inkubation für 1 h bei 4 °C unter sanfter Rührung.
- Ersetzen Sie die PBS durch 5 ml 30% Saccharose, inkubieren für 12-24 h.
HINWEIS: Diese Methode hilft, die Gewebemorphologie zu erhalten. Nach der Inkubation mit Saccharoselösung sollte das Organ am Boden des Brunnens sinken.
6. Einfrieren und Sektionieren
- Trockeneis in eine große Aufnahme geben und einen kleineren Behälter mit ca. 50 ml reinem Methanol und ein paar Stück Trockeneis ins Innere stellen.
- Die Milz vorsichtig auf einem fusselfreien Tuch trocknen.
- Legen Sie die Milz in eine Basisform, die einen Tropfen der optimalen Schnitttemperatur (OCT) Verbindung an der Unterseite enthält. Achten Sie darauf, keine Blasen zu produzieren. Fügen Sie OCT über die Milz.
- Mit Zangen, legen Sie die Basisform auf der Oberfläche des Methanols, um sicherzustellen, dass es nicht das OCT berühren. Einfrieren des Gewebes so schnell wie möglich, um Artefakte zu minimieren.
- Wenn es eingefroren ist, fahren Sie mit dem Schnitt fort.
HINWEIS: Die gefrorene Milz kann mehrere Monate bei -80 °C aufbewahrt werden. -
Abschnitt des Gewebes mit einem Kryomikrotom.
- Stellen Sie die Kammertemperatur am Kryostat auf -21 °C ein. Schnittabschnitte der gewünschten Dicke (in der Regel ca. 10 m). Dieses Protokoll arbeitet mit Dicken bis zu 30 m.
- Sammeln Sie Abschnitte auf Glasmikroskop-Dias (siehe Materialtabelle) und überprüfen Sie visuell.
HINWEIS: Abschnitte können mehrere Monate bei -80 °C aufbewahrt werden.
7. Immunfluoreszierende Färbung
- Lassen Sie den Abschnitt zu RT kommen.
- Zeichnen Sie einen Kreis mit einem Flüssigkeitsblocker (z. B. PAP-Stift) um den Gewebeabschnitt. Zeichnen Sie außerhalb des OCT oder es wird nicht kleben.
- Nach dem Trocknen rehydrieren Sie die Probe, indem Sie PBS für 5 min auf den Gewebeabschnitt legen.
HINWEIS: Das auf den Abschnitt gesetzte Volumen hängt von der Größe des Abschnitts ab. In der Regel verwenden wir 100–300 l. Lassen Sie die Abschnitte nicht trocknen, sobald sie rehydriert sind. - Spülen Sie mindestens zweimal mit PBS ab, um sicherzustellen, dass der Abschnitt gut an der Folie verhält.
-
Fügen Sie dem Abschnitt eine Blockierungslösung hinzu, um die unspezifische Bindung von Antikörpern zu verringern.
- Bereiten Sie die Blockierlösung wie folgt vor: PBS mit 0,1% Triton X100, 2% fetales Kalbsserum (FCS), 2,5 g/ml Fc-Rezeptorblocker (Anti-Maus-CD16/32). Fügen Sie dann 2–5% normales Serum der Spezies jedes sekundären Antikörpers der Färbeplatte hinzu.
HINWEIS: Wenn die Antikörper direkt konjugiert/biotinyliert sind, fügen Sie 5% normales Serum der Arten jedes primären Antikörpers hinzu. Wenn ein Primärantikörper und einer der sekundären Antikörper von derselben Art stammen (z. B. Primärantikörper, die bei Kaninchen und Sekundärkaninchen gegen Ratten aufgezogen werden), verwenden Sie nicht die normale Serumart, da sie das Hintergrundsignal erhöht. - Entfernen Sie den PBS vorsichtig aus dem Abschnitt durch Aspiration, und fügen Sie 100 L der Blockierlösung pro Probenabschnitt hinzu. Inkubieren Sie in einer überdachten Nasskammer für mindestens 1 h bei RT.
- Bereiten Sie die Blockierlösung wie folgt vor: PBS mit 0,1% Triton X100, 2% fetales Kalbsserum (FCS), 2,5 g/ml Fc-Rezeptorblocker (Anti-Maus-CD16/32). Fügen Sie dann 2–5% normales Serum der Spezies jedes sekundären Antikörpers der Färbeplatte hinzu.
-
Stain mit primären Antikörpern.
- Verdünnen Sie die primären Antikörper in der optimalen Konzentration in der Blockierlösung. Die allgemeine Ausgangs-Antikörper-Konzentration beträgt 5 g/ml, sollte aber für jeden Antikörper und jedes Gewebe optimiert werden.
HINWEIS: Wenn die Antikörper direkt konjugiert sind, zentrifugieren Sie den Antikörper bei 17.135 x g (13.500 U/min) für 15 min bei 4 °C, bevor Sie ihn verwenden. Fluorophore können ausfallen. Dieser Schritt wird die Ausscheidungen pellet und somit eine unspezifische Ablagerung der gefällten Antikörper auf dem Dia verhindern. - Ersetzen Sie die Blockierlösung durch den primären Antikörpermix für jede Probe.
- 4 h bei RT oder über Nacht (OVN) bei 4 °C in einer überdachten Nasskammer inkubieren.
- Verdünnen Sie die primären Antikörper in der optimalen Konzentration in der Blockierlösung. Die allgemeine Ausgangs-Antikörper-Konzentration beträgt 5 g/ml, sollte aber für jeden Antikörper und jedes Gewebe optimiert werden.
-
Waschen.
- Bereiten Sie Denwaschpuffer vor, indem Sie PBS 2% FCS hinzufügen.
- 4 Mal mit Waschpuffer waschen: eine schnelle (ohne Inkubation), eine für 10 min und zwei für 5 min. Dann führen Sie eine letzte Wäsche mit PBS für 5 min.
-
Färbung mit sekundären Antikörpern.
- Verdünnen Sie die sekundären Antikörper von Interesse bei der optimalen Konzentration in der Blockierlösung. Zentrifuge die Mischung, wie für die primären Antikörper beschrieben.
- Entfernen Sie die endende Waschlösung. Fügen Sie die sekundäre Antikörpermischung auf den Abschnitt und inkubieren Für 1-4 h bei RT in einer überdachten Nasskammer.
- 4 Mal mit Waschpuffer waschen: eine schnelle (ohne Inkubation), eine für 10 min und zwei für 5 min. Dann führen Sie eine letzte Wäsche mit PBS für 5 min.
- Entfernen Sie die endende Waschlösung. Lassen Sie die PBS verdampfen, aber trocknen Sie den Abschnitt nicht übertrocknen. Legen Sie einen Tropfen des Montagemediums auf die Probe und legen Sie das Deckglas vorsichtig darauf. Das Montagemedium muss den gesamten Abschnitt wiederherstellen. Lassen Sie es OVN bei RT vor Licht geschützt.
HINWEIS: Zeichnen Sie einen Kreis um den Abschnitt auf der Rückseite des Schlittens, bevor Sie das Montagemedium anwenden. Sobald das Montagemedium aufgetragen wird, kann das Gewebe schwer zu sehen sein. - Bewahren Sie die Dias im Dunkeln bei 4 °C auf, bis sie bildbereit sind.
8. Bildgebung und Analyse
- Bildgebung der Färbung mit einem konfokalen Mikroskop durchführen.
HINWEIS: In diesem Protokoll wurde ein invertiertes spektrales Laserscanning-Mikroskop verwendet (siehe Materialtabelle), zusammen mit den Objektiven 10x/NA 0.40 oder 60x/NA 1.4 (zur Analyse der subzellulären Lokalisierung von Zytokin). Wellenlängen der Anregung und Emission werden für jedes Fluorophor und fluoreszierendes Protein im Materialverzeichnisangezeigt. - Führen Sie die Analyse und Quantifizierung nach Bedarf mit Bildverarbeitungssoftware (z.B. Imaris oder Fidschi) durch.
Ergebnisse
Die innerhalb der ersten 24 h nach Listeria monocytogenes-Infektion produzierte IFN ist entscheidend, um die Ausbreitung dieses Erregers zu kontrollieren. Mit diesem Protokoll können wir nicht nur visualisieren, welche Zellen IFN produzieren, sondern auch, ob sie sich in einer bestimmten Mikroumgebung befinden. Um uns zu helfen, die Architektur der Milz abzugrenzen, haben wir Zellen, von denen bekannt ist, dass sie eine bestimmte Position innerhalb der Milz haben, als bestimmte Position bezeichnet. Der Marker F4/80 beschriftet alle Makrophagen und hebt das rote Fruchtfleisch hervor. Der Marker B220 beschriftet B-Zellen und hebt B-Zellfollikel um die T-Zellzone hervor. Der Marker CD169 kennzeichnet Makrophagen der Randzone, die den weißen Zellstoff umgeben (Abbildung 1). Die meisten OTI-Zellen, ob sie IFN ausdrücken oder nicht, sind im weißen Zellstoff vorhanden und als solche sind alle Bilder die des weißen Zellstoffs, sofern nicht angegeben.
Ein wichtiger Schritt in diesem Protokoll ist die Verwendung von BFA, um die Zytokinsekretion zu hemmen. Tatsächlich war der Nachweis von IFN durch NK-Zellen stark beeinträchtigt, wenn Mäuse nicht mit BFA behandelt wurden (Abbildung 2). Anhand unseres Protokolls konnten wir feststellen, dass mindestens zwei Zelltypen nach einer Infektion IFN-24 h erzeugen – NK-Zellen und antigenspezifische CD8+ T-Zellen (Abbildung 3) – ähnlich wie bisher durch Durchflusszytometrie3gefunden wurde.
Die In-situ-Bildgebung der IFN-produzierenden Zellen zeigte, dass die IFN-Produktion nicht über die Milz verteilt ist, sondern sich auf diskrete Bereiche konzentriert (Abbildung 4). Tatsächlich fanden wir heraus, dass T-Zellen während der gesamten Milz aktiviert wurden (hervorgehoben durch T-Zell-Clustering), und dies korrelierte nicht unbedingt mit der IFN-Produktion. Eine wahrscheinliche Erklärung ist, dass die IFN-Produktion auf den Standort der infizierten Zellen15,16und T-Zellaktivierung – dargestellt durch Clustering – sowohl von Infizierten (IFN-positiv) als auch von nicht infizierten (IFN-positiven) unterstützt werden kann. negative) antigenpräsentierende Zellen. Andere Flecken werden erforderlich sein, um den genauen Ort zu bestimmen und einen Hinweis auf den Mechanismus zu erhalten, der die IFN-Produktion auf diesen Bereich und seine Beziehung zur Antigenübertragung beschränkt. Interessanterweise fanden wir heraus, dass sich aktivierte, gruppierte, antigenspezifische T-Zellen im weißen Zellstoff der Milz befinden, aber sie produzieren IFN nur in Regionen, in denen NK-Zellen mit ihnen koexistieren (Abbildung 5). Daher umfliegt das Vorhandensein von NK-Zellen eine bestimmte Mikroumgebung im weißen Zellstoff, in der gruppierte T-Zellen IFN-Zellen produzieren, im Gegensatz zu gruppierten T-Zellen im anderen Teil des weißen Zellstoffs. Dies deutet darauf hin, dass die T-Zellaktivierung nicht ausreicht, um die IFN-Produktion zu diesem Zeitpunkt zu diktieren.
Ein weiteres interessantes Merkmal, das durch unser Protokoll hervorgehoben wird, ist die unterschiedliche subzelluläre Lokalisierung von IFN in NK im Vergleich zu CD8+ T-Zellen5. Wie in Abbildung 6dargestellt, während die IFN-Lokalisierung in NK-Zellen im Zytosol diffundiert wird, rekrutieren CD8+ T-Zellen oft IFN in eine andere T-Zelle.

Abbildung 1: Marker, die die Milzarchitektur hervorheben. Mäuse wurden mit 2 x 104 CFU LM-OVA infiziert und 24 h nach der Infektion eingeschläfert. Spleen wurde wie im Protokoll beschrieben explantiert und verarbeitet. (A) Abschnitte wurden für NK-Zellen (Anti-NCR1 gefolgt von Anti-Ziegen IgG-FITC; grün), OTI-RFP-Zellen (rot) und Makrophagen (Anti-F4/80-APC; Magenta) gefärbt. RP = Roter Zellstoff; WP = Weißer Zellstoff. Skala bar = 200 m. (B) Abschnitte wurden für B-Zellen (Anti-B220-Pacific Blue; Blau), OTI-GFP-Zellen (GFP-Signal rot dargestellt) und Marginalzonenmakrophagen (Anti-CD169-Alexa647; Magenta). RP = Roter Zellstoff; BF = B-Zellfollikel; TZ = T-Zellenzone. Skalenbalken = 50 m. Dies ist ein repräsentatives Bild von 3 unabhängigen Experimenten (N = 4). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
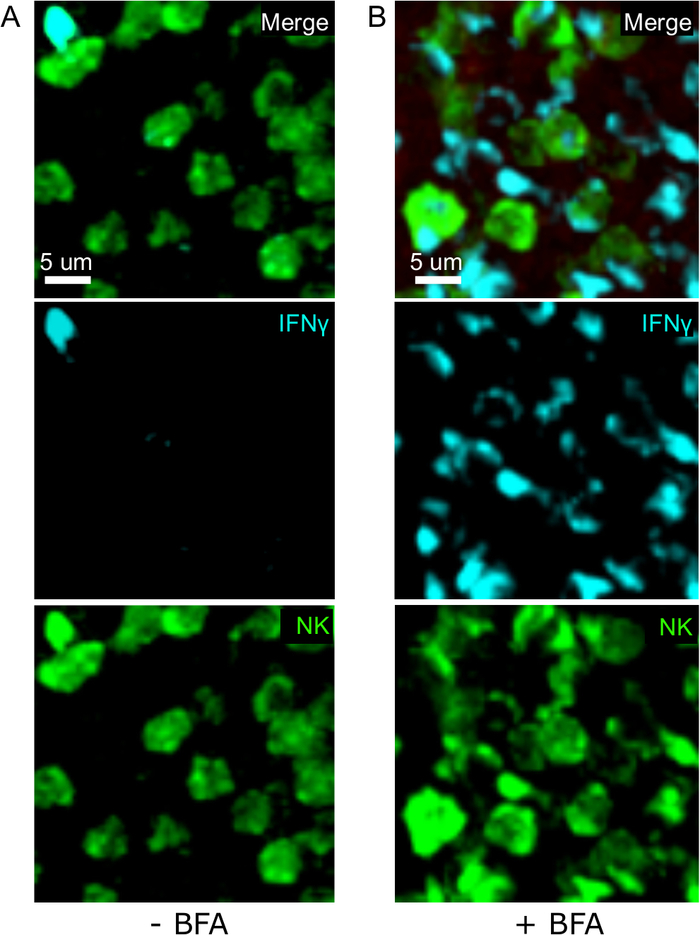
Abbildung 2: Die BFA-Behandlung ermöglicht den Nachweis von intrazellulärem IFN-In-situ. Die Produktion ist auf bestimmte Bereiche in der Milz beschränkt Mäuse wurden mit 2 x 104 CFU LM-OVA infiziert und mit BFA behandelt (A) oder unbehandelt (B) nach 18 h. Mäuse wurden 24 h nach der Infektion eingeschläfert. Spleen wurde wie im Protokoll beschrieben explantiert und verarbeitet. Abschnitte wurden für NK-Zellen (Anti-NCR1 gefolgt von Anti-Ziegen-IgG-FITC; grün), OTI-RFP-Zellen (rot) und IFN (Anti-IFN-BV421; Cyan) gefärbt. Skalenbalken = 5 m. Dies ist ein repräsentatives Bild von NK-Zellreichen Bereichen aus 3 unabhängigen Experimenten (N = 3). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
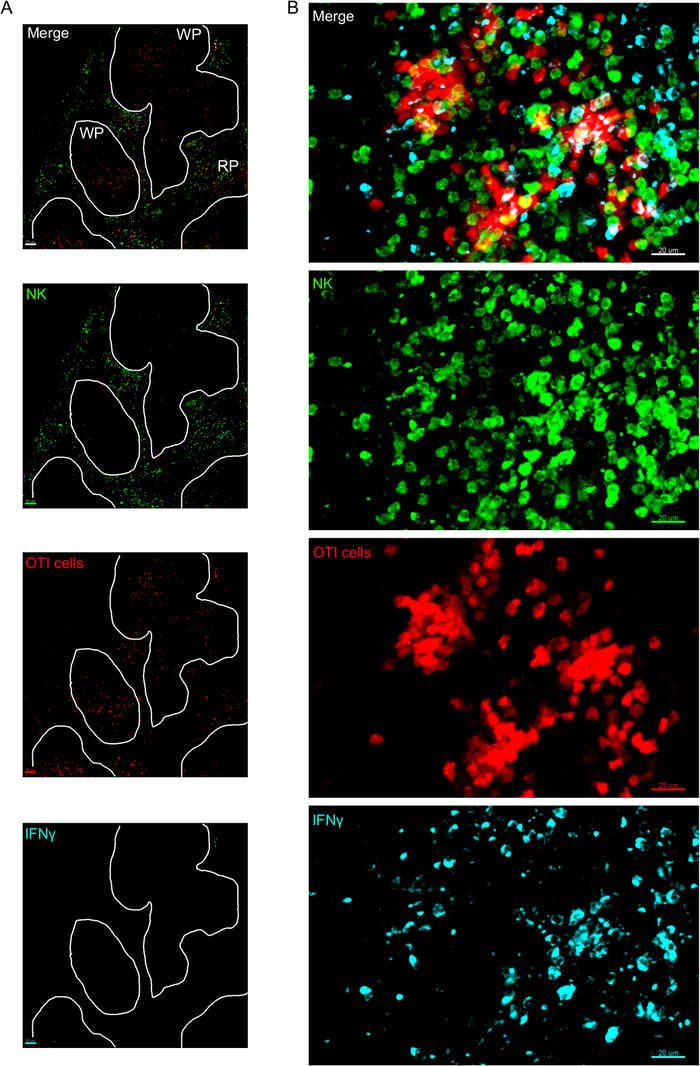
Abbildung 3: IFN-produzierende Zellen in der Milz. Mäuse wurden mit 2 x 104 CFU LM-OVA infiziert, wenn sie nach 18 h mit BFA indiziert und behandelt wurden. Mäuse wurden 24 h nach einer Infektion eingeschläfert. Spleen wurde wie im Protokoll beschrieben explantiert und verarbeitet. Abschnitte wurden für NK-Zellen (Anti-NCR1 gefolgt von Anti-Ziegen-IgG-FITC; grün), OTI-RFP-Zellen (rot) und IFN (Anti-IFN-BV421; Cyan) gefärbt. (A) Repräsentatives Bild einer Milz einer nicht infizierten naiven Maus, um das Fehlen einer unspezifischen Färbung von IFN nachzuweisen. Weiße Linien umleiten den weißen Zellstoff. WP = Weißer Zellstoff; RP = Roter Zellstoff. (B) Repräsentatives Bild des weißen Zellstoffs aus der Milz einer mit LM-OVA infizierten Maus, die die Invasion von NK-Zellen in den weißen Zellstoff und die Produktion von IFN durch NK-Zellen, OTI-Zellen und nicht gekennzeichnete Zellen zeigt. Die Bilder sind repräsentativ aus 4 unabhängigen Experimenten (N = 4). Skalenbalken = 70 m (A); und 20 m (B). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
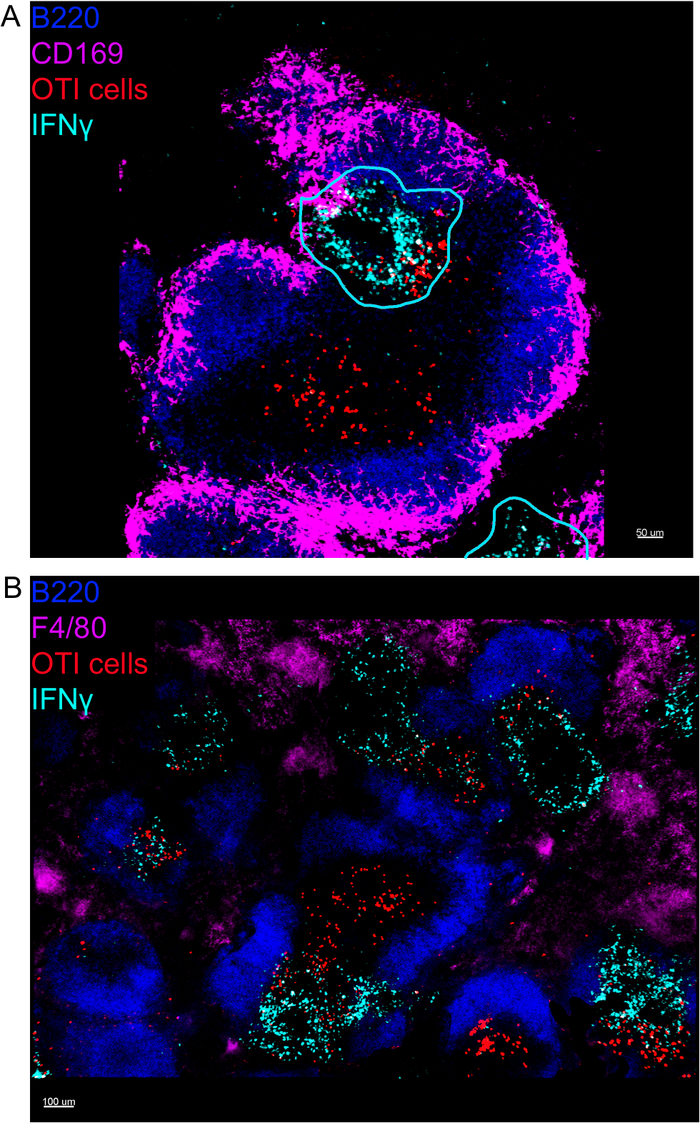
Abbildung 4: Die IFN-Produktion ist nach der LM-OVA-Infektion auf bestimmte Bereiche in der Milz beschränkt. Mäuse wurden mit 2 x 104 CFU LM-OVA infiziert und nach 18 h mit BFA behandelt. Mäuse wurden 24 h nach einer Infektion eingeschläfert. Spleen wurde wie im Protokoll beschrieben explantiert und verarbeitet. Alle Abschnitte wurden für B-Zellen (B220-Pacific Blue Ab, Blue) und IFN(Anti-IFN-Biotin gefolgt von Streptavidin-PE; Cyan) gefärbt. OTI-GFP-Zellen (GFP-Signal rot dargestellt). Cyan-Linien entsprechen Gebieten mit hoher IFN-Produktion. Dabei handelt es sich um repräsentative Bilder von 4 unabhängigen Experimenten (N = 4). (A) Abschnitte wurden für Makrophagen der Grenzzone (Anti-CD169-Alexa 647, Magenta) gefärbt. Skalenbalken = 50 m (B) Abschnitte wurden für alle Makrophagen gebeizt (F4/80). Maßstabsleiste = 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
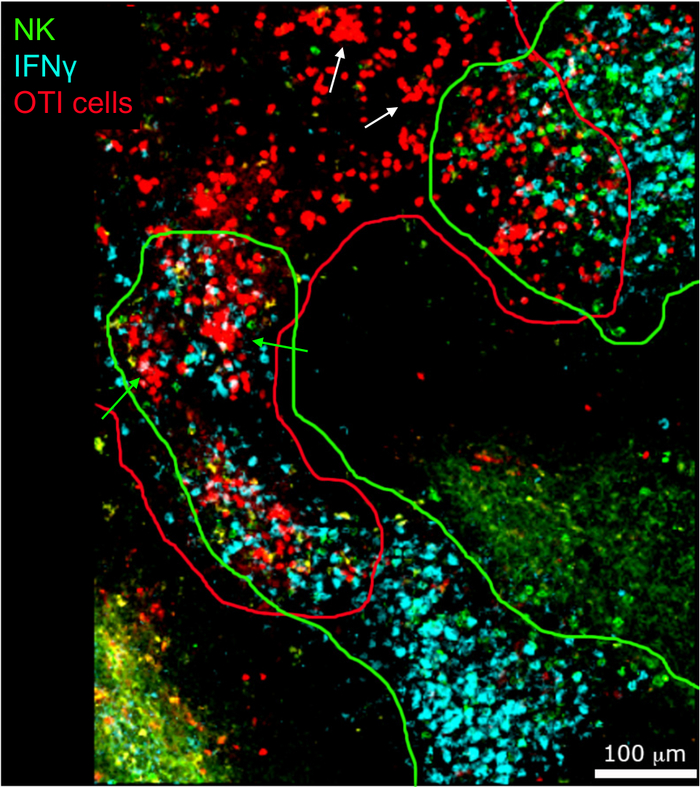
Abbildung 5: Die IFN-Produktion durch aktivierte OTI-Zellen findet in einer bestimmten Mikroumgebung statt. Mäuse wurden mit 2 x 104 CFU LM-OVA infiziert und nach 18 h mit BFA behandelt. Mäuse wurden 24 h nach einer Infektion eingeschläfert. Milz wurden explantiert und verarbeitet, wie im Protokoll beschrieben. Abschnitte wurden für NK-Zellen (Anti-NCR1 gefolgt von Anti-Ziegen-IgG-FITC; grün), OTI-RFP-Zellen (rot) und IFN (Anti-IFN-BV421; Cyan) gefärbt. Grüne und rote Linien markieren NK- bzw. OTI-Zellenzonen. Weißer Pfeil zeigt Beispiele für T-Zellcluster an, die keine IFN erzeugen. Beispiele für grüne Pfeile von T-Zellenclustern, die IFN erzeugen. Skalenbalken = 100 m. Dies ist ein repräsentatives Bild von vier unabhängigen Experimenten (N = 4). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
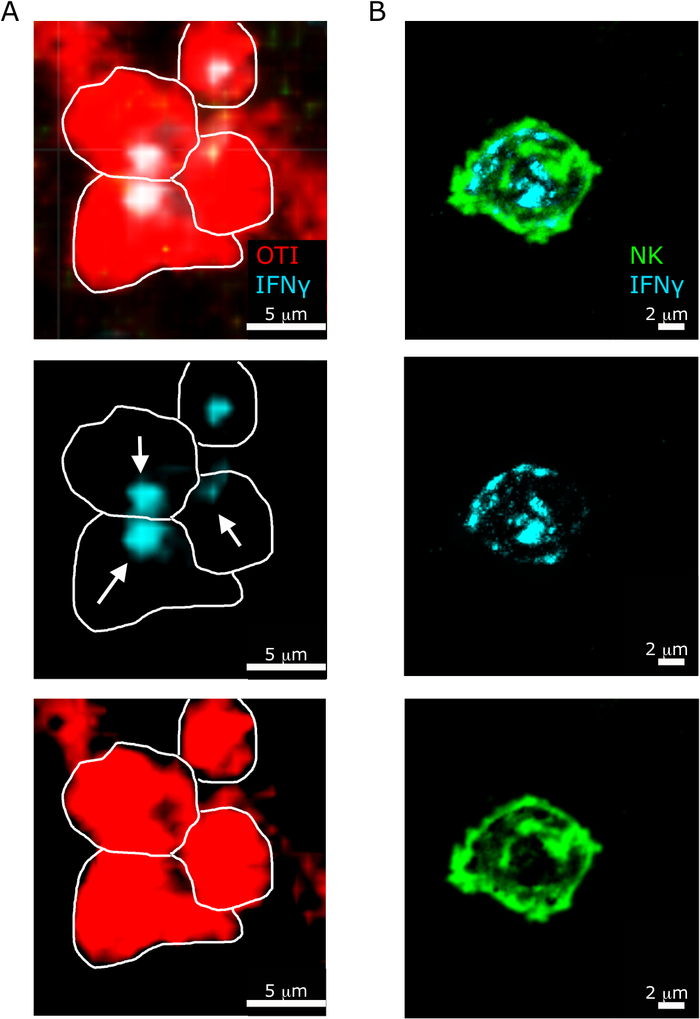
Abbildung 6: Subzelluläre Lokalisierung von IFN in NK-Zellen und T-Zellen. Mäuse wurden mit 2 x 104 CFU LM-OVA infiziert und nach 18 h mit BFA behandelt. Mäuse wurden 24 h nach einer Infektion eingeschläfert. Spleen wurde wie im Protokoll beschrieben explantiert und verarbeitet. Alle Abschnitte wurden für IFN (Anti-IFN-BV421; Cyan) gefärbt. Weiße Linien umgrenzen Zellkanten und weiße Pfeile zeigen die Richtung der Sekretion an. Dies ist ein repräsentatives Bild von zwei unabhängigen Experimenten (N = 5). (A)- OTI-RFP-Zellen sind rot dargestellt. Skala bar = 5 m. (B) Abschnitte wurden für NK-Zellen gefärbt (Anti-NCR1 gefolgt von Anti-Ziegen IgG-FITC; grün. Maßstabsleiste = 2 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
In diesem Manuskript stellen wir eine Methode vor, um die IFN-Produktion in der Milz nach der L. monocytogenes-Infektion bei Mäusen zu visualisieren. Dieses Protokoll ist einfach und kann an andere Gewebe und Zytokin-Trigger angepasst werden, aber die folgenden Aspekte müssen berücksichtigt werden. Zellen sezernieren oft schnell die Zytokine, die sie produzieren, und Zytokine werden schnell von benachbarten Zellen aufgenommen. Es ist als solches schwierig, Zytokine vor Ort zu erkennen. Eine gängige Methode zur schnellen Wiederbelebung der Zytokinproduktion besteht darin, die Zellen ex vivo, gefolgt von Zytokindetektion in den Medien durch enzymgebundenen Immunsorbent-Assay, neu zu stimulieren. In diesem Zusammenhang gehen alle Informationen über die räumliche Lokalisierung der Zytokin produzierenden Zellen verloren. Darüber hinaus spiegelt die Zytokinproduktion nach Restimulation nicht notwendigerweise wider, ob Zytokine tatsächlich in vivo produziert und abgesondert werden, sondern zeigt vielmehr die Fähigkeit einer bestimmten Zellpopulation an, Zytokine herzustellen. Daher werden beide Methoden unterschiedliche Informationen liefern, und man sollte sich überlegen, welche Informationen für ihr Experiment am wertvollsten sind.
Um intrazelluläre Zytokine zu detektieren, verwendet unsere Methode einen intrazellulären Proteintransporthemmer, um Zytokine in Zellen zu fangen und die Signalerkennung zu erhöhen. Es ist jedoch wichtig zu beachten, dass diese Inhibitoren den normalen Transport von Proteinen vom endotheliaalen Retikulum (RE) zum Golgi-Apparat und zum sekretorischen Vesikel beeinflussen, das ihre Freisetzung beeinträchtigt, was Toxizität verursachen könnte. Infolgedessen sollte BFA, oder andere Inhibitor, für einen kurzen Zeitraum verwendet werden, in der Regel nicht mehr als ein paar Stunden. Daher ist es wichtig, das richtige Gleichgewicht zwischen der Inhibitordosis und dem Zeitpunkt der Behandlung zu finden, um das Niveau der in der Zelle eingeschlossenen Zytokine zu optimieren, ohne schwerwiegende zytotoxische Wirkungen zu verursachen. Diese Variablen können zwischen Zytokinen und dem Verabreichungsweg für den BFA unterschiedlich sein. In unserem Infektionsmodell wurde die BFA intraperitonely verwaltet, um eine schnelle systemische Dispersion zu gewährleisten, aber sie kann auch intravenös geliefert werden.
Die am häufigsten verwendeten intrazellulären Proteintransportinhibitoren sind BFA, hier verwendet, und Monensin (MN). Diese Inhibitoren werden oft undeutlich verwendet, um die Zytokinproduktion anzusammeln und zu untersuchen, aber sie haben leichte Unterschiede in ihren Wirkmechanismen. MN hemmt den Transport von Proteinen innerhalb des Golgi-Apparats und sammelt somit Proteine im Golgi17 an, während BFA die Rekrutierung von Coatomer-Protein-Komplex-I verhindert und die retrograde Bewegung von Proteinen zum endoplasmatischen Retikulum hemmt (ER) und fördert damit die Anhäufung von Zytokinen im ER18. Daher hängt die Wahl des besten intrazellulären Proteintransportinhibitors von verschiedenen Faktoren ab, wie z. B. dem zu detektierenden Zytokin. Zum Beispiel wurde bei Lipopolysaccharid-induzierten intrazellulären Färbungen von Monozyten gezeigt, dass BFA effizienter ist, um die Zytokine IL-1, IL-6 und TNF als MN19zu messen.
Dieses Protokoll beinhaltet die Visualisierung des Zytokins in situ durch konfokale Mikroskopie und daher gibt es nur eine begrenzte Anzahl von Markern, die zur Untersuchung der Zytokin produzierenden Zellen und ihrer Mikroumgebung verwendet werden können. Es ist auch zu berücksichtigen, dass Proteintransportinhibitoren wie BFA oder MN die normale Expression mehrerer Proteine stören und daher ihre Verwendung bei der Untersuchung der gleichzeitigen Expression bestimmter Aktivierungszelloberflächenmarker angegangen werden muss. sorgfältig. Zum Beispiel blockiert BFA, aber nicht MN die Expression von CD69 in murinen Lymphozyten20. Trotz dieser Einschränkung ermöglicht die konfokale Bildgebung die subzelluläre Lokalisierung von Zytokinen sowie die Richtung der Zytokinsekretion innerhalb der Zelle. Die mit diesem Protokoll generierten Daten deuten darauf hin, dass NK-Zellen dazu neigen, IFN-y in einem diffusen Muster zu sezernieren, während CD8+ T-Zellen die IFN-Sekretion auf andere CD8+ T-Zellen zu lenken scheinen, die in direkter Interaktion mit ihnen stehen5.
Abschließend möchte ich sagen, dass dieses Protokoll geeignet ist, eine Vielzahl von Zytokinen vor Ort zu visualisieren und produzierende Zellen und ihre Mikroumgebung nach vielen Auslösern wie Infektion oder Autoimmunität zu identifizieren. Die erhaltenen Informationen sind entscheidend, um die Bedeutung der in vivo räumlichen Orchestrierung verschiedener Zelltypen und des von ihnen erzeugten Zytokins zu verstehen, das für eine effiziente Immunantwort notwendig ist.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken dem Personal des Kennedy Institute Imaging Facility für die technische Unterstützung bei der Bildgebung. Diese Arbeit wurde durch Stipendien des Kennedy Trust (an A.G.) und des Biotechnology and Biological Sciences Research Council (BB/R015651/1 bis A.G.) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Brefeldin A | Cambridge bioscience | CAY11861 | |
| Paraformaldehyde | Agar scientific | R1018 | |
| L-Lysin dihydrochloride | Sigma lifescience | L5751 | |
| Sodium meta-periodate | Thermo Scientific | 20504 | |
| D(+)-saccharose | VWR Chemicals | 27480.294 | |
| Precision wipes paper Kimtech science | Kimberly-Clark Professional | 75512 | |
| O.C.T. compound, mounting medium for cryotomy | VWR Chemicals | 361603E | |
| Fc block, purified anti-mouse CD16/32, clone 93 | Biolegend | 101302 | Antibody clone and Concentration used: 2.5 mg/ml |
| Microscope slides - Superfrost Plus | VWR Chemicals | 631-0108 | |
| anti-CD169 - AF647 | Biolegend | 142407 | Antibody clone and Concentration used: clone 3D6.112 1.6 mg/ml Excitation wavelength: 650 Emission wavelength: 65 |
| anti-F4/80 - APC | Biolegend | 123115 | Antibody clone and Concentration used: clone BM8 2.5 mg/ml Excitation wavelength: 650 Emission wavelength: 660 |
| anti-B220 - PB | Biolegend | 103230 | Antibody clone and Concentration used: clone RA3-6B2 1.6 mg/mL Excitation wavelength: 410 Emission wavelength: 455 |
| anti-IFNg - biotin | Biolegend | 505804 | Antibody clone and Concentration used: clone XMG1.2 5 mg/mL |
| anti-IFNg - BV421 | Biolegend | 505829 | Antibody clone and Concentration used: clone XMG1.2 5 mg/mL Excitation wavelength: 405 Emission wavelength: 436 |
| anti-Nkp46/NCRI | R&D Systems | AF2225 | Antibody clone and Concentration used: goat 2.5 mg/mL |
| anti-goat IgG-FITC | Novusbio | NPp 1-74814 | Antibody clone and Concentration used: 1 mg/mL Excitation wavelength: 490 Emission wavelength: 525 |
| Streptavidin - PE | Biolegend | 405203 | Antibody clone and Concentration used: 2.5 mg/mL Excitation wavelength: 565 Emission wavelength: 578 |
| Streptavidin - FITC | Biolegend | 405201 | Antibody clone and Concentration used: 2.5 mg/mlL Excitation wavelength: 490 Emission wavelength: 525 |
| Fluoromount G | SouthernBiotech | 0100-01 | |
| Cover glasses 22 mm x 40 mm | Menzel-Glazer | 12352128 | |
| Liquid blocker super PAP PEN mini | Axxora | CAC-DAI-PAP-S-M | |
| Imaris - Microscopy Image Analysis Software | Bitplane | ||
| Confocal microscope - Olympus FV1200 Laser scanning microscope | Olympus | ||
| Cryostat - CM 1900 UV | Leica | ||
| Base mould disposable | Fisher Scientific UK Ltd | 11670990 | |
| PBS 1x | Life Technologies Ltd | 20012068 | |
| BHI Broth | VWR Brand | 303415ZA | |
| GFP | Excitation wavelength: 484 Emission wavelength: 507 | ||
| RFP | Excitation wavelength: 558 Emission wavelength: 583 | ||
| Insulin syringe, with needle, 29 G | VWR International | BDAM324824 | |
| C57BL/6 wild type mice | Charles River |
Referenzen
- Iwasaki, A., Medzhitov, R. Control of adaptive immunity by the innate immune system. Nature Immunology. 16 (4), 343-353 (2015).
- Harty, J. T., Bevan, M. J. Specific immunity to Listeria monocytogenes in the absence of IFN gamma. Immunity. 3 (1), 109-117 (1995).
- Kubota, K., Kadoya, Y. Innate IFN-gamma-producing cells in the spleen of mice early after Listeria monocytogenes infection: importance of microenvironment of the cells involved in the production of innate IFN-gamma. Frontiers in Immunology. 2 (26), (2011).
- Dunn, P. L., North, R. J. Early gamma interferon production by natural killer cells is important in defense against murine listeriosis. Infection and Immunity. 59 (9), 2892-2900 (1991).
- Krummel, M. F., et al. Paracrine costimulation of IFN-gamma signaling by integrins modulates CD8+ T cell differentiation. Proceedings of the National Academy of Sciences of the United States of America. 115 (45), 11585-11590 (2018).
- Curtsinger, J. M., Agarwal, P., Lins, D. C., Mescher, M. F. Autocrine IFN-gamma promotes naive CD8+ T cell differentiation and synergizes with IFN-alpha to stimulate strong function. Journal of Immunology. 189 (2), 659-668 (2012).
- Hosking, M. P., Flynn, C. T., Whitton, J. L. Antigen-specific naive CD8++ T cells produce a single pulse of IFN-gamma in vivo within hours of infection, but without antiviral effect. Journal of Immunology. 193 (4), 1873-1885 (2014).
- Croxford, A. L., Buch, T. Cytokine reporter mice in immunological research: perspectives and lessons learned. Immunology. 132 (1), 1-8 (2011).
- Gerard, A., et al. Secondary T cell-T cell synaptic interactions drive the differentiation of protective CD8++ T cells. Nature Immunology. 14 (4), 356-363 (2013).
- Engelhardt, J. J., et al. Marginating dendritic cells of the tumor microenvironment cross-present tumor antigens and stably engage tumor-specific T cells. Cancer Cell. 21 (3), 402-417 (2012).
- Matheu, M. P., Cahalan, M. D. Isolation of CD4+ T cells from mouse lymph nodes using Miltenyi MACS purification. Journal of Visualized Experiments. (9), 409 (2007).
- Matheu, M. P., Parker, I., Cahalan, M. D. Dissection and 2-photon imaging of peripheral lymph nodes in mice. Journal of Visualized Experiments. (7), 265 (2007).
- Pope, C., et al. Organ-specific regulation of the CD8+ T cell response to Listeria monocytogenes infection. Journal of Immunology. 166 (5), 3402-3409 (2001).
- Jones, G. S., D'Orazio, S. E. Listeria monocytogenes: cultivation and laboratory maintenance. Current Protocols in Microbiology. 31, 1-7 (2013).
- Kang, S. J., Liang, H. E., Reizis, B., Locksley, R. M. Regulation of hierarchical clustering and activation of innate immune cells by dendritic cells. Immunity. 29 (5), 819-833 (2008).
- Chang, S. R., et al. Characterization of early gamma interferon (IFN-gamma) expression during murine listeriosis: identification of NK1.1+ CD11c+ cells as the primary IFN-gamma-expressing cells. Infection and Immunity. 75 (3), 1167-1176 (2007).
- Mollenhauer, H. H., Morre, D. J., Rowe, L. D. Alteration of intracellular traffic by monensin; mechanism, specificity and relationship to toxicity. Biochimica et Biophysica Acta. 1031 (2), 225-246 (1990).
- Helms, J. B., Rothman, J. E. Inhibition by brefeldin A of a Golgi membrane enzyme that catalyses exchange of guanine nucleotide bound to ARF. Nature. 360 (6402), 352-354 (1992).
- Schuerwegh, A. J., Stevens, W. J., Bridts, C. H., De Clerck, L. S. Evaluation of monensin and brefeldin A for flow cytometric determination of interleukin-1 beta, interleukin-6, and tumor necrosis factor-alpha in monocytes. Cytometry. 46 (3), 172-176 (2001).
- Nylander, S., Kalies, I. Brefeldin A, but not monensin, completely blocks CD69 expression on mouse lymphocytes: efficacy of inhibitors of protein secretion in protocols for intracellular cytokine staining by flow cytometry. Journal of Immunology Methods. 224 (1-2), 69-76 (1999).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten