Method Article
Imaging di In Situ Interferon Gamma Production nella Spleen del topo dopo l'infezione da Listeria monocytogenes
In questo articolo
Riepilogo
Qui, descriviamo un semplice metodo di imaging confocale per visualizzare la localizzazione in situ delle cellule che secernono la gamma di interferone citochina negli organi linfoidi secondari murini. Questo protocollo può essere esteso per la visualizzazione di altre citochine in tessuti diversi.
Abstract
Le citochine sono piccole proteine secrete dalle cellule, che mediano le comunicazioni cellulari che sono cruciali per risposte immunitarie efficaci. Una caratteristica delle citochine è il loro pleiotropismo, in quanto sono prodotti da e possono influenzare una moltitudine di tipi di cellule. Come tale, è importante capire non solo quali cellule producono citochine, ma anche in quale ambiente lo fanno, al fine di definire terapie più specifiche. Qui, descriviamo un metodo per visualizzare la produzione di citochine in situ a seguito di infezione batterica. Questa tecnica si basa sull'imaging di cellule che producono citochine nel loro ambiente nativo mediante microscopia confocale. Per farlo, le sezioni di tessuto sono colorate per marcatori di più tipi di cellule insieme a una macchia di citochina. Chiave di questo metodo, la secrezione di citochine è bloccata direttamente in vivo prima di raccogliere il tessuto di interesse, consentendo il rilevamento della citochina che si accumulava all'interno delle cellule produttrici. I vantaggi di questo metodo sono molteplici. In primo luogo, viene conservato il microambiente in cui vengono prodotte le citochine, che potrebbe in ultima analisi informare sui segnali necessari per la produzione di citochine e sulle cellule colpite da tali citochine. Inoltre, questo metodo fornisce un'indicazione della posizione della produzione di citochine in vivo, in quanto non si basa sulla ristimolazione artificiale in vitro delle cellule produttrici. Tuttavia, non è possibile analizzare contemporaneamente la segnalazione a valle della citochina nelle celle che ricevono la citochina. Allo stesso modo, i segnali di citochina osservati corrispondono solo alla finestra temporale durante la quale è stata bloccata la secrezione della citochina. Mentre descriviamo la visualizzazione della gamma dell'interferone citochina (IFN) nella milza in seguito all'infezione da topo da parte dei batteri intracellulari Listeria monocytogenes, questo metodo potrebbe essere potenzialmente adattato alla visualizzazione di qualsiasi citochina in la maggior parte degli organi.
Introduzione
Orchestrare una risposta immunitaria efficiente contro un agente patogeno richiede una complessa integrazione dei segnali visualizzati da una varietà di cellule immunitarie che sono spesso disperse tra l'organismo. Per comunicare, queste cellule producono piccole proteine solubili con molteplici funzioni biologiche che fungono da immunomodulatori chiamati citochine. Le citochine controllano il reclutamento, l'attivazione e la proliferazione delle cellule e quindi sono note per essere attori chiave nella promozione delle risposte immunitarie1. Risposte immunitarie efficaci richiedono il rilascio delle citochine in un modello patiotemporale molto organizzato che collega cellule specifiche per indurre segnali specifici. Pertanto, è fondamentale studiare la produzione di citochine e la sua segnalazione in situ, tenendo conto del microambiente in cui vengono prodotte le citochine.
Listeria monocytogenes (L. monocytogenes) è un batterio intracellulare Gram-positivo utilizzato come modello primario per studiare le risposte immunitarie agli agenti patogeni intracellulari nei topi. Una citochina, IFN gamma (IFN) viene prodotta rapidamente, entro 24 h dopo l'infezione da L. monocytogenes. È necessario per lo sgombero degli agenti patogeni, in quanto i topi eliminati per IFN sono altamente sensibili all'infezione da L. monocytogenes 2. L'IFN è pleiotropico e prodotto da più cellule a seguito dell'infezione3. Mentre l'IFN, prodotto da cellule natural killer (NK), è necessario per l'attività antibatterica diretta4, è stato dimostrato che l'IFN, proveniente da altre fonti, ha dimostrato di avere altre funzioni. Infatti, noi e altri recentemente abbiamo scoperto che IFNs prodotto da cellule T CD8, ha una funzione specifica nella regolazione diretta della differenziazione delle cellule T5,6,7. In quanto tale, comprendere quali cellule producono IFN (e in quale microambiente) è fondamentale per sezionarne la funzione.
La tecnica più comune per studiare la produzione di citochine si basa sulla colorazione intracellulare delle citochine analizzate dalla citometria di flusso. Questo metodo consente il rilevamento simultaneo di più citochine combinate con marcatori di superficie cellulare all'interno di un singolo campione, fornendo uno strumento estremamente utile per studiare la produzione di citochine. Tuttavia, l'utilizzo della tecnica di cui sopra implica la perdita di qualsiasi informazione spaziale. Inoltre, il rilevamento delle citochine spesso si basa sulla ristimolazione in vitro per consentire il rilevamento delle citochine. Come tale, viene analizzata la capacità di una determinata cellula di produrre una citochina e non è necessariamente correlata alla secrezione di citochina effettiva in situ. Altri metodi utilizzano topi reporter per i quali l'espressione proteica fluorescente è correlata alla trascrizione della citochina e consente la visualizzazione a livello una singola cellula8. Anche se questo metodo può tenere traccia della trascrizione della citochina in situ, ci sono un numero limitato di topi citochine-reporter disponibili. Inoltre, la trascrizione, la traduzione e la secrezione a volte possono essere scollegate, e le proteine fluorescenti hanno un'emivita diversa rispetto alla citochina che riportano, rendendo questo metodo a volte non adeguato per la visualizzazione in situ cytokine.
Qui, descriviamo un metodo per visualizzare la produzione di citochine in situ mediante microscopia confocale a risoluzione a cella singola. Questa tecnica consente la visualizzazione della sorgente cellulare e della nicchia circostante all'interno del tessuto. Questo protocollo descrive in particolare la visualizzazione della produzione di IFN z nella milza di topi infetti da L. monocytogenes, concentrandosi qui sulla produzione IFN, da parte delle cellule NK e delle cellule T specifiche dell'antigene. Tuttavia, può essere esteso e adattato alla caratterizzazione di qualsiasi produzione di citochine nel contesto di altre situazioni in cui vengono prodotte citochine come infezioni, infiammazioni o malattie autoimmuni, a condizione che la citochina mirata possa essere conservata nelle cellule dall'inibitore del trasporto di proteine intracellulari.
Protocollo
Tutti gli esperimenti riguardanti i topi erano in accordo con il UK Scientific Procedures Act del 1986.
1. Trasferimento adottivo di cellule CD8 specifiche per l'antigenico- T nei topi
- Specifico di isolare l'ovalbumina (OVA) CD8- cellule T (OTI) che esprime proteine fluorescenti verdi (OTI-GFP) o proteine fluorescenti rosse (OTI-RFP) dalla sospensione dei linfonodi dei topi transgenici del recettore delle cellule T9,10 utilizzando un topo CD8- Kit di isolamento delle celle T come da istruzioni di fabbricazione. Preparare la sospensione cellulare fracassando i linfonodi utilizzando uno stantuffo di siringa, come descritto in precedenza11.
- Trasferire le cellule OTI-GFP o OTI-RFP (3 x 106 cellule) in C57BL/6 topi di tipo selvatico destinatari di iniezione endovenosa come descritto da Cahalan, et al.12. Utilizzare topi che sono in genere 6-12 settimana di età.
NOT: Questo passaggio è facoltativo e deve essere richiesto solo per tenere traccia delle cellule CD8- T specifiche dell'antigene.
2. Infezione da Listeria monocytogenes
- Espandere L. monocytogenes geneticamente modificato per esprimere OVA (LM-OVA)13 ad una fase esponenziale di crescita dell'infusione del cuore di brodo a 37 gradi centigradi in agitazione delicata fino a quando l'OD600 raggiunge 0,08–0,1, come descritto in precedenza in riferimento 14.
- Iniettare 100 L (volume massimo - 200 L) di 0,1–0,5 LD50 LM-OVA diluito in salina fosfati-buffered (PBS) per iniezione endovenosa utilizzando una siringa insulinica da 29 G, in capi di topi di tipo selvatico C57BL/6 che producono cellule OTI-GFP o OTI-RFP quando indicato.
NOT: Nelle nostre mani, 0,1 x LD50 LM-OVA corrisponde a 2 x 104 unità formanti colonia (CFU). L. monocytogenes geneticamente modificato per esprimere OVA viene utilizzato per attivare le cellule OTI CD8- T precedentemente trasferite, ma possono essere utilizzati altri ceppi di L. monocytogenes.
3. Trattamento con Brefeldin A (BFA) per bloccare la secrezione Cytokine
- Iniettare 250 g di BFA in 200 -L di PBS intraperitonealmente 6 h prima del sacrificio del topo utilizzando una siringa di insulina da 29 G.
NOT: La BFA liofilata viene prima sospesa nel solfuro dimetilo (DMSO) per preparare uno stock di concentrazione di 25 mg/mL. L'BFA viene quindi diluito in PBS a temperatura ambiente (RT) per evitare la cristallizzazione prima dell'iniezione. L'inibizione della secrezione di citochine induce l'accumulo di IFN nelle cellule. Questo è fondamentale per il rilevamento delle citochine.
4. Raccogliere la milza
- Eutanasia i topi utilizzando l'aumento della concentrazione di CO2 seguita da lussazione cervicale.
NOT: Seguire le linee guida dell'istituzione locale per l'eutanasia umana dei topi. - Pulire l'addome con il 70% di etanolo, fare un'incisione con le forbici per fare un 1-2 cm tagliato attraverso la pelle sul lato sinistro del mouse, dove si trova la milza. Fare con attenzione un'incisione nel peritoneo per esporre la milza e tirarla fuori con una pinzetta. Raccogliere la milza, facendo attenzione a non spremere con pinze o tagliarla per evitare di interrompere l'architettura della milza.
5. Fissazione dello spleen con paraformaldeide (PFA)
- Preparare la soluzione fissativa mescolando 3,75 mL di PBS e 3,75 mL di 0,2 M L-lysine. Aggiungere 21 mg di m-periodo di sodio e mescolare bene. Quindi aggiungere 2,5 mL di 4% PFA e 20 luna di 12 N NaOH.
NOT: Utilizzare la soluzione fissativa nello stesso giorno e scartare l'eccesso. Non conservarlo. Questa fase di fissazione è importante se il campione contiene proteine fluorescenti come GFP. Non utilizzare PFA contenente tracce di metanolo, in quanto denatura le proteine fluorescenti.
ATTENZIONE: La PFA è tossica e deve essere maneggiata con cautela. - Sommerse la milza nel fissativo e fissa per un minimo di 4 h, in genere 16-20 h a 4oC in agitazione delicata.
- Eliminare la soluzione fissativa e aggiungere 5 mL di PBS per 5 min a RT in leggera agitazione.
- Sostituire il PBS con 5 mL di incubazione PBS fresca per 1 h a 4 gradi centigradi in agitazione delicata.
- Sostituire il PBS con 5 mL del 30% di saccarosio, incubare per 12-24 h.
NOT: Questo metodo aiuta a mantenere la morfologia dei tessuti. Dopo l'incubazione con soluzione di saccarosio, l'organo dovrebbe affondare sul fondo del pozzo.
6. Congelamento e sezionamento
- Mettere il ghiaccio secco in un grande recipiente e mettere all'interno un recipiente più piccolo contenente circa 50 mL di metanolo puro e alcuni pezzi di ghiaccio secco.
- Asciugare delicatamente la milza su una salvietta senza laminetta.
- Posizionare la milza all'interno di uno stampo di base contenente una goccia di composto di temperatura di taglio ottimale (OCT) nella parte inferiore. Fare attenzione a non produrre bolle. Aggiungere OCT sulla milza.
- Con le pinze, depositare lo stampo di base sulla superficie del metanolo, assicurandosi che non tocchi lo strumento OCT. Congelare il tessuto il più rapidamente possibile per ridurre al minimo gli artefatti.
- Quando è congelato, procedere con il sezionamento.
NOT: La milza congelata può essere conservata a -80 gradi centigradi per diversi mesi. -
Sezionare il tessuto usando un criomicrotoma.
- Impostare la temperatura della camera sul criostato dovrebbe essere a -21 gradi centigradi. Tagliare le sezioni dello spessore desiderato (di solito intorno ai 10 m). Questo protocollo funziona con spessori fino a 30 m.
- Raccogliere le sezioni sui vetrini del microscopio in vetro (vedere la tabella deimateriali) e ispezionare visivamente.
NOT: Le sezioni possono essere conservate a -80 gradi centigradi per diversi mesi.
7. Colorazione immunofluorescente
- Lasciare che la sezione venga a RT.
- Disegnare un cerchio con un bloccante liquido (ad esempio, penna PAP) intorno alla sezione del tessuto. Disegnare all'esterno dello OCT o non si attaccherà.
- Una volta essiccato, reidratare il campione posizionando PBS sulla sezione del tessuto per 5 min.
NOT: Il volume inserito nella sezione dipende dalle dimensioni della sezione. In genere si usano 100-300 l. Non lasciare asciugare le sezioni una volta reidratate. - Risciacquare con PBS almeno due volte per assicurarsi che la sezione sia ben aderito al vetrino.
-
Aggiungere la soluzione di blocco alla sezione per diminuire il legame non specifico degli anticorpi.
- Preparare la soluzione di blocco come segue: PBS con 0,1% Triton X100, 2% siero di vitello fetale (FCS), 2,5 g/mL del recettore Fc (anti-mouse CD16/32). Quindi aggiungere il 2-5% del siero normale della specie di ogni anticorpo secondario del pannello di colorazione.
NOT: Se gli anticorpi sono direttamente coniugati/biotinylati, aggiungere il 5% del siero normale delle specie di ogni anticorpo primario. Se un anticorpo primario e uno degli anticorpi secondari provengono dalla stessa specie (ad esempio, anticorpi primari allevati nell'anti-ratto del coniglio e del coniglio secondario), non utilizzare la normale specie di siero in quanto aumenterà il segnale di fondo. - Rimuovere delicatamente il PBS dalla sezione per aspirazione e aggiungere 100 l della soluzione di blocco per sezione campione. Incubare in una camera bagnata coperta per un minimo di 1 h a RT.
- Preparare la soluzione di blocco come segue: PBS con 0,1% Triton X100, 2% siero di vitello fetale (FCS), 2,5 g/mL del recettore Fc (anti-mouse CD16/32). Quindi aggiungere il 2-5% del siero normale della specie di ogni anticorpo secondario del pannello di colorazione.
-
Macchie con anticorpi primari.
- Diluire gli anticorpi primari alla concentrazione ottimale nella soluzione di blocco. La concentrazione generale di anticorpi del punto di partenza è di 5 g/mL, ma dovrebbe essere ottimizzata per ogni anticorpo e tessuto.
NOT: Se gli anticorpi sono coniugati direttamente, centrificare la miscela di anticorpi a 17,135 x g (13.500 giri/) per 15 min a 4 gradi centigradi prima di usarlo. I fluorofori possono precipitare. Questo passo avrà spinto i precipitati e quindi prevenire la deposizione non specifica degli anticorpi precipitati sul vetrino. - Sostituire la soluzione di blocco con la combinazione di anticorpi primaria per ogni campione.
- Incubare per 4 ore a RT o durante la notte (OVN) a 4 gradi centigradi in una camera bagnata coperta.
- Diluire gli anticorpi primari alla concentrazione ottimale nella soluzione di blocco. La concentrazione generale di anticorpi del punto di partenza è di 5 g/mL, ma dovrebbe essere ottimizzata per ogni anticorpo e tessuto.
-
Eseguire il lavaggio.
- Preparare Wash Buffer aggiungendo 2% FCS a PBS.
- Lavare 4 volte con il tampone di lavaggio: uno veloce (senza incubazione), uno per 10 min e due per 5 min. Quindi eseguire un lavaggio finale con PBS per 5 min.
-
Colorazione con anticorpi secondari.
- Diluire gli anticorpi secondari di interesse alla concentrazione ottimale nella soluzione di blocco. Centrifugare il mix, come descritto per gli anticorpi primari.
- Rimuovere la soluzione di lavaggio finale. Aggiungere la miscela di anticorpi secondari sulla parte superiore della sezione e incubare per 1-4 h a RT in una camera bagnata coperta.
- Lavare 4 volte con il tampone di lavaggio: uno veloce (senza incubazione), uno per 10 min e due per 5 min. Quindi eseguire un lavaggio finale con PBS per 5 min.
- Rimuovere la soluzione di lavaggio finale. Lasciare evaporare il PBS ma non asciugare eccessivamente la sezione. Posizionare una goccia del supporto di montaggio sopra il campione e posizionare con attenzione il vetro di copertura su di esso. Il supporto di montaggio deve recuperare l'intera sezione. Lasciate che polimerasi OVN a RT protetto dalla luce.
NOT: Disegnare un cerchio intorno alla sezione sul lato opposto della diapositiva prima di applicare il supporto di montaggio. Una volta applicato il supporto di montaggio, il tessuto potrebbe diventare difficile da vedere. - Conservare le diapositive al buio a 4 gradi centigradi fino a quando non viene eseguito il tempo necessario per l'immagine.
8. Imaging e analisi
- Eseguire l'imaging della colorazione con un microscopio confocale.
NOT: In questo protocollo è stato utilizzato un microscopio a scansione laser invertito (vedere la Tabella dei Materiali), insieme agli obiettivi 10x/NA 0.40 o 60x/NA 1.4 (per l'analisi della localizzazione subcellulare citochina). Lunghezze d'onda di eccitazione ed emissione sono visualizzate per ogni proteina fluoroforo e fluorescente nella Tabella dei Materiali. - Eseguire l'analisi e la quantificazione come richiesto utilizzando il software di elaborazione delle immagini (ad esempio, Imaris o Fiji).
Risultati
L'IFN- prodotto entro le prime 24 h dopo l'infezione da Listeria monocytogenes è fondamentale per controllare la diffusione di questo agente patogeno. Utilizzando questo protocollo, possiamo visualizzare non solo quali cellule producono IFN, ma anche se si trovano in un microambiente specifico. Per aiutarci a delineare l'architettura della milza, abbiamo etichettato le cellule note per avere una posizione particolare all'interno della milza. Il marcatore F4/80 etichetta tutti i macrofagi e mette in evidenza la polpa rossa. Il marcatore B220 etichetta le cellule B ed evidenzia i follicoli cellulari B che circondano la zona delle cellule T. Il marcatore CD169 etichetta i macrofagi di zona marginale, che circondano la polpa bianca (Figura1). La maggior parte delle cellule OTI, che esprimano o meno, sono presenti nella polpa bianca e come tali, tutte le immagini sono quelle della polpa bianca, a meno che non siano indicate.
Un passo critico in questo protocollo è l'uso di BFA per inibire la secrezione di citochina. In effetti, l'individuazione delle cellule IFN da parte delle cellule NK è stata notevolmente compromessa quando i topi non venivano trattati con BFA (Figura2). Utilizzando il nostro protocollo, potremmo scoprire che almeno due tipi di cellule producono IFN , 24 h dopo l'infezione - cellule NK e antigene specifico CD8- cellule T (Figura3) - in modo simile a quanto è stato trovato in precedenza dalla citometria di flusso3.
L'imaging in situ delle cellule che producono IFN z ha rivelato che la produzione di IFN non è diffusa in tutta la milza, ma concentrata in aree discrete (Figura4). Infatti, abbiamo scoperto che le cellule T sono state attivate in tutta la milza (evidenziata dal clustering di cellule T), e questo non era necessariamente correlato con la produzione di IFN. Una spiegazione probabile è che la produzione di IFN è limitata all'ubicazione delle cellule infette15,16e l'attivazione delle cellule T, rappresentate dal clustering, possono essere supportate sia da infette (IFN - positivo) che non infette (IFN negativo) cellule che presentano l'antigene. Altre macchie saranno necessarie per individuare l'esatta posizione e ottenere un'indicazione del meccanismo che limita la produzione di IFN a quest'area e il suo rapporto con il trasferimento dell'antigene. È interessante notare che abbiamo scoperto che le cellule T attivate, raggruppate e specifiche dell'antigene si trovano in tutta la polpa bianca della milza, ma producono IFN, solo nelle regioni in cui le cellule NK sono coesistenti con loro (Figura 5). Come tale, la presenza di cellule NK delinea un microambiente specifico nella polpa bianca, in cui le cellule T raggruppate producono IFN, in contrasto con le cellule T raggruppate nell'altra parte della polpa bianca. Ciò suggerisce che l'attivazione delle cellule T non è sufficiente per dettare la produzione di IFN , a questo punto.
Un'altra caratteristica interessante evidenziata dal nostro protocollo è la diversa localizzazione subcellulare di IFN , in NK rispetto a CD8, cellule T5. Come mostrato nella Figura 6, mentre la localizzazione dell'IFN z nelle cellule NK è diffusa nel citosol, le cellule T CD8- T spesso reclutano IFN , verso un'altra cellula T.

Figura 1: Marcatori che evidenziano l'architettura della milza. I topi sono stati infettati con 2 x 104 CFU LM-OVA e eutanasia 24 h post infezione. Spleen è stato espiantato ed elaborato come descritto nel protocollo. (A) Le sezioni sono state macchiate per le cellule NK (anti-NCR1 seguite da IgG-FITC anti-capra; verde), cellule OTI-RFP (rosse) e macrofagi (anti-F4/80-APC; magenta). RP - Polpa Rossa; WP - Polpa Bianca. Barra di scala - 200 m.(B) Le sezioni sono state macchiate per le cellule B (anti-B220-Pacific Blue; Blu), cellule OTI-GFP (segnale GFP mostrato in rosso) e macrofagi di zona marginale (anti-CD169-Alexa647; magenta). RP - Polpa Rossa; BF - follicolo a cellule B; T - zona di cella T. Barra della scala: 50 m. Si tratta di un'immagine rappresentativa di 3 esperimenti indipendenti (N ) . Fare clic qui per visualizzare una versione più grande di questa figura.
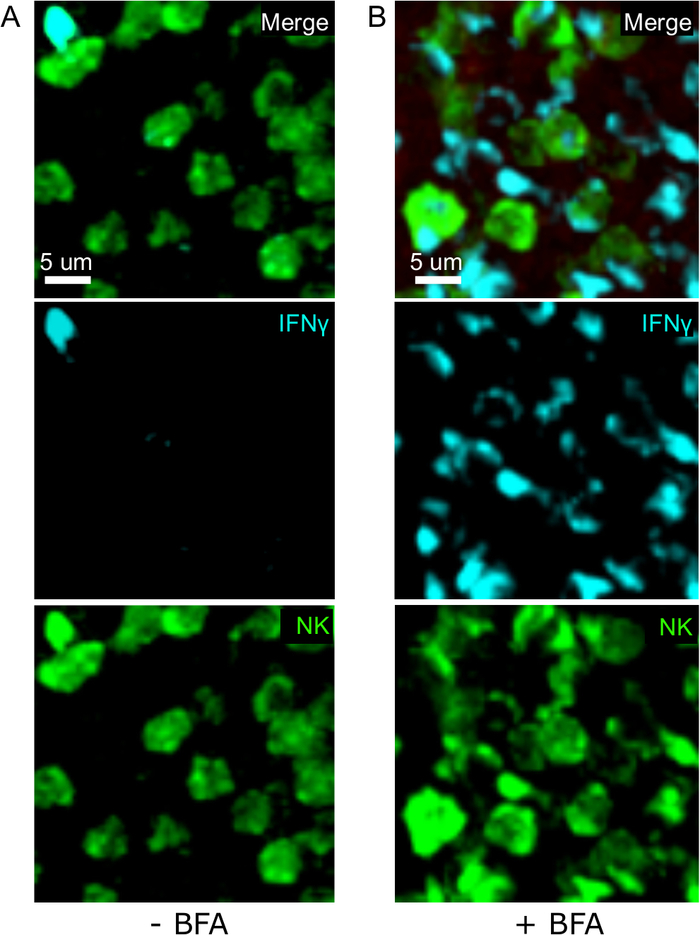
Figura 2: Il trattamento BFA consente l'individuazione di IFN intracellulare in situ. La produzione è limitata a specifiche aree della spleria I topi sono stati infettati con 2 x 104 CFU LM-OVA e trattati con BFA (A) o lasciati non trattati (B) dopo 18 h. I topi sono stati eutanasia 24 h dopo l'infezione. Spleen è stato espiantato ed elaborato come descritto nel protocollo. Le sezioni sono state macchiate per le cellule NK (anti-NCR1 seguite da Anti-goat IgG-FITC; verde), le celle OTI-RFP (rosso) e IFN (anti-IFN -BV421; ciano). Barra della scala: 5 m. Si tratta di un'immagine rappresentativa delle aree ricche di cellule NK provenienti da 3 esperimenti indipendenti (N - 3). Fare clic qui per visualizzare una versione più grande di questa figura.
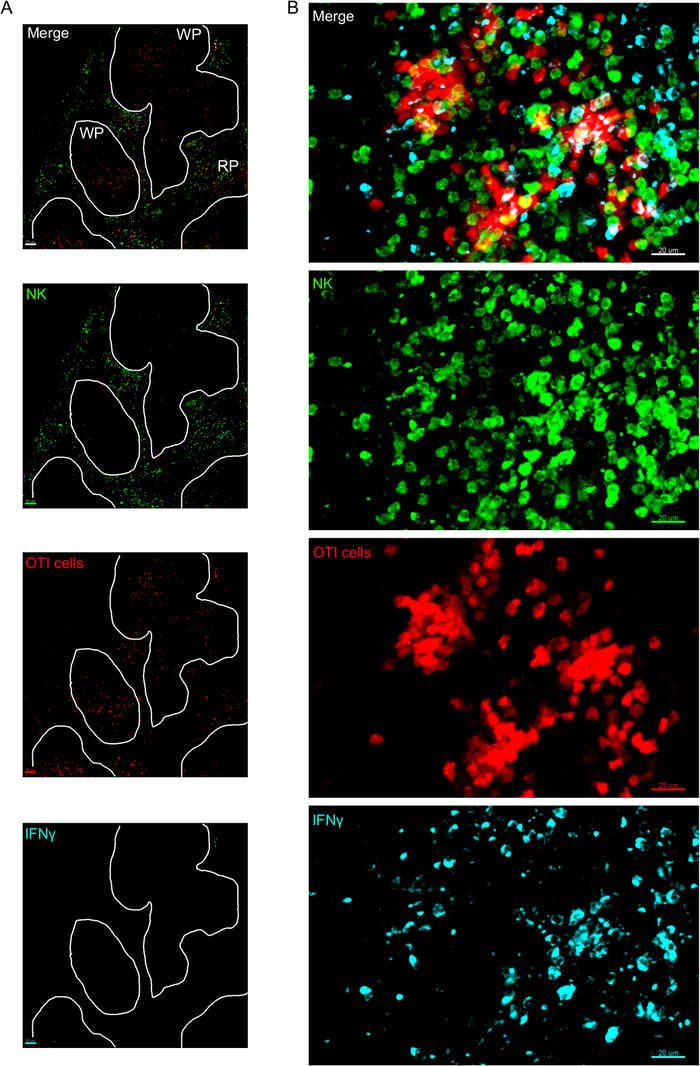
Figura 3: Cellule produttrici di IFN nella milza. I topi sono stati infettati con 2 x 104 CFU LM-OVA quando indicato e trattato con BFA dopo 18 h. I topi sono stati eutanasia 24 h dopo l'infezione. Spleen è stato espiantato ed elaborato come descritto nel protocollo. Le sezioni sono state macchiate per le cellule NK (anti-NCR1 seguite da Anti-goat IgG-FITC; verde), le celle OTI-RFP (rosso) e IFN (anti-IFN -BV421; ciano). (A) Immagine rappresentativa di una milza proveniente da un topo ingenuo non infetto per dimostrare l'assenza di colorazione non specifica dell'IFN. Linee bianche delineano la polpa bianca. WP - Polpa bianca; RP - Polpa rossa. (B) Immagine rappresentativa della polpa bianca dalla milza di un topo infettato da LM-OVA, che mostra l'invasione delle cellule NK nella polpa bianca e la produzione di IFN - da cellule NK, cellule OTI e cellule non etichettate. Le immagini sono rappresentative di 4 esperimenti indipendenti (N ) . Barre della scala: 70 m (A); e 20 m (B). Fare clic qui per visualizzare una versione più grande di questa figura.
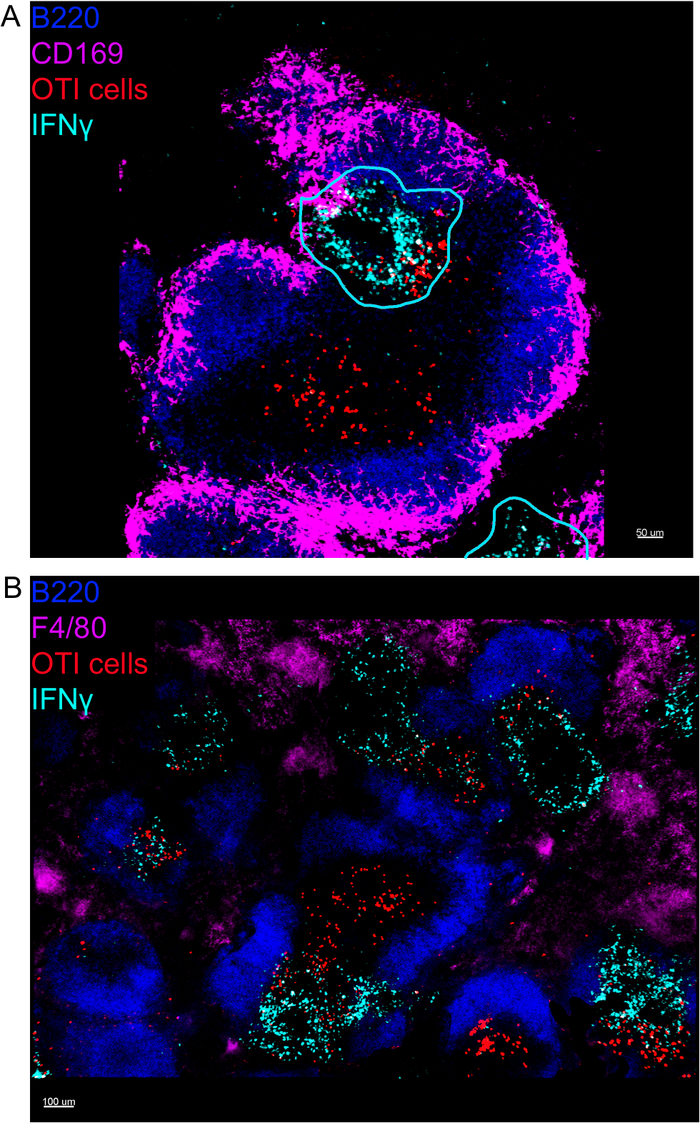
Figura 4: La produzione di IFN è limitata ad aree specifiche della milza in seguito all'infezione da LM-OVA. I topi sono stati infettati da 2 x 104 CFU LM-OVA e trattati con BFA dopo 18 h. I topi sono stati eutanasia 24 h post infezione. Spleen è stato espiantato ed elaborato come descritto nel protocollo. Tutte le sezioni sono state macchiate per le cellule B (B220-Pacific Blue Ab, Blue) e IFNs (anti-IFN-biotina seguita da streptavidin-PE; ciano). Celle OTI-GFP (segnale GFP mostrato in rosso). Le linee ciano corrispondono ad aree di alta produzione di IFN. Si tratta di immagini rappresentative di 4 esperimenti indipendenti (N . 4). (A) Le sezioni sono state macchiate per i macrofagi delle zone marginali (anti-CD169-Alexa 647, magenta). La barra di scala èstata macchiata per tutte le macrofagi (F4/80). Barra di scala: 100 m. Fare clic qui per visualizzare una versione più grande di questa figura.
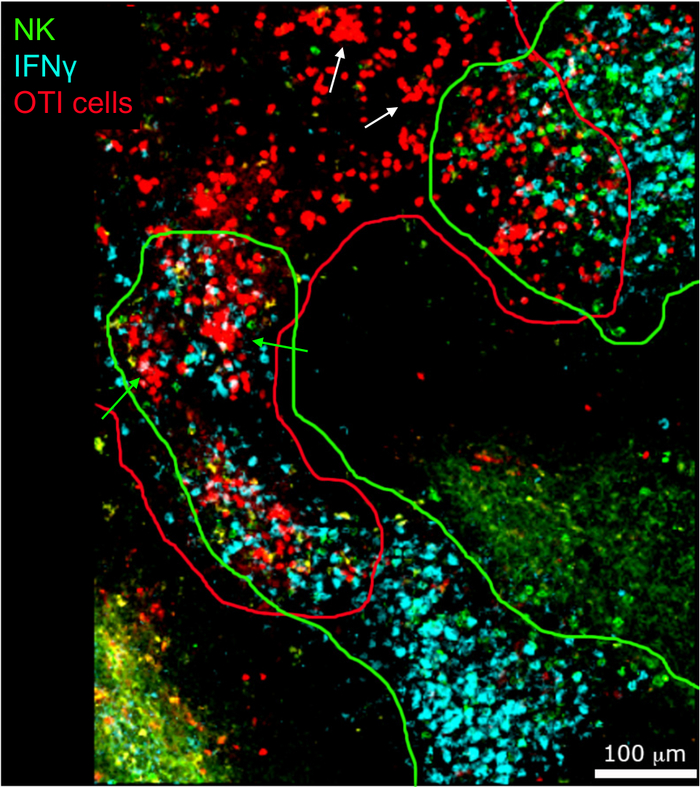
Figura 5: La produzione di IFN e cellule OTI attivate avviene in un microambiente specifico. I topi sono stati infettati da 2 x 104 CFU LM-OVA e trattati con BFA dopo 18 h. I topi sono stati eutanasia 24 h post infezione. Le spleens sono state espiantate ed elaborate come descritto nel protocollo. Le sezioni sono state macchiate per le cellule NK (anti-NCR1 seguite da Anti-goat IgG-FITC; verde), le celle OTI-RFP (rosso) e IFN (anti-IFN -BV421; ciano). Le linee verdi e rosse evidenziano rispettivamente le zone delle celle NK e OTI. La freccia bianca indica esempi di cluster di celle T che non producono IFN. Frecce verdi esempi di gruppi di cellule T che producono IFN. Barra della scala: 100 m. Si tratta di un'immagine rappresentativa di quattro esperimenti indipendenti (N ) . Fare clic qui per visualizzare una versione più grande di questa figura.
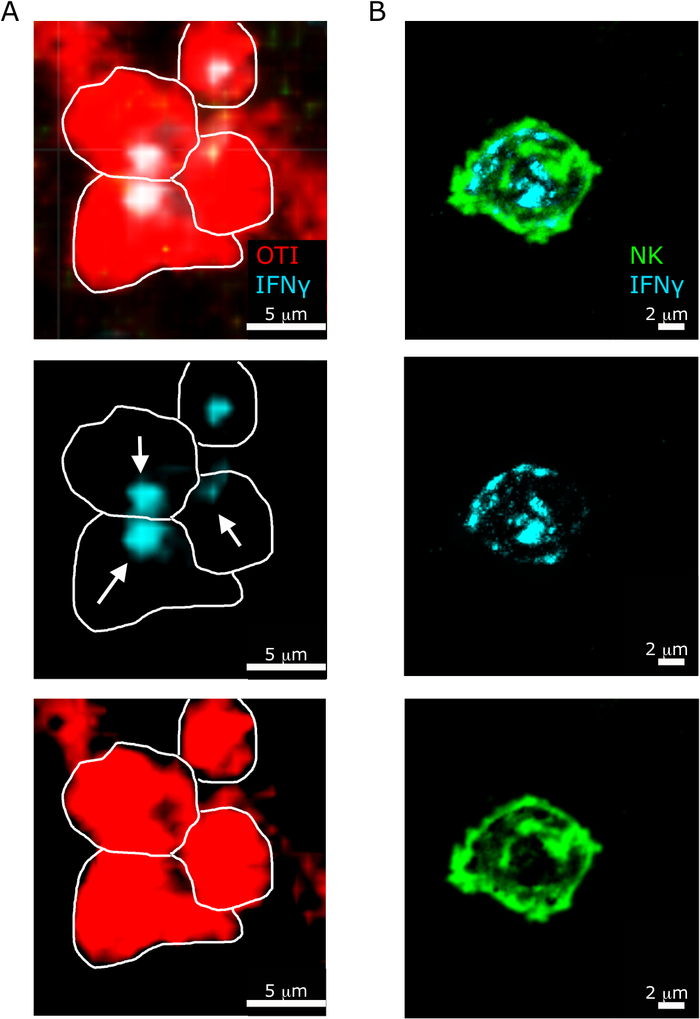
Figura 6: Localizzazione subcellulare di IFN, nelle cellule NK e T. I topi sono stati infettati da 2 x 104 CFU LM-OVA e trattati con BFA dopo 18 h. I topi sono stati eutanasia 24 h post infezione. Spleen è stato espiantato ed elaborato come descritto nel protocollo. Tutte le sezioni sono state macchiate per ifNs (anti-IFN - BV421; ciano). Le linee bianche delineano i bordi delle celle e le frecce bianche mostrano la direzionalità della secrezione. Si tratta di un'immagine rappresentativa di due esperimenti indipendenti (N . 5). (A)- Le celle OTI-RFP sono visualizzate in rosso. Barra di scala - 5 m. (B) Le sezioni sono state macchiate per le cellule NK (anti-NCR1 seguite da IgG-FITC anti-capra; verde. Barra di scala : 2 m. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
In questo manoscritto, presentiamo un metodo per visualizzare la produzione di IFN nella milza dopo l'infezione da L. monocytogenes nei topi. Questo protocollo è semplice e può essere adattato ad altri tessuti e trigger di citochina, ma i seguenti aspetti devono essere considerati. Le cellule spesso secernono rapidamente le citochine che producono, e le citochine vengono rapidamente captate dalle cellule vicine. È così difficile rilevare le citochine in situ. Un metodo comune per re-avviare rapidamente la produzione di citochine è quello di ri-stimolare le cellule ex vivo seguite dal rilevamento di citochine nei media da un saggio immunosorbente collegato agli enzimi. In questo contesto, tutte le informazioni sulla localizzazione spaziale delle cellule che producono citochine vengono perse. Inoltre, la produzione di citochine dopo la ri-stimolazione non riflette necessariamente se le citochine siano effettivamente prodotte e secrete in vivo, ma piuttosto indica la capacità di una determinata popolazione cellulare di produrre citochine. Pertanto, entrambi i metodi forniranno informazioni diverse e si dovrebbe considerare quali informazioni sono più preziose per il loro esperimento.
Al fine di rilevare le citochine intracellulari, il nostro metodo utilizza un inibitore del trasporto di proteine intracellulare per intrappolare le citochine all'interno delle cellule e aumentare il rilevamento del segnale. Tuttavia, è importante notare che questi inibitori influenzano il normale trasporto di proteine dal reticolo endoteliale (RE) all'apparato di Golgi e alla vescicola secretoria che ne altera il rilascio, che potrebbe causare tossicità. Di conseguenza, BFA, o altro inibitore, dovrebbe essere utilizzato per un breve periodo di tempo, in genere non più di un paio d'ore. Quindi, è importante trovare il giusto equilibrio tra la dose inibitore e il tempo di trattamento al fine di ottimizzare il livello di citochine intrappolate all'interno della cellula senza causare gravi effetti citotossici. Queste variabili possono differire tra le citochine e la via di somministrazione per il BFA. Nel nostro modello di infezione, il BFA è stato amministrato intraperitoneally al fine di fornire una rapida dispersione sistemica, ma può anche essere consegnato per via endovenosa.
Gli inibitori del trasporto di proteine intracellulari più comunemente utilizzati sono BFA, usato qui, e monensina (MN). Questi inibitori sono spesso usati indistintamente per accumulare e studiare la produzione di citochine, ma hanno lievi differenze nei loro meccanismi di azione. MN inibisce il trasporto di proteine all'interno dell'apparato Golgi accumulando così proteine nel Golgi17, mentre BFA impedisce il reclutamento di proteine coatomer complex-I, inibendo il movimento retrogrado delle proteine al reticolo endosmico (ER) promuovendo così l'accumulo di citochine nel ER18. Come tale, la scelta del miglior inibitore del trasporto proteico intracellulare dipenderà da diversi fattori, come la citochina da rilevare. Ad esempio, è stato dimostrato nella colorazione intracellulare indotta da lipopolisaccaride dei monociti che la BFA è più efficiente per misurare le citochine IL-1, IL-6 e TNF rispetto a MN19.
Questo protocollo prevede la visualizzazione della citochina in situ mediante microscopia confocale e quindi ci sono solo un numero limitato di marcatori che possono essere utilizzati per studiare le cellule che producono citochine e il loro microambiente. È inoltre necessario considerare che gli inibitori del trasporto proteico come la BFA o il MN disturbano la normale espressione di diverse proteine e quindi il loro utilizzo quando studiano l'espressione simultanea di alcuni marcatori di superficie delle cellule di attivazione deve essere affrontato Attentamente. Ad esempio, BFA ma non MN blocca l'espressione di CD69 nei linfociti murini20. Nonostante questa limitazione, l'imaging confocale consente la localizzazione subcellulare delle citochine, così come la direzione della secrezione di citochina all'interno della cellula. I dati generati utilizzando questo protocollo suggeriscono che le cellule NK tendono a secernere IFN-y in un modello diffuso, mentre le cellule CD8- T sembrano dirigere la secrezione IFN -ion verso altre cellule CD8- T che sono in interazione diretta con loro5.
Per concludere, questo protocollo è adatto per visualizzare una varietà di citochine in situ e identificare le cellule produttrici e il loro microambiente seguendo molti fattori scatenanti come l'infezione o l'autoimmunità. Le informazioni ottenute sono strumentali per comprendere l'importanza dell'orchestrazione spaziale in vivo di diversi tipi di cellule e della citochina che producono, necessaria per una risposta immunitaria efficiente.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo il personale della Kennedy Institute Imaging Facility per l'assistenza tecnica con l'imaging. Questo lavoro è stato sostenuto dalle sovvenzioni del Kennedy Trust (a.G.) e del Biotechnology and Biological Sciences Research Council (BB/R015651/1 a A.G.).
Materiali
| Name | Company | Catalog Number | Comments |
| Brefeldin A | Cambridge bioscience | CAY11861 | |
| Paraformaldehyde | Agar scientific | R1018 | |
| L-Lysin dihydrochloride | Sigma lifescience | L5751 | |
| Sodium meta-periodate | Thermo Scientific | 20504 | |
| D(+)-saccharose | VWR Chemicals | 27480.294 | |
| Precision wipes paper Kimtech science | Kimberly-Clark Professional | 75512 | |
| O.C.T. compound, mounting medium for cryotomy | VWR Chemicals | 361603E | |
| Fc block, purified anti-mouse CD16/32, clone 93 | Biolegend | 101302 | Antibody clone and Concentration used: 2.5 mg/ml |
| Microscope slides - Superfrost Plus | VWR Chemicals | 631-0108 | |
| anti-CD169 - AF647 | Biolegend | 142407 | Antibody clone and Concentration used: clone 3D6.112 1.6 mg/ml Excitation wavelength: 650 Emission wavelength: 65 |
| anti-F4/80 - APC | Biolegend | 123115 | Antibody clone and Concentration used: clone BM8 2.5 mg/ml Excitation wavelength: 650 Emission wavelength: 660 |
| anti-B220 - PB | Biolegend | 103230 | Antibody clone and Concentration used: clone RA3-6B2 1.6 mg/mL Excitation wavelength: 410 Emission wavelength: 455 |
| anti-IFNg - biotin | Biolegend | 505804 | Antibody clone and Concentration used: clone XMG1.2 5 mg/mL |
| anti-IFNg - BV421 | Biolegend | 505829 | Antibody clone and Concentration used: clone XMG1.2 5 mg/mL Excitation wavelength: 405 Emission wavelength: 436 |
| anti-Nkp46/NCRI | R&D Systems | AF2225 | Antibody clone and Concentration used: goat 2.5 mg/mL |
| anti-goat IgG-FITC | Novusbio | NPp 1-74814 | Antibody clone and Concentration used: 1 mg/mL Excitation wavelength: 490 Emission wavelength: 525 |
| Streptavidin - PE | Biolegend | 405203 | Antibody clone and Concentration used: 2.5 mg/mL Excitation wavelength: 565 Emission wavelength: 578 |
| Streptavidin - FITC | Biolegend | 405201 | Antibody clone and Concentration used: 2.5 mg/mlL Excitation wavelength: 490 Emission wavelength: 525 |
| Fluoromount G | SouthernBiotech | 0100-01 | |
| Cover glasses 22 mm x 40 mm | Menzel-Glazer | 12352128 | |
| Liquid blocker super PAP PEN mini | Axxora | CAC-DAI-PAP-S-M | |
| Imaris - Microscopy Image Analysis Software | Bitplane | ||
| Confocal microscope - Olympus FV1200 Laser scanning microscope | Olympus | ||
| Cryostat - CM 1900 UV | Leica | ||
| Base mould disposable | Fisher Scientific UK Ltd | 11670990 | |
| PBS 1x | Life Technologies Ltd | 20012068 | |
| BHI Broth | VWR Brand | 303415ZA | |
| GFP | Excitation wavelength: 484 Emission wavelength: 507 | ||
| RFP | Excitation wavelength: 558 Emission wavelength: 583 | ||
| Insulin syringe, with needle, 29 G | VWR International | BDAM324824 | |
| C57BL/6 wild type mice | Charles River |
Riferimenti
- Iwasaki, A., Medzhitov, R. Control of adaptive immunity by the innate immune system. Nature Immunology. 16 (4), 343-353 (2015).
- Harty, J. T., Bevan, M. J. Specific immunity to Listeria monocytogenes in the absence of IFN gamma. Immunity. 3 (1), 109-117 (1995).
- Kubota, K., Kadoya, Y. Innate IFN-gamma-producing cells in the spleen of mice early after Listeria monocytogenes infection: importance of microenvironment of the cells involved in the production of innate IFN-gamma. Frontiers in Immunology. 2 (26), (2011).
- Dunn, P. L., North, R. J. Early gamma interferon production by natural killer cells is important in defense against murine listeriosis. Infection and Immunity. 59 (9), 2892-2900 (1991).
- Krummel, M. F., et al. Paracrine costimulation of IFN-gamma signaling by integrins modulates CD8+ T cell differentiation. Proceedings of the National Academy of Sciences of the United States of America. 115 (45), 11585-11590 (2018).
- Curtsinger, J. M., Agarwal, P., Lins, D. C., Mescher, M. F. Autocrine IFN-gamma promotes naive CD8+ T cell differentiation and synergizes with IFN-alpha to stimulate strong function. Journal of Immunology. 189 (2), 659-668 (2012).
- Hosking, M. P., Flynn, C. T., Whitton, J. L. Antigen-specific naive CD8++ T cells produce a single pulse of IFN-gamma in vivo within hours of infection, but without antiviral effect. Journal of Immunology. 193 (4), 1873-1885 (2014).
- Croxford, A. L., Buch, T. Cytokine reporter mice in immunological research: perspectives and lessons learned. Immunology. 132 (1), 1-8 (2011).
- Gerard, A., et al. Secondary T cell-T cell synaptic interactions drive the differentiation of protective CD8++ T cells. Nature Immunology. 14 (4), 356-363 (2013).
- Engelhardt, J. J., et al. Marginating dendritic cells of the tumor microenvironment cross-present tumor antigens and stably engage tumor-specific T cells. Cancer Cell. 21 (3), 402-417 (2012).
- Matheu, M. P., Cahalan, M. D. Isolation of CD4+ T cells from mouse lymph nodes using Miltenyi MACS purification. Journal of Visualized Experiments. (9), 409 (2007).
- Matheu, M. P., Parker, I., Cahalan, M. D. Dissection and 2-photon imaging of peripheral lymph nodes in mice. Journal of Visualized Experiments. (7), 265 (2007).
- Pope, C., et al. Organ-specific regulation of the CD8+ T cell response to Listeria monocytogenes infection. Journal of Immunology. 166 (5), 3402-3409 (2001).
- Jones, G. S., D'Orazio, S. E. Listeria monocytogenes: cultivation and laboratory maintenance. Current Protocols in Microbiology. 31, 1-7 (2013).
- Kang, S. J., Liang, H. E., Reizis, B., Locksley, R. M. Regulation of hierarchical clustering and activation of innate immune cells by dendritic cells. Immunity. 29 (5), 819-833 (2008).
- Chang, S. R., et al. Characterization of early gamma interferon (IFN-gamma) expression during murine listeriosis: identification of NK1.1+ CD11c+ cells as the primary IFN-gamma-expressing cells. Infection and Immunity. 75 (3), 1167-1176 (2007).
- Mollenhauer, H. H., Morre, D. J., Rowe, L. D. Alteration of intracellular traffic by monensin; mechanism, specificity and relationship to toxicity. Biochimica et Biophysica Acta. 1031 (2), 225-246 (1990).
- Helms, J. B., Rothman, J. E. Inhibition by brefeldin A of a Golgi membrane enzyme that catalyses exchange of guanine nucleotide bound to ARF. Nature. 360 (6402), 352-354 (1992).
- Schuerwegh, A. J., Stevens, W. J., Bridts, C. H., De Clerck, L. S. Evaluation of monensin and brefeldin A for flow cytometric determination of interleukin-1 beta, interleukin-6, and tumor necrosis factor-alpha in monocytes. Cytometry. 46 (3), 172-176 (2001).
- Nylander, S., Kalies, I. Brefeldin A, but not monensin, completely blocks CD69 expression on mouse lymphocytes: efficacy of inhibitors of protein secretion in protocols for intracellular cytokine staining by flow cytometry. Journal of Immunology Methods. 224 (1-2), 69-76 (1999).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon