Method Article
Proteinextrakt-Präparation und Co-Immunpräzipitation von Caenorhabditis elegans
In diesem Artikel
Zusammenfassung
Diese Methode beschreibt ein Protokoll zur Herstellung von Proteinextrakten mit hohem Durchsatz aus Caenorhabditis elegans Proben und anschließender Co-Immunpräzipation.
Zusammenfassung
Co-Immunpräzipitationsmethoden werden häufig verwendet, um Protein-Protein-Wechselwirkungen zu untersuchen. Die Bestätigung von hypothetischen Protein-Protein-Wechselwirkungen oder die Identifizierung neuer Protein-Wechselwirkungen können unschätzbare Informationen über die Funktion eines von Interesse sindden Proteins liefern. Einige der traditionellen Methoden zur Extraktzubereitung erfordern häufig arbeitsintensive und zeitaufwändige Techniken. Hier wird ein modifiziertes Extraktvorbereitungsprotokoll mit einem Perlenmühlenhomogenisator und Metallperlen als schnelle Alternative zu herkömmlichen Proteinaufbereitungsmethoden beschrieben. Diese Extraktpräparationsmethode ist mit nachgeschalteten Co-Immunpräzipitationsstudien kompatibel. Als Beispiel wurde die Methode verwendet, um C. elegans microRNA Argonaute ALG-1 und zwei bekannte ALG-1 Interaktoren erfolgreich zu koimmunisieren: AIN-1 und HRPK-1. Dieses Protokoll enthält Beschreibungen der Probenentnahme, der Extraktzubereitung, der Extraktklärung und der Proteinimmunpräzipitation. Das beschriebene Protokoll kann angepasst werden, um Wechselwirkungen zwischen zwei oder mehr endogenen, endogen markierten oder überexprimierten C. elegans Proteinen in einer Vielzahl genetischer Hintergründe zu testen.
Einleitung
Die Identifizierung der makromolekularen Wechselwirkungen eines Proteins von Interesse kann der Schlüssel sein, um mehr über seine Funktion zu lernen. Immunpräzipitations- und Co-Immunpräzipitationsexperimente können verwendet werden, um das gesamte Interom eines Proteins durch groß angelegte proteomische Ansätze1 zu identifizieren oder speziell die Fähigkeit eines Proteins zu testen, mit einem hypothetischen Interaktor zu koprezipieren. In C. eleganswurden beide Methoden erfolgreich eingesetzt, um mehr über die Aktivität einer Vielzahl von Proteinen zu erfahren, einschließlich derjenigen, die eng mit microRNAs zusammenarbeiten, um die Genexpression2,3,4zu regulieren. Co-Immunpräzipitationsexperimente haben den Vorteil, die Protein-Protein-Wechselwirkungen in ihrer nativen zellulären Umgebung zu testen, aber die Extraktpräparation kann herausfordernd und zeitaufwändig sein. Eine effiziente Lyse der Probe ist notwendig, aber es muss darauf geachtet werden, die Störung von Protein-Protein-Wechselwirkungen zu minimieren. Methoden wie Douncing5, Beschallung6, Balch Homogenisierung7und Zirkonia Perlen-Homogenisierung8,9 wurden verwendet, um C. elegans Gesamtproteinextrakte erfolgreich vorzubereiten. Diese Methoden, mit Ausnahme der Zirkonia-Perlenhomogenisierung, haben Einschränkungen in Bezug auf die Anzahl der Proben, die gleichzeitig verarbeitet werden können. Präsentiert ist eine alternative Methode, die leicht skaliert werden kann, um eine schnelle Proteinextrakt-Präparation mit hohem Durchsatz aus C. elegans-Proben mit anschließender Co-Immunpräzipation zuermöglichen. Insbesondere kann die Methode bis zu 24 Proben gleichzeitig vorbereiten, wodurch die Zeit für die Extraktvorbereitung erheblich reduziert wird. Im Gegensatz dazu erlaubt z. B. das Douncing in der Regel nur eine Probenvorbereitung nach der anderen. Diese Extraktmethode kann verwendet werden, um Extrakte aus jeder Entwicklungsphase von C. elegansvorzubereiten.
Beschrieben wird ein schrittweises Verfahren zur Entnahme von Tierproben, zur Extraktpräparation, Immunpräzipitation und zur Darstellung von Western Blotting-Daten, um einen erfolgreichen Proteinabzug und den Nachweis des mitimmunisierenden Proteins zu bestätigen. Um die Wirksamkeit des Protokolls zu demonstrieren, wurden zwei Co-Immunpräzipationsexperimente zwischen 1) microRNA Argonaute ALG-1 und AIN-1, einem GW182-Homolog, durchgeführt; und 2) ALG-1 und HRPK-1, ein neu identifizierter ALG-1-Interaktor2. ALG-1 und AIN-1 sind Kernproteine, die den microRNA-induzierten Silencing-Komplex (miRISC) bilden und die Wechselwirkung zwischen diesen beiden Proteinen ist gut etabliert10,11. Das Extraktpräparationsprotokoll war im ALG-1-AIN-1-Koimmunpräzipitationsexperiment wirksam. Dieses Protokoll bestätigte auch erfolgreich die Interaktion zwischen ALG-1 und seinem neu identifizierten Interaktor HRPK-12.
Zusammenfassend beschreibt das Manuskript ein C. elegans Extrakt-Vorbereitungsprotokoll, das bis zur gleichzeitigen Verarbeitung von 24 Proben skaliert werden kann, zusammen mit einem Co-Immuno-Blutungsprotokoll, das verwendet werden kann, um neue oder hypothetische Wechselwirkungen zwischen Proteinen zu identifizieren oder zu bestätigen. Das Extraktvorbereitungsprotokoll ist mit einer Reihe von nachgeschalteten Experimenten kompatibel, einschließlich Proteinimmunpräzipitation2 und microRNA Pulldowns12. Darüber hinaus kann das Immunpräzipitationsprotokoll angepasst werden, um Wechselwirkungen zwischen zwei oder mehr endogenen, endogen markierten oder überexprimierten C. elegans-Proteinen in einer Vielzahl genetischer Hintergründe zu testen.
Protokoll
1. Wurmprobensammlung
- Samen gemischte Stufe oder synchronisiert13 Würmer auf NGM Feststoffplatten bei der erforderlichen Temperatur und lassen Sie die Würmer bis zum gewünschten Stadium wachsen. Grundlegende s. C. elegans Wachstum und Wartung finden Sie unter Stiernagle et al. und Porta-de-la-Riva et al.14,13.
- Sammeln Sie Würmer in einem 15 ml konischen Zentrifugenrohr, indem Sie die Schneckenplatten mit M9-Puffer waschen.
- Pellet die Würmer durch Zentrifugieren bei 400 x g bei Raumtemperatur (RT) für 2 min und entsorgen Sie den Überstand.
HINWEIS: Die Schneckenpelletgröße für die Extraktzubereitung liegt zwischen 100 und 500 l. Für nachgelagerte Immunpräzipationsexperimente wird ein 300-L-Pellet verpackter Würmer empfohlen, der in der Regel 4,5 mg Gesamtprotein liefert, während ein 500-L-Pellet 7,5 mg Gesamtprotein ergibt. - Führen Sie zusätzliche 3:5-Waschungen mit M9-Puffer (siehe Tabelle 1) oder bis der Überstand nicht mehr bewölkt ist.
- Führen Sie eine letzte Wäsche mit ddH2O durch.
- Das lose Schneckenpellet auf ein 1,5 ml Mikrozentrifugenrohr verschieben und bei RT 2 min bei 400 x g drehen. Entsorgen Sie den verbleibenden Überstand, um ein gepacktes Wurmpellet zu erhalten, und fahren Sie mit der Extraktion der Zubereitung fort.
HINWEIS: Das Protokoll kann hier angehalten werden. Wurmpellets können sofort in flüssigem Stickstoff eingefroren und bei -80 °C oder in flüssigem Stickstoff gelagert werden. Bitte beachten Sie, dass Wurmpellets nur einmal aufgetaut werden können und nicht eingefroren werden können.
2. Extraktvorbereitung des Schneckenpellets
HINWEIS: Die Extraktzubereitung sollte auf Eis oder bei 4 °C erfolgen.
- Wenn gefroren, tauen Wurm Pellet auf Eis.
HINWEIS: Wenn die gewünschte verpackte Schneckenpelletgröße von 300 l während der Probenentnahme nicht erreicht wurde, können mehrere kleinere Pellets kombiniert werden, bis genügend Material für die weitere Extraktion vorhanden ist. - Fügen Sie ein gleiches Volumen eiskalter 2x Lysepuffer (60 mM HEPES, pH = 7,4, 100 mM Kaliumchlorid, 0,1% Triton X, 4 mM Magnesiumchlorid, 10% Glycerin, 2 mM DTT mit RNase-Hemmer, Protease-Inhibitor und Phosphatase-Inhibitoren hinzu; siehe Tabelle 1) und Wirbel oder Pipetten nach oben und unten, um sie zu mischen. Drehen Sie die Rohre nach unten, um das Gemisch an der Unterseite des Rohres zu sammeln.
- Die Mischung in ein 1,5 ml RNase-freies Rohr mit Metallperlen (siehe Materialtabelle)bewegen und die Probe in den Perlenmühlenhomogenisator (siehe Materialtabelle) bei 4 °C geben. Stellen Sie sicher, dass die Rohrkappen angezogen und die Proben innerhalb des Homogenisators ausbalanciert sind.
- Homogenisieren Sie die Probe mit der höchsten Geschwindigkeit (Einstellung 12) für 4 min.
- Entfernen Sie die Probe aus den Perlen und legen Sie sie in ein neues 1,5 ml Mikrozentrifugenrohr. Alternativ kann ein mit dem Homogenisator versehener Magnet verwendet werden, um die Perlen aus der Probe zu entfernen.
- Drehen Sie den Extrakt bei 19.000 x g für 20 min bei 4 °C, um den Proteinextrakt zu klären.
- Übertragen Sie den Überstand in eine frische 1,5 ml Röhre auf Eis, während Sie die Übertragung des weißen, trüben Niederschlags vermeiden, der sich auf der Oberseite der Probe bildet. Der Überstand ist nun der geklärte Auszug.
HINWEIS: Speichern Sie 10 l des geklärten Extraktes, um die Gesamtproteinkonzentration zu bestimmen. - Verwenden Sie den Extrakt sofort für die folgenden Experimente oder blitzen Sie den Extrakt in flüssigem Stickstoff einfrieren und lagern Sie ihn bei -80 °C.
HINWEIS: Das Protokoll kann hier angehalten werden. Extrakte können bei extrem niedriger Temperatur gelagert werden (-80 °C Gefrierschrank oder flüssiger Stickstoff für 6 Monate). Gefrorene Extrakte können einmal aufgetaut und nicht eingefroren werden. - Bestimmen Sie die Gesamtproteinkonzentration des Extrakts anhand eines mit Reinigungsmitteln kompatiblen Proteinkonzentrations-Assay-Kits (siehe Materialtabelle)gemäß den Anweisungen des Herstellers.
3. Immunitizipation
HINWEIS: Alle Immunpräzipitungsschritte zur Extraktpräparation sollten auf Eis oder bei 4 °C durchgeführt werden. Es wird empfohlen, 2 mg Gesamtprotein für jede Immunpräzipitation zu verwenden. Es wurden jedoch erfolgreiche Immunpräzipitationen mit 0,8–1 mg Gesamtprotein durchgeführt. Verwenden Sie immer frische oder frisch aufgetaute Proteinextrakte. Das folgende Protokoll wird beschrieben, um Immunpräzipitation von 2 mg Gesamtprotein oder ein einzelnes Immunpräzipitationsexperiment durchzuführen. Die Menge an Perlen und Antikörpern kann bei mehreren Proben oder bei Verwendung einer anderen Menge an Proteinextrakt entsprechend erhöht oder verringert werden.
- Den Proteinextrakt auf Eis legen oder auftauen. Die Probe kann auf 10 mg/ml oder 5 mg/ml mit eiskaltem 1x Lysepuffer verdünnt werden (siehe Tabelle 1).
- Setzen Sie die magnetischen Perlen durch Inversion wieder auf und übertragen Sie 150 l der 50%igen Perlensuspension in ein 1,5 ml Rohr. Magnetisieren Sie die Perlen auf Eis gegen einen magnetischen Ständer für 1 min oder bis die Lösung klar ist. Entsorgen Sie den Überstand.
- Entfernen Sie das Rohr aus dem magnetischen Ständer und waschen Sie die Perlen in 1x Lysepuffer mit 2 Bänden (d. h. 300 l) Perlenschlämme. Wiederholen Sie die Wäsche 2x für insgesamt drei Wäschen.
- Setzen Sie die Perlen in 150 l eiskalten Lysepuffer aus.
- 75 l der Perlenschlämme auf 2 mg Proteinextrakt übertragen und bei 4 °C für 1 h mit sanfter Rührung inkubieren. Speichern Sie die verbleibende Perlensuspension auf Eis für die spätere Verwendung.
HINWEIS: Dieser Schritt wird durchgeführt, um die unspezifische Proteinbindung an Perlen während des Immunpräzipitationsschritts zu reduzieren. - Legen Sie das Rohr mit Probe auf den magnetischen Ständer auf Eis für 1 min oder bis die Perlen vollständig magnetisiert sind und die Probe klar ist. Übertragen Sie den Überstand auf ein neues 1,5 ml Rohr; die Perlen nicht stören. Dies ist das vorgereinigte Proteinlysat. Sparen Sie 10 % der Stichprobe für die Western Blot-Analyse.
- Fügen Sie dem vorgereinigten Lysat 20 g affinitätsgereinigten Antikörper hinzu und inkubieren Sie bei 4 °C für 1 h mit sanfter Rührung.
HINWEIS: Die Menge an Antikörpern, die für Die Immunpräzipitation verwendet werden, ist spezifisch für Antikörper und Protein eisern und sollte empirisch bestimmt werden, um eine wirksame Immunpräzipation des Zielproteins zu gewährleisten. - Die restlichen 75 l der vorgewaschenen Perlensuspension (Schritt 3.5) in das Antikörper-Lysat-Gemisch geben und bei 4 °C mit sanfter Rührung 1 h bebrüten.
- Legen Sie das Rohr in den magnetischen Ständer auf Eis für 1 min oder bis die Perlen vollständig magnetisiert sind und die Probe klar ist. Speichern Sie den Überstand für die Western Blot-Analyse (optional).
- Waschen Sie die Perlen, die das Immunprecipitat 3x in 450 l Waschpuffer (30 mM HEPES, pH = 7,4, 100 mM Kaliumchlorid, 0,1% Triton X, 2 mM Magnesiumchlorid, 10% Glycerin, 1 mM DTT; siehe Tabelle 1) auf Eis enthalten.
HINWEIS: Zusätzliche Waschungen können durchgeführt werden, wenn strengere Waschbedingungen bevorzugt werden. - Setzen Sie das Perlenpellet in 20 l 2x SDS/BME-Proteingel-Ladepuffer (siehe Materialtabelle)wieder auf und denaturieren Sie das Perlenpellet 5 min vor dem Laden auf ein SDS-PAGE-Gel. Alternativ können denaturierte Proben mehrere Monate bei -20 °C gelagert werden.
HINWEIS: Ein Teil des Perlenimmunprecipitats kann auf Wunsch für die nachgeschaltete RNA-Isolierung gespeichert werden.
4. Western Blot-Erkennung von IP-Proben
- Laden Sie die IP-Proben auf das SDS-PAGE-Gel (siehe Materialtabelle). Vermeiden Sie die Übertragung der Perlen, indem Sie die IP-Röhren für 1 min vor der Aspiration der Probe auf den magnetischen Ständer legen.
- Führen Sie die Western Blotting15,16 und Antikörper Färbung16 mit den folgenden Modifikationen: verdünnen Sie den ALG-1 Antikörper17 1:500 in 5% fettfreie Trockenmilch (NFDM); den HRPK-1-Antikörper2 1:1.000 in 5% NFDM verdünnen; und verdünnen Sie den AIN-1-Antikörper18 1:10.000 in 5% NFDM. Sekundäre Antikörper (siehe Materialtabelle) wurden gemäß den Anweisungen des Herstellers verwendet.
- Erkennen Sie die Bänder mit HRP-basierter Chemilumineszenz (siehe Materialtabelle).
Ergebnisse
Dieses Protokoll (schematisiert in Abbildung 1) wurde erfolgreich verwendet, um C. elegans Gesamtproteinextrakte zu erhalten (Abbildung 2) für die nachgeschaltete Immunitizipation mehrerer Proteine2 (Abbildung 3 und Abbildung 4). Das vorgestellte K.o.-Protokoll zur Homogenisatoren-Gesamtmenge war mit douncebasierten Methoden (Abbildung 2) und effizient extrahierten Kernkernen (COL-19::GFP(NLS) (Abbildung 2) und zytoplasmatischen Proteinen ( Abbildung3 und Abbildung 4) vergleichbar. Mehrere Proben unterschiedlicher Größe wurden gleichzeitig extrahiert (Abbildung 2). Argonaute-Proteine interagieren mit Mitgliedern der GW182-Proteinfamilie und bilden die miRISCs, die an den Zielboten RNAs binden und ihre Expression unterdrücken10. Abbildung 3 zeigt die erfolgreiche Koimmunpräzipation der kernigen miRISC-Komponenten ALG-1 und AIN-1, im Einklang mit früheren Berichten11,17. In jüngerer Zeit wurden Anstrengungen unternommen, um zusätzliche Proteininteraktoren von Argonaute ALG-13 zu identifizieren, um mehr darüber zu erfahren, wie die MikroRNA-Biogenese und -aktivität durch Hilfsfaktoren reguliert werden könnte. Das RNA-bindende Protein HRPK-1 wurde in ALG-1-Immunpräcipitaten3identifiziert. Diese Wechselwirkung wurde kürzlich in einem reziprokierten HRPK-1-Immunpräzipitationsexperimentbestätigt 2. Die vorgestellten Extrakt- und Immunpräzipitationsprotokolle haben ALG-1 erfolgreich in HRPK-1-spezifischen Co-Immunpräcipitaten wiederhergestellt (Abbildung 4). Darüber hinaus wurde die ALG-1-AIN-1-Wechselwirkung in einer Vielzahl von genetischen Hintergründen getestet und HRPK-1 erwies sich für die ALG-1/AIN-1 miRISC-Baugruppe2 (Abbildung 3) als unnötig. Zusätzliche Zahlen werden zur Verfügung gestellt, um die vollständige Membransonde zu zeigen (Ergänzende Abbildung 1).
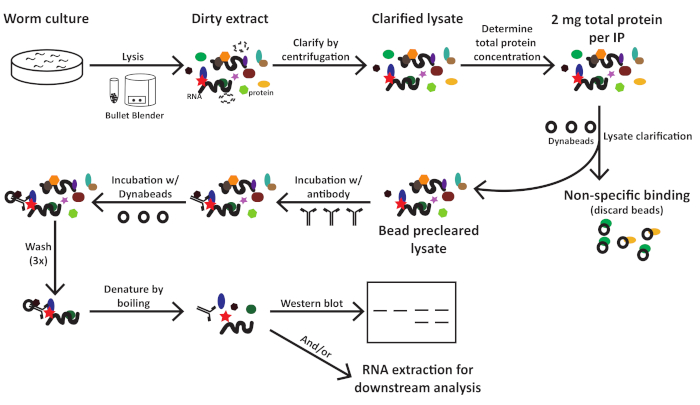
Abbildung 1: Workflow-Schema für C. elegans Extrakt-Präparation und Immunpräzipitation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
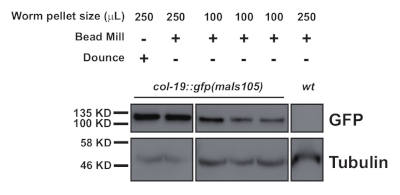
Abbildung 2: Western Blot Vergleich eines nuklear lokalisierten GFP, COL-19::GFP(NLS), Gehalte in dounce-prepared und homogenisierten Proben aus 250 l und 100 l Wurmpellets. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
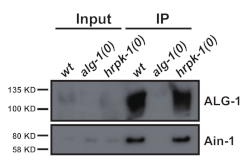
Abbildung 3: GW182 Homolog AIN-1 co-immunoprecipitates mit ALG-1. Western Blotting für ALG-1- und AIN-1-Proteine in ALG-1-Immunpräcipitaten. Die ALG-1/AIN-1 Co-Immunpräzipitation wurde durch das Fehlen von hrpk-1 nicht beeinflusst. Eingang = 10% der IP. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: ALG-1-Co-Immunpräcipitate mit HRPK-1. Western Blotting für HRPK-1 und ALG-1 in HRPK-1-Immunpräcipitaten wird gezeigt. Eingang = 10% der IP. * zeigt eine schwere Antikörperkette an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| M9-Puffer (1 L) | |
| KH2PO4 | 3 g |
| Na2HPO4 | 6 g |
| Nacl | 5 g |
| 1 M MgSO4 | 1 ml |
| ddH2O | bis 1 L |
| 2x Lysispuffer (5 ml) | |
| HEPES (pH 7.4) | 200 l |
| 2 M KCl | 250 l |
| 10% TritonX | 100 l |
| 1 M MgCl2 | 20 l |
| 100% Glycerin | 1 ml |
| ddH2O | bis zu 5 ml |
| Frisch hinzufügen: | |
| 1 M DTT | 20 l |
| EDTA-freier Protease-Inhibitor | 1 Tablette |
| Phosphatase-Inhibitor-Cocktail 2 | 100 l |
| Phosphatase-Inhibitor-Cocktail 3 | 100 l |
| 1x Lysis-Puffer | |
| 2x Lysispuffer mit gleichem Volumen von ddH20 verdünnen. | |
| 1x Waschpuffer 10 ml) | |
| HEPES (pH 7.4) | 300 l |
| 2 M KCl | 500 l |
| 10% TritonX | 100 l |
| 1 M MgCl2 | 20 l |
| 100% Glycerin | 1 ml |
| ddH2O | bis 10 ml |
| 1 M DTT | 20 l (frisch hinzufügen) |
Tabelle 1: Rezepte
Ergänzende Abbildung 1. Es werden vollständig sonsonierte westliche Blotmembranen gezeigt, die zur Erzeugung von Figuren 2-4 verwendet werden. (A) Sondenmembran für Abbildung 2. Beachten Sie, dass die Membran geschnitten wurde, um gleichzeitige Sondierungen für GFP und Tubulin zu ermöglichen, wodurch die Gesamtfleckgröße reduziert wurde. (B) Sondenmembran für Abbildung 3. (C) Sondenmembran für Abbildung 3. *bezeichnet die schwere Kette des Antikörpers. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
C. elegans ist ein ausgezeichnetes Modell für die Untersuchung grundlegender Fragen in der Zell-, Molekular- und Entwicklungsbiologie19. Zusätzlich zu seiner Macht als genetisches Modellsystem ist C. elegans für biochemische Ansätze geeignet, einschließlich, aber nicht beschränkt auf, Protein-Immunpräzipitation und Co-Immunpräzipation. Eine mögliche Hürde bei der Durchführung von Immunpräzipitationsexperimenten ist der Mangel an Antikörpern, die für die Proteine von Interesse spezifisch sind. Wenn kein Antikörper verfügbar ist, können benutzerdefinierte polyklonale oder monoklonale Antikörper erzeugt werden. Jüngste Innovationen in der Genom-Editing-Technologie haben es Forschern jedoch ermöglicht, schnell Mutationen einzuführen oder endogene C. elegans Gene20,21zu markieren, was Studien erleichtert, die die genetischen, funktionellen und physikalischen Wechselwirkungen zwischen den Genen und den kodierten Proteinen entwirren. Insbesondere die CRISPR/Cas9-vermittelte Kennzeichnung von C. elegans-Genen an den endogenen Loci hat die Abhängigkeit von Immunpräzipitationsexperimenten von der Verfügbarkeit von Antikörpern reduziert und Co-Immunpräzipitationsexperimente viel realisierbarer gemacht. C. elegans Gene können mit einer Vielzahl von Tags getaggt werden, die von fluoreszierenden Tags wie GFP oder mCherry bis hin zu kleinen Tags wie FLAG und HA reichen. Antikörper, die diese Tags erkennen, sind leicht kommerziell verfügbar, was die Studien von Protein-Protein-Wechselwirkungen über Immunpräzipationsansätze erleichtert.
Das vorgelegte Protokoll, das in Abbildung 1beschrieben ist, kann für eine kleine Anzahl von Proben durchgeführt oder skaliert werden, so dass bis zu 24 Probenpräparate gleichzeitig möglich sind. Während die anfänglichen Charakterisierungen von Protein-Protein-Wechselwirkungen über Immunpräzipitation enthoben in der Regel in wilden Hintergründen unter normalen Wachstumsbedingungen erfolgen, erfordern Folgestudien häufig Tests der Protein-Protein-Wechselwirkungen in einer Vielzahl von genetischen Hintergründen oder unter verschiedenen Wachstumsbedingungen. Die Möglichkeit, mehrere Extrakte gleichzeitig vorzubereiten, spart Zeit und sorgt vor allem für die Konsistenz der Extraktvorbereitung zwischen den verschiedenen Proben. Eine negative Kontrolle ist immer erforderlich, wobei die ideale Kontrolle eine Nullmutation in der Genkodierung für das immunvorzeichnetse Protein ist (Beispiele siehe Abbildung 3 und Abbildung 4).
Dieses Extraktprotokoll ermöglicht eine schnelle Proteinextrakt-Präparation aus C. elegans Proben und ist vergleichbar mit der zirkoniumperlenbasierten Homogenisierung8. Die Perlenhomogenisierung kann im Allgemeinen mit einer Vielzahl von Perlenmühlenhomogenisatoren oder ähnlichen Geräten auf mehrere gleichzeitige Probenpräparate skaliert werden. Einige wirtschaftlichere Perlenmühlenhomogenisatoren können jedoch die Anzahl der Proben reduzieren, die gleichzeitig verarbeitet werden können. Alternativ ist das vorgestellte Extraktprotokoll mit der dounce-basierten Extraktzubereitung kompatibel, was eine wirtschaftliche Alternative darstellt. Während verschiedene Perlenmühlenhomogenisatoren nicht getestet wurden, sind die meisten wahrscheinlich mit diesem Proteinextrakt-Protokoll kompatibel, solange eine vollständige Störung der C. elegans Proben erreicht wird.
Wie dargestellt, ist dieses Extraktvorbereitungsprotokoll mit mehreren nachgeschalteten Experimenten kompatibel, einschließlich Proteinimmunpräzipitation2 und microRNA Pull-down12 und ermöglicht die nachgelagerte Sammlung von Protein- und RNA-Komponenten. Es extrahiert auch effizient sowohl kerntechnische als auch zytoplasmatische Proteine (Abbildung 2, Abbildung 3und Abbildung 4). In ähnlicher Weise ermöglicht das vorgestellte Immunpräzipitationsprotokoll die RNA-Isolierung von proteinassoziierten Immunpräcipitaten. Während das Immunpräzipitationsprotokoll ursprünglich entwickelt wurde, um ALG-1-Proteininterakisatoren zu identifizieren, kann die Methode angepasst werden, um Wechselwirkungen zwischen beliebigen Proteinen zu testen. Tatsächlich funktionierten die verwendeten Immunpräzipitationsbedingungen bei der Immunpräzipierung von ALG-1 (Abbildung 3) und HRPK-1 (Abbildung 4) gleichermaßen gut. Dieses Protokoll ist ein ausgezeichneter Ausgangspunkt für die Immunreinigung von RNA-bindenden Proteinen. Es sollte jedoch beachtet werden, dass einige Änderungen in der Pufferzusammensetzung für andere Proteine von Interesse erforderlich sein können. Die Veränderungen können von den physikalischen und biochemischen Eigenschaften des von Interesse sindden Proteins abhängen und müssen von Fall zu Fall umgesetzt werden.
Sobald das Zielprotein (hier ALG-1 oder HRPK-1) immunprezipiert ist, kann Western Blotting verwendet werden, um das Co-Immunoprecipitat auf bestimmte Proteininteraktoren zu testen.
Alternativ kann das korifizierte Immunprecipitat einer Massenspektrometrieanalyse unterzogen werden, um alle vermeintlich interagierenden Proteine zu identifizieren. Bestätigte Co-Immunpräzipitations-Wechselwirkungen können dann in einer Vielzahl von genetischen Hintergründen oder Bedingungen untersucht werden, um eine mögliche Regulierung der spezifischen Wechselwirkung zu identifizieren. Um beispielsweise festzustellen, ob hrpk-1 eine Rolle in der ALG-1/AIN-1 miRISC-Assembly spielt, wurde die ALG-1-AIN-1-Kopräzipation sowohl in einem wilden Typ-Hintergrund als auch in Abwesenheit von HRPK-1 (Abbildung 3) bewertet. hrpk-1 erwies sich als entbehrlich für DIE ALG-1/AIN-1-Wechselwirkung2 (Abbildung 3). Darüber hinaus kann die CRISPR/Cas9 Genom-Editing-Technologie eingesetzt werden, um Single-Point- oder Domain-Löschmutationen in den proteinen von Interesse zu erzeugen. Das erneute Testen der Fähigkeit der erzeugten Mutanten, mit ihren Proteininteraktoren zu koprezipieren, kann zeigen, welche Domänen oder Rückstände die physikalische Interaktion vermitteln. Solche zukünftigen Studien können unschätzbare Informationen über den Mechanismus der Proteinfunktion und -regulierung liefern. Diese Ansätze, kombiniert mit der Kraft der C. elegans Genetik, können wichtige Einblicke in die grundlegenden molekularen Prozesse liefern, die die Entwicklung und Zellfunktion von Tieren steuern.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde teilweise unterstützt von Kansas INBRE, P20GM103418 an Li und Zinovyeva und R35GM124828 an Zinovyeva. Wir danken Min Han für die großzügige gemeinsame Nutzung des Anti-AIN-1-Antikörpers. Einige der im Laufe dieser Arbeit verwendeten Stämme wurden vom Caenorhabditis Genetics Center (CGC) bereitgestellt, das vom NIH Office of Research Infrastructure Programs (P40 OD010440) finanziert wurde.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL tube | VWR | 89039-664 | STEP 1.2 |
| 2x Laemmli Sample Buffer | BioRed | 1610737 | STEP 3.11 |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels | BioRed | 4561096 | STEP 4.1 |
| anti-AIN-1 monoclonal antibody | custom generated | n/a | STEP 4.2, see ref. Zhang et al. 2007 |
| anti-ALG-1 monoclonal antibody | custom generated by PRF&L | n/a | STEP 4.2 |
| anti-HRPK-1 monoclonal antibody | custom generated by PRF&L | n/a | STEP 4.2 |
| Bullet Blender Storm Homogenizer | MidSci | BBY24M | STEP 2.3 |
| DL-Dithiothreitol (DTT) | Sigma | D9779-5G | Table 1 |
| Dynabeads Protein A for Immunoprecipitation | Thermo Fisher | 10002D | STEP 3.2 |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | STEP 3.2 |
| EDTA-free protease inhibitors | Roche | 11836170001 | Table 1 |
| GFP antibody (FL) | Santa Cruz Biotechnology | sc-8334 | Figure 2 |
| Glycerol | Thermo Fisher | G33-500 | Table 1 |
| Goat Anti-Rabbit Secondary Antibody, HRP | BioRed | 1662408 | STEP 4.2 |
| Goat anti-Rat IgG (H+L) Secondary Antibody, HRP | Thermo Fisher | 31470 | STEP 4.2 |
| HEPES | Sigma | H4034-500G | Table 1 |
| LICOR WesternSure PREMIUM Chemiluminescent Substrate, 100 mL Kit | LI-COR | 926-95000 | STEP 4.3 |
| Magnesium chloride hexahydrate ACS | VWR | VWRV0288-500G | Table 1 |
| Magnesium Sulfate Anhydrous | Thermo Fisher | M65-500 | Table 1 |
| Microcentrifuge Tubes, 1.5 mL | VWR | 20170-333 | STEP 1.6 |
| N2 wild type | CGC | ||
| Navy RINO RNA Lysis Kit 50 pack (1.5 mL) | MidSci | NAVYR1-RNA | STEP 2.3 |
| Phosphatase inhibitor cocktail 2 | Sigma | P5726-1ML | Table 1 |
| Phosphatase inhibitor cocktail 3 | Sigma | P0044-1ML | Table 1 |
| Potassium Chloride | Thermo Fisher | P217-500 | Table 1 |
| Potassium phosphate monobasic | Thermo Fisher | P285-3 | Table 1 |
| RC DC Protein Assay Kit I | BioRed | 5000121 | STEP 2.9 |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Thermo Fisher | 10777019 | Table 1 |
| Sodium Chloride | Thermo Fisher | S271-500 | Table 1 |
| Sodium Phosphate Dibasic Anhydrous | Thermo Fisher | S374-500 | Table 1 |
| TritionX-100 | Sigma | X100-500ML | Table 1 |
| UY38 hrpk-1(zen17) | available upon request | ||
| VT1367 col-19::gfp(maIS105) | available upon request | ||
| VT3841 alg-1(tm492) | available upon request |
Referenzen
- Liu, X., et al. An AP-MS- and BioID-compatible MAC-tag enables comprehensive mapping of protein interactions and subcellular localizations. Nature Communications. 9 (1), 1188-1216 (2018).
- Li, L., Veksler-Lublinsky, I., Zinovyeva, A. HRPK-1, a conserved KH-domain protein, modulates microRNA activity during Caenorhabditis elegans development. PLoS Genetics. 15 (10), 1008067(2019).
- Zinovyeva, A. Y., Veksler-Lublinsky, I., Vashisht, A. A., Wohlschlegel, J. A., Ambros, V. R. Caenorhabditis elegans ALG-1 antimorphic mutations uncover functions for Argonaute in microRNA guide strand selection and passenger strand disposal. Proceedings of the National Academy of Sciences of the United States of America. 112 (38), 5271-5280 (2015).
- Hammell, C. M., Lubin, I., Boag, P. R., Blackwell, T. K., Ambros, V. nhl-2 Modulates microRNA activity in Caenorhabditis elegans. Cell. 136 (5), 926-938 (2009).
- Zou, Y., et al. Developmental decline in neuronal regeneration by the progressive change of two intrinsic timers. Science. 340 (6130), 372-376 (2013).
- Zanin, E., Dumont, J., et al. Affinity purification of protein complexes in C. elegans. Methods in Cell Biology. 106, 289-322 (2011).
- Bhaskaran, S., et al. Breaking Caenorhabditis elegans the easy way using the Balch homogenizer: an old tool for a new application. Analytical Biochemistry. 413 (2), 123-132 (2011).
- Kohl, K., et al. Plate-based Large-scale Cultivation of Caenorhabditis elegans: Sample Preparation for the Study of Metabolic Alterations in Diabetes. Journal of Visualized Experiments. (138), e58117(2018).
- Larance, M., Bailly, A. P., et al. Stable-isotope labeling with amino acids in nematodes. Nature Methods. 8 (10), 849-851 (2011).
- Ding, L., Han, M. GW182 family proteins are crucial for microRNA-mediated gene silencing. Trends in Cell Biology. 17 (8), 411-416 (2007).
- Ding, L., Spencer, A., Morita, K., Han, M. The Developmental Timing Regulator AIN-1 Interacts with miRISCs and May Target the Argonaute Protein ALG-1 to Cytoplasmic P Bodies in C. elegans. Molecular Cell. 19 (4), 437-447 (2005).
- Jannot, G., Vasquez-Rifo, A., Simard, M. J. Argonaute Pull-Down and RISC Analysis Using 2’-O-Methylated Oligonucleotides Affinity Matrices. Methods in Molecular Biology. 725, Chapter 16 233-249 (2011).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019(2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-11 (2006).
- Gallagher, S., Chakavarti, D. Immunoblot analysis. Journal of Visualized Experiments. (16), e759(2008).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. Journal of Visualized Experiments. (44), e2359(2010).
- Zinovyeva, A. Y., Bouasker, S., Simard, M. J., Hammell, C. M., Ambros, V. Mutations in conserved residues of the C. elegans microRNA Argonaute ALG-1 identify separable functions in ALG-1 miRISC loading and target repression. PLoS Genetics. 10 (4), 1004286(2014).
- Zhang, L., et al. Systematic Identification of C. miRISC Proteins, miRNAs, and mRNA Targets by Their Interactions with GW182 Proteins AIN-1 and AIN-2. Molecular Cell. 28 (4), 598-613 (2007).
- Corsi, A. K., Wightman, B., Chalfie, M. A Transparent Window into Biology: A Primer on Caenorhabditis elegans. Genetics. 200 (2), 387-407 (2015).
- Farboud, B., et al. Enhanced Genome Editing with Cas9 Ribonucleoprotein in Diverse Cells and Organisms. Journal of Visualized Experiments. (135), e57350(2018).
- Dickinson, D. J., Goldstein, B. CRISPR-Based Methods for Caenorhabditis elegans Genome Engineering. Genetics. 202 (3), 885-901 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten