Method Article
Ableitung von Proteinkristallen mit I3C mit Random Microseed Matrix Screening
In diesem Artikel
Zusammenfassung
Dieser Artikel stellt eine Methode zur Erzeugung von Proteinkristallen dar, die mit I3C (5-Amino-2,4,6-Triiodoisophthalsäure) mit Mikrosaatierung zur Erzeugung neuer Kristallisationsbedingungen in spärlichen Matrix-Bildschirmen deaktiviert werden. Die Schalen können mit Flüssigkeitsdosierrobotern oder von Hand aufgestellt werden.
Zusammenfassung
Die Proteinstrukturaufklärung mittels Röntgenkristallographie erfordert sowohl hochwertige diffrierende Kristalle als auch eine rechnerische Lösung des Beugungsphasenproblems. Neuartige Strukturen, denen ein geeignetes Homologiemodell fehlt, werden oft mit schweren Atomen ableitungsiert, um experimentelle Phaseninformationen zu liefern. Das vorgestellte Protokoll erzeugt effizient derivatisierte Proteinkristalle, indem zufälliges Mikrosaating-Matrix-Screening mit Derivatisierung mit einem schweren Atommolekül I3C (5-Amino-2,4,6-Triiodoisophthalsäure) kombiniert wird. Durch die Einbindung von I3C in das Kristallgitter kann das Problem der Beugungsphase effizient mit einer einzigen Wellenlängen-Anomaldispersion (SAD) gelöst werden. Die gleichseitige Dreiecksanordnung von Jodatomen in I3C ermöglicht eine schnelle Validierung einer korrekten anomalen Unterkonstruktion. Dieses Protokoll wird für Strukturbiologen nützlich sein, die makromolekulare Strukturen mit kristallographischen Techniken lösen, die an experimentellem Phasing interessiert sind.
Einleitung
Im Bereich der Strukturbiologie gilt die Röntgenkristallographie als die Goldstandardtechnik zur Bestimmung der atomaren Auflösungsstrukturen von Makromolekülen. Es wurde ausgiebig genutzt, um die molekulare Basis von Krankheiten zu verstehen, rationale Arzneimitteldesign-Projekte zu leiten und den katalytischen Mechanismus der Enzyme1,2aufzuklären. Obwohl Strukturelle Daten eine Fülle von Wissen liefern, kann der Prozess der Proteinexpression und -reinigung, Kristallisation und Strukturbestimmung äußerst mühsam sein. Häufig treten mehrere Engpässe auf, die den Fortschritt dieser Projekte behindern, und dies muss angegangen werden, um die Pipeline zur Bestimmung der Kristallstruktur effizient zu rationalisieren.
Nach rekombinanter Expression und Reinigung müssen vorgegebene Bedingungen identifiziert werden, die der Kristallisation förderlich sind, was oft ein mühsamer und zeitaufwändiger Aspekt der Röntgenkristallographie ist. Kommerzielle spärliche Matrix-Bildschirme, die bekannte und veröffentlichte Bedingungen konsolidieren, wurden entwickelt, um diesen Engpass zu lindern3,4. Es ist jedoch üblich, trotz der Verwendung von hochreinen und konzentrierten Proteinproben nur wenige Treffer aus diesen Anfangsbildschirmen zu generieren. Die Beobachtung klarer Tropfen deutet darauf hin, dass das Protein möglicherweise nicht die Übersättigungswerte erreicht, die erforderlich sind, um einen Kristall zu nukleieren. Um die Kristallkernbildung und das Wachstum zu fördern, können Samen aus bereits vorhandenen Kristallen zu den Bedingungen hinzugefügt werden, was eine erhöhte Probenahme des Kristallisationsraums ermöglicht. Ireton und Stoddard führten erstmals das Mikrosaat-Matrix-Screening-Verfahren5ein. Minderwertige Kristalle wurden zerkleinert, um einen Samenbestand herzustellen, und dann systematisch zu Kristallisationsbedingungen hinzugefügt, die verschiedene Salze enthalten, um neue Kristalle in Beugungsqualität zu erzeugen, die sich sonst nicht gebildet hätten. Diese Technik wurde weiter verbessert durch D'Arcy et al., die zufälliges Mikrosaatmatrix-Screening (rMMS) entwickelten, bei dem Samen in einen Ersatzmatrixkristallisationssieb 6,7eingeführt wurden. Dies verbesserte die Qualität der Kristalle und erhöhte die Anzahl der Kristallisationstreffer im Durchschnitt um den Faktor 7.
Nachdem Kristalle erfolgreich hergestellt und ein Röntgenbeugungsmuster erhalten ist, tritt ein weiterer Engpass in Form der Lösung des "Phasenproblems" auf. Während des Datenerfassungsprozesses wird die Intensität der Beugung (proportional zum Quadrat der Amplitude) aufgezeichnet, aber die Phaseninformationen gehen verloren, was zu dem Phasenproblem führt, das die sofortige Strukturbestimmungstoppt 8. Wenn das Zielprotein eine hohe Sequenzidentität zu einem Protein mit einer zuvor festgelegten Struktur teilt, kann der molekulare Ersatz verwendet werden, um die Phaseninformationen9,10,11,12zu schätzen. Obwohl diese Methode schnell und kostengünstig ist, sind Modellstrukturen möglicherweise nicht verfügbar oder ungeeignet. Der Erfolg der homologischen modellbasierten molekularen Ersatzmethode sinkt deutlich, da die Sequenzidentität unter 35%13fällt. In Ermangelung eines geeigneten Homologiemodells können ab initio Methoden wie ARCIMBOLDO14,15 und AMPLE16getestet werden. Diese Methoden verwenden rechnerisch vorhergesagte Modelle oder Fragmente als Ausgangspunktfürbeinen für den molekularen Ersatz. AMPLE, das vorhergesagte Ködermodelle als Ausgangspunkte verwendet, kämpft darum, Strukturen großer (>100 Rückstände) Proteine und Proteine zu lösen, die überwiegend β-Blätter enthalten. ARCIMBOLDO, das versucht, kleine Fragmente in eine größere Struktur zu passen, ist auf hochauflösende Daten (≤2) und durch die Fähigkeit von Algorithmen beschränkt, die Fragmente zu einer vollständigen Struktur zu erweitern.
Wenn molekulare Ersatzmethoden fehlschlägt, müssen direkte Methoden wie isomorpher Ersatz17,18 und anomale Streuung bei einer einzigen Wellenlänge (SAD19) oder mehreren Wellenlängen (MAD20) verwendet werden. Dies ist oft der Fall bei wirklich neuartigen Strukturen, bei denen der Kristall mit einem schweren Atom gebildet oder ableitungen muss. Dies kann durch Einweichen oder Kokristallisieren mit einer schweren Atomverbindung, chemische Modifikation (wie 5-Bromouracil-Inkorporation in RNA) oder markierte Proteinexpression (wie die Einbeziehung von Selenomethionin oder Selenocystein-Aminosäuren in die primäre Struktur) erreicht werden21,22. Dies erschwert den Kristallisationsprozess zusätzlich und erfordert zusätzliches Screening und Optimierung.
Eine neue Klasse von Phasing-Verbindungen, einschließlich I3C (5-Amino-2,4,6-Triiodoisophthalsäure) und B3C (5-Amino-2,4,6-Tribromoisophthalsäure), bieten spannende Vorteile gegenüber bereits bestehenden Phasing-Verbindungen23,24,25. Sowohl I3C als auch B3C verfügen über ein aromatisches Ringgerüst mit einer abwechselnden Anordnung von anomalen Streuungen, die für direkte Phasing-Methoden und Amino- oder Carboxylat-Funktionsgruppen erforderlich sind, die speziell mit dem Protein interagieren und eine Bindungs-Standortspezifität bieten. Die anschließende gleichseitige dreieckige Anordnung von Schwermetallgruppen ermöglicht eine vereinfachte Validierung des phasing-Unterbaus. Zum Zeitpunkt des Schreibens gibt es 26 I3C-gebundene Strukturen in der Protein Data Bank (PDB), von denen 20 mit SAD phasing26gelöst wurden.
Dieses Protokoll verbessert die Wirksamkeit der Strukturbestimmungspipeline, indem die Methoden der Schwermetall-Derivatisierung und des rMMS-Screenings kombiniert werden, um gleichzeitig die Anzahl der Kristallisationstreffer zu erhöhen und den Kristallderivatisierungsprozess zu vereinfachen. Wir haben gezeigt, dass diese Methode extrem effektiv mit Hühnerei-Weiß-Lysozym und einer Domäne eines neuartigen Lysinproteins aus Bakteriophagen P6827ist. Die Strukturlösung mit der hochautomatisierten Auto-Rickshaw-Strukturbestimmungspipeline wird speziell auf die Phasing-Verbindung I3C zugeschnitten beschrieben. Es gibt andere automatisierte Pipelines, die verwendet werden können, wie AutoSol28, ELVES29 und CRANK230. Nicht vollautomatisierte Pakete wie SHELXC/D/E können auch31,32,33verwendet werden. Diese Methode ist besonders vorteilhaft für Forscher, die Proteine ohne homologe Modelle im PDB untersuchen, indem sie die Anzahl der Screening- und Optimierungsschritte signifikant reduziert. Voraussetzung für diese Methode sind Proteinkristalle oder ein kristalliner Niederschlag des Zielproteins, das aus früheren Kristallisationsversuchen gewonnen wurde.
Protokoll
1. Experimentelle Planung und Überlegungen
- Verwenden Sie bereits vorhandene Kristalle des Proteins von Interesse, vorzugsweise durch Dampfdiffusionskristallisation erzeugt. Für ein verallgemeinertes Protokoll der Dampfdiffusionskristallisation siehe Benvenuti und Mangani34. Andere Methoden der Kristallisation wie Mikrobatch unter Öl und freie Grenzflächendiffusion erfordern die Ernte der Kristalle vor dem Zerkleinern, um Mikrosaaten zu erzeugen.
- Bei der Herstellung eines Samenbestandes verwenden Sie die hochwertigsten Kristalle, die geopfert werden können. Der Kristall höchster Qualität kann visuell anhand der Morphologie beurteilt werden oder der beste diffrierende Kristall kann ausgewählt werden, wenn solche Daten verfügbar sind. Es ist sehr wahrscheinlich, dass noch bessere Kristalle nach der Optimierung durch Aussaat erhalten werden. In dem Fall, in dem keine Kristalle verfügbar sind, können kristalline Niederschlagsstoffe wie Spherulite und Nadeln verwendet werden.
- Identifizieren Sie Salzkristalle. Salzkristalle können in Kristallisationssieben wachsen und wie Proteinkristalle aussehen. Die Verwendung von Salzkristallen in rMMS bringt keinen Nutzen und verschwendet wertvolle Proben, daher ist es wichtig, Falschpositive von Salz zu beseitigen.
- Salzkristalle sind laut, wenn sie zerkleinert werden. Kristalle müssen zerkleinert werden, um einen Saatgutbestand zu erzeugen, daher ist diese Strategie besonders relevant. Wenn beim Zerkleinern der Kristalle ein hörbarer Risston zu hören ist, ist der Kristall wahrscheinlich Salz.
- Wenn das Protein Tryptophan- und Tyrosinrückstände enthält, verwenden Sie die ultraviolette Fluoreszenzmikroskopie, um Proteinkristalle zu identifizieren, die unter diesen Lichtbedingungen fluoreszieren.
- Verwenden Sie Izit-Farbstoff (Methylenblau), um Proteinkristalle zu färben, um sie zu Salzkristallen zu unterscheiden, die relativ unbefleckt bleiben. Dieses Verfahren ist jedoch zerstörerischer und wird nur empfohlen, wenn man Kristalle von Replikationen desselben Tropfens zu verschonen hat.
HINWEIS: Obwohl die oben genannten Tests vielversprechende Ergebnisse liefern können, können Salzkristalle immer noch mit Proteinkristallen verwechselt werden. In diesem Fall können Beugungsexperimente verwendet werden, um definitiv zwischen einem Protein und einem Salzkristall zu unterscheiden.
2. Herstellung von Lithium I3C Lager
- Messen Sie 120 mg I3C (5-Amino-2,4,6-Triiodoisophthalsäure) in ein 1,5 ml Mikrozentrifugenrohr.
- Lösen Sie I3C in 200 l von 2 M Lithiumhydroxid auf. Die Lösung kann mit einem Wärmeblock bei 40-60 °C sanft erhitzt werden, um die Auflösung zu fördern. Die resultierende Lithium-I3C-Lösung sollte braun sein und eine Konzentration von 1 M haben.
VORSICHT: Lithiumhydroxid ist korrosiv. Eine Schutzbrille, Handschuhe und ein Labormantel sollten getragen werden. - Messen Sie den pH-Wert der Lösung. Falls erforderlich, fügen Sie kleine Mengen von 1 M Salzsäure oder 2 M Lithiumhydroxid hinzu, um den pH-Wert auf 7 bis 8 einzustellen. Fügen Sie MilliQ-Wasser hinzu, um das endgültige Lösungsvolumen auf 400 l zu erhöhen. Die Konzentration der I3C-Lagerlösung beträgt 0,5 M.
HINWEIS: Schritt 2.3 ist optional. Der pH-Wert der Lösung sollte vor jeder pH-Einstellung zwischen pH 7-8 liegen. Dieser Schritt sollte durchgeführt werden, wenn das Protein von Interesse stark durch pH beeinflusst wird. Das Protokoll kann hier angehalten werden. Lithium I3C kann mindestens zwei Wochen im Dunkeln gehalten werden35.
3. Zugabe von I3C zum Proteinbestand
- Methode 1
- Fügen Sie den Vorrat Lithium I3C zu einem 150-L-Aliquot des Zielproteins hinzu. Die Endkonzentration sollte zwischen 5-40 mM Lithium I3C liegen.
- Methode 2 (gentler Methode)
- Bereiten Sie einen Proteinverdünnungspuffer vor, der dem Puffer des Zielproteins entspricht. Zu diesem Verdünnungspuffer fügen Sie den Vorrat Lithium I3C hinzu, um eine Konzentration von Lithium I3C zwischen 10-80 mM zu geben.
- Das Protein 1:1 mit Proteinverdünnungspuffer verdünnen, um eine endgültige Konzentration von Lithium I3C zwischen 5-40 mM zu geben.
HINWEIS: Einige Proteine fallen aus, wenn sie mit hohen Konzentrationen von Lithium I3C in Methode 1 in Kontakt kommen, während andere Proteine es tolerieren können. Methode 2 reduziert die Niederschlagswahrscheinlichkeit. Diese Methode halbiert jedoch die Proteinkonzentration. Für Proteine, die kein etabliertes Kristallisationsprotokoll haben, wird im Allgemeinen eine Proteinkonzentration von 10 mg/ml für das erste Kristallisationsscreening empfohlen. Ein anfängliches Molverhältnis von I3C zu Protein von 8 wird empfohlen. Die Proteinkonzentration und das Molverhältnis von I3C zu Protein können nach dem ersten Bildschirm optimiert werden.
4. Herstellung eines Saatgutbestands
- Machen Sie eine abgerundete Sonde zum Zerkleinern von Kristallen.
- Mit einem Bunsenbrenner auf der blauen Flamme eine PasteurPipette in die Mitte erhitzen. Ziehen Sie mit einer Pinzette das Ende der Pasteur-Pipette, um sie in einen dünnen Durchmesser von weniger als 0,3 mm zu ziehen.
- Sobald der Mittelteil dünn genug ist, halten Sie dieses Segment in der Flamme, um die Pipette an dieser Stelle zu trennen und um das Ende der Pipette zu runden, um die Glassonde zu beenden.
HINWEIS: Abgerundete Sondenkristallbrecher werden von Drittanbietern verkauft. Diese sind eine Alternative zur Herstellung von abgerundeten Sonden.
- Fünf 1,5 ml Mikrozentrifugenrohre auf Eis legen.
- Untersuchen Sie unter einem Lichtmikroskop die Kristallisationsschale auf einen geeigneten Zustand zur Erzeugung von Mikrokristallen. Idealerweise werden gute Morphologie große Kristalle ausgewählt. Diese Technik funktioniert jedoch auch mit schlechten Morphologiekristallen, Nadeln, Platten, Mikrokristallen und Spheruliten.
- Öffnen Sie das Kristallisationstablett gut. Für 96 Brunnenkristallisationsschalen, die mit Kunststoff versiegelt sind, verwenden Sie ein Skalpell, um den Kunststoff zu schneiden, der den Brunnen versiegelt. Zum Aufhängen von mit Fett verschlossenen Fallschalen kann der Deckelrutsch mit einer Pinzette entfernt und auf eine ebene Oberfläche gedreht werden.
- 70 L Reservoirlösung auf ein Mikrozentrifugenrohr übertragen und auf Eis abkühlen lassen. Zu den anderen Mikrozentrifugenröhren 90 L Reservoirlösung hinzufügen und zum Abkühlen ins Eis zurückkehren.
HINWEIS: Wenn das Reservoir nicht genügend Volumen hat oder nicht vorhanden ist (bei Mikrobatch unter Öl), erstellen Sie Kristallisationsreservoir durch Mischen der entsprechenden Reagenzien. - Den Kristall im Tropfen mit der Kristallsonde gründlich zerkleinern. Der Kristall muss vollständig zerkleinert werden, der unter dem Mikroskop überwacht werden kann.
- Entfernen Sie die gesamte Flüssigkeit aus dem Tropfen und übertragen Sie sie mit der Reservoirlösung in das Mikrozentrifugenrohr. Mischen und anschließend 2 l Mischung aus dem Mikrozentrifugenrohr nehmen und wieder in den Brunnen geben. Spülen Sie den Brunnen mit der Lösung und übertragen Sie ihn in das Mikrozentrifugenrohr. Wiederholen Sie diesen Spülschritt noch einmal. Halten Sie ab diesem Zeitpunkt das Mikrozentrifugenrohr kalt, um das Schmelzen der Mikrosaaten in der Mischung zu vermeiden.
- Wirbeln Sie das Rohr mit maximaler Geschwindigkeit bei 4 °C für 3 min, halten Sie regelmäßig an, um die Röhre auf Eis zu kühlen, um eine Überhitzung zu verhindern.
HINWEIS: Einige Mikrosaatierungsprotokolle fügen dem Mikrozentrifugenrohr eine Polytetrafluorethylen-Samenperle hinzu, um die Kristallzerkleinerung7,36zu unterstützen. Wir haben die Technik ohne die Verwendung einer Samenperle mit Erfolg verwendet, aber sehen keine Probleme mit der Verwendung einer Samenperle, um Kristalle zu zerquetschen. - Machen Sie eine 1 in 10 serielle Verdünnung des Saatgutbestands, indem Sie sequenziell 10 l zwischen den gekühlten Reservoirlösungen übertragen.
- Saatgutbestände lagern, die nicht sofort bei -80 °C verwendet werden.
5. Einrichten eines rMMS-Bildschirms
- Einrichten einer 96 Well-Siebplatte mit einem Flüssigkeitsdosierroboter. In Ermangelung eines Roboters kann auch eine Mehrkanalpipette verwendet werden.
- Übertragen Sie 75 l von einem tiefen Brunnenblock auf ein 96 Brunnenkristallisationstablett. Fügen Sie dem Kristallisationstropfen 1 L und dem Reservoir 74 l hinzu.
- Übertragen Sie 1 L Protein, das mit Lithium I3C, in Schritt 2, ergänzt wird, auf den Kristallisationstropfen.
- Übertragen Sie 0,1 l des Saatgutbestands auf den Kristallisationsabfall.
- Versiegeln Sie die Platte mit klarem Dichtband und inkubieren Sie die Platte bei konstanter Temperatur, um Kristallwachstum zu ermöglichen.
- Einrichten eines hängenden Fallbildschirms
- Fetten Sie die Ränder der hängenden Fallbrunnen (hängende Tropfenkristallisationsschalen finden Sie in 24 und 48 Brunnenformaten).
- Übertragen Sie die Kristallisationslösung 500 l in das Reservoir.
- In der Mitte einer Glasabdeckung sypfe einen 1 L-Tropfen Protein, ergänzt mit Lithium I3C, in Schritt 2.
- Fügen Sie dem Tropfen 1 L der Kristallisationslösung hinzu.
- Übertragen Sie 0,1 l des Saatgutbestands auf den Kristallisationsabfall.
- Invertieren Sie den Deckelschlitten und versiegeln Sie die Kristallisation gut, indem Sie den Deckelschlitten in das Fett schieben.
- Inkubieren Sie die Platte bei konstanter Temperatur, um Kristallwachstum zu ermöglichen.
HINWEIS: Bei neuen und ungetesteten Saatgutbeständen wird empfohlen, den konzentriertesten Saatgutbestand zu verwenden, um die Chancen auf Kristallisationstreffer zu maximieren. Nachfolgende Bedingungen können mit reduzierter Saatgutkonzentration eingerichtet werden, um die Anzahl der Kristalle zu optimieren.
- Untersuchen Sie Kristallschalen unter dem Mikroskop regelmäßig auf Kristallwachstum. Wenn Kristalle von ausreichender Qualität sind, können sie für die Datenerfassung geerntet werden. Kristalle können auch verwendet werden, um neue Saatgutbestände und neue rMMS-Bildschirme zu generieren, um eine iterative Optimierung zu ermöglichen.
6. Datenerhebung
- Ernten Sie Kristalle mit Kryoloops, kryoprotect die Kristalle und Blitz kühlen sie in flüssigem Stickstoff. Weitere Informationen zu Blitzkühlkristallen finden Sie unter Teng37 und Garman und Mitchell38.
- Während der Kryoschutzphase kann I3C aus dem Kristall verloren gehen, wenn er durch eine neue wässrige Lösung geleitet wird, da er in die Kryoschutzlösung einfließt. Verwenden Sie Lithium I3C in der Kryoschutzlösung in einer Konzentration, die der Kristallisationsbedingung entspricht, um dies zu mildern.
- Kristalle, die mit diesem Protokoll angebaut wurden, wurden erfolgreich mit Parabar 10312 Ölbasis Kryoprotektor (Hampton Research) kryogeschützt.
HINWEIS: Das Protokoll kann hier angehalten werden, während Kristalle in flüssigem Stickstoff gespeichert werden.
VORSICHT: Flüssiger Stickstoff kann zu kalten Verbrennungen führen. Flüssiger Stickstoff kann auch erstickungsbewirken, wenn er in geschlossenen Räumen verwendet wird.
- Montieren Sie den Kristall auf dem Röntgenquellen-Goniometer und sammeln Sie Beugungsdaten mithilfe des für die Röntgenquelle spezifischen Protokolls.
- Diese Technik beruht auf anomalen Signalen von Jodatomen in I3C. Wählen Sie daher die Energie des Röntgens aus, um dieses Signal zu maximieren.
- Stellen Sie Synchrotron-Röntgenquellen mit einstellbaren Energien so niedrig wie möglich ein. Für viele makromolekulare Kristallographie-Strahllinien ist die niedrigste konfigurierbare Energie 8000 bis 8500 eV.
- Rotierende Anoden-Röntgenquellen können nicht gestimmt werden. Häufig verwendete Anodenquellen mit Kupfer haben die K-Kante bei 8046 eV, die ein gutes anomales Signal für Jod liefert (f" = 6,9 e). Anodenquellen mit Chrom haben eine K-Kante bei 5415 eV, die ein großes anomales Signal für Jod liefert (f" = 12,6 e).
- Strahlungsschäden sind ein erhebliches Problem bei der Datenerfassung, da sie das anomale Signal39abbauen. Wählen Sie die Belichtungszeit und Diedämpfung des Strahls, um die beste Beugung zu erreichen und gleichzeitig die Strahlendosis zu minimieren.
HINWEIS: In einer ähnlichen Phasing-Verbindung mit den Jodatomen, die durch Bromatome ersetzt werden, hat sich gezeigt, dass Strahlenschäden die Radiolyse der Kohlenstoffbrombindung und eine Verringerung der Belegung der Bromatome24verursachen.- Verwenden Sie die inverse Balken-SAD-Datensammlung als Sammlungsstrategie. Die Daten werden in Keilen gesammelt, wobei entgegengesetzte Keile hintereinander gesammelt werden. Dadurch können Friedel-Paare mit einer äquivalenten Dosis gesammelt werden, was zu einer verbesserten Messung des anomalen Signals führt, das weniger von Strahlenschäden betroffen ist. Zum Beispiel, eine Strategie von acht Keilen zur Erfassung von 360° würde die Erfassung der Daten in der Reihenfolge von Keil 1 (0°-45°), Keil 2 (180°-225°), Keil 3 (46°-90°), Keil 4 (225°) beinhalten. -270°), Keil 5 (90°-135°), Keil 6 (270°-315°), Keil 7 (135°-180°) und Keil 8 (315°-360°).
HINWEIS: Kontinuierliche Rotation ist eine alternative Erfassungsstrategie zu der Sammlung von Inversen Strahlendaten. Einen aktuellen Vergleich der Sammlungsstrategien finden Sie unter Garcie-Bonte & Katona40.
- Verwenden Sie die inverse Balken-SAD-Datensammlung als Sammlungsstrategie. Die Daten werden in Keilen gesammelt, wobei entgegengesetzte Keile hintereinander gesammelt werden. Dadurch können Friedel-Paare mit einer äquivalenten Dosis gesammelt werden, was zu einer verbesserten Messung des anomalen Signals führt, das weniger von Strahlenschäden betroffen ist. Zum Beispiel, eine Strategie von acht Keilen zur Erfassung von 360° würde die Erfassung der Daten in der Reihenfolge von Keil 1 (0°-45°), Keil 2 (180°-225°), Keil 3 (46°-90°), Keil 4 (225°) beinhalten. -270°), Keil 5 (90°-135°), Keil 6 (270°-315°), Keil 7 (135°-180°) und Keil 8 (315°-360°).
7. Datenverarbeitungs- und Strukturlösung
- Führen Sie die Datenreduktion der Beugungsdaten mit XDS41durch, um das anomale Signal zu maximieren. Eingabeparameter zur Datenreduktion sind spezifisch für das Dataset und erfordern möglicherweise einige Test- und Fehler. Hier sind einige Empfehlungen, um zu beginnen.
- Setzen Sie FRIEDEL'S LAW=FALSE ein. Führen Sie CORRECT zweimal aus, indem Sie STRICT_ABSORPTION_CORRECT = TRUE und STRICT_ABSORPTION_CORRECT = FALSE festlegen. Ein Lauf kann ein höheres anomales Signal haben als der andere. Vergleichen Sie die anomalen Signale zwischen den Läufen mit den Disziplinen "Anomal Corr" und "SigAno" im Ausgang. Dies liefert einen Indikator für die Datenqualität.
- Führen Sie SHELXC auf der XDS_ASCII aus. HKL-Datei für eine genauere Anzeige des anomalen Signals. Die Disziplin "Ranom" wird in verschiedenen Entschließungen auf das anomale Signal hinweisen.
- Führen Sie POINTLESS42 und AIMLESS43 aus, um die Daten zu skalieren. Legen Sie in AIMLESS den Parameter ANOMALOUS ON fest. Wenn die GUI verwendet wird, wählen Sie die Option Anomale Paare trennen für Ausreißerabweisung und Zusammenführen von Statistiken. Das Testen verschiedener Auflösungsabschaltungen kann erforderlich sein, um das anomale Signal zu maximieren.
- Lösen Sie die Proteinstruktur mit Auto-Rickshaw automatisierte Kristallstruktur Bestimmung Pipeline44. Auto-Rickshaw wird versuchen, das Phasenproblem zu lösen und die Kristallstruktur des Proteins automatisch mit Proteinmodellierungs- und Verfeinerungssoftware aufzubauen.
- Führen Sie für Proteine ohne Homologiemodellvorlage das SAD-Protokoll von Auto-Rickshaw im erweiterten Modus aus. Geben Sie die erforderlichen Parameter ein.
- Wählen Sie PROTEIN als Molekültyp aus.
- Geben Sie die Wellenlänge der Datensammlung in angstroms ein.
- Wählen Sie "I" als Unterstrukturelement aus, um anzugeben, dass Jodatome verwendet wurden.
- Wählen Sie "i3c" als Unterstrukturtyp aus, um anzugeben, dass I3C das Phasing-Molekül war.
- Wählen Sie "sub_direct" als Unterstrukturbestimmungsmethode aus. Diese Methode verwendet SHELXD32, um nach der Unterstruktur zu suchen.
- Wählen Sie "3" als Anzahl der erwarteten Unterstruktur pro Monomer aus.
- Geben Sie "1" als Auflösungsschnitt der Unterstruktursuche ein. Dadurch kann Auto-Rickshaw automatisch einen geeigneten Auflösungsschnitt bestimmen.
- Geben Sie die Anzahl der Rückstände in einem einzigen Monomer, die Raumgruppe des Datasets und die Anzahl der Moleküle in der asymmetrischen Einheit basierend auf dem Matthews-Koeffizienten ein.
- Wählen Sie die geeignete Verbreitungsstufe von Röntgendaten aus, die den Anforderungen entspricht. Wenn Sie "AutoRickshaw-Entwickler" auswählen, können Auto-Rickshaw-Entwickler den Lauf beheben, wenn Probleme auftreten.
- Geben Sie die anomalen Daten als mtz-Datei ein.
- Geben Sie die Proteinsequenz als Seq-, Pir- oder Txt-Datei ein. Eine Seq-Datei kann in einem Texteditor generiert werden (z. B. Notepad++9 unter Windows oder nano unter Linux). Erstellen Sie eine neue Datei, geben Sie die primäre Sequenz des Proteins als eine lange Linie oder durch Zeilenumbrüche getrennt ein. Speichern Sie die Datei mit der Dateierweiterung .seq.
- Geben Sie eine institutionelle E-Mail-Adresse ein.
- Führen Sie für Proteine ohne Homologiemodellvorlage das SAD-Protokoll von Auto-Rickshaw im erweiterten Modus aus. Geben Sie die erforderlichen Parameter ein.
- Die Ergebnisse werden über einen Web-Link an die angegebene E-Mail-Adresse übermittelt.
HINWEIS: AutoRickshaw ist eine automatisierte Pipeline, die verschiedene Kristallographie-Softwarepakete aufruft, um eine Röntgenkristallstruktur32,33,45,46,47,48,49,50,51,52,53,54,55,56,57,58zulösen. Wenn der Auto-Rickshaw-Lauf die Struktur nicht löst, können andere Auto-Rickshaw-Einstellungen getestet werden. Die Strukturbestimmungsmethode kann in "sub_phassade" geändert werden, um Phaser59 anstelle von SHELXD32zu verwenden. Die Anzahl der erwarteten Unterstrukturen pro Monomer kann ebenfalls erhöht oder verringert werden. - Während der experimentellen Phasing der Kristallstruktur wird Auto-Rickshaw versuchen, schwere Atome in der Einheitszelle zu positionieren, wodurch eine Unterstruktur entsteht. Die gleichseitige Dreiecksanordnung von Jodatomen in I3C stellt eine effiziente Möglichkeit dar, den Unterbau zu validieren. Wenn Schritt 6.3 fehlschlägt, kann die Überprüfung der Unterstruktur bei der Fehlerbehebung bei der Lösung der Fehlerbehebung helfen.
- Laden Sie die Liste der schweren Atom-Sites von der Auto-Rickshaw-Ergebnisseite herunter. Es ist ein Hyperlink, der als "schwere Atomstandorte" bezeichnet wird. Dadurch wird eine Textdatei mit den schweren Atom-Sites heruntergeladen.
- Ändern Sie die Dateierweiterung der Datei von .txt in .pdb.
- Öffnen Sie die PDB-Datei in Coot60. Aktivieren Sie die Symmetrie, um andere schwere Atome benachbarter asymmetrischer Einheiten zu sehen.
- Messen Sie die Abstände zwischen den schweren Atomen, auch über asymmetrische Einheiten hinweg. I3C erscheint als gleichseitiges Dreieck mit einer Seitenlänge von 6 Angstroms. Das Vorhandensein eines Dreiecks mit diesen Dimensionen zeigt an, dass die Platzierungen dieser schweren Atome korrekt sind.
Ergebnisse
Die Integration von I3C in rMMS kann neue Bedingungen generieren, die das derivatisierte Kristallwachstum unterstützen
Die Wirksamkeit des gleichzeitigen rMMS-Screenings und der I3C-Derivatisierung wurde in zwei Proteinen nachgewiesen, in Dem Eiweißlysozym (HEWL, als lyophilisiertes Pulver gewonnen) und der vermeintlichen Orf11-Lysin-N-Terminal-Domäne (Orf11 NTD) aus Bakteriophagen P68. Jedes Protein wurde gegen PEG/ION HT unter vier verschiedenen Bedingungen gescreent, darunter: ungesättigt, gesät, mit I3C ungesät und mit I3C gesät (Abbildung 1). Bei beiden Proteinen erhöht die einzige Zugabe von I3C nicht die Anzahl der Bedingungen, die der Kristallisation förderlich sind. Im Fall von Orf11 NTD wurde nur eine geeignete Bedingung mit und ohne I3C identifiziert (Abbildung 1B). Als I3C den HEWL-Bildschirmen hinzugefügt wurde, wurde die Anzahl der Treffer von 31 auf 26 reduziert, was die zusätzlichen Komplexitäten der Kristallisation bei der Einführung von Phasing-Compounds hervorhebt (Abbildung 1A). In Übereinstimmung mit anderen Studien erhöhte das Hinzufügen von Saatgut zu kommerziellen spärlichen Matrix-Bildschirmen, um einen rMMS-Bildschirm zu erzeugen, die Anzahl der möglichen Kristallisationsbedingungen für beide Proteine signifikant, was zu einer 2,1- und 6-fachen Erhöhung für HEWL und Orf11 NTD bzw.6,61 (Abbildung 1) führte. Am wichtigsten ist, dass die gleichzeitige Zugabe von I3C und Saatgut die Anzahl der Treffer im Vergleich zu einem nicht gesäten Bildschirm erhöhte, was eine 2,3- bzw. 7-fache Erhöhung für HEWL bzw. Orf11 NTD zeigt. Viele der Kristalle von rMMS in Gegenwart von I3C zeigen eine ausgezeichnete Kristallmorphologie (Abbildung 2).
Seeding ermöglicht eine sorgfältige Kontrolle der Kristallzahl in I3C rMMS-Bildschirmen
In Mikrosaatierungsexperimenten kann die Anzahl der Samen, die in eine Kristallisationsstudie eingeführt werden, durch Verdünnung des Samenbestands gesteuert werden, was eine präzise Kontrolle der Keimbildung im Tropfen7,36ermöglicht. Dies ermöglicht oft die Bildung größerer Kristalle, da der Wettbewerb von Proteinmolekülen an Keimbildungsstätten eingeschränkt ist. Dieser Vorteil erstreckt sich auch auf die I3C-rMMS-Methode und wurde sowohl in HEWL als auch in Orf11 NTD erfolgreich nachgewiesen. Die Nachbildung eines Kristallisationszustandes, der vom I3C-rMMS-Bildschirm mit einem verdünnten Saatgutbestand identifiziert wurde, ergab weniger, aber größere Kristalle(Abbildung 3).
SAD Phasing kann verwendet werden, um die Strukturen aus Kristallen aus rMMS I3C Bildschirm abgeleitet zu lösen
Kristalle, die mit dem in Abbildung 3 dargestellten verdünnten Saatgut angebaut wurden, wurden verwendet, um die Struktur der Proteine mithilfe von SAD-Phasing mit Beugungsdaten aus einem einzigen Kristall zu lösen ( Abbildung4). Daten wurden über die australische Synchrotron MX1 Beamline62gesammelt. Detaillierte Details zur Datenerfassung und Strukturlösung werden an anderer Stelle beschrieben27.
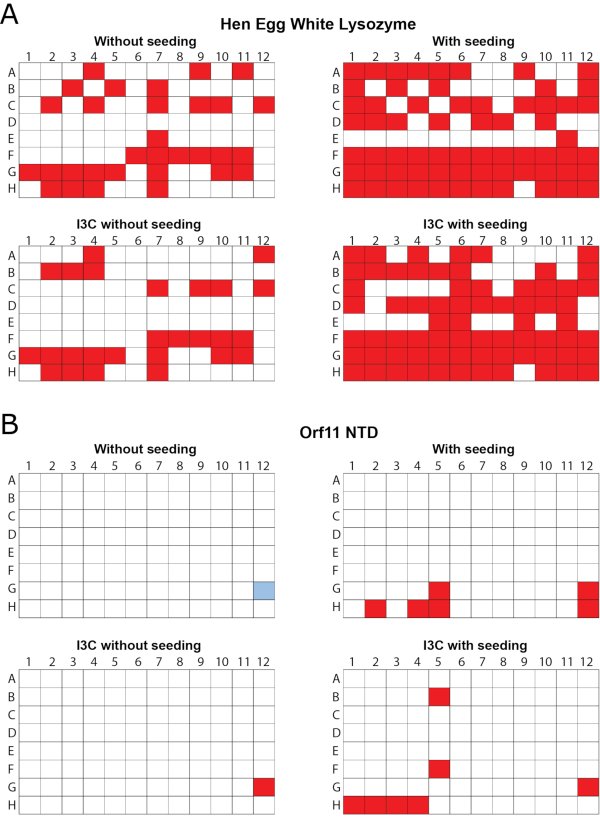
Abbildung 1: rMMS wurde verwendet, um neue Bedingungen für das Kristallwachstum in Gegenwart von I3C für zwei Testproteine zu erzeugen. 96 Well-Dampf-Diffusionskristallisationssiebe wurden mit kommerziellen spärlichen Matrix-Sieben durchgeführt. (A) Hen Eiweiß-Lysozym wurde mit dem Index HT-Bildschirm getestet. Die Schalen wurden mit HEWL-Kristallen aus 0,2 M Ammoniumtartrat-Dibasis-pH 7,0, 20 % (w/v) Polyethylenglykol 3350 gesät. (B) Orf11 NTD aus Bakteriophagen P68 wurde mit dem PEG/ION-Bildschirm getestet. Orf11 NTD-Trays wurden aus Kristallen aus dem Zustand G12 aus dem nicht ausgesäten Sieb gesät, blau dargestellt. Bedingungen, die das Kristallwachstum unterstützen, sind rot dargestellt. rMMS-Saat in Gegenwart und Abwesenheit von I3C gab sowohl deutlich mehr Kristalltreffer als ungesättigte Schalen. Figur angepasst von Truong et al.27. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
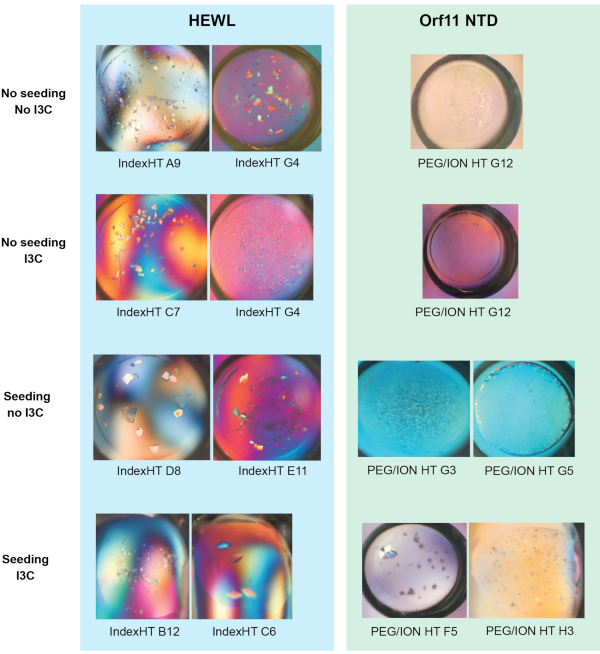
Abbildung 2 : Repräsentative Bilder von Kristallen, die aus den Dampfdiffusionsversuchen in Abbildung 1 a) und b) herangewachsen sind. Figur angepasst von Truong et al.27. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Die Verdünnung des Saatgutbestands ist ein effektiver Weg, um die Keimbildung in einem Kristallisationszustand zu reduzieren, der mit der I3C-rMMS-Methode gefunden wurde, um die Anzahl der Kristalle zu kontrollieren, die sich bilden. Die Reduzierung der Keimbildung innerhalb eines Tropfens führt oft dazu, dass Kristalle zu größeren Dimensionen wachsen. Figur angepasst von Truong et al.27. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4- Orf11 NTD (PDB ID 6O43) und HEWL (PDB ID 6PBB) wurden mit der I3C-rMMS-Methode kristallisiert und mit Auto-Rickshaw SAD-Phasing gelöst. (A) Bandstrukturen von HEWL und Orf11 NTD durch experimentelles Phasing gelöst. (B) I3C Molekül gebunden an HEWL und Orf11 NTD. (C) Anomale Jodatome in I3C sind in einem gleichseitigen Dreieck von 6 ° angeordnet. So weist das Vorhandensein dieses Dreiecks in der phasing Unterstruktur darauf hin, dass sich ein I3C-Molekül in dieser Position befindet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Strukturbestimmung eines neuartigen Proteins in Ermangelung eines geeigneten Homologiemodells für den molekularen Ersatz erfordert eine experimentelle Phasing. Diese Methoden erfordern die Einbindung schwerer Atome in den Proteinkristall, was der Strukturbestimmungspipeline eine zusätzliche Komplexität verleiht und zahlreiche Hindernisse mit sich bringen kann, die angegangen werden müssen. Schwere Atome können durch etikettierte Expression mit Selenomethionin und Selenocystein direkt in das Protein eingearbeitet werden. Da diese Methode kostspielig, mühsam ist und zu niedrigeren Proteinerträgen führen kann, wird markiertes Protein oft ausgedrückt, nachdem Kristallisationsbedingungen gefunden und mit nicht gekennzeichnetem Protein optimiert wurden. Alternativ können Kristalle durch Einweichen in eine Lösung mit schweren Atomen22,63,64absaugt werden. Diese Methode verwendet häufig hochwertige Kristalle und wird daher durchgeführt, nachdem bereits eine robuste Kristallisationsmethode entwickelt wurde. Die erfolgreiche Beschaffung eines derivatisierten Kristalls mit dieser Methode erfordert eine weitere Optimierung der Einweichverfahren und das Screening verschiedener Phasing-Verbindungen, wodurch einem bereits mühsamen Prozess mehr Zeit eingezäutt wird.
Die Kokristallisation des Proteins mit dem schweren Atom kann im Screening-Stadium durchgeführt werden, wodurch der Prozess effizient gestrafft und Kristallmanipulationsschritte reduziert werden, die Schäden verursachen können. Es gibt jedoch immer noch das mögliche Szenario, nur wenige anfängliche Kristallisationstreffer zu erhalten, und das Problem der Wahl einer kompatiblen schweren Atomverbindung. Viele derzeit verfügbare Phasing-Compounds sind inkompatibel mit Niederschlagsmitteln, Puffern und Additiven, die häufig unter Kristallisationsbedingungen zu finden sind. Sie können in Sulfat- und Phosphatpuffern unlöslich sein, zu Citrat und Acetat chelaten, ungünstig mit HEPES- und Tris-Puffern reagieren oder sich durch DTT und β-Mercaptoethanol21sequestrieren lassen. Da die Phasing-Verbindung I3C nicht unter diesen Inkompatibilitäten leidet, ist es eine robuste Phasing-Verbindung, die für viele verschiedene Bedingungen zugänglich sein könnte.
In dieser Studie wird eine schlanke Methode zur Herstellung von derivatisierten Kristallen vorgestellt, die für die SAD-Phasing durch gleichzeitige Kokristallisation der I3C-Phasing-Verbindung und rMMS bereit sind. Die Kombination beider Techniken erhöht die Anzahl der Kristallisationstreffer, wobei viele der Bedingungen eine verbesserte Morphologie und Beugungseigenschaften aufweisen. In orf11 NTD- und HEWL-Testfällen wurden neue Bedingungen im I3C-rMMS-Bildschirm identifiziert, die fehlten, als I3C nicht vorhanden war. Potenziell kann I3C günstig an das Protein binden, was die Bildung und Stabilisierung von Kristallkontakten erleichtert27. Dies wiederum kann Kristallisation induzieren und möglicherweise die Beugungseigenschaften verbessern. I3C ist nicht nur eine Verbindung, die mit spärlichen Matrix-Bildschirmen kompatibel ist, sondern ist aufgrund seiner intrinsischen Eigenschaften auch eine attraktive Phasing-Verbindung. Die funktionellen Gruppen, die sich mit Jod auf dem aromatischen Ringgerüst abwechseln, ermöglichen eine spezifische Bindung an Proteine. Dies führt zu einer höheren Belegung und reduziert möglicherweise das Hintergrundsignal23. Darüber hinaus ist die Anordnung anomaler Streuer in einem gleichseitigen Dreieck im Unterbau offensichtlich und kann verwendet werden, um die Bindung von I3C schnell zu validieren (Abbildung 4B und 4C). Schließlich kann es ein anomales Signal mit abstimmbarer Synchrotronstrahlung sowie Chrom- und Kupfer-rotierenden Anoden-Röntgenquellen erzeugen. Somit kann es auf viele verschiedene Workflows angewendet werden. Da I3C weit verbreitet und kostengünstig zu erwerben ist, ist dieser Ansatz für die meisten laboratorien für Strukturbiologie in Reichweite.
Es gibt mehrere experimentelle Überlegungen, die bei der Verwendung der I3C-rMMS-Methode berücksichtigt werden müssen. Diese Methode kann nicht angewendet werden, wenn das ursprüngliche kristalline Material des Proteins nicht gewonnen werden kann. In schwierigen Fällen kann kristallines Material aus einem homologen Protein auch zur Erzeugung von Saatgutverwendet werden. Dieser Cross-Seeding-Ansatz für rMMS hat einige vielversprechende Ergebnisse gezeigt7. Die Optimierung der Kristallzahl durch Verdünnung des Saatgutbestands ist ein entscheidender Schritt, der nicht übersehen werden sollte, um die Chance zu maximieren, hochwertige große Kristalle zu produzieren und geeignete Beugungsdaten zu erhalten. Wenn in der asymmetrischen Einheit nur wenige I3C-Standorte identifiziert werden, sollten die für die Kristallisation förderlichen Bedingungen mit einer erhöhten Konzentration von I3C weiter optimiert werden. Dies kann die Belegung von I3C erhöhen, um das anomale Signal zu maximieren und die Kristallableitung zu unterstützen.
Es kann Fälle geben, in denen diese Technik möglicherweise nicht die optimale Methode zur Ableitung von Proteinkristallen ist. Da die Größe eines Protein- oder Proteinkomplexes zunimmt, bietet die begrenzte Anzahl von I3C-Standorten auf der Proteinoberfläche möglicherweise nicht genügend Phasing Power, um die Struktur zu lösen. In diesen Szenarien, in denen die Proteingröße im Verdacht steht, die Phasing-Zeit zu behindern, kann die Selenomethionin-Kennzeichnung des Proteins ein praktikablerer Ansatz für die Phasing des Proteins sein. Wenn das Protein eine ausreichende Anzahl von Methioninrückständen im Protein aufweist (empfohlen mit mindestens einem Methionin pro 100 Rückstände65) und eine hocheffiziente Selenomethionin-Inkorporation in ein Protein erreicht werden kann (z. B. in bakteriellen Expressionssystemen66), werden mehrere Selenatome mit hoher Belegung in den Kristallen vorhanden sein, um die Struktur zu phasen.
Darüber hinaus können einige Proteine von Natur aus ungeeignet für die Ableitung mit I3C sein. I3C-Bindungsstellen an Proteinen sind von der Proteinstruktur abhängig. Es kann Proteine geben, die natürlich nur wenige exponierte Pflaster haben, die mit der I3C-Bindung kompatibel sind. Daher ist es nicht unvorhersehbar, dass es Schwierigkeiten bei der Kokristallisation einiger Zielproteine mit I3C geben kann.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Forschung wurde an der MX1-Beamline am Australian Synchrotron, einem Teil von ANSTO, durchgeführt. Die Autoren möchten die Mitglieder der Laboratorien Shearwin und Bruning für die Diskussionen über diese Arbeit würdigen. Die Autoren möchten auch Dr. Santosh Panjikar und Dr. Linda Whyatt-Shearwin würdigen, die zu der ursprünglichen Arbeit beigetragen haben, die dieses Protokoll als Pioniere war.
Folgende Finanzierung wird anerkannt: Australian Research Council (Grant No. DP150103009 und DP160101450 an Keith E. Shearwin); University of Adelaide (Australian Government Research Training Program Stipendium an Jia Quyen Truong und Stephanie Nguyen).
Materialien
| Name | Company | Catalog Number | Comments |
| 10 mL disposable luer lock syringes | Adelab Scientific | T3SS10LAT | Used for dispensing vacuum grease for hanging drop crystal tray wells |
| 24 well tissue culture plate | Sigma Aldrich | CLS3527 | Used for hanging drop crystal tray |
| 3 inch wide Crystal Clear Sealing Tape | Hampton Research | HR4-506 | For 96 well crystallization screens set up by robot |
| 5-amino-2,4,6-triiodoisophthalic acid | Alfa Aesar | B22178 | Commonly referred to as I3C in the article |
| Art Robbins Intelli-Plate 96-2 Original | Hampton Research | HR3-297 | For 96 well crystallization screens set up by robot |
| Coverslips | Thermo Fisher Scientific | 18X18-2 | Coverslips for hanging drop crystal tray wells |
| Dow Corning vacuum grease | Hampton Research | HR3-510 | Used for sealing hanging drop crystal tray wells |
| Eppendorf Pipette 0.1 μL-2.5 μL | Eppendorf | 3120000011 | |
| Gilson Pipette 2 μL-20 μL | John Morris Group | 1153247 | |
| Gilson Pipette 20 μL-200 μL | John Morris Group | 1152006 | |
| Glass pasteur pipettes | Adelab Scientific | HIR92601.01 | |
| Hen Egg White Lysozyme | Sigma-Aldrich | L6876 | Approximately 95% pure |
| IndexHT screen | Hampton Research | HR2-134 | |
| Microscope illuminator | Meiji Techno | FT192/230 | Light source to illuminate crystallography experiments |
| PEG/ION HT screen | Hampton Research | HR2-139 | |
| Phoenix Liquid Dispenser | Art Robbins Instruments | 602-0001-10 | |
| Scalpel with scalpel blade no. 15 | Adelab Scientific | LV-SMSCPO15 | |
| Seed bead kit | Hampton Research | HR2-320 | Kit contains a glass probe for crushing crystals. A PTFE seed bead, designed for crushing crystals, is also part of the kit but not used in this protocol. |
| Stereo microscope | Meiji Techno | EMZ-5TR | Microscope for visualising crystallography experiments |
| Tweezers | Sigma-Aldrich | T5415 | |
| Vortex mixer | Adelab Scientific | RAVM1 |
Referenzen
- Zheng, H., Hou, J., Zimmerman, M. D., Wlodawer, A., Minor, W. The future of crystallography in drug discovery. Expert Opinion on Drug Discovery. 9 (2), 125-137 (2014).
- Oakley, A. J., Wilce, M. C. J. Macromolecular crystallography as a tool for investigating drug, enzyme and receptor interactions. Clinical and Experimental Pharmacology and Physiology. 27 (3), 145-151 (2000).
- Jancarik, J., Kim, S. H. Sparse matrix sampling. A screening method for crystallization of proteins. Journal of Applied Crystallography. 24, 409-411 (1991).
- Newman, J., et al. Towards rationalization of crystallization screening for small- To medium-sized academic laboratories: The PACT/JCSG+ strategy. Acta Crystallographica Section D: Biological Crystallography. 61 (10), 1426-1431 (2005).
- Ireton, G. C., Stoddard, B. L. Microseed matrix screening to improve crystals of yeast cytosine deaminase. Acta Crystallographica Section D: Biological Crystallography. 60 (3), 601-605 (2004).
- D'Arcy, A., Villard, F., Marsh, M. An automated microseed matrix-screening method for protein crystallization. Acta Crystallographica Section D: Biological Crystallography. 63 (4), 550-554 (2007).
- D'Arcy, A., Bergfors, T., Cowan-Jacob, S. W., Marsh, M. Microseed matrix screening for optimization in protein crystallization: What have we learned. Acta Crystallographica Section:F Structural Biology Communications. 70 (9), 1117-1126 (2014).
- Taylor, G. The phase problem. Acta Crystallographica - Section D Biological Crystallography. 59 (11), 1881-1890 (2003).
- Rossmann, M. G. The molecular replacement method. Acta Crystallographica Section A. 46 (2), 73-82 (1990).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. Phaser crystallographic software. Journal of Applied Crystallography. 40 (4), 658-674 (2007).
- Millán, C., Jiménez, E., Schuster, A., Diederichs, K., Usón, I. ALIXE: a phase-combination tool for fragment-based molecular replacement. Acta Crystallographica Section D. 76 (3), 209-220 (2020).
- Liebschner, D., et al. Macromolecular structure determination using X-rays, neutrons and electrons: Recent developments in Phenix. Acta Crystallographica Section D: Structural Biology. 75, 861-877 (2019).
- Abergel, C. Molecular replacement: Tricks and treats. Acta Crystallographica Section D: Biological Crystallography. 69 (11), 2167-2173 (2013).
- Pröpper, K., et al. Structure solution of DNA-binding proteins and complexes with ARCIMBOLDO libraries. Acta Crystallographica Section D: Biological Crystallography. 70 (6), 1743-1757 (2014).
- Rodríguez, D. D., et al. Crystallographic ab initio protein structure solution below atomic resolution. Nature Methods. 6 (9), 651-653 (2009).
- Bibby, J., Keegan, R. M., Mayans, O., Winn, M. D., Rigden, D. J. AMPLE: A cluster-and-truncate approach to solve the crystal structures of small proteins using rapidly computed ab initio models. Acta Crystallographica Section D: Biological Crystallography. 68 (12), 1622-1631 (2012).
- Green, D. W., Ingram, V. M., Perutz, M. F. The structure of haemoglobin - IV. Sign determination by the isomorphous replacement method. Proceedings of the Royal Society of London. Series A. Mathematical and Physical Sciences. 225 (1162), 287-307 (1954).
- Blow, D. M., Rossmann, M. G. The single isomorphous replacement method. Acta Crystallographica. 14 (11), 1195-1202 (1961).
- Wang, B. C. Resolution of phase ambiguity in macromolecular crystallography. Methods in Enzymology. 115, 90-112 (1985).
- Hendrickson, W. A. Determination of macromolecular structures from anomalous diffraction of synchrotron radiation. Science. 254 (5028), 51-58 (1991).
- Pike, A. C. W., Garman, E. F., Krojer, T., Von Delft, F., Carpenter, E. P. An overview of heavy-atom derivatization of protein crystals. Acta Crystallographica Section D: Structural Biology. 72 (3), 303-318 (2016).
- Dauter, Z., Dauter, M., Rajashankar, K. R. Novel approach to phasing proteins: Derivatization by short cryo-soaking with halides. Acta Crystallographica Section D: Biological Crystallography. 56 (2), 232-237 (2000).
- Beck, T., Krasauskas, A., Gruene, T., Sheldrick, G. M. A magic triangle for experimental phasing of macromolecules. Acta Crystallographica Section D: Biological Crystallography. 64 (11), 1179-1182 (2008).
- Beck, T., Gruene, T., Sheldrick, G. M. The magic triangle goes MAD: Experimental phasing with a bromine derivative. Acta Crystallographica Section D: Biological Crystallography. 66 (4), 374-380 (2010).
- Beck, T., Da Cunha, C. E., Sheldrick, G. M. How to get the magic triangle and the MAD triangle into your protein crystal. Acta Crystallographica Section F: Structural Biology and Crystallization Communications. 65 (10), 1068-1070 (2009).
- Berman, H. M., et al. The Protein Data Bank. Nucleic Acids Research. , (2000).
- Truong, J. Q., Panjikar, S., Shearwin-Whyatt, L., Bruning, J. B., Shearwin, K. E. Combining random microseed matrix screening and the magic triangle for the efficient structure solution of a potential lysin from bacteriophage P68. Acta Crystallographica Section D: Structural Biology. 75 (7), 670-681 (2019).
- Terwilliger, T. C., et al. Decision-making in structure solution using Bayesian estimates of map quality: The PHENIX AutoSol wizard. Acta Crystallographica Section D: Biological Crystallography. 65 (6), 582-601 (2009).
- Holton, J., Alber, T. Automated protein crystal structure determination using ELVES. Proceedings of the National Academy of Sciences of the United States of America. 101 (6), 1537-1542 (2004).
- Skubák, P., Pannu, N. S. Automatic protein structure solution from weak X-ray data. Nature Communications. 4, (2013).
- Sheldrick, G. M. Crystal structure refinement with SHELXL. Acta Crystallographica Section C: Structural Chemistry. 71, 3-8 (2015).
- Schneider, T. R., Sheldrick, G. M. Substructure solution with SHELXD. Acta Crystallographica Section D: Biological Crystallography. 58 (10), 1772-1779 (2002).
- Sheldrick, G. M. Macromolecular phasing with SHELXE. Zeitschrift fur Kristallographie. 217 (12), 644-650 (2002).
- Benvenuti, M., Mangani, S. Crystallization of soluble proteins in vapor diffusion for x-ray crystallography. Nature Protocols. , (2007).
- Beck, T. . Sticky triangles New tools for experimental phasing of biological macromolecules. , (2010).
- Luft, J. R., DeTitta, G. T. A method to produce microseed stock for use in the crystallization of biological macromolecules. Acta Crystallographica Section D: Biological Crystallography. 55 (5), 988-993 (1999).
- Teng, T. -. Y. Mounting of crystals for macromolecular crystallography in a free-standing thin film. Journal of Applied Crystallography. 23 (5), 387-391 (1990).
- Garman, E. F., Mitchell, E. P. Glycerol concentrations required for cryoprotection of 50 typical protein crystallization solutions. Journal of Applied Crystallography. 29, 584-587 (1996).
- Garman, E. F., Weik, M. X-ray radiation damage to biological samples: recent progress. Journal of Synchrotron Radiation. 26 (4), 907-911 (2019).
- Garcia-Bonete, M. J., Katona, G. Bayesian machine learning improves single-wavelength anomalous diffraction phasing. Acta Crystallographica Section A: Foundations and Advances. 75, 851-860 (2019).
- Kabsch, W. XDS. Acta Crystallographica Section D Biological Crystallography. 66 (2), 125-132 (2010).
- Evans, P. Scaling and assessment of data quality. Acta Crystallographica Section D: Biological Crystallography. 62 (1), 72-82 (2006).
- Evans, P. R., Murshudov, G. N. How good are my data and what is the resolution. Acta Crystallographica Section D: Biological Crystallography. 69 (7), 1204-1214 (2013).
- Panjikar, S., Parthasarathy, V., Lamzin, V. S., Weiss, M. S., Tucker, P. A. Auto-Rickshaw: An automated crystal structure determination platform as an efficient tool for the validation of an X-ray diffraction experiment. Acta Crystallographica Section D: Biological Crystallography. 61 (4), 449-457 (2005).
- Jones, T. A., Thirup, S. Using known substructures in protein model building and crystallography. The EMBO journal. 5 (4), 819-822 (1986).
- Kleywegt, G. J., Jones, T. A. Template convolution to enhance or detect structural features in macromolecular electron-density maps. Acta Crystallographica Section D: Biological Crystallography. 53 (2), 179-185 (1997).
- Perrakis, A., Morris, R., Lamzin, V. S. Automated protein model building combined with iterative structure refinement. Nature Structural Biology. 6 (5), 458-463 (1999).
- Morris, R. J., et al. Breaking good resolutions with ARP/wARP. Journal of Synchrotron Radiation. 11 (1), 56-59 (2004).
- Yao, D. Q., et al. SAD phasing by OASIS-2004: Case studies of dual-space fragment extension. Acta Crystallographica Section D: Biological Crystallography. 62 (8), 883-890 (2006).
- Hao, Q. ABS: A program to determine absolute configuration and evaluate anomalous scatterer substructure. Journal of Applied Crystallography. 37 (3), 498-499 (2004).
- Collaborative Computational Project Number 4. The CCP4 suite: Programs for protein crystallography. Acta Crystallographica Section D: Biological Crystallography. 50 (5), 760-763 (1994).
- Sheldrick, G. M., Hauptman, H. A., Weeks, C. M., Miller, R., Usón, I. Ab initio phasing. International Tables for Crystallography. , 333-345 (2006).
- Smith, G. D. Matching selenium-atom peak positions with a different hand or origin. Journal of Applied Crystallography. 35 (3), 368-370 (2002).
- Pannu, N. S., McCoy, A. J., Read, R. J. Application of the complex multivariate normal distribution to crystallographic methods with insights into multiple isomorphous replacement phasing. Acta Crystallographica - Section D Biological Crystallography. 59 (10), 1801-1808 (2003).
- Pannu, N. S., Read, R. J. The application of multivariate statistical techniques improves single-wavelength anomalous diffraction phasing. Acta Crystallographica Section D: Biological Crystallography. 60 (1), 22-27 (2004).
- De La Fortelle, E., Bricogne, G. Maximum-likelihood heavy-atom parameter refinement for multiple isomorphous replacement and multiwavelength anomalous diffraction methods. Methods in Enzymology. 276, 472-494 (1997).
- Cowtan, K. Joint CCP4 and ESF-EACBM Newsletter on Protein. Crystallography. 31, 34-38 (1994).
- Terwilliger, T. C. Maximum-likelihood density modification. Acta Crystallographica Section D: Biological Crystallography. 56 (8), 965-972 (2000).
- Read, R. J., McCoy, A. J. Maximum-likelihood determination of anomalous substructures. Acta Crystallographica Section D: Structural Biology. , (2018).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallographica Section D: Biological Crystallography. 66 (4), 486-501 (2010).
- Till, M., et al. Improving the success rate of protein crystallization by random microseed matrix screening. Journal of visualized experiments JoVE. (78), e50548 (2013).
- McPhillips, T. M., et al. Blu-Ice and the distributed control system: Software for data acquisition and instrument control at macromolecular crystallography beamlines. Journal of Synchrotron Radiation. 9 (6), 401-406 (2002).
- Nagem, R. A. P., Polikarpov, I., Dauter, Z. Phasing on Rapidly Soaked Ions. Methods in Enzymology. 374, 120-137 (2003).
- Taylor, G. L. Introduction to phasing. Acta Crystallographica Section D: Biological Crystallography. 66 (4), 325-338 (2010).
- Hendrickson, W. A., Ogata, C. M. Phase determination from multiwavelength anomalous diffraction measurements. Methods in Enzymology. 276, 494-523 (1997).
- Doublié, S. Production of Selenomethionyl Proteins in Prokaryotic and Eukaryotic Expression Systems. Macromolecular Crystallography Protocols. Methods in Molecular Biology. , 91-108 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten