Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verwendung der Next-Generation-Sequenzierung zur Identifizierung von Mutationen, die mit der Reparatur eines CAS9-induzierten Doppelstrangbruchs in der Nähe des CD4-Promotors verbunden sind
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier wird das sgRNA/CAS9-Endonuklease- und Next-Generation-Sequenzierungsprotokoll vorgestellt, das verwendet werden kann, um die Mutationen zu identifizieren, die mit der Reparatur von Doppelstrangbrüchen in der Nähe des CD4-Promotors verbunden sind.
Zusammenfassung
Doppelstrangbrüche (DSBs) in der DNA sind die zytotoxischste Art von DNA-Schäden. Da eine Vielzahl von Beleidigungen zu diesen Läsionen führen können (z. B. Replikationsstress, ionisierende Strahlung, nicht reparierte UV-Schäden), treten DSBs jeden Tag in den meisten Zellen auf. Zusätzlich zum Zelltod reduzieren nicht reparierte DSBs die Genomintegrität und die daraus resultierenden Mutationen können die Tumorgenese vorantreiben. Diese Risiken und die Prävalenz von DSBs motivieren zu Untersuchungen der Mechanismen, mit denen Zellen diese Läsionen reparieren. Die Sequenzierung der nächsten Generation kann mit der Induktion von DSBs durch ionisierende Strahlung gepaart werden, um ein leistungsfähiges Werkzeug zur genauen Definition der Mutationen bereitzustellen, die mit DSB-Reparaturdefekten verbunden sind. Dieser Ansatz erfordert jedoch eine rechenintensive und kostenintensive Sequenzierung des gesamten Genoms, um die Reparatur der zufällig auftretenden DSBs im Zusammenhang mit ionisierender Strahlung nachzuweisen. Seltene Schneideendonukleasen wie I-Sce1 bieten die Möglichkeit, ein einzelnes DSB zu erzeugen, aber ihre Erkennungsstellen müssen in das Genom von Interesse eingefügt werden. Infolgedessen ist der Ort der Reparatur von Natur aus künstlich. Jüngste Fortschritte ermöglichen es der Guide-RNA (sgRNA), eine Cas9-Endonuklease an jeden Genomort von Interesse zu leiten. Dies könnte auf die Untersuchung der DSB-Reparatur angewendet werden, die die Sequenzierung der nächsten Generation kostengünstiger macht, indem sie sich auf die DNA konzentriert, die das Cas9-induzierte DSB flankiert. Ziel des Manuskripts ist es, die Machbarkeit dieses Ansatzes zu demonstrieren, indem ein Protokoll vorgestellt wird, das Mutationen definieren kann, die aus der Reparatur eines DSB vor dem CD4-Gen stammen. Das Protokoll kann angepasst werden, um Veränderungen des mutagenen Potenzials von DSB zu bestimmen, die mit exogenen Faktoren wie Reparaturinhibitoren, viraler Proteinexpression, Mutationen und Umweltexpositionen mit relativ begrenzten Rechenanforderungen verbunden sind. Sobald das Genom eines Organismus sequenziert wurde, kann diese Methode theoretisch an jedem genomischen Ort und in jedem Zellkulturmodell dieses Organismus, der transfiziert werden kann, angewendet werden. Ähnliche Anpassungen des Ansatzes könnten Vergleiche der Reparaturtreue zwischen verschiedenen Loci im selben genetischen Hintergrund ermöglichen.
Einleitung
Die Aufrechterhaltung der genomischen Stabilität ist für alle lebenden Organismen von entscheidender Bedeutung. Eine genaue DNA-Replikation und eine robuste DNA-Schadensantwort (DDR) sind notwendig, um das genetische Material originalgetreu zu verbreiten 1,2. DNA-Schäden treten in den meisten Zellen regelmäßigauf 2,3. Wenn diese Schäden erkannt werden, wird der Zellzyklusverlauf gestoppt und DNA-Reparaturmechanismen aktiviert. Doppelstrangbrüche in DNA oder DSBs sind die giftigste und mutagenste Art von DNA-Schäden 3,4.
Während mehrere DDR-Signalwege diese Läsionen reparieren können, sind die am gründlichsten untersuchten DSB-Reparaturwege homologe Rekombination (HR) und nicht-homologe Endverbindung (NHEJ). HR ist ein weitgehend fehlerfreier Weg, der eine DSB mit einer Schwesterchromatide als homologe Vorlage repariert. Dies geschieht tendenziell in der S- und G2-Phase eines Zellzyklus 5,6,7. NHEJ ist fehleranfälliger, kann aber während des gesamten Zellzyklusauftreten 8,9. Verschiedene Reporter-Assays wurden entwickelt, um die Effizienz spezifischer Reparaturmechanismen zu messen10,11,12. Diese Assays stützen sich in der Regel auf die Durchflusszytometrie für eine Hochdurchsatzmessung der DSB-Reparaturpfadaktivität unter Verwendung von GFP oder mCherry als Auslesung11,13. Obwohl sie sehr effizient sind, verlassen sie sich auf eine kanonische Reparatur, die bei einem künstlich eingeführten DSB auftritt.
Es gibt eine Vielzahl anderer Methoden, die verwendet werden, um die DSB-Reparatur zu untersuchen. Viele davon beruhen auf Immunfluoreszenzmikroskopie (IF) 1,14. Die IF Mikroskopie weist diskrete Kernherde nach, die für Reparaturkomplexe repräsentativ sind, nachdem DSBs durch Exposition gegenüber genotoxischen Chemikalien oder ionisierender Strahlung induziert wurden15,16. Die Verfolgung der Bildung und Auflösung dieser Herde liefert einen Hinweis auf die Einleitung bzw. den Abschluss der Reparatur14,17. Diese Methoden der DSB-Induktion (d.h. Chemikalien oder ionisierende Strahlung) verursachen jedoch keine DSBs an definierten Stellen im Genom. Es ist auch funktional unmöglich, sie zu verwenden, um konsistent nur eine kleine Anzahl (z. B. 2-4) von DSBs zu induzieren. Infolgedessen verursachen die am häufigsten verwendeten Methoden zur Induktion von DSBs eine Vielzahl von Läsionen, die zufällig im gesamten Genom verteiltsind 18. Eine kleine Anzahl von DSBs kann eingeführt werden, indem die Erkennungsstelle für eine Endonuklease mit seltenem Schneiden eingefügt und die entsprechende Endonuklease wie I-Sce119 ausgedrückt wird. Leider verhindert die erforderliche Integration eines Zielortes die Untersuchung von DSB an endogenen genomischen Loci.
Dieses Manuskript beschreibt eine Methode zur Erkennung von Mutationen, die mit der Reparatur eines an einem benutzerdefinierten Ort generierten DSB verbunden sind. Wir liefern ein repräsentatives Beispiel für den Ansatz, der angewendet wird, um die Fähigkeit eines viralen Proteins zu bewerten, die Anzahl der mit einem DSB assoziierten Mutationen zu erhöhen. Insbesondere beschreibt dieses Manuskript die Verwendung einer Single-Guide-RNA (sgRNA), um eine CAS9-Endonuklease anzuordnen, um einen DSB bei menschlichem CD4-offenem Leserahmen in menschlichen Vorhautkeratinozyten zu induzieren, die Vektorkontrolle (HFK LXSN) exprimieren, und HFK, das das E6-Protein des humanen Papillomavirus Typ 8 (HFK 8E6) exprimiert. Gezielte Next-Generation-Sequenzierung (NGS) der Region, die den Bruch umgibt, ermöglicht es, Mutationen, die mit der Reparatur der Läsion verbunden sind, streng zu definieren. Diese Daten zeigen, dass das virale Protein während der DSB-Reparatur einen etwa 20-fachen Anstieg der Mutationen verursacht. Es bietet auch eine unvoreingenommene Charakterisierung der mutagenen Folgen von DSBs an einem einzigen Ort, ohne dass eine Sequenzierung des gesamten Genoms erforderlich ist. Prinzipiell könnte das Protokoll leicht angepasst werden, um das relative Risiko von Mutationen zwischen Genomloci oder Zelllinien zu vergleichen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Zellenbeschichtung
- Züchten Sie HFK LXSN- und HFK 8E6-Zellen in 10-cm-Platten in Keratinozytenkulturmedien (10 ml / Platte) mit humanem Keratinozyten-Wachstumspräparat (HKGS) und 1% Penicillin / Streptomycin. Züchten Sie Zellen zu etwa 80% Konfluenz bei 37 °C in einem Jacket-Inkubator mit 5% CO2.
- Ersetzen Sie Kulturmedien durch 3 ml Trypsin-EDTA (0,05%, Ethylendiamintetraessigsäure). 3 min bei 37 °C inkubieren. Neutralisieren Sie Trypsin mit gleichem Volumen des mit fetalem Rinderserum (FBS) ergänzten Mediums und übertragen Sie die Zellen auf ein 15-ml-Zentrifugenröhrchen. Zentrifuge bei 300 x g für 5 min.
- Resuspendiert Zellen mit 10 ml Keratinozytenkulturmedien mit HKGS. Bestimmen Sie die Konzentration der Zellen mit einem Hämozytometer.
- Platte 4 x 105 Zellen/6-cm-Platten (Samen Sie zwei Platten für HFK LXSN und zwei Platten für HFK 8E6) in 4 ml Keratinozytenkulturmedien mit HKGS und 1% Penicillin und Streptomycin. Bei 37 °C in einem Jacken-Inkubator mit 5% CO2 wachsen.
HINWEIS: Die Analyse von HFK-Zellen wurde für dieses Protokoll aus zwei Gründen ausgewählt. Erstens ist HFK eine schwer zu transfizierende Zelllinie. Durch den Nachweis, dass das Protokoll in dieser Zelllinie funktioniert, wird daher der Nachweis erbracht, dass es wahrscheinlich in häufiger verwendeten und leichter transfizierten Zellen funktioniert. Zweitens zeigen bereits veröffentlichte Daten, dass ein virales Protein (8E6) die Reparatur von Doppelstrangbrüchen in DNA20,21,22 behindert. Der Vergleich von HFK LXSN und HFK 8E6 ermöglicht es uns daher, die Fähigkeit des Assays zu demonstrieren, eine Zunahme von Mutationen im Zusammenhang mit einer Verringerung der zellulären Reparaturkapazität zu erkennen.
2. Transfektion
- Ersetzen Sie am Tag der Transfektion (24 h nach der Beschichtung) die Medien durch 3 ml antibiotikafreie Ergänzungsmedien. Inkubieren Sie für 2 h bei 37 °C in einem Jackeninkubator.
- Transfekte Zellen mit geeigneten lipidbasierten Transfektionsreagenzien gemäß den Anweisungen des Herstellers.
- Transfektionsreagenzien auf Raumtemperatur erwärmen und vor Gebrauch vorsichtig pipettieren.
- Geben Sie für jede Zelllinie (HFK LXSN und HFK 8E6) eine geeignete Menge Transfektionspuffer (gemäß Anweisung des Herstellers) in ein steriles 1,5-ml-Zentrifugenröhrchen (Tube 1). Fügen Sie eine weitere Röhre mit der gleichen Menge an Transfektionspuffer (Scheintransfektion oder Röhre 2) hinzu.
- 2 μg Plasmid-DNA, die CAS9/sgRNA exprimiert, die auf menschliches CD4 abzielt (px330-CD4, 5'- GGCGTATCTGTGTGAGGACT), in Tube 1 aus Schritt 2.2.2 geben. Pipette vorsichtig zum vollständigen Mischen auftischen. Fügen Sie Tube 2 die gleiche Menge steriles Wasser hinzu.
- Fügen Sie für jede Zelllinie eine Kontrollplatte mit Transfektionsreagenzien allein (kein Plasmid) hinzu.
HINWEIS: Die zweite Platte dient als Negativkontrolle im Experiment, so dass der Benutzer bestätigen kann, dass die Transfektion mit der CAS9 / sgRNA nicht für Mutationen verantwortlich ist. - Dem Röhrchen mit DNA-Gemisch (Tube 1) aus Schritt 2.2.3 und der simulierten Transfektion (Tube 2) aus Schritt 2.2.2 ist eine angemessene Menge des Transfektionsreagenzes (nach Anweisung des Herstellers) zugesetzt. Pipette vorsichtig zum vollständigen Mischen auftischen. Inkubieren Sie bei Raumtemperatur für 15-30 Minuten, um genügend Zeit für die Bildung von Komplexen zu haben.
- Die Transfektionsmischung tropfenweise auf die Platte geben. Schaukeln Sie die Kultur vorsichtig für 1 Minute, um die Transfektionsmischung gleichmäßig zu verteilen.
- Nach der Transfektion 48 h inkubieren, um die CAS9-Expression zu ermöglichen.
- Zellen durch Trypsinisierung ernten.
- Ersetzen Sie Kulturmedien durch 1 ml Trypsin-EDTA (0,05%, Ethylendiamintetraessigsäure). 3 min bei 37 °C inkubieren. Neutralisieren Sie Trypsin mit gleichem Volumen an FBS-ergänzten Medien.
- Übertragen Sie die Zellsuspension für jede Zellplatte auf zwei Mikrozentrifugenröhrchen mit gleichen Aliquoten. Zentrifuge bei 300 x g für 5 min.
- Resuspendiert das Zellpellet aus einem Schlauch in Schritt 2.4.2 in 1 ml phosphatgepufferter Kochsalzlösung (PBS) zur Sequenzierung. Resuspendieren Sie die andere Röhre aus Schritt 2.4.2 mit eiskaltem PBS für Immunoblot.
- Ernten Sie die Ganzzelllysate für Immunoblot.
- Zentrieren Sie das Röhrchen bei 300 x g für 5 min. Verwerfen Sie den Überstand.
- 100 μL Radioimmunpräzipitations-Assay-Puffer (RIPA-Lysepuffer) gemischt mit 1% Protease-Inhibitor und 1% Phosphatase-Inhibitor in die Tube geben, gründlich mit einer Pipette mischen und 10 min auf Eis inkubieren.
HINWEIS: RIPA-Lysepuffer enthält 10 mM Tris-HCl, pH 8,0; 1 mM EDTA; 0,5 mM EGTA; 1% Triton X-100; 0,1% Natriumdesoxycholat; 0,1% SDB; 140 mM NaCl und deionisiertes Wasser. - Zentrifugenlysate bei 13.000 x g für 10 min. Sammeln Sie Überstände für Immunoblot.
3. Messung der CAS9-Expression über Immunoblot
- Bestimmen Sie die Proteinkonzentration mit einem Bicchinchinsäure (BCA) Assay gemäß den Anweisungen des Herstellers.
- Lassen Sie 20 μg Protein jeder Probe in den Vertiefungen eines 3%-8% Tris-Acetat-Gels für 150 min laufen und halbtrockenen Transfer (10 V für 30 min und dann 25 V für 12 min) auf die Polyvinylidendifluoridmembran.
- Nachdem Sie die Membran in 5% fettfreier Trockenmilch in PBS mit 0,1% Tween (PBST) für 1 h bei Raumtemperatur blockiert haben, fügen Sie Anti-CAS9 (1:1000) und Anti-GAPDH (1:1000) Antikörper hinzu. Über Nacht bei 4 °C inkubieren.
- Nach dem Waschen der Membran mit PBST die Membran mit sekundärem Antikörper in 5% fettfreier Trockenmilch in PBST für 1 h bei Raumtemperatur inkubieren.
- Stellen Sie den Blot ab und bestimmen Sie den CAS9-Spiegeldurch Densitometrie 23. Siehe Abbildung 1 für einen repräsentativen Blot.
HINWEIS: Der Nachweis der phosphorylierten H2AX (S139) -Herkbildung durch Immunfluoreszenzmikroskopie kann zur Validierung der CAS9-Aktivität14 verwendet werden. Eine geringe Anzahl unterschiedlicher Herde (typischerweise 1-4 Herde) wird erwartet, abhängig von der Zellzyklusposition, ob Mutationen in der CAS9-Zielstelle ein weiteres Schneiden verhindern und wie viele Kopien der CAS9-Schnittstelle im Genom von Interesse vorhanden sind. Ein repräsentatives Bild ist in Abbildung 2 dargestellt.
4. Nukleinsäureextraktion und Amplikonerzeugung
- Extrahieren Sie DNA aus Zellproben aus Schritt 2.4.3 mit einem hochmolekularen DNA-Extraktionskit, wie vom Hersteller angegeben.
- Resuspendieren Sie Primer mit dem angegebenen Lösungsmittel gemäß dem Datenblatt. Mit dem gleichen Reagenz auf 20 μM verdünnen und 20 μM Primer in die angegebenen Pools legen.
HINWEIS: Der Primer-Pool ist in der Ergänzenden Tabelle 1 aufgeführt. - Erstellen Sie ein PCR-Master-Mix mit einer Taq-Polymerase mit langer Amplifikation für jeden 20-μM-Primerpool, wie in Tabelle 1 angegeben.
- Geben Sie 21 μL der Mastermixe zu separaten PCR-Röhrchen.
- 4 μL der Zielprobe (100 ng/μL) aus Schritt 4.1 in PCR-Assay-Röhrchen mit Mastermix- und Cap-Assay-Röhrchen geben. Stellen Sie für jeden Primerpool separate Reaktionen sicher.
- Vortex zum Mischen von PCR-Assay-Röhrchen und Zentrifuge (Quick Spin), um Tröpfchen von Röhrchendeckeln zu entfernen.
- Legen Sie die PCR-Röhrchen auf eine herkömmliche Thermocycler-Maschine.
- Programmieren Sie PCR-Maschine wie in Tabelle 2 angegeben.
- Führen Sie das Programm auf einem Thermocycler aus.
5. PCR-Bereinigung
- Entfernen Sie Primer aus PCR-Reaktionen mit einem perlenbasierten PCR-Reinigungssystem.
- Entfernen Sie die Reinigungsperlen 30 Minuten vor Gebrauch aus dem Kühlschrank.
- Wirbelperlen lange vor Gebrauch und stellen Sie sicher, dass alle Perlen wieder aufgehängt sind.
- Fügen Sie 30 μL (1,2x) resuspendierte Perlen zu jeder Vertiefung einer Tiefbrunnenplatte mit 96 Vertiefungen hinzu.
- Fügen Sie 25 μL PCR-Reaktion zu Wells hinzu, die Perlen enthalten.
- Legen Sie die Platte bei 2000 U / min für 2 min auf einen Plattenschüttler.
- Lassen Sie die Platte nach dem Schütteln 5 Minuten bei Raumtemperatur bleiben.
- Legen Sie die Tieflochplatte auf einen 96-Well-Plattenmagneten und inkubieren Sie für 2 min.
- Entfernen und entsorgen Sie den Überstand ohne die störenden Perlen.
- Während die Platte auf dem Magneten verbleibt, fügen Sie 180 μL 80% Ethanol hinzu und inkubieren Sie für 30 s. Entfernen und verwerfen Sie den Überstand.
- Wiederholen Sie Schritt 5.1.9.
- Entfernen und entsorgen Sie mit einer 10-μL-Pipette die verbleibende Flüssigkeit aus den Vertiefungen.
- Perlen bei Raumtemperatur 10 min trocknen lassen.
- Geben Sie 20 μL nukleasefreies Wasser in die Brunnen, die Perlen enthalten, und entfernen Sie die Platte vom Magneten.
- Schütteln Sie die Platte bei 2000 U / min für 2 min bei Raumtemperatur.
- Bebrüten Sie die Platte bei Raumtemperatur für 5 min.
- Legen Sie die Platte auf einen Magnetständer und inkubieren Sie sie für 2 min bei Raumtemperatur.
- Entfernen Sie den Überstand in eine zweite, beschriftete PCR-Platte. Diese enthält die gereinigte DNA.
- Messen Sie die Konzentration jeder Reaktion mit einem Fluorometer.
- Stellen Sie sicher, dass dsDNA-Fluorometer-Reagenzien Raumtemperatur haben.
- Richten Sie fluorometrische Assay-Röhren sowie zwei zusätzliche Röhren für Standards ein.
- Fügen Sie 199 μL 1x dsDNA-Arbeitslösung zu allen bis auf zwei Röhrchen hinzu. 190 μL der Arbeitslösung zu den letzten beiden Röhrchen geben.
- Fügen Sie 10 μL der beiden Standards (in der Materialtabelle enthalten) hinzu, um die Assay-Röhrchen zu trennen.
- Fügen Sie 1 μL jeder PCR-Reaktion zu Fluorometer-Mastermix-Röhrchen hinzu.
- Wirbelrohre zum Mischen und Inkubieren bei Raumtemperatur für 2 min.
- Wählen Sie auf dem Startbildschirm des Fluorometers die Schaltfläche mit dem Assay-Kitin (1x dsDNA) und dann Read Standards und Run Samples aus.
- Setzen Sie Standard 1 Tube ein, wählen Sie die Schaltfläche Lesen und wiederholen Sie dann den Vorgang für Standard 2.
- Nach Schritt 5.2.6 wiederholen Sie den Vorgang für eine Probe, wählen Sie ein Probenvolumen von 1 μL aus, und die resultierende Konzentration wird angegeben.
- Wiederholen Sie Schritt 5.2.7 für die verbleibenden Stichproben.
- Berechnen Sie die projizierte Molarität aller Reaktionen und die gleichen Konzentrationen von Reaktionen aus jeder einzelnen Probe separat (ein endgültiger Pool pro Probe), indem Sie die folgende Gleichung verwenden.
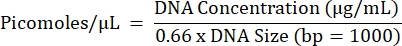
- Wiederholen Sie die Schritte 5.2.1 bis 5.2.7, um die endgültige Poolkonzentration zu erhalten.
- Überprüfen Sie den Amplicon-Pool auf einer Kapillarelektrophoresemaschine / Agarosegel, wie vom Hersteller angegeben.
- Bereiten Sie Kapillarelektrophoreseröhrchen für die entsprechende Anzahl von Proben vor. Fügen Sie 7 μL des DNA-Puffers hinzu, wie vom Hersteller angegeben.
- Geben Sie 4 μL des Amplikonpools in die Röhre, die den DNA-Puffer enthält.
- Legen Sie die Röhrchen auf die Elektrophoresemaschine und führen Sie die Maschine gemäß den Angaben des Herstellers für dsDNA aus.
- Sehen Sie sich die Elektrophorese-Gelbilder an, um sicherzustellen, dass die Bänder auf ~ 5 kb (Größe der Amplikonen) lokalisiert werden.
- Berechnen Sie die projizierte Molarität aller Reaktionen und die gleichen Konzentrationen von Reaktionen aus jeder einzelnen Probe separat (ein endgültiger Pool pro Probe), indem Sie die folgende Gleichung verwenden.
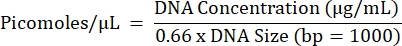
6. Bibliotheksvorbereitung
- Verdünnen Sie Probenpools von Schritt 5.3.1 auf 0,2 ng/μL für die Bibliotheksvorbereitung.
- Bereiten Sie Bibliotheken mithilfe eines Bibliotheksvorbereitungskits mit geringem Input, das mit kurzen Sequenzen (300 bp) kompatibel ist, mithilfe eindeutiger Indexkombinationen für jeden in Schritt 5.3 erstellten Beispielpool gemäß den Anweisungen des Herstellers vor.
HINWEIS: Befolgen Sie die Anweisungen des Herstellers, um Indexsequenzen auszuwählen. Alle Indizes, die für das Bibliotheksvorbereitungskit zugänglich sind, funktionieren für die Beispiele. - Nach der Bibliotheksvorbereitung alle Proben nach den Anweisungen des Herstellers zusammenführen.
HINWEIS: Berechnen Sie vor dem Erstellen des Bibliothekspools die Anzahl der Lesevorgänge, die für die 250-fache Abdeckung Ihrer Zielsequenz erforderlich sind, und stellen Sie sicher, dass die ausgewählte Sequenzierungskassette eine ausreichende Abdeckung für jede enthaltene Probe bieten kann. Bei insgesamt 0,5 MB entspricht dies 1 Mio. Lesevorgängen.
- Bereiten Sie Bibliotheken mithilfe eines Bibliotheksvorbereitungskits mit geringem Input, das mit kurzen Sequenzen (300 bp) kompatibel ist, mithilfe eindeutiger Indexkombinationen für jeden in Schritt 5.3 erstellten Beispielpool gemäß den Anweisungen des Herstellers vor.
- Bereiten Sie den Bibliothekspool für die Sequenzierung vor.
- Tauen Sie auf und bereiten Sie eine Kartusche mit 300 Zyklen und Sequenzierungsreagenzien vor.
- Denaturieren und verdünnen Sie den in Schritt 5.1.1 erstellten Sequenzierungspool gemäß den Anweisungen des Herstellers des Sequenzers.
- Fügen Sie denaturierten und verdünnten Bibliothekspool zu Sequenzierungsreagenzien hinzu, und führen Sie den Sequenziercomputer gemäß den Angaben des Herstellers aus.
HINWEIS: Fehlerbehebungen finden Sie in der beigefügten Tabelle 3.
7. Datenanalyse
HINWEIS: Alle Datenschritte werden in der genomischen Datenanalysesoftware durchgeführt. Klammern zeigen Benutzereingaben an. Das Größer-als-Zeichen gibt die Reihenfolge der Mausklicks für einen bestimmten Schritt an (z. B. 1. Mausklick>2. Mausklick)
- Importieren Sie die Lesevorgänge, indem Sie auf Software öffnen > > Illumina importieren klicken, > Dateien auswählen > Weiter > Speicherort auswählen > fertig stellen. Die Lesevorgänge werden nun in der Software angezeigt.
- Kürzen und filtern Sie die Lesevorgänge.
- Schneiden Sie in der Deep-Sequence-Datenanalysesoftware die Standardparameter für unformatierte Lesevorgänge zu.
- Markieren Sie die Lesevorgänge, und klicken Sie auf Toolbox > Sequenzierungsdaten vorbereiten > Zuschneidelesevorgänge > Nächste > Nächste > Nächste > Nächste > Speichern > Nächste > (Speicherort auswählen) > Fertig stellen
- Ordnen Sie die zugeschnittenen Lesevorgänge der Referenz zu.
- Zuordnen Sie getrimmte Lesevorgänge auf die in Schritt 4.1 verwendete Referenzsequenz mit einer Übereinstimmungspunktzahl von 2, Mismatch-Kosten von 3 und Einfüge-/Löschkosten von 2. Stellen Sie sicher, dass der Längenanteil über 0,7 und der Ähnlichkeitsanteil bei oder über 0,8 liegt.
- Markieren Sie die zugeschnittene Lesedatei, und klicken Sie auf Toolbox > Resequencing Analysis > Map Reads to Reference > Next > (Referenzsequenz auswählen) > Nächste > (Stellen Sie sicher, dass die Parameter wie oben angegeben sind) > Weiter > > speichern (Speicherort auswählen) > Fertig stellen.
- Extrahieren Sie Varianten und Indels.
- Extrahieren Sie mit einem geeigneten indel-Aufrufer indels mit einem p-Wert-Schwellenwert von 0,005 oder niedriger und einer maximalen Anzahl von Inkongruenzen von 3.
- Markieren Sie die zugeordnete Lesedatei, und klicken Sie auf Toolbox > Sequenzierungsanalyse > Variantenerkennung > Indels und Strukturvarianten > Nächste > (Geben Sie sicher, dass die erforderliche Signifikanz eingegeben wird> Weiter > > speichern (Speicherort zum Speichern auswählen) > Fertig stellen.
- Rufen Sie mit einem geeigneten Variantenaufrufer Varianten aus dem Read Mapping mit einer Signifikanz von 5% auf.
- Markieren Sie die zugeordnete gelesene Datei, und klicken Sie auf Toolbox > Resequencing Analysis > Variant Detection > Low frequency variant detection > Next > (Sicherstellen, dass die erforderliche Signifikanz eingegeben wird, > Weiter > Weiter > Save > (Speicherort auswählen) > Fertig stellen.
HINWEIS: Stellen Sie sicher, dass die Ploidie des Wirtsgenoms in den Indel- und Variantenaufrufern berücksichtigt wird. Extrahieren Sie keine Mutanten unter 5%. Dieser Schwellenwert berücksichtigt PCR- und Sequenzierungsfehler im Zusammenhang mit dem Assay. Die Normalisierung (basierend auf dem Immunoblot-Nachweis von CAS9) sollte durch Anpassen der Sequenzierungsabdeckung erfolgen. Wenn beispielsweise Probe A die doppelte Transfektionseffizienz von Probe B aufweist, sollten 50 % der Messwerte aus Probe A für die Analyse verwendet werden. Dies sollte durch Stichproben erfolgen und die Abdeckung für eine Stichprobe nicht unter 100x reduzieren.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Für dieses Protokoll werden drei repräsentative Ergebnisse vorgestellt. Abbildung 1 ist ein Immunoblot, der die Expression von CAS9 in der HFK-Kontrolle (LXSN) und die HFK-Expression von Beta-HPV 8E6 (8E6) bestätigt. 48 h nach der Transfektion wurden Ganzzelllysate entnommen und anschließend mit einem Anti-CAS9-Antikörper (oder GAPDH als Belastungskontrolle) untersucht. Das Ergebnis zeigt, dass HFK LXSN und HFK 8E6 eine ähnliche Menge an CAS9 exprimiere...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Neben der Tiefe der bereitgestellten Informationen bietet diese Methode mehrere Vorteile. Erstens kann die DSB-Reparatur theoretisch an jedem genomischen Loci beurteilt werden, ohne das Genom der interessierenden Zelle zu verändern. Zweitens wird der Zugang zur NGS-Analyse der Reparatur durch die reduzierten Kosten und den geringeren Rechenaufwand erhöht, die durch die Erstellung und Analyse eines einzelnen DSB für einen definierten Bereich erforderlich sind. Da die Genome zusätzlicher Organismen routinemäßig verf?...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die in diesem Manuskript berichtete Forschung wurde vom National Institute of General Medical Sciences der National Institutes of Health (P20GM130448) (NAW und RP) unterstützt; Nationales Krebsinstitut der National Institutes of Health (NCI R15 CA242057 01A1); Johnson Cancer Research Center an der Kansas State University; und dem US-Verteidigungsministerium (CMDRP PRCRP CA160224 (NAW)). Wir danken KSU-CVM Confocal Core und Joel Sanneman für unsere Immunfluoreszenzmikroskopie. Der Inhalt liegt allein in der Verantwortung der Autoren und stellt nicht notwendigerweise die offizielle Meinung dieser Förderagenturen dar.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 6 Well Tissue Culture Plate | Celltreat | 229106 | Cell culture plate |
| BCA Kit | VWR | 89167-794 | BCA assay kit |
| Centrifuge 5910 R | Eppendorf | 2231000772 | Tabletop Centrifuge |
| CLC Genomic Workbench | Qiagen | 832001 | deep sequence data analysis software/indel caller/variant caller |
| Digital Microplate Genie pulse | Scientific industries | SI-400A | Plate shaker |
| DYKDDDDK Tag Monoclonal Antibody (FG4R) | ThermoFisher Scientific | MA191878 | Anti-FLAG antibody |
| Epilife CF Kit | ThermoFisher Scientific | MEPICF500 | Cell cultrue media and supplements |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | Cell culture supplement |
| Goat anti-Rabbit IgG | ThermoFisher Scientific | A-11012 | Secondary antibody |
| HighPrep PCR Clean-up system | MagBio | AC-60005 | Bead-based PCR cleanup kit |
| KAPA HiFi HotStart ReadyMix PCR Kit | KAPA Biosystems | KK2600 | PCR mastermix/PCR assay |
| MagAttract HMW DNA kit | Qiagen | 67563 | High Molecular Weight DNA extraction kit |
| Magnetic Stand-96 | Thermo Fisher Scientific | AM10027 | 96-Well Magnetic Rack |
| MiniAmp Thermal Cycler | Applied Biosystems | A37834 | Thermal Cycler |
| Miseq | Illumina | SY-410-1003 | Sequencer |
| Miseq v2 300 cycle reagent kit | Illumina | MS-102-2002 | 300-cycle cartridge/sequencing reagents |
| Nextera XT DNA Library Prep kit | Illumina | FC-131-1024 | Library preparation kit |
| Nextera XT Kit v2 Set A | Illumina | 20027215 | Indexes |
| Nunc 96-well polypropylene DeepWell Stroage plates | Thermo Fisher Scientific | 260251 | deep well 96-well plates |
| Penicillin-Streptomycin Solution (100X) | Calsson Labs | PSL02-6X100ML | Antibiotics for cell culture |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | PBS |
| px330-CD4 | Addgen | 136938 | SgRNA/CAS9 plasmids targeting 5’- GGCGTATCTGTGTGAGGACT |
| QIAxcel Advanced System | Qiagen | 9001941 | capillary electrophersis machine |
| QIAxcel DNA screening kit | Qiagen | 929004 | DNA buffer/ capillary electrophersis tubes |
| Qubit 1x ds HS Assay Kit | ThermoFisher Scientific | Q23851 | Fluorometer reagents/1x dsDNA solution |
| Qubit 4 Fluorometer | ThermoFisher Scientific | Q33238 | Fluorometer |
| Qubit Assay Tubes | Thermo Fisher Scientific | Q32856 | Fluorometer assay tubes |
| RIPA Lysis Buffer | VWR | VWRVN653-100ML | Lysis buffer for protein extraction |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | Trypsin |
| Vortex-Genie 2 | Scientific industries | SI-0236 | Vortex |
| Xfect Transfection Reagent | Takara Bio | 631318 | Transfection reagent |
| genomic data analysis software | QIAGEN | CLC Workbench v21.0. | Data analysis software |
Referenzen
- Vítor, A. C., Huertas, P., Legube, G., de Almeida, S. F. Studying DNA double-strand break repair: an ever-growing toolbox. Frontiers in Molecular Biosciences. 7, (2020).
- Giglia-Mari, G., Zotter, A., Vermeulen, W. DNA damage response. Cold Spring Harbor Perspectives in Biology. 3 (1), 000745(2011).
- Khanna, K. K., Jackson, S. P. DNA double-strand breaks: Signaling, repair and the cancer connection. Nature Genetics. 27 (3), 247-254 (2001).
- vanden Berg, J. G., et al. A limited number of double-strand DNA breaks is sufficient to delay cell cycle progression. Nucleic Acids Research. 46 (19), 10132-10144 (2018).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews Molecular Cell Biology. 18 (8), 495-506 (2017).
- Daley, J. M., Sung, P. 53B. P. 1 BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Molecular and Cellular Biology. 34 (8), 1380-1388 (2014).
- Godin, S. K., Sullivan, M. R., Bernstein, K. A. Novel insights into RAD51 activity and regulation during homologous recombination and DNA replication. Biochemistry and Cell Biology = Biochimie et Biologie Cellulaire. 94 (5), 407-418 (2016).
- Jette, N., Lees-Miller, S. P. The DNA-dependent protein kinase: a multifunctional protein kinase with roles in DNA double strand break repair and mitosis. Progress in Biophysics and Molecular Biology. 117 (0), 194-205 (2015).
- Weterings, E., van Gent, D. C. The mechanism of non-homologous end-joining: A synopsis of synapsis. DNA Repair. 3 (11), 1425-1435 (2004).
- Bhargava, R., Lopezcolorado, F. W., Tsai, L. J., Stark, J. M. The canonical non-homologous end joining factor XLF promotes chromosomal deletion rearrangements in human cells. Journal of Biological Chemistry. 295 (1), 125-137 (2020).
- Gunn, A., Bennardo, N., Cheng, A., Stark, J. M. Correct end use during end joining of multiple chromosomal double strand breaks is influenced by repair protein RAD50, DNA-dependent protein kinase DNA-PKcs, and transcription context. Journal of Biological Chemistry. 286 (49), 42470-42482 (2011).
- Simsek, D., Jasin, M. Alternative end-joining is suppressed by the canonical NHEJ component Xrcc4-ligase IV during chromosomal translocation formation. Nature Structural & Molecular Biology. 17 (4), 410-416 (2010).
- Certo, M. T., et al. Tracking genome engineering outcome at individual DNA breakpoints. Nature Methods. 8 (8), 671-676 (2011).
- Murthy, V., et al. Characterizing DNA repair processes at transient and long-lasting double-strand DNA breaks by immunofluorescence microscopy. JoVE Journal of Visualized Experiments. (136), e57653(2018).
- Wang, J. L., et al. Dissection of DNA double-strand-break repair using novel single-molecule forceps. Nature Structural & Molecular Biology. , (2018).
- Azzam, E. I., Jay-Gerin, J. -P., Pain, D. Ionizing radiation-induced metabolic oxidative stress and prolonged cell injury. Cancer Letters. 327 (0), 48-60 (2012).
- Kuo, L. J., Yang, L. -X. γ-H2AX - A novel biomarker for DNA double-strand breaks. In Vivo. 5, (2008).
- Sanders, J. T., et al. Radiation-induced DNA damage and repair effects on 3D genome organization. Nature Communications. 11 (1), 6178(2020).
- Bellaiche, Y., Mogila, V., Perrimon, N. I-SceI endonuclease, a new tool for studying DNA double-strand break repair mechanisms in Drosophila. Genetics. 152 (3), 1037-1044 (1999).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 disrupt homology dependent double strand break repair by attenuating BRCA1 and BRCA2 expression and foci formation. PLOS Pathogens. 11 (3), 1004687(2015).
- Hu, C., Bugbee, T., Gamez, M., Wallace, N. A. Beta human papillomavirus 8E6 attenuates non-homologous end joining by hindering DNA-PKcs activity. Cancers. 12 (9), 2356(2020).
- Hu, C., Bugbee, T., Dacus, D., Palinski, R., Wallace, N. A. Beta human papillomavirus 8 E6 allows colocalization of non-homologous end joining and homologous recombination repair factors. PLOS Pathogens. 18 (3), 1010275(2022).
- Butler, T. A. J., Paul, J. W., Chan, E. -C., Smith, R., Tolosa, J. M. Misleading westerns: Common quantification mistakes in western blot densitometry and proposed corrective measures. BioMed Research International. 2019, 5214821(2019).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX Phosphorylation on serine 139. Journal of Biological Chemistry. 273 (10), 5858-5868 (1998).
- Taning, C. N. T., Van Eynde, B., Yu, N., Ma, S., Smagghe, G. CRISPR/Cas9 in insects: Applications, best practices and biosafety concerns. Journal of Insect Physiology. 98, 245-257 (2017).
- Ghezraoui, H., et al. Chromosomal translocations in human cells are generated by canonical nonhomologous end-joining. Molecular Cell. 55 (6), 829-842 (2014).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten