Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Использование секвенирования следующего поколения для выявления мутаций, связанных с восстановлением CAS9-индуцированного разрыва двойной цепи вблизи промотора CD4
В этой статье
Резюме
Здесь представлена эндонуклеаза sgRNA/CAS9 и протокол секвенирования следующего поколения, который может быть использован для идентификации мутаций, связанных с восстановлением двухцепочечного разрыва вблизи промотора CD4.
Аннотация
Двухцепочечные разрывы (DSB) в ДНК являются наиболее цитотоксическим типом повреждения ДНК. Поскольку множество оскорблений может привести к этим поражениям (например, стресс репликации, ионизирующее излучение, невосстановленное УФ-повреждение), DSB возникают в большинстве клеток каждый день. В дополнение к гибели клеток, невосстановленные DSB снижают целостность генома, и возникающие мутации могут привести к опухолевому генезу. Эти риски и распространенность DSB мотивируют исследования механизмов, с помощью которых клетки восстанавливают эти поражения. Секвенирование следующего поколения может быть сопряжено с индукцией DSB ионизирующим излучением, чтобы обеспечить мощный инструмент для точного определения мутаций, связанных с дефектами восстановления DSB. Однако этот подход требует вычислительно сложного и дорогостоящего секвенирования всего генома для обнаружения восстановления случайно возникающих DSB, связанных с ионизирующим излучением. Редкие режущие эндонуклеазы, такие как I-Sce1, обеспечивают способность генерировать один DSB, но их сайты распознавания должны быть вставлены в интересующий геном. В результате место ремонта по своей сути является искусственным. Последние достижения позволяют направлять РНК (sgRNA) направлять эндонуклеазу Cas9 в любой интересующий его локус генома. Это может быть применено к исследованию восстановления DSB, что делает секвенирование следующего поколения более экономически эффективным, позволяя ему сосредоточиться на ДНК, фланкирующей CAS9-индуцированный DSB. Цель рукописи состоит в том, чтобы продемонстрировать осуществимость этого подхода путем представления протокола, который может определить мутации, возникающие в результате восстановления DSB перед геном CD4. Протокол может быть адаптирован для определения изменений мутагенного потенциала DSB, связанных с экзогенными факторами, такими как ингибиторы репарации, экспрессия вирусного белка, мутации и воздействие окружающей среды с относительно ограниченными требованиями к вычислениям. После того, как геном организма был секвенирован, этот метод теоретически может быть использован в любом геномном локусе и в любой модели клеточной культуры этого организма, которая может быть трансфектирована. Подобные адаптации подхода могут позволить сравнивать точность репарации между различными локусами в одном и том же генетическом фоне.
Введение
Поддержание геномной стабильности имеет решающее значение для всех живых организмов. Точная репликация ДНК и надежный ответ на повреждение ДНК (DDR) необходимы для добросовестного распространения генетического материала 1,2. Повреждения ДНК происходят регулярно в большинстве клеток 2,3. Когда эти повреждения ощущаются, прогрессирование клеточного цикла останавливается, и активируются механизмы репарации ДНК. Двухцепочечные разрывы в ДНК или DSB являются наиболее токсичным и мутагенным типом повреждения ДНК 3,4.
В то время как несколько сигнальных путей DDR могут восстанавливать эти поражения, наиболее тщательно изученными путями восстановления DSB являются гомологичная рекомбинация (HR) и негомологичное соединение конца (NHEJ). HR - это в значительной степени безошибочный путь, который восстанавливает DSB, используя сестринскую хроматиду в качестве гомологичного шаблона. Это имеет тенденцию происходить в фазе S и фазе G2 клеточного цикла 5,6,7. NHEJ более подвержен ошибкам, но это может произойти на протяжении всего клеточного цикла 8,9. Для измерения эффективности конкретных ремонтных механизмов были разработаны различные репортерные анализы 10,11,12. Эти анализы, как правило, полагаются на проточную цитометрию для высокопроизводительного измерения активности пути восстановления DSB с использованием GFP или mCherry в качестве считывания11,13. Несмотря на высокую эффективность, они полагаются на канонический ремонт, происходящий на искусственно введенном DSB.
Существует множество других методов, используемых для изучения ремонта DSB. Многие из них полагаются на иммунофлуоресцентную (IF) микроскопию 1,14. Микроскопия ПЧ обнаруживает дискретные ядерные очаги, представляющие ремонтные комплексы, после того, как ДСП индуцируются воздействием генотоксических химических веществ или ионизирующего излучения15,16. Отслеживание образования и разрешения этих очагов дает представление о начале и завершении ремонта соответственно14,17. Однако эти методы индукции DSB (т.е. химических веществ или ионизирующего излучения) не вызывают DSB в определенных местах генома. Также функционально невозможно использовать их для последовательного индуцирования только небольшого количества (например, 2-4) DSB. В результате наиболее часто используемые методы индуцирования DSB вызывают множество поражений, случайным образом распределенных по всему геному18. Небольшое количество DSB может быть введено путем вставки сайта распознавания для редкорезающей эндонуклеазы и экспрессии соответствующей эндонуклеазы, такой как I-Sce119. К сожалению, необходимая интеграция целевого участка препятствует исследованию DSB в эндогенных геномных локусах.
В этой рукописи описывается метод обнаружения мутаций, связанных с восстановлением DSB, сгенерированного в определяемом пользователем локусе. Приведен репрезентативный пример подхода, применяемого для оценки способности вирусного белка увеличивать количество мутаций, связанных с DSB. В частности, в этой рукописи описывается использование одной направляющей РНК (sgRNA) для направления эндонуклеазы CAS9 для индуцирования DSB в открытой рамке чтения CD4 человека в кератиноцитах крайней плоти человека, экспрессирующих векторный контроль (HFK LXSN) и HFK, который экспрессирует белок E6 вируса папилломы человека типа 8 (HFK 8E6). Целенаправленное секвенирование следующего поколения (NGS) области, окружающей разрыв, позволяет строго определять мутации, связанные с восстановлением поражения. Эти данные демонстрируют, что вирусный белок вызывает примерно 20-кратное увеличение мутаций во время восстановления DSB. Он также обеспечивает объективную характеристику мутагенных последствий DSB в одном локусе без необходимости секвенирования всего генома. В принципе, протокол может быть легко адаптирован для сравнения относительного риска мутаций между локусами генома или клеточными линиями.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Покрытие ячеек
- Выращивайте клетки HFK LXSN и HFK 8E6 в 10 см пластинах в кератиноцитарной культуральной среде (10 мл / пластина) с добавкой роста кератиноцитов человека (HKGS) и 1% пенициллином / стрептомицином. Вырастите клетки примерно до 80% слияния при 37 °C в оболочке инкубатора с 5% CO2.
- Заменить культуральную среду 3 мл трипсина-ЭДТА (0,05%, этилендиамин тетрауксусной кислоты). Инкубировать при 37 °C в течение 3 мин. Нейтрализуют трипсин равным объемом фетальной бычьей сыворотки (FBS), дополненной средой, и переносят клетки в центрифужную трубку объемом 15 мл. Центрифуга при 300 х г в течение 5 мин.
- Повторное суспендирование клеток с 10 мл кератиноцитарной культуральной среды с помощью HKGS. Определяют концентрацию клеток с помощью гемоцитометра.
- Пластина 4 x 105 клеток/6 см пластин (семена двух пластин для HFK LXSN и двух пластин для HFK 8E6) в 4 мл кератиноцитарной культуральной среды с HKGS и 1% пенициллина и стрептомицина. Растут при 37 °C в инкубаторе с 5% CO2.
ПРИМЕЧАНИЕ: Анализ клеток HFK был выбран для этого протокола по двум причинам. Во-первых, HFK - это трудно трансфектируемая клеточная линия. Таким образом, демонстрируя, что протокол работает в этой клеточной линии, предоставляются доказательства того, что он, вероятно, будет работать в более часто используемых и более легко трансфектируемых клетках. Во-вторых, ранее опубликованные данные демонстрируют, что вирусный белок (8E6) препятствует восстановлению двухцепочечных разрывов в ДНК 20,21,22. Таким образом, сравнение HFK LXSN и HFK 8E6 позволяет продемонстрировать способность анализа обнаруживать увеличение мутаций, связанных со снижением способности клеточного репарации.
2. Трансфекция
- В день трансфекции (через 24 ч после нанесения покрытия) замените среду 3 мл безантибиотиков. Инкубировать в течение 2 ч при 37 °C в инкубаторе рубашки.
- Трансфектируйте клетки соответствующими трансфекционными реагентами на основе липидов в соответствии с инструкциями производителя.
- Перед использованием осторожно нагревайте трансфекционные реагенты до комнатной температуры и пипетку.
- Для каждой клеточной линии (HFK LXSN и HFK 8E6) поместите соответствующее количество трансфекционного буфера (в соответствии с указаниями производителя) в стерильную центрифужную трубку объемом 1,5 мл (трубка 1). Включите другую трубку с таким же количеством трансфекционного буфера (имитация трансфекции или трубка 2).
- Добавьте 2 мкг плазмидной ДНК, экспрессирующей CAS9/sgRNA, нацеленную на человеческий CD4 (px330-CD4, 5'- GGCGTATCTGTGTGAGGACT) в трубку 1 из шага 2.2.2. Пипетку аккуратно перемешать полностью. Добавьте равный объем стерильной воды в трубку 2.
- Включите контрольную пластину с только трансфекционными реагентами (без плазмиды) для каждой клеточной линии.
ПРИМЕЧАНИЕ: Вторая пластина служит отрицательным контролем в эксперименте, позволяя пользователю подтвердить, что трансфекция с CAS9/sgRNA не несет ответственности за какие-либо мутации. - Добавьте соответствующее количество трансфекционного реагента (в соответствии с указаниями завода-изготовителя) в пробирку со смесью ДНК (трубка 1) со стадии 2.2.3 и пробную трансфекцию (трубку 2) из стадии 2.2.2. Пипетку аккуратно перемешать полностью. Инкубировать при комнатной температуре в течение 15-30 мин, чтобы дать достаточно времени для образования комплексов.
- Добавьте трансфекционную смесь по каплям в тарелку. Аккуратно раскачиваем культуру в течение 1 мин, чтобы равномерно распределить трансфекционную смесь.
- Инкубировать в течение 48 ч после трансфекции, чтобы обеспечить экспрессию CAS9.
- Собирают клетки путем трипсинизации.
- Заменить культуральную среду 1 мл трипсина-ЭДТА (0,05%, этилендиамин тетрауксусной кислоты). Инкубировать при 37 °C в течение 3 мин. Нейтрализуют трипсин равным объемом добавляемых ФБС сред.
- Для каждой пластинки клеток переложите клеточную суспензию на две микроцентрифужные трубки с равными аликвотами. Центрифуга при 300 х г в течение 5 мин.
- Повторное суспендирование клеточной гранулы из одной трубки на стадии 2.4.2 в 1 мл фосфатного буферного физиологического раствора (PBS) для секвенирования. Повторно суспендируйте другую трубку из стадии 2.4.2 ледяной PBS для иммуноблота.
- Соберите целоклеточные лизаты для иммуноблота.
- Центрифугируйте трубку при 300 х г в течение 5 мин. Выбросьте супернатант.
- Добавьте в пробирку 100 мкл радиоиммунопреципитационный буфер анализа (буфер лизиса RIPA), смешанный с 1% ингибитором протеазы и 1% ингибитором фосфатазы, тщательно перемешайте с пипеткой и инкубируйте в течение 10 мин на льду.
ПРИМЕЧАНИЕ: Буфер лизиса RIPA содержит 10 мМ Tris-HCl, рН 8,0; 1 мМ ЭДТА; 0,5 мМ ЭГТА; 1% Тритон Х-100; 0,1% дезоксихолат натрия; 0,1% SDS; 140 мМ NaCl и деионизированная вода. - Центрифужные лизаты по 13 000 х г в течение 10 мин. Соберите супернатанты для иммуноблота.
3. Измерение экспрессии CAS9 с помощью иммуноблота
- Определить концентрацию белка с помощью анализа бицинхониновой кислоты (BCA) в соответствии с инструкциями производителя.
- Запускают 20 мкг белка каждого образца в лунках 3%-8% трисацетатного геля в течение 150 мин и полусухой перенос (10 В в течение 30 мин и затем 25 В в течение 12 мин) в поливинилидендифторидную мембрану.
- После блокировки мембраны в 5% обезжиренном сухом молоке в PBS с 0,1% анимацией (PBST) в течение 1 ч при комнатной температуре добавляют антитела анти-CAS9 (1:1000) и анти-GAPDH (1:1000). Инкубировать при 4 °C в течение ночи.
- После промывки мембраны ПБСТ инкубируют мембрану с вторичным антителом в 5% обезжиренном сухом молоке в ПБСТ в течение 1 ч при комнатной температуре.
- Изобразите пятно и определите уровень CAS9 с помощью денситометрии23. Репрезентативную пятно см. на рисунке 1 .
ПРИМЕЧАНИЕ: Обнаружение образования фосфорилированных очагов H2AX (S139) с помощью иммунофлуоресцентной микроскопии может быть использовано для подтверждения активности CAS914. Ожидается небольшое количество отчетливых очагов (обычно 1-4 очага) в зависимости от положения клеточного цикла, от того, препятствуют ли мутации в целевом участке CAS9 дальнейшему вырезанию и сколько копий сайта резания CAS9 существует в интересующем геноме. Репрезентативное изображение показано на рисунке 2.
4. Экстракция нуклеиновых кислот и генерация ампликонов
- Извлеките ДНК из образцов клеток с этапа 2.4.3 с использованием набора для экстракции ДНК с высоким молекулярным весом, как указано производителем.
- Грунтовки повторного суспендирования с указанным растворителем согласно техническому описанию. Разбавить тем же реагентом до 20 мкМ и объединить 20 мкМ грунтовки в указанные бассейны.
ПРИМЕЧАНИЕ: Праймер пул указан в Дополнительной таблице 1. - Создайте смесь PCR Master с использованием полимера Taq с длинным усилением для каждого пула грунтовок 20 мкМ, как указано в таблице 1.
- Добавьте 21 мкл мастермиксов в отдельные трубки ПЦР.
- Добавьте 4 мкл целевого образца (100 нг/мкл) со стадии 4.1 в пробирки для анализа ПЦР, содержащие мастермиксные и колпачковые пробирки. Обеспечьте отдельные реакции для каждого пула грунтовок.
- Вихрь для смешивания пробирок для анализа ПЦР и центрифуги (быстрый отжим) для удаления капель с крышек трубок.
- Поместите трубки ПЦР на обычную термовекторную машину.
- Программа ПЦР-аппарата, указанная в таблице 2.
- Запустите программу на тепловом цикле.
5. Очистка ПЦР
- Удалите праймеры из реакций ПЦР с помощью системы очистки ПЦР на основе шариков.
- Выньте бусины для очистки из холодильника за 30 минут до использования.
- Вихревые бусины задолго до использования и убедитесь, что все бусины повторно суспендированы.
- Добавьте 30 мкл (1,2x) повторно суспендированных шариков в каждую скважину глубокой 96-луночной плиты.
- Добавьте 25 мкл реакции ПЦР к скважинам, содержащим шарики.
- Поместите пластину на шейкер для пластин при 2000 об/мин в течение 2 мин.
- Дайте пластине остаться при комнатной температуре в течение 5 минут после встряхивания.
- Поместите глубокую пластину скважины на магнит из 96 лунок и инкубируйте в течение 2 минут.
- Удалите и выбросьте супернатант без мешающих бусин.
- Пока пластина остается на магните, добавьте 180 мкл 80% этанола и инкубируйте в течение 30 с. Удалите и выбросьте супернатант.
- Повторите шаг 5.1.9.
- Используя пипетку объемом 10 мкл, удалите и выбросьте оставшуюся жидкость из скважин.
- Дайте бусинам высохнуть при комнатной температуре в течение 10 мин.
- Добавьте 20 мкл воды без нуклеазы в колодцы, содержащие шарики, и извлеките пластину из магнита.
- Встряхните пластину при 2000 об/мин в течение 2 мин при комнатной температуре.
- Инкубировать пластину при комнатной температуре в течение 5 мин.
- Поместите пластину на магнитную подставку и высиживайте в течение 2 мин при комнатной температуре.
- Удалите супернатант во вторую, помеченную ПЦР-пластину. Он содержит очищенную ДНК.
- Измерьте концентрацию каждой реакции с помощью флуорометра.
- Убедитесь, что реагенты фторометра dsDNA имеют комнатную температуру.
- Установите флуорометрические пробирные трубки плюс две дополнительные трубки для стандартов.
- Добавьте 199 мкл 1x рабочего раствора dsDNA во все трубки, кроме двух. Добавьте 190 мкл рабочего раствора в последние две пробирки.
- Добавьте 10 мкл двух стандартов ( включенных в Таблицу материалов) для разделения пробирных трубок.
- Добавьте 1 мкл каждой реакции ПЦР в пробирки мастермикса флуорометра.
- Вихревые трубки для смешивания и инкубации при комнатной температуре в течение 2 мин.
- На начальном экране флуорометра нажмите кнопку с анализом использования китина (1x dsDNA), затем выберите «Чтение стандартов и запуск образцов».
- Вставьте стандартную трубку 1, нажмите кнопку «Чтение» и повторите для стандарта 2.
- После этапа 5.2.6 повторите для одного образца, выберите объем образца в 1 мкл и будет указана полученная концентрация.
- Повторите шаг 5.2.7 для остальных образцов.
- Рассчитайте прогнозируемую молярность всех реакций и пул равных концентраций реакций из каждого отдельного образца в отдельности (один конечный пул на образец), используя уравнение ниже.
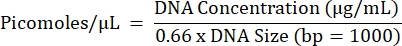
- Повторите шаги 5.2.1-5.2.7 для получения конечной концентрации в бассейне.
- Проверьте пул ампликонов на аппарате капиллярного электрофореза / агарозном геле в соответствии с указаниями производителя.
- Подготовьте капиллярные электрофорезные трубки для соответствующего количества образцов. Добавьте 7 мкл буфера ДНК, как указано производителем.
- Добавьте 4 мкл пула ампликонов в трубку, содержащую буфер ДНК.
- Поместите трубки на машину для электрофореза и запустите машину, как указано производителем для dsDNA.
- Просмотрите изображения геля электрофореза, гарантируя, что полосы локализуются до ~5 кб (размер ампликонов).
- Рассчитайте прогнозируемую молярность всех реакций и пул равных концентраций реакций из каждого отдельного образца в отдельности (один конечный пул на образец), используя уравнение ниже.
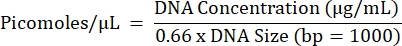
6. Подготовка библиотеки
- Разбавляйте пулы проб от стадии 5.3.1 до 0,2 нг/мкл для подготовки библиотеки.
- С помощью комплекта подготовки библиотек с низким входом, совместимого с короткими последовательностями (300bp), подготовьте библиотеки с использованием уникальных комбинаций индексов для каждого пула образцов, созданного на шаге 5.3 в соответствии с инструкциями производителя.
ПРИМЕЧАНИЕ: Следуйте инструкциям производителя для выбора последовательностей индексов. Все индексы, поддающиеся набору подготовки библиотеки, будут работать для образцов. - После подготовки библиотеки объедините все образцы в соответствии с инструкциями производителя.
ПРИМЕЧАНИЕ: Перед созданием библиотечного пула рассчитайте количество считываний, необходимое для 250-кратного покрытия целевой последовательности, и убедитесь, что выбранный картридж последовательности может обеспечить адекватное покрытие для каждого включенного образца. Для общей суммы 0,5 Мб это будет приравниваться к 1 МБ чтения.
- С помощью комплекта подготовки библиотек с низким входом, совместимого с короткими последовательностями (300bp), подготовьте библиотеки с использованием уникальных комбинаций индексов для каждого пула образцов, созданного на шаге 5.3 в соответствии с инструкциями производителя.
- Подготовьте пул библиотек к виртуализации.
- Разморозьте и подготовьте 300-цикловый картридж и реагенты для секвенирования.
- Денатурируйте и разбавляйте пул секвенирования, созданный на шаге 5.1.1, в соответствии с инструкциями производителя секвенсора.
- Добавьте денатурированный и разбавленный пул библиотек в реагенты секвенирования и запустите машину секвенирования, как указано производителем.
ПРИМЕЧАНИЕ: См. прилагаемую таблицу 3 для устранения неполадок.
7. Анализ данных
ПРИМЕЧАНИЕ: Все этапы обработки данных выполняются в программном обеспечении для анализа геномных данных. Скобки обозначают вводимые пользователем данные. Знак больше, указывает порядок щелчков мыши для любого заданного шага (например,1-й щелчок мыши>2-й щелчок мыши)
- Импортируйте прочитанные данные, щелкнув Открыть программное обеспечение > Импортировать > Illumina > Выбрать файлы > Далее > Выбрать местоположение, чтобы сохранить > Готово. Чтение теперь появится в программном обеспечении.
- Обрежьте и отфильтруйте чтение.
- В программном обеспечении для анализа данных глубокой последовательности обрежьте необработанные параметры чтения по умолчанию.
- Выделите данные для чтения и щелкните Панель элементов > Подготовка данных виртуализации > Обрезка чтения > Следующая > Следующая > Следующая > Следующая > Сохранить > Следующая > (Выберите расположение для сохранения) > Готово
- Сопоставьте обрезанные чтения со ссылкой.
- Сопоставление обрезанных чтений с эталонной последовательностью, используемой на шаге 4.1, с использованием оценки соответствия 2, стоимости несоответствия 3 и затрат на вставку/удаление 2. Убедитесь, что доля длины выше 0,7, а доля подобия находится на уровне или выше 0,8.
- Выделите обрезанный прочитанный файл и щелкните Панель элементов > Повторное сопоставление анализа > сопоставление чтения со ссылкой > Следующая > (Выбор ссылочной последовательности) > Следующая > (Убедитесь, что параметры указаны, как указано выше) > Далее > Сохранить > (Выберите местоположение для сохранения) > Готово.
- Извлекайте варианты и инделы.
- Используя соответствующий вызывающий объект indel, извлеките indels, используя пороговое значение p 0,005 или ниже и максимальное число несоответствий 3.
- Выделите сопоставленный прочитанный файл и щелкните Панель элементов > Анализ повторного > Обнаружение вариантов > Indels и Варианты конструкций > Следующая > (Убедитесь, что необходимая значимость введена > Следующая > Сохранить > (Выбрать местоположение для сохранения) > Готово.
- Используя соответствующий вызывающий вариант, вызовите варианты из сопоставления чтения, используя значение 5%.
- Выделите сопоставленный прочитанный файл и щелкните Панель элементов > Анализ > обнаружение вариантов > Обнаружение низкочастотных вариантов > Следующая > (Убедитесь, что необходимая значимость введена > Следующая > Следующая > Сохранить > (Выбрать местоположение для сохранения) > Готово.
ПРИМЕЧАНИЕ: Убедитесь, что учитывается плоидность генома хозяина в индельных и вариантных вызывающих абонентах. Не извлекайте мутантов ниже 5%. Этот порог учитывает ошибки ПЦР и секвенирования, связанные с анализом. Нормализация (на основе иммуноблоттического обнаружения CAS9) должна осуществляться путем корректировки охвата секвенированием. Например, если образец А имеет в два раза большую эффективность трансфекции, чем образец В, то для анализа следует использовать 50% показаний из образца А. Это должно быть сделано путем случайной выборки и не уменьшать охват для любой выборки ниже 100x.
Access restricted. Please log in or start a trial to view this content.
Результаты
Для этого протокола представлены три репрезентативных результата. Рисунок 1 представляет собой иммуноблот, подтверждающий экспрессию CAS9 в контроле HFK (LXSN) и HFK, экспрессирующем бета-ВПЧ 8E6 (8E6). Через 48 ч после трансфекции цельклеточные лизаты собирали и ?...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Помимо глубины предоставляемой информации, у этого метода есть несколько преимуществ. Во-первых, репарация DSB, теоретически, может быть оценена в любом геномном локусе без изменения генома интересующей клетки. Во-вторых, доступ к анализу ремонта NGS увеличивается за счет снижения затрат ...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Исследования, о которых сообщается в этой рукописи, были поддержаны Национальным институтом общих медицинских наук Национальных институтов здравоохранения (P20GM130448) (NAW и RP); Национальный институт рака Национальных институтов здравоохранения (NCI R15 CA242057 01A1); Исследовательский центр рака Джонсона в Университете штата Канзас; и Министерство обороны США (CMDRP PRCRP CA160224 (NAW)). Мы ценим KSU-CVM Confocal Core и Джоэла Саннемана за нашу иммунофлуоресцентную микроскопию. Содержание является исключительной ответственностью авторов и не обязательно отражает официальную точку зрения этих финансирующих учреждений.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 6 Well Tissue Culture Plate | Celltreat | 229106 | Cell culture plate |
| BCA Kit | VWR | 89167-794 | BCA assay kit |
| Centrifuge 5910 R | Eppendorf | 2231000772 | Tabletop Centrifuge |
| CLC Genomic Workbench | Qiagen | 832001 | deep sequence data analysis software/indel caller/variant caller |
| Digital Microplate Genie pulse | Scientific industries | SI-400A | Plate shaker |
| DYKDDDDK Tag Monoclonal Antibody (FG4R) | ThermoFisher Scientific | MA191878 | Anti-FLAG antibody |
| Epilife CF Kit | ThermoFisher Scientific | MEPICF500 | Cell cultrue media and supplements |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | Cell culture supplement |
| Goat anti-Rabbit IgG | ThermoFisher Scientific | A-11012 | Secondary antibody |
| HighPrep PCR Clean-up system | MagBio | AC-60005 | Bead-based PCR cleanup kit |
| KAPA HiFi HotStart ReadyMix PCR Kit | KAPA Biosystems | KK2600 | PCR mastermix/PCR assay |
| MagAttract HMW DNA kit | Qiagen | 67563 | High Molecular Weight DNA extraction kit |
| Magnetic Stand-96 | Thermo Fisher Scientific | AM10027 | 96-Well Magnetic Rack |
| MiniAmp Thermal Cycler | Applied Biosystems | A37834 | Thermal Cycler |
| Miseq | Illumina | SY-410-1003 | Sequencer |
| Miseq v2 300 cycle reagent kit | Illumina | MS-102-2002 | 300-cycle cartridge/sequencing reagents |
| Nextera XT DNA Library Prep kit | Illumina | FC-131-1024 | Library preparation kit |
| Nextera XT Kit v2 Set A | Illumina | 20027215 | Indexes |
| Nunc 96-well polypropylene DeepWell Stroage plates | Thermo Fisher Scientific | 260251 | deep well 96-well plates |
| Penicillin-Streptomycin Solution (100X) | Calsson Labs | PSL02-6X100ML | Antibiotics for cell culture |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | PBS |
| px330-CD4 | Addgen | 136938 | SgRNA/CAS9 plasmids targeting 5’- GGCGTATCTGTGTGAGGACT |
| QIAxcel Advanced System | Qiagen | 9001941 | capillary electrophersis machine |
| QIAxcel DNA screening kit | Qiagen | 929004 | DNA buffer/ capillary electrophersis tubes |
| Qubit 1x ds HS Assay Kit | ThermoFisher Scientific | Q23851 | Fluorometer reagents/1x dsDNA solution |
| Qubit 4 Fluorometer | ThermoFisher Scientific | Q33238 | Fluorometer |
| Qubit Assay Tubes | Thermo Fisher Scientific | Q32856 | Fluorometer assay tubes |
| RIPA Lysis Buffer | VWR | VWRVN653-100ML | Lysis buffer for protein extraction |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | Trypsin |
| Vortex-Genie 2 | Scientific industries | SI-0236 | Vortex |
| Xfect Transfection Reagent | Takara Bio | 631318 | Transfection reagent |
| genomic data analysis software | QIAGEN | CLC Workbench v21.0. | Data analysis software |
Ссылки
- Vítor, A. C., Huertas, P., Legube, G., de Almeida, S. F. Studying DNA double-strand break repair: an ever-growing toolbox. Frontiers in Molecular Biosciences. 7, (2020).
- Giglia-Mari, G., Zotter, A., Vermeulen, W. DNA damage response. Cold Spring Harbor Perspectives in Biology. 3 (1), 000745(2011).
- Khanna, K. K., Jackson, S. P. DNA double-strand breaks: Signaling, repair and the cancer connection. Nature Genetics. 27 (3), 247-254 (2001).
- vanden Berg, J. G., et al. A limited number of double-strand DNA breaks is sufficient to delay cell cycle progression. Nucleic Acids Research. 46 (19), 10132-10144 (2018).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews Molecular Cell Biology. 18 (8), 495-506 (2017).
- Daley, J. M., Sung, P. 53B. P. 1 BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Molecular and Cellular Biology. 34 (8), 1380-1388 (2014).
- Godin, S. K., Sullivan, M. R., Bernstein, K. A. Novel insights into RAD51 activity and regulation during homologous recombination and DNA replication. Biochemistry and Cell Biology = Biochimie et Biologie Cellulaire. 94 (5), 407-418 (2016).
- Jette, N., Lees-Miller, S. P. The DNA-dependent protein kinase: a multifunctional protein kinase with roles in DNA double strand break repair and mitosis. Progress in Biophysics and Molecular Biology. 117 (0), 194-205 (2015).
- Weterings, E., van Gent, D. C. The mechanism of non-homologous end-joining: A synopsis of synapsis. DNA Repair. 3 (11), 1425-1435 (2004).
- Bhargava, R., Lopezcolorado, F. W., Tsai, L. J., Stark, J. M. The canonical non-homologous end joining factor XLF promotes chromosomal deletion rearrangements in human cells. Journal of Biological Chemistry. 295 (1), 125-137 (2020).
- Gunn, A., Bennardo, N., Cheng, A., Stark, J. M. Correct end use during end joining of multiple chromosomal double strand breaks is influenced by repair protein RAD50, DNA-dependent protein kinase DNA-PKcs, and transcription context. Journal of Biological Chemistry. 286 (49), 42470-42482 (2011).
- Simsek, D., Jasin, M. Alternative end-joining is suppressed by the canonical NHEJ component Xrcc4-ligase IV during chromosomal translocation formation. Nature Structural & Molecular Biology. 17 (4), 410-416 (2010).
- Certo, M. T., et al. Tracking genome engineering outcome at individual DNA breakpoints. Nature Methods. 8 (8), 671-676 (2011).
- Murthy, V., et al. Characterizing DNA repair processes at transient and long-lasting double-strand DNA breaks by immunofluorescence microscopy. JoVE Journal of Visualized Experiments. (136), e57653(2018).
- Wang, J. L., et al. Dissection of DNA double-strand-break repair using novel single-molecule forceps. Nature Structural & Molecular Biology. , (2018).
- Azzam, E. I., Jay-Gerin, J. -P., Pain, D. Ionizing radiation-induced metabolic oxidative stress and prolonged cell injury. Cancer Letters. 327 (0), 48-60 (2012).
- Kuo, L. J., Yang, L. -X. γ-H2AX - A novel biomarker for DNA double-strand breaks. In Vivo. 5, (2008).
- Sanders, J. T., et al. Radiation-induced DNA damage and repair effects on 3D genome organization. Nature Communications. 11 (1), 6178(2020).
- Bellaiche, Y., Mogila, V., Perrimon, N. I-SceI endonuclease, a new tool for studying DNA double-strand break repair mechanisms in Drosophila. Genetics. 152 (3), 1037-1044 (1999).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 disrupt homology dependent double strand break repair by attenuating BRCA1 and BRCA2 expression and foci formation. PLOS Pathogens. 11 (3), 1004687(2015).
- Hu, C., Bugbee, T., Gamez, M., Wallace, N. A. Beta human papillomavirus 8E6 attenuates non-homologous end joining by hindering DNA-PKcs activity. Cancers. 12 (9), 2356(2020).
- Hu, C., Bugbee, T., Dacus, D., Palinski, R., Wallace, N. A. Beta human papillomavirus 8 E6 allows colocalization of non-homologous end joining and homologous recombination repair factors. PLOS Pathogens. 18 (3), 1010275(2022).
- Butler, T. A. J., Paul, J. W., Chan, E. -C., Smith, R., Tolosa, J. M. Misleading westerns: Common quantification mistakes in western blot densitometry and proposed corrective measures. BioMed Research International. 2019, 5214821(2019).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX Phosphorylation on serine 139. Journal of Biological Chemistry. 273 (10), 5858-5868 (1998).
- Taning, C. N. T., Van Eynde, B., Yu, N., Ma, S., Smagghe, G. CRISPR/Cas9 in insects: Applications, best practices and biosafety concerns. Journal of Insect Physiology. 98, 245-257 (2017).
- Ghezraoui, H., et al. Chromosomal translocations in human cells are generated by canonical nonhomologous end-joining. Molecular Cell. 55 (6), 829-842 (2014).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены