Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Utilisation du séquençage de nouvelle génération pour identifier les mutations associées à la réparation d’une rupture double brin induite par CAS9 près du promoteur CD4
Dans cet article
Résumé
Présenté ici est sgRNA/CAS9 endonucléase et protocole de séquençage de nouvelle génération qui peut être utilisé pour identifier les mutations associées à la réparation de rupture double brin près du promoteur CD4.
Résumé
Les cassures double brin (DSB) dans l’ADN sont le type de dommage à l’ADN le plus cytotoxique. Parce qu’une myriade d’insultes peuvent entraîner ces lésions (par exemple, stress de réplication, rayonnements ionisants, dommages UV non réparés), les DSB se produisent dans la plupart des cellules chaque jour. En plus de la mort cellulaire, les DSB non réparés réduisent l’intégrité du génome et les mutations qui en résultent peuvent conduire à la tumorigenèse. Ces risques et la prévalence des DSB motivent les recherches sur les mécanismes par lesquels les cellules réparent ces lésions. Le séquençage de nouvelle génération peut être associé à l’induction de DSB par rayonnement ionisant pour fournir un outil puissant permettant de définir avec précision les mutations associées aux défauts de réparation des DSB. Cependant, cette approche nécessite un séquençage du génome entier difficile sur le plan informatique et d’un coût prohibitif pour détecter la réparation des DSB aléatoires associés aux rayonnements ionisants. Les endonucléases coupantes rares, telles que I-Sce1, permettent de générer un seul DSB, mais leurs sites de reconnaissance doivent être insérés dans le génome d’intérêt. En conséquence, le site de réparation est intrinsèquement artificiel. Des avancées récentes permettent à l’ARN guide (ARNg) de diriger une endonucléase Cas9 vers n’importe quel locus du génome d’intérêt. Cela pourrait être appliqué à l’étude de la réparation du DSB rendant le séquençage de nouvelle génération plus rentable en lui permettant de se concentrer sur l’ADN flanquant le DSB induit par Cas9. L’objectif du manuscrit est de démontrer la faisabilité de cette approche en présentant un protocole permettant de définir des mutations issues de la réparation d’un DSB en amont du gène CD4. Le protocole peut être adapté pour déterminer les changements dans le potentiel mutagène du DSB associés à des facteurs exogènes, tels que les inhibiteurs de réparation, l’expression des protéines virales, les mutations et les expositions environnementales avec des exigences de calcul relativement limitées. Une fois que le génome d’un organisme a été séquencé, cette méthode peut théoriquement être utilisée à n’importe quel locus génomique et dans n’importe quel modèle de culture cellulaire de cet organisme qui peut être transfecté. Des adaptations similaires de l’approche pourraient permettre des comparaisons de la fidélité de réparation entre différents loci dans le même fond génétique.
Introduction
Le maintien de la stabilité génomique est essentiel pour tous les organismes vivants. Une réplication précise de l’ADN et une réponse robuste aux dommages à l’ADN (DDR) sont nécessaires pour propager fidèlement le matériel génétique 1,2. Les dommages à l’ADN se produisent régulièrement dans la plupart des cellules 2,3. Lorsque ces dommages sont détectés, la progression du cycle cellulaire est arrêtée et les mécanismes de réparation de l’ADN sont activés. Les ruptures double brin dans l’ADN ou les DSB sont le type de dommages à l’ADN le plus toxique et le plus mutagène 3,4.
Alors que plusieurs voies de signalisation DDR peuvent réparer ces lésions, les voies de réparation DSB les plus étudiées sont la recombinaison homologue (HR) et l’assemblage final non homologue (NHEJ). HR est une voie largement sans erreur qui répare un DSB en utilisant une chromatide sœur comme modèle homologue. Cela a tendance à se produire dans la phase S et la phase G2 d’un cycle cellulaire 5,6,7. NhEJ est plus sujet aux erreurs, mais cela peut se produire tout au long du cycle cellulaire 8,9. Divers essais de rapporteur ont été mis au point pour mesurer l’efficacité de mécanismes de réparation spécifiques 10,11,12. Ces essais ont tendance à s’appuyer sur la cytométrie en flux pour une mesure à haut débit de l’activité de la voie de réparation DSB en utilisant GFP ou mCherry comme lecture11,13. Bien que très efficaces, ils reposent sur une réparation canonique se produisant dans un DSB introduit artificiellement.
Il existe une variété d’autres méthodes utilisées pour étudier la réparation DSB. Beaucoup d’entre eux reposent sur la microscopie par immunofluorescence (FI) 1,14. La microscopie IF détecte des foyers nucléaires discrets représentatifs des complexes de réparation après que les DSB sont induits par l’exposition à des produits chimiques génotoxiques ou àdes rayonnements ionisants 15,16. Le suivi de la formation et de la résolution de ces foyers fournit une indication du début et de l’achèvement des réparations, respectivement14,17. Cependant, ces méthodes d’induction du DSB (c.-à-d. les produits chimiques ou les rayonnements ionisants) ne causent pas de DSB à des endroits définis du génome. Il est également fonctionnellement impossible de les utiliser pour induire systématiquement seulement un petit nombre (par exemple, 2-4) de DSB. En conséquence, les méthodes les plus couramment utilisées pour induire les DSB provoquent une multitude de lésions réparties aléatoirement dans tout le génome18. Un petit nombre d’ADB peut être introduit en insérant le site de reconnaissance d’une endonucléase coupant rare et en exprimant l’endonucléase pertinente, telle que I-Sce119. Malheureusement, l’intégration requise d’un site cible empêche l’examen du DSB aux loci génomiques endogènes.
Ce manuscrit décrit une méthode pour détecter les mutations associées à la réparation d’un DSB généré à un locus défini par l’utilisateur. Nous fournissons un exemple représentatif de l’approche appliquée pour évaluer la capacité d’une protéine virale à augmenter le nombre de mutations associées à un DSB. Plus précisément, ce manuscrit décrit l’utilisation d’un ARN guide unique (ARNg) pour diriger une endonucléase CAS9 afin d’induire un DSB au cadre de lecture ouvert CD4 humain dans les kératinocytes du prépuce humain exprimant la lutte antivectorielle (HFK LXSN) et HFK qui exprime la protéine E6 du virus du papillome humain de type 8 (HFK 8E6). Le séquençage ciblé de nouvelle génération (NGS) de la région entourant la cassure permet de définir rigoureusement les mutations associées à la réparation de la lésion. Ces données démontrent que la protéine virale provoque une augmentation d’environ 20 fois des mutations pendant la réparation du DSB. Il fournit également une caractérisation impartiale des conséquences mutagènes des DSB à un seul locus sans avoir besoin d’un séquençage du génome entier. En principe, le protocole pourrait être facilement adapté pour comparer le risque relatif de mutations entre les loci du génome ou les lignées cellulaires.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Placage cellulaire
- Cultiver des cellules HFK LXSN et HFK 8E6 dans des plaques de 10 cm dans des milieux de culture de kératinocytes (10 mL / plaque) avec un supplément de croissance des kératinocytes humains (HKGS) et 1% de pénicilline / streptomycine. Cultiver des cellules à environ 80% de confluence à 37 °C dans un incubateur à enveloppe avec 5% de CO2.
- Remplacer les milieux de culture par 3 mL de trypsine-EDTA (0,05 %, acide tétraacétique d’éthylènediamine). Incuber à 37 °C pendant 3 min. Neutraliser la trypsine avec un volume égal de sérum fœtal bovin (FBS) complété par un milieu et transférer les cellules dans un tube de centrifugeuse de 15 mL. Centrifuger à 300 x g pendant 5 min.
- Remettre en suspension des cellules avec 10 mL de milieux de culture de kératinocytes avec HKGS. Déterminer la concentration de cellules avec un hémocytomètre.
- Plaque 4 x 10plaques de 5 cellules/6 cm (graine deux plaques pour HFK LXSN et deux plaques pour HFK 8E6) dans 4 ml de milieux de culture de kératinocytes avec HKGS et 1% de pénicilline et de streptomycine. Cultivez à 37 °C dans un incubateur à enveloppe avec 5% de CO2.
NOTE: L’analyse des cellules HFK a été choisie pour ce protocole pour deux raisons. Tout d’abord, HFK est une lignée cellulaire difficile à transfecter. Ainsi, en démontrant que le protocole fonctionne dans cette lignée cellulaire, il est prouvé qu’il fonctionnera probablement dans des cellules plus couramment utilisées et plus facilement transfectées. Deuxièmement, des données publiées précédemment démontrent qu’une protéine virale (8E6) entrave la réparation des cassures double brin dans l’ADN 20,21,22. Ainsi, la comparaison de HFK LXSN et HFK 8E6 nous permet de démontrer la capacité du test à détecter des augmentations de mutations associées à une réduction de la capacité de réparation cellulaire.
2. Transfection
- Le jour de la transfection (24 h après le placage), remplacer le milieu par 3 mL de milieux supplémentés sans antibiotiques. Incuber pendant 2 h à 37 °C dans un incubateur à veste.
- Transfecter les cellules avec des réactifs de transfection à base de lipides appropriés conformément aux instructions du fabricant.
- Réchauffez les réactifs de transfection à température ambiante et pipetez doucement avant utilisation.
- Pour chaque lignée cellulaire (HFK LXSN et HFK 8E6), placez la quantité appropriée de tampon de transfection (selon les directives du fabricant) dans un tube centrifuge stérile de 1,5 mL (tube 1). Inclure un autre tube avec la même quantité de tampon de transfection (transfection simulée ou tube 2).
- Ajouter 2 μg d’ADN plasmidique exprimant CAS9/sgRNA ciblant le CD4 humain (px330-CD4, 5'- GGCGTATCTGTGTGAGGACT) au tube 1 à partir de l’étape 2.2.2. Pipette doucement pour bien mélanger. Ajouter un volume égal d’eau stérile au tube 2.
- Inclure une plaque témoin avec des réactifs de transfection seuls (sans plasmide) pour chaque lignée cellulaire.
REMARQUE: La deuxième plaque sert de contrôle négatif dans l’expérience, permettant à l’utilisateur de confirmer que la transfection avec le CAS9 / SGRNA n’est responsable d’aucune mutation. - Ajouter la quantité appropriée du réactif de transfection (selon les directives du fabricant) dans le tube avec le mélange d’ADN (tube 1) de l’étape 2.2.3 et la transfection simulée (tube 2) de l’étape 2.2.2. Pipette doucement pour bien mélanger. Incuber à température ambiante pendant 15-30 min pour laisser suffisamment de temps pour que les complexes se forment.
- Ajouter le mélange de transfection à la plaque. Bercez doucement la culture pendant 1 min pour répartir uniformément le mélange de transfection.
- Incuber pendant 48 h après la transfection pour permettre l’expression de CAS9.
- Récolter des cellules par trypsinisation.
- Remplacer les milieux de culture par 1 mL de trypsine-EDTA (0,05 %, acide tétraacétique d’éthylènediamine). Incuber à 37 °C pendant 3 min. Neutraliser la trypsine avec un volume égal de milieux supplémentés FBS.
- Pour chaque plaque de cellules, transférer la suspension cellulaire dans deux tubes de microcentrifugation avec des aliquotes égales. Centrifuger à 300 x g pendant 5 min.
- Remettre en suspension la pastille cellulaire d’un tube à l’étape 2.4.2 dans 1 mL de solution saline tamponnée au phosphate (PBS) pour le séquençage. Remettre en suspension l’autre tube de l’étape 2.4.2 avec du PBS glacé pour l’immunoblot.
- Récoltez les lysats cellulaires entiers pour l’immunoblot.
- Centrifuger le tube à 300 x g pendant 5 min. Jetez le surnageant.
- Ajouter 100 μL de tampon de dosage de radioimmunoprécipitation (tampon de lyse RIPA) mélangé à 1 % d’inhibiteur de protéase et à 1 % d’inhibiteur de phosphatase dans le tube, bien mélanger avec une pipette et incuber pendant 10 min sur de la glace.
REMARQUE: Le tampon de lyse RIPA contient 10 mM de Tris-HCl, pH 8,0; 1 mM EDTA; 0,5 mM EGTA; 1 % Triton X-100; Désoxycholate de sodium à 0,1 %; 0,1 % de FDS; 140 mM de NaCl, et eau désionisée. - Centrifugeuse lysats à 13 000 x g pendant 10 min. Recueillir des surnageants pour l’immunoblot.
3. Mesure de l’expression de CAS9 par immunoblot
- Déterminer la concentration en protéines à l’aide d’un test d’acide bicinchoninique (ACB) conformément aux instructions du fabricant.
- Faire fonctionner 20 μg de protéines de chaque échantillon dans les puits d’un gel de tris-acétate à 3% à 8% pendant 150 min et transfert semi-sec (10 V pendant 30 min puis 25 V pendant 12 min) vers la membrane de difluorure de polyvinylidène.
- Après avoir bloqué la membrane dans du lait sec non gras à 5 % dans du PBS avec 0,1 % d’interpolation (PBST) pendant 1 h à température ambiante, ajouter les anticorps anti-CAS9 (1:1000) et anti-GAPDH (1:1000). Incuber à 4 °C pendant la nuit.
- Après avoir lavé la membrane avec du PBST, incuber la membrane avec un anticorps secondaire dans du lait sec non gras à 5% dans du PBST pendant 1 h à température ambiante.
- Imagez le blot et déterminez le niveau CAS9 par densitométrie23. Reportez-vous à la figure 1 pour un transfert représentatif.
REMARQUE: La détection de la formation de foyers H2AX phosphorylés (S139) par microscopie par immunofluorescence peut être utilisée pour valider l’activité CAS914. Un faible nombre de foyers distincts (généralement 1 à 4 foyers) est attendu en fonction de la position du cycle cellulaire, si des mutations dans le site cible CAS9 empêchent une coupe supplémentaire et combien de copies du site de coupe CAS9 existent dans le génome d’intérêt. Une image représentative est illustrée à la figure 2.
4. Extraction d’acides nucléiques et génération d’amplicons
- Extraire l’ADN d’échantillons de cellules de l’étape 2.4.3 à l’aide d’un kit d’extraction d’ADN de haut poids moléculaire, tel que spécifié par le fabricant.
- Remettre en suspension les apprêts avec le solvant indiqué selon la fiche technique. Diluer avec le même réactif à 20 μM et regrouper les amorces de 20 μM dans les piscines indiquées.
REMARQUE : Le pool d’amorces est répertorié dans le tableau supplémentaire 1. - Créez un mélange maître PCR à l’aide d’une Taq polymérase à amplification longue pour chaque pool d’amorces de 20 μM, comme spécifié dans le tableau 1.
- Ajouter 21 μL des mélanges maîtres pour séparer les tubes de PCR.
- Ajouter 4 μL de l’échantillon cible (100 ng/μL) de l’étape 4.1 aux tubes d’essai PCR contenant des tubes d’essai mastermix et capuchon. Assurez des réactions distinctes pour chaque pool d’amorces.
- Vortex pour mélanger les tubes de dosage PCR et la centrifugeuse (rotation rapide) pour éliminer les gouttelettes des couvercles des tubes.
- Placez les tubes PCR sur une machine de cycle thermique conventionnelle.
- Programmer la machine PCR comme spécifié dans le tableau 2.
- Exécutez le programme sur un cycleur thermique.
5. Nettoyage par PCR
- Retirez les amorces des réactions de PCR à l’aide d’un système de nettoyage par PCR à base de billes.
- Retirez les billes de nettoyage du réfrigérateur 30 minutes avant utilisation.
- Vortex perles bien avant utilisation et s’assurer que toutes les perles sont remises en suspension.
- Ajouter 30 μL (1,2x) de billes remises en suspension à chaque puits d’une plaque profonde de 96 puits.
- Ajouter 25 μL de réaction PCR aux puits contenant des billes.
- Placez la plaque sur un agitateur à plaque à 2000 tr/min pendant 2 min.
- Laisser la plaque rester à température ambiante pendant 5 min après agitation.
- Placez la plaque de puits profond sur un aimant à plaque de 96 puits et incubez pendant 2 min.
- Retirez et jetez le surnageant sans les perles dérangeantes.
- Pendant que la plaque reste sur l’aimant, ajoutez 180 μL d’éthanol à 80% et incubez pendant 30 s. Retirez et jetez le surnageant.
- Répétez l’étape 5.1.9.
- À l’aide d’une pipette de 10 μL, retirez et jetez tout liquide restant des puits.
- Laisser sécher les perles à température ambiante pendant 10 min.
- Ajouter 20 μL d’eau sans nucléase aux puits contenant des billes et retirer la plaque de l’aimant.
- Agiter la plaque à 2000 tr/min pendant 2 min à température ambiante.
- Incuber la plaque à température ambiante pendant 5 min.
- Placez la plaque sur un support magnétique et incubez pendant 2 min à température ambiante.
- Retirez le surnageant dans une deuxième plaque PCR étiquetée. Celui-ci contient l’ADN nettoyé.
- Mesurez la concentration de chaque réaction avec un fluoromètre.
- Assurez-vous que les réactifs du fluoromètre dsDNA sont à température ambiante.
- Installez des tubes de dosage fluorométrique plus deux tubes supplémentaires pour les étalons.
- Ajouter 199 μL de solution de travail 1x dsDNA à tous les tubes sauf deux. Ajouter 190 μL de la solution de travail aux deux derniers tubes.
- Ajouter 10 μL des deux étalons (inclus dans la Table des matériaux) pour séparer les tubes d’essai.
- Ajouter 1 μL de chaque réaction PCR aux tubes mastermix du fluoromètre.
- Tubes vortex à mélanger et à incuber à température ambiante pendant 2 min.
- Sur l’écran d’accueil du fluoromètre, sélectionnez le bouton avec le kitin d’essai (1x dsDNA), puis sélectionnez Lire les normes et Exécuter les échantillons.
- Insérez le tube standard 1, sélectionnez le bouton Lire , puis répétez l’opération pour le tube standard 2.
- Après l’étape 5.2.6, répéter pour un échantillon, sélectionner un volume d’échantillon de 1 μL et la concentration résultante sera fournie.
- Répétez l’étape 5.2.7 pour les échantillons restants.
- Calculer la molarité projetée de toutes les réactions et regrouper les concentrations égales de réactions de chaque échantillon individuel séparément (un pool final par échantillon) en utilisant l’équation ci-dessous.
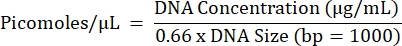
- Répétez les étapes 5.2.1 à 5.2.7 pour obtenir la concentration finale du pool.
- Vérifiez la piscine d’amplicon sur une machine d’électrophorèse capillaire / gel d’agarose comme spécifié par le fabricant.
- Préparer des tubes d’électrophorèse capillaire pour le nombre approprié d’échantillons. Ajouter 7 μL du tampon d’ADN tel que spécifié par le fabricant.
- Ajouter 4 μL du pool d’amplicons au tube contenant un tampon d’ADN.
- Placez les tubes sur la machine d’électrophorèse et faites fonctionner la machine comme spécifié par le fabricant pour dsDNA.
- Visualisez les images du gel d’électrophorèse en vous assurant que les bandes se localisent à ~ 5 kb (taille des amplicons).
- Calculer la molarité projetée de toutes les réactions et regrouper les concentrations égales de réactions de chaque échantillon individuel séparément (un pool final par échantillon) en utilisant l’équation ci-dessous.
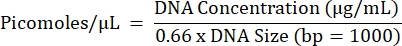
6. Préparation de la bibliothèque
- Diluer les pools d’échantillons de l’étape 5.3.1 à 0,2 ng/μL pour la préparation de la bibliothèque.
- À l’aide d’un kit de préparation de bibliothèque à faible entrée compatible avec de courtes séquences (300 bp), préparez les bibliothèques à l’aide de combinaisons d’index uniques pour chaque pool d’échantillons créé à l’étape 5.3 en suivant les instructions du fabricant.
REMARQUE: Suivez les instructions du fabricant pour sélectionner des séquences d’index. Tous les index compatibles avec le kit de préparation de la bibliothèque fonctionneront pour les échantillons. - Après la préparation de la bibliothèque, regroupez tous les échantillons conformément aux instructions du fabricant.
REMARQUE : Avant de créer le pool de bibliothèques, calculez le nombre de lectures nécessaires pour une couverture 250x de votre séquence cible et assurez-vous que la cartouche de séquençage sélectionnée peut fournir une couverture adéquate pour chaque échantillon inclus. Pour un total de 0,5 Mo, cela équivaudra à 1 million de lectures.
- À l’aide d’un kit de préparation de bibliothèque à faible entrée compatible avec de courtes séquences (300 bp), préparez les bibliothèques à l’aide de combinaisons d’index uniques pour chaque pool d’échantillons créé à l’étape 5.3 en suivant les instructions du fabricant.
- Préparez le pool de bibliothèques pour le séquençage.
- Décongeler et préparer une cartouche à 300 cycles et des réactifs de séquençage.
- Dénaturer et diluer le pool de séquençage créé à l’étape 5.1.1 conformément aux instructions du fabricant du séquenceur.
- Ajoutez un pool de bibliothèques dénaturé et dilué aux réactifs de séquençage et exécutez la machine de séquençage comme spécifié par le fabricant.
REMARQUE : Voir le tableau 3 ci-joint pour les dépannages.
7. Analyse des données
REMARQUE : Toutes les étapes des données sont effectuées dans le logiciel d’analyse des données génomiques. Les parenthèses indiquent l’entrée de l’utilisateur. Le signe supérieur indique l’ordre des clics de souris pour une étape donnée (par exemple, 1e clic de souris>2e clic de souris)
- Importez les lectures en cliquant sur Logiciel ouvert > Importer > Illumina > Sélectionner les fichiers > Suivant > Sélectionnez l’emplacement pour enregistrer > terminer. Les lectures vont maintenant apparaître dans le logiciel.
- Coupez et filtrez les lectures.
- Dans le logiciel d’analyse de données de séquence profonde, coupez les paramètres par défaut des lectures brutes.
- Mettez en surbrillance les lectures et cliquez sur Boîte à outils > Préparer les données de séquençage > Ajuster les lectures > Lectures > suivant > suivant > suivant > Enregistrer > > suivant (Sélectionnez l’emplacement à enregistrer) > Terminer
- Mappez les lectures découpées à la référence.
- La carte rognée lit la séquence de référence utilisée à l’étape 4.1 en utilisant un score de correspondance de 2, un coût d’incompatibilité de 3 et des coûts d’insertion/suppression de 2. Assurez-vous que la fraction de longueur est supérieure à 0,7 et que la fraction de similitude est égale ou supérieure à 0,8.
- Mettez en surbrillance le fichier de lecture découpé et cliquez sur Boîte à outils > Rééquençage analyse > lectures de carte à référence > > suivant (Sélectionner la séquence de référence) > > suivant (Assurez-vous que les paramètres sont indiqués comme ci-dessus) > Suivant > Enregistrer > (Sélectionner l’emplacement à enregistrer) > Terminer.
- Extraire les variantes et les indels.
- À l’aide d’un appelant indel approprié, extrayez les indels en utilisant un seuil de valeur de p de 0,005 ou moins et un nombre maximal d’incohérences de 3.
- Mettez en surbrillance le fichier de lecture mappé et cliquez sur Boîte à outils > Analyse de reséquençage > Détection des variantes > Indels et variantes structurelles > > suivant (Assurez-vous que l’importance requise est la saisie > > suivant Enregistrer > (Sélectionnez l’emplacement à enregistrer) > Terminer.
- À l’aide d’une variante appropriée de l’appelant, appelez les variantes à partir du mappage de lecture en utilisant une signification de 5 %.
- Mettez en surbrillance le fichier de lecture mappé et cliquez sur Boîte à outils > Analyse de reséquençage > Détection de variante > Détection de variante basse fréquence > > suivant (Assurez-vous que l’importance requise est entrée > > suivant > Enregistrer > (Sélectionnez l’emplacement à enregistrer) > Terminer.
REMARQUE: Assurez-vous de tenir compte de la ploïdie du génome de l’hôte dans les appelants indel et variants. Ne pas extraire les mutants en dessous de 5%. Ce seuil tient compte des erreurs de PCR et de séquençage associées au test. La normalisation (basée sur la détection immunoblot de CAS9) doit être effectuée en ajustant la couverture de séquençage. Par exemple, si l’échantillon A a une efficacité de transfection deux fois supérieure à celle de l’échantillon B, 50 % des lectures de l’échantillon A doivent être utilisées pour l’analyse. Cela devrait être fait par échantillonnage aléatoire et ne pas réduire la couverture pour tout échantillon en dessous de 100x.
Access restricted. Please log in or start a trial to view this content.
Résultats
Trois résultats représentatifs sont présentés pour ce protocole. La figure 1 est un immunoblot confirmant l’expression de CAS9 dans le contrôle HFK (LXSN) et HFK exprimant le bêta-HPV 8E6 (8E6). 48 heures après la transfection, des lysats de cellules entières ont été récoltés et ensuite sondés avec un anticorps anti-CAS9 (ou GAPDH comme contrôle de charge). Le résultat montre que HFK LXSN et HFK 8E6 expriment une quantité similaire de CAS9, ...
Access restricted. Please log in or start a trial to view this content.
Discussion
En plus de la profondeur des informations fournies, cette méthode présente plusieurs avantages. Tout d’abord, la réparation DSB, en théorie, peut être évaluée à n’importe quel loci génomique sans modifier le génome de la cellule d’intérêt. Deuxièmement, l’accès à l’analyse NGS de la réparation est accru par la réduction des coûts et des efforts de calcul offerts par la création et l’analyse d’un seul DSB ciblé sur une zone définie. Enfin, étant donné que les génomes d’organismes su...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les recherches rapportées dans ce manuscrit ont été soutenues par l’Institut national des sciences médicales générales des National Institutes of Health (P20GM130448) (NAW et RP); Institut national du cancer des National Institutes of Health (NCI R15 CA242057 01A1); Johnson Cancer Research Center à l’Université d’État du Kansas; et le département de la Défense des États-Unis (CMDRP PRCRP CA160224 (NAW)). Nous apprécions KSU-CVM Confocal Core et Joel Sanneman pour notre microscopie à immunofluorescence. Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les points de vue officiels de ces organismes de financement.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 6 Well Tissue Culture Plate | Celltreat | 229106 | Cell culture plate |
| BCA Kit | VWR | 89167-794 | BCA assay kit |
| Centrifuge 5910 R | Eppendorf | 2231000772 | Tabletop Centrifuge |
| CLC Genomic Workbench | Qiagen | 832001 | deep sequence data analysis software/indel caller/variant caller |
| Digital Microplate Genie pulse | Scientific industries | SI-400A | Plate shaker |
| DYKDDDDK Tag Monoclonal Antibody (FG4R) | ThermoFisher Scientific | MA191878 | Anti-FLAG antibody |
| Epilife CF Kit | ThermoFisher Scientific | MEPICF500 | Cell cultrue media and supplements |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | Cell culture supplement |
| Goat anti-Rabbit IgG | ThermoFisher Scientific | A-11012 | Secondary antibody |
| HighPrep PCR Clean-up system | MagBio | AC-60005 | Bead-based PCR cleanup kit |
| KAPA HiFi HotStart ReadyMix PCR Kit | KAPA Biosystems | KK2600 | PCR mastermix/PCR assay |
| MagAttract HMW DNA kit | Qiagen | 67563 | High Molecular Weight DNA extraction kit |
| Magnetic Stand-96 | Thermo Fisher Scientific | AM10027 | 96-Well Magnetic Rack |
| MiniAmp Thermal Cycler | Applied Biosystems | A37834 | Thermal Cycler |
| Miseq | Illumina | SY-410-1003 | Sequencer |
| Miseq v2 300 cycle reagent kit | Illumina | MS-102-2002 | 300-cycle cartridge/sequencing reagents |
| Nextera XT DNA Library Prep kit | Illumina | FC-131-1024 | Library preparation kit |
| Nextera XT Kit v2 Set A | Illumina | 20027215 | Indexes |
| Nunc 96-well polypropylene DeepWell Stroage plates | Thermo Fisher Scientific | 260251 | deep well 96-well plates |
| Penicillin-Streptomycin Solution (100X) | Calsson Labs | PSL02-6X100ML | Antibiotics for cell culture |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | PBS |
| px330-CD4 | Addgen | 136938 | SgRNA/CAS9 plasmids targeting 5’- GGCGTATCTGTGTGAGGACT |
| QIAxcel Advanced System | Qiagen | 9001941 | capillary electrophersis machine |
| QIAxcel DNA screening kit | Qiagen | 929004 | DNA buffer/ capillary electrophersis tubes |
| Qubit 1x ds HS Assay Kit | ThermoFisher Scientific | Q23851 | Fluorometer reagents/1x dsDNA solution |
| Qubit 4 Fluorometer | ThermoFisher Scientific | Q33238 | Fluorometer |
| Qubit Assay Tubes | Thermo Fisher Scientific | Q32856 | Fluorometer assay tubes |
| RIPA Lysis Buffer | VWR | VWRVN653-100ML | Lysis buffer for protein extraction |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | Trypsin |
| Vortex-Genie 2 | Scientific industries | SI-0236 | Vortex |
| Xfect Transfection Reagent | Takara Bio | 631318 | Transfection reagent |
| genomic data analysis software | QIAGEN | CLC Workbench v21.0. | Data analysis software |
Références
- Vítor, A. C., Huertas, P., Legube, G., de Almeida, S. F. Studying DNA double-strand break repair: an ever-growing toolbox. Frontiers in Molecular Biosciences. 7, (2020).
- Giglia-Mari, G., Zotter, A., Vermeulen, W. DNA damage response. Cold Spring Harbor Perspectives in Biology. 3 (1), 000745(2011).
- Khanna, K. K., Jackson, S. P. DNA double-strand breaks: Signaling, repair and the cancer connection. Nature Genetics. 27 (3), 247-254 (2001).
- vanden Berg, J. G., et al. A limited number of double-strand DNA breaks is sufficient to delay cell cycle progression. Nucleic Acids Research. 46 (19), 10132-10144 (2018).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews Molecular Cell Biology. 18 (8), 495-506 (2017).
- Daley, J. M., Sung, P. 53B. P. 1 BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Molecular and Cellular Biology. 34 (8), 1380-1388 (2014).
- Godin, S. K., Sullivan, M. R., Bernstein, K. A. Novel insights into RAD51 activity and regulation during homologous recombination and DNA replication. Biochemistry and Cell Biology = Biochimie et Biologie Cellulaire. 94 (5), 407-418 (2016).
- Jette, N., Lees-Miller, S. P. The DNA-dependent protein kinase: a multifunctional protein kinase with roles in DNA double strand break repair and mitosis. Progress in Biophysics and Molecular Biology. 117 (0), 194-205 (2015).
- Weterings, E., van Gent, D. C. The mechanism of non-homologous end-joining: A synopsis of synapsis. DNA Repair. 3 (11), 1425-1435 (2004).
- Bhargava, R., Lopezcolorado, F. W., Tsai, L. J., Stark, J. M. The canonical non-homologous end joining factor XLF promotes chromosomal deletion rearrangements in human cells. Journal of Biological Chemistry. 295 (1), 125-137 (2020).
- Gunn, A., Bennardo, N., Cheng, A., Stark, J. M. Correct end use during end joining of multiple chromosomal double strand breaks is influenced by repair protein RAD50, DNA-dependent protein kinase DNA-PKcs, and transcription context. Journal of Biological Chemistry. 286 (49), 42470-42482 (2011).
- Simsek, D., Jasin, M. Alternative end-joining is suppressed by the canonical NHEJ component Xrcc4-ligase IV during chromosomal translocation formation. Nature Structural & Molecular Biology. 17 (4), 410-416 (2010).
- Certo, M. T., et al. Tracking genome engineering outcome at individual DNA breakpoints. Nature Methods. 8 (8), 671-676 (2011).
- Murthy, V., et al. Characterizing DNA repair processes at transient and long-lasting double-strand DNA breaks by immunofluorescence microscopy. JoVE Journal of Visualized Experiments. (136), e57653(2018).
- Wang, J. L., et al. Dissection of DNA double-strand-break repair using novel single-molecule forceps. Nature Structural & Molecular Biology. , (2018).
- Azzam, E. I., Jay-Gerin, J. -P., Pain, D. Ionizing radiation-induced metabolic oxidative stress and prolonged cell injury. Cancer Letters. 327 (0), 48-60 (2012).
- Kuo, L. J., Yang, L. -X. γ-H2AX - A novel biomarker for DNA double-strand breaks. In Vivo. 5, (2008).
- Sanders, J. T., et al. Radiation-induced DNA damage and repair effects on 3D genome organization. Nature Communications. 11 (1), 6178(2020).
- Bellaiche, Y., Mogila, V., Perrimon, N. I-SceI endonuclease, a new tool for studying DNA double-strand break repair mechanisms in Drosophila. Genetics. 152 (3), 1037-1044 (1999).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 disrupt homology dependent double strand break repair by attenuating BRCA1 and BRCA2 expression and foci formation. PLOS Pathogens. 11 (3), 1004687(2015).
- Hu, C., Bugbee, T., Gamez, M., Wallace, N. A. Beta human papillomavirus 8E6 attenuates non-homologous end joining by hindering DNA-PKcs activity. Cancers. 12 (9), 2356(2020).
- Hu, C., Bugbee, T., Dacus, D., Palinski, R., Wallace, N. A. Beta human papillomavirus 8 E6 allows colocalization of non-homologous end joining and homologous recombination repair factors. PLOS Pathogens. 18 (3), 1010275(2022).
- Butler, T. A. J., Paul, J. W., Chan, E. -C., Smith, R., Tolosa, J. M. Misleading westerns: Common quantification mistakes in western blot densitometry and proposed corrective measures. BioMed Research International. 2019, 5214821(2019).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX Phosphorylation on serine 139. Journal of Biological Chemistry. 273 (10), 5858-5868 (1998).
- Taning, C. N. T., Van Eynde, B., Yu, N., Ma, S., Smagghe, G. CRISPR/Cas9 in insects: Applications, best practices and biosafety concerns. Journal of Insect Physiology. 98, 245-257 (2017).
- Ghezraoui, H., et al. Chromosomal translocations in human cells are generated by canonical nonhomologous end-joining. Molecular Cell. 55 (6), 829-842 (2014).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon