É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Usando o sequenciamento da próxima geração para identificar mutações associadas ao reparo de uma quebra de fio dupla induzida pelo CAS9 perto do promotor cd4
Neste Artigo
Resumo
Apresentado aqui é o endonuclease sgRNA/CAS9 e o protocolo de sequenciamento de próxima geração que pode ser usado para identificar as mutações associadas ao reparo de quebra de fios duplos perto do promotor CD4.
Resumo
Quebras duplas de fios (DSBs) no DNA são o tipo mais citotóxico de dano de DNA. Como uma miríade de insultos pode resultar nessas lesões (por exemplo, estresse de replicação, radiação ionizante, dano UV não reparado), os DSBs ocorrem na maioria das células todos os dias. Além da morte celular, DSBs não reparados reduzem a integridade do genoma e as mutações resultantes podem impulsionar a tumorigênese. Esses riscos e a prevalência de DSBs motivam investigações sobre os mecanismos pelos quais as células reparam essas lesões. O sequenciamento da próxima geração pode ser emparelhado com a indução de DSBs por radiação ionizante para fornecer uma ferramenta poderosa para definir precisamente as mutações associadas aos defeitos de reparo do DSB. No entanto, essa abordagem requer sequenciamento de genoma inteiro e proibitivo de custos computacionalmente para detectar o reparo dos DSBs que ocorrem aleatoriamente associados à radiação ionizante. Endonucleases de corte raros, como o I-Sce1, fornecem a capacidade de gerar um único DSB, mas seus locais de reconhecimento devem ser inseridos no genoma do interesse. Como resultado, o local de reparo é inerentemente artificial. Os avanços recentes permitem orientar o RNA (sgRNA) a direcionar uma endonuclease Cas9 para qualquer lócus de genoma de interesse. Isso poderia ser aplicado ao estudo do reparo do DSB tornando o sequenciamento da próxima geração mais econômico, permitindo que ele se concentrasse no DNA flanqueando o DSB induzido pelo Cas9. O objetivo do manuscrito é demonstrar a viabilidade dessa abordagem apresentando um protocolo que pode definir mutações que decorrem da reparação de um DSB upstream do gene CD4. O protocolo pode ser adaptado para determinar mudanças no potencial mutagênico do DSB associados a fatores exógenos, como inibidores de reparação, expressão de proteína viral, mutações e exposições ambientais com requisitos de computação relativamente limitados. Uma vez que o genoma de um organismo tenha sido sequenciado, este método pode ser teoricamente empregado em qualquer lócus genômico e em qualquer modelo de cultura celular desse organismo que pode ser transfeminado. Adaptações semelhantes da abordagem poderiam permitir comparações de fidelidade de reparo entre diferentes loci no mesmo fundo genético.
Introdução
Manter a estabilidade genômica é fundamental para todos os organismos vivos. A replicação precisa do DNA e uma resposta robusta de dano de DNA (DDR) são necessárias para propagar fielmente o material genético 1,2. Os danos no DNA ocorrem regularmente na maioria das células 2,3. Quando esses danos são sentidos, a progressão do ciclo celular é interrompida, e os mecanismos de reparação do DNA são ativados. Quebras duplas de fios no DNA ou DSBs são o tipo mais tóxico e mutagênico de dano de DNA 3,4.
Embora várias vias de sinalização DDR possam reparar essas lesões, as vias de reparo DSB mais estudadas são a recombinação homologous (RH) e a junção final não homóloga (NHEJ). O RH é um caminho em grande parte livre de erros que repara um DSB usando um cromátide irmã como modelo homólogo. Isso tende a acontecer na fase S e G2 de um ciclocelular 5,6,7. O NHEJ é mais propenso a erros, mas pode acontecer ao longo do ciclocelular 8,9. Vários ensaios de repórteres foram desenvolvidos para medir a eficiência de mecanismos específicos de reparo 10,11,12. Esses ensaios tendem a depender da citometria de fluxo para uma medição de alto rendimento da atividade da via de reparo DSB usando GFP ou mCherry como leitura11,13. Embora altamente eficientes, eles dependem de reparos canônicos que ocorrem em um DSB artificialmente introduzido.
Há uma variedade de outros métodos usados para estudar o reparo do DSB. Muitos deles dependem da microscopia de imunofluorescência (IF) 1,14. Se a microscopia detectar focos nucleares discretos representativos de complexos de reparo após os DSBs serem induzidos pela exposição a produtos químicos genotoxicos ou radiação ionizante15,16. O acompanhamento da formação e resolução desses focos fornece uma indicação de iniciação e conclusão do reparo, respectivamente14,17. No entanto, esses métodos de indução de DSB (ou seja, produtos químicos ou radiação ionizante) não causam DSBs em locais definidos no genoma. Também é funcionalmente impossível usá-los para induzir consistentemente apenas um pequeno número (por exemplo, 2-4) de DSBs. Como resultado, os métodos mais comumente utilizados de induzir DSBs causam uma infinidade de lesões distribuídas aleatoriamente ao longo do genoma18. Um pequeno número de DSBs pode ser introduzido inserindo o local de reconhecimento de uma endonuclease de corte raro e expressando a endonuclease pertinente, como o I-Sce119. Infelizmente, a necessária integração de um local de destino impede o exame de DSB em loci genômico endógeno.
Este manuscrito descreve um método para detectar mutações associadas ao reparo de um DSB gerado em um lócus definido pelo usuário. Fornecemos um exemplo representativo da abordagem aplicada para avaliar a capacidade de uma proteína viral de aumentar o número de mutações associadas a um DSB. Especificamente, este manuscrito descreve o uso de um único guia RNA (sgRNA) para direcionar uma endonuclease CAS9 para induzir um DSB no quadro de leitura aberta CD4 humano em queratinócitos de prepúcio humano expressando controle vetorial (HFK LXSN) e HFK que expressa a proteína E6 do vírus papiloma humano tipo 8 (HFK 8E6). O sequenciamento de última geração (NGS) da região em torno do rompimento permite que mutações associadas à reparação da lesão sejam rigorosamente definidas. Esses dados demonstram que a proteína viral causa um aumento de aproximadamente 20 vezes nas mutações durante o reparo do DSB. Também fornece uma caracterização imparcial das consequências mutagênicas dos DSBs em um único lócus sem a necessidade de sequenciamento de genoma inteiro. Em princípio, o protocolo poderia ser facilmente adaptado para comparar o risco relativo de mutações entre loci genoma ou linhas celulares.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Revestimento de células
- Cresça células HFK LXSN e HFK 8E6 em placas de 10 cm em mídia de cultura queratinócito (10 mL/placa) com suplemento de crescimento de queratinócito humano (HKGS) e 1% penicilina/estreptomicina. Cresça células para cerca de 80% de confluência a 37 °C em uma incubadora de jaquetas com 5% de CO2.
- Substitua a mídia cultural por 3 mL de trippsin-EDTA (0,05%, ácido tetraácético de etilenodiamina). Incubar a 37 °C por 3 min. Neutralizar a trippsina com igual volume de soro bovino fetal (FBS) complementou a mídia e transfira células para um tubo de centrífuga de 15 mL. Centrifugar a 300 x g por 5 min.
- Células resuspend com 10 mL de mídia de cultura queratinócito com HKGS. Determine a concentração de células com um hemócito.
- Placa 4 x 105 células/6 cm de placas (semente duas placas para HFK LXSN e duas placas para HFK 8E6) em 4mL de mídia de cultura queratinócito com HKGS e 1% penicilina e estreptomicina. Cresça a 37 °C em uma incubadora de jaquetas com 5% de CO2.
NOTA: A análise das células HFK foi escolhida para este protocolo por duas razões. Primeiro, hfk é uma linha celular difícil de transfectar. Assim, demonstrando que o protocolo funciona nesta linha celular, são fornecidas evidências de que ele provavelmente funcionará em células mais comumente utilizadas e mais facilmente transfectadas. Em segundo lugar, dados publicados anteriormente demonstram que uma proteína viral (8E6) dificulta a reparação de quebras duplas de fios no DNA 20,21,22. Assim, comparar HFK LXSN e HFK 8E6 nos permite demonstrar a capacidade do ensaio de detectar aumentos de mutações associadas a uma redução na capacidade de reparo celular.
2. Transfecção
- No dia da transfecção (24h após o revestimento), substitua a mídia por 3 mL de mídia suplementada sem antibióticos. Incubar por 2 h a 37 °C em uma incubadora de jaquetas.
- Células transfeitidentais com reagentes de transfecção adequados à base de lipídios de acordo com as instruções do fabricante.
- Reagentes de transfecção quentes à temperatura ambiente e pipeta suavemente antes de usar.
- Para cada linha celular (HFK LXSN e HFK 8E6), coloque a quantidade apropriada de tampão de transfecção (conforme orientado pelo fabricante) em um tubo de centrífuga estéril de 1,5 mL (Tubo 1). Inclua outro tubo com a mesma quantidade de tampão de transfecção (transfecção simulada ou Tubo 2).
- Adicione 2 μg de DNA plasmídeo expressando CAS9/sgRNA visando CD4 humano (px330-CD4, 5'- GGCGTATCTGTGTGAGGACT) ao Tubo 1 a partir da etapa 2.2.2. Pipeta suavemente para misturar completamente. Adicione o volume igual de água estéril ao tubo 2.
- Inclua uma placa de controle com reagentes de transfecção sozinhos (sem plasmídeo) para cada linha celular.
NOTA: A segunda placa serve como um controle negativo no experimento, permitindo que o usuário confirme que a transfecção com o CAS9/sgRNA não é responsável por quaisquer mutações. - Adicione a quantidade apropriada do reagente de transfecção (conforme indicado pelo fabricante) ao tubo com mistura de DNA (Tubo 1) da etapa 2.2.3 e a transfecção simulada (Tubo 2) da etapa 2.2.2. Pipeta suavemente para misturar completamente. Incubar à temperatura ambiente por 15-30 min para permitir tempo suficiente para que os complexos se formem.
- Adicione a mistura de transfecção em termos de gota à placa. Abale suavemente a cultura por 1 min para distribuir uniformemente a mistura de transfecção.
- Incubar por 48 horas após a transfecção para permitir a expressão CAS9.
- Células de colheita por trippsinização.
- Substitua a mídia cultural por 1 mL de trippsin-EDTA (0,05%, ácido tetraácético de etilenodiamina). Incubar a 37 °C por 3 min. Neutralizar a trippsina com o mesmo volume de mídia suplementada da FBS.
- Para cada placa de células, transfira a suspensão celular para dois tubos de microcentrifuuagem com alíquotas iguais. Centrifugar a 300 x g por 5 min.
- Resuspenque a pelota celular de um tubo na etapa 2.4.2 em 1 mL de salina tamponada de fosfato (PBS) para sequenciamento. Resuspend o outro tubo a partir do passo 2.4.2 com PBS gelado para imunoblot.
- Colhe as células inteiras para imunoblot.
- Centrifugar o tubo a 300 x g por 5 min. Descarte o supernatante.
- Adicione 100 μL de tampão de ensaio de radioimunoprecipitação (tampão de lise RIPA) misturado com 1 % inibidor de protease e 1% inibidor de fosfatase no tubo, misture bem com uma pipeta e incubar por 10 minutos no gelo.
NOTA: O buffer de lise RIPA contém 10 mM Tris-HCl, pH 8.0; 1 mM EDTA; 0,5 mM EGTA; 1% Tritão X-100; Desoxicolato de sódio de 0,1%; 0,1% SDS; 140 mM NaCl, e água deionizada. - Centrifugar lysates a 13.000 x g por 10 min. Colete supernantes para imunoblot.
3. Medindo a expressão CAS9 via imunoblot
- Determine a concentração proteica com um ensaio de ácido bicinchonínico (BCA) de acordo com as instruções do fabricante.
- Execute 20 μg de proteína de cada amostra nos poços de um gel de tris-acetato tris-acetato de 3%-8% para 150 min e transferência semidária (10 V por 30 min e depois 25 V por 12 min) para a membrana de difluoreto polivinino.
- Depois de bloquear a membrana em 5% de leite seco sem gordura em PBS com 0,1% de interpolação (PBST) por 1h à temperatura ambiente, adicione anticorpos anti-CAS9 (1:1000) e anti-GAPDH (1:1000). Incubar a 4 °C durante a noite.
- Depois de lavar a membrana com PBST, incubar a membrana com anticorpo secundário em 5% de leite seco sem gordura em PBST por 1h em temperatura ambiente.
- Imagem a mancha e determinar o nível CAS9 por densitometria23. Veja a Figura 1 para uma mancha representativa.
NOTA: A detecção de focos H2AX (S139) fosforiladas por microscopia de imunofluorescência pode ser usada para validar a atividade CAS914. Um baixo número de focos distintos (tipicamente 1-4 focos) são esperados dependendo da posição do ciclo celular, se mutações no local alvo CAS9 impedem novos cortes e quantas cópias do local de corte CAS9 existem no genoma de interesse. Uma imagem representativa é mostrada na Figura 2.
4. Extração de ácido nucleico e geração de amplicon
- Extrair DNA de amostras de células da etapa 2.4.3 usando um kit de extração de DNA de alto peso molecular, conforme especificado pelo fabricante.
- Resuspend primers com solvente indicado de acordo com a folha de dados. Diluir com o mesmo reagente a 20 μM e agrupar primers de 20 μM nas piscinas indicadas.
NOTA: O pool primer está listado na Tabela Suplementar 1. - Crie uma mistura PCR Master usando uma polimerase Taq de amplificação longa para cada pool de primer de 20 μM conforme especificado na Tabela 1.
- Adicione 21 μL dos misturas principais para separar os tubos PCR.
- Adicione 4 μL da amostra alvo (100 ng/μL) da etapa 4.1 aos tubos de ensaio PCR contendo tubos de ensaio mastermix e cap. Certifique-se de reações separadas para cada pool de primer.
- Vórtice para misturar tubos de ensaio PCR e centrífuga (giro rápido) para remover gotículas das tampas do tubo.
- Coloque os tubos PCR em uma máquina de cicloviário térmico convencional.
- Programe a máquina PCR conforme especificado na Tabela 2.
- Execute o programa em um cicloviário térmico.
5. Limpeza do PCR
- Remova os primers das reações do PCR usando um sistema de limpeza PCR baseado em contas.
- Remova as contas de limpeza da geladeira 30 minutos antes de usar.
- As contas de vórtice bem antes de usar e garantem que todas as contas sejam resuspendidas.
- Adicione 30 μL (1,2x) de contas resuspended a cada poço de uma placa de poço profundo de 96 poços.
- Adicione 25 μL de reação pcr a poços que contêm contas.
- Coloque a placa em um agitador de pratos a 2000 rpm por 2 min.
- Deixe a placa permanecer em temperatura ambiente por 5 minutos após o tremor.
- Coloque a placa de poço profundo em um ímã de placa de 96 poços e incubar por 2 minutos.
- Remova e descarte o supernatante sem as contas perturbadoras.
- Enquanto a placa permanece no ímã, adicione 180 μL de 80% de etanol e incuba para 30 s. Remova e descarte o supernatante.
- Repita o passo 5.1.9.
- Com uma pipeta de 10 μL, remova e descarte qualquer líquido restante dos poços.
- Deixe as contas secarem à temperatura ambiente por 10 minutos.
- Adicione 20 μL de água livre de nuclease aos poços que contêm contas e remova a placa do ímã.
- Agite a placa a 2000 rpm por 2 min à temperatura ambiente.
- Incubar a placa em temperatura ambiente por 5 minutos.
- Coloque a placa em um suporte de ímã e incubar por 2 minutos em temperatura ambiente.
- Remova o supernatante em uma segunda placa PCR rotulada. Isto contém o DNA limpo.
- Meça a concentração de cada reação com um Fluorômetro.
- Certifique-se de que os reagentes do fluorômetro dsDNA estão em temperatura ambiente.
- Configure tubos de ensaio fluorométricos mais dois tubos adicionais para padrões.
- Adicione 199 μL de 1x dsDNA solução de trabalho para todos, exceto dois tubos. Adicione 190 μL da solução de trabalho aos dois últimos tubos.
- Adicione 10 μL das duas normas (incluídas na Tabela de Materiais) para separar os tubos de ensaio.
- Adicione 1 μL de cada reação pcr aos tubos de mastermix fluorômetro.
- Tubos de vórtice para misturar e incubar à temperatura ambiente por 2 minutos.
- Na tela inicial do fluorômetro, selecione o botão com o uso do kitin de ensaio (1x dsDNA) e selecione Ler Padrões e Executar Amostras.
- Insira o tubo padrão 1, selecione o botão Ler e repita para o padrão 2.
- Após a etapa 5.2.6, repita para uma amostra, selecione um volume amostral de 1 μL e a concentração resultante será fornecida.
- Repita o passo 5.2.7 para as amostras restantes.
- Calcule a molaridade projetada de todas as reações e concentrações iguais de reações de cada amostra individual separadamente (um último pool por amostra) usando a equação abaixo.
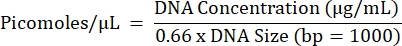
- Repita os passos 5.2.1 a 5.2.7 para obter a concentração final do pool.
- Verifique a piscina de amplicon em uma máquina de eletroforese capilar/gel de agarose conforme especificado pelo fabricante.
- Prepare tubos de eletroforese capilar para o número apropriado de amostras. Adicione 7 μL do buffer de DNA especificado pelo fabricante.
- Adicione 4 μL da piscina de amplicon ao tubo contendo tampão de DNA.
- Coloque tubos na máquina de eletroforese e execute a máquina conforme especificado pelo fabricante para dsDNA.
- Veja as imagens de gel de eletroforese garantindo que as bandas se localizem para ~5 kb (tamanho de amplicons).
- Calcule a molaridade projetada de todas as reações e concentrações iguais de reações de cada amostra individual separadamente (um último pool por amostra) usando a equação abaixo.
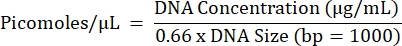
6. Preparação da biblioteca
- Diluir piscinas de amostras da etapa 5.3.1 para 0,2 ng/μL para preparação da biblioteca.
- Usando um kit de preparação de biblioteca de baixa entrada compatível com sequências curtas (300bp) prepare bibliotecas usando combinações de índice únicas para cada pool de amostras criado na etapa 5.3 seguindo as instruções do fabricante.
NOTA: Siga as instruções do fabricante para selecionar sequências de índice. Todos os índices favoráveis ao kit de preparação da biblioteca funcionarão para as amostras. - Após a preparação da biblioteca, acumule todas as amostras de acordo com as instruções do fabricante.
NOTA: Antes de criar o pool de biblioteca, calcule o número de leituras necessárias para a cobertura de 250x da sequência de destino e certifique-se de que o cartucho de sequenciamento selecionado possa fornecer cobertura adequada para cada amostra incluída. Para o total de 0,5Mb, isso equivalerá a leituras de 1M.
- Usando um kit de preparação de biblioteca de baixa entrada compatível com sequências curtas (300bp) prepare bibliotecas usando combinações de índice únicas para cada pool de amostras criado na etapa 5.3 seguindo as instruções do fabricante.
- Prepare a piscina da biblioteca para sequenciamento.
- Descongele e prepare um cartucho de 300 ciclos e reagentes de sequenciamento.
- Desnaturar e diluir a piscina de sequenciamento criada na etapa 5.1.1 de acordo com as instruções do fabricante do sequenciador.
- Adicione piscina de biblioteca desnaturada e diluída para sequenciar reagentes e Execute a máquina de sequenciamento conforme especificado pelo fabricante.
NOTA: Consulte a tabela anexada 3 para obter problemas de tiroteios.
7. Análise de dados
NOTA: Todas as etapas de dados são realizadas no software de análise de dados genômicos. Os parênteses indicam a entrada do usuário. Maior que o sinal indica a ordem dos cliques do mouse para qualquer passo (por exemplo,1º clique do mouse>2º clique do mouse)
- Importe as leituras clicando em Criar Software > Importar > Illumina > Selecionar arquivos > próximo > Selecione o local para salvar > acabamento. As leituras agora aparecerão no software.
- Aparar e filtrar as leituras.
- No software de análise de dados de sequência profunda, corte os parâmetros padrão de leituras brutas.
- Destaque as leituras e clique em Caixa de ferramentas > Prepare dados de sequenciamento > Trim Reads > Próximo > Próxima > Próxima > Próxima > Salve > Próximo > (Selecione local a salvar) > Acabamento
- Mapear as leituras aparadas para referência.
- Mapa aparado lê para a sequência de referência usada na etapa 4.1 usando uma pontuação de correspondência de 2, custo de incompatibilidade de 3 e custos de inserção/exclusão de 2. Certifique-se de que a fração de comprimento está acima de 0,7 e que a fração de similaridade está igual ou superior a 0,8.
- Destaque o arquivo de leitura aparada e clique em Caixa de ferramentas > Análise de Resequenciamento > Mapa Lê para Referência > Próxima > (Selecione sequência de referência) > Próxima > (Garantir que os parâmetros sejam indicados acima) > Próxima > Salvar > (Selecionar local para salvar) > Acabamento.
- Extrair variantes e indels.
- Usando um chamador indel apropriado, extraia indels usando um limiar de valor p de 0,005 ou inferior e um número máximo de incompatibilidades de 3.
- Destaque o arquivo de leitura mapeado e clique em Caixa de ferramentas > Análise de Resequenciamento > Detecção de Variantes > Indels e Variantes Estruturais > Próxima > (Certifique-se de que o significado necessário é a entrada > Próxima > Salvar > (Selecione local a salvar) > Acabamento.
- Usando um chamador de variante apropriado, ligue para variantes do mapeamento de leitura usando um significado de 5%.
- Destaque o arquivo de leitura mapeado e clique em Caixa de ferramentas > Análise de Resequenciamento > Detecção de Variantes > detecção de variantes de baixa frequência > Próxima > (Certifique-se de que o significado necessário é a entrada > Próxima > Próxima > Salvar > (Selecione local a salvar) > Acabamento.
NOTA: Certifique-se de prestar contas da ploididade do genoma hospedeiro nos chamadores indel e variantes. Não extraia mutantes abaixo de 5%. Esse limite é responsável por erros de PCR e sequenciamento associados ao ensaio. A normalização (com base na detecção de imunoblot de CAS9) deve ser feita ajustando a cobertura de sequenciamento. Por exemplo, se a amostra A tiver o dobro da eficiência de transfecção da amostra B, então 50% das leituras da amostra A devem ser usadas para análise. Isso deve ser feito por amostragem aleatória e não reduzir a cobertura para qualquer amostra abaixo de 100x.
Access restricted. Please log in or start a trial to view this content.
Resultados
Três resultados representativos são apresentados para este protocolo. Figura 1 é um imunoblot confirmando expressão de CAS9 no controle HFK (LXSN) e HFK expressando beta-HPV 8E6 (8E6). 48 h após a transfecção, os lises celulares inteiros foram colhidos e, posteriormente, sondados com um anticorpo anti-CAS9 (ou GAPDH como controle de carga). O resultado mostra que hfk LXSN e HFK 8E6 estão expressando quantidade semelhante de CAS9 indicando que a efici?...
Access restricted. Please log in or start a trial to view this content.
Discussão
Além da profundidade das informações fornecidas, existem várias vantagens para este método. Primeiro, o reparo do DSB, em teoria, pode ser avaliado em qualquer loci genômico sem modificar o genoma da célula de interesse. Em segundo lugar, o acesso à análise de reparo do NGS é aumentado pelo custo reduzido e pelo esforço computacional proporcionado pela fabricação e análise de um único DSB direcionado a uma área definida. Finalmente, com os genomas de organismos adicionais se tornando rotineiramente dispon...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
A pesquisa relatada neste manuscrito foi apoiada pelo Instituto Nacional de Ciências Médicas Gerais dos Institutos Nacionais de Saúde (P20GM130448) (NAW e RP); Instituto Nacional de Câncer dos Institutos Nacionais de Saúde (NCI R15 CA242057 01A1); Centro de Pesquisa do Câncer Johnson na Universidade Estadual do Kansas; e o Departamento de Defesa dos EUA (CMDRP PRCRP CA160224 (NAW)). Agradecemos a KSU-CVM Confocal Core e Joel Sanneman pela nossa microscopia de imunofluorescência. O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais dessas agências de financiamento.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 6 Well Tissue Culture Plate | Celltreat | 229106 | Cell culture plate |
| BCA Kit | VWR | 89167-794 | BCA assay kit |
| Centrifuge 5910 R | Eppendorf | 2231000772 | Tabletop Centrifuge |
| CLC Genomic Workbench | Qiagen | 832001 | deep sequence data analysis software/indel caller/variant caller |
| Digital Microplate Genie pulse | Scientific industries | SI-400A | Plate shaker |
| DYKDDDDK Tag Monoclonal Antibody (FG4R) | ThermoFisher Scientific | MA191878 | Anti-FLAG antibody |
| Epilife CF Kit | ThermoFisher Scientific | MEPICF500 | Cell cultrue media and supplements |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | Cell culture supplement |
| Goat anti-Rabbit IgG | ThermoFisher Scientific | A-11012 | Secondary antibody |
| HighPrep PCR Clean-up system | MagBio | AC-60005 | Bead-based PCR cleanup kit |
| KAPA HiFi HotStart ReadyMix PCR Kit | KAPA Biosystems | KK2600 | PCR mastermix/PCR assay |
| MagAttract HMW DNA kit | Qiagen | 67563 | High Molecular Weight DNA extraction kit |
| Magnetic Stand-96 | Thermo Fisher Scientific | AM10027 | 96-Well Magnetic Rack |
| MiniAmp Thermal Cycler | Applied Biosystems | A37834 | Thermal Cycler |
| Miseq | Illumina | SY-410-1003 | Sequencer |
| Miseq v2 300 cycle reagent kit | Illumina | MS-102-2002 | 300-cycle cartridge/sequencing reagents |
| Nextera XT DNA Library Prep kit | Illumina | FC-131-1024 | Library preparation kit |
| Nextera XT Kit v2 Set A | Illumina | 20027215 | Indexes |
| Nunc 96-well polypropylene DeepWell Stroage plates | Thermo Fisher Scientific | 260251 | deep well 96-well plates |
| Penicillin-Streptomycin Solution (100X) | Calsson Labs | PSL02-6X100ML | Antibiotics for cell culture |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | PBS |
| px330-CD4 | Addgen | 136938 | SgRNA/CAS9 plasmids targeting 5’- GGCGTATCTGTGTGAGGACT |
| QIAxcel Advanced System | Qiagen | 9001941 | capillary electrophersis machine |
| QIAxcel DNA screening kit | Qiagen | 929004 | DNA buffer/ capillary electrophersis tubes |
| Qubit 1x ds HS Assay Kit | ThermoFisher Scientific | Q23851 | Fluorometer reagents/1x dsDNA solution |
| Qubit 4 Fluorometer | ThermoFisher Scientific | Q33238 | Fluorometer |
| Qubit Assay Tubes | Thermo Fisher Scientific | Q32856 | Fluorometer assay tubes |
| RIPA Lysis Buffer | VWR | VWRVN653-100ML | Lysis buffer for protein extraction |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | Trypsin |
| Vortex-Genie 2 | Scientific industries | SI-0236 | Vortex |
| Xfect Transfection Reagent | Takara Bio | 631318 | Transfection reagent |
| genomic data analysis software | QIAGEN | CLC Workbench v21.0. | Data analysis software |
Referências
- Vítor, A. C., Huertas, P., Legube, G., de Almeida, S. F. Studying DNA double-strand break repair: an ever-growing toolbox. Frontiers in Molecular Biosciences. 7, (2020).
- Giglia-Mari, G., Zotter, A., Vermeulen, W. DNA damage response. Cold Spring Harbor Perspectives in Biology. 3 (1), 000745(2011).
- Khanna, K. K., Jackson, S. P. DNA double-strand breaks: Signaling, repair and the cancer connection. Nature Genetics. 27 (3), 247-254 (2001).
- vanden Berg, J. G., et al. A limited number of double-strand DNA breaks is sufficient to delay cell cycle progression. Nucleic Acids Research. 46 (19), 10132-10144 (2018).
- Chang, H. H. Y., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nature Reviews Molecular Cell Biology. 18 (8), 495-506 (2017).
- Daley, J. M., Sung, P. 53B. P. 1 BRCA1, and the choice between recombination and end joining at DNA double-strand breaks. Molecular and Cellular Biology. 34 (8), 1380-1388 (2014).
- Godin, S. K., Sullivan, M. R., Bernstein, K. A. Novel insights into RAD51 activity and regulation during homologous recombination and DNA replication. Biochemistry and Cell Biology = Biochimie et Biologie Cellulaire. 94 (5), 407-418 (2016).
- Jette, N., Lees-Miller, S. P. The DNA-dependent protein kinase: a multifunctional protein kinase with roles in DNA double strand break repair and mitosis. Progress in Biophysics and Molecular Biology. 117 (0), 194-205 (2015).
- Weterings, E., van Gent, D. C. The mechanism of non-homologous end-joining: A synopsis of synapsis. DNA Repair. 3 (11), 1425-1435 (2004).
- Bhargava, R., Lopezcolorado, F. W., Tsai, L. J., Stark, J. M. The canonical non-homologous end joining factor XLF promotes chromosomal deletion rearrangements in human cells. Journal of Biological Chemistry. 295 (1), 125-137 (2020).
- Gunn, A., Bennardo, N., Cheng, A., Stark, J. M. Correct end use during end joining of multiple chromosomal double strand breaks is influenced by repair protein RAD50, DNA-dependent protein kinase DNA-PKcs, and transcription context. Journal of Biological Chemistry. 286 (49), 42470-42482 (2011).
- Simsek, D., Jasin, M. Alternative end-joining is suppressed by the canonical NHEJ component Xrcc4-ligase IV during chromosomal translocation formation. Nature Structural & Molecular Biology. 17 (4), 410-416 (2010).
- Certo, M. T., et al. Tracking genome engineering outcome at individual DNA breakpoints. Nature Methods. 8 (8), 671-676 (2011).
- Murthy, V., et al. Characterizing DNA repair processes at transient and long-lasting double-strand DNA breaks by immunofluorescence microscopy. JoVE Journal of Visualized Experiments. (136), e57653(2018).
- Wang, J. L., et al. Dissection of DNA double-strand-break repair using novel single-molecule forceps. Nature Structural & Molecular Biology. , (2018).
- Azzam, E. I., Jay-Gerin, J. -P., Pain, D. Ionizing radiation-induced metabolic oxidative stress and prolonged cell injury. Cancer Letters. 327 (0), 48-60 (2012).
- Kuo, L. J., Yang, L. -X. γ-H2AX - A novel biomarker for DNA double-strand breaks. In Vivo. 5, (2008).
- Sanders, J. T., et al. Radiation-induced DNA damage and repair effects on 3D genome organization. Nature Communications. 11 (1), 6178(2020).
- Bellaiche, Y., Mogila, V., Perrimon, N. I-SceI endonuclease, a new tool for studying DNA double-strand break repair mechanisms in Drosophila. Genetics. 152 (3), 1037-1044 (1999).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 disrupt homology dependent double strand break repair by attenuating BRCA1 and BRCA2 expression and foci formation. PLOS Pathogens. 11 (3), 1004687(2015).
- Hu, C., Bugbee, T., Gamez, M., Wallace, N. A. Beta human papillomavirus 8E6 attenuates non-homologous end joining by hindering DNA-PKcs activity. Cancers. 12 (9), 2356(2020).
- Hu, C., Bugbee, T., Dacus, D., Palinski, R., Wallace, N. A. Beta human papillomavirus 8 E6 allows colocalization of non-homologous end joining and homologous recombination repair factors. PLOS Pathogens. 18 (3), 1010275(2022).
- Butler, T. A. J., Paul, J. W., Chan, E. -C., Smith, R., Tolosa, J. M. Misleading westerns: Common quantification mistakes in western blot densitometry and proposed corrective measures. BioMed Research International. 2019, 5214821(2019).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX Phosphorylation on serine 139. Journal of Biological Chemistry. 273 (10), 5858-5868 (1998).
- Taning, C. N. T., Van Eynde, B., Yu, N., Ma, S., Smagghe, G. CRISPR/Cas9 in insects: Applications, best practices and biosafety concerns. Journal of Insect Physiology. 98, 245-257 (2017).
- Ghezraoui, H., et al. Chromosomal translocations in human cells are generated by canonical nonhomologous end-joining. Molecular Cell. 55 (6), 829-842 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados