Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Bestimmung der dreigliedrigen Wechselwirkung zwischen zwei Monomeren eines MADS-Box-Transkriptionsfaktors und eines Calcium-Sensorproteins mittels BiFC-FRET-FLIM-Assay
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir eine Methode zur Visualisierung der ternären Komplexbildung zwischen drei Proteinpartnern unter Verwendung fluoreszenzmarkierter Proteine durch BiFC-basierten FRET-FLIM-Assay vor. Diese Methode ist wertvoll für die Untersuchung von Protein-Protein-Interaktionskomplexen in vivo.
Zusammenfassung
Protein-Protein-Interaktionen sind ein integraler Bestandteil aller biologischen Prozesse in den Zellen, da sie eine entscheidende Rolle bei der Regulierung, Aufrechterhaltung und Änderung zellulärer Funktionen spielen. Diese Wechselwirkungen sind an einer Vielzahl von Phänomenen wie Signaltransduktion, Pathogenantwort, Zell-Zell-Interaktionen, Stoffwechsel- und Entwicklungsprozessen beteiligt. Im Falle von Transkriptionsfaktoren können diese Wechselwirkungen zu einer Oligomerisierung von Untereinheiten führen, die in spezifischen subzellulären Kontexten wie Kern, Zytoplasma usw. sequestriert werden, was wiederum einen tiefgreifenderen Einfluss auf die Expression der nachgeschalteten Gene haben könnte. Hier demonstrieren wir eine Methodik zur Visualisierung der dreigliedrigen In-vivo-Interaktion unter Verwendung der bimolekulären Fluoreszenzkomplementierung (BiFC) basierenden Förster-Resonanz-Energieübertragung (FRET) mit Fluoreszenz-Lifetime-Imaging (FLIM). Zwei der für diese Demonstration ausgewählten Proteine interagieren als BiFC-Partner, und ihre rekonstituierte Fluoreszenzaktivität wird verwendet, um FRET-FLIM mit dem dritten Partner zu testen. Vier bis fünf Wochen alte, in der Wachstumskammer angebaute Nicotiana benthamiana-Pflanzen wurden als Modellpflanzensystem für diese Demonstration verwendet.
Einleitung
Protein-Protein-Interaktionen (PPIs) bilden die Grundlage für das reibungslose Funktionieren der eukaryotischen Zellen, indem sie verschiedene Stoffwechsel- und Entwicklungsprozesse regulieren. Einige PPIs sind stabil, während andere vorübergehender Natur sind. Die Wechselwirkungen können basierend auf der Anzahl und Art der Mitglieder in der Wechselwirkung kategorisiert werden, z. B. dimer, trimer, tetramer homomer und heteromer1. Die Identifizierung und Charakterisierung von Proteininteraktionen kann zu einem besseren Verständnis von Proteinfunktionen und regulatorischen Netzwerken führen.
Transkriptionsfaktoren sind Proteine, die an regulatorischen Funktionen beteiligt sind. Sie regulieren die Transkriptionsrate ihrer nachgeschalteten Gene, indem sie an die DNA binden. Manchmal ist die Oligomerisierung oder Bildung von Komplexen höherer Ordnung durch Proteine eine Voraussetzung für die Ausübung ihrer Funktionen2. Pflanzliche MADS-Box-Transkriptionsfaktoren sind homöotische Gene, die verschiedene Prozesse wie Blütenübergang, Blütenorganentwicklung, Befruchtung, Samenentwicklung, Seneszenz und vegetative Entwicklung regulieren. Es ist bekannt, dass sie Komplexe höherer Ordnung bilden, die an die DNAbinden 3,4. Die Untersuchung von PPI-Netzwerken zwischen Transkriptionsfaktoren und ihren Interaktoren liefert Einblicke in die Komplexität, die der transkriptionellen Regulation zugrunde liegt.
Die transiente Proteinexpression in Nicotiana benthamiana war ein beliebter Ansatz, um proteinlokalisation oder Protein-Protein-Interaktionen in vivozuuntersuchen 5. BiFC und FRET sind Methoden zur Untersuchung von Protein-Protein-Interaktionen in vivo unter Verwendung fluoreszierender Reportersysteme6. Es wurde gezeigt, dass eine Kombination dieser beiden Techniken die Wechselwirkung zwischen drei Proteinen aufdeckt7. FRET wird mit Akzeptor-Photobleiching, sensibilisierter Emission und Fluoreszenz-Lifetime-Imaging (FLIM) -Technik gemessen. Der FLIM-basierte FRET-Assay hat sich zu einem Werkzeug entwickelt, das eine genaue Quantifizierung und raumzeitliche Spezifität für Energietransfermessungen zwischen zwei Molekülen basierend auf ihrer Fluoreszenzlebensdauer ermöglicht8. FLIM misst die Zeit, in der ein Fluorophor in einem angeregten Zustand bleibt, bevor es ein Photon emittiert, und ist besser als Techniken, die nur Intensitätsmessungen verwenden9,10. Neben heterologen Systemen wie Nicotiana benthamiana und Zwiebel-Epidermisschalen haben neuere Berichte die Verwendung von Arabidopsis-Wurzeln und jungen Reissämlingen usw. für die In-vivo-Analyse von Protein-Protein-Interaktionen unter nativen Bedingungen gezeigt11,12.
Neben einem geeigneten Expressionssystem ist auch die Auswahl der interagierenden Partner für BiFC- und FRET-Assays entscheidend für den Erfolg dieses Experiments. Der PPI zwischen Partnern, die in der BiFC-Konfiguration verwendet werden, sollte vor ihrer Verwendung als konjugierter Partner im FRET-Experiment13mit geeigneten Kontrollen validiert werden. BiFC nutzt die strukturelle Komplementierung von N- und C-terminalen Teilen des fluoreszierenden Proteins. Eine häufige Einschränkung in den meisten, wenn nicht allen fluoreszierenden Proteinen, die in BiFC-Assays verwendet werden, war die Selbstorganisation zwischen den beiden abgeleiteten nichtfluoreszierenden Fragmenten, die zu einer falsch positiven Fluoreszenz beitrug und das Signal-Rausch-Verhältnis (S / N)14verringerte. Jüngste Entwicklungen, einschließlich Punktmutationen oder der Position der Spaltung des fluoreszierenden Proteins, haben zu BiFC-Paaren mit erhöhter Intensität, höherer Spezifität, hohem S /N-Verhältnis15,16geführt. Diese fluoreszierenden Proteine können je nach Eignung des Experiments auch zur Durchführung von BiFC verwendet werden.
Traditionell wurden CFP und YFP als Donor- und Akzeptorpaar in FRET-Experimentenverwendet 17. Es wurde jedoch festgestellt, dass YFP oder m-Citrin aufgrund der hohen Quantenausbeute (QY) während der nativen Expression von Zielproteinen im Arabidopsis-Wurzelsystem bessere FRET-Donatoren sind (wenn sie mit RFP als Akzeptor verwendet werden). Die Auswahl von Promotoren (konstitutiv versus native/endogen) und Fluorophoren spielt ebenfalls eine entscheidende Rolle bei der Entwicklung eines erfolgreichen BiFC-FRET-FLIM-Experiments. Es ist wichtig zu beachten, dass sich die Effizienz von FRET-Donatoren und die Eignung von FRET-Paaren tendenziell mit der Änderung des Promotors und des biologischen Systems, das für die Expression verwendet wird, ändern. Die QY des Fluorophors, die sich auf seine Helligkeit bezieht, hängt vom pH-Wert, der Temperatur und dem verwendeten biologischen System ab. Wir schlagen vor, dass diese Kriterien gründlich berücksichtigt werden, bevor das Fluorophorpaar für das FRET-Experiment ausgewählt wird. Das biologische System, die Promotoren und die Proteine, die für dieses Protokoll verwendet wurden, funktionierten gut mit CFP-YFP-Fluorophoren für das BiFC FRET-FLIM-Experiment.
In der vorliegenden Studie integrieren wir die Funktion von FLIM, um die Wechselwirkung zwischen drei Proteinmolekülen mit BiFC-basierter FRET zu visualisieren. Bei dieser Technik werden zwei Proteine mit gespaltenem YFP-Protein und das dritte Protein mit CFP markiert. Da wir daran interessiert waren, die Interaktion eines MADS-Box-Protein (M) Homodimers mit einem Calcium-Sensorprotein (C) zu untersuchen, wurden diese Proteine mit fluoreszierenden Proteinen in pSITE-1CA- und pSITE-3CA-Vektorenmarkiert 18. Zwei der interagierenden Partner wurden in diesem Assay mit N- und C-terminalen Teilen des YFP in pSPYNE-35S- und pSPYCE-35S-Vektoren19markiert, und ihre Interaktion führt zur Rekonstitution des funktionellen YFP, das als FRET-Akzeptor fungiert, zum dritten interagierenden Partner, der mit CFP (als FRET-Donor fungierend) markiert ist(Abbildung 1 ). In diesem speziellen Fall wurde der PPI zwischen zwei M-Monomeren und zwischen M und C validiert, indem BiFC in drei verschiedenen Systemen zusammen mit dem Hefe-Zwei-Hybrid-System durchgeführt wurde. Diese Vektoren wurden durch Elektroporation in den Stamm Agrobacterium tumifaciens GV3101 mobilisiert. Der Stamm GV3101 hat ein entwaffnetes Ti-Plasmid pMP90 (pTiC58DT-DNA) mit Gentamicin-Resistenz20. Ein p19 Agrobacterium-Stamm wurde zusammen mit allen Infiltrationen hinzugefügt, um eine Transgen-Stummschaltung zu verhindern21. Wir empfehlen, dass die drei Proteine auch in entgegengesetzten Konformationen verwendet werden sollten, um die dreigliedrigen Wechselwirkungen zu validieren.
Bei dieser Technik haben wir FLIM eingesetzt, wobei zunächst die Fluoreszenzlebensdauer des Spenders (ungequetschte Spenderlebensdauer) in Abwesenheit eines Akzeptors gemessen wird. Danach wird seine Lebensdauer in Gegenwart des Akzeptors gemessen (abgeschreckte Spenderlebensdauer). Dieser Unterschied in der Donorfluoreszenzlebensdauer wird verwendet, um die FRET-Effizienz zu berechnen, die von der Anzahl der Photonen abhängt, die eine Verringerung der Fluoreszenzlebensdauer aufweisen. Im Folgenden finden Sie ein detailliertes Protokoll zur Bestimmung der Bildung eines ternären Komplexes zwischen drei beliebigen Proteinen, indem die fluoreszierenden markierten Proteine in Nicotiana benthamiana vorübergehend exprimiert und ihre Interaktion durch BiFC-FRET-FLIM untersucht werden.
Protokoll
1. Klonen von Genen in Eintrags- und Zielvektoren (Abbildung 2)
- Amplifizieren Sie die kodierende Sequenz (CDS) der interessierenden Gene (in unserem Fall M- und C-Gene) durch PCR und klonieren Sie sie in geeigneten Eingangsvektoren (z. B. pENTR/D-TOPO-Vektor; siehe Tabelle 1 für die in diesem Experiment verwendeten Vektoren).
- Züchten Sie die Klone auf Platten, die Antibiotika enthalten. Validierung von Klonen, die auf den Antibiotika ausgewählt werden, durch Restriktionsverdauung und DNA-Sequenzierung22,23.
- Mobilisieren Sie die rekonstituierten CDS von Eingangsklons zu den Zielvektoren (pSPYNE-35S, pSPYCE-35S, pSITE-1CA und pSITE-3CA) und bestätigen Sie den Transfer von Sequenzen vom Eingang zu den Zielvektoren durch Restriktionsenzymaufschluss.
ANMERKUNG: Alle in diesem Versuch verwendeten Vektoren sind in Tabelle 1aufgeführt. - Schließlich transformieren Agrobacterium GV3101 (pMP90 (GentR)) Zellen mit den Zielvektoren durch Elektroporation (Abbildung 3)24.
2. Wachstumsbedingungen für Nicotiana benthamiana Pflanzen
HINWEIS: Wachsen Sie Nicotiana-Pflanzen bis zum 4-6-Blatt-Stadium unter Kontrollbedingungen.
- Um Nicotiana-Pflanzen anzubauen, bereiten Sie die Bodenmischung vor, indem Sie handelsübliche Bodenmischungen mit Kokos und Kompost im Verhältnis 2: 1: 1 mischen.
- Verteilen Sie eine 1 Zoll dicke Schicht dieser Bodenmischung in einer Kunststoffschale, um das Bodenbett zu bilden und mit entionisiertem Wasser zu sättigen. Streuen Sie etwa 200 Samen in dieses Bodenbett.
- Geben Sie es in ein größeres Tablett mit 1 cm stehendem Wasser. Decken Sie dieses Tablett mit Plastikfolie ab, um eine Feuchtigkeitskammer zu schaffen.
- Übertragen Sie diesen Aufbau auf eine Wachstumskammer, die auf 23 °C mit 16 h Licht und 8h Dunkelzyklus mit 150-170 μmol/m2 s Lichtintensität eingestellt ist.
- Nach zwei Wochen junge Sämlinge in kleine, 3-4-Zoll-Töpfe mit wassergesättigter Bodenmischung übertragen.
- Legen Sie diese Töpfe in Plastikschalen und geben Sie sie für weitere vier Wochen in die Wachstumskammer.
3. Bakterienstämme für die Agro-Infiltration vorbereiten
HINWEIS: Für die Agro-Infiltration müssen Bakterienstämme frisch subkulturiert und zusammen mit dem p19-Stamm von Agrobacterium in geeigneten Verhältnissen gemischt werden.
- Bereiten Sie 2xYT-Agarplatten mit Rifampicin (100 μg/ml), Gentamicin (25 μg/ml) und Kanamycin (50 μg/ml) für Agrobacterium mit pSPYNE-35S- und pSPYCE-35S-Vektor vor. Verwenden Sie für pSITE-Vektor-haltige Stämme Rifampicin (100 μg/ml), Gentamicin (25 μg/ml) und Spectinomycin (50 μg/ml).
- Streifen Sie die Agrobacterium-Stämme, die die Plasmide enthalten, auf diesen Platten mit sterilen Impfschleifen in einer Laminar-Flow-Haube ab.
- Inkubieren Sie diese bei 28 °C für 48 h im Dunkeln.
- Beginnen Sie dieses Verfahren, indem Sie den Agrobacterium GV3101-Stamm mit BiFC- und FRET-Konstrukten (hergestellt in pSPYNE-35S-, pSPYCE-35S- und pSITE-Vektoren) aus gestreiften Platten in 10 ml 2xYT-Brühe mit geeigneten Antibiotika (Rifampicin (100 μg / ml), Gentamicin (25 μg / ml), Kanamycin (50 μg / ml) oder Spectinomycin (50 μg / ml)) impfen.
- Initiieren Sie zusätzlich eine Kultur des p19-Stammes von Agrobakterien, indem Sie 10 ml 2xYT-Brühe mit Rifampicin (100 μg / ml) und Kanamycin (50 μg / ml) impfen.
HINWEIS: p19-Stamm wird hinzugefügt, um transgene Schalldämpfung zu verhindern. - Decken Sie den Kolben mit einer Aluminiumfolie ab und bewahren Sie ihn im Inkubator-Shaker bei 28 °C und 170 U / min für 16 h im Dunkeln auf.
- Nach dem Wachstum über Nacht 1 ml dieser Kultur auf eine Einwegküvette übertragen, um die optische Dichte (O.D.) der Kulturen bei 600 nm mit einem Spektralphotometer zu messen.
- Mischen Sie die Kulturen geeigneter BiFC- und FRET-Partner-haltiger Stämme, so dass der endgültige O.D. jeder Kultur 0,5 und der von p19 0,3 in einem Gesamtvolumen von 2 ml beträgt.
- Um diese Verhältnisse zu erreichen, verwenden Sie die unten genannte Formel:
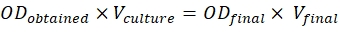
ERHALTENES OD = O.D. der bei 600 nm gemessenen Kultur
V-Kultur = Volumen der gewünschten Kultur
ODfinal = 0,5 für Konstrukte und 0,3 für p19
Vfinal = Endvolumen für die Infiltration, das 2 ml beträgt
ANMERKUNG: Die in dieser Studie verwendeten Konstruktkombinationen sind in Tabelle 2aufgeführt. - Die gemischten Agrobacterium-Kulturen bei 3.000 x g für 5 min bei Raumtemperatur zentrifugieren und den Überstand vorsichtig entsorgen. Resuspendieren Sie das Pellet in 2 ml frisch zubereitetem Infiltrationspuffer (10 mM MES, 100 μM Acetosyringon und 10 mMMgCl2). Verwenden Sie einen Wirbelmischer, um eine homogene Zellsuspension herzustellen.
- Inkubieren Sie die Röhrchen, die resuspendierte Zellen enthalten, im Dunkeln bei Raumtemperatur für 3 h.
- Beschriften Sie in der Zwischenzeit jeden Pflanzentopf mit der Konstruktmischung, mit der er infiltriert wird. Verwenden Sie zwei Pflanzen für jede Infiltrationsmischung.
- Füllen Sie eine 1 ml nadellose Spritze mit der agrobakteriellen Mischung. Drücken Sie die Spritze vorsichtig, aber fest auf die Abaxialseite des vollständig expandierten Blattes, während Sie das Blatt von der anderen Seite abstützen. Drücken Sie den Kolben vorsichtig, bis sich die Lösungen im Blattbereich füllen, was dem 2-3-fachen der Spritzenspitze entspricht.
- Infiltrieren Sie bis zu vier Flecken auf einem Blatt und 3-4 Blätter pro Pflanze, wie in Abbildung 4 gezeigt.
HINWEIS: Wechseln Sie die Handschuhe oder wischen Sie die Handschuhe mit 70% Alkohol zwischen den Proben ab, um eine Kreuzkontamination zu vermeiden. - Alle Töpfe in ein Tablett geben und in einer Wachstumskammer unter den gleichen Bedingungen wie in Schritt 2 inkubieren.
- Überprüfen Sie einen kleinen Teil des agroinfiltrierten Blattes zu verschiedenen Zeitpunkten mit einem Fluoreszenzmikroskop. Wenn die Fluoreszenz von YFP und CFP in Zellen nachweisbar ist, fahren Sie mit dem konfokalen Mikroskop für den BiFC-FRET FLIM-Assay fort. In diesem Experiment wurde die Analyse 3 Tage nach der Agroinfiltration durchgeführt.
HINWEIS: Legen Sie die Inkubationszeit nach der Agroinfiltration für jede Promotor- und Genkombination individuell fest, um eine Überexpression der im BiFC-FRET FLIM-Assay verwendeten chimären Proteine zu vermeiden. Die Überexpression der Partnerproteine kann zu falsch-positiven Wechselwirkungen führen.
4. Objektträger für die Fluoreszenzvisualisierung vorbereiten
- Wenn die Pflanzen für die Visualisierung bereit sind, schneiden Sie quadratische Blattproben, 5-8 mm von der Infiltrationswunde entfernt, und montieren Sie sie in destilliertem Wasser auf sauberen Objektträgern.
HINWEIS: Um die Hintergrundfluoreszenz zu minimieren, reinigen Sie die Objektträger mit 80% Ethanol, gefolgt von destilliertem Wasser 3-4 Mal, trocknen Sie sie an der Luft und bewahren Sie sie auf einer saugfähigen Folie auf. - Decken Sie die Blattprobe mit einem sauberen Deckglas ab und verschließen Sie sie mit einem Nagellack.
- Visualisieren Sie diese Proben unter einem konfokalen Laser-Scanning-Mikroskop.
5. FRET-FLIM-Analyse mit einem konfokalen Laser-Scanning-Mikroskop
ANMERKUNG: In diesem Verfahren ist die Grundlage für die Bestimmung und Quantifizierung der Wechselwirkung zwischen zwei Proteinen die Verringerung der Fluoreszenzlebensdauer des FRET-Donor-Partners bei seiner Wechselwirkung mit dem Akzeptor, die zur Berechnung der Effizienz von FRET verwendet wird. Die Komplexität im Falle der dreigliedrigen Wechselwirkung nimmt weiter zu, da der FRET-Akzeptor in diesem Fall kein einzelnes Molekül ist, sondern ein gespaltenes YFP-BiFC-Paar, das zunächst in vivo rekonstituiert werden sollte, um ein funktioneller FRET-Akzeptor-Fluorophor zu werden. Um FRET-FLIM durchzuführen, muss man die Fluoreszenz des Spendermoleküls lebenslang bestimmen - zuerst allein und dann in Anwesenheit eines FRET-Partners.
- Öffnen Sie die FLIM-Anwendung im konfokalen Laser-Scanning-Mikroskop, starten Sie die Konsole und verwenden Sie die Photonenzählung zur Mustererkennung, um die Fluoreszenzlebensdauer zu messen. Wählen Sie den Standard-Messmodus "Alle Photonenzählung".
- Analysieren Sie Proben von zwei Arten von agroinfiltrierten Pflanzen: eine nur mit dem Spender (C-CFP) und die andere mit dem Spender und dem Akzeptor (C-CFP, zusammen mit M-YFP).
HINWEIS: Da die Wechselwirkung des M-Proteins mit dem C-Protein bereits unter Verwendung von BiFC und Y2H validiert wurde, wird bei diesem interagierenden Paar eine gute FRET-Effizienz erwartet. - Scannen Sie als Nächstes das C-CFP-Agro-infiltrierte Blatt und konzentrieren Sie sich auf eine Zelle, die eine gute CFP-Fluoreszenz aufweist. Starten Sie den Laserscanning-Modus und stellen Sie das System für CFP-Visualisierung und FLIM-Messungen ein (λex 440 nm Pulslaser,λ em 480-520 nm durch Hybriddetektoren, Scangeschwindigkeit 512 x 512 Pixel bei 400 Hz).
- Passen Sie den Fokus, den Zoom und die intelligente Verstärkung an, um sich auf den Bereich zu konzentrieren, der erfasst werden muss.
- Beleuchten Sie die Probe mit ausreichender Laserleistung, um die Erfassung von etwa 0,1 Photonen pro Puls zu erreichen. Erfassen Sie bei Proben mit variabler Fluoreszenzintensität 50 Frames, um die für die Lebensdauermessung erforderlichen Photonen zu sammeln. CFP weist aufgrund seiner Konformationsanpassung zwei Fluoreszenzlebensdauern auf; Passen Sie daher die Daten mit dem n-exponentiellen Rekonvolutionsmodell an, während der Wert von n gleich 2 bleibt.
- Bei diesen Einstellungen zeigt die CFP zwei Lebensdauern von 1,0 und 3,2 ns. Hier wird die höhere, 3,2 ns, Lebensdauer für alle nachfolgenden Berechnungenverwendet 25,26.
- Um die FRET-Effizienz mit FLIM zu berechnen, das das Maß für den Grad der Interaktion zwischen zwei Proteinen ist, nehmen Sie eine Blattprobe, die mit C-CFP und M-YFP co-infiltriert wurde. Suchen Sie nach einer Zelle, die sowohl C-CFP als auch M-YFP exprimiert, und bestätigen Sie ihre jeweiligen Emissionsmuster, indem Sie sie mit λex 440 nm Pulslaser, λem 480-520 nm und λex 514 nm Weißlichtlaser mit λem 526-550 nm anregen. Scannen und identifizieren Sie nacheinander eine Zelle, die sowohl CFP- als auch YFP-Fluoreszenz zeigt.
- Nachdem Sie die Fluoreszenz beider Proteine bestätigt haben, wechseln Sie zur FLIM-Konsole, um die Lebensdauer von CFP mit den gleichen Einstellungen zu messen, die wir zuvor für die Messung der Lebensdauer von C-CFP verwendet haben (Schritt 5.5).
HINWEIS: Diese Zelle exprimiert auch M-YFP, das möglicherweise mit C-CFP interagieren und eine Verringerung der Lebensdauer von C-CFP verursachen kann. - Passen Sie den Graphen an, der mit dem n-exponentiellen Rekonvolutionsmodell erhalten wurde, wobei n = 2 ist. Es wurde eine Abnahme der CFP-Lebensdauer von 3,2 auf 2,6 ns beobachtet, was auf einen Förster-Resonanzenergietransfer zwischen CFP und YFP hinweist (Abbildung 5A).
- Starten Sie nun die FRET-Konsole in der Software und berechnen Sie die FRET-Effizienz, indem Sie die ungestillte Spenderlebensdauer manuell in die in der Software bereitgestellte Gleichung eingeben. Und der beobachtete FRET-Wirkungsgrad ist: 56%.
- Dreigliedrige Interaktion
- Um die Wechselwirkungen zwischen drei Partnern zu visualisieren, nehmen Sie schließlich die Blattprobe von einer Pflanze, die mit C-CFP, M-YFPn und M-YFPc co-infiltriert wurde.
- Scannen Sie das Blattexplantat nach einer Zelle, die sowohl CFP als auch rekonstituierte YFP-Fluoreszenz zeigt, die von der BiFC-Interaktion zwischen zwei M-Proteinen ausgeht. Verwenden Sie die gleichen Laser- und Emissionswellenlängen wie zuvor.
- Schalten Sie anschließend den 514-nm-Laser aus und wechseln Sie zur FLIM-Konsole.
HINWEIS: Wenn das M-YFP-Dimer mit C-CFP interagiert, sollte man eine Verringerung der Lebensdauer von C-CFP sehen, wie sie während seiner Wechselwirkung mit M-YFP beobachtet wurde. Wenn das C-CFP jedoch nicht mit dem M-YFP-Dimer interagiert, sollte seine Fluoreszenzlebensdauer bei 3,2 ns bleiben. - Messen Sie mit ähnlichen Einstellungen wie oben erwähnt die CFP-Lebensdauer in Gegenwart von rekonstituiertem YFP. Passen Sie den mit dem n-exponentiellen Rekonvolutionsmodell erhaltenen Graphen mit n = 2 an, und wechseln Sie zur FRET-Konsole.
ANMERKUNG: Die Lebensdauer der GFP ist von 3,2 auf 2,3 ns zurückgegangen. Berechnen Sie den FRET-Wirkungsgrad wie oben beschrieben. Der berechnete FRET-Wirkungsgrad beträgt 55%. Die Verkürzung der Spenderlebensdauer und die gute FRET-Effizienz von 55% bestätigen die dreigliedrige Interaktion zwischen zwei M-Proteinen und dem C-Protein in vivo (siehe Abbildung 5B).
Ergebnisse
Dieses Protokoll stellt eine optimierte Methode zur Untersuchung von in vivo dreigliedrigen Protein-Protein-Interaktionen in Pflanzen dar. Das Grundprinzip des Protokolls besteht darin, zwei fluoreszenzmarkierte Protein-Interaktionstechniken, d.h. BiFC und FRET, zu kombinieren, um einen Assay zur Messung der ternären Komplexbildung zwischen drei Proteinpartnern zu erstellen. Hier haben wir FLIM verwendet, um die Fluoreszenzlebensdauer des FRET-Donorpartners in Gegenwart und Abwesenheit des FRET-Akzeptors zu mes...
Diskussion
Das vorliegende Protokoll demonstriert die Verwendung eines BiFC-basierten FRET-FLIM-Assays zur Bestimmung der Bildung eines ternären Komplexes zwischen zwei Monomeren eines MADS-Box-Proteins und eines Calciumsensorproteins. Das Protokoll basiert auf einem Bericht von Y. John Shyu et al., wo sie eine BiFC-basierte FRET-Methode entwickelt haben, um ternäre Komplexe zu visualisieren, die zwischen Fos-Jun-Heterodimeren und NFAT oder p65 unter Verwendung der sensibilisierten Emissionsmethode gebildet werden
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
NB, GG, SB, KC danken der University Grants Commission (UGC), UGC-BSR, DBT-INSPIRE und dem Council for Scientific and Industrial Research (CSIR) herzlich für ihre Forschungsstipendien. Wir danken dem Department of Biotechnology (DBT), der indischen Regierung, dem Department of Science and Technology (DST-FIST) und der indischen Regierung für die finanzielle Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
Referenzen
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1 (2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11 (2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747 (1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten