JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
BIFC-FRET-FLIM 분석체에 의한 MADS 박스 전사 계수의 두 단백제와 칼슘 센서 단백질 간의 삼자 상호 작용 결정
* 이 저자들은 동등하게 기여했습니다
요약
여기서 우리는 BiFC 기반 FRET-FLIM 분석법에 의한 형광 태그 단백질을 사용하여 3개의 단백질 파트너 간의 삼차 복합 형성을 시각화하는 방법을 제시합니다. 이 방법은 생체 내에서단백질 단백질 상호 작용 복합체를 연구하는 데 유용합니다.
초록
단백질 단백질 상호 작용은 세포 기능을 조절, 유지 및 수정하는 데 중요한 역할을 할 때 세포의 모든 생물학적 과정의 필수적인 부분입니다. 이러한 상호 작용은 신호 트랜스듀션, 병원체 반응, 세포 세포 상호 작용, 대사 및 발달 과정과 같은 광범위한 현상에 관여합니다. 전사 요인의 경우, 이러한 상호 작용은 핵, 세포질 등과 같은 특정 세포 외 맥락에서 분리된 하위 단위의 올리고머화로 이어질 수 있으며, 이는 차례로 다운스트림 유전자의 발현에 더 심오한 영향을 미칠 수 있다. 여기서는 형광 수명 이미징(FLIM)을 포함하는 이중 분자 형광 보완(BiFC) 기반 Förster 공명 에너지 전송(FRET)을 사용하여 생체 삼자 상호 작용을 시각화하는 방법론을 시연합니다. 이 데모를 위해 선택된 단백질의 2개는 BiFC 파트너로 상호 작용하고, 그들의 재구성한 형광 활동은 제 3 파트너와 FRET-FLIM를 분석하기 위하여 이용됩니다. 4~5주 된 성장 챔버 에서 자란 니코티아나 벤타미안나 식물은 이 데모의 모델 플랜트 시스템으로 사용되었습니다.
서문
단백질 단백질 상호 작용(PPI)은 다양한 대사 및 발달 과정을 조절하여 진핵 세포의 적절한 기능의 기초를 형성한다. 일부 PPI는 안정적이며 다른 PPI는 자연에서 일시적입니다. 상호 작용은 조광, 트리메릭, 테트라메릭 균동체 및 이종성1과같은 상호 작용에서 구성원의 수와 유형에 따라 분류될 수 있다. 단백질 상호 작용의 식별 및 특성화는 단백질 기능 및 규제 네트워크의 더 나은 이해로 이어질 수 있습니다.
전사 요인은 규제 기능에 관여하는 단백질입니다. 그(것)들은 DNA에 결합하여 그들의 다운스트림 유전자의 전사의 비율을 통제합니다. 때로는 단백질에 의한 고수 복합체의 올리고머화 또는 형성은 그들의 기능을 수행하기 위한 전제조건2. 식물 MADS 박스 전사 요인은 꽃 전환, 꽃 장기 발달, 수정, 종자 발달, 노화 및 식물 발달과 같은 다양한 과정을 조절하는 동종 유전자입니다. 그들은 DNA3에결합 하는 높은 순서 복합체를 형성 하는 것으로 알려져 있다3,4. 전사 요인과 그 상호 작용자 중 PPI 네트워크를 연구하면 기본 전사 규정의 복잡성에 대한 통찰력을 얻을 수 있습니다.
니코티아나 벤타미안나에서의 과도 단백질 발현은 생체 내에서단백질 국소화 또는 단백질-단백질 상호 작용을 연구하기 위한 인기 있는 접근법이다5. BiFC 및 FRET는 형광 리포터 시스템6을사용하여 생체 내에서 단백질-단백질 상호 작용을 연구하는 방법이다. 이들 두 기술의 조합은 3개의 단백질7사이의 상호작용을 드러내는 것으로 나타났다. FRET는 수용조 광표백, 감감 방출 및 형광 평생 이미징(FLIM) 기술을 사용하여 측정됩니다. FLIM 기반 FRET 분석법은 형광 수명8을기준으로 두 분자 간의 에너지 전달 측정에 대한 정확한 정량화 및 현면 특이성을 제공하는 도구로 부상했다. FLIM은 광자 방출 전에 플루오로포어가 흥분된 상태로 머무르는 시간을 측정하며 강도 측정을 단독으로 사용하는 기술보다9,10이더 낫다. 니코티아나 벤타미안나 및 양파 표피 껍질과 같은 이종 시스템 외에도, 최근 보고서는 네이티브 조건 하에서 단백질-단백질 상호 작용의 생체 분석을 위해 아라비도시스 뿌리와 젊은 쌀 모종 등의 사용을입증했다11,12.
적절한 식 시스템 이외에 BiFC 및 FRET 에세이에 대한 상호 작용 파트너의 선택은 이 실험의 성공에 매우 중요합니다. BiFC 구성에 사용되는 파트너 간의 PPI는 FRET 실험13에서컨쥬게이션 파트너로 사용하기 전에 적절한 컨트롤을 사용하여 유효성을 검사해야 한다. BiFC는 형광 단백질의 N 및 C 단말 부분의 구조적 보완을 활용합니다. BiFC 분석서에 사용되는 모든 형광 단백질이 아닌 대부분의 일반적인 제한은 두 유도체 비형광 단편 간의 자체 조립으로, 거짓 양성 형광에 기여하고 신호-잡음(S/N)비율(14)을감소시한다. 최근 개발, 점 돌연변이 또는 형광 단백질분할의 위치를 포함하는, 증가 강도와 BiFC 쌍상승을 주었다, 높은 특이성, 높은 S/N 비율15,16. 이러한 형광성 단백질은 또한 실험의 적합성에 따라 BiFC를 수행하기 위해 사용될 수 있다.
전통적으로 CFP와 YFP는 FRET 실험17에서기증자 및 수용자 쌍으로 사용되어 왔다. 그러나, YFP 또는 m-Citrine은 애비독증 뿌리 시스템에서 표적 단백질의 원발현 동안 높은 양자 수율(QY)으로 인해 더 나은 FRET 기증자(RFP를 수용자로 사용할 경우)인 것으로 나타났다. 발기인(구성대 네이티브/내생)과 형광호는 성공적인 BiFC-FRET-FLIM 실험을 설계하는 데 중요한 역할을 합니다. FRET 기증자의 효율성과 FRET 쌍의 적합성은 발기인의 변화와 발현에 사용되는 생물학적 시스템의 변화와 함께 변화하는 경향이 있음을 주목하는 것이 필수적입니다. 그것의 밝기에 관련되는 불소호의 QY는, 사용 중인 pH, 온도 및 생물학 시스템에 달려 있습니다. FRET 실험을 위한 플루오로포레 쌍을 선택하기 전에 이러한 기준을 철저히 고려하는 것이 좋습니다. 이 프로토콜에 사용되는 생물학적 시스템, 프로모터 및 단백질은 BiFC FRET-FLIM 실험을 위한 CFP-YFP 형광과 잘 작용했습니다.
본 연구에서는, 우리는 BiFC 기지를 둔 FRET를 사용하여 3개의 단백질 분자 사이 상호 작용을 구상하기 위하여 FLIM의 특징을 통합합니다. 이 기술에서, 2개의 단백질은 분할 YFP 단백질 및 CFP를 가진 세 번째 단백질로 태그됩니다. 우리는 칼슘 센서 단백질 (C)와 MADS 박스 단백질 (M) 호모이머의 상호 작용을 연구하는 데 관심이 있었기 때문에, 이러한 단백질은 pSITE-1CA 및 pSITE-3CA 벡터18에서형광 단백질로 태그되었다. 상호 작용 파트너 중 2개는 pSPYNE-35S 및 pSPYCE-35S 벡터19에서YFP의 N-및 C 단말 부분과 태그되었으며, 이들의 상호 작용은 CFP(FRET 기증자로 작용)와 태그되는 제3 상호작용 파트너에게 FRET 수락자 역할을 하는 기능성 YFP의 재구성(FRET 기증자의 경우)1(FRET 기증자의 역할)로 태그됩니다( FRET 기증자로 작용) (1) ). 이 경우, 두 M 단량체와 M과 C 사이의 PPI는 효모-2 하이브리드 시스템과 함께 세 가지 시스템에서 BiFC를 수행하여 검증되었습니다. 이러한 벡터는 전기화에 의해 Agrobacterium tumifaciens GV3101 균주로 동원되었다. GV3101 균주는 젠타미신저항(20)을가진 무장 해제 된 티 플라스미드 pMP90 (pTiC58DT-DNA)을 갖는다. p19 아그로박테리움 균주는 모든 침투와 함께 첨가되어 유전자침묵(21)을방지하였다. 3개의 단백질은 삼자 상호 작용을 확인하기 위하여 반대 순응에 이용되어야 한다는 것을 추천합니다.
이 기술에서, 우리는 FLIM를 채택했습니다, 여기서 첫째로, 기증자의 형광 수명 (불순종한 기증자 수명)는 수용자의 부재에서 측정됩니다. 그 후, 그 수명은 수용자의 존재에서 측정됩니다 (담금질 기증자 수명). 기증자 형광 수명에 있는 이 다름은 형광 수명에 있는 감소를 나타내는 광자의 수에 의존하는 FRET 효율성을 계산하기 위하여 이용됩니다. 아래에 언급된 상세한 프로토콜은 니코티아나 벤타미안에서 형광 태그 단백질을 일시적으로 표현하고 BiFC-FRET-FLIM에 의한 상호 작용을 말함으로써 어떤 3개의 단백질 사이 대동맥 복합체의 형성을 결정하기 위한 상세한 프로토콜입니다.
프로토콜
1. 항목 및 대상 벡터에서 유전자의 복제 (그림2)
- 관심 있는 유전자의 코딩 서열(CDS)을 PCR에 의해 증폭시키고 적절한 진입 벡터(예를 들어, pENTR/D-TOPO 벡터)에서 복제하는; 이 실험에 사용된 벡터의 표 1을 참조하십시오.).
- 항생제를 함유 한 접시에 클론을 성장. 제한 소화 및 DNA 염기서열22,23에의해 항생제에 선택되는 클론을 검증한다.
- 재구성된 CDS를 엔트리 클론에서 대상 벡터(pSPYNE-35S, pSPYCE-35S, pSITE-1CA 및 pSITE-3CA)로 동원하고 효소 소화제한에 의해 대상 벡터로의 항목으로부터 서열의 전달을 확인한다.
참고: 이 실험에 사용된 모든 벡터는 표 1에나열됩니다. - 마지막으로, 멸균 GV3101(pMP90(GentR))세포를 전기포레이션에 의한 대상 벡터로 변환(도3)24.
2. 니코티아나 벤타미안 식물의 성장 조건
참고: 니코티아나 식물을 제어 조건에서 4-6 잎 스테이지까지 재배합니다.
- 니코티아나 식물을 재배하려면 2:1:1의 비율로 코코파트와 퇴비와 시판되는 토양 믹스를 혼합하여 토양 믹스를 준비한다.
- 이 토양 혼합물의 1 인치 두께층을 플라스틱 트레이에 펴서 토양 침대를 만들고 탈온화 된 물로 포화시하십시오. 이 토양 침대에 약 200 씨앗을 뿌립니다.
- 1cm의 서있는 물이 들어 있는 더 큰 트레이로 옮기습니다. 이 트레이를 플라스틱 랩으로 덮어 수분 챔버를 만듭니다.
- 16h 광및 150-170 μmol/m2의광강도로 8h 다크 사이클로 23°C로 설정된 성장 챔버로 설정된 이 설정을 전송합니다.
- 2 주 후, 물 포화 토양 믹스를 포함하는 작은, 3-4 인치 냄비에 젊은 묘목을 전송합니다.
- 이 냄비를 플라스틱 트레이에 놓고 4 주 동안 성장 챔버로 옮기십시오.
3. 농약 침투를 위한 세균성 균주 준비
참고: 농약 침투의 경우, 세균균은 적절한 비율로 Agrobacterium의 p19 균주와 함께 신선하게 하위 배양되고 혼합되어야 합니다.
- 리팜피신(100 μg/mL), 젠타미신(25 μg/mL), 카나마이신(50 μg/mL)을 함유한 2xYT 아가르 플레이트를 아그로바테리움 항구 pSPYNE-35S 및 pSPYCE-35S 벡터용으로 준비한다. 균주를 포함하는 pSITE 벡터의 경우 리팜피신(100 μg/mL), 젠타미신(25 μg/mL) 및 스펙티노마이신(50 μg/mL)을 사용하십시오.
- 라미나르 플로우 후드에 멸균 접종 루프를 사용하여 이 플레이트의 플라스미드를 함유한 아그로박테리움 균주를 줄입니다.
- 어둠 속에서 48 h에 대한 28 °C에서 이들을 배양.
- BiFC 및 FRET 구조물을 수용하는 Agrobacterium GV3101 균주를 접종하여이 절차를 시작합니다 (pSPYNE-35S에서 제조, pSPYCE-35S 및 pSITE 벡터)는 적절한 항생제(리팜피신(100 μg/mL), 젠타미신(25 μg/mL), 카나마이신(50 μg/mL) 또는 스펙티노미신(50 μg/mL)을 함유한 2xYT 국물의 10mL의 줄무늬 플레이트에서 나온 것입니다.
- 또한, 리팜피신(100 μg/mL) 및 카나마이신(50 μg/mL)을 함유한 2xYT 국물의 10mL를 접종하여 농균의 p19 균주의 배양을 개시한다.
참고: p19 균주가 첨가되어 유전자 침묵을 방지합니다. - 플라스크를 알루미늄 호일로 덮고 28°C로 설정된 인큐베이터 셰이커와 어둠 속에서 16시간 동안 170rpm으로 보관하십시오.
- 하룻밤 동안 성장한 후, 이 배양물의 1mL를 일회용 큐벳으로 이송하여 분광계를 이용하여 600nm에서 배양물의 광학 밀도(O.D.)를 측정한다.
- 각 배양물의 최종 O.D.가 0.5이고 p19의 최종 O.D.가 총 2mL의 0.3이 되도록 균주를 포함하는 적절한 BiFC 및 FRET 파트너의 배양을 혼합합니다.
- 이러한 비율을 얻으려면 아래에 언급된 공식을 사용하십시오.
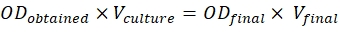
OD 획득 = 600 nm에서 측정된 배양물의 O.D.
V배양 = 필요한 배양량
OD최종 = 구문에 대한 0.5, p19의 경우 0.3
V최종 = 침투에 대한 최종 볼륨, 이는 2 mL입니다
참고: 이 스터디에 사용된 구성 조합은 표 2에지정됩니다. - 상온에서 5 분 동안 3,000 x g의 혼합 Agrobacterium 배양원 심분리기및 조심스럽게 상류체를 폐기하십시오. 새로 준비된 침투 버퍼(10m M MES, 아세토시링본 100μM, MgCl210m)의 2mL에서 펠릿을 재연한다. 소용돌이 믹서를 사용하여 균질성 세포 현탁액을 만듭니다.
- 3 시간 동안 실온에서 어둠 속에서 다시 중단 된 세포를 포함하는 튜브를 배양합니다.
- 한편, 각 식물 냄비에 물입이 침투될 구조 혼합물로 라벨을 부착합니다. 각 침투 혼합물에 두 개의 식물을 사용하십시오.
- 농균 혼합물로 1mL 바늘없는 주사기를 채웁니다. 주사기를 완전히 확장된 잎의 축축 측에 부드럽게 누르고 다른 쪽에서 잎을 지지합니다. 주사기 팁의 2-3배에 해당하는 리프 영역에 용액이 채워날 때까지 플런저를 부드럽게 밀어 넣습니다.
- 그림 4에도시된 바와 같이 잎에 최대 4개의 반점과 식물당 3-4잎에 침투한다.
참고: 장갑을 교체하거나 샘플 사이에 70%의 알코올로 장갑을 닦아 교차 오염을 방지합니다. - 모든 냄비를 트레이로 옮기고 2단계에서 언급한 것과 동일한 조건하에서 성장 챔버에서 배양한다.
- 형광 현미경을 사용하여 다른 시간 지점에서 농약침 잎의 작은 부분을 확인하십시오. YFP와 CFP 모두에서 형광이 세포에서 검출될 때, BiFC-FRET FLIM 분석기에 대한 공초점 현미경으로 진행한다. 이 실험에서, 분석은 농침 후 3일 후에 수행되었다.
참고: BiFC-FRET FLIM 분석에서 사용되는 키메라 단백질의 과발현을 피하기 위해 모든 프로모터 및 유전자 조합에 대해 개별적으로 인큐베이션후 의 침투 기간을 설정합니다. 파트너 단백질의 과발현은 거짓 양성 상호 작용으로 이어질 수 있습니다.
4. 형광 시각화슬라이드 준비
- 식물이 시각화할 준비가 되면, 각당 잎 샘플을 5-8mm 떨어진 침수상처에서 자르고 깨끗한 슬라이드에 증류수에 장착하십시오.
참고: 배경 형광을 최소화하기 위해 80%의 에탄올로 슬라이드를 청소하고 증류수 3-4회, 공기 건조, 흡수시트에 보관하십시오. - 잎 샘플을 깨끗한 커버슬립으로 덮고 손톱 에나멜을 사용하여 밀봉하십시오.
- 공초점 레이저 스캐닝 현미경으로 이러한 샘플을 시각화합니다.
5. 공초점 레이저 스캐닝 현미경을 이용한 FRET-FLIM 분석
참고: 이 절차에서, 두 단백질 간의 상호 작용을 결정하고 정량화하는 기초는 FRET의 효율성을 계산하는 데 사용되는 수용자와의 상호 작용에 따라 FRET-기증자 파트너의 형광 수명 감소입니다. 삼자상호작용의 복잡성은 FRET-acceptor가 단일 분자가 아니라 분할 YFP-BiFC 쌍이기 때문에 더욱 증가하며, 이는 먼저 기능적인 FRET-acceptor fluorophore가 되기 위해 생체 내에서 재구성되어야 한다. FRET-FLIM을 수행하기 위해, 하나는 기증자 분자의 형광 일생일대의 일생 을 단독으로 결정하고 FRET 파트너의 존재에서 결정해야합니다.
- 공초점 레이저 스캐닝 현미경에서 FLIM 응용 프로그램을 열고 콘솔을 시작하고 패턴 인식 광자 계수를 사용하여 형광 수명을 측정합니다. 표준 '모든 광자 계수' 측정 모드를 선택합니다.
- 두 가지 유형의 농약 침투 식물의 샘플을 분석합니다: 기증자전용(C-CFP)을 가진 식물과 기증자 및 수용자(C-CFP, M-YFP)와 함께 다른 식물을 분석합니다.
참고: M 단백질의 상호 작용은 이미 BiFC 및 Y2H를 사용하여 C 단백질로 검증되었기 때문에, 이 상호 작용 쌍으로 좋은 FRET 효율이 예상됩니다. - 다음으로, C-CFP 농경에 침투한 잎을 스캔하고 좋은 CFP 형광을 나타내는 세포에 초점을 맞춥니다. 레이저 스캐닝 모드를 시작하고 CFP 시각화 및 FLIM 측정 시스템 설정(λex 440 nm 펄스 레이저, 하이브리드 검출기로 λem 480-520 nm, 400Hz에서 스캔 속도 512 x 512 픽셀).
- 초점, 줌 및 스마트 게인을 조정하여 캡처해야 하는 영역에 초점을 맞춥니다.
- 펄스당 약 0.1 광자의 포획을 달성하기에 충분한 레이저 전력으로 샘플을 조명합니다. 가변 형광 강도를 가진 샘플의 경우 50 프레임을 캡처하여 수명 측정에 필요한 적절한 광자를 수집합니다. CFP는 그것의 형성 적응 때문에 2개의 형광 수명을 전시합니다; 따라서 n-지수 재수렴 모델을 사용하여 데이터를 적어 n값을 2와 동일하게 유지합니다.
- 이러한 설정에서 CFP는 1.0및 3.2 ns의 두 수명을 보여줍니다. 여기서 더 높은, 3.2 ns, 수명은 모든 후속 계산에 사용된다25,26.
- 두 단백질 간의 상호 작용 정도의 척도인 FLIM을 사용하여 FRET 효율을 계산하려면 C-CFP 및 M-YFP와 공동 침투한 잎 샘플을 채취하십시오. C-CFP와 M-YFP를 모두 표현하고 λex 440 nm 펄스 레이저, λem 480-520 nm 및 λem 526-550 nm와 함께 λex 514 nm 백색 광 레이저를 사용하여 흥미 진진한 그들 각각의 방출 패턴을 확인하는 세포를 찾아보십시오. CFP와 YFP 형광을 모두 보여주는 세포를 순차적으로 스캔하고 식별합니다.
- 두 단백질 모두에서 형광을 확인한 후, C-CFP의 수명을 측정하기 위해 이전에 사용한 것과 동일한 설정을 사용하여 CfP의 수명을 측정하기 위해 FLIM 콘솔로 전환합니다(단계 5.5).
참고: 이 셀은 또한 잠재적으로 C-CFP와 상호 작용하고 C-CFP의 수명 감소를 일으키는 M-YFP를 표현하고 있습니다. - n-지수 재수렴 모델을 사용하여 얻은 그래프에 n=2를 맞춥시게 한다. CFP 수명 수명이 3.2에서 2.6ns로 감소하여 CFP와 YFP(그림5A)간의 Förster 공명 에너지 전달을 나타낸다.
- 이제 소프트웨어에서 FRET 콘솔을 시작하고 소프트웨어에 제공된 방정식에서 수동으로 언크되지 않은 기증자 수명을 입력하여 FRET 효율성을 계산합니다. 그리고 관찰 된 FRET 효율은 : 56 %입니다.
- 삼자 상호 작용
- 마지막으로, 세 파트너 간의 상호 작용을 시각화하려면 C-CFP, M-YFPn 및 M-YFPc와 공동 침투한 공장에서 리프 샘플을 채취하십시오.
- 두 M 단백질 간의 BiFC 상호 작용에서 방출되는 CFP와 재구성된 YFP 형광을 모두 보여주는 세포를 위해 잎 을 스캔합니다. 이전에 사용했던 것과 동일한 레이저 및 방출 파장을 사용합니다.
- 그 후, 514 nm 레이저를 끄고 FLIM 콘솔로 이동합니다.
참고: M-YFP 이머가 C-CFP와 상호 작용하는 경우 M-YFP와의 상호 작용 중에 관찰된 C-CFP 수명 감소가 표시됩니다. 그러나 C-CFP가 M-YFP 이머와 상호 작용하지 못하면 형광 수명은 3.2ns에 머물러야 합니다. - 위에서 언급한 것과 유사한 설정을 사용하여 재구성된 YFP가 있는 경우 CFP 수명을 측정합니다. n=2와 함께 n-지수 재수렴 모델을 사용하여 얻은 그래프에 맞추고 FRET 콘솔로 이동합니다.
참고: CFP 수명 수는 3.2에서 2.3ns로 감소합니다. 위에서 설명한 대로 FRET 효율을 계산합니다. 계산된 FRET 효율은 55%입니다. 기증자 수명 감소 및 55%의 양호한 FRET 효율은 두 M 단백질과 생체 내 C 단백질 간의 삼자 상호 작용을 확인합니다(그림 5B참조).
결과
이 프로토콜은 식물에서 생체 삼자 단백질 상호 작용을 연구하는 최적화된 방법을 나타냅니다. 프로토콜의 기본 원리는 두 개의 형광 태그 단백질 상호 작용 기술, 즉 BiFC 및 FRET를 결합하여 세 가지 단백질 파트너 간의 삼차 복합 형성을 측정하는 분석법을 만드는 것입니다. 여기에서, 우리는 FRET 수락자의 존재와 부재에서 FRET 기증자 파트너의 형광 수명을 측정하기 위하여 FLIM를 이용했습?...
토론
본 프로토콜은 MADS 박스 단백질의 두 단량체와 칼슘 센서 단백질 사이의 대동맥 복합체의 형성을 확인하기 위해 BiFC 기반 FRET-FLIM 분석의 사용을 보여줍니다. 이 프로토콜은 Y. John Shyu 등의 보고서에서 적용되어 Fos-Jun heterodimers와 NFAT 또는 p65 사이에 형성된 삼각 복합체를 민감하게 하여 7을 사용하여 시각화하는 BiFC 기반 FRET방법을개발하였다. 이전에는 2004년29년...
공개
저자는 이해 상충을 선언하지 않습니다.
감사의 말
NB, GG, SB, KC는 대학 보조금 위원회 (UGC), UGC-BSR, DBT-INSPIRE, 과학 및 산업 연구위원회 (CSIR)의 연구 펠로우십에 진심으로 감사드립니다. 우리는 고맙게도 생명공학부 (DBT), 인도 정부, 과학 기술부 (DST-FIST), 재정 지원을 위한 인도 정부를 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
참고문헌
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1 (2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11 (2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747 (1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유