Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung und Quantifizierung des Epstein-Barr-Virus aus der P3HR1-Zelllinie
In diesem Artikel
Zusammenfassung
Dieses Protokoll ermöglicht die Isolierung von Epstein-Barr-Viruspartikeln aus der menschlichen P3HR1-Zelllinie nach Induktion des viralen lytischen Zyklus mit Phorbol-12-Myristat-13-acetat. Anschließend wird DNA aus der viralen Zubereitung extrahiert und einer Real-Time-PCR unterzogen, um die Viruspartikelkonzentration zu quantifizieren.
Zusammenfassung
Das Epstein-Barr-Virus (EBV), offiziell als Humanes Herpesvirus 4 (HHV-4) bezeichnet, ist das erste isolierte humane Tumorvirus. Fast 90-95% der erwachsenen Weltbevölkerung sind mit EBV infiziert. Mit den jüngsten Fortschritten in der Molekularbiologie und Immunologie hat die Anwendung von experimentellen In-vitro - und In-vivo-Modellen tiefe und aussagekräftige Einblicke in die Pathogenese von EBV bei vielen Krankheiten sowie in die EBV-assoziierte Tumorgenese ermöglicht. Ziel dieser visualisierten Experimentierarbeit ist es, einen Überblick über die Isolierung von EBV-Viruspartikeln aus Zellen der P3HR1-Zelllinie zu geben, gefolgt von einer Quantifizierung der viralen Zubereitung. P3HR1-Zellen, die ursprünglich aus einem menschlichen Burkitt-Lymphom isoliert wurden, können ein P3HR1-Virus produzieren, das ein EBV-Stamm vom Typ 2 ist. Der EBV-lytische Zyklus kann in diesen P3HR1-Zellen durch Behandlung mit Phorbol-12-Myristat-13-acetat (PMA) induziert werden, wodurch EBV-Viruspartikel entstehen.
Unter Verwendung dieses Protokolls zur Isolierung von EBV-Partikeln werden P3HR1-Zellen für 5 Tage bei 37 °C und 5%CO2 in vollständigem RPMI-1640-Medium kultiviert, das 35 ng/ml PMA enthält. Anschließend wird das Kulturmedium mit einer Geschwindigkeit von 120 x g für 8 min zentrifugiert, um die Zellen zu pelletieren. Der virushaltige Überstand wird dann gesammelt und mit einer Geschwindigkeit von 16.000 x g für 90 min heruntergeschleudert, um die EBV-Partikel zu pelletieren. Das virale Pellet wird dann in einem vollständigen RPMI-1640-Medium resuspendiert. Es folgen DNA-Extraktion und quantitative real-time PCR, um die Konzentration von EBV-Partikeln in der Zubereitung zu bestimmen.
Einleitung
Das Epstein-Barr-Virus (EBV) ist das erste humane Tumorvirus, das isoliert wurde1. EBV, offiziell als Humanes Herpesvirus 4 (HHV-4)2 bezeichnet, ist Teil der Gamma-Herpesvirus-Unterfamilie der Herpesvirusfamilie und ist der Prototyp der Lymphocryptovirus-Gattung. Fast 90-95% der erwachsenen Weltbevölkerung sind mit dem Virus infiziert3. In den meisten Fällen tritt die Erstinfektion innerhalb der ersten 3 Lebensjahre auf und ist asymptomatisch, wenn die Infektion jedoch später während der Adoleszenz auftritt, kann dies zu einer Krankheit führen, die als infektiöse Mononukleosebezeichnet wird 4. EBV ist in der Lage, ruhende B-Zellen zu infizieren, wodurch sie zu proliferativen B-Lymphoblasten werden, in denen das Virus einen latent infizierten Zustand aufbaut und aufrechterhält5. EBV kann jederzeit reaktiviert werden und so zu wiederkehrenden Infektionen führen6.
In den letzten 50 Jahren ist der Zusammenhang zwischen einigen Viren und der Entwicklung menschlicher Malignome immer deutlicher geworden, und heute wird geschätzt, dass 15% bis 20% aller menschlichen Krebserkrankungen mit Virusinfektionen zusammenhängen7. Die Herpesviren, einschließlich EBV, sind einige der am besten untersuchten Beispiele für diese Art von Tumorviren8. Tatsächlich kann EBV viele Arten von menschlichen Malignomen verursachen, wie Burkitt-Lymphom (BL), Hodgkin-Lymphom (HL), diffuses großzelliges B-Zell-Lymphom und lymphoproliferative Erkrankungen in immungeschwächten Wirten 9,10. Es wurde auch gezeigt, dass EBV mit der Entwicklung systemischer Autoimmunerkrankungen assoziiert ist. Einige Beispiele für diese Autoimmunerkrankungen sind rheumatoide Arthritis (RA), Polymyositis-Dermatomyositis (PM-DM), systemischer Lupus erythematodes (SLE), gemischte Bindegewebserkrankung (MCTD) und Sjögren-Syndrom (SS)11. EBV ist auch mit der Entwicklung von entzündlichen Darmerkrankungen (IBD) verbunden12.
Viele dieser Krankheiten können mit Zellkulturen, Mäusen oder anderen Organismen, die mit EBV infiziert sind, untersucht oder modelliert werden. Aus diesem Grund werden EBV-Partikel benötigt, um Zellen oder Organismen zu infizieren, sei es in vitro oder in vivo Modellen13,14,15,16, daher die Notwendigkeit, eine Technik zu entwickeln, die die Isolierung von Viruspartikeln zu niedrigen Kosten ermöglicht. Das hier beschriebene Protokoll bietet Richtlinien für eine einfache Möglichkeit, EBV-Partikel zuverlässig aus einer relativ zugänglichen Zelllinie zu isolieren und die Partikel mittels real-time PCR zu quantifizieren, die kostengünstig und für die meisten Labore leicht verfügbar ist. Dies steht im Vergleich zu mehreren anderen Methoden, die beschrieben wurden, um EBV aus verschiedenen Zelllinien zu isolieren17,18,19,20.
P3HR-1 ist eine BL-Zelllinie, die in Suspension wächst und latent mit einem EBV-Typ-2-Stamm infiziert ist. Diese Zelllinie ist ein EBV-Produzent und kann dazu gebracht werden, virale Partikel zu produzieren. Das Ziel dieses Manuskripts ist es, eine Methode zu demonstrieren, die die Isolierung von EBV-Partikeln aus der P3HR-1-Zelllinie ermöglicht, gefolgt von einer Quantifizierung des Virusbestands, die später sowohl für in vitro als auch in vivo EBV-Versuchsmodelle verwendet werden könnte.
Protokoll
HINWEIS: EBV sollte als potenziell biogefährliches Material betrachtet werden und daher unter der Eindämmung der Biosicherheitsstufe 2 oder höher behandelt werden. Ein Laborkittel sowie Handschuhe sollten getragen werden. Wenn eine mögliche Exposition gegenüber Spritzern besteht, sollte auch ein Augenschutz in Betracht gezogen werden. Das folgende Verfahren sollte in einer biologischen Sicherheitswerkbank durchgeführt werden.
1. Zählen der P3HR1-Zellen
- Zentrifugieren und Resuspendieren von Zellen
- Die Zellsuspension wird von einer 100-mm-Kulturplatte (oder einem T-25-Kolben) einer laufenden P3HR1-Zellkultur bei 80%iger Konfluenz in ein konisches 15-ml-Röhrchen überführt. Die Seeding-Dichte von P3HR-1-Zellen beträgt 1 x 106 Zellen/ml. In vollständigem RPMI-Kulturmedium (79 % RPMI-Kulturmedium, 20 % Fetales Rinderserum, 1 % Pen-Streptokokken-Antibiotikum) bei 37 °C und 5 %CO2 aufbewahren und alle 3-4 Tage durchgehen.
- Zentrifugieren für 8 min bei 120 x g. Nach der Zentrifugation den Überstand verwerfen, das Zellpellet in 1 ml vollständigem RPMI-Kulturmedium resuspendieren und gut mischen (diese Lösung wird als Suspension A bezeichnet).
- Vorbereitung der Zellsuspension zum Zählen
- Herstellen einer verdünnten Zellsuspension B durch Mischen von 2 μL Zellsuspension A mit 8 μL Kulturmedium. Fügen Sie 10 μL 0,4% Trypanblau zu Suspension B hinzu, um Suspension C zu erhalten.
- Zählen von Zellen mit einem Hämozytometer
- Legen Sie ein Glasdeckglas auf das Hämozytometer und laden Sie 10 μL Suspension C. Stellen Sie das Hämozytometer unter ein Lichtmikroskop und zählen Sie die Anzahl der Zellen, die nicht blau gefärbt sind, in jedem der vier Quadranten der Hämozytometerkammer mit dem 40-fachen Mikroskopobjektiv (Abbildung 1).
- Trypanblau färbt tote Zellen blau; Zählen Sie weder diese Zellen noch Zellen, die einen der oberen, unteren, rechten oder linken Ränder jedes Hämozytometerquadranten berühren.
- Berechnen Sie die Konzentration der Zellen pro ml mit der folgenden Formel:
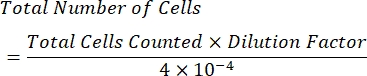
HINWEIS: Die Anzahl der gezählten Quadranten beträgt vier und 10-4 ml ist das Volumen der Quadrate auf dem Hämozytometer. Die vier Quadranten, die gezählt werden sollen, sind in Abbildung 1 grün dargestellt. Gesamtzahl der in der Formel gezählten Zellen ist die Summe der Anzahl der Zellen in den Quadranten 1, 2, 3 und 4. Die in Abbildung 1 blau dargestellten Zellen sollten gezählt werden, während rot dargestellte Zellen nicht gezählt werden sollten, da sie den oberen, rechten, unteren oder linken Rand des Quadranten berühren.
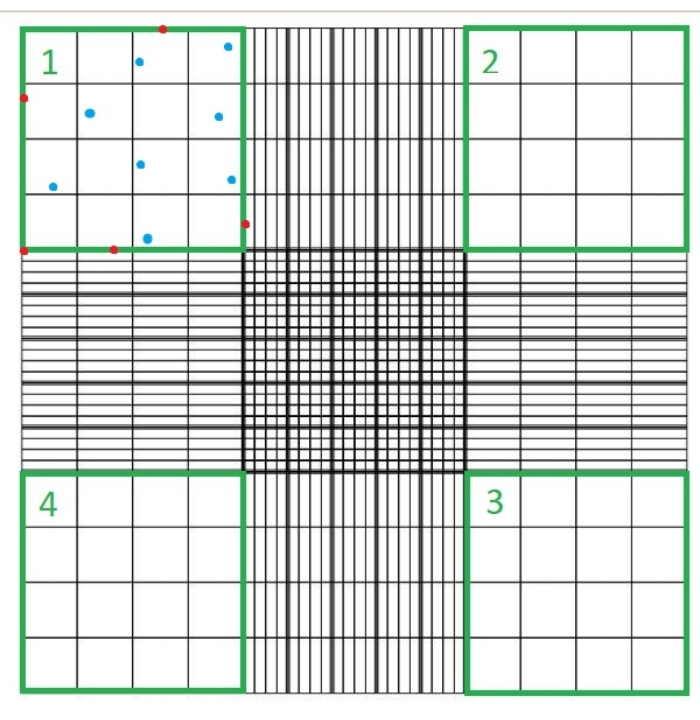
Abbildung 1: Zellzählung mit einer Hämozytometerkammer. Vier Quadranten werden mit einem Lichtmikroskop gezählt; Diese Quadranten sind grün dargestellt. Gesamtzahl der Zellen In der in Schritt 1.3.3 des hier beschriebenen Protokolls angegebenen Formel wird die Summe der Zellen in den Quadranten 1, 2, 3 und 4 gezählt. Blau markierte Zellen sollten gezählt werden, während rot markierte Zellen nicht gezählt werden sollten, da sie den oberen, rechten, unteren oder linken Rand des Quadranten berühren Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Den Teller für die Kultur vorbereiten
- Geben Sie ein Volumen der Zellsuspension A in ein 15-ml-konisches Röhrchen. Das benötigte Volumen ist abhängig von der Zellzahl. Stellen Sie das Volumen so ein, dass die Anzahl der Zellen ungefähr 2,2 x 106 Zellen für eine 100 mm Kulturplatte beträgt.
- 5 mL des kompletten Kulturmediums in das Röhrchen geben und dann den Inhalt des Röhrchens auf eine 100 mm Kulturplatte überführen. 80 μL Dimethylsulfoxid (DMSO) in die Kulturplatte geben.
HINWEIS: DMSO ist lichtempfindlich und sollte daher in einem lichtbeständigen Behälter aufbewahrt oder mit einem undurchsichtigen Material wie Aluminiumfolie bedeckt werden. - 350 μL 1 mg/ml Phorbol-12-Myristat-13-acetat (PMA) in die Tube geben. Die Endkonzentration von PMA sollte 35 ng/ml und die von DMSO 0,08% betragen.
ACHTUNG: PMA ist giftig, ätzend und krebserregend und sollte daher mit äußerster Vorsicht behandelt werden. PMA ist lichtempfindlich und sollte daher in einem lichtbeständigen Behälter gelagert oder mit einem undurchsichtigen Material wie Aluminiumfolie bedeckt werden. - Fügen Sie 4,27 ml Kulturmedium hinzu, so dass das Gesamtvolumen 10 ml beträgt. Mischen Sie den Inhalt des Röhrchens durch Kippen und geben Sie den Inhalt dann auf eine 100 mm Kulturplatte. Lassen Sie die Platte 5 Tage in einem Zellkulturinkubator bei 37 °C, 5% CO2.
3. Induktion und Isolierung von Epstein-Barr-Viruspartikeln
- Nach 5 Tagen im Inkubator zentrifugieren Sie den Inhalt der Platte bei 120 x g für 8 min, um die Zellen zu pelletieren. Sammeln Sie den zellfreien, virushaltigen Überstand und entsorgen Sie das Zellpellet.
- Zentrifugieren Sie den Überstand bei 16.000 x g für 90 min bei 4 °C, um die Viruspartikel zu pelletieren. Verwerfen Sie den Überstand und resuspendieren Sie das Viruspellet in 5 ml Kulturmedium.
HINWEIS: Phosphatgepufferte Kochsalzlösung (PBS) kann verwendet werden, um das Viruspellet anstelle des Kulturmediums zu resuspendieren. - Aliquot erhält man die virale Suspension in 20 Röhrchen, die jeweils 250 μL der Virussuspension enthalten. Lagern Sie die Suspension bei -80 °C.
4. DNA-Extraktion aus Viruspartikeln
VORSICHT: Beim Umgang mit Phenol ist äußerste Vorsicht geboten, da es giftig und ätzend ist und schwere Verbrennungen verursachen kann. Phenol ist lichtempfindlich und oxidiert bei Kontakt mit Licht oder Luft. Lagern Sie es in einem lichtbeständigen Behälter oder bedecken Sie den Phenolschlauch alternativ mit einem undurchsichtigen Material wie Aluminiumfolie.
- Trennung von Proteinen aus der DNA
- Fügen Sie 500 μL TrisCl-gesättigtes Phenol in eines der 250 μL-Röhrchen hinzu. Fügen Sie 100 μL Wasser hinzu (um das Volumen der wässrigen Phase zu erhöhen) und wirbeln Sie gut, um eine rosa Emulsion zu erhalten. 15 min bei 9650 x g zentrifugieren.
- DNA-Fällung
- Sammeln Sie den Überstand (transparente, wässrige Phase) und geben Sie ihn in ein neues 1,5-ml-Mikrozentrifugenröhrchen.
- Fügen Sie ein Äquivalent von 1/10 des Volumens des Überstands an kaltem Natriumacetat (3 M, pH 5,2) hinzu und mischen Sie es durch Pipettieren auf und ab. Fügen Sie 1 μL 20 mg / ml Glykogen hinzu und mischen Sie durch Pipettieren.
HINWEIS: Dieser Schritt ist optional, aber die Zugabe von Glykogen verbessert die DNA-Fällung und erleichtert die Visualisierung des DNA-Pellets. - Fügen Sie das dreifache Volumen des Überstands an kaltem 100% igem Ethanol hinzu. Über Nacht bei -80 °C lagern.
HINWEIS: Der Vorgang kann hier beendet werden, um zu einem späteren Zeitpunkt fortgesetzt zu werden. Die Probe kann bei -80 °C für 1 h oder alternativ über Nacht gelagert werden. Die Lagerung über Nacht erhöht die DNA-Fällung und wird daher empfohlen.
- Isolierung der viralen DNA
- Am nächsten Tag bei 9650 x g für 15 min bei 4 °C zentrifugieren und den Überstand verwerfen, um ein DNA-Pellet zu erhalten.
- Waschen Sie das Pellet dreimal mit 1 ml kaltem 70% Ethanol, zentrifugieren Sie es bei 9650 x g für 15 min und entsorgen Sie den Überstand.
- Trocknen Sie das Pellet ca. 10 min an der Luft. Resuspendieren Sie das Pellet in 10-50 μL nukleasefreiem destilliertem Wasser (abhängig von der Größe des DNA-Pellets).
- Lagern Sie die Proben bei -20 °C für die spätere Verarbeitung oder bei 4 °C über Nacht, um eine maximale DNA-Auflösung zu gewährleisten, gefolgt von einer Lagerung bei -20 °C. Alternativ können Sie auch direkt mit Schritt 5 fortfahren.
5. Überprüfung der DNA-Konzentration und -Reinheit
- Nach dem Reinigen des Sockels eines Mikrospektralphotometers mit einem empfindlichen Aufgabenwischer 1 μL der DNA-Präparation einladen.
- Beachten Sie die Konzentration der DNA in der Probe. Überprüfen Sie die Absorptionsverhältnisse sowohl bei als
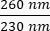 auch
auch 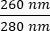 . DNA absorbiert bei 260 nm, Proteine bei 280 nm und organische Substanzen wie Phenol bei 230 nm. Ein Verhältnis von 1,8-2 wird für die real-time PCR als ausreichend angesehen.
. DNA absorbiert bei 260 nm, Proteine bei 280 nm und organische Substanzen wie Phenol bei 230 nm. Ein Verhältnis von 1,8-2 wird für die real-time PCR als ausreichend angesehen.
6. Quantifizierung durch Echtzeit-Polymerase-Kettenreaktion
- Herstellung der PCR-Reaktionsmischungen
- Geben Sie 5 μL einer grünen SYBR real-time PCR-Mischung in 0,2 ml PCR-Röhrchen. Fügen Sie jedem Röhrchen 1 μL 7,5 pmol/μL Vorwärtsprimer und 1 μL 7,5 pmol/μL Reverse-Primer hinzu. Vorwärts-Primer-Sequenz: 5'-CCCTAGTGGTTTCGGACACA-3'; umgekehrte Primersequenz: 5'-ACTTGCAAATGCTCTAGGCG-3'. Das Gen, das zur Bestimmung der EBV-Genomkopienzahl amplifiziert wurde, ist das Epstein-Barr-Virus Small RNA 2 (EBER-2) - Gen.
- In eines der Röhrchen werden 2 μL nukleasefreies Wasser und 1 μL virale DNA aus Schritt 4 gegeben. Das Gesamtvolumen des Reaktionsgemisches beträgt 10 μL.
HINWEIS: Abhängig von der Konzentration der DNA in der Probe kann ein Verdünnungsschritt erforderlich sein. Der Verdünnungsfaktor F ist zu beachten und wird in Schritt 6.3 in die Formel integriert. - Bereiten Sie andere Röhrchen vor, die als Standards mit bekannten EBV-Genomkopiennummern (1000, 2000, 5000, 10.000 und 54.000 Kopien) verwendet werden.
- Führen Sie die PCR-Gemische beginnend mit einem ersten Aktivierungsschritt bei 95 °C für 5 min, dann 40 Zyklen bei 95 °C und 58 °C (Glühen) für 15 s bzw. 30 s durch.
- Generieren Sie eine qPCR-Standardkurve, indem Sie die Ct-Werte der Standards gegen den Logarithmus der Anzahl der EBV-Genomkopien pro Standardröhrchen aufzeichnen. Unter Verwendung der Standardkurvendiagrammgleichung wird die Anzahl der EBV-Genomkopien in der PCR-Röhre abgeleitet, die die induzierte virale DNA enthält. Verwenden Sie dann die folgende Formel, um die Konzentration der induzierten viralen Zubereitung zu berechnen:

Dabei ist X die Anzahl der EBV-Genomkopien, die von der Standardkurve abgeleitet werden, und F der Verdünnungsfaktor, der zum Aufbau der pro PCR-Reaktion verwendeten DNA verwendet wird.
7. Überprüfung auf biologische Aktivität/Infektiosität der Viruspartikel
- Übertragen Sie BC-3-Zellen von einer 100-mm-Kulturplatte bei 80% Konfluenz in ein 15-ml-konisches Röhrchen. Die Seeding-Dichte von P3HR-1-Zellen beträgt 1 x 106 Zellen/ml. In vollständigem RPMI-Kulturmedium (79 % RPMI-Kulturmedium, 20 % Fetales Rinderserum, 1 % Pen-Streptokokken-Antibiotikum) bei 37 °C und 5 %CO2 aufbewahren und alle 3-4 Tage durchgehen.
- Zählen Sie die BC-3-Zellen auf die gleiche Weise, wie die P3HR-1-Zellen mit Schritt 1 gezählt wurden. Fügen Sie zu einer 96-Well-Platte 105 BC-3-Zellen zu jeder Vertiefung hinzu. Verwenden Sie drei, sechs, neun oder 12 Wells, um die Signifikanz der Ergebnisse sicherzustellen und Fehler zu minimieren.
- In jede Vertiefung wird ein Volumen Virusmaterial gegeben, abhängig von der Konzentration der in Schritt 6.3 bestimmten Viruszubereitung. Das MOI kann variieren, und die Optimierung dieses Protokolls zeigt das beste MOI für Infektionen (ein MOI-Bereich von 2-50 wird empfohlen). Stellen Sie sicher, dass das Gesamtvolumen der in jeder Vertiefung vorhandenen Mischung 250 μL beträgt.
- Um das erforderliche Volumen zu bestimmen, sollte die Konzentration des Virusvorrats bekannt sein und ein MOI ausgewählt werden. Wenn der MOI beispielsweise 2 ist, sollte die Anzahl der hinzugefügten Viruspartikel doppelt so hoch sein wie die Anzahl der Zellen. Berechnen Sie das Volumen V des benötigten Virusvorrats nach folgender Formel: , wobei n die Anzahl der benötigten Viruspartikel und C die bekannte Konzentration des Virusvorrats ist.
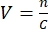
- Um das erforderliche Volumen zu bestimmen, sollte die Konzentration des Virusvorrats bekannt sein und ein MOI ausgewählt werden. Wenn der MOI beispielsweise 2 ist, sollte die Anzahl der hinzugefügten Viruspartikel doppelt so hoch sein wie die Anzahl der Zellen. Berechnen Sie das Volumen V des benötigten Virusvorrats nach folgender Formel: , wobei n die Anzahl der benötigten Viruspartikel und C die bekannte Konzentration des Virusvorrats ist.
- Inkubieren Sie den Inhalt der 96-Well-Platte für 5 Tage. Nach 5 Tagen wird der Inhalt jeder Vertiefung in ein 1,5-ml-Röhrchen überführt.
- Zentrifugieren Sie die Röhrchen bei 120 x g für 8 min, um die Zellen vom virushaltigen Kulturmedium zu trennen. Unterziehen Sie die Zellpellets sowie die Überstände einer DNA-Extraktion, gefolgt von einer Echtzeit-PCR-Quantifizierung, um das Vorhandensein viraler Genome in jeder Fraktion zu überprüfen und so die Infektiosität der Viruspartikel zu beurteilen.
Ergebnisse
Ziel dieses Verfahrens ist es, EBV-Partikel in einer Suspension mit bekanntem Virustiter zu isolieren, die anschließend zur Modellierung der EBV-Infektion verwendet werden könnten. Daher ist es von größter Bedeutung, optimale Konzentrationen der verschiedenen Reagenzien zu verwenden, um die höchste EBV-Ausbeute aus dem Verfahren zu erzielen.
Ein Optimierungsversuch wurde durchgeführt, um die Konzentrationen von PMA und DMSO zu bestimmen, die die höchste Anzahl von EBV-Partikeln ergeben ...
Diskussion
Die Produktion von EBV-Partikeln ist notwendig, um die Biologie dieses Virus sowie die damit verbundenen Krankheiten zu verstehen. Hier haben wir die Herstellung dieser Partikel aus der P3HR-1-Zelllinie beschrieben. Diese Zelllinie ist nicht die einzige EBV-Produktionslinie; Tatsächlich wurden EBV-Partikel auch aus B95-8-Zellen21,22 sowie der Raji-Zelllinie18,19 isoliert. Der EBV-lytische Zyklus wurde in...
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Die Finanzierung dieser Arbeit wurde durch Zuschüsse des Asmar Research Fund, des Lebanese National Council for Scientific Research (L-CNRS) und des Medical Practice Plan (MPP) an der American University of Beirut unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2 mL thin-walled PCR tubes | Thermo Scientific | AB0620 | Should be autoclaved before use |
| 0.2-10 µL Microvolume Filter Tips | Corning | 4807 | Should be autoclaved before use |
| 0.5-10 µL Pipette | BrandTech | 704770 | |
| 10 mL Disposable Serological Pipette | Corning | 4488 | |
| 1000 µL Filtered Pipette Tips | QSP | TF-112-1000-Q | |
| 100-1000 µL Pipette | Eppendorf | 3123000063 | |
| 100x20 mm Cuture Plates | Sarstedt | 83.1802 | |
| 10-100 µL Pipette | BrandTech | 704774 | |
| 15 mL Conical Tubes | Corning | 430791 | |
| 200 µL Filtered Pipette Tips | QSP | TF-108-200-Q | |
| 20-200 µL Pipette | Eppendorf | 3123000055 | |
| 50 mL Conical Tubes | Corning | 430828 | |
| CFX96 Real-Time C-1000 Thermal Cycler | Bio-Rad | 184-1000 | |
| DMSO | Amresco | 0231 | |
| DNase/RNase Free Water | Zymo Research | W1001-1 | |
| EBER Primers | Macrogen | N/A | Custom Made Primers |
| EBV DNA Control (Standards) | Vircell | MBC065 | |
| Ethanol (Laboratory Reagent Grade) | Fischer Chemical | E/0600DF/17 | |
| Fetal Bovine Serum | Sigma | F9665 | |
| Fresco 21 MicroCentrifuge | Thermo Scientific | 10651805 | |
| Glycogen Solution | Qiagen | 158930 | |
| Hemocytometer | BOECO | BOE 01 | |
| Inverted Light Microscope | Zeiss | Axiovert 25 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5121 | |
| Microcentrifuge Tube | Costar (Corning) | 3621 | Should be autoclaved before use |
| P3HR-1 Cell Line | ATCC | HTB-62 | |
| Penicillin-Streptomycin Solution | Biowest | L0022 | |
| Phenol | VWR | 20599.297 | |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma-Aldrich | P8139 | |
| Pipette Filler | Thermo Scientific | 9501 | |
| Precision Wipes | Kimtech | 7552 | |
| RPMI-1640 Culture Medium | Sigma | R7388 | |
| SL 16R Centrifuge | Thermo Scientific | 75004030 | |
| Sodium Acetate | Riedel-de Haën (Honeywell) | 25022 | |
| Spectrophotomer | DeNovix | DS-11 | |
| Tris-HCl | Sigma | T-3253 | |
| Trypan Blue Solution | Sigma | T8154 | |
| Water Jacketed CO2 Incubator | Thermo Scientific | 4121 |
Referenzen
- Epstein, M. A., Achong, B. G., Barr, Y. M. Virus particles in cultured lymphoblasts from Burkitt's lymphoma. Lancet. 1 (7335), 702-703 (1964).
- Sample, J., et al. Epstein-Barr virus types 1 and 2 differ in their EBNA-3A, EBNA-3B, and EBNA-3C genes. Journal of Virology. 64 (9), 4084-4092 (1990).
- Chang, M. S., Kim, W. H. Epstein-Barr virus in human malignancy: a special reference to Epstein-Barr virus associated gastric carcinoma. Cancer Research and Treatment. 37 (5), 257-267 (2005).
- Manet, E., Schwab, M. . Encyclopedia of Cancer. , 1602-1607 (2017).
- Babcock, G. J., Decker, L. L., Volk, M., Thorley-Lawson, D. A. EBV persistence in memory B cells in vivo. Immunity. 9 (3), 395-404 (1998).
- Khan, G., Miyashita, E. M., Yang, B., Babcock, G. J., Thorley-Lawson, D. A. Is EBV persistence in vivo a model for B cell homeostasis. Immunity. 5 (2), 173-179 (1996).
- Jha, H. C., Banerjee, S., Robertson, E. S. The role of gammaherpesviruses in cancer pathogenesis. Pathogens. 5 (1), 18 (2016).
- El-Sharkawy, A., Al Zaidan, L., Malki, A. Epstein-Barr virus-associated malignancies: roles of viral oncoproteins in carcinogenesis. Frontiers in Oncology. 8, 265 (2018).
- Vereide, D., Sugden, B. Insights into the evolution of lymphomas induced by Epstein-Barr virus. Advances in Cancer Research. 108, 1-19 (2010).
- Vereide, D. T., Sugden, B. Lymphomas differ in their dependence on Epstein-Barr virus. Blood. 117 (6), 1977-1985 (2011).
- Houen, G., Trier, N. H. Epstein-Barr virus and systemic autoimmune diseases. Frontiers in Immunology. 11, 587380 (2020).
- Ortiz, A. N., et al. Impact of Epstein-Barr virus infection on inflammatory bowel disease (IBD) clinical outcomes. Revista Espanola de Enfermedades Digestivas. 114 (5), 259-265 (2021).
- Caplazi, P., et al. Mouse models of rheumatoid arthritis. Veterinary Pathology. 52 (5), 819-826 (2015).
- Kiesler, P., Fuss, I. J., Strober, W. Experimental models of inflammatory bowel diseases. Cellular and Molecular Gastroenterology and Hepatology. 1 (2), 154-170 (2015).
- Warde, N. Experimental arthritis: EBV induces arthritis in mice. Nature Reviews Rheumatology. 7 (12), 683 (2011).
- Jog, N. R., James, J. A. Epstein Barr virus and autoimmune responses in systemic lupus erythematosus. Frontiers in Immunology. 11, 623944 (2020).
- Shimizu, N., Yoshiyama, H., Takada, K. Clonal propagation of Epstein-Barr virus (EBV) recombinants in EBV-negative Akata cells. Journal of Virology. 70 (10), 7260-7263 (1996).
- Hsu, C. H., et al. Induction of Epstein-Barr virus (EBV) reactivation in Raji cells by doxorubicin and cisplatin. Anticancer Research. 22, 4065-4071 (2002).
- Nutter, L. M., Grill, S. P., Li, J. S., Tan, R. S., Cheng, Y. C. Induction of virus enzymes by phorbol esters and n-butyrate in Epstein-Barr virus genome-carrying Raji cells. Cancer Research. 47 (16), 4407-4412 (1987).
- Fresen, K. O., Cho, M. S., zur Hausen, H. Recovery of transforming EBV from non-producer cells after superinfection with non-transforming P3HR-1 EBV. International Journal of Cancer. 22 (4), 378-383 (1978).
- Glaser, R., Tarr, K. L., Dangel, A. W. The transforming prototype of Epstein-Barr virus (B95-8) is also a lytic virus. International Journal of Cancer. 44 (1), 95-100 (1989).
- Sairenji, T., et al. Inhibition of Epstein-Barr virus (EBV) release from P3HR-1 and B95-8 cell lines by monoclonal antibodies to EBV membrane antigen gp350/220. Journal of Virology. 62 (8), 2614-2621 (1988).
- Savage, A., et al. An assessment of the population of cotton-top tamarins (Saguinus oedipus) and their habitat in Colombia. PLoS one. 11 (12), 0168324 (2016).
- Kallin, B., Klein, G. Epstein-Barr virus carried by raji cells: a mutant in early functions. Intervirology. 19 (1), 47-51 (1983).
- Fresen, K. O., Cho, M. S., Hausen, H. Z. Recovery of transforming EBV from non-producer cells after superinfection with non-transforming P3HR-1 EBV. International Journal of Cancer. 22 (4), 378-383 (1978).
- Bounaadja, L., Piret, J., Goyette, N., Boivin, G. Evaluation of Epstein-Barr virus, human herpesvirus 6 (HHV-6), and HHV-8 antiviral drug susceptibilities by use of real-time-PCR-based assays. Journal of Clinical Microbiology. 51 (4), 1244-1246 (2013).
- Buelow, D., et al. Comparative evaluation of four real-time PCR methods for the quantitative detection of Epstein-Barr virus from whole blood specimens. Journal of Molecular Diagnostics. 18 (4), 527-534 (2016).
- Wu, D. Y., Kalpana, G. V., Goff, S. P., Schubach, W. H. Epstein-Barr virus nuclear protein 2 (EBNA2) binds to a component of the human SNF-SWI complex, hSNF5/Ini1. Journal of Virology. 70 (9), 6020-6028 (1996).
- Li, C., et al. EBNA2-deleted Epstein-Barr virus (EBV) isolate, P3HR1, causes Hodgkin-like lymphomas and diffuse large B cell lymphomas with type II and Wp-restricted latency types in humanized mice. PLoS Pathogens. 16 (6), 1008590 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten